
Zestawienia sekwencji homologicznych
(dopasowanych)
Multiple Sequence Alignment
Zestawienia sekwencji homologicznych
(dopasowanych)
Multiple Sequence Alignment
H U M A N - - M A S V S E A C I Y S A
H D D E T T E D K N A
K A A G N - E P F W P G F A K A A N V N G S
C N V G A G G P A P A A G A A P A G G P A P S T A A A P A - - E E K K V E A K K E E S E E
R A T - - M A S V S E A C I Y S A
H D D E T T E D K N A
K A A G N - E P F W P G F A K A A N V N G S
C N V G A G G P A P A A G A A P A G G P A P S A A A A P A - - E E K K V E A K K E E S E E
D I C D I M S E I K T E E A C I Y S G
Q D D G E T A D K K T
E A A N T - A S H W P G Y A R S A K V N P E
L N A G S S G - - - - - A A G A A P V A A A T S A A A P A A A A K K E T K K E E V K K E E
D R O M E - - M S T K A E A C V Y A S
V D D D A T G E K N T
K A A N E - E P Y W P G F A K A E G I N K D
T N I G S G V G - - - - A A P A G G A A P A A A A A A P A A E S K K E E K K K E E E S D Q
A R A T H - - M S T V G E A C S Y A V M
E D E G A T I A T V K
G V E I E S Y W P M L F A K M A E K R N V T D L M N G A - - - G G G G G G A P V S - - - A A A P A A A G G A A A A A P A - K E E K K D E P A E E
G
Y E A S T - A - - M S T E S A
S Y A A
A D S E E S S E K L T
N A A N P - D E N I W A D F A K A D G Q N K D
V N F S A G A A - - - - A P A G V A G G V A G G - - - - E A G E A E A E K E E E E A K E E
Y E A S T - B - - - M S D S I
S F A A
A D A G E T S D N L T
K A A G N - D N V W A D Y A K A E G K D K E
S G F H N A G P - - - - V A G A G A A S G A A A A G G D A A A - - - E E E K E E E A A E E
C L A H E - - - M S A A E A S S Y A A
A D E G E T A D K Q A
S A A K P E E P I W T S F A K A E G K D K D
L N V G S G G G - - - - A A P A A G G A A A G G A A A V L D A P A - - E E K A E E E K E E
T R Y C R - - M S S K Q Q A C T Y A A
A D S G K T D - M D S L K
K A A G D - S K G M A S F A S I K N V D N D
S K V S F G G - - - - - V A P A A G G A T A A P A A A A A A A A P A A A A A K - K E E E E E
L E I D O - - - M S T K Y - A A Y A L S S K A S P S Q - - A D E A
K A V H D - D Q A T L A V M E S T G R D A T
A E G A A K M S A M P A A S S G A A A G V T A S A A G D A A P A A A A A K K D - E P E E E A
- -
- -
L
L I L
V V
I
L I
V
V
L
L
I
L I
L
L I L
V V
I
L I
V
V
L
L
I
L I
L
L L L
I I
I
L L
I
V
L
L
I
L L
L
L I L
V V
I
I L
V
V
L
L
V
L I
L
I L
I I
L
A A
I
V
L
L I L
I I
L
L T
V
I
L
L
L L
I
F I L
L I
L
I T
A
V
V
L
L
I L
L
L I L
L I
L
L I
V
I
L
L
V
L L
L
L I L
L
V T
V
V
A
L
I
V L
L
A L
V
I C
I
V
F
V
V
L I
S D D D M G F G L F D
S E D D M G F G L F D
S D D D M G M G L F D
S D D D M G F G L F D
S D D L G F G L F D
S D D D M G F G L F D
S D D D M G F G L F D
S D D D M G F G L F D
D D D M G F G L F D
D D D M G F G L F D
H U M A N M R Y A S Y
A G G N S S P S A K D K K
D S V G E A D D D R N K
S E N G K - N E D
A Q G I G K A S V P A - G G A V A V S A A P G S A A P A A G S A P A A A - - E E K K D E K K E E S E E
R A T M R Y A S Y
A G G N S N P S A K D K K
D S V G E A D D E R N K
S E N G K - N E D
A Q G V G K A S V P A - G G A V A V S A A P G S A A P A A G S A P A A A - - E E K K D E K K E E S E E
E E
M
D R O M E M R Y A A Y
V G G K D S P A N S D E K
S S V G E V D A E R T K
K E A G K - S D D
K E G R E K S S M P V - G G G G A V A A A D A A P A - - - - - - - - - - K K E A K K E E K K E E S E S E
A R A T H M K V A A Y
V S G K A S P T S A D K T
G S V G E T E D S Q E L
K E K G K D L E L
A G R E K L S V P S G G G G G V A V A S A T S G G G G G G G A S A A - - - - E S K K E E K K E E K E E
Y E A S T - A M K Y A A Y
N A G - N T P D A T K K A
E S V G E I E D E K S S
S A E G K - S D E
T E G N E K
A A V P A - A G P A S A G G A A A A S G D A A A - - - - - - E E E K E E E A A E E
Y E A S T - B M K Y A A Y
V Q G G N A A P S A A D K A
E S V G E V D E A R N E
S S E G K G S E E
A E G Q K K A T V P T - G G A S S A A A G A A G A A A G G D A A - - - - - - - - E E E K E E E A K E E
C L A H E M K Y A A F
G A G N S S P S A E D K T
S S V G D A D E E R S S
K E E G K - D N E
S S G S E K A S V P S - G G A G A A S A G G A A A A G G A A E A A - - - - - - P E A E R A E E E K E E
T R Y C R
E
L E I D O
A
V
L L A L
I
I L
I
L
V I
L
I
V I
L
V
L L A L
I
I L
I
L
V I
L
I
V I
L
L
L L A L
I L
V
V
V
L
V
L I
V
V
L L A L
L
I L
V
L
V I
L
I
L I
L
V
L L A L
I
I L
A
I
L L
V
A
I A
A
L
L L L A
I
I L
I
V
V L
L
V
L I
L
L
L L L
I
V V
A
I
L L
L
L
I I
F
L
L L L L
I
V L
I
L
L L
L
I
L I
L
L
A L V L
V
V L
V
V
L F
F
F
V
L
L
A L V L
V
V L
V
V
V F
L
F
L V
L
S D D D M G F G L F D
S D D D M G F G L F D
S D D D M G G L F D
D D D M G F A L F E
S D D D M G F S L F E
S D D D M G F G L F D
S D D D M G F G L F D
S D D D M G F G L F D
D D D M G F G L F D
D D D M G F G L F D
D I C D I M K Y A A Y
S S G N A N A A S V - - T K
Q S V G E V D A A R E S C K E D G K - D Q A
A A G K S K G S V - A A A A A P A A A T S A A P A A A A A A P A - - - - - - - - - K K V V E E K K
M K Y A A Y
G S G G T - P S K S A E A
K A A G P V D P S R D A
A E A G K - D D T C T E G K S K V G G V T R P N A A T A S A P T A A A A A S S G A A A P A A A A - - - - - - - - - - E E E
M Q Y A A Y
A S G K T - P S K A A E A
K A A G A V D A S R D A
Q E E G K - S D A
A E G R A K V G S G S A A P A A A A S T A A A A A A V V A - - - - - - - - - - - - E A K K E E P E E E
- - - -
H U M A N - - M A S V S E A C I Y S A
H D D E T T E D K N A
K A A G N - E P F W P G F A K A A N V N G S
C N V G A G G P A P A A G A A P A G G P A P S T A A A P A - - E E K K V E A K K E E S E E
R A T - - M A S V S E A C I Y S A
H D D E T T E D K N A
K A A G N - E P F W P G F A K A A N V N G S
C N V G A G G P A P A A G A A P A G G P A P S A A A A P A - - E E K K V E A K K E E S E E
D I C D I M S E I K T E E A C I Y S G
Q D D G E T A D K K T
E A A N T - A S H W P G Y A R S A K V N P E
L N A G S S G - - - - - A A G A A P V A A A T S A A A P A A A A K K E T K K E E V K K E E
D R O M E - - M S T K A E A C V Y A S
V D D D A T G E K N T
K A A N E - E P Y W P G F A K A E G I N K D
T N I G S G V G - - - - A A P A G G A A P A A A A A A P A A E S K K E E K K K E E E S D Q
A R A T H - - M S T V G E A C S Y A V M
E D E G A T I A T V K
G V E I E S Y W P M L F A K M A E K R N V T D L M N G A - - - G G G G G G A P V S - - - A A A P A A A G G A A A A A P A - K E E K K D E P A E E
G
Y E A S T - A - - M S T E S A
S Y A A
A D S E E S S E K L T
N A A N P - D E N I W A D F A K A D G Q N K D
V N F S A G A A - - - - A P A G V A G G V A G G - - - - E A G E A E A E K E E E E A K E E
Y E A S T - B - - - M S D S I
S F A A
A D A G E T S D N L T
K A A G N - D N V W A D Y A K A E G K D K E
S G F H N A G P - - - - V A G A G A A S G A A A A G G D A A A - - - E E E K E E E A A E E
C L A H E - - - M S A A E A S S Y A A
A D E G E T A D K Q A
S A A K P E E P I W T S F A K A E G K D K D
L N V G S G G G - - - - A A P A A G G A A A G G A A A V L D A P A - - E E K A E E E K E E
T R Y C R - - M S S K Q Q A C T Y A A
A D S G K T D - M D S L K
K A A G D - S K G M A S F A S I K N V D N D
S K V S F G G - - - - - V A P A A G G A T A A P A A A A A A A A P A A A A A K - K E E E E E
L E I D O - - - M S T K Y - A A Y A L S S K A S P S Q - - A D E A
K A V H D - D Q A T L A V M E S T G R D A T
A E G A A K M S A M P A A S S G A A A G V T A S A A G D A A P A A A A A K K D - E P E E E A
- -
- -
L
L I L
V V
I
L I
V
V
L
L
I
L I
L
L I L
V V
I
L I
V
V
L
L
I
L I
L
L L L
I I
I
L L
I
V
L
L
I
L L
L
L I L
V V
I
I L
V
V
L
L
V
L I
L
I L
I I
L
A A
I
V
L
L I L
I I
L
L T
V
I
L
L
L L
I
F I L
L I
L
I T
A
V
V
L
L
I L
L
L I L
L I
L
L I
V
I
L
L
V
L L
L
L I L
L
V T
V
V
A
L
I
V L
L
A L
V
I C
I
V
F
V
V
L I
S D D D M G F G L F D
S E D D M G F G L F D
S D D D M G M G L F D
S D D D M G F G L F D
S D D L G F G L F D
S D D D M G F G L F D
S D D D M G F G L F D
S D D D M G F G L F D
D D D M G F G L F D
D D D M G F G L F D
H U M A N M R Y A S Y
A G G N S S P S A K D K K
D S V G E A D D D R N K
S E N G K - N E D
A Q G I G K A S V P A - G G A V A V S A A P G S A A P A A G S A P A A A - - E E K K D E K K E E S E E
R A T M R Y A S Y
A G G N S N P S A K D K K
D S V G E A D D E R N K
S E N G K - N E D
A Q G V G K A S V P A - G G A V A V S A A P G S A A P A A G S A P A A A - - E E K K D E K K E E S E E
E E
M
D R O M E M R Y A A Y
V G G K D S P A N S D E K
S S V G E V D A E R T K
K E A G K - S D D
K E G R E K S S M P V - G G G G A V A A A D A A P A - - - - - - - - - - K K E A K K E E K K E E S E S E
A R A T H M K V A A Y
V S G K A S P T S A D K T
G S V G E T E D S Q E L
K E K G K D L E L
A G R E K L S V P S G G G G G V A V A S A T S G G G G G G G A S A A - - - - E S K K E E K K E E K E E
Y E A S T - A M K Y A A Y
N A G - N T P D A T K K A
E S V G E I E D E K S S
S A E G K - S D E
T E G N E K
A A V P A - A G P A S A G G A A A A S G D A A A - - - - - - E E E K E E E A A E E
Y E A S T - B M K Y A A Y
V Q G G N A A P S A A D K A
E S V G E V D E A R N E
S S E G K G S E E
A E G Q K K A T V P T - G G A S S A A A G A A G A A A G G D A A - - - - - - - - E E E K E E E A K E E
C L A H E M K Y A A F
G A G N S S P S A E D K T
S S V G D A D E E R S S
K E E G K - D N E
S S G S E K A S V P S - G G A G A A S A G G A A A A G G A A E A A - - - - - - P E A E R A E E E K E E
T R Y C R
E
L E I D O
A
V
L L A L
I
I L
I
L
V I
L
I
V I
L
V
L L A L
I
I L
I
L
V I
L
I
V I
L
L
L L A L
I L
V
V
V
L
V
L I
V
V
L L A L
L
I L
V
L
V I
L
I
L I
L
V
L L A L
I
I L
A
I
L L
V
A
I A
A
L
L L L A
I
I L
I
V
V L
L
V
L I
L
L
L L L
I
V V
A
I
L L
L
L
I I
F
L
L L L L
I
V L
I
L
L L
L
I
L I
L
L
A L V L
V
V L
V
V
L F
F
F
V
L
L
A L V L
V
V L
V
V
V F
L
F
L V
L
S D D D M G F G L F D
S D D D M G F G L F D
S D D D M G G L F D
D D D M G F A L F E
S D D D M G F S L F E
S D D D M G F G L F D
S D D D M G F G L F D
S D D D M G F G L F D
D D D M G F G L F D
D D D M G F G L F D
D I C D I M K Y A A Y
S S G N A N A A S V - - T K
Q S V G E V D A A R E S C K E D G K - D Q A
A A G K S K G S V - A A A A A P A A A T S A A P A A A A A A P A - - - - - - - - - K K V V E E K K
M K Y A A Y
G S G G T - P S K S A E A
K A A G P V D P S R D A
A E A G K - D D T C T E G K S K V G G V T R P N A A T A S A P T A A A A A S S G A A A P A A A A - - - - - - - - - - E E E
M Q Y A A Y
A S G K T - P S K A A E A
K A A G A V D A S R D A
Q E E G K - S D A
A E G R A K V G S G S A A P A A A A S T A A A A A A V V A - - - - - - - - - - - - E A K K E E P E E E
- - - -

Zestawienia sekwencji
homologicznych
Zestawienia sekwencji
homologicznych
Zestawienie wielu sekwencji aminkwasowej wielu białek stanowi
rozszerzenie analizy w których stosowano takie programy jak
FASTA lub BLAST.
Zestawienie wielu sekwencji to dopasowanie więcej niż dwóch
sekwencji aminokwasowych. Czym więcej sekwencji jest użyte to
zestawienia, tym analiza jest bardziej precyzyjna i ujawnia
pozycje jednoznacznie konserwatywne.
Dzięki tej metodzie uzyskujemy dodatkową wiedzę na temat
białka które jest analizowane. Informacje jakie można uzyskać
to:
- pokrewieństwo ewolucyjne białek (ewolucja molekularna)
- określenie biologiczne ważnych fragmentów białka
odpowiedzialnych za właściwości biologiczne jak i
strukturalne.
Zestawienie wielu sekwencji aminkwasowej wielu białek stanowi
rozszerzenie analizy w których stosowano takie programy jak
FASTA lub BLAST.
Zestawienie wielu sekwencji to dopasowanie więcej niż dwóch
sekwencji aminokwasowych. Czym więcej sekwencji jest użyte to
zestawienia, tym analiza jest bardziej precyzyjna i ujawnia
pozycje jednoznacznie konserwatywne.
Dzięki tej metodzie uzyskujemy dodatkową wiedzę na temat
białka które jest analizowane. Informacje jakie można uzyskać
to:
- pokrewieństwo ewolucyjne białek (ewolucja molekularna)
- określenie biologiczne ważnych fragmentów białka
odpowiedzialnych za właściwości biologiczne jak i
strukturalne.

• Podstawą dla porównania białek z tej samej rodziny jest
fakt, że białka są powiązane ewolucyjnie, tzn. posiadają
wspólnego przodka. W procesie ewolucji, białka (geny)
akumulują mutacje, które z reguły mają charakter
przypadkowy i stanowią one podstawowy mechanizm
ewolucji (genów) białek. W rezultacie, zmiany w białkach
prowadzą do utworzenia się rodzin czyli grup białek o tych
samych aktywnościach biologicznych, występujących w
różnych organizmach. Białka te są często znacznie
zróżnicowane pod względem sekwencji aminokwasowej a
metoda zestawienia sekwencji homologicznych jest
jedyną możliwością zestawienia wszystkich białek w jedną
rodzinę i scharakteryzowanie ich pod względem
funkcjonalnym i strukturalnym.
• Zestawienia sekwencji homologicznych
stało
się obecnie jedna z podstawowych metod w analizie
sekwencji aminokwasowych w naukach biologicznych
.
• Podstawą dla porównania białek z tej samej rodziny jest
fakt, że białka są powiązane ewolucyjnie, tzn. posiadają
wspólnego przodka. W procesie ewolucji, białka (geny)
akumulują mutacje, które z reguły mają charakter
przypadkowy i stanowią one podstawowy mechanizm
ewolucji (genów) białek. W rezultacie, zmiany w białkach
prowadzą do utworzenia się rodzin czyli grup białek o tych
samych aktywnościach biologicznych, występujących w
różnych organizmach. Białka te są często znacznie
zróżnicowane pod względem sekwencji aminokwasowej a
metoda zestawienia sekwencji homologicznych jest
jedyną możliwością zestawienia wszystkich białek w jedną
rodzinę i scharakteryzowanie ich pod względem
funkcjonalnym i strukturalnym.
• Zestawienia sekwencji homologicznych
stało
się obecnie jedna z podstawowych metod w analizie
sekwencji aminokwasowych w naukach biologicznych
.

Jak zdefiniować białka
homologiczne?
Jak zdefiniować białka
homologiczne?
•Podstawowym kryterium w ocenie podobieństwa białek jest
analiza procentowa identycznych aminokwasów
znajdujących się w podobnej lokalizacji w analizowanym
polipeptydzie. Uważa się, jeśli podobieństwo wynosi
minimum 25% (na całej długości polipeptydu, np. 200
aminokwasów) to białka takie prawdopodobnie są
homologiczne.
•Podstawowymi narzędziami w takich analizach są
programy FASTA i BLAST służące do analizy tylko dwóch
białek jednocześnie.
•Do bardzo skomplikowanych analiz gdzie wysoka czułość i
specyficzność analizy jest wymagana służy PSI-BLAST.
•Podstawowym kryterium w ocenie podobieństwa białek jest
analiza procentowa identycznych aminokwasów
znajdujących się w podobnej lokalizacji w analizowanym
polipeptydzie. Uważa się, jeśli podobieństwo wynosi
minimum 25% (na całej długości polipeptydu, np. 200
aminokwasów) to białka takie prawdopodobnie są
homologiczne.
•Podstawowymi narzędziami w takich analizach są
programy FASTA i BLAST służące do analizy tylko dwóch
białek jednocześnie.
•Do bardzo skomplikowanych analiz gdzie wysoka czułość i
specyficzność analizy jest wymagana służy PSI-BLAST.
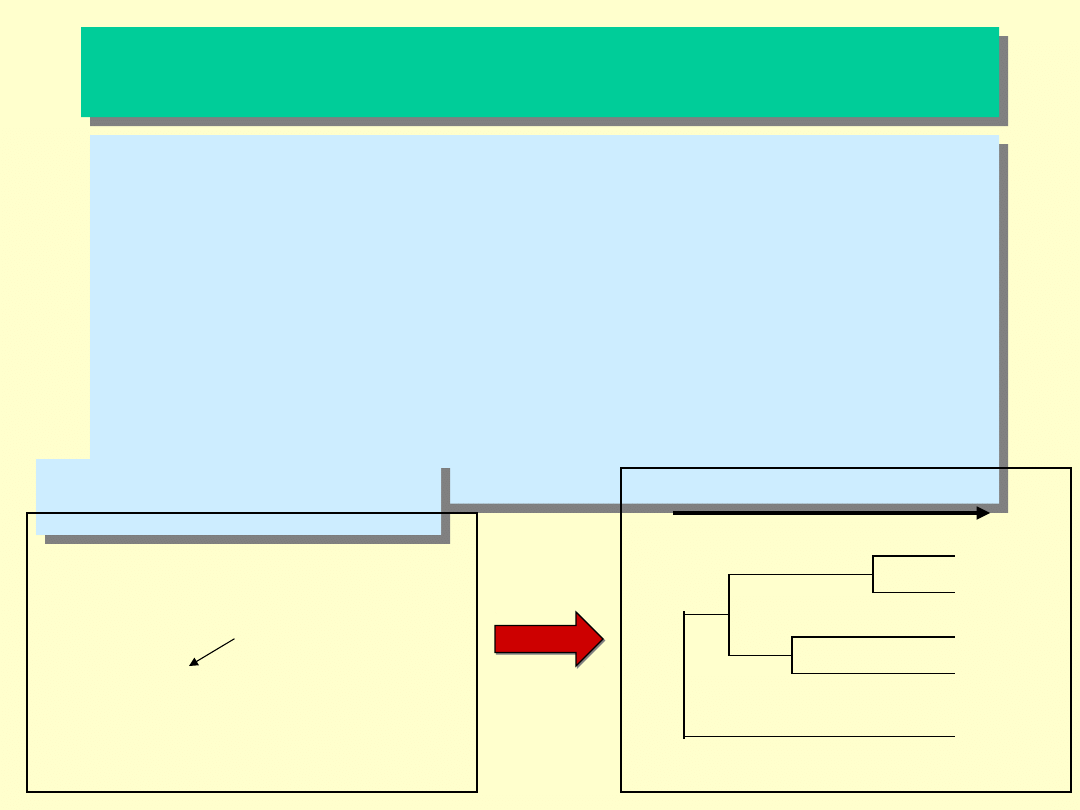
Narzędzia do zestawienia wielu sekwencji
homologicznych
Narzędzia do zestawienia wielu sekwencji
homologicznych
Metody hierarchiczne
-oparte są na znalezieniu tzw. drzewa przewodniego a następnie
dopasowanie sekwencji według tego drzewa.
Pierwszy etap to dopasowywanie sekwencji w parach,
każda z każdą. Tworzy się ranking podobnych par, i pary
podobne umieszcza się bliżej siebie niż sekwencje o mniejszym
podobieństwie. Zestawienie sekwencji dopasowanych powstaje
poprzez uszeregowanie najpierw najbardziej podobnych par,
następnie dostawienie kolejnych sekwencji o coraz mniejszym
stopniu podobieństwa.
Metody hierarchiczne
-oparte są na znalezieniu tzw. drzewa przewodniego a następnie
dopasowanie sekwencji według tego drzewa.
Pierwszy etap to dopasowywanie sekwencji w parach,
każda z każdą. Tworzy się ranking podobnych par, i pary
podobne umieszcza się bliżej siebie niż sekwencje o mniejszym
podobieństwie. Zestawienie sekwencji dopasowanych powstaje
poprzez uszeregowanie najpierw najbardziej podobnych par,
następnie dostawienie kolejnych sekwencji o coraz mniejszym
stopniu podobieństwa.
#1 #2
#5
#3 #4
#1
#2
#5
#3
#4
21
32
20
11
19
39
10
20
10
9
Oszacowanie jakości
dopasowania
Oszacowanie jakości
dopasowania
Wartość Z – (określa tzw.
test
istotności
dopasowania)
Hierarchiczne
zestawienie
sekwencji
#4
#2
#3
#1
#5
Zwiększające się podobieństwo
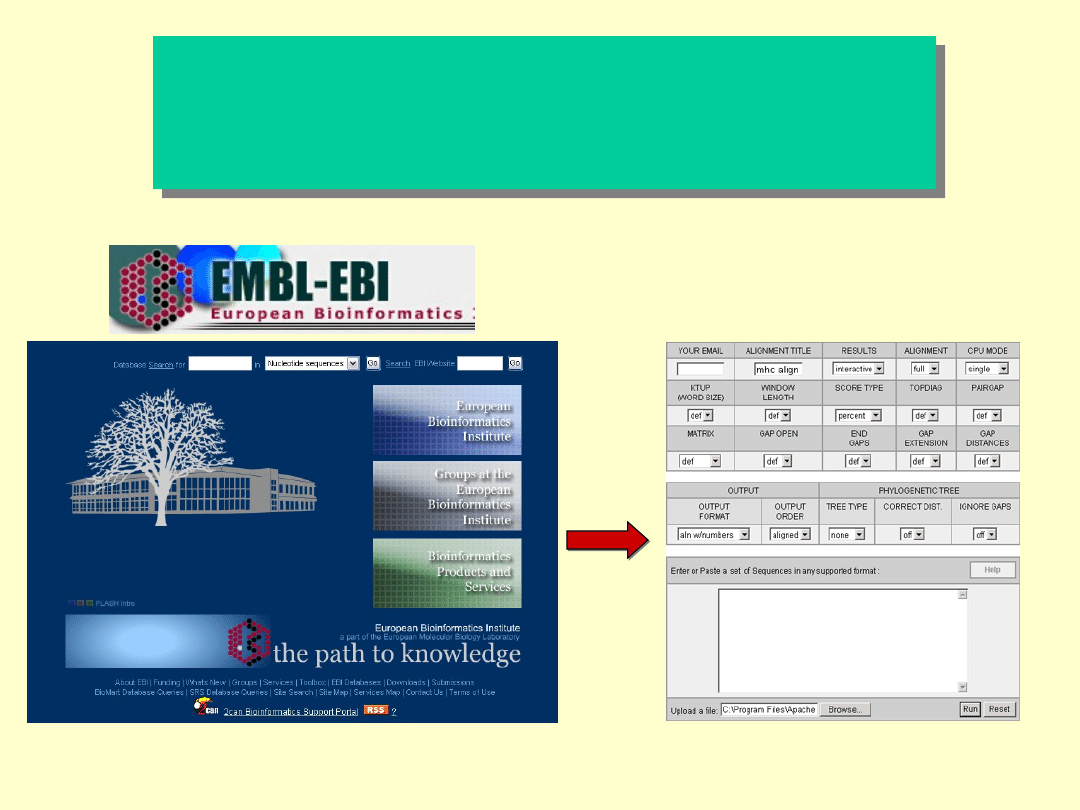
Clustal W jako uniwersalny program do
zestawień sekwencji aminokwasowych
oparty na metodzie hierarchicznej
Clustal W jako uniwersalny program do
zestawień sekwencji aminokwasowych
oparty na metodzie hierarchicznej
http://www.ebi.ac.uk/clustalw/
Clustal W
>RLA1_HUMAN
MASVSELACIYSALILHDDEVTVTEDKINALIKAAGVNVEPFWPGLFAKALANVNIG
SLI
CNVGAGGPAPAAGAAPAGGPAPSTAAAPAEEKKVEAKKEESEESDDDMGFGLFD
>RLA1_MAIZE
MASGELACRYAALILSDDGIAITAEKIATIVKAANIKVESYWPALFAKLLEKRNVEDLI
L
SVGSGGGAAPVAAAAPAGGAAAAAAPAVEEKKEEAKEESDDDMGFSLFD
>RLA1_MOUSE
MASVSELACIYSALILHDDEVTVTEDKINALIKAAGVSVEPFWPGLFAKALANVNIG
SLI
CNVGAGGPAPAAGAAPAGGAAPSTAAAPAEEKKVEAKKEESEESEDDMGFGLFD
>RLA1_HYDRO
MADSSTSELACVYSALILHDDAITAEKMNKIISAANVNVEPYWPGLFALEGKNIGD
LICN
VGSSGPAAGAPAAGAAGGAVEEKKEEKKAESEDESDDDMGLFD
>RLA1_RAT
MASVSELACIYSALILHDDEVTVTEDKINALIKAAGVNVEPFWPGLFAKALANVNIG
SLI
CNVGAGGPAPAAGAAPAGGPAPSAAAAPAEEKKVEAKKEESEESEDDMGFGLFD
>RLA1_DROME
MSTKAELACVYASLILVDDDVAVTGEKINTILKAANVEVEPYWPGLFAKALEGINVK
DLI
TNIGSGVGAAPAGGAAPAAAAAAPAAESKKEEKKKEEESDQSDDDMGFGLFD
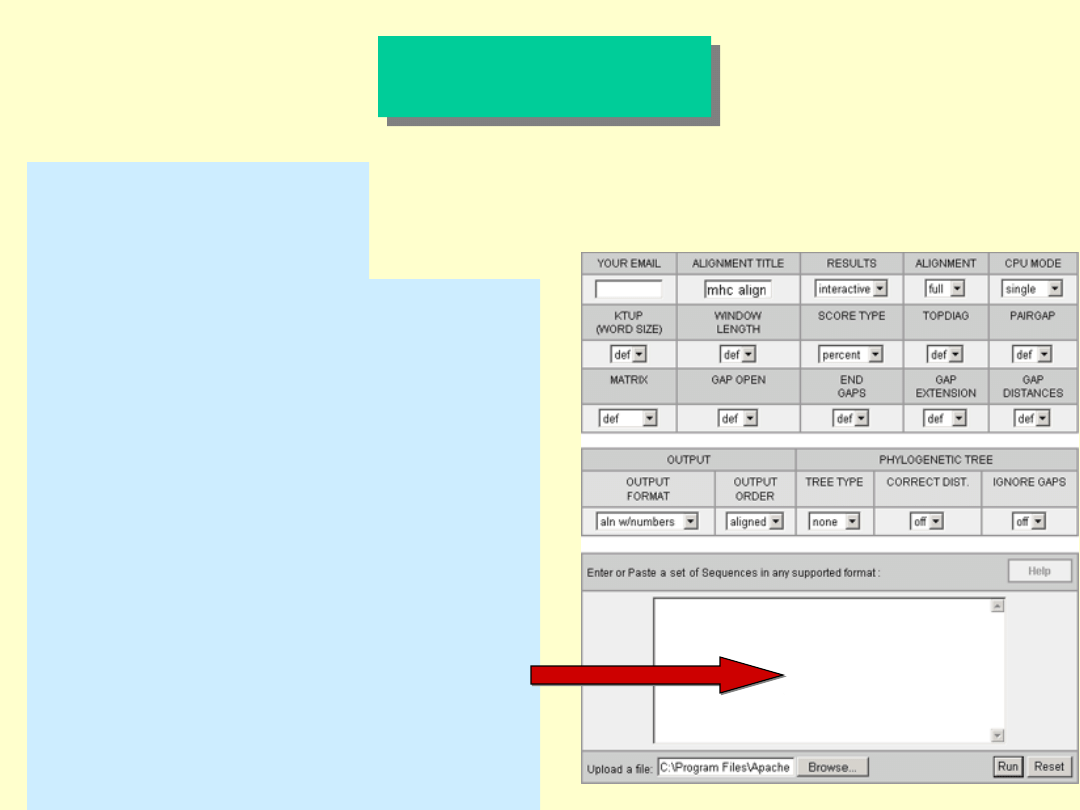
Dane do analizy
występują w jednym
pliku w formacie
FASTA.
Clustal W
Clustal W
Clustal W
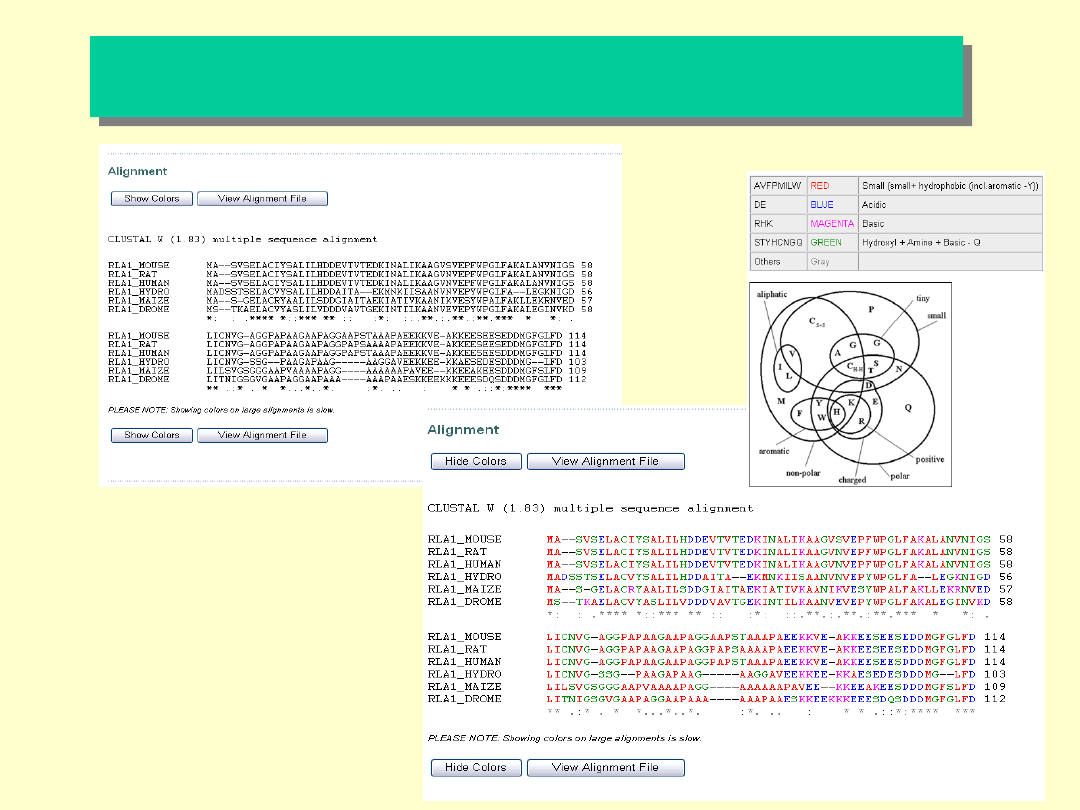
Zestawienie sekwencji aminokwasowych dla białek
rybosomalnych
Clustal W
Zestawienie sekwencji aminokwasowych dla białek
rybosomalnych
Clustal W

Drzewo filogenetyczne białek
rybosomalnych
Drzewo filogenetyczne białek
rybosomalnych
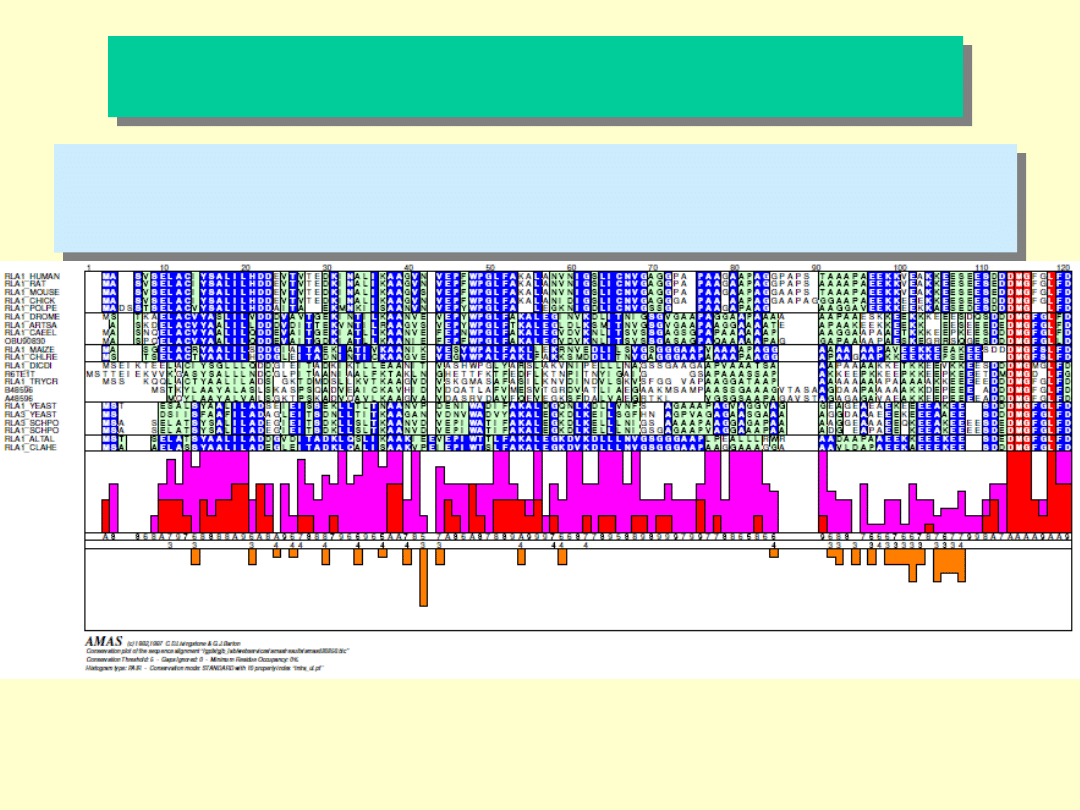
Wtórne dopasowanie sekwencji – AMAS
http://www.compbio.dundee.ac.uk/amas
Wtórne dopasowanie sekwencji – AMAS
http://www.compbio.dundee.ac.uk/amas
AMAS – służy do badania wzajemnych zależności między
sekwencjami oraz do identyfikacji reszt aminokwasowych o istotnym
znaczeniu funkcjonalnym.
AMAS – służy do badania wzajemnych zależności między
sekwencjami oraz do identyfikacji reszt aminokwasowych o istotnym
znaczeniu funkcjonalnym.
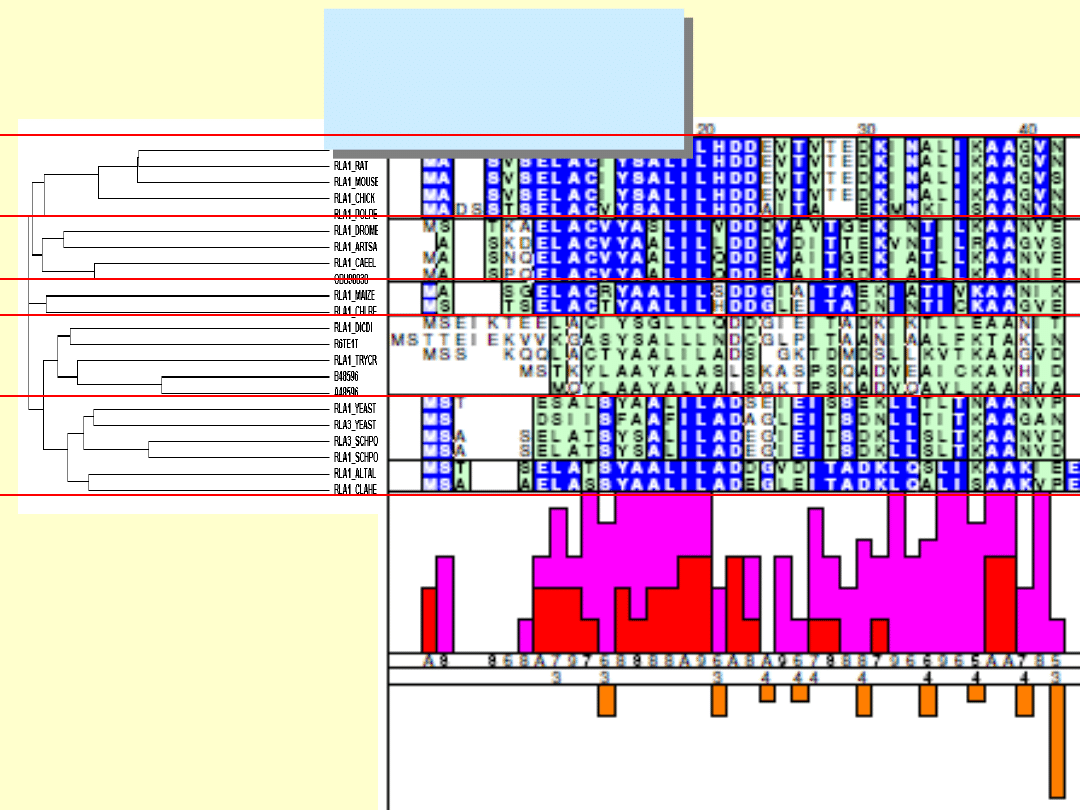
Zestawienie wyników
drzewa
filogenetycznego z
analizą AMAS.
Zestawienie wyników
drzewa
filogenetycznego z
analizą AMAS.
P
osition
S
pecific
I
terative - BLAST (PSI-
BLAST)
P
osition
S
pecific
I
terative - BLAST (
PSI
-
BLAST)
PSI-BLAST jest to program do poszukiwania białek homologicznych.
Odznacza się on wysoką czułością i specyficznością w działaniu. Program ten
wykorzystuje protokół PSSM (position-specific scoring matrix)
nazywanym także profilem aminokwasowym. W procesie analizy PSI-
BLAST tworzone jest automatycznie zestawienie wielu białek (MSA) i na
podstawie analizy dla każdego aminokwasu przyporządkowana jest wartość
liczbowa określająca częstotliwość występowania danego aminokwasu w
określonej pozycji polipeptydu. Aminokwasy bardzo konserwatywne otrzymują
wysoką wartość zaś aminokwasy nie konserwatywne otrzymują wartości
zbliżone często do zera. Utworzony w ten sposób profil aminokwasowy służy
do ponownego przeszukiwania bazy danych.
PSI-BLAST jest to program do poszukiwania białek homologicznych.
Odznacza się on wysoką czułością i specyficznością w działaniu. Program ten
wykorzystuje protokół PSSM (position-specific scoring matrix)
nazywanym także profilem aminokwasowym. W procesie analizy PSI-
BLAST tworzone jest automatycznie zestawienie wielu białek (MSA) i na
podstawie analizy dla każdego aminokwasu przyporządkowana jest wartość
liczbowa określająca częstotliwość występowania danego aminokwasu w
określonej pozycji polipeptydu. Aminokwasy bardzo konserwatywne otrzymują
wysoką wartość zaś aminokwasy nie konserwatywne otrzymują wartości
zbliżone często do zera. Utworzony w ten sposób profil aminokwasowy służy
do ponownego przeszukiwania bazy danych.

2-NITROPROPANE DIOXYGENASE (NITROALKANE
OXIDASE) (2-NPD) z Williopsis saturnus var.
mrakii
2-NITROPROPANE DIOXYGENASE (NITROALKANE
OXIDASE) (2-NPD) z Williopsis saturnus var.
mrakii
MRSQIQSFLK TFEVRYPIIQ APMAGASTLE LAATVTRLGG IGSIPMGSLS EKCDAIETQL
ENFDELVGDS GRIVNLNFFA HKEPRSGRAD VNEEWLKKYD KIYGKAGIEF DKKELKLLYP
SFRSIVDPQH PTVRLLKNLK PKIVSFHFGL PHEAVIESLQ ASDIKIFVTV TNLQEFQQAY
ESKLDGVVLQ GWEAGGHRGN FKANDVEDGQ LKTLDLVSTI VDYIDSASIS NPPFIIAAGG
IHDDESIKEL LQFNIAAVQL GTVWLPSSQA TISPEHLKMF QSPKSDTMMT AAISGRNLRT
ISTPFLRDLH QSSPLASIPD YPLPYDSFKS LANDAKQSGK GPQYSAFLAG SNYHKSWKDT
RSTEEIFSIL VQDL
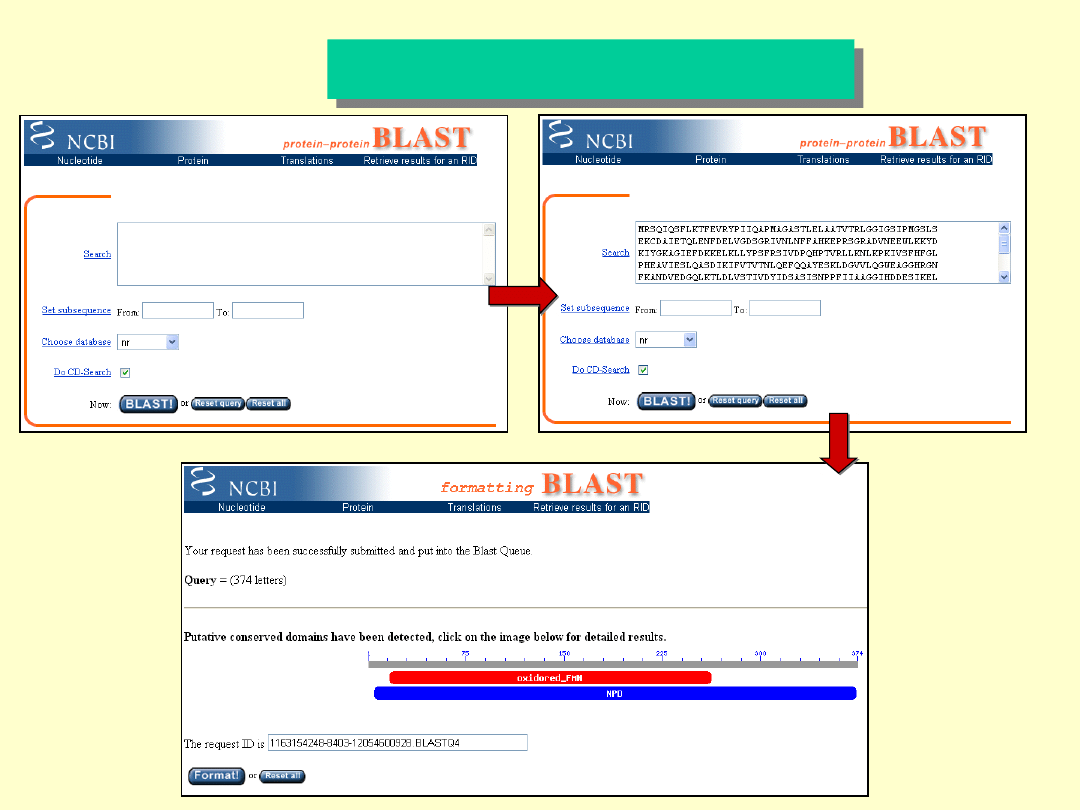
PSI-BLAST
PSI-BLAST
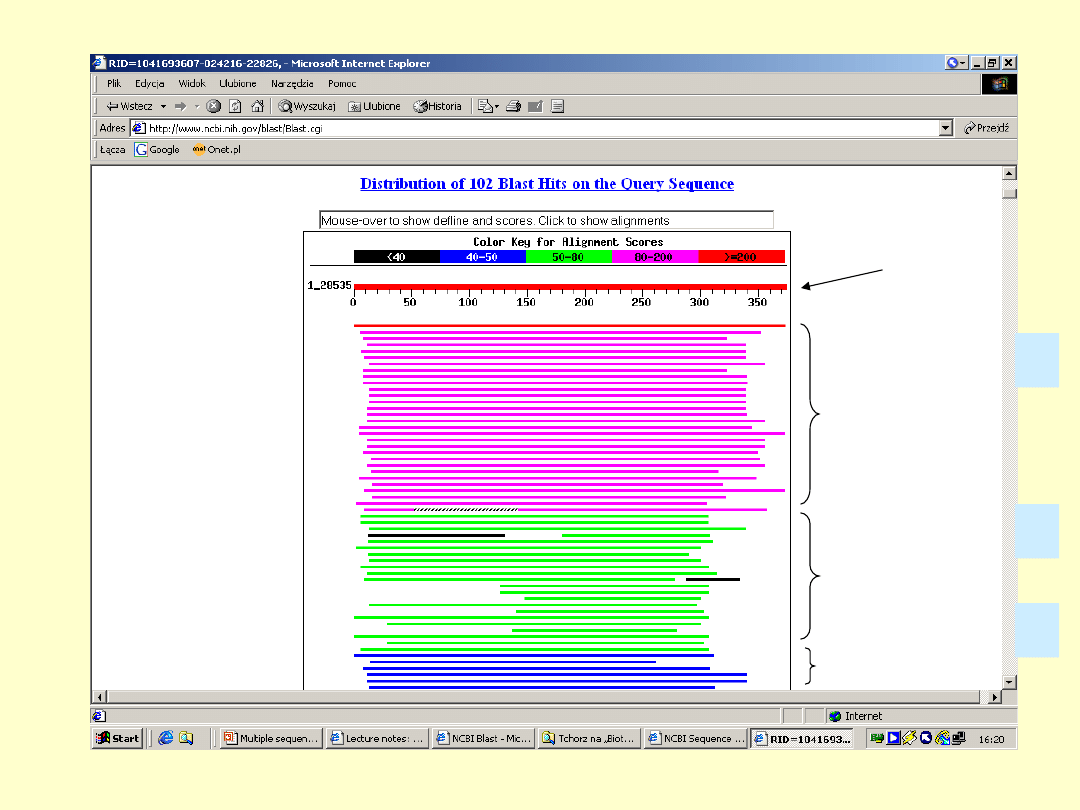
2-
NPD
Sekwencje o wysokim stopniu
podobieństwa (prawdopodobnie
białka homologiczne).
Sekwencje o niskim stopniu
podobieństwa (mogą należeć do
białek homologicznych).
Sekwencje o niskim stopniu
podobieństwa (prawdopodobnie
białka nie homologiczne).
1
2
3
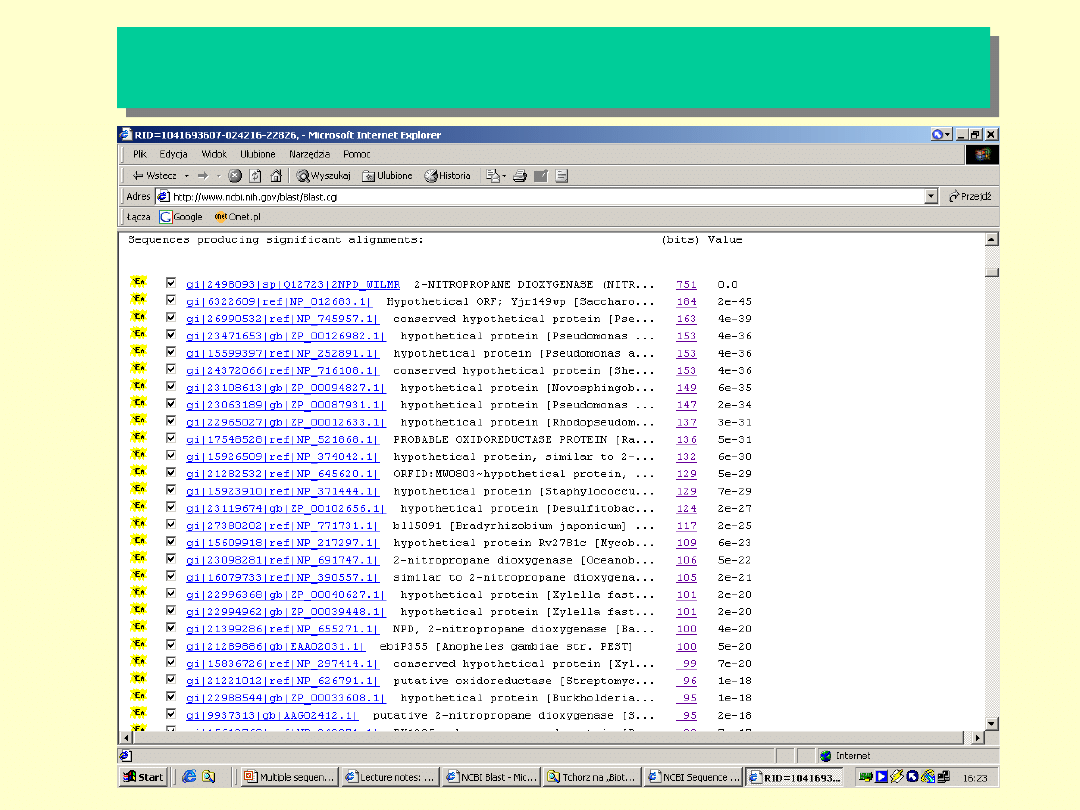
Lista sekwencji uzyskana w procesie
pierwszej analizy
Lista sekwencji uzyskana w procesie
pierwszej analizy
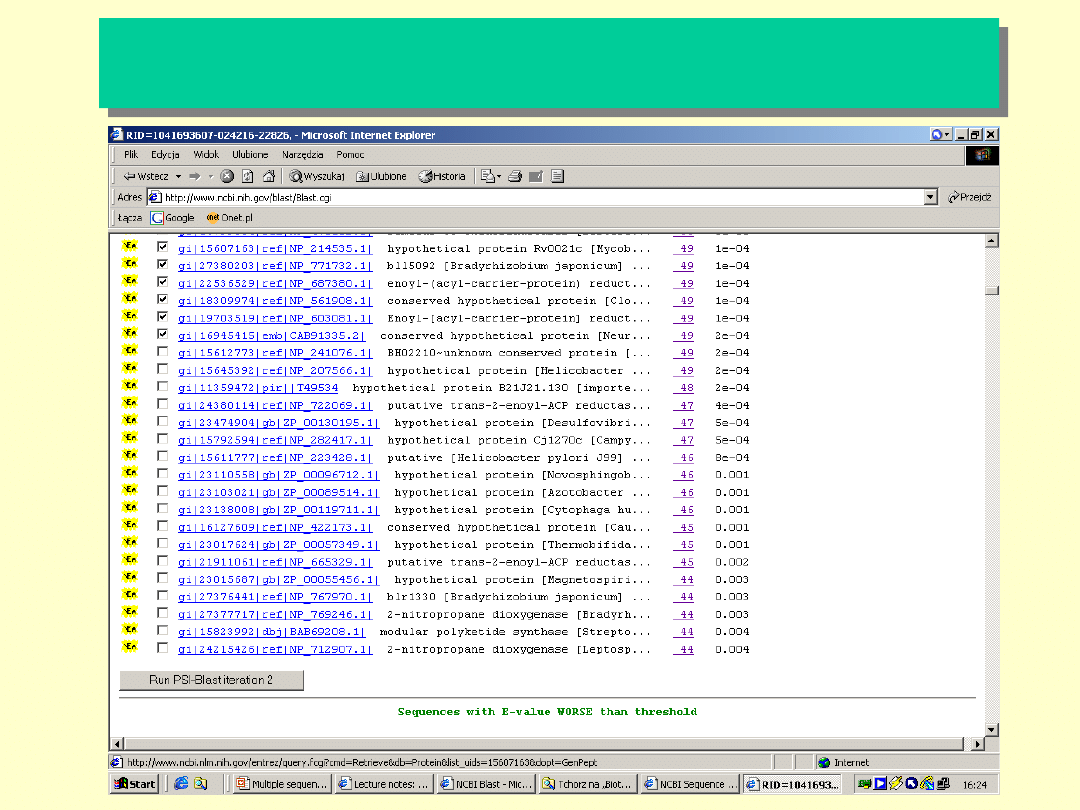
Selekcja sekwencji na podstawie wartości E
(E
xpectation value
) określającej stopień
podobieństwa
Selekcja sekwencji na podstawie wartości E
(E
xpectation value
) określającej stopień
podobieństwa
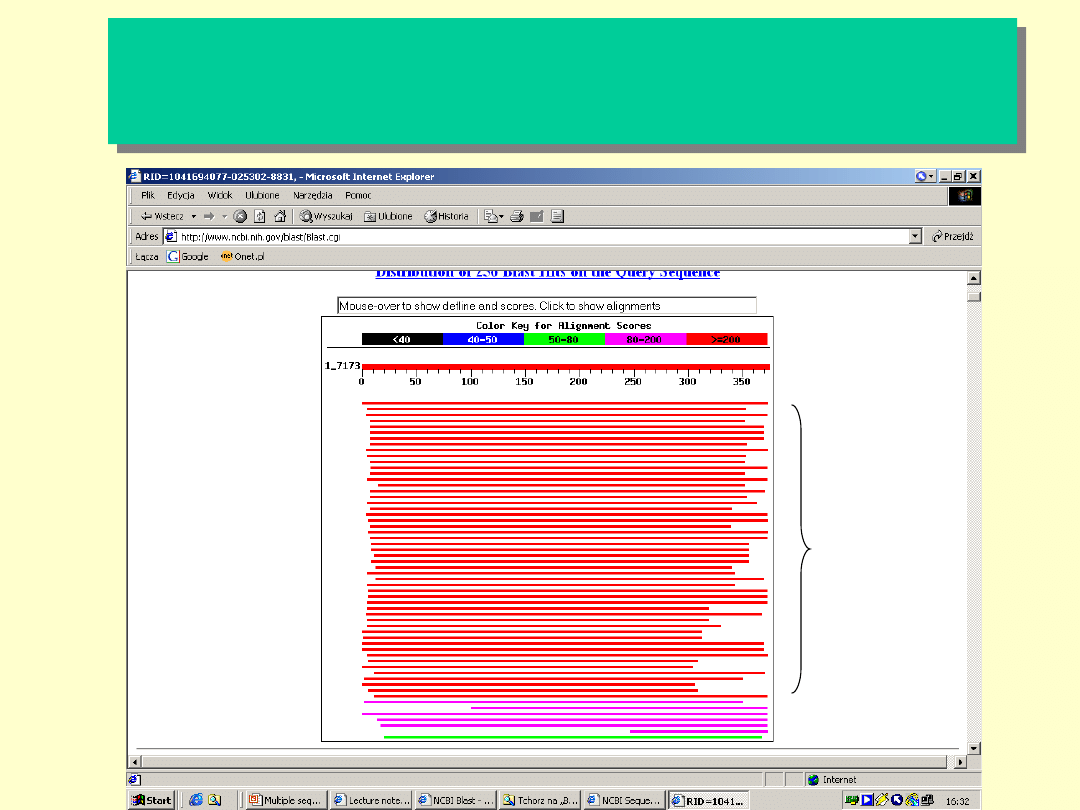
Graficzny obraz sekwencji otrzymany po
drugiej rundzie analiz za pomocą PSI-BLAST
Graficzny obraz sekwencji otrzymany po
drugiej rundzie analiz za pomocą PSI-BLAST
Sekwencje o wysokim stopniu
podobieństwa (prawdopodobnie
białka homologiczne).
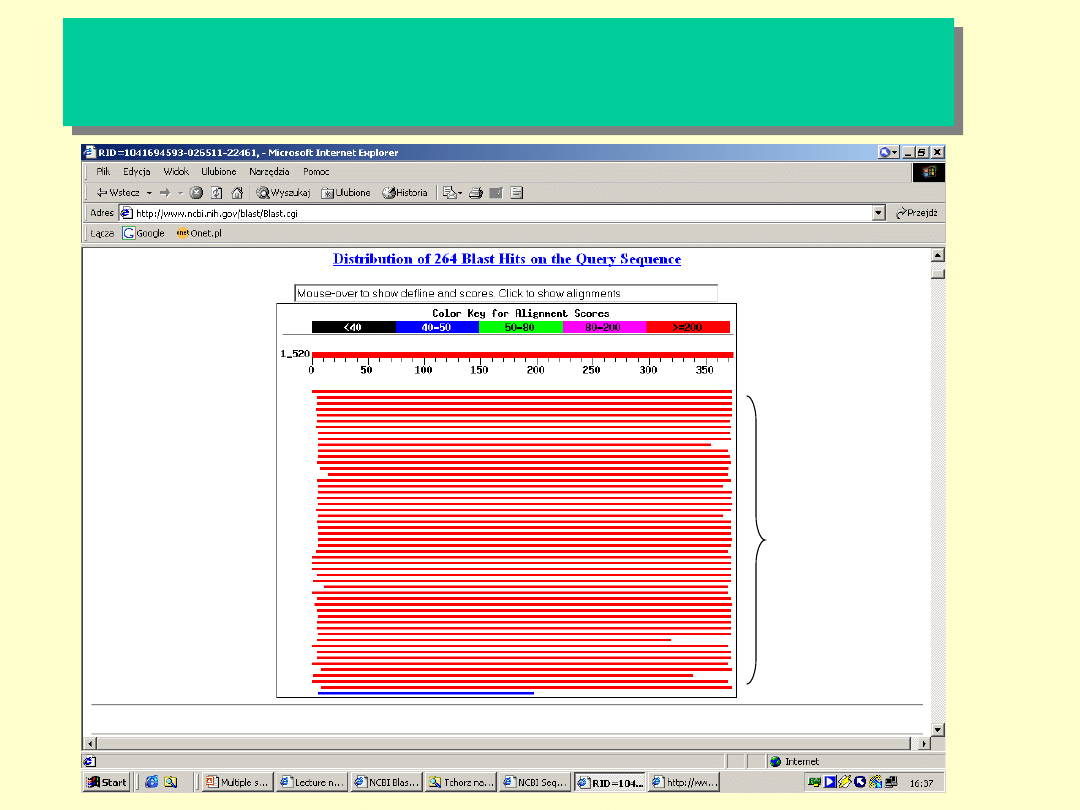
Graficzny obraz sekwencji otrzymany po
trzeciej rundzie analiz za pomocą PSI-BLAST
Graficzny obraz sekwencji otrzymany po
trzeciej rundzie analiz za pomocą PSI-BLAST
Sekwencje o wysokim stopniu
podobieństwa (prawdopodobnie
białka homologiczne).

Globalne dopasowanie dwóch sekwencji
aminokwasowych
Globalne dopasowanie dwóch sekwencji
aminokwasowych
HYGQCGGI...GYSGPTVCASGTTCQVLNPYY
HWGQCGGI...GYSGCKTCTSGTTCQYSNDYY
QWGQCGGI...GYTGSTTCASPYTCHVLNPYY
VWGQCGGI...GWSGPTNCAPGSACSTLNPYY
VWGQCGGQ...NWSGPTCCASGSTCVYSNDYY
LYGQCGGA...GWTGPTTCQAPGTCKVQNQWY
IWGQCGGN...GWTGATTCASGLKCEKINDWY
VWGQCGGN...GWTGPTTCASGSTCVKQNDFY
DWAQCGGN...GWTGPTTCVSPYTCTKQNDWY
QWGQCGGQ...NYSGPTTCKSPFTCKKINDFY
RWQQCGGI...GFTGPTQCEEPYICTKLNDWY
HWAQCGGI...GFSGPTTCPEPYTCAKDHDIY
LYEQCGGI...GFDGVTCCSEGLMCMKMGPYY
VWAQCGGQ...NWSGTPCCTSGNKCVKLNDFY
PYGQCGGM...NYSGKTMCSPGFKCVELNEFF
AYYQCGGSKSAYPNGNLACATGSKCVKQNEYY
RYAQCGGM...GYMGSTMCVGGYKCMAISEGS
EYAACGGE...MFMGAKCCKFGLVCYETSGKW
Aminokwasy konserwatywne ...QCGG.......G...C.....C.......
HYGQCGGI...GYSGPTVCASGTTCQVLNPYY
HWGQCGGI...GYSGCKTCTSGTTCQYSNDYY
QWGQCGGI...GYTGSTTCASPYTCHVLNPYY
VWGQCGGI...GWSGPTNCAPGSACSTLNPYY
VWGQCGGQ...NWSGPTCCASGSTCVYSNDYY
LYGQCGGA...GWTGPTTCQAPGTCKVQNQWY
IWGQCGGN...GWTGATTCASGLKCEKINDWY
VWGQCGGN...GWTGPTTCASGSTCVKQNDFY
DWAQCGGN...GWTGPTTCVSPYTCTKQNDWY
QWGQCGGQ...NYSGPTTCKSPFTCKKINDFY
RWQQCGGI...GFTGPTQCEEPYICTKLNDWY
HWAQCGGI...GFSGPTTCPEPYTCAKDHDIY
LYEQCGGI...GFDGVTCCSEGLMCMKMGPYY
VWAQCGGQ...NWSGTPCCTSGNKCVKLNDFY
PYGQCGGM...NYSGKTMCSPGFKCVELNEFF
AYYQCGGSKSAYPNGNLACATGSKCVKQNEYY
RYAQCGGM...GYMGSTMCVGGYKCMAISEGS
EYAACGGE...MFMGAKCCKFGLVCYETSGKW
Aminokwasy konserwatywne ...QCGG.......G...C.....C.......
Przykład analizy sekwencji aminokwasowej
białka cellobiohydrolazy (fragment
wiążący celuloze)
Przykład analizy sekwencji aminokwasowej
białka cellobiohydrolazy (fragment
wiążący celuloze)
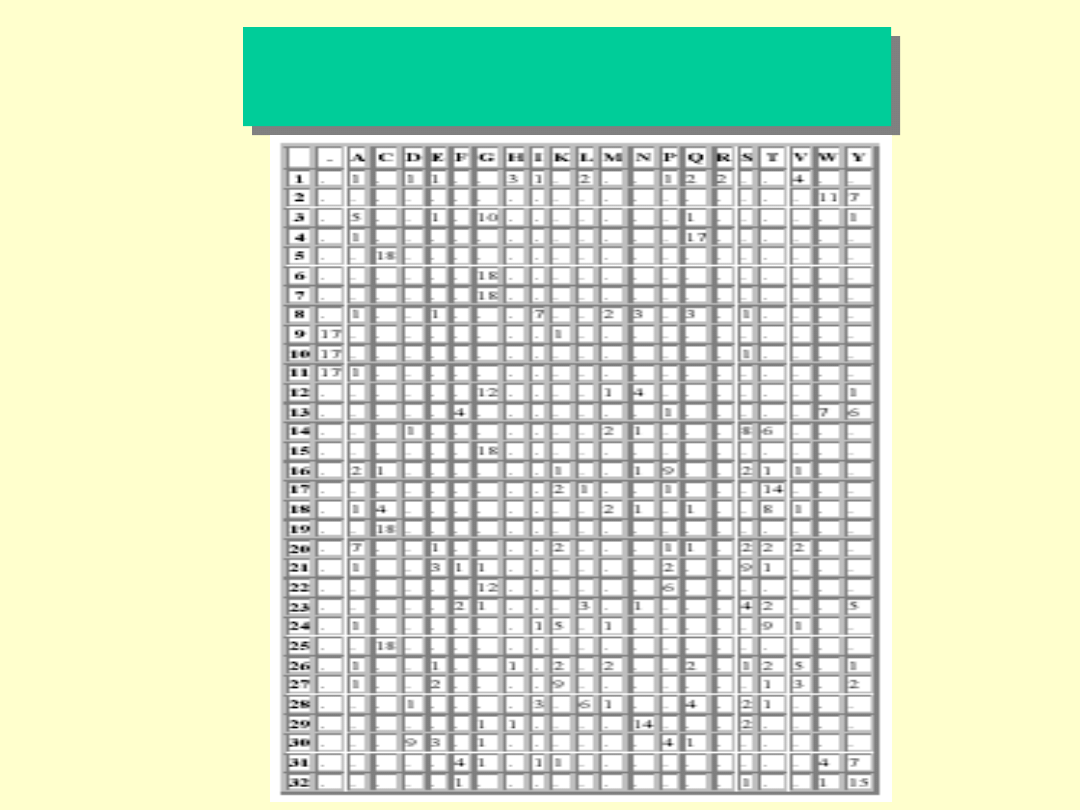
Profil aminokwasowy dla
cellobiohydrolazy
(fragment wiążący celuloze)
Profil aminokwasowy dla
cellobiohydrolazy
(fragment wiążący celuloze)
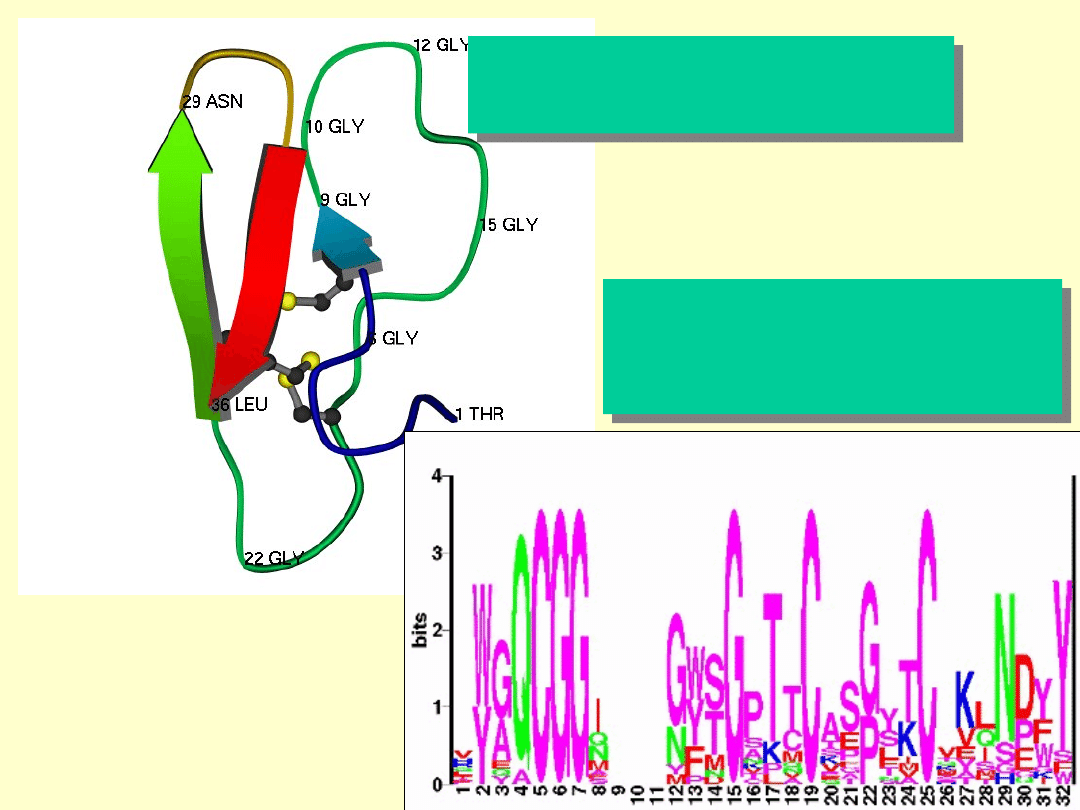
Graficzny obraz profilu
aminokwasowego dla
cellobiohydrolazy
Graficzny obraz profilu
aminokwasowego dla
cellobiohydrolazy
Struktura 3D dla cellobiohydrolazy
(fragment odpowiedzialny za
wiązanie się do celulozy)
Struktura 3D dla cellobiohydrolazy
(fragment odpowiedzialny za
wiązanie się do celulozy)
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
Wyszukiwarka
Podobne podstrony:
elementy bioinformatyki wyklad2
Bioinformatyka wykład 1
Bioinformatyka wykład 3
elementy bioinformatyki wyklad4
bioinformatyka wyklad #6
bioinformatyka wyklad #3
Bioinformatyka wykłady
bioinfoI wyklad01
elementy bioinformatyki wyklad3
bioinfoI wyklad03
bioinfoI wyklad02
elementy bioinformatyki wyklad1
Bioinformatyka wykładMocx
bioinformatyka wyklad #2
Bioinformatyka wykład 5
bioinfoI wyklad04
Bioinformatyka wykład ocx
elementy bioinformatyki wyklad2
więcej podobnych podstron