SPRAWOZDANIE
Inżynieria genetyczna - Laboratorium
Ćwiczenie nr 4 i 5:
Przygotowanie insertu EcRDBD do klonowania (generowanie miejsc restrykcyjnych BamHI) - reakcja PCR
Przygotowanie plazmidu pGEX - 2T do klonowania - elektroforetyczna analiza plazmidowego DNA oczyszczonego po trawieniu i defosforylacji oraz szacowanie jego stężenia
Elektroforeza analityczna produktów PCR
Izolacja produktu PCR (protokół Clean - up) (A&A Biotechnology)
Ligacja wektora pGEX - 2T zlinearyzowanego enzymem BamHI i defosforylowanego
(pGEX - 2T/BamHI) z fragmentem DNA kodującym domenę wiążącą DNA receptora ekdysteroidowego (EcRDBD/BamHI) przy użyciu ligazy T4Transformacja komórek kompetentnych (Maniatis i wsp. 1989)
1. Procedura i metodyka
W celu przeprowadzenia reakcji PCR przygotowano następujące roztwory w probówkach Eppendorfa:
Roztwór 1 - mieszanina reakcyjna:
3 μl pBS - EcR/EcoRI (około 15ng) - matryca DNA
4,1 μl WR5 (50 pmoli) - starter przedni („forward”)
4,2 μl WR6 (50 pmoli) - starter tylni (”reverse”)
8 μl dNTP (mieszanina nukleotydów, C = 1,25mM każdego)
5 μl bufor dla polimerazy [10x]
1 μl polimerazy - dodawanej po tzw. „gorącym starcie”
24,7 μl H2O
= 50 μl razem
Roztwór 2 - kontrola (K):
4,1 μl WR5 (50 pmoli)
4,2 μl WR6 (50 pmoli)
8 μl dNTP (mieszanina nukleotydów, C = 1,25mM każdego)
5 μl bufor dla polimerazy [10x]
1 μl polimerazy - dodawanej po tzw. „gorącym starcie”
27,7 μl H2O
= 50 μl razem
Na powierzchnię roztworów nałożono 50 μl oleju parafinowego.
Tak przygotowane próbki umieszczono w termocyklerze i ustawiono program reakcji PCR zgodnie z instrukcją.
Po „gorącym starcie” dodano 1 μl termostabilnej polimerazy, po czym „puszczono” reakcję dalej według nastawionych wcześniej cyklów.
W następnym kroku zlinearyzowany, oczyszczony i defosforylowany wektora pGEX - 2T poddano analizie densytometrycznej; w tym celu przeprowadzono horyzontalną elektroforezę analityczną w żelu agarozowym o stężeniu 0,8% przygotowanym w 1xTBE
i zawierającym bromek etydyny w stężeniu 0,4 μg/ml. Elektroforezę prowadzono w buforze 1xTBE przy stałym napięciu 75 V przez około 50min.W celu przeprowadzenia powyżej opisanej elektroforezy przygotowano następujący roztwór:
Roztwór do elektroforezy analitycznej:
3 μl roztworu plazmidowego DNA po oczyszczeniu
7 μl TE
2 μl 5x buforu do próbek
= 12 μl razem
Oprócz analizowanych próbek, na żel naniesiono również 5 μl markera wagowego FastRuler™ DNA Ladder, Middle Range, (MBI Fermentas).
Kolejnym krokiem było wykonanie elektroforezy analitycznej produktów przeprowadzonej reakcji PCR. W tym celu przygotowano następujące roztwory:
Roztwór 1 - próbka:
5 μl próbki po reakcji PCR (pobranej spod oleju)
5 μl TE
2 μl 6xSB
= 12 μl razem
Roztwór 2 - kontrola (K):
5 μl próbki po reakcji PCR (spod oleju z roztworu kontroli)
3 μl TE
2 μl 6xSB
= 12 μl razem
Oprócz analizowanych próbek na żel naniesiono też 5 μl markera wagowego FastRuler™ DNA Ladder, Middle Range, (MBI Fermentas).
Elektroforeza prowadzona była w żelu agarozowym o stężeniu agarozy równym 2%, przygotowanym w 1xTBE i zawierającym bromek etydyny w stężeniu 0,4 μg/ml. Elektroforezę prowadzono w buforze 1xTBE przy stałym napięciu 75 V przez około 50min.
Następnie przeprowadzono izolację produktu PCR z pozostałej części mieszaniny niepoddanej elektroforezie. W tym celu opisano nową probówkę Eppendorfa i przeniesiono do niej pozostałą część mieszniany po PCR spod oleju.
Do takiego roztworu dodano pięciokrotny nadmiar roztworu G (223 μl).
Mieszaninę krótko odwirowano w celu usunięcia resztek roztworu z wieczka i ścianek probówki.
Mieszaninę naniesiono na kolumnę do oczyszczania DNA i wirowano przez 30 s przy około 10 000 x g.
Następnie wyciągnięto kolumnę z probówki, przesącz wylano i kolumnę ponownie włożono do probówki.
Na złoże w kolumnie naniesiono 600 μl roztworu A1 do płukania.
Wirowano przez 30 s przu 10 000 x g.
Wyciągnięto kolumnę z probówki, odrzucono przesącz i kolumnę ponownie włożono do probówki.
Na złoże w kolumnie naniesiono 300 μl roztworu A1 do płukania.
Wirowano przez 2 min przy 10 000 x g.
Wyjęto kolumnę z probówki, przesącz ponownie odrzucono i kolumnę ponownie włożono do probówki.
Kolumnę z probówką inkubowano przez 3 min w temperaturze pokojowej w celu osuszenia złoża.
Osuszoną kolumnę umieszczono w nowej probówce Eppendorfa i naniesiono na złożę 30 μl buforu TE tak, by pokrył on całkowicie złoże.
Ponownie inkubowano w temperaturze pokojowej przez 3 min.
Następnie odwirowano przez 1 min przy 10 000 x g.
Przesącz na dnie probówki, zawierający docelowe DNA, przeniesiono do czystej opisanej probówki Eppendorfa i zanotowano jego objętość.
Tak oczyszczony produkt PCR znajdujący się w buforze TE przechowywano w temperaturze
-20ᴼ C.
Kolejnym krokiem doświadczenia było przeprowadzenie ligacji wektora pGEX - 2T zlinearyzowanego BamHI i defosforylowanego (pGEX - 2T/BamHI) z fragmentem DNA kodującym domenę wiążącą DNA receptora ekdysteroidewego (EcRDBD/BamHI) przy użyciu ligazy T4. W tym celu przygotowano następujące roztwory:
Roztwór 1 - reakcja na ligację, próbka:
1,5 μl pGEX-2T/BamHI (C = 15 ng/μl) (wektor)
1,65 μl EcRDBD/BamHI (C = 3 ng/μl) (insert)
1 μl ligazy T4 (1U) (MBI Fermentas) (enzym)
2 μl buforu do ligazy T4 [10x] (MBI Fermentas)
13,85 μl H2O
= 20 μl razem
Roztwór 2 - kontrola (K):
1,5 μl pGEX-2T/BamHI (C = 15 ng/μl) (wektor)
1 μl ligazy T4 (1U) (MBI Fermentas) (enzym)
2 μl buforu do ligazy T4 [10x] (MBI Fermentas)
15,5 μl H2O
= 20 μl razem
Ligację prowadzono w stosunku molowym 1:4, ninsertu = 4*nwektora , w sterylnej i opisanej probówce Eppendorfa. Reakcja była prowadzona w temperaturze pokojowej ~20ᴼ C przez 1h i 20 min.
Kolejnym etapem była transformacja komórek kompetentnych. W tym celu rozmrożono na lodzie 100 μl komórek kompetentych (XL1 - Blue).
Natychmiast przeniesiono je do schłodzonej sterylnej probówki Eppendorfa, która zawierała mieszaninę po ligacji.
Całość delikatnie wymieszano i inkubowano przez 20 min na lodzie.
Następnie próbki poddano szokowi cieplnemu w 37ᴼC przez 5 min w termomikserze.
Dodano 90 μl pożywki płynnej LB i inkubowano w termomikserze w temperaturze 37ᴼC przez 20 min.
W ostatnim kroku przeniesiono 200 μl zawiesiny transformowanych komórek na szalkę Petriego z podłożem LB-agar, zawierającym karbenicylinę w stężeniu końcowym 100 μg/ml.
Wykonano posiew dywanowy za pomocą sterylnej głaszczki.
Następnie prowadzono inkubację płytek przez 12h w 37ᴼC, po czym policzono kolonie transformantów wyrosłe na płytce po ligacji i na płytce kontrolnej.
2. Wyniki i obserwacje
Objętości starterów użytych w reakcji PCR w próbce i kontroli otrzymano w wyniku poniższych obliczeń:
Starter WR5 (forward)
MW = 29 nukleotydów x 320 g/mol nul. = 9280 g/mol
9280 g - 1 mol
x1 - 50 x 10-12 mola
x1 = 4,64 x 10-7 g masa startera WR5 [g]
C1 = 1,14 x 10-7 g/μl stąd:
1,14 x 10-7 g - 1 μl
4,64 x 10-7 - V1
V1 = 4,1 μl objętość startera WR5 [μl]
Starter WR6 (reverse)
MW = 30 nukleotydów x 320 g/mol nul. = 9600 g/mol
9600 g - 1 mol
x2 - 50 x 10-12 mola
x2 = 4,80 x 10-7 g masa startera WR5 [g]
C2 = 1,14 x 10-7 g/μl stąd:
1,14 x 10-7 g - 1 μl
4,80 x 10-7 - V2
V2 = 4,2 μl objętość startera WR6 [μl]
Przeprowadzony program PCR:
Temp 1 94ᴼC
Czas 1 5 min „gorący start”
Cykl 1 -----
Dodanie 1 μl termostabilnej polimerazy DNA
Temp 2 94ᴼC
Czas 2 1 min denaturacja DNA
Cykl 2 -----
Temp 3 58ᴼC
Czas 3 0,5 min 5x stapianie MATRYCA
Cykl 3 ----- PIERWOTNA
Temp 4 72ᴼC
Czas 4 0,5 min polimeryzacja
Cykl 4 do 2-giego
Temp 5 94ᴼC
Czas 5 1 min denaturacja
Cykl 5 -----
15x
Temp 6 72ᴼC
Czas 6 1 min stapianie i polimeryzacja
Cykl 6 do 5-ego MATRYCA
WTÓRNA
Temp 7 72ᴼC
Czas 7 2 min dokończenie polimeryzacji
Cykl 7 -----
Temp 8 4ᴼC
Czas 8 PAUZA zakończenie (schłodzenie mieszaniny reakcyjnej)
Cykl 8 -----
Do przygotowana próbki do elektroforezy plazmidu pGEX-2T zastosowano końcową objętość 12 μl, tak aby roztwor 2 μl 6xSB rozcieńczony był w niej 6 razy (do stężenia 1x) i to z niej wynikają ilości pozostałych składników. W ten sposób użyto 3 μl plazmidowego DNA i 7 μl roztworu TE.
Wyniki elektroforetycznej analizy plazmidowego DNA, oczyszczonego po trawieniu i defosforylacji przedstawione są poniżej. Żel analizowano w świetle UV przy krótszej długości fali 254 nm.
No. |
Label |
Type |
Mean Bkgd. (Int) |
Abs. Quant. (ng) |
Rel. Quant. |
# of Pixels |
Area (mm2) |
1 |
U1 |
Unknown |
489,61 |
103,23 |
263,87 |
5885,00 |
24,60 |
2 |
U2 |
Unknown |
338,98 |
0,39 |
1,00 |
2592,00 |
10,84 |
3 |
U3 |
Unknown |
332,32 |
0,93 |
2,38 |
2574,00 |
10,76 |
4 |
U4 |
Unknown |
330,42 |
0,66 |
1,70 |
3162,00 |
13,22 |
5 |
U5 |
Unknown |
325,43 |
0,46 |
1,18 |
3605,00 |
15,07 |
6 |
U6 |
Unknown |
330,27 |
3,12 |
7,99 |
3003,00 |
12,55 |
7 |
B1 |
Background |
344,04 |
N/A |
N/A |
20496,00 |
85,68 |
8 |
Std1 |
Standard |
364,50 |
20,00 |
51,12 |
4158,00 |
17,38 |
Wymagana była minimalna ilość 5 ng wektora, dlatego też do dalszych badań użyto próbki grupy I (U1), gdzie wektor znajdował się w najwyższym stężeniu. Elektroforezę przeprowadzono przy 3 μl roztworu plazmidowego. Stężenie to policzono wg. poniższego schematu:
![]()
[ng] ![]()
[μl] 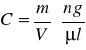
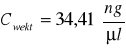
Podobnie, w elektroforezie analitycznej produktów PCR zastosowano końcową objętość 12 μl. Pobrano 5 μl próbki po PCR spod oleju, 5 μl roztworu TE i 2 μl 6xSB (aby jego końcowe stężenie
w 12 μl wynosiło 1x). Jednak po nabraniu całej zawartości do pipety okazało się, że dokładna ilość mieszaniny reakcyjnej wynosi 11,7 μl.Kontrola przygotowana została tak samo, jak próbka, jednak 5 μl próbki po PCR pobierano
z kontroli z reakcji PCR. Końcowa objętość wynosiła 12 μl.Elektroforeza analityczna produktów PCR niestety nie przyniosła pożądanych rezultatów. Widoczna jest jedynie smuga po użytych starterach przy każdej studzience. Żel analizowano
w świetle UV przy krótszej długości fali 254 nm.
Podczas izolacji produktu PCR zastosowano 5-krotny nadmiar roztworu G (223 μl). Dokładną objętość wyliczono na podstawie posiadanych ilości produktu PCR: 39,3 μl i 5,3 μl - razem 44,6 μl.
![]()
μl roztworu G
Do ligacji wektora pGEX-2T, zlinearyzowanego enzymem BamHI i defosforylowanego
(pGEX-2T/BamHI) fragmentem DNA kodującym domenę wiążącą DNA receptora ekdysteroidowego (EcRDBD/BamHI) przy użyciu ligazy T4 użyto następujących ilości poszczególnych składników:
Wyjściowe stężenie wektora, obliczone na podstawie wyników elektroforezy, wynosiło
34,41 ng/μl. Wymagane było rozcieńczenie do stężenia 15 ng/μl, zatem policzono to rozcieńczenie: 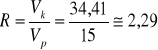
Zatem mamy objętość 7,5 μl wektora
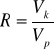
![]()
![]()
μl
Objętość roztworu TE: ![]()
μl
(wtedy właśnie stężenie wektora wynosi 15 ng/μl)
Wektor pGEX-2T:
![]()
ng/μl
![]()
ng (20 x 10-9 g) ![]()
![]()
![]()
W przypadku insertu DNA stosowany był 4-krotny nadmiar molowy (ninsertu = 4nwektora):
![]()
ng/μl
![]()
pz
![]()
pz
![]()
g/mol pz
![]()
g/mol
![]()
g/mol
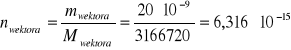
mol![]()
mol
![]()
ng
![]()
![]()
Po inkubacji bakterii w 37ᴼC policzono kolonie transformantów wyrosłe na płytce po ligacji i płytce kontrolnej. Stopień religacji wektora policzono, dzieląc liczbę kolonii wyrosłych na płytce kontrolnej przez liczbę kolonii na płytce „po ligacji”. Wynik podano w procentach:
Liczba kolonii na płytce kontrolnej: 2
Liczba kolonii na płytce „po ligacji”: 14
Stopień religacji: ![]()
3. Wnioski
Zastosowane zostały startery WR5 i WR6 o długości odpowiednio 29 i 30 nukleotydów o temperaturze stapiania równej 58ᴼC, wyliczonej na podstawie obecności poszczególnych par zasad we fragmentach. Im starter jest dłuższy, tym specyficzność reakcji PCR jest wyższa.
Jony Mg2+ obecne w mieszaninie reakcyjnej PCR w buforze tworzą kompleksy z DTP, starterami i matrycą DNA, stąd też wymagane jest ich odpowiednie stężenie. Jeśli jest ono zbyt niskie, produktów PCR jest mało i wydajność reakcji spada. Z kolei, jeśli występuje zbyt wysokie stężenie, może powstać wiele niespecyficznych produktów.
Ważne są równe ilości dNTP (dATP, dCTP, dGTP i dTTP) w mieszaninie reakcyjnej, gdyż różnica w stężeniu nawet jednego z deoksynukleotydów drastycznie zwiększa poziom źle włączonych zasad.
50 μl oleju parafinowego dodano na powierzchnię mieszaniny reakcyjnej w celu zapobieżenia jej parowaniu. Tzw. „gorący start” zastosowany został w celu stworzenia optymalnych warunków dla działania polimerazy DNA. Nastąpiła denaturacja ewentualnych zanieczyszczeń białkowych i zwiększona została wydajność enzymu. Dopiero po przeprowadzeniu „gorącego startu” dodano 1 μl enzymu.
Z racji wprowadzania miejsca restrykcyjnego, przeprowadzone zostały 2 cykle podczas reakcji PCR. Wynika to z faktu, że użyta matryca nie zawierała wcześniej miejsca restrykcyjnego.
W wyniku elektroforezy analitycznej nie otrzymano produktów PCR. Możliwe jest, że do mieszaniny reakcyjnej nie dodano matrycy DNA, co objawia się właśnie brakiem tej matrycy na żelu. Widoczna jest jedynie smuga po użytych starterach.
Maciej Duda 168516
Wojciech Ślęczek 168560
2000 bp
100 bp
5000 bp
850 bp
400 bp
Wyszukiwarka
Podobne podstrony:
IG, inżynieria genetyczna
IG.7 - Detekcja zakażeń w hodowlach komórkowych techniką PCR, Genetyka, Inżynieria genetyczna
IG.4 - Uszkodzenia i naprawa DNA w komórkach nowotworowych, Genetyka, Inżynieria genetyczna
IG.6 - Oznaczanie aktywności enzymatycznej metaloproteinaz komórkowych, Genetyka, Inżynieria genetyc
IG notatki LAB, inżynieria genetyczna, laboratorium
IG.2 - Ocena stopnia degradacji kwasów nukleinowych poprzez rozdział elektroforetyczny, Genetyka, In
IG sprawozdanie 1, inżynieria genetyczna
IG.1 - Techniki izolacji kwasów nukleinowych, Genetyka, Inżynieria genetyczna
IG.3 - Indukcja i detekcja uszkodzeń oksydacyjnych w komórkach eukariotycznych HPLC-ECD, Genetyka, I
inżynieria genetyczna
Inzynieria genetyczna roslin i jej wykorzystanie w rolnictwie
bioetyka inzynieria genetyczna
Obliczenia cw 2, studia, materiały od roku wyżej, Inżynieria genetyczna, inżynieria
rozwi-zania, inżynieria genetyczna, inż genetyczna, Inzynieria genetyczna - zagadnienia
ZAGADNIENIA DO KOLOKWIUM 2, Genetyka, Inżynieria genetyczna
SPRAWOZDANIE Z BIOLOGII KOMÓRKI I INŻYNIERII GENETYCZNEJ I
Inżynieria genetyczna
więcej podobnych podstron