Kinetyka reakcji: reakcje biotechn.: ΣνiAi=0, νi>0 produkty, νi<0 substraty, νi- współcz. stech. Stopień konwersji (przemiany )-stosunek przereagow. Liczby moli do pierwotnej liczby moli, można go odnosić do dowolnego składnika: α=Δn/no=(nso-ns)/nso (substrat), liczba postępu reakcji (liczba przereagowanej liczby moli do współcz. stechiom.): γi=Δni/νi.
Równowaga chemiczna - każda r. jest równowagowa, może być przesunięta w prawo, lub w lewo, ale ukł. dąży zawsze do stanu równowagi. Stałą równ. można uzależnić od temp. np. przez potencjał Gibbsa (entalpię swobodną). Równanie van't Hoffa. K=exp(-ΔG/RT), ΔG >0 r.praktycznie niemożliwa, ΔG <0 r. zach. z dużą wydajn., K-stała równowagi. Entalpia: ΣνiHi=ΔH, ΔH<0 egzo, wydziela się ciepło, entalpia r. jest istotna, bo stałą równowagi często podaje się w zależności od niej: K=Aexp(-ΔH/RT), wraz ze wzrostem T spada stała szybk. r. ale ΔH>0, wraz ze wzrostem T rośnie stała szybk. r. ale ΔH<0. Reakcje w biotechnologii są heterogeniczne(przebieg. W ukł. Kilkufazowych). W roztworach heterogen. Mamy do czynienia z dyfuzją. Najwolnieszy etap procesu determinuje szybk. Całego proc. (bottle neck effect. Dyfuzja ogranicza szybk. Procesu, bo to ona kontrol. Jego szybk.
Szybk. reakcji: 1) szybk. obj.- zmiana ilości moli w czasie odniesiona do jednostki obj.: rA![]()
(1/V)*(dnA/dt)=dcA/dt, „+'dnA dla produktów, „-'dnA dla substratów, szybkość porównujemy przez współcz. stech.: rk/νk= rn/νn=…= ri/νi = ident., szybkość jest f-cją: r=f(T, skład, pH, p), najczęściej pracuje się pod ciśnieniem atmosferycznym, ale czasem trzeba je uwzględnić, np. przy procesach w wysokich kolumnach-ciśnienie słupa cieczy. Zależność od składu określa pr. działania mas Guldberga- Waagego - Szybkość r. chem. jest proporc. do efektywnego stęż. wszystkich uczestnicz. w niej reagentów. nA+mB→P: -rA=kCAn*CBm, k - stała szybk. Poszczeg. Składników, n,m - rząd reakcji, nie zawsze równy współcz. stech. Zależnośc od T: rown. Arrheniusa : k=koe-Eo/RT, teoria zderzeń i teoria kompleksów aktywnych : k=koTme -Ea/RT, 0≤m≤1, bariera energet. : dla r.chem: 40-300kJ/mol, dla biochem.: 5kJ/mol ( bo są katalizowane - enzymat.), wyznacz. Energii aktywacji: k=K0e-Ea/RT.
Kinetyka wzrostu drobnoustrojów: rozpatrujemy b. dużą populacje , musimy odnosić szybk. do aktualnej liczby drobnoustr. lub stęż. biomasy, Cx - aktualne stęż. biomasy, μ![]()
1dCx/Cxdt, rx=dCx/dt, obecność katalizatora obniża energię aktyw, ale nie zmienia stanu równowagi - enzym zmienia kinetykę, a nie termodynamikę reakcji.
Reakcje enzymat. ![]()
, r=d[P]/dt=k3[ES](zgodnie z pr. działania mas). Metoda równowagowa : na początku zaproponował ją Henry, a do końcowego etapu przyczynił się Michaelis i Menten. KM - stała Michaelisa - Menten, KM=[E][S]/[ES]=k2/k1, [Eo]=[E]+[ES] → [E], KM=(( [Eo]-[ES])*[S])/[ES] → [ES], r=k3*(([Eo][S])/KM+[S])= rmax([S]/(KM+[S])) równ. Michaelisa -Ment. Stała M-M opisuje powinowacto enzymu do subst. im wyższe KM tym większe powinowactwo. Z tego równania wynikają 2 opisy asymptotyczne: [S]<<KM: lim r ,[S]→0,(rmax/KM)*[S]=K[S] - r. I rzędu, [S]>>KM: lim r [S]∞ =rmax([S]/[S])=rmax[So] - r. 0 rzędu. Wyrażenie opis. Szybkość r. dla poszczeg. Składników: robimy układ równań: d[E]/dt=(k2+k3)[ES]-k1[E][S], d[ES]/dt=k1[E][S]-(k2+k3)[ES], d[S]/dt=k2[ES]-k1[E][S], d[P]/dt=k3[ES].
Założenie Bodensteina: zmiana stęż. Kompleksu w czasie jest =0 d[ES]/dt=0. Jednostka eznymat. (unit) 1U - ilość enzymu katal. Przekształcenie 1 umola substratu w produkt w czasie 1 min.: 1U=1umol/1min, S>>KM_. Jednostka w ukł. SI (katal)- ilość enzymu kataliz. Przekształc. 1 mola substr. W produkt w czasie 1 s.
Aktywność specyf.- miara czystości enzymu, im bardziej czysty enzym, tym wyższa aktywn. Specyf.
Liczba obrotów enzymu-max. liczba molekuł substratu obrócona w produkt w jednostce czasu w war. Nasycenia. Miara stałej szybk. r.K3.
Wyznacz. szybk. max i stałej Michaelisa:
1)linearyzacja Lineweaver-Burk → met. podwójnych odwrotności: 1/r=(KM+[S])/(rmax[S])= (KM / rmax)*(1/[S])+(1/ rmax); 2)met. Eodie - Hofstee: rmax/r =KM(1/[S])+1, rmax= KM(r/[S])+r → r; 3)Hanesa - Woolfa : [S]/r=(KM/rmax)+[(1/rmax)*[S]]
Enzymy allosteryczne,równ. Hilla: allosteria - zjawisko przyłącz. Się enzymu do innego miejsca niż centrum aktywne, np. hemoglobina(ma 4 podjednostki mogące przyłączyć substrat). W przebiegu r. udział bierze nie tylko jedna cząsteczka substratu ale n: E+nS <->[ES]n→E+P, k1[E][S]n=(k2+k3)[ES]n, KM=(k2+k3)/k1=([E][S]n)/[ES]n (stałe dysocjacji), [ES]n=([E][S]n)/KM, r=k3[ES]n, rmax=k3[Eo]=k3([E]+[ES]n), [Eo]=[E]+[ES]n, r/rmax= (k3[ES]n)/(k3([E]+[ES]n)=
[E][S]n/(KM[E]+([E][S]n/KM))= [E][S]n/(KM[E](1+[S]n/KM)=[S]n/(KM+[S]n), r=rmax*([S]n/(KM+[S]n)) <-- równ. Hilla. Enzymy alloster. Są b.często spotykane w praktyce. Oparte o nie jest praktycznie cała regulacja.
wyznaczenie współcz. kooperat.(n): rmax/r=(KM+[S]n)/[S]n=(KM/[S]n)+1, (rmax-r)/r=KM/[S]n, r/(rmax-r)=[S]n/KM, ln r/(rmax-r)=nln[S]-ln KM → n z nachylenia prostej ln (r/(rmax-r))=f(ln[S])
Inhibicja:
Prędkość r. zal. Od stęż. Substratu, enzymu, a także od temp., i pH. Subst. Wpływaj. Na szybk. r. nazywamy aktywatorami i inhibitorami.
i.kompetycyjna: r=rmax([S]/(KM(1+([I]/KI))+[S]), i.niekompetencyjna: w przyp. Np. przyłączenia się metalu ciężkiego (Hg,Cu) na stałe do grup przede wszystkim tiolowych inhibicja nieodwracalna.r=rmax ([S]/(KM+[S]))*(KI/(KI+[I])), wart. stałej KM pozostaje niezmieniona, ale spada wart. prędkości max. i. akompetycyjna: gdy inhibitor łączy się tylko z kompleksem ES: r=rmax [S]/(KM(1+([I]/KI)+[S]))*(KI/(KI+[I])), zmianie ulega zarówno KM jak i r max ,i. substratowa: nadmiar substratu wpływa niekorzystnie np. podczas ferment. Alkohol. (gdy dodamy za dużo cukru to ferment. Nie ruszy), cząst. Substr. Przyłącz. Się nie tylko do centr. Aktywnego, ale też do miejsc regulatorowych, co prowadzi do zmian konformacji w kompleksie, ES zmiana szybk. przemiany w produkt, r=rmax ([S]/(KM+[S]+([S]2/KIS))) równ. Haldane'a;
Wyznaczenie stężenia substratu, przy którym prędkość osiąga max. wart. [S]max: dr/d[S]=0, dr/d[S]={rmax(KM+[S]+([S]2/KIS))-rmax[S](1+(2[S]/KIS))}/{KM+[S]+[S]2/KIS}2=0, mianownik możemy zaniedbać: KM+[S]+([S]2/KIS)=[S]+(2[S]2/KIS), [S]2/KIS=KM, [S]max =![]()
, r=rmax*![]()
/(2KM+![]()
), zauważono, że spadek szybkości jest tak gwałtowny, że wykładnik potęgi powinien być wyższy niż 2: r=rmax*([S]/(KM+[S]+([S]n/KIS))) równ. Neufelda, i. produktem: przykład: ferment. Alkoholowa gdy stęż. Etanolu jest wyższe niż 6%, to fermentacja staje. r=rmax* ([S]/(KM+[S]))*(1-([P]/[Pmax])), Pmax- wart., przy której proces idzie, czasem zdarza się, że inhibicja jest zarówno substr. Jak i produktem, wtedy w równaniu M-M należy uwzgl. Oba człony.
Wpływ temp. Na szybk. r. enzymat.: im wyższa temp., tym większa szybk. r., jednak białko ulega denat. r=r max exp (-E/RT), reakcja denaturacji ma swoją stałą równowagi. Forma natywna zamienia się w zdegenerowaną. [N]↔[D], z równ. Van't Hoffa KD=[D]/[N]=exp (-ΔG/RT), KD=exp (-(ΔH-TΔS)/RT)=exp (-ΔH/RT)*exp (ΔS/R)=Aexp(-ΔH/RT), [D]/[N]=KD i Eo=[N]+[D]=[N](1+KD); [N]=Eo/(1+KD), r = rmax* ([Eo]exp (-E/RT))/(1+Aexp (-ΔH/RT))
Wpływ pH: rmax=k3[Eo], r=rmax[S]/([S]+KM*(1+(K1/[H+])+([H+]/K2))), stała KM zal. od stęż. jonów wodorowych. Pozorna stała KM zależna jest od pK, log KMpoz=logKM-pH+pK1
Faza wykładnicza -gdybyśmy chcieli opisać zmianę stęż. biomasy w czasie d[X]/dt , w fazie logarytm., to szybk. ta jest proporcj. do stężenia (r. autokatalityczne):
d[X]/dt=μ[X], μ-specyficzna szybkość wzrostu biomasy, μ![]()
(1/[X])*(d[X]/dt),r=d[X]/dt=μ[X] → szybkość objętościowa, całk. (x0 -x) d[X]/[X]= μ całk.(0-t) dt, ln ([X]/[Xo])=μt, [X]=[Xo]exp(μmax*t), μmax-w fazie logaryt. szybkość jest największa. Czas podwojenia biomasy ([X]=2[Xo]): 2[Xo]=[Xo]exp(μmax*τd), ln2=μmax*τd, →τd (np. dla bakterii rosnących w optym. war. E.coli: τd=20min, μmax=2h-1, Saccharomyces cerevisiae: τd=1,5, μmax=0,5h-1)
Model logistyczny - do równ. dokłada się jeszcze jeden człon - człon śmierci, bo równ. dla fazy wykładn. zakłada, że wzrost jest nieskończony: d[X]/dt= μmax[X]*(1-([X]/[Xmax])), d[X]/dt= μmax[X]- μmax *([X]2/[Xmax]), ([X]2/[Xmax])-człon śmierci, Po scałkowaniu w granicach Xo→X, 0→t mamy: [X]= [Xmax]/(1+exp(([Xo]/[Xmax])- μmaxt)), f-cja ta wygląda jak logistyczna stąd nazwa modelu: [X]=A/(1+exp (f(t)).
Model Monoda - monod zaproponował bardzo proste doświadcz. ukaz. zależność szybkości wzrostu od stęż substr. →obserwował krzywe wzrostu E.coli w zależności od stęż glukozy. Mają charakter. wielkośc μmax - max. specyficzna szybkość wzrostu biomasy, niezależna od stęż. Zależność μ[S]: μ=μmax[S]/(KS+[S]), KS-stała Monoda, w pełni analogiczna do równ. Michaelisa-Menten, KS określa przyswajalność danego substratu (np. dla glukozy KS=2-4mg/l. Model ten słuszny jest w dużym zakresie,bo wszystko odbywa się na drodze enzymat. i przez białka transportujące substrat do komórki, μmax jest miarą tego enzymu, który katalizuje najwolniejszy etap. Szukanie najwolniejszego etapu przez rozwiąz. ukł. równań.
d[X]/dt=μmax([S]/KS+[S])[X], d[S]/dt=(-1/Yxs)*μmax ([S]/KS+[S])[X], gdzie Y-współcz. wydajności
faza adapt. (lag faza): d[X]/dt=μmax*([S]/KS+[S])[X](1-exp(-t/tL))
Inne modele niestrukturalne(dotyczą tylko 1 substratu limitującego): 1. równ. Monoda: μ=μmax([S]/(Ks+ Ks[S0]+[S])); 2. równ. Contois: (uwzględnia stęż. biomasy) μ=μmax([S]/(Ksx[X]+[S])); 3. równ.Moser'a: μ=μmax([S]n/(Ks+[S]n)); 4. równ. Teissier'a: μ=μmax(1-exp(-Ks[S])); 5. równ. Konak'a: dμ/d[S]=k(μmax-μ)m dla m=1-równ. Teissier'a; dla m=2→ μ=μmax([S]/((1/kμmax)+[S]))
Model diauksji (uwzgl. większą ilość substratów limitujących): μ=μmax1([S1]/(KS1+[S1]+α2[S2])) + μmax2([S2]/(KS2+[S2]+α1[S1])), α1,α2-stałe
Równ. Haldey'a-Monoda (dla inhibicji substratowej): μ=μmax([S]/(Ks+ [S]+( [S]2/ KIS)))
Inhibicja produktem: 1.współzawodnicza μ=μmax([S]/(S+(1+([P]/ KIP))); 2.niewspółzawodnicza: μ=μmax(1/((1+(KS/[S])+(1+([P]/ KIP)))
Uwzględnienie metabolizmu endogennego; podejście: 1.Herbert'a: rx'= rx+μe[X]; wykorzystując model Monoda: rx= d[X]/dt=μmax[X]*([S]/(KS+[S]))+μ e[X], szybkość konsumpcji: rs= -d[S]/dt= rx'/YXS=(1/YXS)*( rx +μ e[X]); 2. Pirt'a: rs= -d[S]/dt=(μmax/YXS)*[X]* ([S]/(KS+[S]))+ms[X], współcz. zachowawczy: ms=μR/YXS,
reaktor z idealnym miesz. CSTR(B) - wszystkie param. są stałe, to co na wylocie=temu co w reaktorze, obj. kontrolna to cała obj. tego reaktora. Rys. V=const. , F0≈F1=const (bo ukł. wodne, rozcieńczone), uwzględniamy stechiometrię reakcji: A→2B, szybkość ubywania substr. A, to szybk. powst. produktu B, podziel. przez współcz. stechiom.: rA/νA= rB/νB, rA=rB/2, d(VCA1)/dt=FoCAo-F1CA1+rAV, CA1 bo rozpatrujemy tylko to co na wylocie, d(VCB1)/dt=FoCBo-F1CB1+rBV, Fo=F1=F, VdCA1/dt=d(VCA1)/dt, VdCB1/dt=d(VCB1)/dt, bo V=const, dCA1/dt=(F/V)(CAo-CA1)+(rA=kcA), dCB1/dt=(F/V)(CBo-CB1)+(rB=2kcA), r=k*CA. Teraz należy określić liczbę zmiennych, gdy mamy układ ustalony to kumulacja wynosi 0. F(CAo-CA1)=VrA
czas przebywania τ: obj. reaktora podzielona przez natęż przepływu (objętościowe), τ=V/F, D-współcz. rozcieńczenia, D=F/V, τ=(CAo-CA1)/rA, ale rA jest funkcją stężenia, można również wprowadzić stopień konwersji α (stosunek różnicy stęż. początk. i stęż. w dowolnym momencie do stęż. początk.): α= (CAo-CA1)/CAo=1-(CA1/ CAo), τ=CAα/r, gdzie r jest funkcją α
reaktor z przepł. tłokowym PFR(B): F=const (przepływ stały), FCA-F(CA+dCA)=rA(CA)dV, rA(CA)-r jest funkcją stęż, -FdCA=rAdV, dV/F=dτ, -dCA=rAdτ, V=całk. dV=-F całk dCA/rA(CA), V/F=τ= - całk. dCA/rA(CA), τ= - całk. dCA/rA(CA); interpret. graf. τ= - całk.(CA1- CAo) dCA/kCA; dla inhibicji substrat. r= rmax(CA/(KM+CA+(CA2/KI)))
Chemostat: Rys. FCi0-FCi±rVR=0, X=CX-steż. biomasy, S=CS-steż. substratu, F(X0-X)+ rxVR=0, X0=0, μ=(1/X)*(dX/dt)→ μX=rx; FX=μXVR, szybkość rozcieńczania: D=F/VR, τ=VR/F; X(F/VR)= μX, D= μ mamy stan ustalony. Ograniczeniem chemostatu jest μmax. FS0-FS-rsVR=0, F(S0-S)=rsVR; YXS=ΔX/|ΔS|= rx/rs, D(S0-S)= rx/YXS= μX/YXS, YXS=X/(S0-S)
Wprowadzamy równ. Monoda: μ=μmax(S/(KM+S); DS+DKS=μmaxS→S; D= μmax(S/(KS+S), S= DKS/μmax-D; X=YXS*(S0 (DKS/μmax-D)); Produktywność biomasy: Pr=DX, d(Pr)/dD=0; Dopt= μmax(1-![]()
), Xopt=YXS(S0+ KS-![]()
). Kinetyka wymywania: dx/dt=-DX+μX, D1>μmax, dx/dt=-D1X+μmaxX, dX/(Xdt)= -D1+μmax, d(lnX)/dt=-D1+μmax, lnX=(-D1+μmax)t; D(S0-S)=DX(1/YXS)+mX, (S0-S)/X=(1/YXS)+(m/D); Prawdziwy współcz. 1/YXS uwzględnia mechanizm endogenny (1/YXS)obserw=(1/YXS)+(m/D)
Bioreaktory z unieruchomionymi enzymami:
Zalety: wielokrotne wykorzystanie drogich enz., stabilny układ. Imobilizacja na nośnikach: porowatych(absorpcja), żelowych(inkluzja),płytkach szklanych Wady: dodatkowe opory, musimy uwzględnić dyfuzję, szybkość zmniejszona.
Efektywny wsp. Dyfuzji Def/D=1/10 Def=D(δ*ε/ζ) ε-porowatość ciała δ-przewężenie pora ζ-krętość
Dyfuzja zew. (do pow pellet):NA*S=(ks*as)(cA0-cAS) gdzie NA=strumień sk. A, as-pow. właściwa , (ks*as)- strumień dyfunduję z cieczy do c.s. (cA0+cA0)-siła napędowa procesu. W stanie ustalonym (pomijając dyfuzję wew.) gdy tylko strumień dotrze do powierzchni zachodzi reakcja. Strumień dyfundujący zew. Jest równy prędkości procesu. Dla kinetyki Menten: (ks*as)(cA0-cAS)= rmax(cAS/(KM+cAS)), ![]()
= cAS/ cA0, KM/ cA0=α, rmax/((ksas)*cA0)=DA-liczba DamKóhler'a, (cA0- cAS)/ cA0=| rmax/((ksas)*cA0)|*( cAS/ cA0)/ (KM+ cAS / cA0)
1-![]()
=DA*(![]()
/(α+![]()
)); DA- stosunek maksymalnej szybkości rekacji chemicznej do max. Szybkośi wnikania masy (gdy cAS=0).
α+![]()
- α![]()
-![]()
2=DA*![]()
; -α-![]()
+α![]()
+![]()
2+DA*![]()
=0; -α+![]()
*(-1+ α +DA)+![]()
2 =0 -otrzymujemy równanie kw. Które da się rozwiązać. 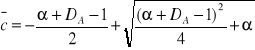
; gdy DA<<1 szybkośc bardzo mała w porównaniu z szybkościa dyzfuzji. Prbieg kontrolowany przez cA0~cAS. Reżim kinetyczny. Gdy DA>>1 szybkość reakcji duża w porównaniu z szybkością dyfuzji, proces limituje dyfuzja zew.
Czynnik efektywności ηz: stopień wykorzystania pow. zew. Katalizatora. Jest to stosunek obserwowanej szybkości do maksymalnej szybkości bioprocesu (tzn. bez wpływu dyfuzji zew.) ηz=rw/rmax rw-obserwowana rw= rmax(![]()
/(α+![]()
)), D<<1, ![]()
=1 ; r= rmax(1/(α+1)),
ηz=rw/rmax= (rmax(![]()
/(α+![]()
)))/( rmax(1/(α+1)))= (![]()
*( α +1))/( α +![]()
)
gdy ηz=1 to reakcja nie jest limitowana dyfuzją zew. D>>1, ![]()
=0
1= DA*(![]()
/(α+![]()
))1/DA=(![]()
/(α+![]()
)) ηz=(![]()
*( α +1))/( α +![]()
)=(1+α)/DA Rys.
Jeżeli liczba DA jest duża DA>>1 to stężenie substartu spada do 0 bo to co się dostaje do powierzchni od razu przeeraguje.
Dyfuzja wew. :o ile dyfuzja zew. i reakcja przechodzi w kolejnośći dyfuzjareakcja tak w dyfuzji wew. Reakcja i dyfuzja zachodzą jednocześnie. Trzeba skorzystać z II prawa Ficka: Def=(d2cA/dx2)=r, gdzie x- droga w głąb kat. Dla niedużych stężeń r=k*c, k=rmax/KM, r= rmax(c/(KM+c)); Wartośći brzegowe (układ jest symetryczny) x=0, stąd (dcA/dx)=0, x=L,
cA= cAS, -D*(dcA/dx)X-L=(ks*as)(cA0-cAS) pierwsze prawo Ficka; (d2cA/dx2)-(k1/Def)=0, c=A1eax+ A2e-ax. Moduł Thiele'go: D=L![]()
(dla r. I rzędu). Dla kinetyki Monoda: 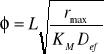
, ![]()
, ηw=(tgh(![]()
))![]()
Rys.
Dla małych pellets- obszar kinetyczny.
Mieszając szybko powodujemy szybsze wnikanie, nie musimy uwzględniać dyf. zew.
Możemy jeszcze rozwiązać uwzględniając warunki brzegowe , -D*(dcA/dx)=(ks*as)(cA0-cAS)
Wtedy otrzymujemy óogólniony współcznynnik dyfuzji 1/ηc=1/ηz+1/ηw
Wyszukiwarka
Podobne podstrony:
w2, studia, bio, 4rok, 7sem, inżynieria bioprocesowa i bioreaktorowa, bioprocesy (1 koło)
w5, studia, bio, 4rok, 7sem, inżynieria bioprocesowa i bioreaktorowa, bioprocesy (1 koło)
w4, studia, bio, 4rok, 7sem, inżynieria bioprocesowa i bioreaktorowa, bioprocesy (1 koło)
w6, studia, bio, 4rok, 7sem, inżynieria bioprocesowa i bioreaktorowa, bioprocesy (1 koło)
w2, studia, bio, 4rok, 7sem, inżynieria bioprocesowa i bioreaktorowa, bioprocesy (1 koło)
cz1, studia, bio, 4rok, 7sem, fakultet chemia żywności, wykład
koło lab, studia, bio, 4rok, 7sem, biotechnologia środowiska, lab, teoria
cz3, studia, bio, 4rok, 7sem, fakultet chemia żywności, wykład
cz4, studia, bio, 4rok, 7sem, fakultet chemia żywności, wykład
czesc-wykladow, studia, bio, 4rok, 7sem, biotechnologia środowiska, wykład
cz2, studia, bio, 4rok, 7sem, fakultet chemia żywności, wykład
spr-kofeina, studia, bio, 4rok, 7sem, fakultet chemia żywności, lab
inżynieria genetyczna, studia, bio, 4rok, 8sem, inzynieria genetyczna, lab
zaliczenie-pytania, studia, bio, 4rok, 7sem, procesy fermentacyjne, wykład
spr-olejki eteryczne, studia, bio, 4rok, 7sem, fakultet chemia żywności, lab
B II W03, studia, bio, 4rok, 8sem, biotechnologia2, wykład
mutagenizacja, studia, bio, 4rok, 8sem, biotechnologia2, lab
B II W12, studia, bio, 4rok, 8sem, biotechnologia2, wykład
więcej podobnych podstron