Spis treści
I) CZĘŚĆ TEORETYCZNA
1) Charakterystyka plazmidów i ich zastosowanie, jako klonujących wektorów.
Plazmidy są to autonomiczne pozachromosomalne elementy genetyczne występujące u wielu organizmów prokariotycznych i niektórych eukariotycznych. Występują w cytoplazmie, o wielkości najczęściej do 100 kbp (znane są również większe, tzw. megaplazmidy osiągające nawet 500 kbp). Występują one w wielu kopiach w komórce. Większość plazmidów zawiera region początku replikacji (ori), pozwalający im na niezależne namnażanie się w komórkach. Plazmidy występują zwykle w postaci superskręconej (CCC - DNA), mogą występować jako otwarta, kolista cząsteczka (OC - DNA) lub w postaci liniowej (L - DNA). Charakterystyczną ich cechą jest: fizyczna odrębność od chromosomu, zdolność do trwałego utrzymywania i replikowania się w komórce. Plazmid nie koduje funkcji, które byłyby konieczne do jej życia. Zwiększa natomiast różnorodność zajmowanych środowisk. Ilość genów zgromadzonych w plazmidach odgrywa znaczącą rolę w ewolucji i zdolności adaptacyjnych komórek, w których się znajdują.
Plazmidy zawierają zazwyczaj kilka genów, do których często zaliczamy geny oporności na antybiotyki. Klasyfikację plazmidów przeprowadzono w oparciu o to, jakie zawierają geny oraz o właściwości, jakie nadają komórkom gospodarza.
Typy plazmidów:
plazmidy typu R - zawierają geny nadające bakterii oporność na antybiotyki (np. ampicylina). Sposób uzyskania oporności jest różnorodny. Plazmidy typu R mogą przenosić między gatunkami bakterii oporność na antybiotyki. Powoduje to ogromne utrudnienia w leczeniu infekcji bakteryjnych,
plazmidy typu F - umożliwiają przenoszenie genów między komórkami bakteryjnymi w procesie zwanym koniugacją. Na plazmidzie F zlokalizowane są geny, które kierują przenoszeniem plazmidu typu F z jednej komórki do drugiej przy użyciu tubularnej struktury zwanej pilusem płciowym. Plazmid ten może zawierać dodatkowe geny, przejęte z chromosomu, które są przenoszone do komórki biorcy podczas koniugacji,
plazmidy coli cynowe - w tych plazmidach zlokalizowane są geny kodujące białka zwane koli cynami, które zabijają inne bakterie. Przykładem jest Col1E1 u E.coli,
plazmidy degradacyjne - kodują białka pozwalające komórce gospodarza metabolizować nietypowe związki, takie jak toluen czy kwas salicylowy,
plazmidy wirulencji - nadają bakteriom zdolność do wywołania chorób. Przykładem jest
plazmid Ti u Agrobacterium tumefaciens powodując chorobę roślin zwaną guzowatością korzeni.
Ze względu na sposób przenoszenia wyróżniamy plazmidy:
koniugacyjne,
niekoniugacyjne.
W plazmidach mogą być zakodowane różne cechy, nie są one jednak niezbędne dla życia komórek. Do cech fenotypowych zakodowanych w plazmidach należą:
fermentacja cukrów,
oporność na działanie metali ciężkich,
wytwarzanie enzymów restrykcyjnych i modyfikujących,
wytwarzanie enterotoksyn,
oporność na działanie antybiotyków,
produkcja antybiotyków,
degradacja związków organicznych.
Plazmidy jako wektory
Wektorem posługujemy się w przypadku gdy chcemy wprowadzić fragment obcego DNA do komórki biorcy, celem uzyskania replikacji i/lub ekspresji, unikając przy tym jego degradacji .
Wektor zapewnia powielanie wprowadzonego fragmentu DNA, czyli klonowanie a czasami także wydajną syntezę kodowanego przez gen białka (transkrypcję i translację oraz jego stabilność) jak to ma miejsce w tzw. wektorach ekspresyjnych. Najważniejszym elementem warunkującym specyficzność wektora są sekwencje odpowiedzialne za inicjację replikacji tzw. sekwencje ori. Najprostsze wektory posiadały wyłącznie jedno, unikalne miejsce restrykcyjne, w które można było wprowadzić obcy DNA. Obecnie najczęściej jest to tzw. polilinker - syntetyczny odcinek DNA, w którym znajduje się zwykle kilkanaście miejsc rozpoznawanych przez różne restryktazy.
Wektory zazwyczaj posiadają również geny markerowe ( geny kodujące białka odpowiedzialne za łatwo wyróżnialne cechy fenotypowe).
Wymogiem konstrukcji wektora jest możliwość selekcji tych komórek, do których został wprowadzony. Jego budowa powinna pozwalać także na odróżnienie wektora zrekombinowanego od takiego, który zamknął się bez ligacji z fragmentem DNA.
Sposób wprowadzania rekombinacyjnych cząstek DNA do komórek gospodarza jest ograniczone przez pojemność wektorów plazmidowych. Przy klasycznej transformacji wielkość wstawki nie powinna być większa niż około 10 kb.
Cechy dobrego wektora:
Mały rozmiar - im wektor jest mniejszy tym większy fragment DNA można w nim umieścić. Wprowadzenie większych fragmentów DNA stanowi dla komórki gospodarza znaczne obciążenie.
Silny promotor - umożliwiający ekspresję na wysokim poziomie białka, którego gen znajduje się na tym wektorze.
Duża liczba kopii - uzależniona jest od rodzaju ori. Im więcej kopii, tym jest więcej matryc dla transkrypcji.
Pojedyncze miejsca restrykcyjne - najprostsze wektory posiadały wyłącznie jedno miejsce restrykcyjne, w które można było wprowadzić obcy DNA. Obecnie najczęściej jest to tzw. polilinker - syntetyczny odcinek DNA, w którym znajduje się zwykle kilkanaście miejsc rozpoznawanych przez różne restryktazy.
Posiadanie markerów selekcyjnych - sekwencje pozwalające odróżnić bakterie posiadające plazmid od tych, które go nie zawierają. Jako markery najczęściej stosuje się geny oporności na antybiotyki (np. tetracyklinę lub ampicylinę).
Posiadanie sekwencji umożliwiających autonomiczną replikację (niezależną od chromosomu bakteryjnego)-region ori.
Najczęściej stosowanym typem wektora do klonowania są plazmidy. Proces ten przeprowadzamy w kilku etapach. Najpierw plazmid trawi się odpowiednim enzymem restrykcyjnym, który przecina go w jednym miejscu. Zmienia się przy tym jego forma z kolistej na liniową z lepkimi końcami. Obcy DNA, który chcemy wklonować poddaje się trawieniu enzymem restrykcyjnym, generującym takie same lepkie końce. Zrekombinowane koliste plazmidy otrzymujemy w wyniku zmieszania plazmidu z obcym DNA (ich cząsteczki łączą się poprzez lepkie końce). Aby związać je kowalencyjnie używa się ligazy DNA. Zrekombinowany plazmid wprowadza się do gospodarza w procesie transformacji.
2) Etapy preparatyki plazmidowego DNA.
Do oczyszczania plazmidowego DNA zastosowaliśmy metodę lizy alkalicznej. Metoda ta wykorzystuje różnice pomiędzy DNA plazmidowym, a genomowym bakterii. DNA genomowy (chromosomalny) występuje u bakterii w postaci pojedynczej, kolistej cząsteczki. DNA plazmidowy jest to natomiast kolista, dwuniciowa cząsteczka w formie CCC, czyli kowalencyjnie zamknięta.
Etapy izolowania kwasów nukleinowych:
Namnożenie komórek (otrzymanie biomasy),
Uwolnienie treści komórkowej na zewnątrz,
Oczyszczenie - oddzielenie kwasów nukleinowych od innych składników komórki,
Zatężanie kwasów nukleinowych do odpowiedniego stężenia roboczego.
W preparatyce plazmidowego DNA zastosowano bakterie E.coli.
1. Pierwszy wysiew bakterii przeprowadzono na stałym podłożu LB z dodatkiem antybiotyków (ampicylina lub tetracyklina), w celu wyselekcjonowania bakterii odpornych na dany antybiotyk. Następnie zaszczepiono płynne podłoże LB (bez agaru) z dodatkiem odpowiedniego antybiotyku komórkami pobranymi z pojedynczej kolonii. Kolejny krok to całonocna inkubacja zaszczepionego płynnego podłoża na wytrząsarce w temp. 37°C. Hodowlę komórek prowadzono do momentu osiągnięcia końca logarytmicznej fazy wzrostu.
2. Otrzymaną hodowlę komórek odwirowano w celu oddzielenia ich od podłoża, a następnie zawieszono w buforze TGE, zawierającym:
Tris-HCl - utrzymuje stałe pH (związek buforujący); dzięki niemu komórki łatwo ulegają rozproszeniu, nie tworzą skupisk,
glukozę - zachowuje właściwe ciśnienie osmotyczne (komórki zostają umieszczone w roztworze izotonicznym),
EDTA - kompleksuje jony dwuwartościowe (Mg2+,Ca2+); stosowany w celu zablokowania aktywności enzymatycznej nukleaz i zmniejszenia stabilności ściany komórkowej bakterii,
pH=8,0.
Kolejno przeprowadzono wirowanie. Następnie usunięto supernatant i zawieszono komórki w buforze TGE. Do tak przygotowanych komórek dodano alkalicznego roztworu SDS- siarczan dodecylu sodu, w celu uwolnienia treści komórkowej. W wyniku tego mieszanina stała się klarowna, o dużej lepkości (w zasadowym środowisku, przy udziale SDS dochodzi do lizy komórek bakteryjnych, tworzą się pory i zawartość komórki wylewa się na zewnątrz. Denaturacja genomowego i plazmidowego DNA zachodzi pod wpływem zasadowego pH, w którym następuje rozerwanie wiązań wodorowych. Tworzą się liniowe fragmenty DNA).
3. Do mieszaniny reakcyjnej dodano octanu potasu o pH 4,8, który spowodował zobojętnienie środowiska (pH 6-7). Nastąpiło obniżenie pH i wytrącił się osad, w którym są agregaty częściowo już denaturowanego genomowego DNA oraz niewielka ilość SDS.
Zawartość probówki mieszano początkowo łagodnie do wystąpienia zwartego osadu, a następnie dokładnie wymieszano w celu zupełnego wytrącenia bakteryjnego genomowego DNA, białek, fragmentów błon i większości SDS-u. Całość następnie odwirowano. Plazmidowy DNA zawarty w supernatancie łatwo zdenaturował po zobojętnieniu środowiska. Oprócz plazmidowego DNA supernatant zawierał RNA i trochę białek. Klarowny supernatant przeniesiono do nowych probówek i ekstrahowano jednokrotnie z równą objętością mieszaniny fenol/chloroform. Mieszanina ta jest czynnikiem denaturującym białka.
Kolejno odwirowano próbę w wyniku czego powstały dwie warstwy: dolna -organiczna i górna - wodna (z kwasami nukleinowymi). Fazę wodną przeniesiono z dwóch probówek do nowej probówki typu eppendorf i dodano izopropanolu, w którym rozpuszcza się reszta fenolu oraz wytrąca plazmidowy DNA. Kolejny raz próbę poddano wirowaniu, po czym usunięto supernatant (zawierający alkohol) i rozpuszczono osad w octanie sodu. Roztwór stał się klarowny i bezbarwny. W wyniku dodania chlorku sodu i zimnego 96% etanolu wytrąca się plazmidowy DNA w postaci koloidu. Etanol stosowany jest w niskiej temperaturze, aby nie denaturował kwasów nukleinowych. Próbę kolejno odwirowano i dokładnie usunięto supernatant w celu otrzymania osadu zawierającego kwasy nukleinowe. Osad wysuszono w suszarce próżniowej (w czasie trwania tego etapu resztki etanolu odparowują) i rozpuszczono go w buforze TE - służy do przechowywania kwasów nukleinowych. Aby z próby pozbyć się RNA dodano RNA-azę wyizolowaną z trzuski wołowej. Następnie przeprowadzono wirowanie w celu zadziałania enzymu w całej objętości zawiesiny. Wykorzystywany termostabilny enzym nie narusza DNA, hydrolizuje natomiast wiązania fosfodiestrowe w RNA. Celem pozbycia się RNA-zy z próby dodano mieszaninę fenol/chloroform, a następnie odwirowano i przeniesiono górną fazę do nowej probówki. Dodano chlorku sodu, a następnie 96% etanolu. Krótkie odcinki DNA pozostają w roztworze, a fragmenty DNA pozbawione otoczki solwatacyjnej zaczynają tworzyć agregaty i wypadają z roztworu. Próbę odwirowano i usunięto supernatant. Powstały osad kwasów nukleinowych przemyto etanolem, odwirowano, wysuszono i zawieszono plazmidowy DNA w buforze TE.
3) Enzymy restrykcyjne.
Enzymy restrykcyjne (endonukleazy restrykcyjne) są enzymami tnącymi wiązania fosfodiestrowe w łańcuchu DNA. Izolowane są głownie z bakterii i sinic, rzadziej z promieniowców. Ich biologiczna rola polega na rozcinaniu cząstek obcego DNA. Są one niezbędne w badaniu struktury chromosomów, sekwencjonowaniu bardzo długich cząsteczek DNA nadających się do klonowania. System ten składa się z dwóch elementów: pierwszym jest endonukleaza restrykcyjna, która rozpoznaje krótką, symetryczną sekwencję DNA i przecina (hydrolizuje) szkielet każdej z nici DNA w specyficznym miejscu w obrębie tej sekwencji. Drugim elementem systemu jest metylaza, która dodaje grupę metylową do zasady C lub A w obrębie tej samej sekwencji komórkowego DNA. Ta modyfikacja sprawia, że DNA gospodarza staje się odporny na degradację. Komórkowy DNA nie ulega degradacji przez własne enzymy restrykcyjne, ponieważ miejsca przez nie rozpoznawane są w tym DNA zmetylowane.
3.1 Podział enzymów restrykcyjnych:
Enzymy klasy I - przecinają cząsteczkę DNA od wewnątrz, nie rozpoznając określonych sekwencji. Nie posiadają zatem zdefiniowanych miejsc cięcia i są mało specyficzne. Oprócz jonów Mg2+ wymagają do działania S-adenozynometioniny (SAM) oraz ATP.
Enzymy klasy II - są zdolne do rozpoznawania specyficznych sekwencji w DNA i przecinania dwuniciowej cząsteczki DNA w dwóch miejscach w obrębie tej sekwencji albo w dokładnie zdefiniowanym miejscu w pewnej odległości od niej. Do swego działania wymagają jonów Mg2+. Mają największe znaczenie praktyczne.
Enzymy klasy III - rozpoznają określone sekwencje w DNA, ale miejsce cięcia nie jest dokładnie zdefiniowane. Wymagają ATP i jonów Mg2+ do działania.
W inżynierii genetycznej powszechnie są stosowane enzymy klasy drugiej. Enzymy pochodzące z różnych szczepów, ale rozpoznające te same sekwencje DNA, nazywają się izoschizomerami. Sekwencje cztero- lub sześcionukleotydowe rozpoznawane przez te enzymy mają najczęściej charakter sekwencji palindromowych (posiadającą oś symetrii), tzn. że jeśli są odczytywane z każdej nici w kierunku np. 5'→3', to są identyczne.
Enzymy restrykcyjne mogą rozcinać DNA na dwa sposoby, pozostawiając dwa rodzaje końców:
Tępe końce - powstają w wyniku nacięcia obu komplementarnych nici w tym samym miejscu.
5' -GGATATCC- 3'
3' -CCTATAGG- 5'
5' -GGAT-OH + P-ATCC- 3'
3' -CCTA-P + OH-TAGG- 5'
Lepkie końce - są efektem nacięcia nici DNA w dwóch różnych miejscach oddalonych od siebie o kilka nukleotydów. Rozcięte w ten sposób nici DNA mają na końcach krótkie 1-niciowe fragmenty.
5' -GGATATCC- 3'
3' -CCTATAGG- 5'
5' -G-OH + P-GATATCC- 3'
3' -CCTATAG-P + OH-G- 5'
Wszystkie enzymy restrykcyjne pozostawiają grupę fosforanową na końcu 5', a grupę hydroksylową na końcu 3'. "Lepkie końce" mają duże znaczenie przy klonowaniu genów: sekwencje "lepkich końców" powstałych w wyniku działania tego samego enzymu są komplementarne. W obecności enzymu ligazy można takie cząsteczki ponownie połączyć ze sobą kowalencyjnie.
Inkubację preparatów DNA z enzymami restrykcyjnymi prowadzi się w temperaturze 37oC w czasie 1 godziny lub dłużej. Przerwanie reakcji (zwykle niekonieczne) można przeprowadzić przez termiczną inaktywację enzymu (15 minut, 65oC) lub dodając EDTA pH 8.0 do końcowego stężenia 10 mM (chelatacja jonów magnezu). Następnie do próby dodawany jest barwnik i próbkę nanosi się na żel agarozowy.
Fragmenty DNA powstałe w wyniku trawienia enzymami restrykcyjnymi dają się rozdzielić na żelach w taki sposób, że w każdym prążku na żelu znajdują się cząsteczki DNA o identycznej sekwencji nukleotydowej. Fakt ten pozwala na precyzyjną obróbkę DNA, m.in. konstruowanie map restrykcyjnych badanego DNA (mapa restrykcyjna wskazuje miejsca cięte przez enzymy restrykcyjne). Analizując na żelach produkty trawienia danego DNA różnymi enzymami restrykcyjnymi, stosowanymi pojedynczo i w kombinacjach, można ustalić wzajemne położenie i odległości pomiędzy sekwencjami rozpoznawanymi przez te enzymy. Oprócz tego, DNA pocięty enzymami restrykcyjnymi można poddawać ligacji
z wektorem i tworzyć zrekombinowane (zawierające sklonowany DNA) plazmidy.
4) Elektroforeza i czynniki wpływające na jej przebieg.
Elektroforeza jest to technika stosowana przy rozdziale, analizie i oczyszczaniu kwasów nukleinowych i białek. DNA i RNA mają ładunek ujemny i zgodnie z tym ładunkiem, nadawanym przez grupy fosforanowe, migrują w polu elektrycznym w kierunku anody. Szybkość migracji zależy od wielkości i kształtu cząsteczki.
4.1 Schemat postępowania.
Żel ulega zestaleniu w formie cienkiej płytki na podstawce tzw. saneczkach wraz z zanurzonym specjalnego typu grzebieniem, który następnie wyciąga się. W zastygłym żelu znajduje się szereg studzienek, do których wprowadzane są próbki DNA oraz z reguły tzw. standard, czyli mieszaninę cząsteczek o znanych masach, dzięki czemu możliwa będzie późniejsza kalibracja żelu. Przed naniesieniem na żel, próbki muszą być odpowiednio przygotowane: dodawany jest barwnik oraz związek interkalujący - bromek etydyny, który fluoryzuje w świetle UV co pozwala na zlokalizowanie prążków. Minimalna ilość DNA, którą widać na żelu w postaci prążka to 20ng. Ilość DNA w prążku nie powinna przekraczać 200ng. Żel umieszczony zostaje w specjalnym aparacie do elektroforezy i zalany buforem przewodzącym prąd. Podłączony zostaje do zasilacza generującego prąd o stałym i określonym napięciu i natężeniu, optymalna wartość to napięcie nie większe niż 5V na 1cm długości żelu.
4.2 Żele używane do elektroforezy:
Żele agarozowe
Powszechnie stosowane. Zdolność rozdzielcza zależy od wielkości porów w żelu, których rozmiary są zdeterminowane przez stężenie agarozy. Żeli bardziej stężonych, tj. 1,4-2,0 %, używa się do rozdziału cząsteczek mniejszych (0,5-2,0 kb). Żele mniej stężone 0,5-0,7 % stosuje się do rozdziału cząsteczek większych (10-20 kb i więcej). Zaletą może być niekiedy fakt, że podczas elektroforezy możliwe jest rozdzielenie cząsteczek o tej samej wielkości, ale zróżnicowanych pod względem kształtu. Doskonałym przykładem jest rozdział trzech różnych form plazmidu: CCC (ang. Covalently closed circle), liniowej i OC (ang. open circle). Pierwsza najszybciej wędruje w żelu, druga nieco wolniej, trzecia najwolniej.
Żele akrylamidowe
Powstają przez polimeryzacje monomerów akrylamidu w długie łańcuchy z wytworzeniem wiązań poprzecznych przez bisakrylamid. Można regulować sieciowanie poprzez manipulację proporcjami stężeń akrylamidu do bisakrylamidu. Do zajścia reakcji niezbędna jest obecność odpowiedniego katalizatora (PERS - nadsiarczan potasowy) i związku inicjującego reakcję (TEMED). Żele te stosuje się przy rozdziale fragmentów o wielkości od kilku par zasad (20% poliakrylamid) do tysiąca par zasad (3% poliakrylamid) a także przy rozdziale jednoniciowych cząsteczek DNA, np. do sekwencjonowania.
Żele denaturujące
Ponieważ szybkość migracji jednoniciowych cząsteczek w żelu, zależy nie tylko od ich wielkości i ładunku, ale także w dużym stopniu od ich struktury przestrzennej. Aby dokładnie określić ich wielkość należy wyeliminować różnice w szybkości migracji wywołane różną konformacją cząsteczki. Jest to szczególnie ważne w przypadku jednoniciowego RNA, który tworzy miejscowe struktury dwuniciowe. W tym właśnie celu stosuje się żele denaturujące agarozowe lub poliakrylamidowe. Najczęściej używanymi czynnikami denaturującymi są: formaldehyd i glioksal w żelach agarozowych (przy analizie preparatów RNA) lub mocznik w żelach poliakrylamidowych (przy analizie jednoniciowych fragmentów DNA).
4.3 Elektroforeza w polu pulsacyjnym.
Stosowana do rozdzielenia dużych cząsteczek DNA - od 2.104 do 107 bp, czyli od 20 kb do 10 Mb. Można w ten sposób rozdzielić całe chromosomy np. drożdżowe. Pole elektryczne jest włączane i wyłączane w krótkich odstępach czasu. Kiedy pole elektryczne jest włączone cząsteczki migrują zgodnie ze swoją wielkością a gdy zostaje wyłączone mają tendencję do relaksacji i zwijania w przypadkowe pętle. Czas wymagany do relaksacji jest wprost proporcjonalny do długości cząsteczki. Następnie kierunek pola elektrycznego jest zmieniany o 90 lub 180 stopni w stosunku do poprzedniego. Dłuższe cząsteczki zaczynają poruszać się wolniej niż krótsze. Powtarzające się zmiany kierunku pola stopniowo powodują rozdzielenie.
Czynniki wpływające na przebieg elektroforezy:
wielkość cząsteczek DNA - cząsteczki liniowego dwuniciowego DNA migrują przez pory żelu z szybkością odwrotnie proporcjonalną do logarytmu dziesiętnego z ilości par zasad. Większe cząsteczki migrują wolniej z powodu większych sił tarcia oraz mniej efektywnego przechodzenia przez pory w żelu.
Szybkość, z jaką przesuwa się w żelu cząsteczka DNA jest odwrotnie proporcjonalna do jej masy cząsteczkowej. Umieszczenie w niektórych studzienkach fragmentów DNA o znanej wielkości pozwala na dokładny pomiar masy cząsteczkowej badanych fragmentów.
stężenie agarozy - przy niższych stężeniach agarozy formowane są pory o większych średnicach, co pozwala na rozdział większych fragmentów. Zależność szybkości migracji od stężenia agarowy opisuje równanie:
log μ = log μ0 - Kr ⋅ τ
gdzie:
μ- szybkość migracji,
μ0 - szybkość swobodnej migracji DNA,
Kr - stała zależna od własności żelu oraz wielkości i kształtu migrującej molekuły,
τ - stężenie agarozy.
Stosowanie różnych stężeń żelu pozwala na uzyskanie optymalnego rozdziału fragmentów o różnej wielkości.
konformacja DNA - forma superskręcona (CCC), zrelaksowana(OC) i liniowa(L) o tej samej masie migrują w żelu z różną prędkością. Względna ruchliwość tych trzech form zależy głównie od stężenia agarozy w żelu, wartości przyłożonego napięcia, siły jonowej buforu i gęstości formy superskręconej. W pewnych warunkach forma CCC migruje szybciej niż forma liniowa a w innych odwrotnie. Aby ustalić konformację DNA można przeprowadzić elektroforezę w obecności zwiększających się ilości bromku etydyny. Gdy stężenie bromku etydyny wzrasta coraz więcej barwnika interkaluje do DNA. Ujemne (prawostronne) skręty superhelisy w formie CCC są stopniowo usuwane, fluorescencja cząsteczek wzrasta a szybkość migracji maleje. Przy krytycznym stężeniu barwnika, gdy nie ma już żadnych form superskręconych, szybkość migracji formy CCC osiąga minimalną wartość. Ponieważ bromek etydyny jest wciąż włączany powstają dodatnie skręty superhelisy, cząsteczki DNA stają się bardziej zwarte i ich ruchliwość gwałtownie wzrasta. Równocześnie ruchliwość formy zrelaksowanej i liniowej zmniejsza się w sposób zróżnicowany z powodu neutralizacji ładunku i większej sztywności uzyskanej na skutek obecności bromku etydyny.
przyłożone napięcie - przy niskim napięciu szybkość migracji liniowych fragmentów DNA jest proporcjonalna do przyłożonego napięcia. Efektywny rozdział fragmentów DNA w żelu agarozowym maleje, jeżeli zwiększa się przyłożone napięcie. Dla uzyskania maksymalnego rozdziału fragmentów większych niż 2000bp przez żel powinien przepływać prąd nie większy niż 5V/cm. Przy większej wartości napięcia rychliwość nie wzrasta już tak proporcjonalnie.
kierunek pola elektrycznego - cząsteczki DNA większe niż 50 - 100 kbp migrują przez żel agarozowy z tą samą prędkością, jeśli kierunek pola elektrycznego pozostaje stały. Kiedy kierunek pola zmienia się okresowo cząsteczki DNA są zmuszone do zmiany kierunku przemieszczania się. Ponieważ większym cząsteczkom DNA potrzeba dłuższego czasu, aby ustawić się zgodnie z nowym kierunkiem pola, elektroforeza w pulsującym polu elektrycznym może być używana do frakcjonowania ekstremalnie dużych cząsteczek DNA (do 10000kbp)
temperatura - w żelach agarozowych względna ruchliwość fragmentów różnej wielkości nie zmienia się w temperaturze 4°C - 30°C. Elektroforezę prowadzi się zwykle w temperaturze pokojowej. Jednakże dla żeli zawierających mniej niż 0,5% agarozy i żeli o niskiej temperaturze topnienia najlepiej stosować temperaturę 4°C.
obecność interkalujących barwników - bromek etydyny będący barwnikiem fluorescencyjnym jest używany do wykrywania DNA w żelach agarozowych i poliakrylamidowych. Obniża on elektroforetyczną ruchliwość liniowego DNA o około 15%. Barwnik interkaluje pomiędzy sąsiednie pary zasad, zwiększając długość form liniowej i zrelaksowanej i czyniąc je bardziej sztywnymi. W zależności od budowy DNA różna ilość barwnika interkaluje więcej EtBr włącza się do cząsteczek liniowych a mniej do formy CCC.
skład buforów do elektroforezy - na ruchliwość DNA wpływa ich skład i siła jonowa. W nieobecności jonów, (jeżeli bufor nie został dodany do żelu) przewodnictwo elektryczne jest minimalne, a DNA migruje bardzo powoli lub wcale. W buforze w wysokiej sile jonowej przewodnictwo jest bardzo wysokie i wydzielane są znaczne ilości ciepła.
W takich warunkach może dojść do stopienia się żelu i denaturowania DNA.
Na elektroforezę wpływa także rodzaj żelu: dla fragmentów DNA powyżej 500bp stosuje się żele poliakrylamidowe, natomiast dla mniejszych cząsteczek - żele agarozowe.
5) Metody transformacji.
Transformacja bakteryjna polega na zmianie genomu komórkowego na skutek pobrania cząsteczek DNA z pożywki. Metodę tę zastosowano przy pierwszych udanych próbach wprowadzenia obcego genu do bakterii. Właśnie to odkrycie stanowiło dowód, że DNA jest nośnikiem informacji genetycznej. W praktyce, gdy populacja komórek z pewnym defektem genetycznym (na przykład niezdolnością do syntezy aminokwasu tryptofanu, metabolizowania cukru, laktozy czy też syntezy jednej z cząsteczek występujących na powierzchni komórki) są wystawiane na działanie DNA wydzielonego z komórek, mogących wykonywać tę funkcję, niektóre z komórek uzyskują zdolności charakterystyczne dla dawcy DNA. Transformacja taka jest trwała i dziedziczona przez komórki potomne, ponieważ nowo nabyty funkcjonalny gen staje się częścią genomowego DNA komórek biorcy. Transformacja bakterii okazała się niemal nieprzydatna w początkowych fazach badań nad genetyką Prokaryota dla kilku przyczyn, które są w tym miejscu mało istotne. Obecnie proces transformacji nabrał wielkiego znaczenia i jest kluczowym etapem wielu doświadczeń ze zrekombinowanym DNA. Termin „transformacja” stosuje się powszechnie dla określenia trwałej zmiany zapisu genetycznego komórki i jego wpływu na cechy komórki tak u Prokaryota, jak i Eukaryota.
5.1 Metody transformacji:
Metoda Chung'a i Miller'a - zaletą tej metody jest możliwość wcześniejszego przygotowania kompetentnych komórek, czyli zdolnych do przyjęcia egzogennego DNA i przechowywanie w dłuższym okresie czasu w głębokim zamrożeniu. W metodzie tej stosujemy glikol etylenowy (PEG 10% o stopniu polimeryzacji 3350, a czasami 6000 lub 8000), DMSO (sulfotlenek dimetylu), jony Mn2+ zawarte w buforze TSB oraz glukozę jako źródło węgla. Aby uniknąć szoku termicznego wszystkie czynności wykonujemy w lodzie. Ostatnim etapem jest przeszczepienie transformantów do świeżego podłożą i inkubacja w 37°C.
Chang'a i Cohen'a - bakterie hodowane są do środkowej fazy logarytmicznego wzrostu, po czym odwirowane komórki zawiesza się w roztworze sacharozy z lizozymem. Po inkubacji powstają koliste formy - protoplasty, które są zabezpieczone przed pęknięciem dzięki obecności sacharozy. Następnie wprowadza się obcy DNA w określonej ilości, po czym zawiesinę przenosi się do glikolu polietylenowego PEG 6000. Przy braku zewnętrznej osłony obcy DNA może bez przeszkód wniknąć do środka. Po inkubacji protoplasty są rozsiewane na podłożą regeneracyjne, na których następuje odbudowanie osłon komórkowych, a następnie na podłoża selekcyjne.
Metoda Mandel'a i Higa - badacze ci stwierdzili, że aby wywołać indukcję kompetencji należy inkubować w probówce komórki E. coli w roztworze chlorku wapnia we łaźni lodowej, po czym dodać roztwór plazmidowego DNA.
50 molowy CaCl2 powoduje rozluźnienie struktur komórkowych. Następnie przenosi się probówkę z łaźni lodowej do łaźni o temp. 42°C (3min) wywołując szok termiczny. W wyniku szoku najprawdopodobniej dzięki rozluźnieniu struktury osłon komórkowych następuje wniknięcie obcego DNA do wnętrza. Przeprowadzamy inkubacje w 37°C w środowisku bez antybiotyku (zaczynają działać nowe cechy wprowadzone z DNA plazmidowego). Rozsiewamy na stałe podłoże z dodatkiem antybiotyku i otrzymujemy transformanty. Inkubacja z CaCl2 i szok termiczny sprawiają, że następuje rozluźnienie osłon komórkowych. Zjawisko to jest nazywane przejściem w stan kompetencji. W wyniku tej metody osiągamy wydajność transformacji 106 jtk (cfu) z 1 μg DNA. Hanahan zmodyfikował tą metodę dodając oprócz CaCl2, także jony magnezowe i manganowe, co zwiększyło wydajność do 108jtk/ μg DNA.Metoda Anagnostopulos'a - polega na otrzymaniu kompetentnych komórek na drodze dwuetapowej hodowli na podłożach ubogich w składniki odżywcze. Powoduje to, że komórki nie wytwarzają do końca składników ściany komórkowej, co zwiększa ich przepuszczalność.
Wydajność transformacji jest to ilość kolonii bakteryjnych otrzymanych, z g DNA użytego do transformacji. Wydajność wyrażana jest w cfu/ μg DNA.
II) CZĘŚĆ DOŚWIADCZALNA
1) Przygotowanie hodowli bakterii do preparatyki plazmidowego DNA.
1. Bakterie E. coli zawierające plazmidowy DNA przechowywane w temperaturze -70°C w glicerolu wysiano na stałe podłoże LB o składzie:
Bactotryptone (Difco) 1,0%
Ekstrakt drożdżowy (Difco) 0,5%
NaCl 0,5%
Agar (Difco) 1,5%
z dodatkiem: ampicyliny 50 μg/ml lub tetracykliny 15 μg/ml
Kolejno pojedyncze kolonie posłużyły do zaszczepienia 10 ml płynnego podłoża LB
(bez agaru) z dodatkiem wyznaczonej uprzednio objętości odpowiedniego antybiotyku (stężenia antybiotyków jak w podłożu stałym).
Podstawowe roztwory antybiotyków:
Ampicylina 50 mg/ml (roztwór wykonany w sterylnej wodzie destylowanej, przechowywany w -20ºC)
Tetracyklina 10 mg/ml (roztwór wykonany bezpośrednio przed użyciem w 96 % etanolu)
Wyznaczenie ilości antybiotyków dodanych do 60 ml podłoża LB:
Ampicylina: Tetracyklina:
50 μg - 1 ml 15 μg - 1 ml
x μg - 60 ml x μg - 60 ml
x = 3000 μg x = 900 μg
50 mg = 50 000 μg 10 mg = 10 000 μg
50 000 μg - 1 ml 10 000 μg - 1 ml
3000 μg - y ml 900 μg - y ml
y = 0,06 ml = 60 μl y = 0,09 ml = 90 μl
W przeprowadzonym przez nas doświadczeniu materiałem biologicznym, który wykorzystywałyśmy były komórki Escherichia coli, u których stwierdzono występowanie następujących plazmidów :
pAT 153 - oporność na działanie ampicyliny,
pBR 322 - oporność na działanie ampicyliny i tetracykliny,
pACYC 184 - oporność na działanie tetracykliny,
pUC 19 - oporność na działanie ampicyliny;
Ampicylina- antybiotyk β-laktamowy z grupy aminopenicylin, charakteryzuje się szerokim spektrum działania, powoduje zakłócenie syntezy ściany komórkowej, hamuje działanie licznych enzymów błony cytoplazmatycznej. Inaktywacja antybiotyku związana jest z rozszczepieniem wiązania β-laktamowego czego dokonuje β-laktamaza kodowana przez gen oporności bla.
Tetracyklina - antybiotyk tetracyklinowy syntetyzowany przez niektóre szczepy Streptomyces. Jest to związek bakteriostatyczny, hamuje biosyntezę białka, poprzez wiązanie się
z podjednostka 30S rybosomu, a także wpływa na procesy fosforylacyjne w komórkach bakteryjnych. Oporność na ten antybiotyk warunkuje gen tet kodujący białko powodujące modyfikujące membrany bakteryjnej, dzięki czemu antybiotyk nie jest transportowany do wnętrza komórki.
2. Po całonocnej inkubacji hodowli na wytrząsarce w temperaturze 37 °C, komórki bakteryjne odwirowano przy 9000 rpm wciągu 5 minut i zawieszono w 200 μl buforu TGE o składzie:
25 mM Tris-HCl
50 mM glukoza
10 mM EDTA
pH = 8.0
Tak przygotowaną zawiesinę przeniesiono do probówki typu eppendorf i ponownie odwirowano komórki przy 12000 rpm przez 30 s.
Następuje rozbicie skupisk komórek, oraz wyrównanie ciśnienia osmotycznego, za co odpowiedzialna jest glukoza ( działanie ochronne). EDTA tworzący kompleksy z jonami Mg²+ uniemożliwia działanie nukleaz, których aktywność zależna jest od obecności w środowisku tych jonów.
3. Próby, po usunięciu supernatantu i zawieszeniu komórek w 200 μl buforu TGE, poddano inkubacji w łaźni lodowej przez 5 minut. Kolejno dodano 400 μl świeżo przygotowanego alkalicznego roztworu SDS ( siarczan dodecylosodu), który jako środek powierzchniowo czynny powoduje niszczenie struktury ściany komórkowej oraz denaturacje białka a tym samym uwolnienie organelli komórkowych, w tym kwasów nukleinowych. Uzyskany w ten sposób roztwór staje się klarowny i charakteryzuje się zwiększoną lepkością. W takich warunkach
( pH = 12) liniowe fragmenty DNA ulegają denaturacji, w przeciwieństwie do kolistych, które są stabilizowane dzięki obecności wiązań wodorowych.
Alkaliczny roztwór SDS-u odpowiednio rozcieńczono, aby otrzymać 6 ml roztworu.
Stężenie SDS-u wynosi 10% - mamy otrzymać 0,6 ml 1% SDS-u.
10% SDS - 0,6 ml
1% SDS - x ⇒ x = 0,06 ml = 60 μl SDS
Stężenie NaOH 2M - mamy otrzymać 0,6 ml 0,2 M NaOH
2 M NaOH - 0,6 ml
0,2 M NaOH - x ⇒ x = 0,06 ml = 60μl NaOH
Objętość dodanej wody wynosi: 6000 μl - 60 μl (SDS) - 60 μl (NaOH) = 480 μl (H2O)
4. Po inkubacji w lodzie (5 minut) do probówki wprowadzono 300 µl 3M octanu potasu o pH 4,8. Kolejno jej zawartość poddano łagodnemu mieszaniu do wystąpienia zwartego osadu, po czym zastosowano intensywne wytrząsanie, aby umożliwić całkowite wytrącenie genomowego DNA oraz polimerów. Liniowe fragmenty DNA ulegają agregacji czego konsekwencją jest ich wytrącanie się w formie osadu. W roztworze pozostaje kolisty DNA oraz kwas rybonukleinowy.
5. Po kolejnej inkubacji w lodzie (5 minut) i odwirowaniu przy 12000 rpm (2 minuty) klarowny supernatant przeniesiono do 2 nowych probówek w równych ilościach (ok. 450 μl). W celu odbiałczenia preparatu przeprowadzono ekstrakcję równą objętością mieszaniny fenol/chloroform. Górna fazę stanowią kwasy nukleinowe ( faza wodna), natomiast dolną - białka (faza organiczna).
6. Po odwirowaniu przy 12000 rpm (1 minuta) przeniesiono fazę wodną z dwóch probówek do jednej nowej i wprowadzono 500 μl alkoholu izopropylowego wspomagającego odbiałczanie i rozdzielenie faz. Następnie wymieszano zawartość probówki, inkubowano w temperaturze pokojowej przez 5 min, po czym ponownie odwirowano przy 13200 rpm przez 5 minut.
7. Następnie po dokładnym usunięciu supernatantu rozpuszczono osad w 100 μl 0,1 M octanu sodu, który wspomaga wytrącanie kwasów nukleinowych w kolejnym etapie.
8. Wprowadzono 10 μl 3 M chlorku sodu oraz 250 μl zimnego (- 20 °C) 96% etanolu,
powoduje on usuniecie otoczki hydratacyjnej, w konsekwencji koagulację kwasów nukleinowych i wytrącenie osadu.
9. Po 20 min inkubacji w temperaturze - 20 °C ponownie wirowano próbę (13200 rpm ,
5 minut), po usunięciu supernatantu przemyto osad kwasów nukleinowych wykorzystując do tego celu 300 μl 96 % etanolu i wirowano jak wyżej.
10. Kolejno, w celu dokładnego usunięcia etanolu suszono osad w suszarce próżniowej przez 20 minut.
11. Wysuszony osad rozpuszczono w 100 µl buforu TE o składzie:
10 mM Tris-HCl,
1 mM EDTA,
pH= 8.0,
który zapobiega fragmentacji plazmidowego DNA.
Roztwór podstawowy TE rozcieńczono 100-krotnie (do 990µl wody wprowadzamy 10 μl
podstawowego roztworu buforu TE).
12. Wprowadzono RNA-zę w ilości 100 µg/ml (1µl na ściankę) hydrolizującą kwas rybonukleinowy na krótsze odcinki, dzięki czemu w kolejnym etapie, nie będą ulegały wytrąceniu w wyniku działania etanolu.
Roztwór podstawowy RNA-zy o stężeniu 10 mg/ml (10000 μg/1000μl) rozcieńczono 100-krotnie.
100 μg - 1 ml = 1000 μl
x μg - 100 μl
x = 10 μg
10 000 μg - 1000 μl
10 μg - y μl
y = 1 μl
Zatem w celu uzyskania 100 μl RNA-zy o stężeniu 100 μg/ml należało pobrać 1 μl RNA-zy
i 99 μl wody.
13. Po 30 minutach inkubacji w temperaturze 37 ºC ekstrahowano z równą objętością mieszaniny fenol/chloroform i odwirowano (12000 rpm, 1 minuta). Górną warstwę układu przeniesiono do czystej probówki eppendorf (zmierzono objętość) i dodano 1/10 objętości 3 M chlorku sodu i 2,5-krotną objętość 96% etanolu. Zawartość probówek wymieszano i pozostawiono w temperaturze - 20ºC przez 30 minut. Powstały osad zawiera wyłącznie plazmidowy DNA. Ponownie przeprowadzono wirowanie, przemywanie osadu 96% etanolem i suszenie w suszarce próżniowej (punkty 9-10 sprawozdania) .
14. Preparat plazmidowego DNA rozpuszczono w 30 μl buforu TE.
2) Przygotowanie żelu i buforu do elektroforezy.
2.1 Przygotowanie buforu do elektroforezy.
W celu przygotowania buforu do elektroforezy rozcieńczono 20-krotnie roztwór podstawowy o składzie:
0,89 M Tris, 0,89 M kwas borowy, 0,02 M EDTA
całkowita objętość buforu: 500 ml
objętość roztworu podstawowego: 500: 20 = 25 ml
objętość wody: 475 ml
Do rozcieńczonego buforu dodano bromek etydyny (EtBr), związek ten umożliwia uwidocznienie na żelu kwasów nukleinowych. Interkaluje między wiązania wodorowe między zasadami w DNA i wykazuje fluorescencję pod wpływem promieniowania UV.
wyjściowe stężenie: 10 mg/ml
końcowe stężenie: 0,5μg/ml
10 mg - 1 ml
0,5 × 10 -3 mg - x ml
x = 5 × 10 -5 ml
objętość 5 × 10 -5 ml odpowiada 1 ml żelu natomiast objętość żelu pobrana do elektroforezy wynosi 500 ml, w związku z tym:
dodana objętość bromku etydyny: 500 × 5 × 10 -5 ml = 0,0025 ml = 25 μl.
2.2 Obliczenie ilości agarozy.
Kolejno przygotowano żel agarozowy:
zakładane stężenie żelu: 0,7 %
objętość buforu: 30 ml
0,7g - 100 ml
x - 30 ml
x = 0,21 g agarozy
1. Odważone 0,21 g agarozy wprowadzono do kolby łącznie z 30 ml buforu, po czym poddano inkubacji we wrzącej łaźni wodnej przez 10 minut.
2. Po przestudzeniu do temperatury ok. 45°C wylano do aparatu instalując następnie grzebyk kształtujący studzienki.
3. Następnie po zestaleniu żelu usunięto grzebyk i tacę wraz z żelem umieszczono w aparacie do elektroforezy zalewając go buforem.
4. Z probówki zawierającej plazmidowy DNA pobrano 1 μl i rozcieńczono 10-krotnie dodając 9 μl buforu TE.
5. Do probówek z 10-krotnym rozcieńczeniem wprowadzono po 1,6 μl roztworu barwnika (0,25% błękit bromofenolowy, 40% sacharoza). Związek ten migruje w żelu z szybkością odpowiadającą cząsteczce DNA o długości 300 bp dając możliwość śledzenia postępu elektroforezy.
6. Elektroforezę prowadzono przy napięciu 5 V/cm (w naszym przypadku było to maksimum 75V/15cm) do momentu osiągnięcia końca żelu przez barwnik, po czym żel przeniesiono do aparatu i obserwowano fluorescencję DNA wobec światła ultrafioletowego.
2.3 Absorbancja.
Wyizolowane plazmidowe DNA poddano pomiarowi absorbancji przy długości fali 260 nm.
Próba pobrana do analizy została 100-krotnie rozcieńczona buforem TE.
Skład próby:
1 μl plazmidowego DNA
99 μl buforu TE
Uzyskane wyniki przedstawiono w tabeli 1.
Tabela 1. Zestawienie wyników absorbancji i stężeń DNA
Plazmid |
Numer próby |
Absorbancja pojedynczej próby |
Stężenie DNA plazmidowego |
|
|
|
[gDNA/l] |
pAT 153 |
1 |
0.035 |
3.5 |
|
2 |
0.023 |
2.3 |
|
3 |
0.041 |
4.1 |
pBR 322 |
4 |
0.113 |
11.3 |
|
5 |
0.204 |
20.4 |
|
6 |
- |
- |
|
7 |
- |
- |
pUC 19 |
8 |
0.047 |
4.7 |
|
9 |
0.055 |
5.5 |
|
10 |
0.092 |
9.2 |
|
11 |
- |
- |
pACYC 184 |
12 |
0.071 |
7.1 |
|
13 |
0.123 |
12.3 |
Przykładowe wyznaczenie stężenia plazmidowego DNA na podstawie absorbancji plazmidu pUC19 (numer próby 10):
przy parametrach standardowej kuwety:
A260 stężenie DNA
1 - 50 μg/ml
0,092 - x μg/ml
x = 4,6 μg/ml
po uwzględnieniu parametrów wykonania ćwiczenia oraz 100-krotnego rozcieńczenia próby:
x × R × K = 4,6 x 100 x 20 =9200 μg/ml = 9,2 μg/μl
gdzie:
właściwe stężenie próby [μg/ml],
x - stężenie próby [μg/ml],
R - rozcieńczenie,
K - przeliczenie drogi światła.
Rysunek 1.
1 2 3 4 5 8 9 10 12 13
Legenda:
pAT 153: 1 - Magdalena H.
2 - Monika K.
3 - Anna P.
pBR322: 4 - Karolina K.
5 - Justyna M.
pUC19: 8 - Maria G.
9 - Katarzyna K.
10 - Katarzyna S.
pACYC184 : 12 - Maciej K.
13 - Anna C.
Analiza wyników elektroforezy.
Podczas elektroforezy w żelu agarozowym plazmidowy DNA rozdziela się na prążki charakterystyczne dla różnych form plazmidu.
Prążek usytuowany najniżej charakterystyczny jest dla plazmidu w formie kolistej super skręconej DNA (CCC), wyizolowanej z komórki w stanie nienaruszonym. Taka forma DNA plazmidowego charakteryzuje się najwyższą ruchliwością wynikającą z jej zwartej struktury. Plazmid o zwartej konformacji migruje najszybciej w żelu agarozowym, co można obserwować podczas elektroforezy.
Wolniej migruje plazmid w formie OC (zrelaksowanej), powstały z formy superhelikalnej po pęknięciu jednej z nici. W wyniku rozluźnienia konformacji ruchliwość plazmidu ulega zmianie - porusza się on wyraźnie wolniej podczas elektroforezy w żelu agarozowym, co uwidacznia się położeniem prążka na żelu powyżej prążka charakterystycznego dla formy superskręconej.
Prążek położony najwyżej na żelu charakteryzuje się najmniejszą ruchliwością. Zawiera on cząsteczki liniowego DNA lub zanieczyszczenia preparatu. Mieszanina ta migruje najwolniej podczas rozdziału elektroforetycznego.
Po przeprowadzonej elektroforezie pewne jest iż najszybciej wędrujący prążek odnosi się do struktury CCC. A kolejności dwóch pierwszym form nie jesteśmy w stanie określić. Będzie to pewne po przeprowadzeniu trawienia enzymami restrykcyjnymi.
Każdy z plazmidów podczas elektroforezy w żelu agarozowym pokonuje drogę o długości charakterystycznej dla jego wielkości. Plazmidy typu pUC, jako najmniejsze, migrowały w żelu najszybciej. Prążki przedstawiające położenia tych plazmidów znajdowały się najniżej w żelu. Plazmidy typu pAT pokonały krótszą drogę, stąd można wywnioskować, że są większe od plazmidów pUC. W przypadku plazmidów pBR obserwujemy nieco krótszą drogę migracji niż w przypadku plazmidów typu pAT. Droga migracji plazmidów pACYC jest najkrótsza. Zapewne jest to spowodowane tworzeniem przez nie multimerów, przez co wędrują one o wiele wolniej niż w przypadku pojedynczych plazmidów. Położenie takich prążków wskazuje na wiele większe rozmiary niż w rzeczywistości. Cecha ta utrudnia wyznaczenie ich wielkości podczas rozdziału w żelu agarozowym.
Wzrost fluorescencji próby jest spowodowany wysokim stężeniem DNA w próbie oraz dzięki bromkowi etydyny, który interkaluje pomiędzy pary zasad cząsteczek dwuniciowego DNA. Wzrost wartości absorbancji dla każdego z preparatów powinien być proporcjonalny do intensywności fluorescencji na żelu. W naszym przypadku preparaty były zanieczyszczone genomowym DNA dlatego nie jest widoczna korelacja pomiędzy stężeniem plazmidowego DNA a intensywnością świecenia w żelu.
3) Trawienie plazmidowego DNA enzymami restrykcyjnymi.
Wypreparowane w pierwszym etapie doświadczenia plazmidowe DNA poddano trawieniu enzymami restrykcyjnymi. Skład przygotowanych mieszanin restrykcyjnych:
|
Pojedyncze trawienie |
Podwójne trawienie |
Roztwór DNA |
1 µl |
2 µl |
Bufor restrykcyjny |
1 µl |
2 µl |
Woda destylowana |
7 µl |
14 µl |
Enzym restrykcyjny |
1 µl |
2 µl |
Podwójne trawienie przeprowadzono w objętości 20 μl, co jest konieczne aby osiągnąć stężenie glicerolu 5 %. Zbyt duże stężenie powoduje zmianę aktywności enzymu.
Wykonaliśmy następujące trawienia plazmidów:
pAT153 (3,7 kbp) |
pACYC184 (4,2 kbp) |
pBR322 (4,4 kbp) |
|||
Enzym |
Bufor |
Enzym |
Bufor |
Enzym |
Bufor |
Eco RI |
Eco RI |
Eco RI |
Eco RI |
Eco RI + Eco 88I |
TangoTMx1,5 |
Bam HI |
Bam HI |
Eco RI + Sal I |
0 (orange) |
Eco RI |
Eco RI |
Hind III |
R (red) |
Eco RI + Hind III |
TangoTMx2 |
Eco RI + Sal I |
0 (orange) |
Eco RI + Bam HI |
TangoTMx2 |
Hind III |
R (red) |
Eco RI + Hind III |
TangoTMx2 |
Eco RI + Sal I |
0 (orange) |
|
|
Eco RI + Bam HI |
TangoTMx2 |
Eco RI + Eco 88I |
TangoTMx1,5 |
|
|
|
|
Po inkubacji probówek w temperaturze 37°C w czasie 1 godziny dodano roztwór barwinka odpowiednio dla trawienia pojedynczego (1,6 µl) i podwójnego (3,2 µl), wymieszano i wprowadzono do studzienek w żelu do elektroforezy.
Rysunek 2. Eksperyment.
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 M
Rysunek 3. Wzorzec.
M 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 M
Legenda:
1- pAT nietrawiony 11- pBR322 Eco RI + Sal I
2- pAT153 Eco RI 12- pBR322 Eco RI + Hind III
3- pAT153 Bam HI 13- pBR322 Eco RI + Bam HI
4- pAT153 Hind III 14- pACYC184 nietrawiony
5- pAT153 Eco RI + Bam HI 15- pACYC184 Eco RI
6- pAT153 Eco RI + Sal I 16 - pACYC184 Eco RI + Sal I
7- pAT153 Eco RI + Eco 88I 17- pACYC184 Eco RI + Hind III
8 -pBR322 nietrawiony 18 - pACYC184 Hind III
9- pBR322 Eco RI + Eco 88I M - markery wielkości ( bakteriofag λ )
10-pBR322 Eco RI
Analiza wyników elektroforezy:
Na podstawie otrzymanego obrazu elektroforezy możemy stwierdzić, że trawienia zaszły. Ostatnia linia migrującego preparatu jest wykrzywiona najprawdopodobniej z powodu zmian natężenia prądu lub nierównomiernie rozłożonego żelu. Obserwujemy również rozmazanie prążków, spowodowane najprawdopodobniej zanieczyszczeniem genomowym DNA.
Ścieżka 1 przedstawia wynik elektroforezy nietrawionego plazmidu pAT153 obserwujemy tutaj 3 prążki, pierwszy z nich przedstawia pozostałości genomowego DNA stanowiącego zanieczyszczenie próbki, drugi przedstawia DNA w konformacji kolistej zrelaksowanej. Ostatni prążek odpowiada plazmidowemu DNA.
Na ścieżkach 2, 3, 4 obserwujemy prążki (po jednym na ścieżce) powstałe w wyniku pojedynczego trawienia enzymami: Eco RI, Bam HI, Hind III. Oznacza to, że każdy z nich ma tylko jedno miejsce rozpoznania i po przecięciu w miejscu restrykcyjnym postaje zlinearyzowane DNA. Wszystkie powstałe fragmenty są podobnej wielkości. Widoczne smugi powstały w wyniku niecałkowitego strawienia DNA genomowego. Są one mniej widoczne na ścieżkach 5 i 6 ( podwójne trawienia). Cząsteczka kolistego DNA została całkowicie strawiona.
Na ścieżce 5 wyraźnie widoczny jest prążek dłuższego fragmentu, natomiast drugi prążek jest niewidoczny gdyż fragment przeciętego DNA był tak krótki, że wyszedł z żelu.
Ścieżki 6 i 7 przedstawiają 2 prążki z podwójnych trawień. Drugi prążek jest widoczny ponieważ odległość między miejscami restrykcyjnymi jest większa.
Kolejna ścieżka odpowiada nie trawionemu pBR 322. Plazmid ten jest największy spośród analizowanych. Najdalej położony prążek obrazuje super skręconą konformacje plazmidu. Zapewne istnieje analogia pomiędzy nim a pAT-em. Jeden z prążków pBR jest widoczny na tej samej wysokości co prążek pAT. Dlatego możemy podejrzewać, że pAT powstaje z pBR.
Na ścieżce 9 i 11 widać dwa prążki - podwójne trawienia, natomiast na 10 jeden - pojedyncze trawienie. W przypadku ścieżek 12 i 13 (podwójne trawienia) widoczny jest tylko jeden prążek, ponieważ miejsca restrykcyjne użytych do trawień enzymów są blisko siebie położone odległość pomiędzy miejscami restrykcyjnymi jest bardzo mała i widoczny jest tylko dłuższy fragment ponieważ krótszy nie został zatrzymany w żelu.
Ostatnie prążki na ścieżkach 7 i 9 potwierdzają, że pAT powstał w wyniku usunięcia fragmentu plazmidu pBR.
Ścieżka 14 przedstawia wynik elektroforezy nie trawionego pACYCA. Widzimy kilka prążków, które mogą świadczyć o zdolności do multimeryzacji, co powoduje wolniejszą migrację w żelu.
Na ścieżce 15 widać jeden prążek, który przedstawia zmultimeryzowaną liniową formę DNA dzięki czemu szybciej migruje w żelu niż w przypadku niemultimeryzującego plazmidu.
Na ścieżce 16 widać 2 nakładające się prążki (podwójne trawienia), co świadczy o tym, że miejsca restrykcyjne trawiących enzymów leżą prawie naprzeciw siebie (naprzeciwlegle).
Na ścieżce 17 widzimy 2 pojedyncze prążki - podwójne trawienie, każdy enzym ma jedno miejsce rozpoznania. Natomiast na ścieżce 18 widoczny jest jeden prążek - pojedyncze trawienie.
Tabela 2. Dane dotyczące faga λ po trawieniu enzymem Hind III.
Liczba par zasad [bp] |
Log par zasad |
Droga [mm] |
23130 |
4,364 |
15 |
9416 |
3,974 |
22 |
6557 |
3,817 |
28 |
4361 |
3,64 |
37 |
2322 |
3,366 |
54 |
2027 |
3,307 |
58 |
564 |
2,751 |
94 |
125 |
2,097 |
- |
Powyższe dane pozwalają na skonstruowanie krzywej wzorcowej przedstawiającej zależność miedzy logarytmem z długości par zasad otrzymanych w wyniku trawienia bakteriofaga λ enzymem restrykcyjnym Hind III, a dystansem migracji fragmentów w żelu.
Rysunek 4. Krzywa wzorcowa.
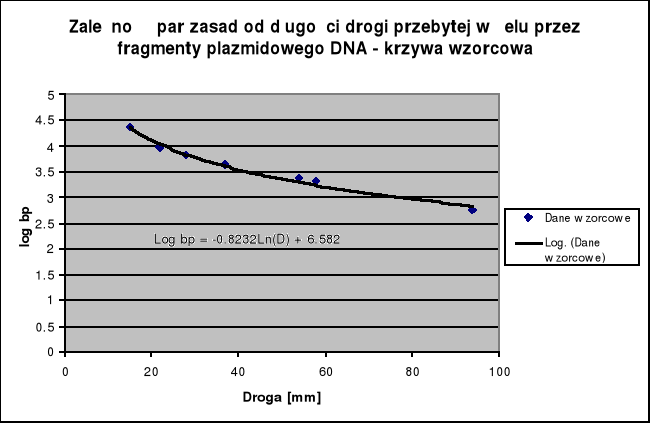
Korzystając z równania krzywej wzorcowej można obliczyć długości fragmentów plazmidowego DNA, które powstały po trawieniu.
Tabela 3. Długości fragmentów restrykcyjnych dla poszczególnych plazmidów:
Plazmid |
Numer ścieżki |
Trawienie |
Droga w żelu [mm] |
Log10pb |
Długość fragmentów pb na podstawie wykresu |
pAT153 |
2 |
Eco RI |
41 |
3.525 |
3349.56 |
|
3 |
Bam HI |
41 |
3.525 |
3349.56 |
|
4 |
Hind III |
41 |
3.525 |
3349.56 |
|
5 |
Eco RI + Bam HI |
44 |
3.467 |
2929.92 |
|
|
|
- |
- |
- |
|
6 |
Eco RI + Sal I |
46 |
3.430 |
2693.16 |
|
|
|
87 |
2.906 |
804.76 |
|
7 |
Eco RI + Eco 88I |
56 |
3.268 |
1854.94 |
|
|
|
69 |
3.096 |
1248.77 |
pBR322 |
9 |
Eco RI + Eco 88I |
47 |
3.413 |
2585.58 |
|
|
|
69 |
3.096 |
1248.77 |
|
10 |
Eco RI |
37 |
3.609 |
4069.04 |
|
11 |
Eco RI + Sal I |
10 |
4.687 |
48586.09 |
|
|
|
87 |
2.906 |
804.76 |
|
12 |
Eco RI + Hind III |
36 |
3.632 |
4285.95 |
|
|
|
- |
- |
- |
|
13 |
Eco RI + Bam HI |
38 |
3.588 |
3868.47 |
|
|
|
- |
- |
- |
pACYC |
15 |
Eco RI |
37 |
3.609 |
4069.04 |
|
16 |
Eco RI + Sal I |
54 |
3.298 |
1987.32 |
|
|
|
56 |
3.268 |
1854.94 |
|
17 |
Eco RI + Hind III |
49 |
3.378 |
2389.21 |
|
|
|
66 |
3.133 |
1358.55 |
|
18 |
Hind III |
37 |
3.609 |
4069.04 |
4) Przygotowanie komórek kompetentnych Escherichia coli.
Bakterie E. Coli (przechowywane w -70°C w glicerolu) wysiano na stałą pożywkę LB i następnie jedną kolonią zaszczepiono 10 ml pożywki LB - hodowla wgłębna.
Po całonocnej inkubacji na wstrząsarce (w temperaturze 37°C) przeszczepiono 0,5 ml hodowli do 50 ml podłoża LB (pH=6,1), następnie inkubowano na wstrząsarce w temperaturze 37°C do osiągnięcia absorbancji OD600 = 0,4- 0,6 (w naszym przypadku inkubacja trwała 2 godziny do osiągnięcia OD600=0,476).
Odwirowano hodowlę (5000 rpm, 10 minut, temperatura 4°C) i po usunięciu supernatantu zawieszono komórki w 1,9 ml zimnego sterylnego roztworu TSB o składzie:
Bactotryptone (Difco) 1gEkstrakt drożdżowy (Difco) 0,5g
NaCl 0,5g
Polietylenowy glikol (PEG, MW = 8000) 10g
- zwiększa lepkość, powoduje powstawanie małych kryształków lodu, chroni przy zamrażaniu
Woda destylowana 93ml
Regulacja pH 6,11M MgSO4 1ml
1M MgCl2 1ml
Mieszaninę umieszczono w łaźni lodowej i dodano 100 μl sulfotlenku dimetylu (DMSO) co ma na celu utrwalenie komórek.
Po 10 min inkubacji mieszaninę rozdzielono na porcje po 100 μl i zamrożono w łaźni z suchym lodem i przechowywano w temperaturze -72°C, co zapobiega powstawaniu zbyt dużych kryształów lodu, które mogą rozerwać komórkę.
5) Transformacja komórek kompetentnych E. coli.
1. Przygotowano odpowiednio 5μl preparatu plazmidowego i 45 μl buforu TE w celu uzyskania 10-krotnego rozcieńczenia, z którego pobrano 10μl preparatu plazmidowego i dodano 90 μl TE uzyskując 100-krotne rozcieńczenie.
2. Do 5 probówek zawierających rozmrożone kompetentne komórki dodano 10 μl zimnego plazmidowego DNA rozcieńczonego 10-cio i 100-krotnie w buforze TE (odpowiednio po 2 probówki i jedną kontrolną do której dodano 10 μl TE). Próbówki inkubowano w lodzie ok.15 minut.
3. Z przygotowanych 100-krotnych rozcieńczeń (pkt. 2) wykonano pomiar absorbancji przy długości fali 260 nm (wyniki w tabeli zbiorczej rys. 7).
4. W sterylnych warunkach dodano 850 μl roztworu TSB, 50 μl DMSO (powoduje perforacje błony komórkowej co pomaga wniknąć plazmidowemu DNA do komórki) i 20 μl roztworu glukozy (stabilizacja ciśnienia osmotycznego), a następnie inkubowano w temperaturze pokojowej 15 minut, przeniesiono na wytrząsarkę i inkubowano w temperaturze 37°C przez godzinę. Po zakończeniu tego etapu plazmidowy DNA powinien znaleźć się w komórce E. coli.
Przygotowano podłoża ( po 150 ml) zawierające odpowiednie stężenia antybiotyków.
Ampicylina: Tetracyklina:
roztwór wyjściowy: 50 mg/ml roztwór wyjściowy: 10 mg/ml
stężenie w pożywce: 50 μg/ml stężenie w pożywce: 15 μg/ml
50 μg - 1 ml 15 μg - 1 ml
x μg - 150 ml x μg - 150 ml
x = 7500 μg x = 2250 μg
50 000 μg - 1 ml 10 000 μg - 1 ml
7500 μg - y ml 2250 μg - y ml
y = 0,15 ml = 150 μl y = 0,225 ml = 225 μl
6. Na podłoża wysiano po 50 μl transformowanych komórek, a następnie zaszczepione płytki pozostawiono na 30 minut w komorze szczepień.
7. Po całonocnej inkubacji w temperaturze 30°C w termostacie, policzono kolonie transformantów (wyniki zebrane w tabeli - rys. 7).
5.1 Obliczenie stężenia DNA [μg DNA/μl] ( dla plazmidu pUC).
Z prawa Lamberta - Beera wiadomo, że: dla absorbancji A260 = 1 stężenie DNA wynosi 50 μg /ml dla drogi światła 1 cm.
A260= 0,114
1 - 50 μg DNA/ml
0,114 - x μg DNA/ml
x = 5,7 μg DNA/ml
Po uwzględnieniu rozcieńczenia do pomiaru absorbancji i przeliczenia drogi światła, która wynosiła 0,5 mm otrzymujemy:
x × R × K = 5,7 μg DNA/ml ×100 × 20 = 11400 μg DNA/ml = 11,4 μg DNA/μl
Uwzględnienie rozcieńczenia prób:
R = 10
11,4 μg DNA/μl / 10 = 1,14 μg DNA/μl
R = 100
11,4 μg DNA/μl / 100 = 0,114 μg DNA/μl
Wysiano 50 μl zawiesiny o składzie:
100 μl - zawiesina komórek
10 μl - DNA
- 859 μl - TSB
- 50 μl - DMSO
- 20 μl - glukoza
suma: 1030 μl
Obliczono, że DNA wprowadzone do zawiesiny ( 1030 μl ),
R = 10
1,14 μg - 1 μl
x - 10 μl
x = 11,4 μg
R = 100
0,114 μg - 1 μl
x - 10 μl
x = 1,14 μg
Obliczono ile DNA wprowadzono w 50 μl zawiesiny:
R = 10
11,4 μg - 1030 μl
x - 50 μl
x = 0,55 μg
R = 100
1,14 μg - 1030 μl
x - 50 μl
x = 0,055 μg
Ilość wyrosłych kolonii transformantów:
R = 10 - 1607 ; 1725
R = 100 - 623 ; 747
Obliczona średnia ilość kolonii:
R = 10 ( 1607 + 1725 ) / 2 = 1666 cfu
R = 100 ( 623 + 747 ) / 2 = 685 cfu
5.2 Obliczenie wydajności transformacji.
R = 10
0,55 μg - 1666 cfu
1 μg - x
x = 3029 cfu/ μg
R = 100
0,055 μg - 685 cfu
1 μg - x
x = 12455 cfu/ μg
Tabela 4. Dane dla wszystkich plazmidów i wyniki wydajności transformacji.
Plazmid |
Rozcieńczenie |
Ilość kolonii [cfu] |
Średnia ilość kolonii |
Absorbancja przed transformacją |
Stężenie DNA [µg/µl] |
Wydajność transformacji [cfu/µg DNA] |
pAT |
10 |
992; 1512 |
1252 |
0,007 |
0,07 |
36845 |
|
100 |
18; 2 |
10 |
|
0,007 |
2943 |
pBR |
10 |
212; 647 |
430 |
0,115 |
1,15 |
771 |
|
100 |
163; 92 |
128 |
|
0,115 |
2293 |
pUC |
10 |
1607; 1725 |
1666 |
0,114 |
1,14 |
3029 |
|
100 |
623; 747 |
685 |
|
0,114 |
12455 |
pACYC |
10 |
37; 39 |
38 |
0,106 |
1,06 |
74 |
|
100 |
7; 13 |
10 |
|
0,106 |
194 |
Wnioski:
Osiągnięta wydajność praktyczna znacznie odbiega od wydajności teoretycznej, która dla tej metody transformacji ( Millera- Chunga) powinna mieścić się w zakresie 107 - 109 jtk/μg DNA. Tak duża rozbieżność spowodowana jest niedoświadczeniem osób wykonujących preparatykę. Do czynników, które głównie wpłynęły na błędy można zaliczyć brak doświadczenia w pracy z bardzo małymi ilościami DNA, a co za tym idzie błędy w rozcieńczeniach, niedokładność w izolowaniu DNA, nieprawidłowa technikę pipetowania, a także zniszczenie kolonii poprzez użycie w trakcie posiewów zbyt gorącego gładzika i ezy. O niedokładnościach tych świadczyć mogą między innymi rozbieżności w ilości wyrosłych kolonii w obrębie tego samego rozcieńczenia.
Wydajność transformacji zależy bezpośrednio od ilości kopii plazmidu na komórkę - im większa jest ilość kopii tym większa wydajność. Teoretycznie liczba kopii plazmidu, a zatem i wydajność powinna wzrastać w kierunku pACYC184, pBR322, pAT153, i pUC19. W naszym przypadku wydajność transformacji pACYC jest najmniejsza ,nastepną w kolejności jest wydajność dla pBR co potwierdza teoretyczną kolejność. Jednak w przypadku wydajności dla transformacji pozostałych plazmidów , dla pAT jest ona wyższa niż w przypadku pUC.
6) Preparatyka plazmidowego DNA z otrzymanych transformantów.
Kwasy nukleinowe izolowano z transformantów w podobny sposób jak na początku ćwiczeń, a następnie rozpuszczono je w 30 μl buforu TE. Różnica w postępowaniu polega na tym, że materiał nie został poddany działaniu RNA-zy. W celu stwierdzenia, czy kompetentne komórki przyjęły plazmid, wykonano elektroforezę prób przed i po transformacji.
Tabela 5. Wyniki pomiaru absorbancji.
Plazmid |
|
pAT |
pBR |
pUC |
pACYC |
Komórki przed transformacją |
Absorbancja |
0,007 |
0,115 |
0,114 |
0,106 |
|
Stężenie DNA [μg DNA/μl] |
0,007 |
0,115 |
0,114 |
0,106 |
Komórki po transformacji |
Absorbancja |
0,335 |
0,273 |
0,177 |
0,285 |
|
Stężenie DNA [μg DNA/μl] |
0,335 |
0,273 |
0,177 |
0,285 |
Rysunek 5. Wyniki elektroforezy.
1 2 3 4 5 6 7 8
Legenda:
1- pAT 153 przed transformacją,
2 - pAT 153 po transformacji
3 - pBR 322 przed transformacją
4 - pBR 322 po transformacji
5 - pUC 19 przed transformacją
6 - pUC 19 po transformacji
7 - pACYC 184 przed transformacją
8 - pACYC 184 po transformacji.
Wnioski:
Na ścieżkach z plazmidami po transformacji widoczne są mocno fluoryzujące prążki, charakterystyczne dla RNA, które są wynikiem nie dodania w traktacie preparatyki RNA-az, (odpowiedzialne za usuwanie kwasu rybonukleinowego). Mimo, że RNA jest jednoniciowy to możemy obserwować go w żelu agarozowym, ponieważ posiada on komplementarne fragmenty tworzące struktury dwuniciowe. Bromek etydyny interkaluje między nie tak jak między dwuniciowe kwasy, dając tym samym obraz widoczny na żelu.
Na podstawie obrazu elektroforezy przed i po transformacji można zauważyć, że droga migracji prążków jest zbliżona. Jednak w przypadku plazmidów po transformacji, DNA migruje minimalnie szybciej, ponieważ plazmidy te nie zostały poddane RNA-zie i RNA wędrujące przed DNA rozpycha pory żelu.
Absorbancje preparatów po transformacji są wyższe niż przed, co świadczy o dodatkowej obecności w próbce RNA.
7) Ustalenie mapy restrykcyjnej.
Opis warunków elektroforezy:
Elektroforeza przy stałym napięciu w żelu agarozowym
Stężenie: 0,7%
Napięcie: 4,0 V/cm
Długość żelu 15 cm
Bufor TBA>
Numer ścieżki |
Używany enzym |
Ilość fragmentów |
Rozmiar fragmentów |
1 |
SPP1-ECORI Marker wielkości |
15 |
8000 |
|
|
|
7100 |
|
|
|
6000 |
|
|
|
4800 |
|
|
|
3500 |
|
|
|
2700 |
|
|
|
1900 |
|
|
|
1850 |
|
|
|
1500 |
|
|
|
1400 |
|
|
|
1150 |
|
|
|
1000 |
|
|
|
680 |
|
|
|
490 |
|
|
|
370 |
2 |
BamHI/Clal |
3 |
4145 |
|
|
|
2261 |
|
|
|
818 |
3 |
BamHI/Smal |
3 |
4 145 |
|
|
|
3076 |
|
|
|
3 |
4 |
Clal/Smal |
2 |
4966 |
|
|
|
2258 |
5 |
Pstl/Smal |
3 |
5178 |
|
|
|
2027 |
|
|
|
19 |
6 |
Pstl/Clal |
3 |
2920 |
|
|
|
2277 |
|
|
|
2027 |
7 |
Aval/Clal |
4 |
4968 |
|
|
|
1443 |
|
|
|
514 |
|
|
|
299 |
8 |
Aval/Smal |
4 |
5480 |
|
|
|
1443 |
|
|
|
299 |
|
|
|
2 |
9 |
Aval |
3 |
5482 |
|
|
|
1443 |
|
|
|
299 |
10 |
Aval/Xmal |
3 |
5482 |
|
|
|
1443 |
|
|
|
299 |
ROZWIĄZANIE: Mapa restrykcyjna.
Na podstawie trawień nr 2 i 3 ustaliłyśmy miejsca restrykcyjne dla enzymów BamHI oraz Clal:
Następnie na podstawie trawień nr 3 i 4 ustaliłyśmy miejsca restrykcyjne dla enzymów Smal i Clal:
Po analizie trawienia nr 5 i 6 ustaliłyśmy miejsca trawienia enzymu Pst oraz Smal.
Trawienie nr 7 i 8 sugeruje nam dwa rozwiązania:
a.)
b.)
Nie możemy ustalić, która mapa restrykcyjna jest poprawna. Aby to ustalić można by poddać plazmid niepełnemu trawieniu enzymami np Aval/Clal, Aval/Smal bądź zastosować inny enzym trawiący na fragmencie 1443 lub 299.
Nie możemy ustalić miejsc restrykcyjnych dla Xmal ponieważ pokrywają się z miejscami restrykcyjnymi Awal. Aby je ustalić można by plazmid poddać trawieniu samym enzymem Xmal.
III) LITERATURA
J. M. Berg, J. L. Tymoczko, L. Stryer; Biochemia ; PWN , 2007;
Red. naukowy P. Węgleński; Genetyka molekularna ; PWN, 2008;
T. A. Brown; Genomy ; PWN , 2001;
W. Gajewski, P. Węgleński; Inżynieria genetyczna ; PWN, 1986.
Strona 20 z 38
Jony Mg2+ zwiększają kompetencję komórek
Składniki pożywki
23130bp
9416bp
6557bp
4361bp
2322bp
2027bp
564bp
Wyszukiwarka