6. Kwasy nukleinowe
Wiadomości wstępne
Budowa
Kwasy nukleinowe są liniowymi polimerami podstawowych jednostek zwanych nukleotydami. Pojedynczy nukleotyd składa się z części powtarzalnej, którą tworzą cząsteczka pentozy (ryboza lub dezoksyryboza) i reszta kwasu fosforowego oraz części specyficznej, którą tworzy cząsteczka zasady azotowej - pochodnej puryny (adenina, guanina) lub pirymidyny (cytozyna, tymina lub uracyl). Stosownie do rodzaju pentozy rozróżniamy kwasy rybonukleinowe - RNA i kwasy dezoksyrybonukleinowe - DNA. W DNA występują zasady: adenina, tymina, cytozyna i guanina, natomiast w RNA zamiast tyminy występuje uracyl. Cząsteczka kwasu nukleinowego powstaje w ten sposób, że tworzy się wiązanie estrowe pomiędzy grupą -OH pentozy jednego nukleotydu i grupą fosforanową nukleotydu sąsiedniego. Specyficzna dla każdego nukleotydu zasada azotowa nie uczestniczy więc w powstawaniu cząsteczki kwasu nukleinowego (por. budowę aminokwasów i powstawanie białek przez łączenie się aminokwasów).
Struktura DNA
DNA stanowi podstawowy magazyn informacji genetycznej w organizmie. Informacja ta zakodowana jest w kolejności czyli sekwencji nukleotydów, a dokładniej w sekwencji zasad azotowych w kolejnych nukleotydach, którą analogicznie jak w białkach, nazywamy strukturą I rzędu. W komórkach eukariotycznych DNA zlokalizowany jest w jądrze komórkowym oraz w mniejszych ilościach w mitochondriach i chloroplastach, zaś w komórkach prokariotycznych (nie posiadających jądra komórkowego) mieści się w centralnej części komórki. Strukturę II rzędu DNA stanowi, podobnie jak w białkach, struktura α, czyli prawoskrętna helisa polinukleotydów, przy czym w odróżnieniu od białka helisa DNA składa się z dwóch nici. Nici te zwinięte są tak, że ich zewnętrzną powierzchnię stanowią grupy fosforowo-cukrowe, zaś do środka helisy wystają prostopadle do jej osi - zasady azotowe. Obydwie nici wiążą się ze sobą przy pomocy wiązań wodorowych wytwarzanych pomiędzy leżącymi naprzeciwko siebie zasadami azotowymi. Wiązania te mogą powstać tylko wtedy, gdy zasady te nie leżą zbyt blisko ani zbyt daleko od siebie. Ten warunek jest spełniony tylko wtedy, gdy naprzeciwko zasady większej (purynowej) znajduje się zasada mniejsza (pirymidynowa) oraz, gdy naprzeciwko zasady azotowej z grupą C=O (z atomem tlenu z wolnymi parami elektronów) znajduje się zasada posiadająca w tym miejscu grupę aminową bogatą w atomy wodoru. Z tych dwu warunków wynika tzw. zasada komplementarności, tj. wymogu, aby naprzeciwko siebie w obydwu niciach występowały tylko dwa rodzaje par zasad azotowych: para AT (adenina - tymina) i para GC (guanina - cytozyna) (Rys. 6.1). Z reguły tej wynika, że struktura I rzędu jednej nici DNA (tj. sekwencja nukleotydów) określa w pełni strukturę drugiej nici. Jedna z tych nici jest nazwana nicią kodującą (sensowną, 5'→3'), ponieważ sekwencja nukleotydów tej nici zwana kodem genetycznym określa kolejność występowania aminokwasów w poszczególnych białkach. Druga nić natomiast - nić matrycowa (3'→5') wykorzystywana jest do przepisywania (transkrypcji) informacji genetycznej. Replikacji (namnażaniu) podlegają obydwie nici.
Jedynie kilka procent sekwencji cząsteczki DNA to sekwencje kodujące, zawierające geny, czyli odcinki, w których zapisana jest struktura różnych rodzajów RNA. Tylko część genów (tzw. geny konstytucyjne) koduje cząsteczki mRNA, na podstawie sekwencji których syntetyzowane są białka. Inne cząsteczki RNA uczestniczą natomiast w biosyntezie białka pośrednio. Najważniejszymi z nich są tRNA (transportowy RNA), rRNA (rybosomalny RNA) oraz miRNA (regulatorowy mikro RNA).
Rys. 6.1. Fragment helisy DNA
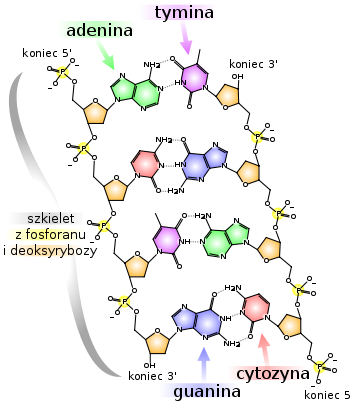
Pozostałej, niekodującej części DNA przypisuje się funkcje regulatorowe lub też inną, nieznaną dotąd rolę, np. ochrony informacji genetycznej przed działaniem mutagenów, czyli czynników zmieniających strukturę DNA, a więc zawartą w nim informację genetyczną. Warto przypomnieć jeszcze, że skok „śrubowo nawiniętych” dwóch nici polinukleotydowych wynosi 3,4 nm, na co składa się 10 par zasad, natomiast średnica helisy wynosi 2 nm.
DNA wytwarza struktury wyższego rzędu, tzw. nukleosomy (Rys. 6.2), obejmujące około 150 par nukleotydów, nawiniętych na rdzeń składający się z 8 cząsteczek białek histonowych (po dwie cząsteczki z czterech rodzajów histonów grupy H2). Cząsteczka innego histonu (H1) zapobiega rozwijaniu się struktury nukleosomu.
Struktury nukleosomowe zwijają się następnie w tzw. superhelisę, która ostatecznie tworzy pętle o końcach przyłączonych do białek nukleoszkieletu (Rys. 6.3). Dzięki temu, pomimo ogromnej ilości informacji zmagazynowanej w DNA (np. sumaryczna długość DNA w jądrze ludzkiej komórki somatycznej sięga 2 m i zawiera 5,8 miliardów par nukleotydów), enzymy odczytujące tę informację bezbłędnie odnajdują i kopiują w procesie transkrypcji każdy potrzebny gen „licząc” nie kolejne nukleotydy, ale „odliczając” całe pętle superhelisy, zawierające tysiące nukleotydów. W okresie poprzedzającym mitozę cząsteczki DNA nadal się kondensują i tworzą jeszcze grubsze struktury, i wskutek tego stają się znacznie krótsze, przyjmując pałeczkowaty kształt chromosomów. W tej „spakowanej” formie chromosomy mogą być rozdzielane do komórek potomnych.
Rys. 6.2. Nukleosomowa struktura DNA
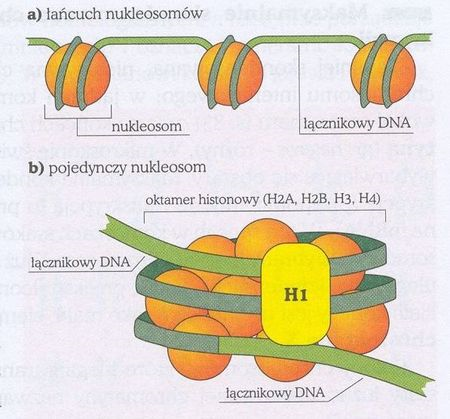
Rys. 6.3. Superhelisa DNA.
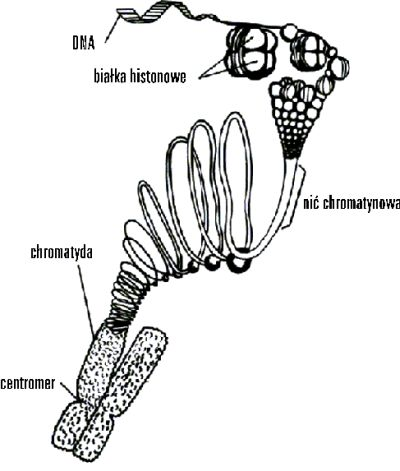
Przed każdym podziałem komórkowym, w tak zwanej fazie S cyklu komórkowego, DNA ulega powieleniu, tak, by każda komórka potomna zawierała taką samą informację genetyczną. Proces powielenia (duplikacji) DNA nazywany jest replikacją. Przed replikacją struktura DNA ulega rozluźnieniu, mostki wodorowe pękają, a obie nici są rozplatane. Następuje wówczas odtworzenie obu nici równocześnie na zasadzie komplementarności zasad i powstają dwie kompletne cząsteczki DNA, przy czym każda składa się z jednaj tzw. starej i jednej dosyntetyzowanej, a więc nowej nici DNA, a każda z tych całych cząsteczek przechodzi do jednej z dwóch komórek potomnych. Ten sposób powielania DNA określany jest jako semikonserwatywne odtwarzanie kodu genetycznego.
Warunkiem zdolności DNA do replikacji jest jego luźna struktura, natomiast forma skondensowana, zwłaszcza do postaci chromosomów, jest nieaktywna transkrypcyjnie i metabolicznie.
Struktura RNA
Kwas RNA jest jednoniciowy, aczkolwiek nić ta zwykle ulega przestrzennemu zwinięciu. W niektórych rodzajach wirusów, tzw. wirusach RNA (retrowirusach i rybowirusach), stanowi on główny magazyn informacji genetycznej. W organizmach natomiast rozróżnianych jest kilka rodzajów kwasu rybonukleinowego. Matrycowy RNA (mRNA) jest syntetyzowany w jądrze komórkowym na podstawie informacji zapisanej na kodującej nici DNA w procesie transkrypcji, tj. przepisania informacji o strukturze konkretnego białka. W pierwszym etapie transkrypcji odpowiedni fragment cząsteczki DNA przepisywany jest na komplementarną strukturę pre-mRNA (hnRNA - heterogenny jądrowy RNA) i po wstępnej obróbce, po wycięciu fragmentów niekodujących tzw. intronów, informacja genetyczna przenoszona jest do cytoplazmy. Do syntezy białek (proces ten nazywa się translacją) potrzebne są dalsze dwa rodzaje RNA: rybosomowy RNA (rRNA, który wytwarza strukturę rybosomów stanowiących środowisko dla syntezy białek w cytoplazmie) i transportujący RNA (tRNA), którego cząsteczki wiążą się specyficznie z określonymi aminokwasami.
Struktura mRNA odczytywana jest sekwencyjnie kolejnymi trójkami nukleotydów, przy czym każda trójka nukleotydów stanowi kodon, czyli informację niezbędną dla określenia rodzaju aminokwasu wbudowywanego w odpowiednie miejsce do syntetyzowanego białka. Ten związek pomiędzy strukturą genu w DNA, strukturą odpowiedniego mRNA oraz strukturą powstającego białka nazywa się kodem genetycznym. Kod ten jest więc sekwencyjny (informacja odczytywana jest w stałej kolejności od aminokwasu N-końcowego do aminokwasu C-końcowego), trójkowy (trzy nukleotydy stanowią informację wystarczającą do zakodowania jednego aminokwasu), uniwersalny (te same trójki nukleotydów kodują te same aminokwasy u wszystkich organizmów od bakterii do człowieka), nieprzekraczający (każdy nukleotyd wchodzi w skład tylko jednego kodonu, a więc nukleotydy w kolejnych kodonach nie nakładają się), oraz zdegenerowany (liczba możliwych trójek nukleotydowych przewyższa liczbę aminokwasów, stąd też niektóre aminokwasy kodowane są przez więcej niż jeden kodon, a poza tym istnieją kodony puste, nie kodujące żadnego aminokwasu i stanowiące sygnał końca struktury białka). Reasumując, kod genetyczny umożliwia translację czyli „przetłumaczenie” informacji genetycznej zawartej w mRNA w postaci sekwencji nukleotydów na sekwencję aminokwasów zawartych w białkach.
Transportowy RNA (tRNA) jest nośnikiem aminokwasów na miejsce syntezy białek. Struktura tego kwasu przypomina kształtem liść koniczynki. W jego cząsteczce można wydzielić cztery nierówne pętle. Na jednym końcu łańcucha tRNA znajdują się trzy nukleotydy, których sekwencja decyduje o rodzaju aminokwasu, który będzie doczepiony do tego rodzaju tRNA. Z kolei na przeciwległym końcu cząsteczki znajduje się trójka nukleotydów nazywana antykodonem. Układ ich zasad jest komplementarny do zasad kodonu znajdującego się na mRNA, dzięki czemu specyficzny tRNA odszukuje właściwe miejsce dla transportowanego przez niego aminokwasu.
Pozostałe rodzaje cząsteczek RNA charakteryzujące się niewielką długością cząsteczek zaliczane do dwóch grup: RNA interferującego (np siRNA - small interfering RNA i miRNA - mikro RNA) oraz nieinterferującego (snRNA - small nuclear RNA, scRNA - small cytoplasmic RNA, snoRNA - small nucleolar RNA,) pełnią doniosłą rolę w regulacji odczytywania informacji genetycznej. Badania ich funkcji oraz znaczenia są obecnie wyznacznikiem w ostatnich latach postępu w dziedzinie biologii molekularnej.
Przyjmuje się, że RNA był pierwszym związkiem warunkującym ewolucję pojawiającym się w prakomórkach. Początkowo cząsteczki RNA realizowały zarówno funkcje gromadzenia jak i przekazywania informacji genetycznej, oraz funkcje katalityczne. Obydwie funkcje były jednak pełnione albo z niewystarczającą precyzją (przekazywanie informacji genetycznej) albo z niedostateczną efektywnością (funkcje katalityczne). Dlatego też w późniejszym etapie ewolucji funkcję przechowywania informacji przejął DNA, zaś funkcje katalityczne - białka enzymatyczne. Jednak do czasów współczesnych RNA spełnia nadal pewne ograniczone funkcje katalityczne (w tzw. rybozymach).
ĆWICZENIA
6.1. Hydroliza, wykrywanie składników kwasów nukleinowych
Zasada:
Po hydrolizie kwasów nukleinowych można wykrywać poszczególne ich składniki jak pentozę, kwas fosforowy i zasady azotowe.
Wykonanie:
Hydroliza kwasów nukleinowych: Do 5 cm3 0,5 % roztworu RNA z drożdży dodać 5 cm3 1 M HCl i ogrzewać na wrzącej łaźni wodnej przez 30 minut od momentu zagotowania.
a) Wykrywanie pentozy:
Do probówki nalać 2 cm3 odczynnika Biala i ogrzewać na łaźni wodnej przez ok. 3 min, a następnie dodać 1 cm3 hydrolizatu. Zamieszać i wstawić do łaźni wodnej. Po kilku minutach zanotować powstające zielononiebieskie zabarwienie.
b) Wykrywanie jonów ortofosforanowych:
Do 1 cm3 hydrolizatu dodać 2 cm3 2,5% roztworu molibdenianu amonowego i całość ogrzać do wrzenia. Wytwarza się żółty osad fosforomolibdenianu amonowego. Po dodaniu paru kropel 1% roztworu kwasu askorbinowego (witamina C) następuje redukcja molibdenianu amonowego do mieszaniny tlenków molibdenu (Mo2O5⋅MoO3), tzw. błękitu molibdenowego.
c) Wykrywanie zasad purynowych:
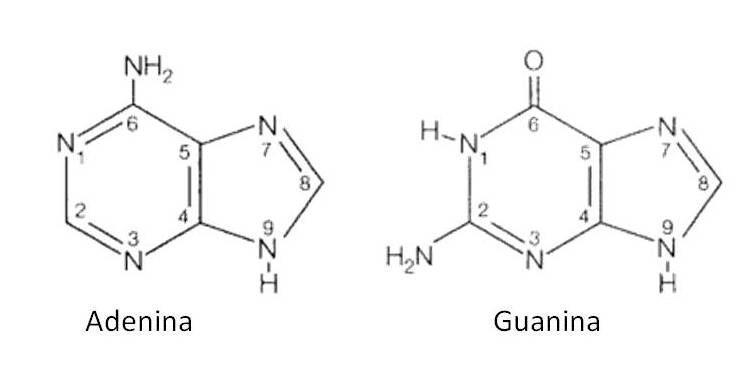
2 cm3 hydrolizatu ogrzać do wrzenia, dodać kilka kropel roztworu 0,25 M CuSO4 i ostrożnie kroplami dodawać nasycony roztwór Na2SO3. W obecności puryn (Rys. 6.4) wytrąca się żółtobiały osad nierozpuszczalnych soli miedziowych I (miedziawych) puryn (Na2SO3 zredukował Cu2+ do Cu+).
Rys. 6.4. Wzory strukturalne zasad purynowych
6.2. Izolacja DNA z warzyw, owoców
Wykonanie:
Pomidor (zgnieść) lub jabłko (zetrzeć na tarce) a następnie umieścić w zlewce i dodać łyżeczkę płynu do mycia naczyń w celu emulgacji lipidów, a przez to dezintegracji błon cytoplazmatycznych. Zlewkę wraz ze zhomogenizowanym materiałem wstawić na 15 minut do łaźni wodnej (60°C) (Uwaga!!! temperatura nie może być zbyt wysoka, bowiem DNA może zdenaturować, z kolei nie może też być za chłodno, bo wtedy nie zdenaturuje RNA co sprawi, że izolowany DNA może być nim zanieczyszczony). Przełożyć do łaźni lodowej (szybko schłodzić) i dodać łyżkę stołową soli zmiękczającej do mięsa firmy „Kamis” (na tym etapie następuje wytrącenie białek, które podobnie jak DNA rozpuszczają się w wodzie). Otrzymany ekstrakt przesączyć przez lejek z sączkiem bibułowym. Wlać przesącz do naczynia (cylindra) zawierającego 50 cm3 alkoholu 96% (może być denaturat). Zaobserwować wytrącone białe „kłaczki” DNA.
6.3. Spektrofotometryczne oznaczanie zawartości kwasów nukleinowych i badanie ich czystości
Zasada:
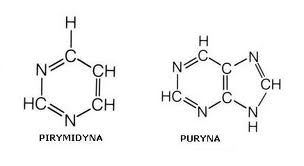
Aromatyczne pierścienie purynowe i pirymidynowe (Rys. 6.5) absorbują światło w zakresie UV. Właściwość tę można zastosować do pomiaru stężenia RNA w roztworze. Wprawdzie poszczególne zasady różnią się nieco pod względem zakresem maksymalnej absorpcji UV, to jednak różnice te są niewielkie i można przyjąć, że 260 nm jest optymalną długością fali przy pomiarze ilościowym RNA.
Rys. 6.5. Wzory strukturalne pirymidyny i puryny
Spektrofotometrię w UV można wykorzystać również do ilościowych oznaczeń białek, jednak jedynie w przypadku, jeśli oznacza się zawartość konkretnego białka względem konkretnego wzorca (białko badane i wzorzec zawierać muszą taką samą ilość absorbujących UV aminokwasów aromatycznych; pomiar wykonuje się przy λ = 280 nm).
Wykonanie:
Sporządzanie krzywej kalibracyjnej
Do probówki wlać 3 cm3 roztworu wzorcowego (w 100 cm3 buforu octanowego o pH = 5,5 rozpuścić na gorąco 10 mg RNA z drożdży) i dolać do niego 3 cm3 buforu octanowego uzyskując stężenie 100 μg RNA w 1 cm3. Po zmieszaniu powtórzyć rozcieńczanie tą samą metodą pobierając do nowej probówki 3 cm3 roztworu z poprzedniej probówki i uzupełnić 3 cm3 buforu octanowego, uzyskując kolejne stężenia RNA: 50; 25; 12,5; 6,25; 3,1; 1,5 μg w 1 cm3. Z każdego rozcieńczenia wzorcowego pobrać po 1 cm3 do kuwetki spektrofotometrycznej ze szkła kwarcowego (przepuszczającego promienie UV) i zmierzyć absorbancję przy długości fali 260 nm względem ślepej próby (sam bufor octanowy).
Pomiar stężenia RNA w nieznanym roztworze
Do kuwetki spektrofotometrycznej wlać 1 cm3 roztworu RNA i zmierzyć absorbancję przy λ=260 nm względme ślepej próby (bufor octanowy).
Sprawozdanie
a) Za pomocą arkusza kalkulacyjnego MS Excel wykreślić krzywą kalibracyjną i wyznaczyć równanie regresji liniowej zależności absorbancji od stężenia RNA (wzorca).
b) Odczytać stężenie RNA nieznanych roztworów z równania regresji liniowej (x), podstawiając pod y odczytaną absorbancję.
96
Wyszukiwarka