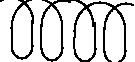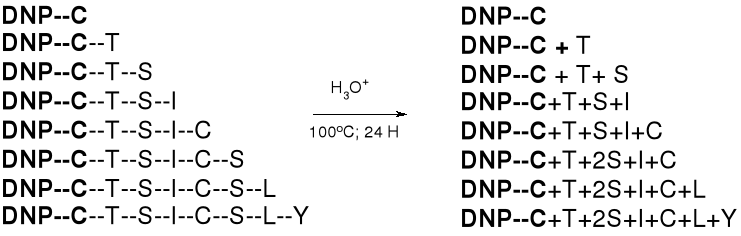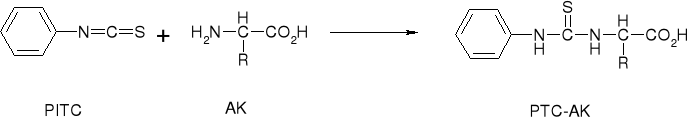STRUKTURY BIAŁEK |
||
LINDSROM-LANG: STRUKTURA I, II & II-RZĘDOWA |
||
STRUKTURA I-RZĘDOWA |
||
OKREŚLA SEKWENCJĘ (LICZBA I KOLEJNOŚĆ) POŁĄCZONYCH AMINOKWASÓW |
||
SEKWENCJA AMINOKWASÓW |
||
|
||
STRUKTURA II-RZĘDOWA: OPISUJE KONFORMACJĘ ŁAŃCUCHA PEPTYDOWEGO: |
||
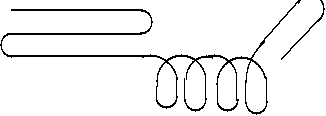
STRUKTURA HELIKALNA vs. POFAŁDOWANY ŁAŃCUCH |
||
|
|
|
HELISA |
WSTĘGA |
|
|
||
STRUKTURA III-RZĘDOWA |
||
|
||
OKREŚLA TRÓJWYMIAROWE POFAŁDOWANIE (WSTĘGA VS HELISA) ŁAŃCUCHA PEPTYDOWEGO |
||
|
||
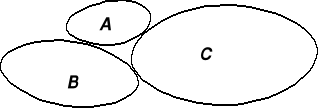
STRUKTURA IV-RZĘDOWA [BERNAL; 1958] |
||
DOTYCZY ASOCJATÓW BIAŁKOWYCH W KOMPLEKSY MOLEKULARNE |
||
|
||
WZAJEMNE USYTUOWANIE PODJEDNOSTEK |
||
|
||
STRUKTURY II#, III# & IV# RZĘDOWE TWORZĄ WSPÓLNIE KONFORMACJĘ BIAŁKA |
||
ANALIZA BIAŁEK/PEPTYDÓW |
||
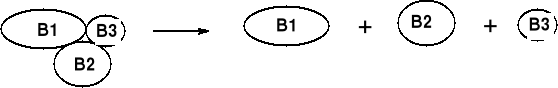
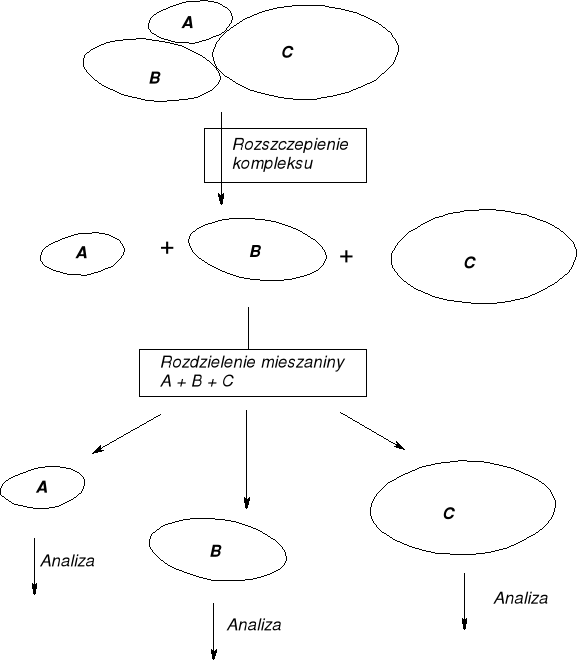
1. ROZSZCZEPIENIE KOMPLEKSU PEPTYDÓW (BIAŁEK) |
||
|
||
2. ROZDZIAŁ SKŁADOWYCH PODJEDNOSTEK PEPTYDOWYCH/ BIAŁKOWYCH |
||
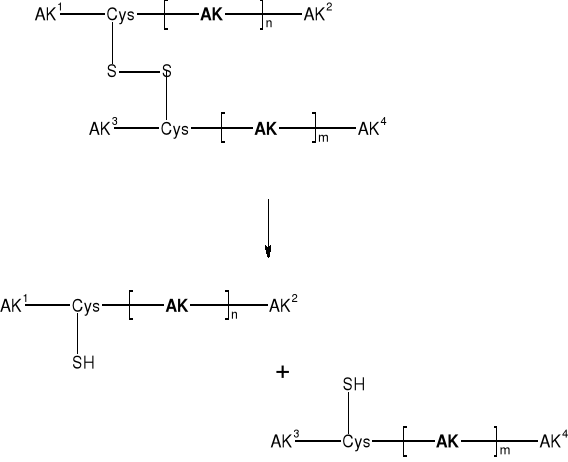
2.1. PRZERWANIE MOSTKÓW DISIARCZKOWYCH |
||
|
||
3. SELEKTYWNA DEGRADACJA ŁAŃCUCHA BIAŁKA/PEPTYDU |
||
4. ANALIZA SKŁADU AMINOKWASOWEGO PEPTYDU/BIAŁKA & MASY CZASTECZKOWEJ |
||
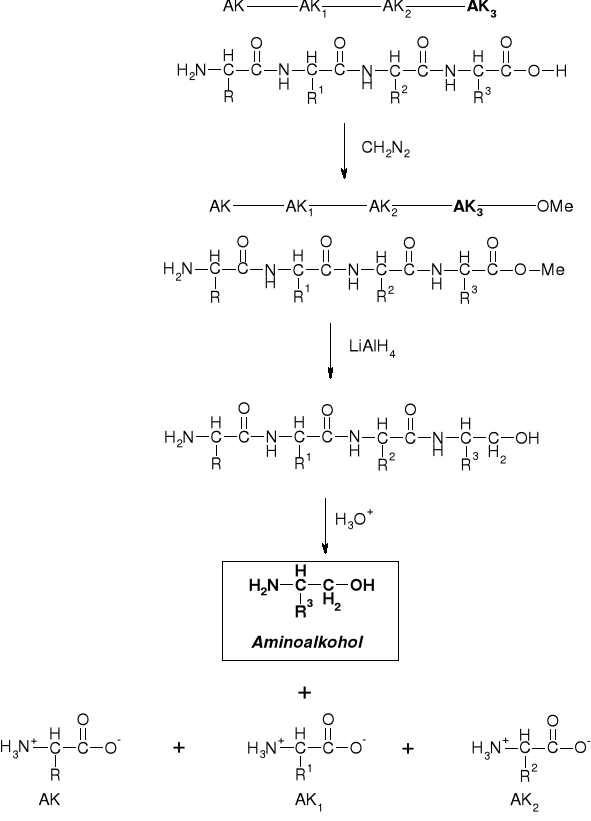
5. OZNACZENIE AMINOKWASU N-KOŃCOWEGO & C-KOŃCOWEGO |
||
6. ANALIZA SEKWENCJI PODJEDNOSTKI |
||
Opisano ponad 1000 struktur białek (15000 prac). |
||
7. WERYFIKACJA SEKWENCJI POPRZEZ SYNTEZĘ |
||
1. ROZSZCZEPIENIE KOMPLEKSU PEPTYDÓW/ BIAŁEK
|
||
|
||
DENATURACJA: DSS, MOCZNIK |
||
|
||||
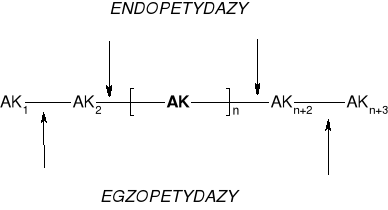
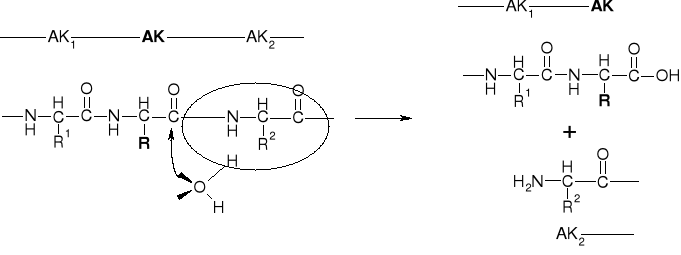
3. HYDROLIZA SPECYFICZNA ŁAŃCUCHÓW PEPTYDOWYCH |
||||
|
||||
3.1. HYDROLIZA ENZYMATYCZNA ŁAŃCUCHÓW PEPTYDOWYCH |
||||
|
||||
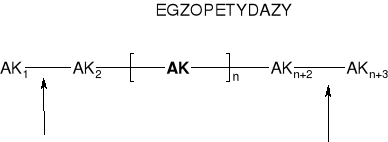
3.1.1. EGZOPEPTYDAZY
|
||||
|
||||
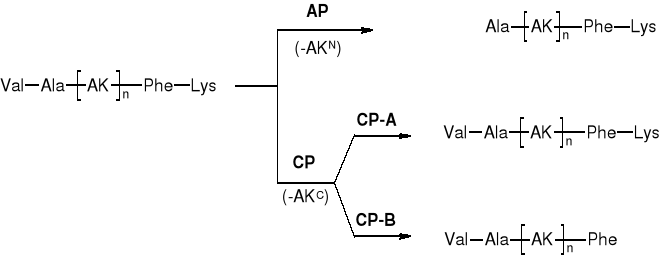
AMINOPEPTYDAZY (AP) |
KARBOKSYPEPTYDAZY (CP) |
|||
np. Leucyloaminopeptydaza (LAP) |
CP-A |
CP-B |
||
|
odcinają AKC z wyjątkiem His, Arg, Lys, Pro |
odcinają AKC: Lys, Arg, Orn |
||
|
||||
3.1.2. ENDOPEPTYDAZY
|
||||
PROTEAZY: trypsyna, chymotrypsyna, pepsyna, papaina, subtylizyna, elastaza, termolizyna
|
||||
|
||||
Trypsyna: |
Chymotrypsyna |
Subtylizyna |
Elastaza |
|
AK: Lys; Arg
|
AK: Phe, Tyr, Trp |
AK: Gly, Ser, Phe, Tyr, Trp |
AK: Aminokwasy obojętne |
|
3.2. HYDROLIZA CHEMICZNA ŁAŃCUCHÓW PEPTYD.
|
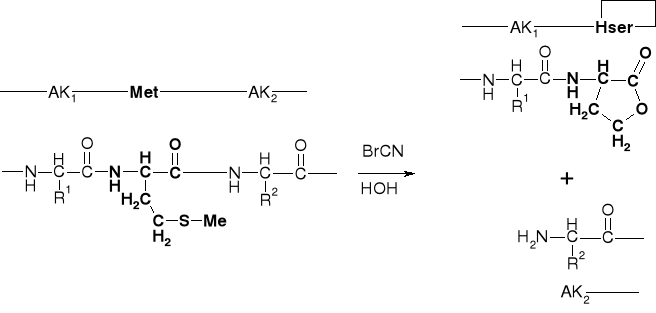
3.2.1. DZIAŁANIE BrCN
|
|
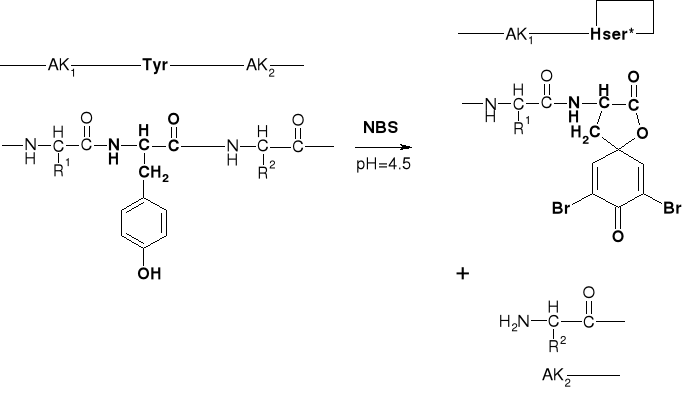
3.2.2. DZIAŁANIE NBS
|
|
|
ANALIZA SEKWENCYJNA PEPTYDÓW |
4. WYZNACZANIE SKŁADU AMINOKWASOWEGO PEPTYDÓW |
4.1. Wyczerpująca degradacja hydrolityczna peptydu do aminokwasów.
|
|
5. OZNACZANIE GRUP KOŃCOWYCH PEPTYDU |
5.1. Oznaczanie Aminokwasu N-Końcowego |
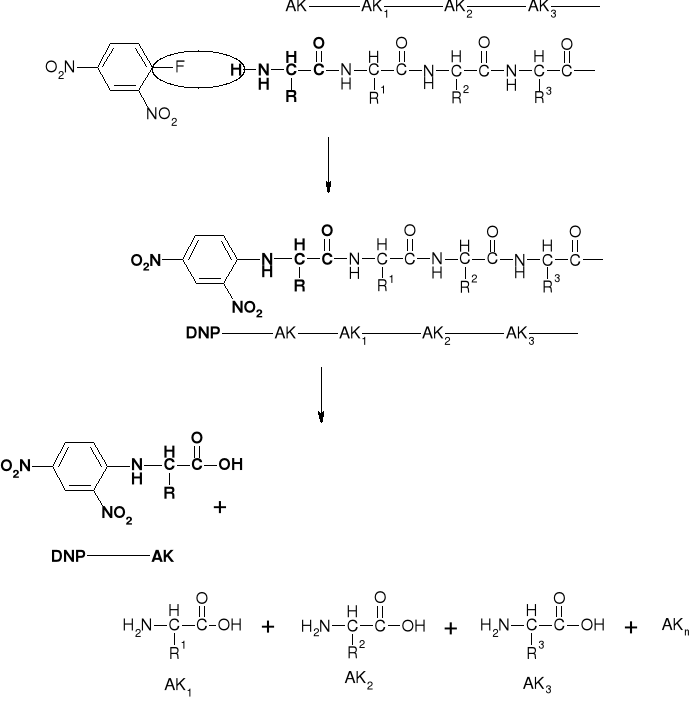
5.1.1. Metoda Sangera |
|
|
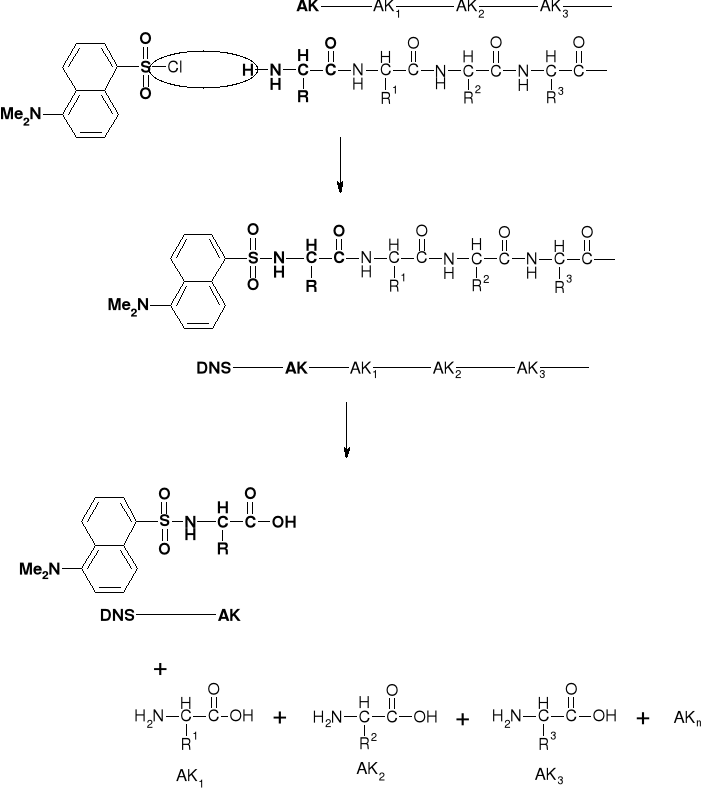
5.1.2. Metoda Dansylowa |
|
|
|
5.1.3. Inne Metody |
|
|
5.2. OZNACZANIE AMINOKWASU C-KOŃCOWEGO |
|
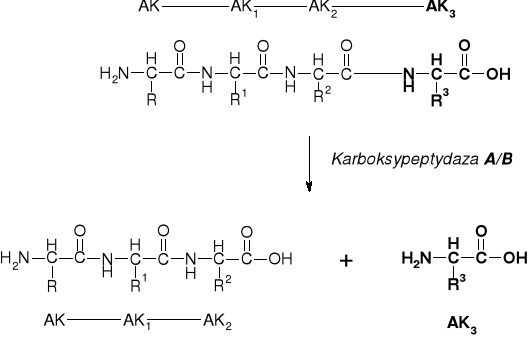
5.2.1. METODY ENZYMATYCZNE |
Egzopeptydazy: Karboksypeptydaza A i B |
|
|
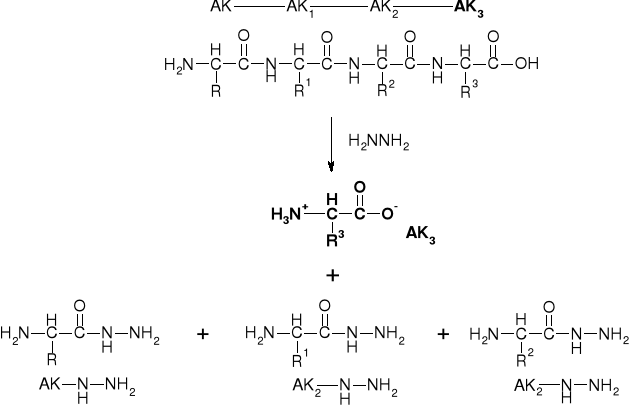
5.2.2. METODY CHEMICZNE |
5.2.2.1. Metoda Akabori'ego |
|
5.2.2.2. Metoda Fromageot'a |
|
|
|
6. ANALIZA SEKWENCJI PODJEDNOSTKI |
|
6.1. STOPNIOWA DEGRADACJA ŁAŃCUCHA PEPTYDOWEGO |
6.1.2. METODY CHEMICZNE |
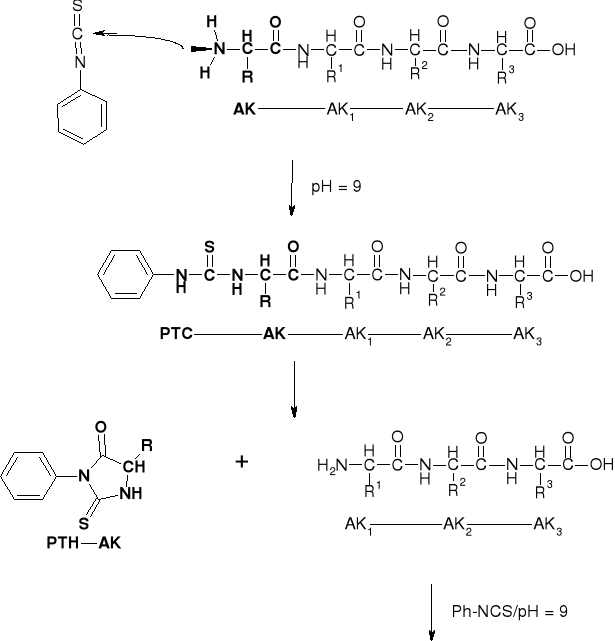
6.1.2.1. METODA EDMANA |
|
|
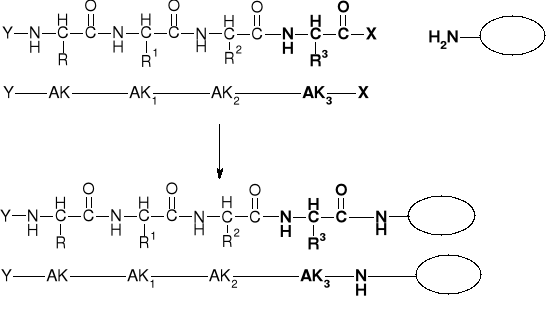
6.1.2.2. Degradacja EDMANA Peptydu Zawieszonego na Nośniku Stałym |
|
1. Zawieszenie peptydu na nośniku |
|
|
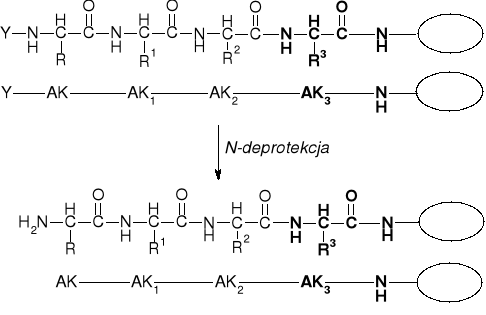
2. N-Deprotekcja peptydu zawieszonego na fazie stałej |
|
|
|
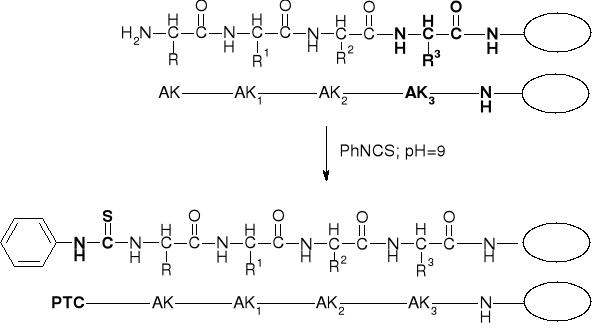
3. Reakcja peptydu zawieszonego na fazie stałej z izotiocyjanianem fenylu (PhNCS) (konwersja peptydu do PTC-peptydu) |
|
|
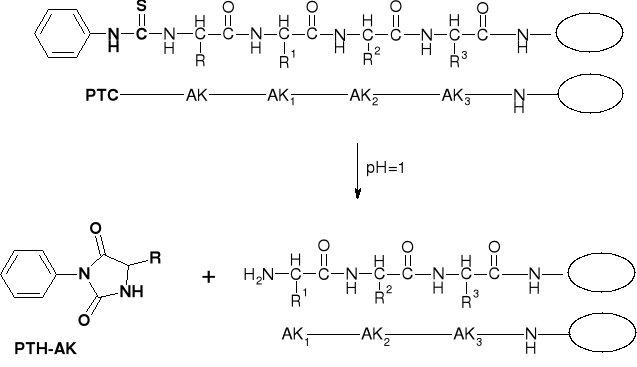
4. Odszczepienie aminokwasu N-końcowego w postaci PTH-AK |
|
|
5. Powtórzenie cyklu. |
|
6.1.2.3. METODA SANGERA SEKWENCJONOWANIA BIAŁEK |
|
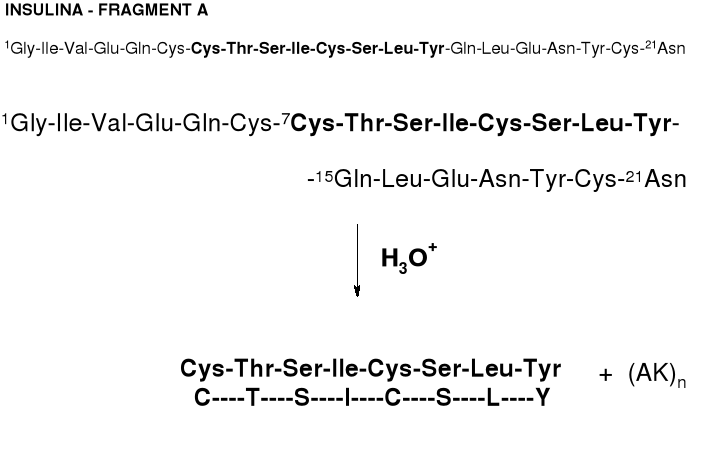
1. INSULINA A PODDAWANY JEST CZĘŚCIOWEJ HYDROLIZIE |
|
|
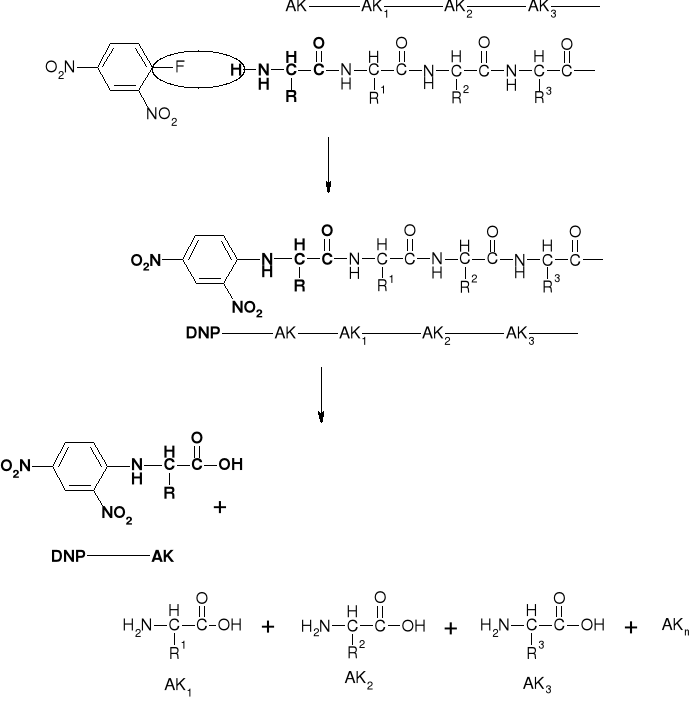
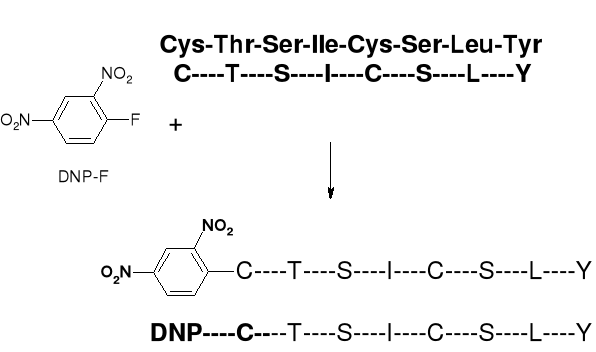
2. FRAGMENT C-T-S-I-C-S-L-Y IZOLOWANY JEST Z MIESZANINY HYDROLITYCZNEJ I PODDAWANY REAKCJI Z ODCZYNNIKIEM SANGERA (DNP-F) |
|
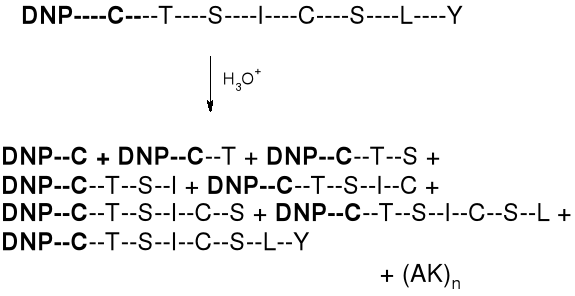
3. ZMODYFIKOWANY DNP-PEPTYD PODDAWANY JEST ŁAGODNEJ (CZĘŚCIOWEJ) HYDROLIZIE
|
|
A. ODDZIELA SIE FRAKCJE DNP-(AK)n OD (AK)n. |
B. MIESZANINA DNP-(AK)n PODDAWANA JEST FRAKCJONOWANIU |
C. FRAKCJE PODDAWANE SĄ WYCZERPUJĄCEJ HYDROLIZIE |
|
4. NA PODSTAWIE ANALIZY HYDROLIZATÓW FRAKCJI DOKONYWANE JEST “SKŁADANIE” SEKWENCJI PEPTYDU: |
DNP-C → DNP-C-T → DNP-C-T-S → DNP-C-T-S-I → DNP-C-T-S-I-C → DNP-C-T-S-I-C-S → DNP-C-T-S-I-C-S-L → DNP-C-T-S-I-C-S-L-Y
|
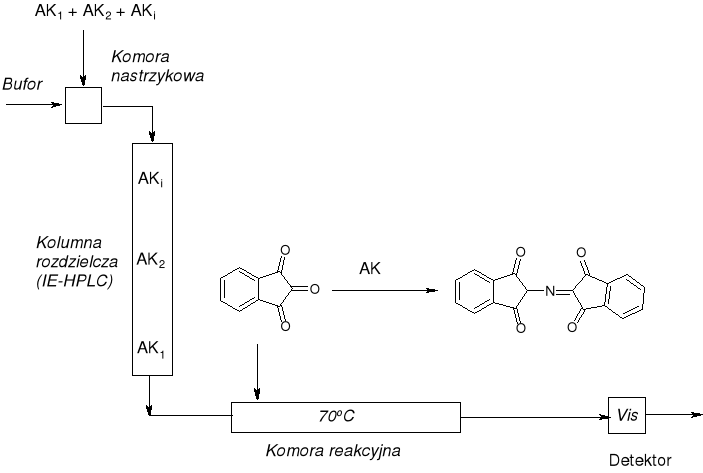
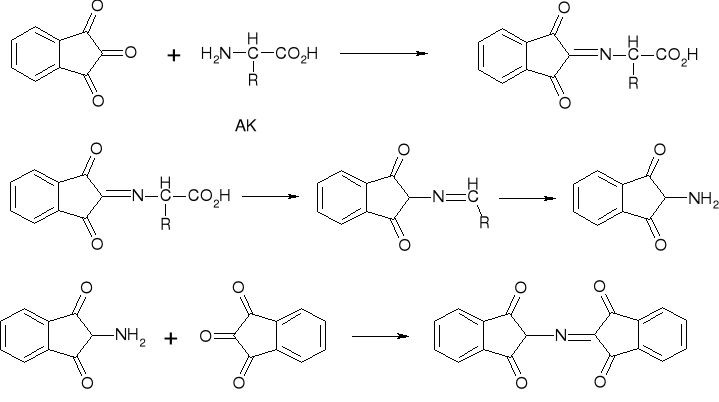
ANALIZATOR AMINOKWASÓW Z DERYWATYZACJĄ POST-KOLUMNOWĄ MOORE'A I STEINA |
|
REAKCJA DETEKCYJNA |
|
λmax = 540 nm, 1 µg (10 nmoli) |
|
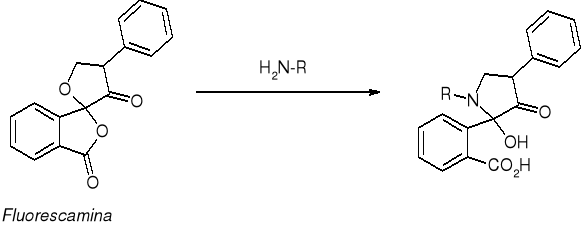
REAKCJA DETEKCYJNA AK Z FLUORESCAMINĄ |
|
λmax = 336 nm, 10 pmoli |
|
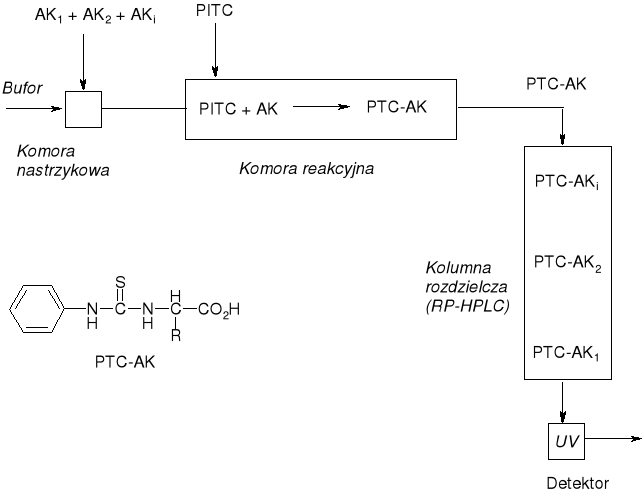
ANALIZATOR AMINOKWASÓW Z DERYWATYZACJĄ PRE-KOLUMNOWĄ |
|
REAKCJA DERYWATYZACYJNA: AK → PTC-AK |
|
λmax = 270 nm, (1 nmol) |
|
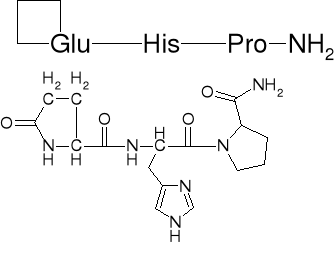
IZOLACJA TRF Z MATERIAŁU BIOLOGICZNEGO |
||
TYREOLIBERYNA (TRF) - HORMON UWALNIAJĄCY TYREOTROPINĘ (STYMULACJA TARCZYCY) |
||
|
||
USTALENIE SEKWENCJI TRF |
||
IZOLACJA TRF Z PODWZGÓRZA OWCY |
||
ETAPY IZOLACJI |
MASA |
U/mg |
MATERIAŁ WYJŚCIOWY: 300 000 podwzgórzy owiec |
25 000 g |
|
1. EKSTRAKCJA CHCl3/EtOH |
294 g |
1 |
2. ULTRAFILTRACJA |
71 g |
3 |
3. SEPHADEX-G-25 |
16 g |
16 |
4. HPLC (×2) |
246 mg |
800 |
5. HPLC (×4) |
2 mg |
58500 |
NIEJEDNOZNACZNE WYNIKI SEKWENCJONOWANIA -
KONIECZNA SYNTEZA TOTALNA PEPTYDU [1969]
Od 1969 r. - ponad 100 publikacji nt. korelacji SAR-TRF
17
Wyszukiwarka
Podobne podstrony:
PEPTYDY-GENERAL, CHEMIA UŁ, 3 rok, Biochemia
równowagi protolityczne aminokwasów, CHEMIA UŁ, 3 rok, Biochemia
AK-REACTIVITY, CHEMIA UŁ, 3 rok, Biochemia
AK-BIOSYNTEZA, CHEMIA UŁ, 3 rok, Biochemia
1a i 1b, CHEMIA UŁ, 2 rok, Analiza instrumentalna
sciaga I koło, CHEMIA UŁ, 4 rok, Krystalografia
krystalografia kolokwium I LAB, CHEMIA UŁ, 4 rok, Krystalografia
Kryształki lab ściąga, CHEMIA UŁ, 4 rok, Krystalografia
spektroskopia ściągi, CHEMIA UŁ, 4 rok, Spektroskopia
Spektroskopia LAB kolokwium, CHEMIA UŁ, 4 rok, Spektroskopia
Peptydy i białka, STOMATOLOGIA, II ROK, Biochemia, zbiorcze
wyklad 1 1 2008, CHEMIA UŁ, 3 rok, Fizyczna, różne
Tabelka LabolatoriumI, ANALITYKA CHEMICZNA- UŁ, Rok I, CHEMIA OGÓLNA I, I- laboratorium
AJ Wykrywanie anionów I-VI grupy – analiza kontrolna, ~FARMACJA, I rok, chemia (ciul wie co), Semest
Deklaracja jezyki, Studia - Chemia kosmetyczna UŁ, I rok
chromatografia analiza jakościowa, II rok, II semestr, Chemia wody i powietrza
Przykładowe egzaminy, Studia - Chemia kosmetyczna UŁ, I rok, II semestr, MATEMATYKA wykłady
AJ Wykrywanie soli - analiza kontrolna, ~FARMACJA, I rok, chemia (ciul wie co), Semestr I
chromatografia analiza ilościowa, II rok, II semestr, Chemia wody i powietrza
więcej podobnych podstron