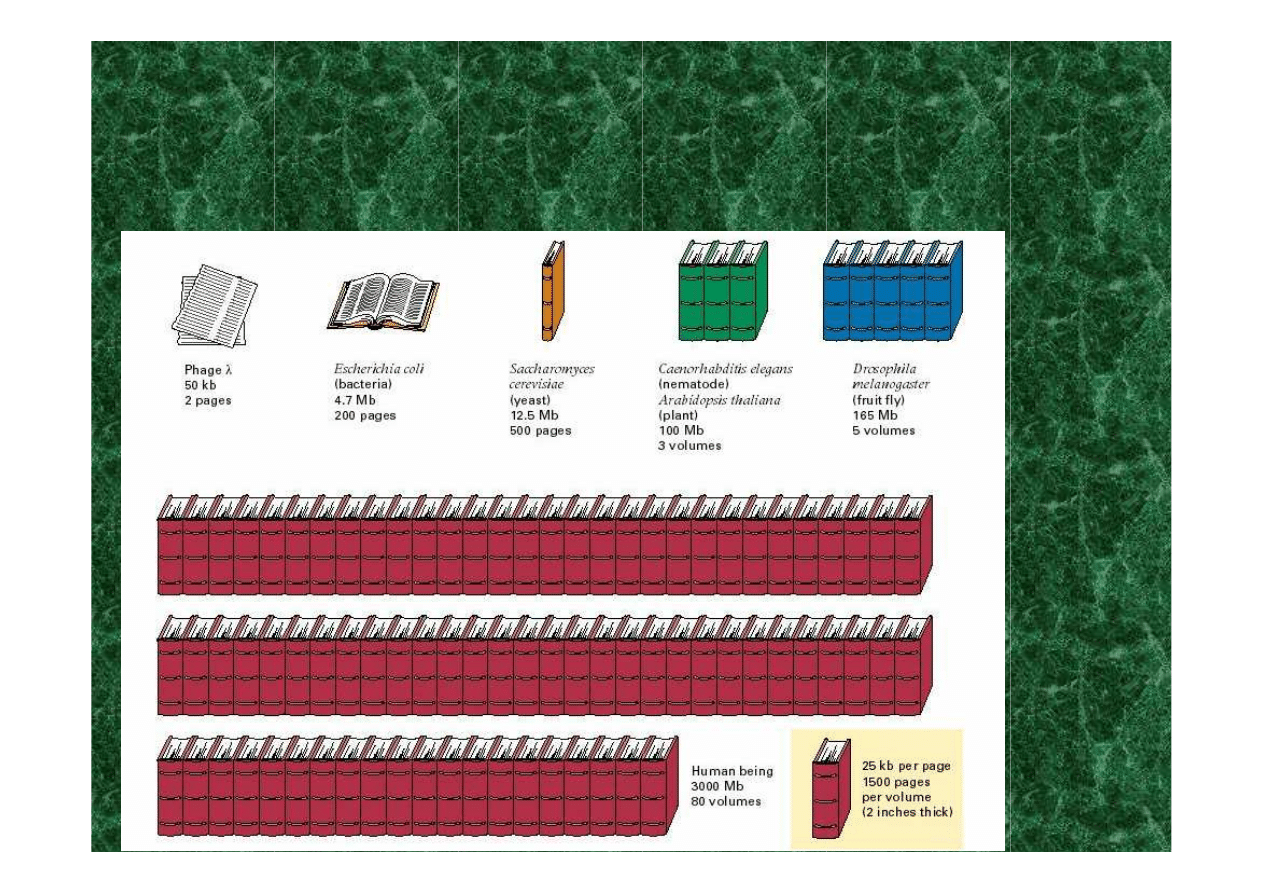
GENOM
- całość informacji genetycznej danego gatunku
1pz (ang. 1 bp, base pair)
1000 pz = 1 kpz (ang. 1 kb, kilobase)
1mln pz = 1 Mpz (ang. 1 Mb, megabase)

J. Craig Venter
Celera Genomics
Francis Collins
Human Genome Project
http://mercury.bio.uaf.edu/~kevin_mccracken/genetics/lectures/chapter_08-09.ppt#331,28,Slajd 28
haploidalny genom jądrowy
24 chromosomy
3200 Mbp; ~25 000 genów
genom mitochondrialny
16,6 kb; 37 genów
genom człowieka =
genom jądrowy + genom mitochondrialny
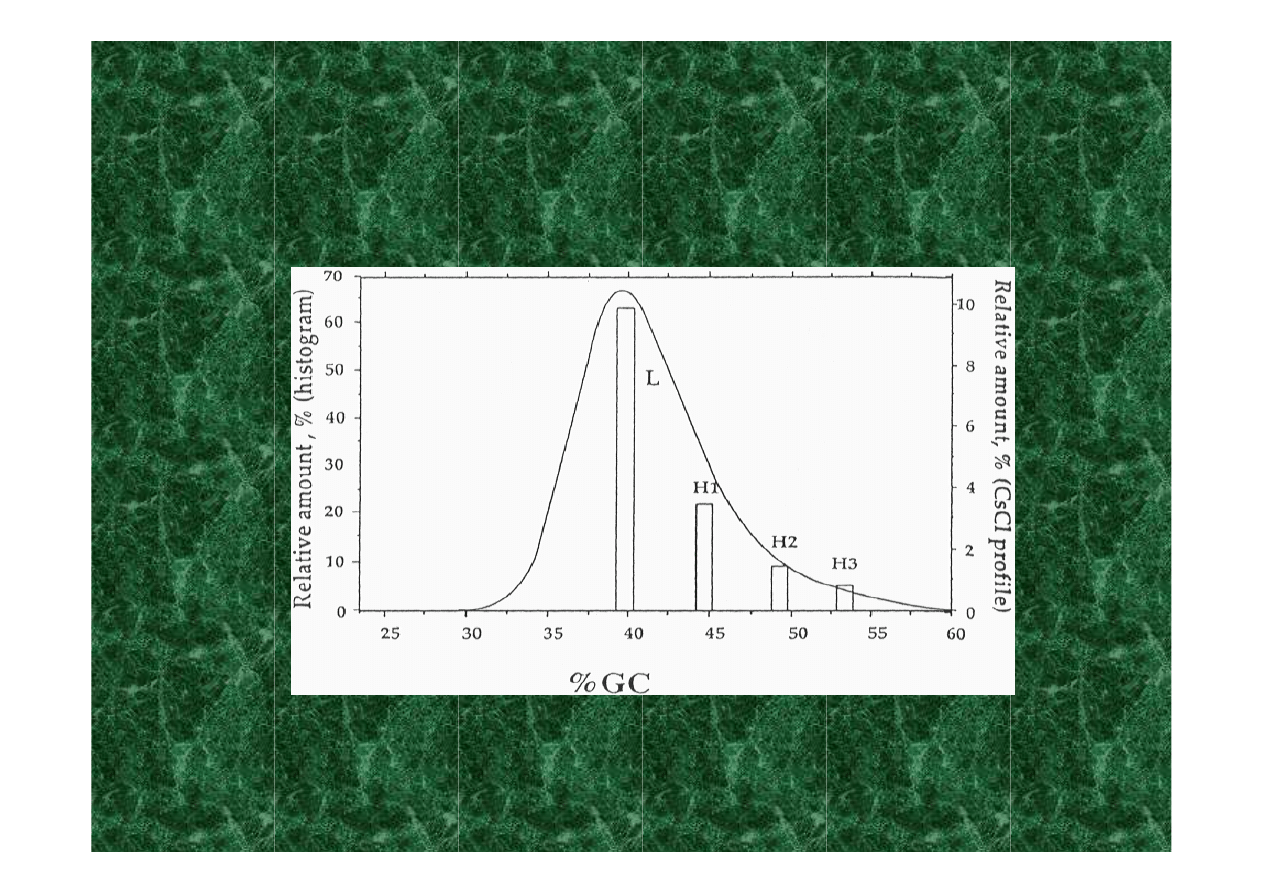
Chromosomy, prążki, izochory, geny
izochory
- długie (>300 kb) regiony o podobnej zawartości
par GC; wyróżnia się rodziny izochor L1-2, H1-4
H.sapiens średnia zawartość CG - 41%
chr. 4 & 13 - 38%
chr. 19
- 49%
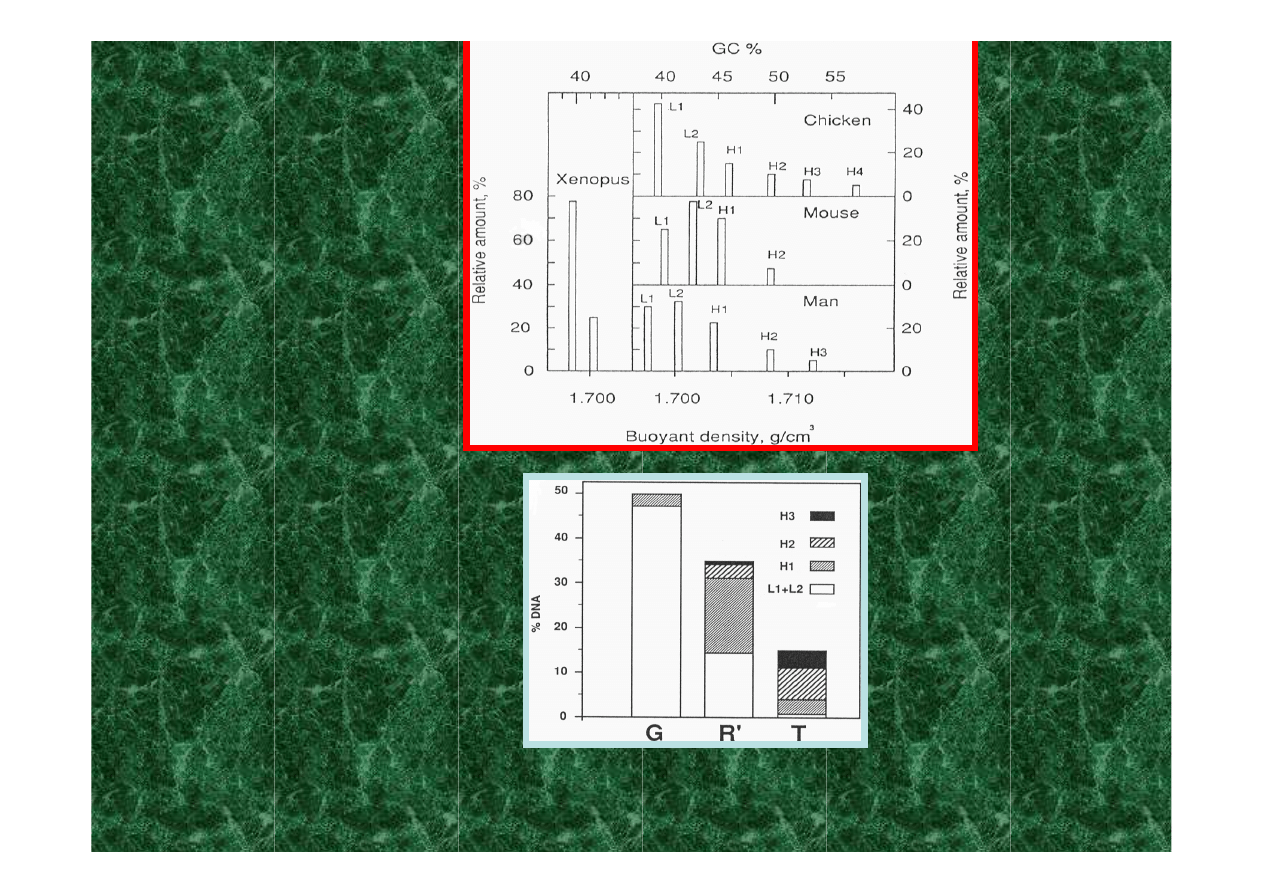
rozkład izochor
u różnych gatunków
rozkład izochor w prążkach
chromosomowych:
G: trypsynizacja, Giemsa
R: denaturacja cieplna, Giemsa
T: denaturacja cieplna +Giemsa
rozmieszczenie genów
:
izochory H
, zwłaszcza H3, są
bogate
w geny
rozmieszczenie genów:
istnieją chromosomy
bogate
(11, 17,19) i
ubogie
w geny (18)
Wielkość genomów
Prokariota
-
od
0,6 Mb
(niektóre obligatoryjne
wewnątrzkomórkowe
pasożyty) do
10 Mb
(niektóre sinice)
liczba genów 500 - 8 000
Eukariota - duża rozpiętość
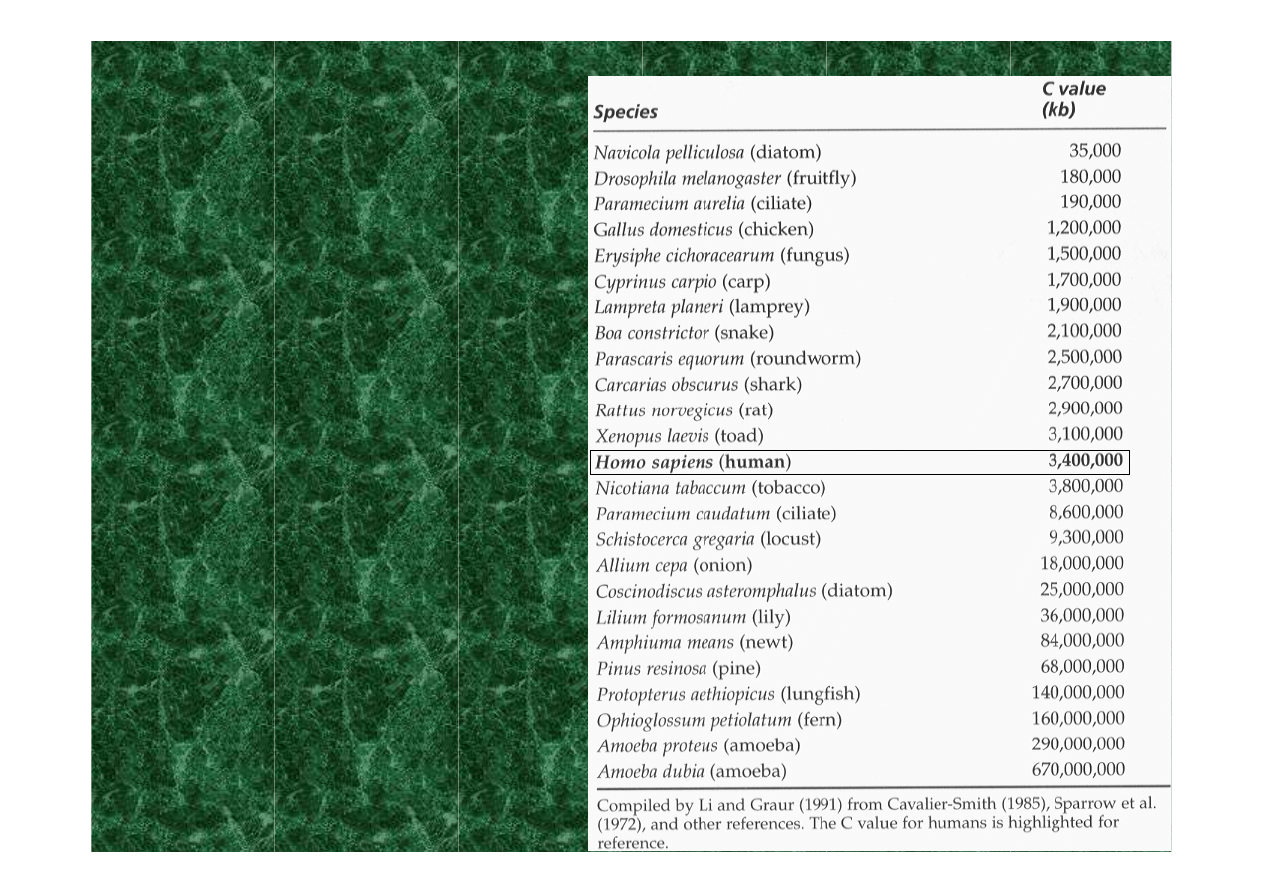
wartość C
- całkowita
długość haploidalnego
genomu
paradoks wartości C
brak korelacji pomiędzy
wielkością genomu
a poziomem złożoności
eukariotycznego
gatunku
Ohno S (1970) Evolution by gene duplication. London: George Allen and Unwin. 160 p.
http://www.gmu.edu/departments/mmb/baranova/pages/ppt/LEC5-organizationOfHumanGenome.ppt#279,17,Slajd 17
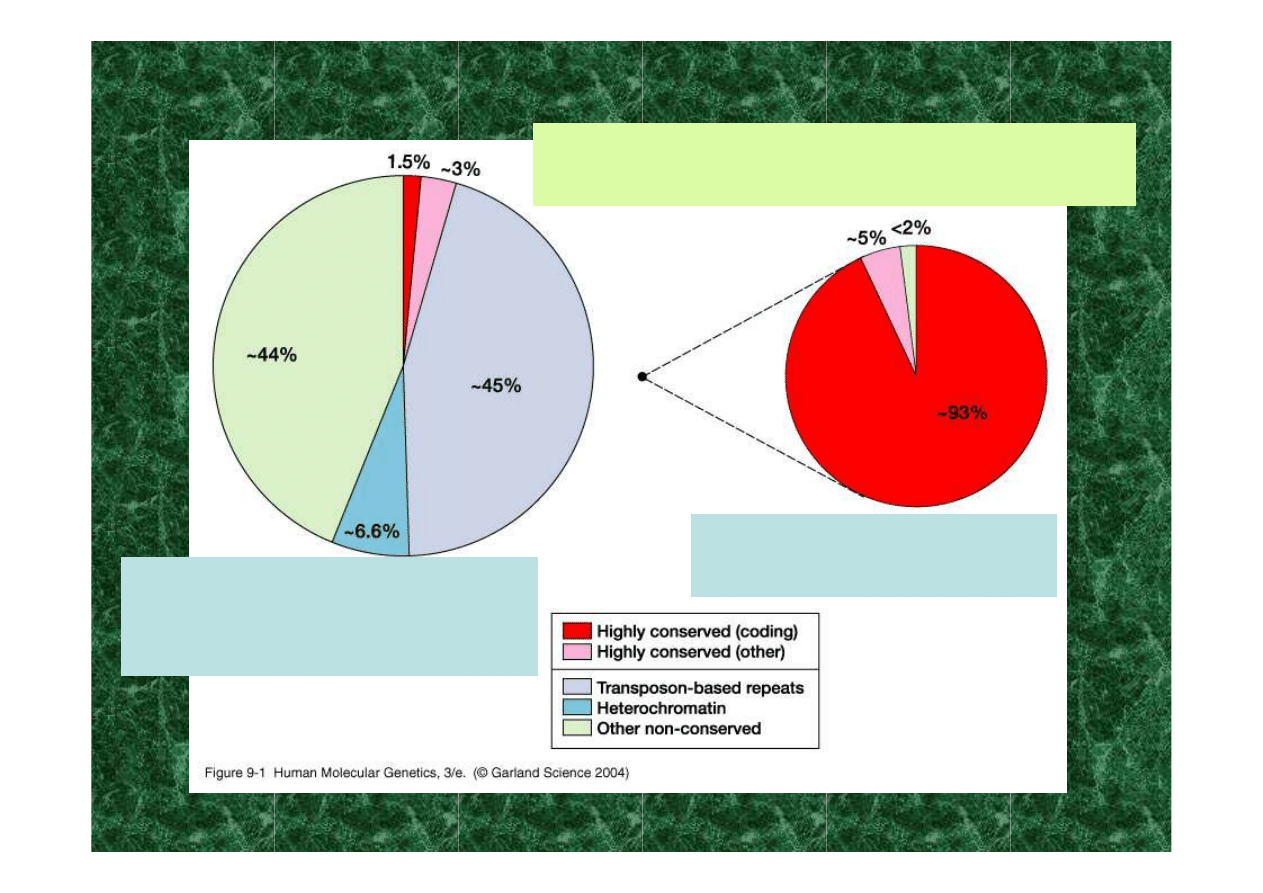
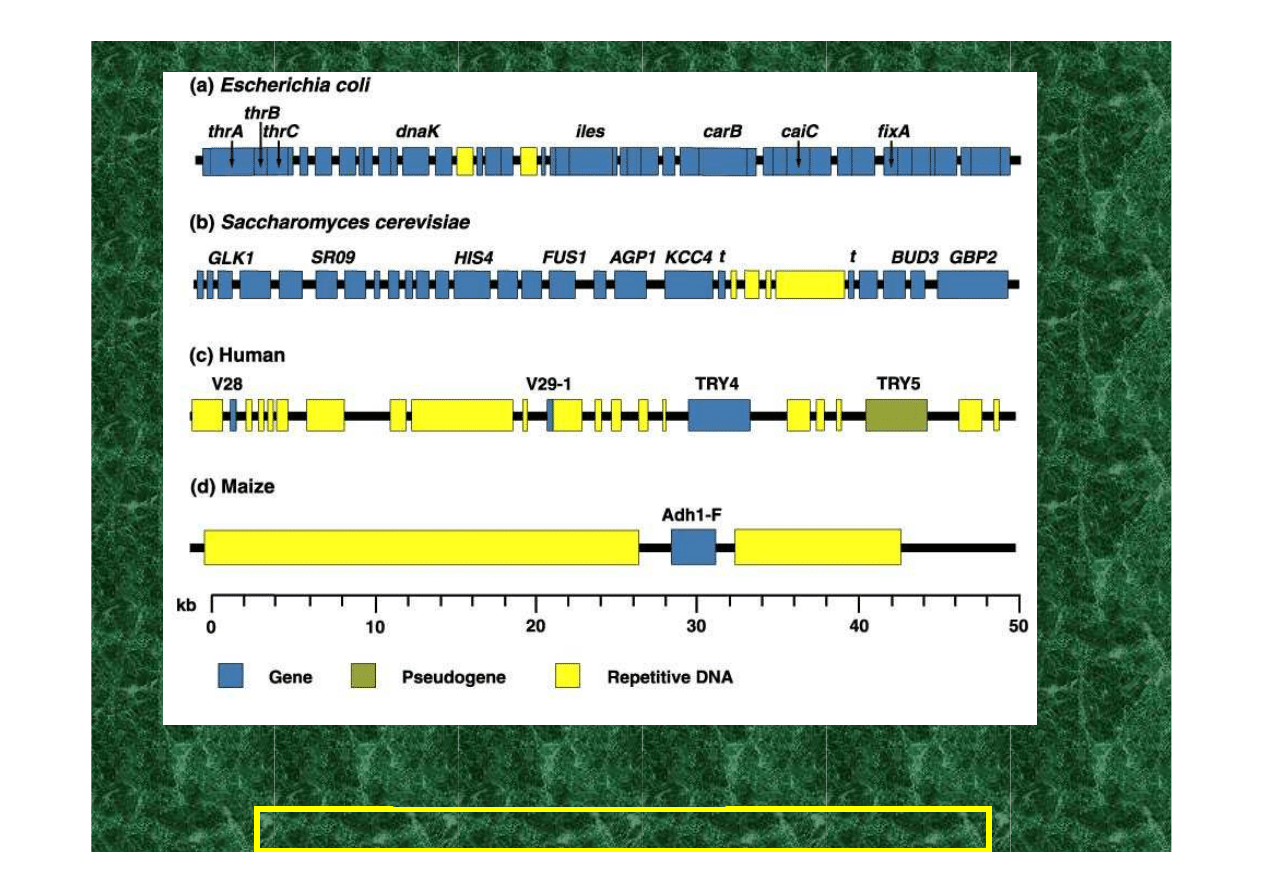
POWTÓRZENIA!!!
tylko 5-10% typowego genomu
kręgowców jest kodujące,
co najmniej 20% stanowią
sekwencje powtórzone
Nonrepetitive
Repetitive
DNA DNA
100
83
17
70
30
64
36
54 46
30
70
E. coli
(bacterium)
G
e
n
o
m
e
s
iz
e
(
b
p
)
10
10
10
9
10
8
10
7
10
6
10
5
C. elegans
(nematode worm)
D. melanogaster
(fruit fly)
M. musculus
(mouse)
X. laevis
(toad)
N. tabacum
(tobacco)
HUMAN GENOME
Genes and gene-
related sequences
Extragenic
DNA
Nuclear genome
3000 Mb
30-40000 genes?
Mitochondrial genome
16.6 kb
37 genes
Coding
DNA
Noncoding
DNA
Unique or
low copy
number
Moderate to
highly
repetitive
Pseudogenes
Gene
fragments
Introns,
untranslated
sequences, etc.
Tandemly
repeated
or clustered
repeats
Interspersed
repeat s
Unique or moderately repetitive
Two rRNA
genes
22 tRNA
genes
13 polypeptide-
encoding genes
~30%
~70%
~10%
~90%
80%
20%
powtórzenia
tandemowe (satelitarne)
rozproszone (LINE, SINE)
zduplikowane geny i pseudogeny
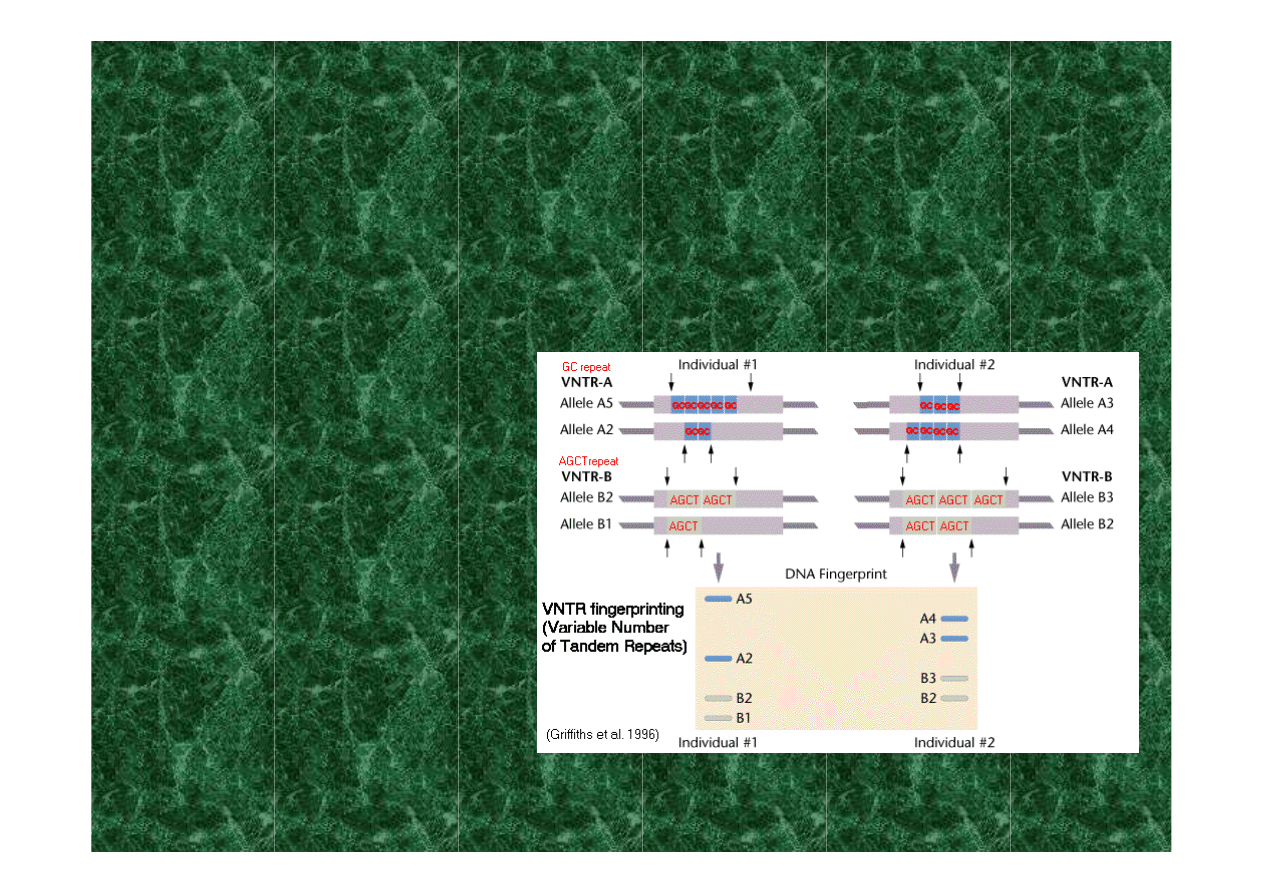
mini
- i
mikro
satelity umożliwiają
identyfikację ludzi w badaniu
genetycznych odcisków palców
(ang. DNA fingerprinting)
powt.
satelitarne:
centromerowe, heterochromatynowe
wlk. powtórzenia 5-200 pz, ilość powtórzeń >1000
powt.
minisatelitarne (30 000 loci u człowieka)
wlk. powtórzenia 15-50 pz, ilość powtórzeń do 1000
[np. sekwencje telomerowe (TTAGGG)
n
stabilizujące chromosomy
i ulegające skróceniu z każdym cyklem komórkowym]
powt.
mikrosatelitarne
(200 000 loci u człowieka)
wlk. powtórzenia 2-6 pz, ilość powtórzeń 10-100
loci rozproszone, powstające w wyniku poślizgu replika-
cyjnego lub nierównego c-o
powt.
specyficzne dla chromosomu
– malowanie chromosomów


powt. rozproszone -
elem. ruchome, transpozony
stanowią 20-40% genomu, czasem >60%
rozproszone między genami, wewnątrz intronów, UTRów i
sekwencji kodujących
klasa I - retro(trans)pozony - przeniesienie wymaga
transkrypcji + odwrotnej transkrypcji + integracji
tak powstałej kopii
(kopiuj poprzez RNA i cDNA & wklej)
SINEs (short interspersed elements)
rodzina Alu - ~ 300 bp x >10
6
kopii w genomie człowieka
rodzina Alphoid - heterochromatyna centromerowa, 170bp x ~10
3
kopii
LINEs (long interspersed elements)
rodzina LI – 6400 bp x 10
4
kopii
ε
G
γ
A
γ
δ
β
ψβ
2
ψβ
1
Alu
repeats
LINEs
10 kb
elem.
Alu
i
LINE
w regionie
genów
ß-globiny
na chromosomie 11 człowieka
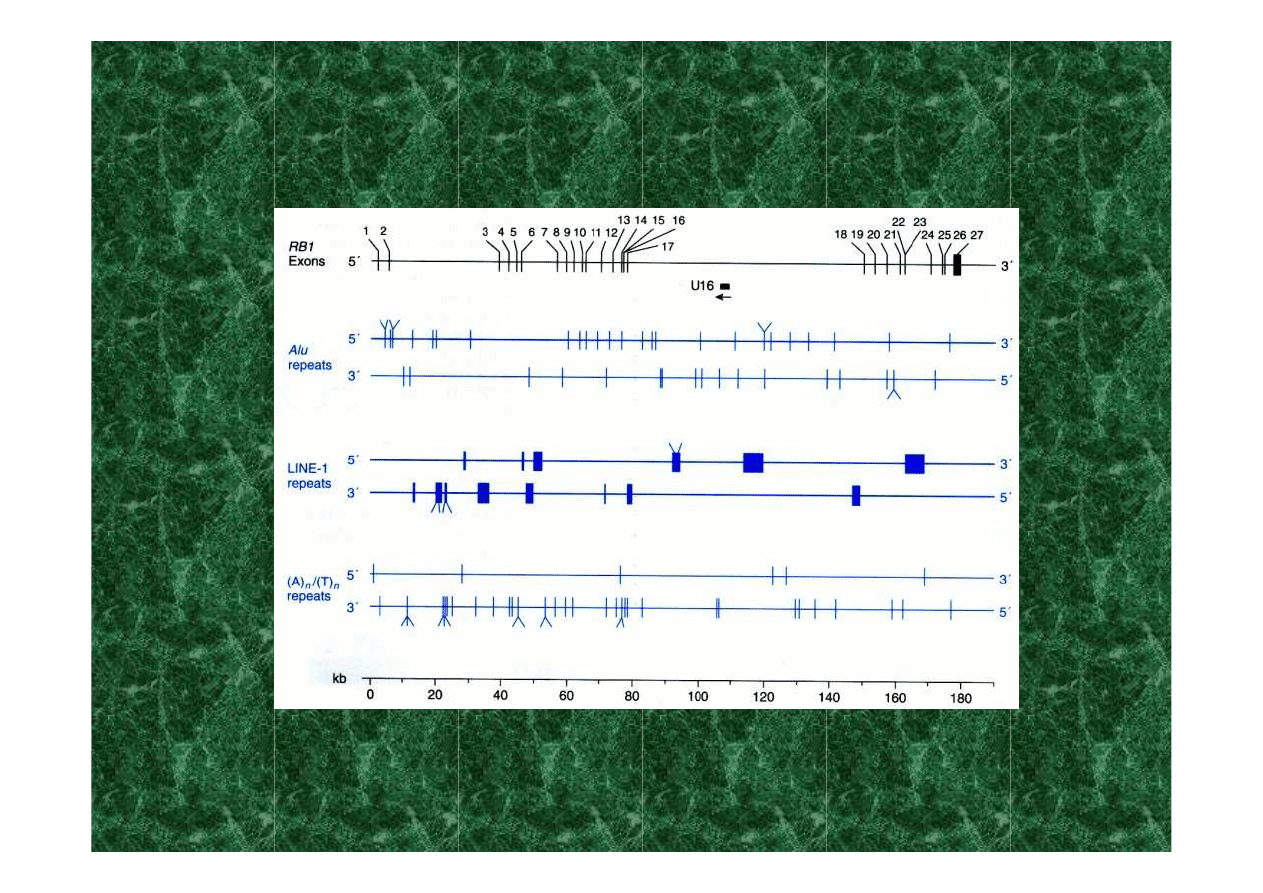
powtórzone elementy rozproszone w lokus genu retinoblastomy

powt. rozproszone -
elem. ruchome, transpozony
stanowią 20-40% genomu, czasem >60%
rozproszone między genami, wewnątrz intronów, UTRów i
sekwencji kodujących
klasa I - retro(trans)pozony - przeniesienie wymaga
transkrypcji + odwrotnej transkrypcji + integracji
tak powstałej kopii
(kopiuj poprzez RNA i cDNA & wklej)
SINEs (short interspersed elements)
rodzina Alu - 200 ~ 300 bp x >10
6
kopii w genomie człowieka
rodzina Alphoid - heterochromatyna centromerowa, 170bp x ~10
3
kopii
LINEs (long interspersed elements)
rodzina LI – 6400 bp x 10
4
kopii
klasa II - transpozony DNA – przeniesienie wymaga
(i) wycięcia z dotychczasowego locus + integracji w nowym miejscu
(wytnij & wklej)
lub (ii) replikacji + integracji kopii w nowym miejscu (kopiuj & wklej)
duplikacje
genów prowadzą do powstawania:
rodzin genów
- rodzin wariantów genów
zróżnicowanych w procesie ewolucji,
rozproszonych lub zgrupowanych
w klastery
pseudogenów
- niefunkcjonalnych kopii genów lub fragmentów genów
powstałych podczas ekspansji rodziny genowej lub retrotranspozycji;
mogą być pozbawione intronów; zawierają insercje, delecje, mutacje
nonsens, zwykle nie są transkrybowane
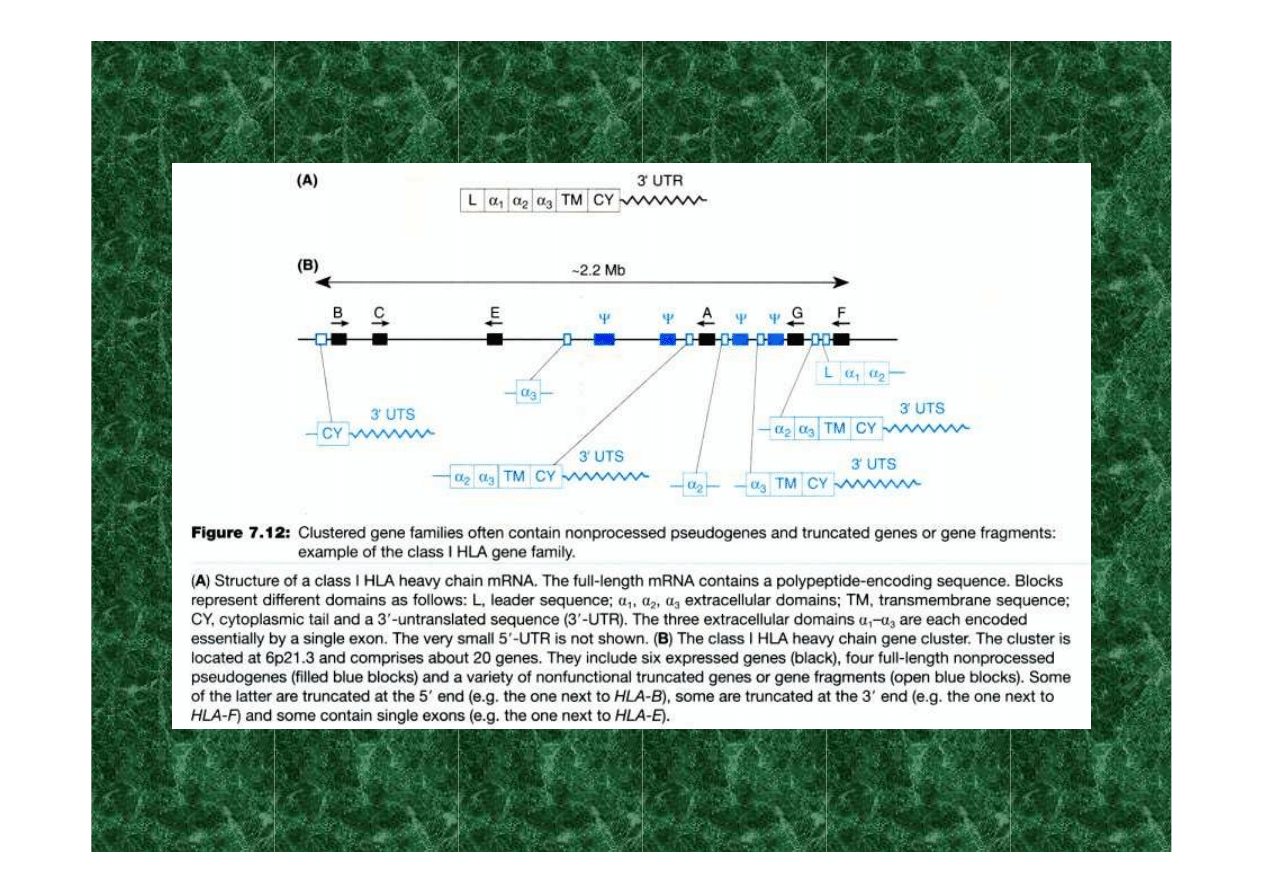
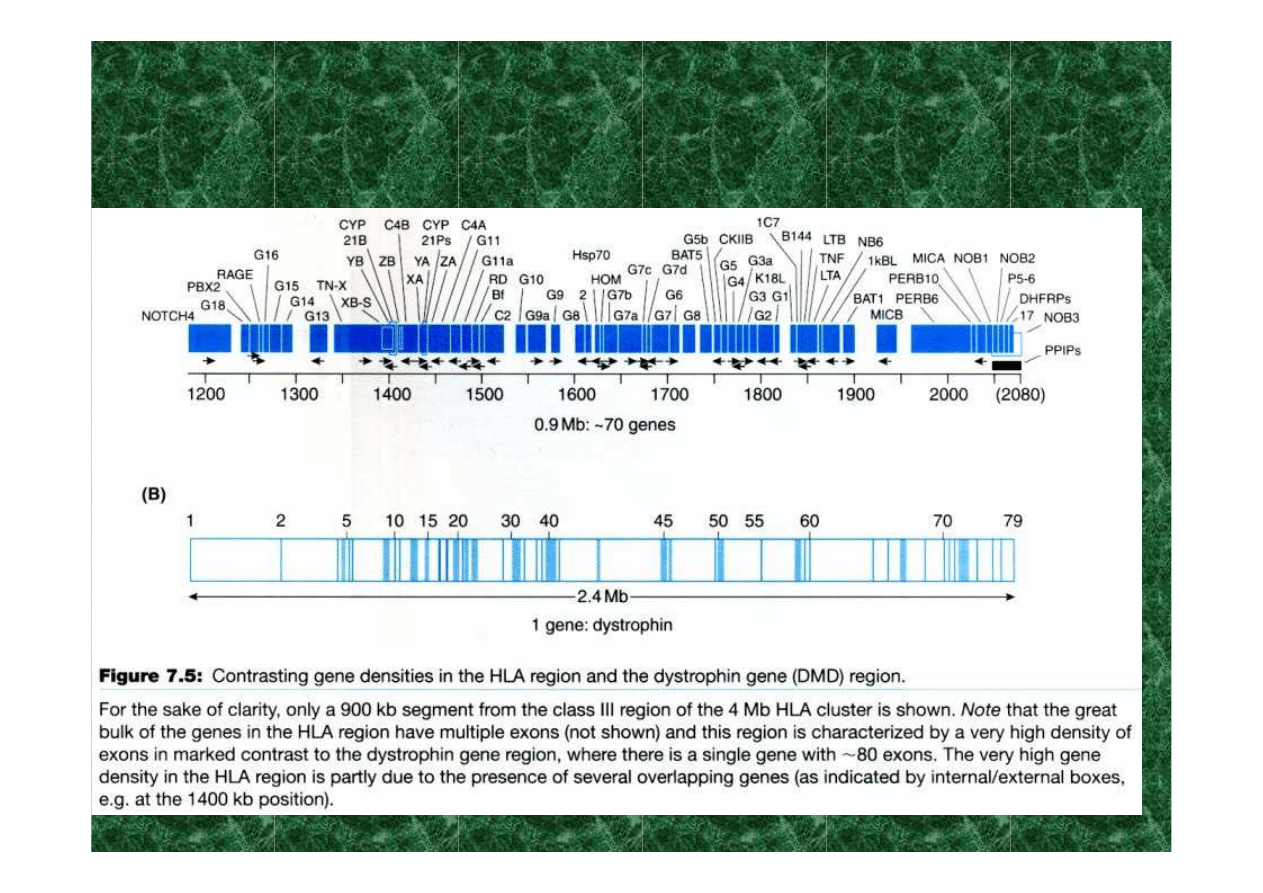
geny i pseudogeny w klasterze genów HLA klasy I
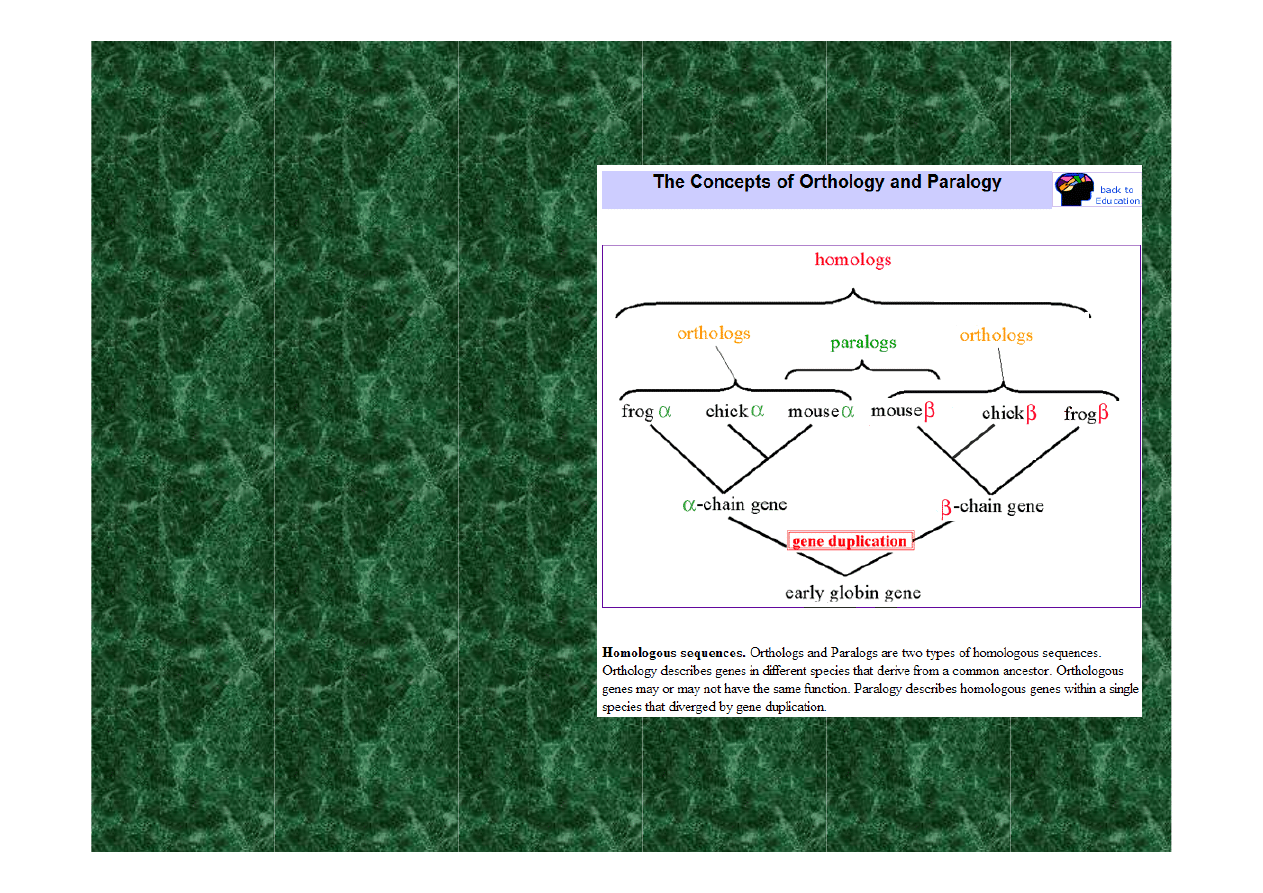
ortologi
– homologiczne
geny występujące
u różnych gatunków
o dużym stopniu
homologii świadczącym
o wspólnym genowym
pochodzeniu,
rozdzielone na skutek
specjacji
paralogi
- homologiczne
geny w tym samym
genomie, pochodzące
od wspólnego przodka,
rozdzielone w wyniku
duplikacji genu
http://angel.elte.hu/~fij/homepage/okt/gbi/kieg/img/ncbi-homolog-ortholog-paralog.gif
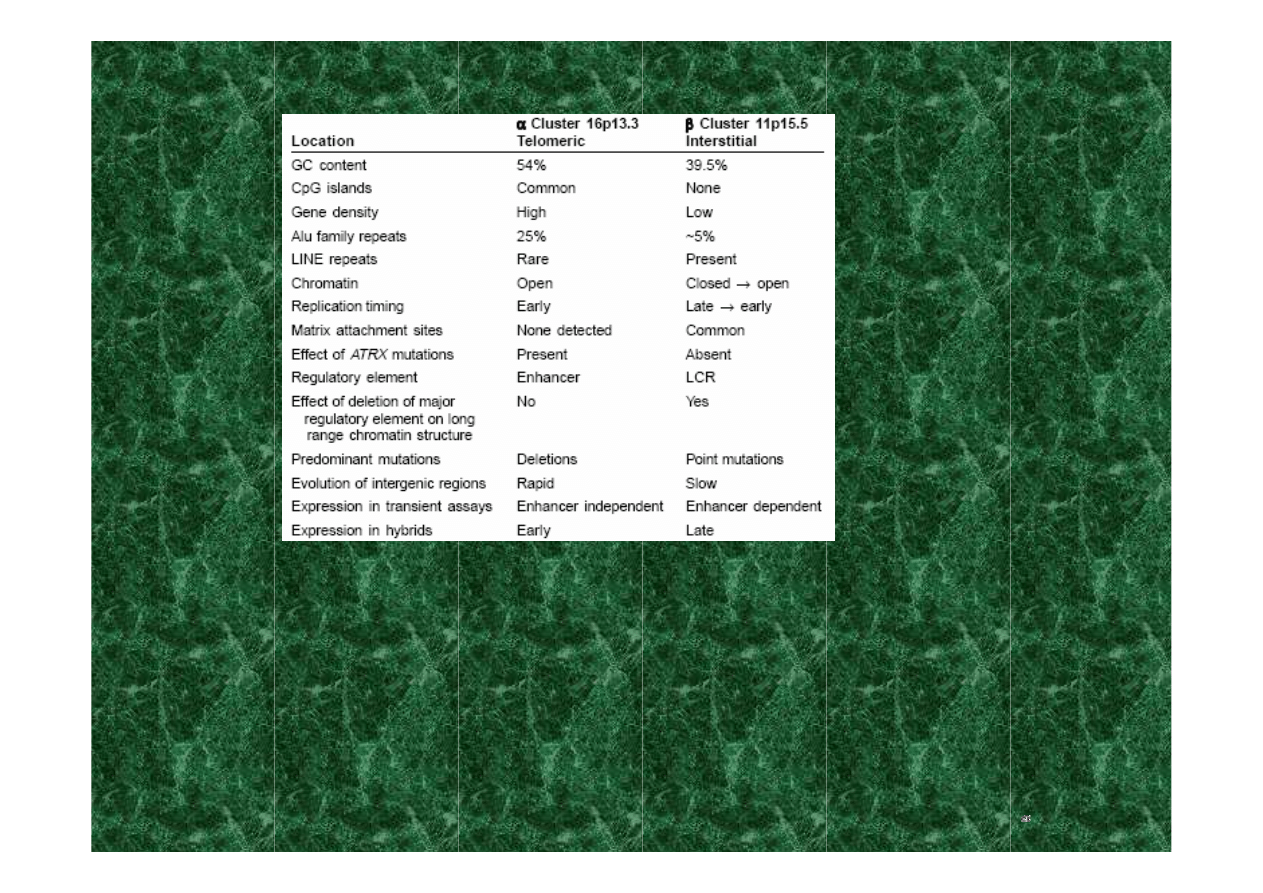
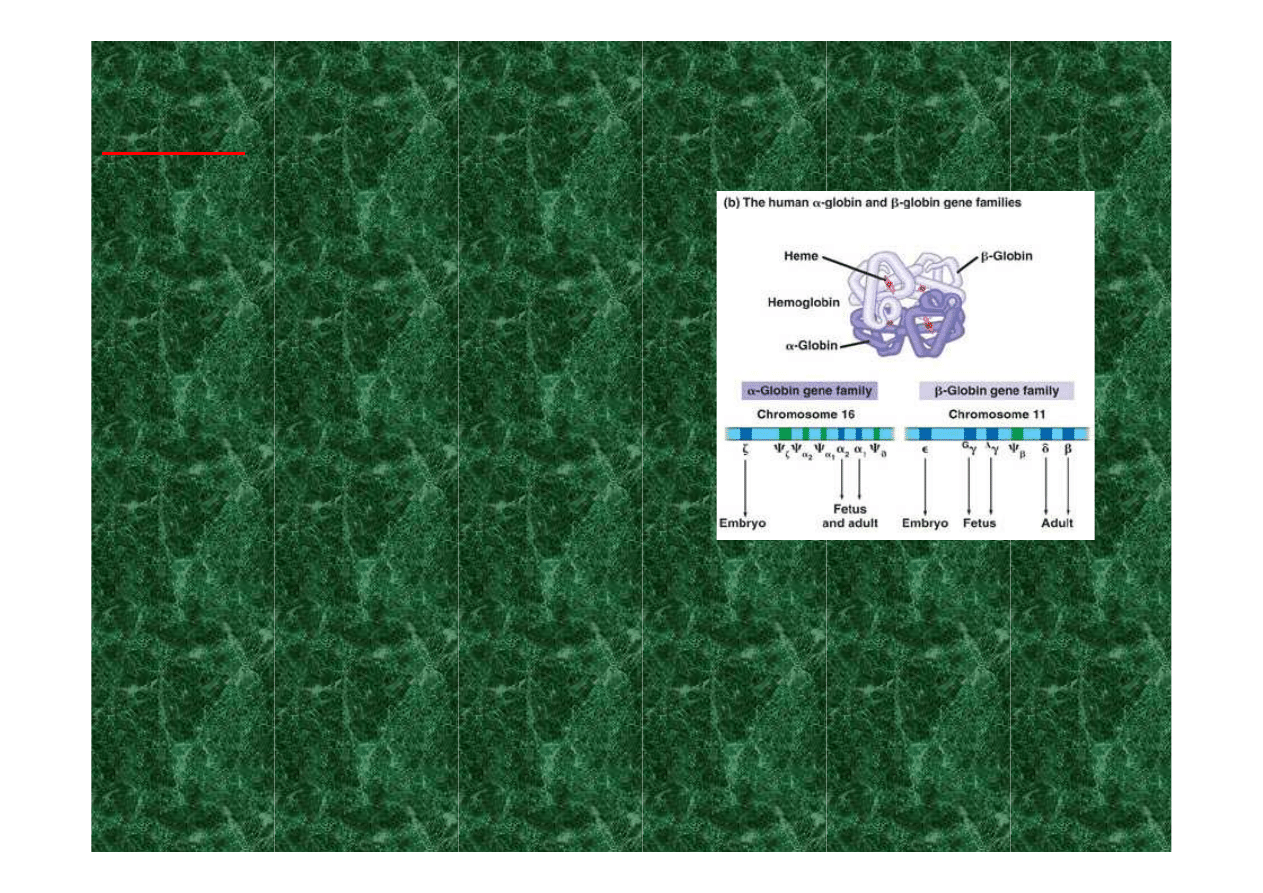
porównanie rodzin paralogicznych genów α- i β-globiny
człowieka sugeruje, że są one strukturalnie różne pod
wieloma względami, różnice mogą wpływać na:
transkrypcję, naprawę DNA, rekombinację i replikację
Higgs 2004 Hematology Am Soc Hematol Educ Program 1: 1-13

Chromosomal distribution of the human histone gene family. Eleven clusters comprising a
total of about 60 histone genes are distributed over seven human chromosomes. The two
clusters on 6p contain the great majority of histone genes. Other clusters contain only one
or two of the histone gene subtypes.
Note
that identical histones can be specified by genes
on different chromosomes.
Strachan & Read 1999 Human molecular genetics 2
John Wiley & Sons, Inc., BIOS Scientific Publishers Ltd
Wyszukiwarka
Podobne podstrony:
P 10 id 343561 Nieznany
dodawanie do 10 4 id 138940 Nieznany
ldm rozmaite 10 id 264068 Nieznany
Dubiel LP01 MRS 10 id 144167 Nieznany
I CSK 305 10 1 id 208211 Nieznany
IMG 10 id 211085 Nieznany
na5 pieszak 03 02 10 1 id 43624 Nieznany
img 10 id 211004 Nieznany
cwicz 10 F id 124010 Nieznany
IMG 10 id 210949 Nieznany
Chemia 10 3 id 111757 Nieznany
IMG 10 id 210983 Nieznany
egzamin09 10 id 153651 Nieznany
ETI Semestr 5 inz 10 10 id 1644 Nieznany
DrgAE Wym 10 id 141914 Nieznany
dodawanie do 10 3 id 138939 Nieznany
czynniki biologiczne 10 id 6672 Nieznany
więcej podobnych podstron