
Biologia molekularna 4
Mutacje i naprawa DNA
Egbert
Piasecki
10-03-2014

Mutacje i naprawa DNA
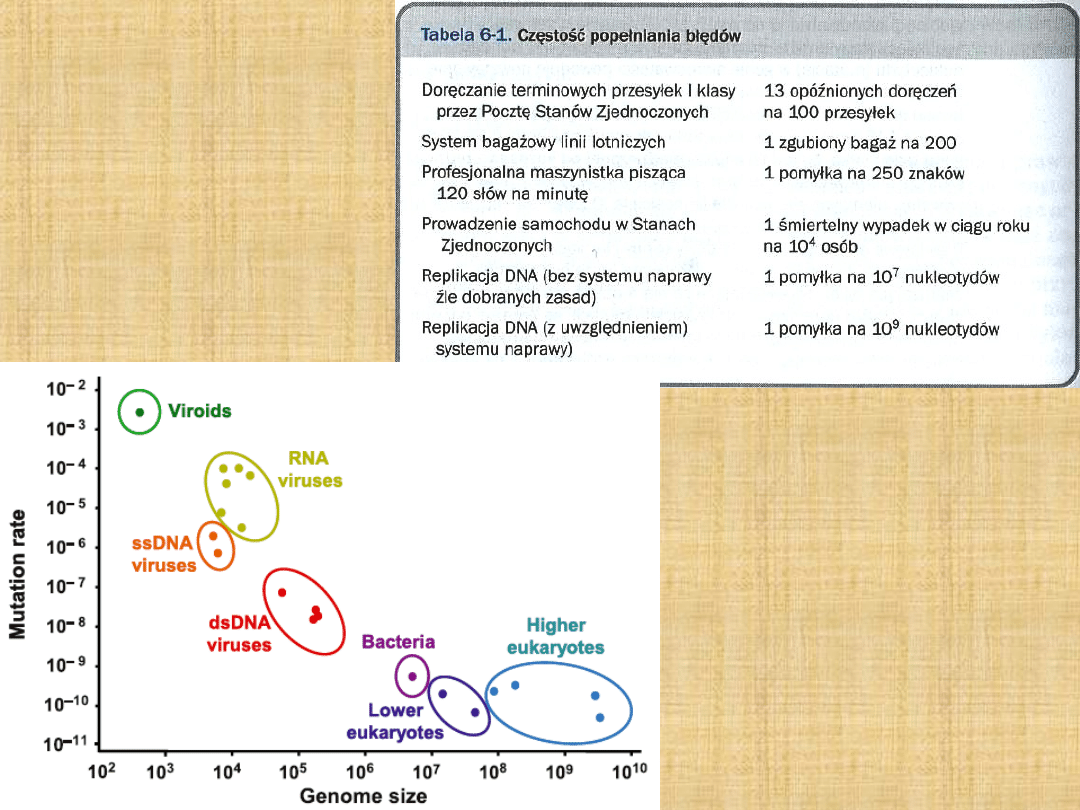
Błędy w sekwencji DNA powstałe w czasie
replikacji lub wskutek uszkodzeń –
mutacje
–
trwałe zmiany w DNA
Mutacje są zwykle niekorzystne, np. choroby
dziedziczne (mutacje w komórkach
płciowych), nowotwory (mutacje w
komórkach somatycznych)
System naprawy błędów
replikacji powstałych
mimo korekcyjnego działania aparatu
replikacyjnego – ang. DNA mismatch repair
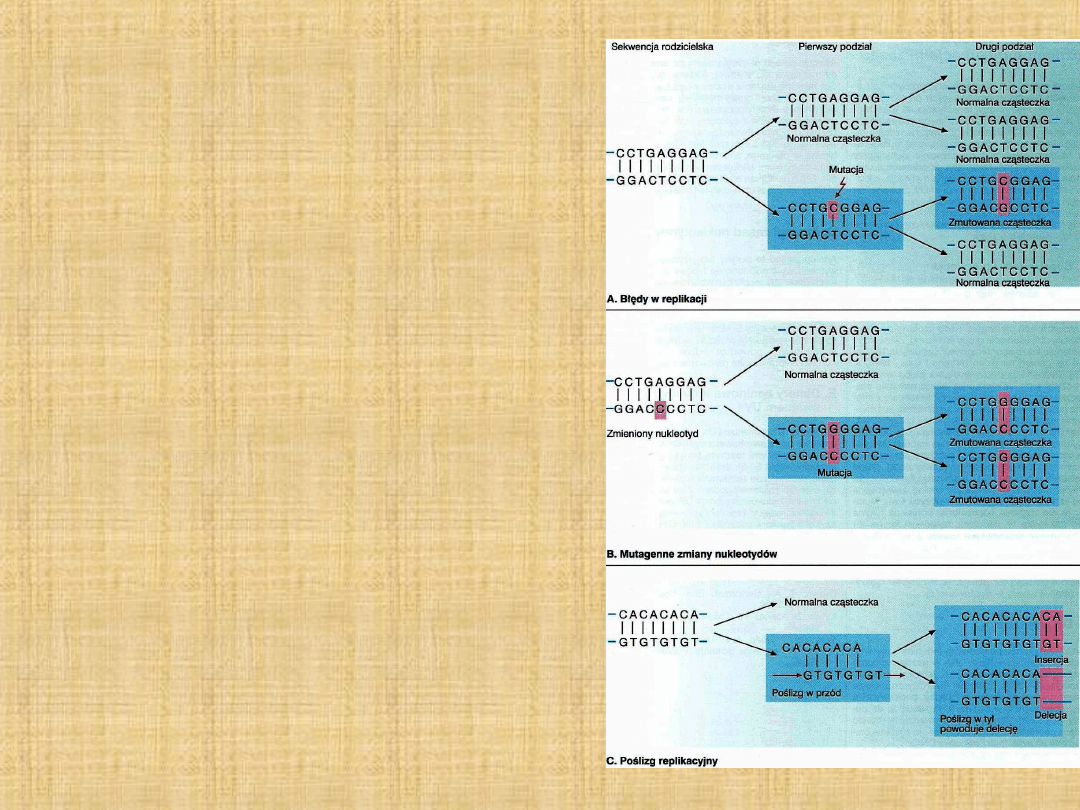
Mutacje
Mutacje
Mutacje
– stałe dziedziczne zmiany
w sekwencji zasad DNA
Mutacje powstają wskutek
spontanicznych błędów replikacji lub
działania czynników mutagennych
Mutacje punktowe:
–
Tranzycja
puryna puryna,
pirymidyna pirymidyna
–
Transwersja
puryna
pirymidyna,
pirymidyna puryna
Mutacja
missensowna
– zmiana sensu =
zmiana aminokwasu
Mutacja
nonsensowna
– powstanie
nowych kodonów stop
Insercje i delecje
często zmiana ramki
odczytu (mutacje „
frameshift
”)
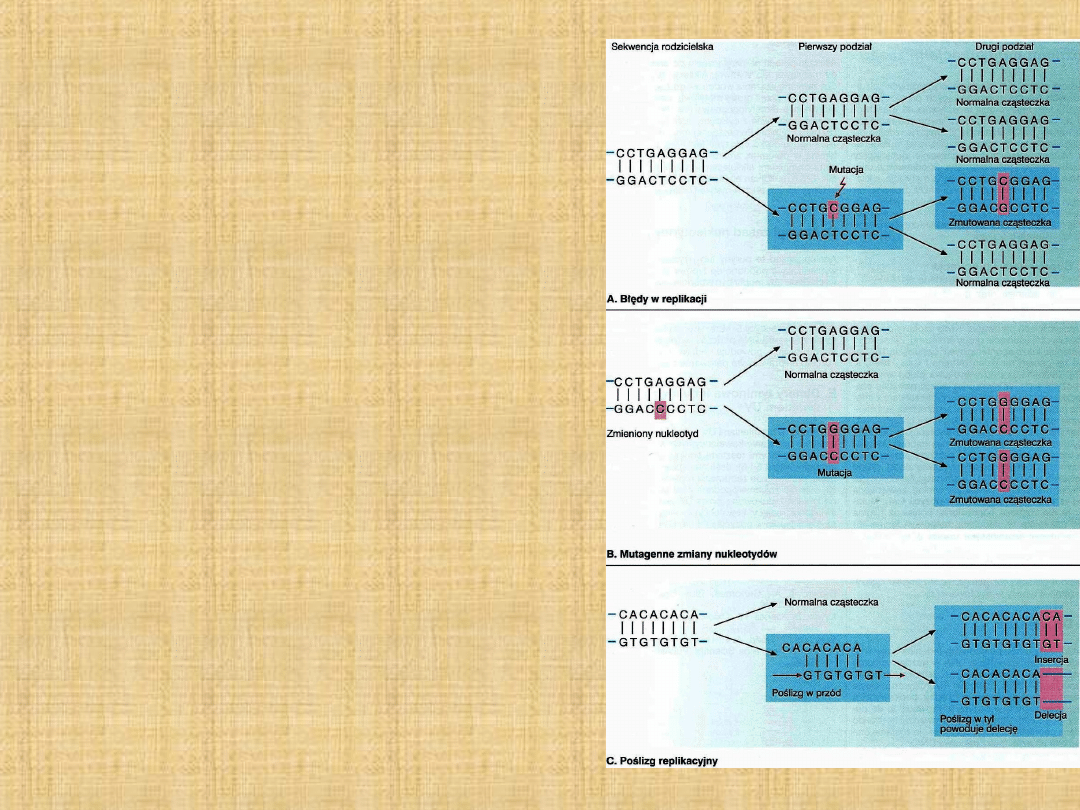
Mutacje
Mutacje ciche i nieletalne
polimorfizm
genetyczny
Uszkodzenia DNA – mutagenne i letalne
Mutageny chemiczne
– w większości
kancerogenne
Benzo[a]piren – wiązanie z guaniną
Aflatoksyna B1 – wiązanie kowalencyjne
z DNA
Interkalatory
– insercje i delecje
Poślizg replikacyjny
– związany z
powtórzeniami tandemowymi:
• poślizg w przód insercja
• poślizg w tył delecja
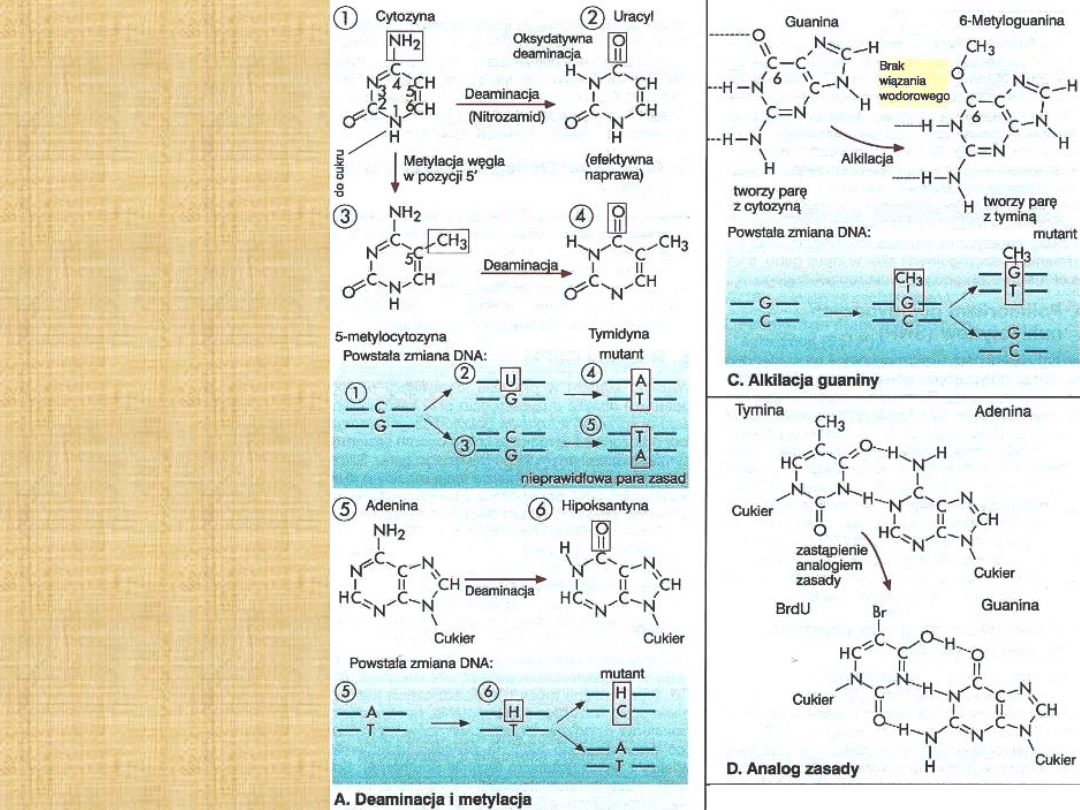
Mutacje
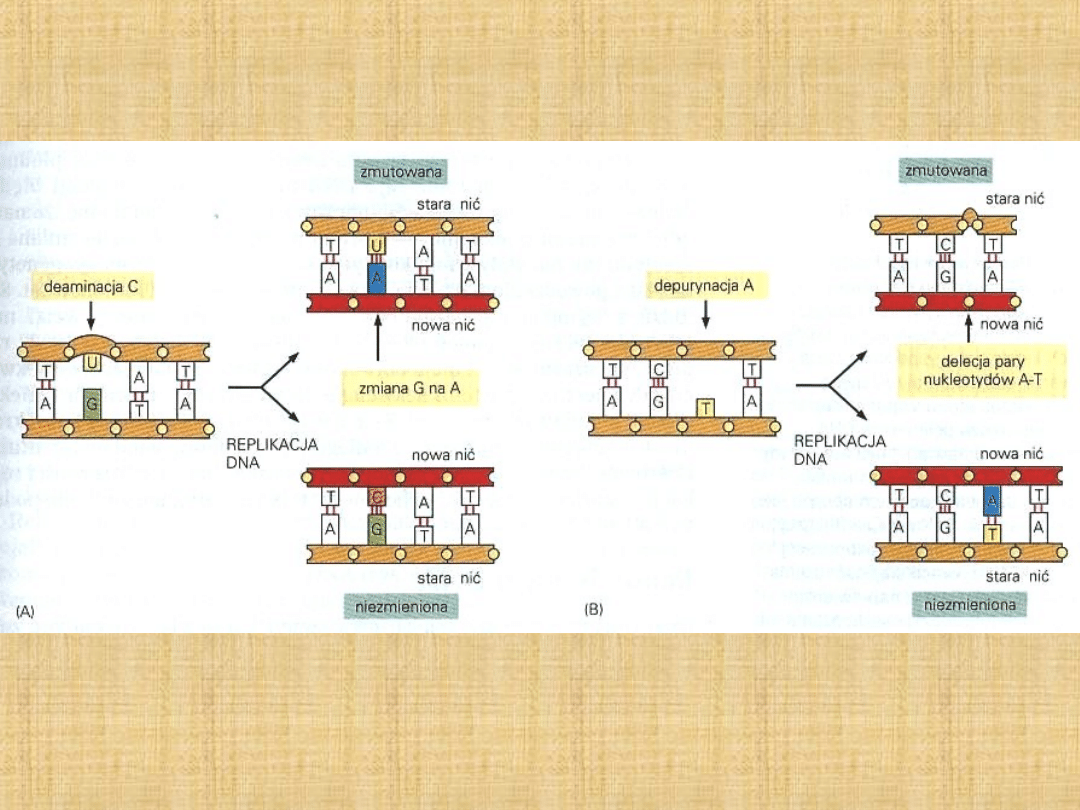
1.
Deaminacja
np.
cytozyny (powstaje
uracyl tranzycja
GCAT)
Deaminacja adeniny
do hipoksantyny
tranzycja ATGC
Deaminacja chemiczna
(kwas azotawy)
Deaminacja
spontaniczna: 100
zasad/genom/ dzień
2.
Alkilacja
–
przeszkadza w
rozplataniu dsDNA
3.
Analogi zasad
, np.
bromouracyl
kot
Mutacje
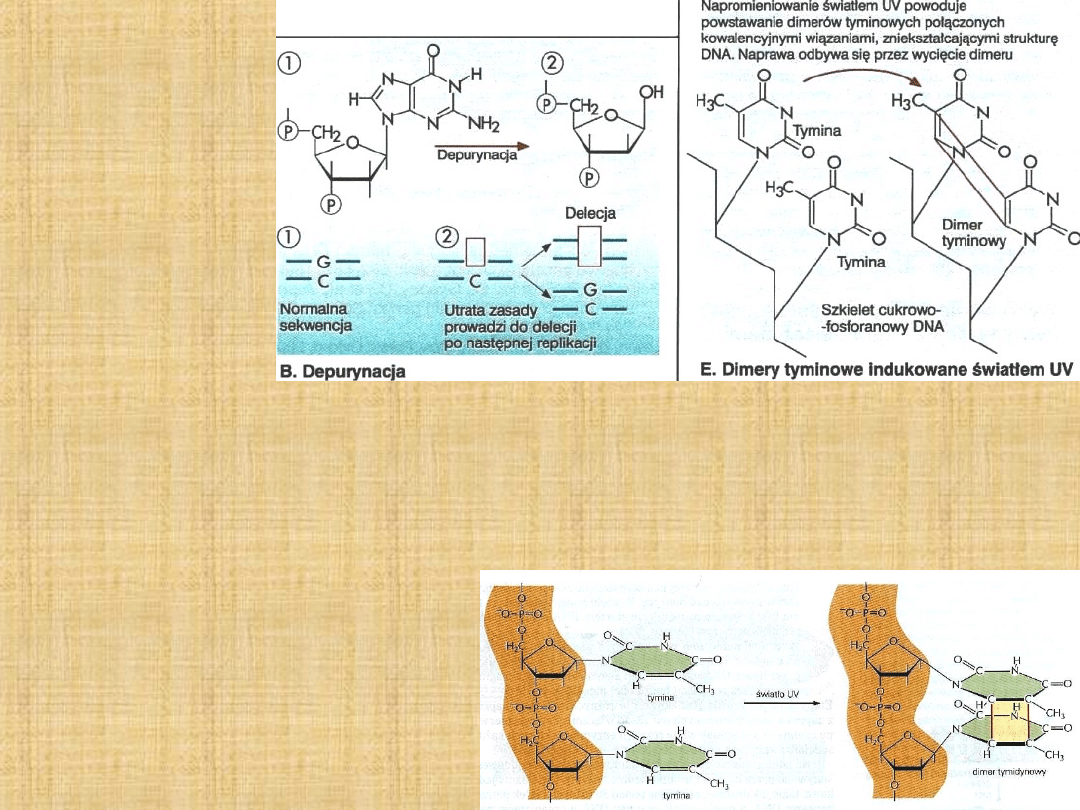
4.
Depurynacja
–
oderwanie guaniny
lub adeniny,
zmiana niekodująca
Częstość: 5000 zasad
dziennie/komórkę
Depirymidyzacja –
znacznie rzadsza
5.
Dimery pirymidynowe
. Promieniowanie UV powoduje tworzenie wiązań
kowalencyjnych sąsiednich pirymidyn, powstają np.
dimery tymidynowe
blokujące aparat replikacyjny, powstaje pierścień cyklobutanowy brak
możliwości parowania = lokalna denaturacja
kot
Mutacje
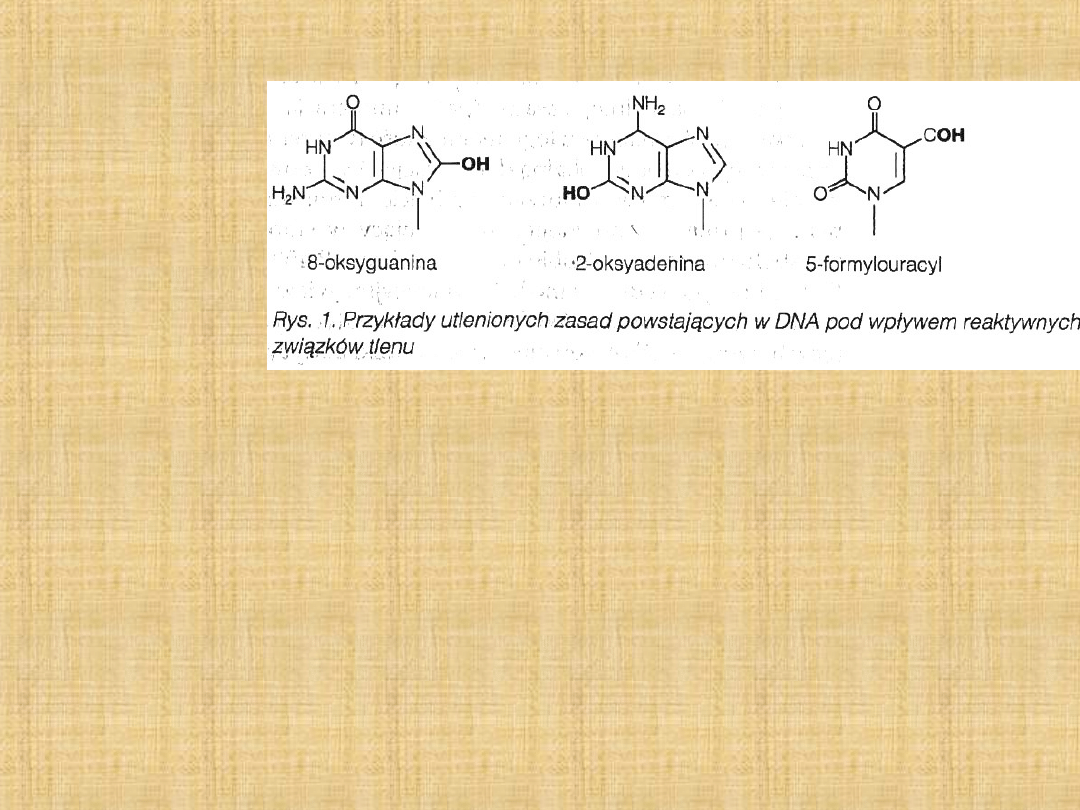
6.
Oksydacja
zasad
---------------------------------------------------------------------------------------------------------
Mutageneza
bezpośrednia
Mutageneza
pośrednia
– wprowadzanie błędnych zasad przez polimerazy w
miejscach uszkodzonych
Mutageneza specyficzna (ukierunkowana) – mutacje tylko na terenie
uszkodzenia
Mutageneza niespecyficzna (nieukierunkowana) – również w innych
miejscach genomu
Naprawa DNA
Konsekwencje braku naprawy uszkodzeń DNA:
Różne mechanizmy naprawcze z odrębnymi zestawami enzymów
Odróżnienie nici zmienionej od pierwotnej – zazwyczaj po zmianie
powstają struktury nietypowe dla normalnego DNA
Naprawa DNA
Naprawa:
1.
Usunięcie
fragmentu nici nowo zsyntetyzowanej
2.
Uzupełnienie
nowymi nukleotydami
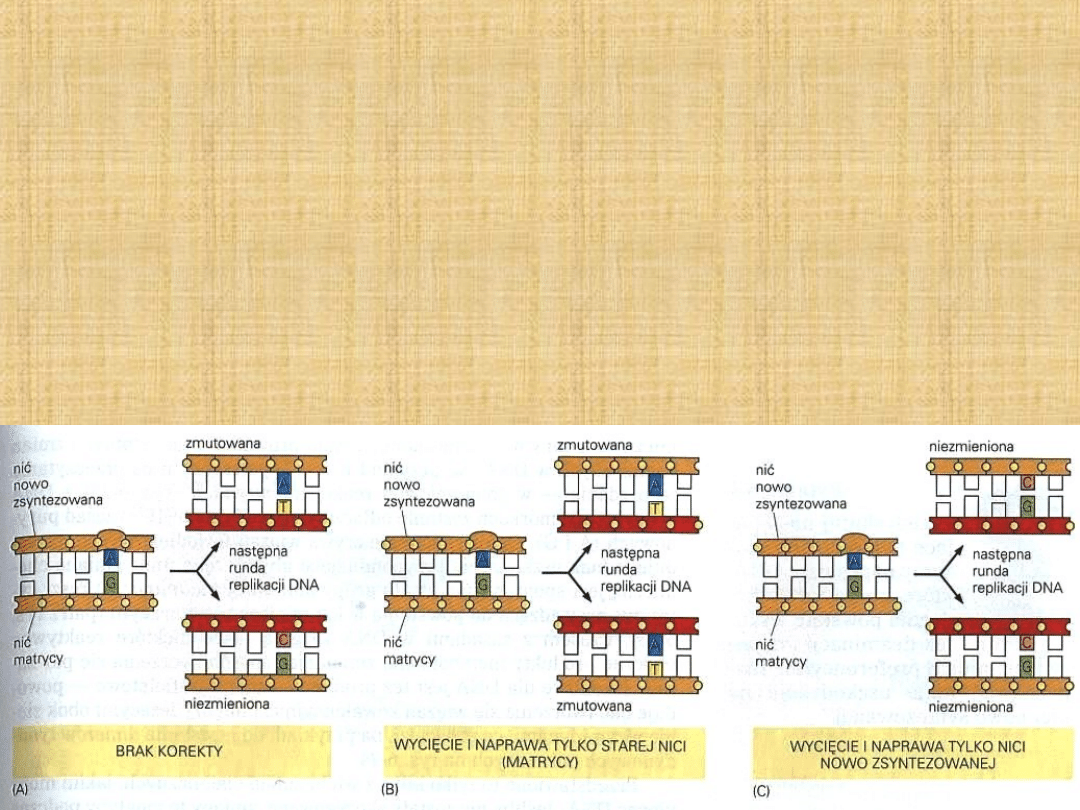
Nie wiadomo dokładnie jaki jest mechanizm odróżniania nici starej i nowej u
eukariontów. U prokariontów nić stara jest metylowana. Nici nowe są
preferencyjnie nacinane. Próba naprawy nici starej doprowadziłaby do
utrwalenia mutacji
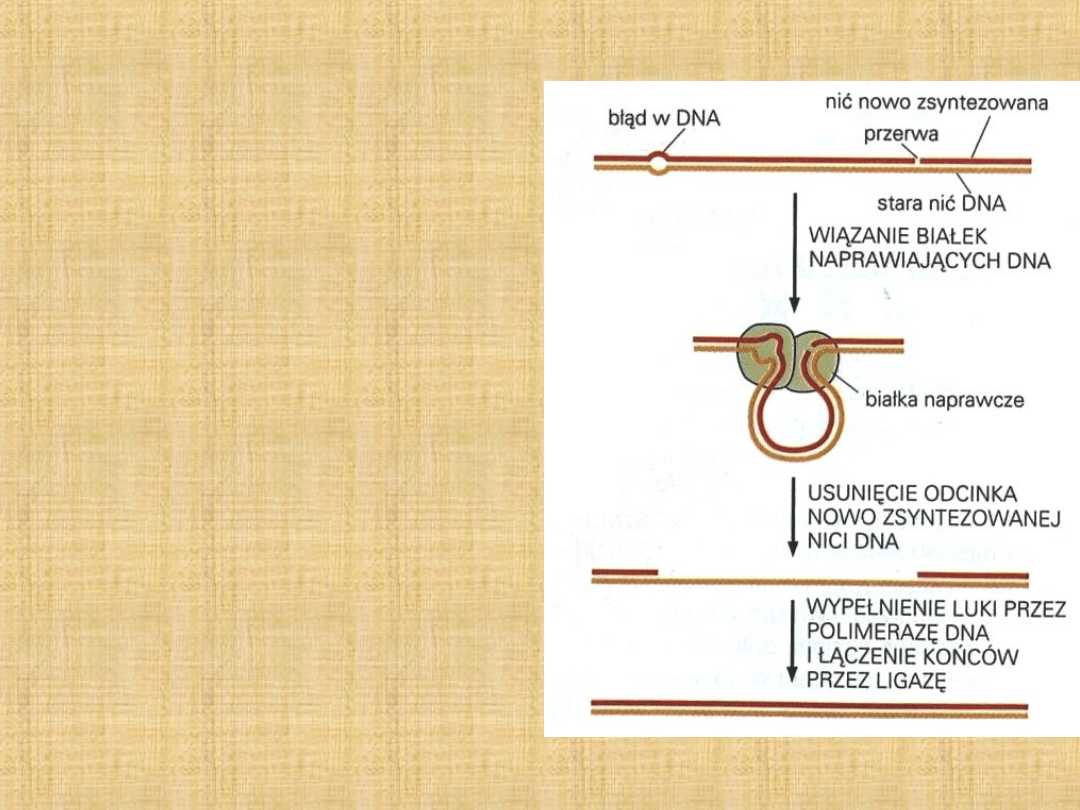
Naprawa DNA
Źle sparowane zasady powodują
zaburzenie geometrii
dwuniciowej helisy
rozpoznawane przez białka
naprawiające
Organizmy jednokomórkowe np.
drożdże mają >50 białek
związanych z naprawą DNA.
Organizmy wyższe – więcej
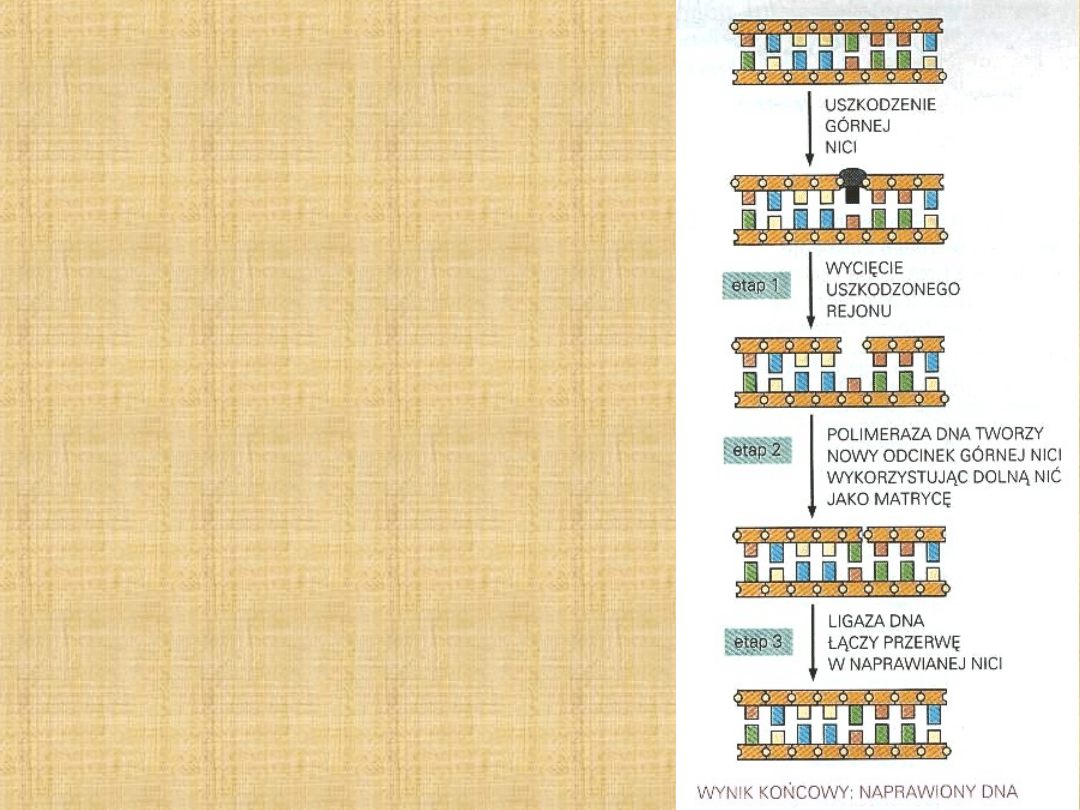
Naprawa DNA
Trzy etapy naprawy DNA:
1.
WYCINANIE
Rozpoznanie uszkodzonego fragmentu i jego
usunięcie przez nukleazę (różne nukleazy w
przypadku różnych uszkodzeń)
Usuwane jest od 1 nt (np. deaminacja
cytozyny) do 20 nt (np. dimery tymidyny)
2.
PONOWNA SYNTEZA
Wypełnienie luki przez naprawczą
polimerazę DNA (wydłuża łańcuch w
kierunku 5’3’, ma aktywność korekcyjną)
3.
LIGACJA
Łączenie łańcucha DNA przez ligazę
Etapy 2 i 3 zwykle jednakowe dla różnych
typów uszkodzeń
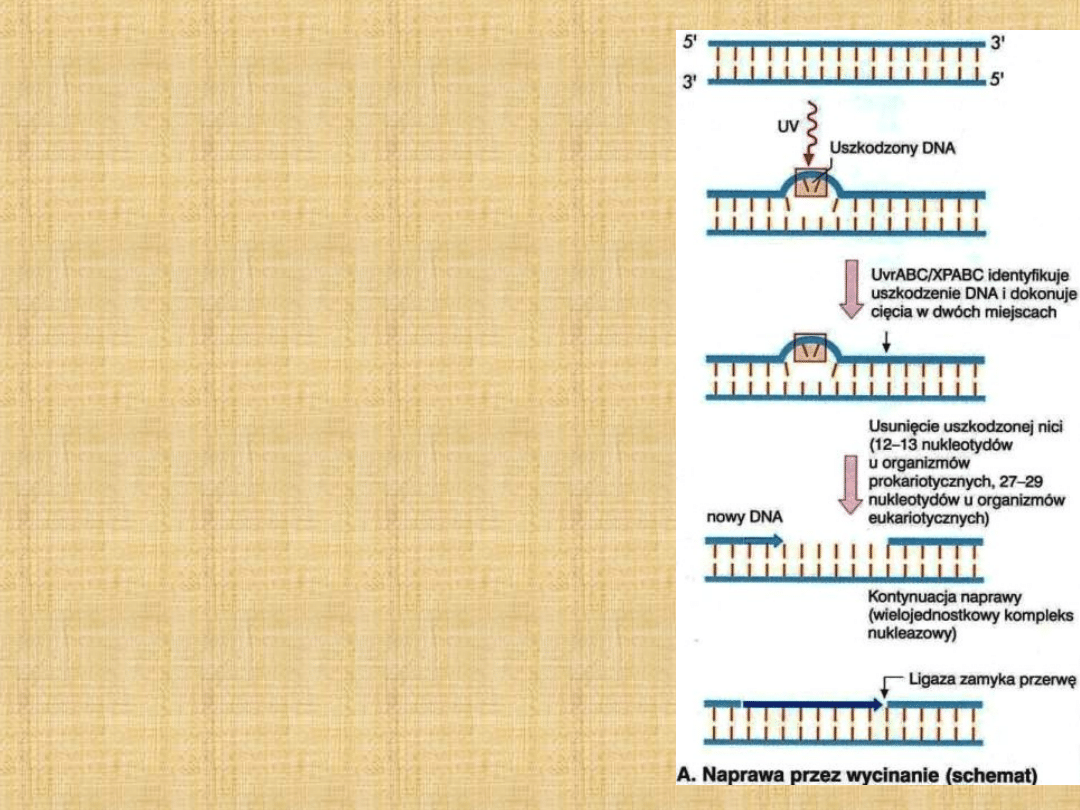
Naprawa DNA
1)
Naprawa błędów przez wycinanie
a)
NER
– nucleotide excision repair – wycięcie
nukleotydu
- endonukleaza wycina fragment obejmujący
uszkodzenie
b)
BER
– base excision repair – wycięcie zasady
- glikozydaza DNA wycina zasady (powstaje AP:
miejsce apurynowe lub apirymidynowe)
- endonukleaza AP rozcina i poszerza lukę (1-
kilka nt)
NER i BER u E. coli: polimeraza DNA I i ligaza DNA
U eukariontów: BER – polimeraza DNA , NER –
polimeraza DNA lub
W NER u eukariontów bierze udział co najmniej
18 czynników białkowych, w tym TFIIH (czynnik
transkrypcyjny).
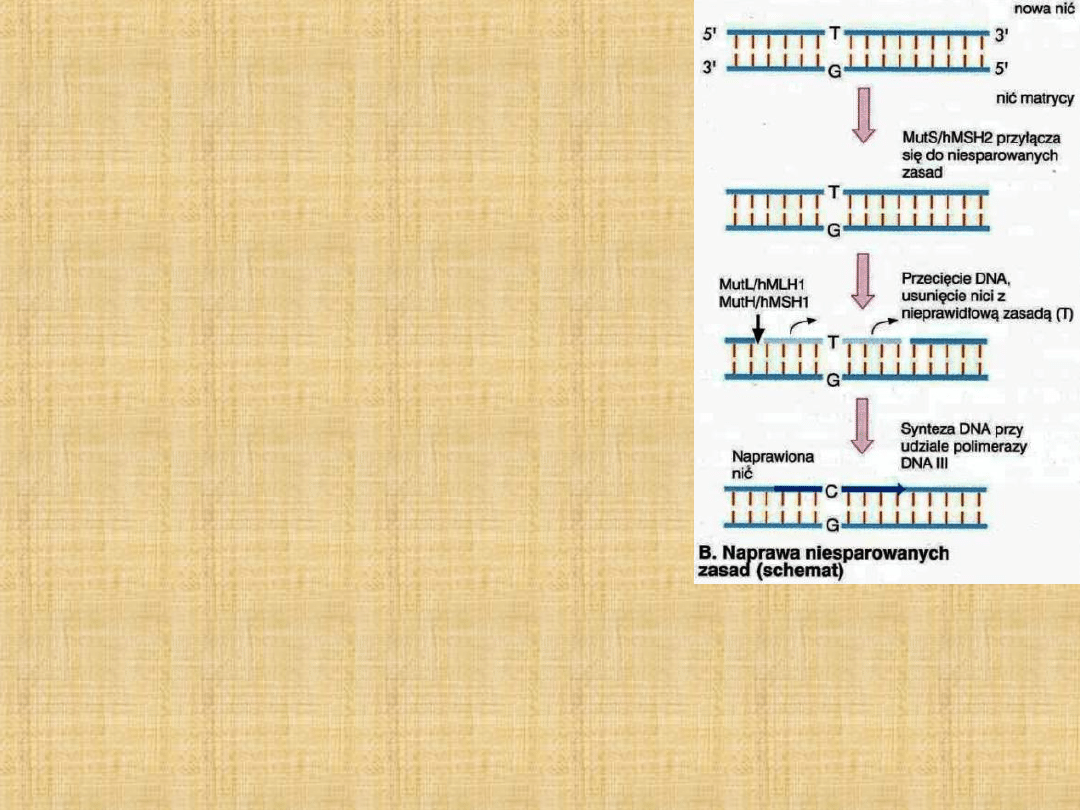
Naprawa DNA
2)
Naprawa żle sparowanych zasad
Po replikacji system naprawczy musi odróżnić nić starą
i nową. U prokariontów adenina jest metylowana w
sekwencji GATC. Opóźnienie metylacji nici potomnej
o kilka minut rozróżnienie nici [Nowo replikowany
DNA jest hemimetylowany]
Endonukleaza MutH nacina nić w pobliżu miejsca GATC
U eukariontów mechanizm nieznany
Brak funkcjonalnego enzymu systemu naprawy źle
sparowanych zasad dziedziczny niepolipowaty
rak okrężnicy
3)
Naprawa przez niehomologiczne łączenie końców (
NHEJ)
– nieligowalne
końce wskutek pęknięcia chromosomów, nieprawidłowego działania
topoizomeraz typu II, rearanżacja przeciwciał (VDJ). Uszkodzenia systemu
NHEJ wrażliwość na promieniowanie jonizujące, SCID

Naprawa DNA
4)
Fotoliazy
E. coli (enzymy fotoreaktywujące) – w obecności światła
widzialnego powodują ponowną monomeryzację dimerów
pirymidynowych bezpośrednie usuwanie uszkodzenia, nie generują
błędów
5)
Alkilotransferaza
E. coli (także ssaki) – usuwa grupy alkilowe, nie
generuje błędów
6)
Synteza DNA ignorująca uszkodzenie
– naprawa wymuszona przez
uszkodzenie (odpowiedź SOS) często mutageneza pośrednia
U prokariontów – kompleks polimerazy DNA V
U eukariontów – polimeraza DNA [zeta] (skłonna do robienia błędów)
–
polimeraza DNA [eta] (zwykle nie robi błędów)
Naprawa DNA
Mutacje w genach systemu naprawczego wzrost skłonności do
nowotworów
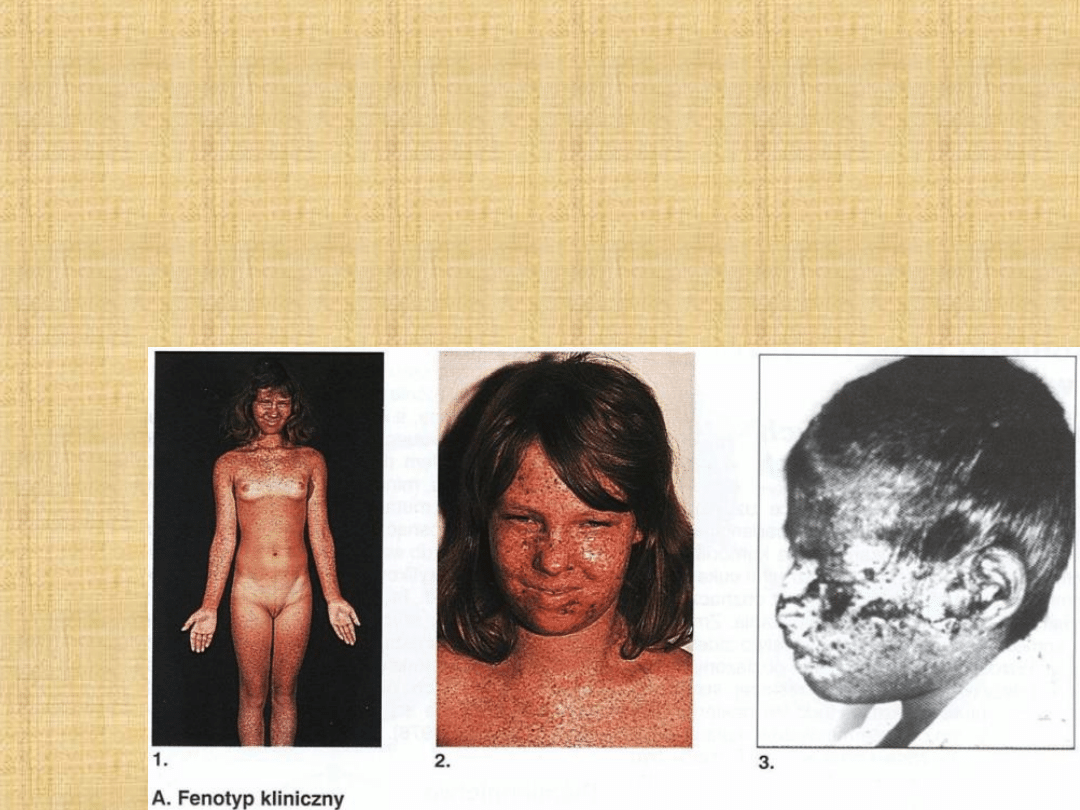
Xeroderma pigmentosum
(XP) – choroba autosomalna, recesywna,
skrajna wrażliwość na światło słoneczne i duża częstość
występowania raków skóry, wskutek braku naprawy dimerów
tymidynowych, wadliwego działania mechanizmu NER (uszkodzenia
różnych genów)
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
Wyszukiwarka
Podobne podstrony:
mutacje i naprawa DNA rozpiska, 08. MEDYCYNA, 1.Analityka medyczna, I rok, Genetyka, mutacje
Materiał genetyczny, mutacje, systemy naprawy DNA, test Amesa
Materiał genetyczny, mutacje, systemy naprawy DNA,
Mutacje i mechanizmy naprawy DNA
Materiał genetyczny, mutacje, systemy naprawy DNA, test Amesa
3 Systemy naprawcze w DNA
Uszkodzenia i naprawa DNA obieralny prof E Pastwa
biologia, NAPRAWA DNA, Mechanizmy naprawy oksydacyjnych uszkodzeń DNA
mutacjei naprawacdHuman Molecular Genetics 2
Naprawa DNA Biotechnologia e biotechnologia
Wpływ poszczególnych?fektów naprawy DNA w nowotworach i starzenia się był przedmiotem?dań
IG.4 - Uszkodzenia i naprawa DNA w komórkach nowotworowych, Genetyka, Inżynieria genetyczna
genetyka, Cwiczenie 8, Wrodzone zaburzenia naprawy DNA predysponujace do wystapienia nowotworów
MUTACJA, Genetyka, DNA, biologia molekularna, techniki
naprawa DNA
naprawa DNA
Mechanizm uszkodzenia i naprawy DNA, Patologia i choroby
więcej podobnych podstron