
Najczęstsze techniki
Najczęstsze techniki
molekularne stosowane
molekularne stosowane
w laboratorium
w laboratorium
klinicznym
klinicznym

Analiza DNA
Analiza DNA
Izolacja materiału genetycznego
Izolacja materiału genetycznego
Elektroforeza
Elektroforeza
Amplifikacja
Amplifikacja
Analiza Restrykcyjna
Analiza Restrykcyjna
Sekwencjonowanie
Sekwencjonowanie
itd
itd

W badaniach molekularnych stosuje
W badaniach molekularnych stosuje
się DNA (RNA) pozyskane z krwi
się DNA (RNA) pozyskane z krwi
obwodowej, komórek naskórka,
obwodowej, komórek naskórka,
komórek pozyskanych z płynu
komórek pozyskanych z płynu
owodniowego, kosmówki, włosów,
owodniowego, kosmówki, włosów,
nasienia….
nasienia….
W przypadku badań nowotworów
W przypadku badań nowotworów
stosuje się komórki uzyskane z
stosuje się komórki uzyskane z
biopsji tkanki nowotworowej.
biopsji tkanki nowotworowej.

Jakość wyniku zależy od jakości
Jakość wyniku zależy od jakości
materiału genetycznego użytego do
materiału genetycznego użytego do
badania.
badania.
Materiał genetyczny zdegradowany i
Materiał genetyczny zdegradowany i
zanieczyszczony nie nadaje się do
zanieczyszczony nie nadaje się do
dalszej analizy (brak wyniku).
dalszej analizy (brak wyniku).

Najwygodniejszym materiałem
Najwygodniejszym materiałem
wyjściowym do dalszej analizy
wyjściowym do dalszej analizy
molekularnej jest krew obwodowa.
molekularnej jest krew obwodowa.
Do wykonania pełnych badań
Do wykonania pełnych badań
diagnostycznych wystarczy 1ml
diagnostycznych wystarczy 1ml
pobranej na EDTA.
pobranej na EDTA.

Inne antykoagulanty mogą być
Inne antykoagulanty mogą być
stosowane pod warunkiem że nie
stosowane pod warunkiem że nie
interferują podczas dalszych prac z
interferują podczas dalszych prac z
materiałem genetycznym.
materiałem genetycznym.
(heparyna inhibuje niektóre enzymy
(heparyna inhibuje niektóre enzymy
restykcyjne)
restykcyjne)

Izolacja
Izolacja
Uzyskanie z maksymalną
Uzyskanie z maksymalną
wydajnością wysokocząsteczkowego
wydajnością wysokocząsteczkowego
materialu genetycznego, przy
materialu genetycznego, przy
jednoczesnym pozbawieniu
jednoczesnym pozbawieniu
preparatu zanieczyszczeń
preparatu zanieczyszczeń

Etapy izolacji
Etapy izolacji
Dezintegracja tkanek i rozbicie błon
Dezintegracja tkanek i rozbicie błon
komórkowych.
komórkowych.
•
Homogenizacja
Homogenizacja
•
Trawienie tkanek trypsyną
Trawienie tkanek trypsyną
•
Bufory lizujące
Bufory lizujące
•
Proteinaza K
Proteinaza K
•
SDS
SDS
•
Triton X / igepal
Triton X / igepal
•
Mocznik
Mocznik

Oczyszczenie preparatu z
Oczyszczenie preparatu z
białek
białek
•
Fenol - chloroform
Fenol - chloroform
•
Wysalanie 5 M NaCl
Wysalanie 5 M NaCl
•
Związanie DNA z nośnikiem i
Związanie DNA z nośnikiem i
wypłukanie zanieczyszczeń
wypłukanie zanieczyszczeń

Pozyskiwanie DNA
Pozyskiwanie DNA
Ekstrakcja alkoholem 99.8%
Ekstrakcja alkoholem 99.8%
Ekstrakcja octanem amonu i
Ekstrakcja octanem amonu i
izopropanolem
izopropanolem

Ocena jakościowa i
Ocena jakościowa i
ilościowa
ilościowa
Stosunek absorbancji fal o długości
Stosunek absorbancji fal o długości
260 nm do 280 nm jest miarą
260 nm do 280 nm jest miarą
czystości uzyskanego roztworu DNA
czystości uzyskanego roztworu DNA
(A260/A280 ). Wartość współczynnika
(A260/A280 ). Wartość współczynnika
A260/A280 mieszcząca się w
A260/A280 mieszcząca się w
przedziale 1,7 - 2,0 świadczy o
przedziale 1,7 - 2,0 świadczy o
prawidłowej czystości roztworu DNA
prawidłowej czystości roztworu DNA

Stężenie dsDNA
Stężenie dsDNA
C(µg/ml) = (A260-A320) x 50 x R (rozcieńczenie)
C(µg/ml) = (A260-A320) x 50 x R (rozcieńczenie)
Stężenie ssDNA
Stężenie ssDNA
C(µg/ml) = (A260-A320) x 33 x R (rozcieńczenie)
C(µg/ml) = (A260-A320) x 33 x R (rozcieńczenie)
Stężenie RNA
Stężenie RNA
C(µg/ml) = (A260-A320) x 44 x R (rozcieńczenie)
C(µg/ml) = (A260-A320) x 44 x R (rozcieńczenie)

Elektroforeza w żelu agarozowym
Elektroforeza w żelu agarozowym
(0.8%) w obecności bromku etydyny
(0.8%) w obecności bromku etydyny
(0.5
(0.5
µ
µ
g/
g/
µ
µ
l)
l)
Porównanie intensywności świecenia
Porównanie intensywności świecenia
badanej próby z próbkami o znanym
badanej próby z próbkami o znanym
steżeniu – ocena ilościowa
steżeniu – ocena ilościowa
Porównanie do wzorca wielkości KBL-
Porównanie do wzorca wielkości KBL-
>50kpz DNA wysokoczasteczkowe
>50kpz DNA wysokoczasteczkowe

Elektroforeza
Elektroforeza
Elektroforeza jest zjawiskiem elektrokinetycznym,
w którym pod wpływem przyłożonego pola
elektrycznego przemieszczają się
makrocząsteczki obdarzone niezrównoważonym
ładunkiem elektrycznym. Prędkość
przemieszczania się naładowanej elektrycznie
makrocząsteczki zależy od jej ładunku, rozmiaru,
kształtu oraz oporów ruchu środowiska.
Wykorzystując te zależności można dokonać
szybkiej separacji różnych makrocząsteczek przy
zastosowaniu stosunkowo prostych urządzeń i
przy relatywnie niskim nakładzie kosztów.

Technika stosowana przy rozdziale ,
Technika stosowana przy rozdziale ,
analizie i oczyszczaniu kwasów
analizie i oczyszczaniu kwasów
nukleinowych i białek.
nukleinowych i białek.
DNA i RNA mają ładunek ujemny i zgodnie
DNA i RNA mają ładunek ujemny i zgodnie
z tym ładunkiem , nadawanym przez grupy
z tym ładunkiem , nadawanym przez grupy
fosforanowe , migrują w polu elektrycznym
fosforanowe , migrują w polu elektrycznym
w kierunku anody. Szybkość migracji
w kierunku anody. Szybkość migracji
zależy od wielkości i kształtu cząsteczki
zależy od wielkości i kształtu cząsteczki
(odwrotnie proporcjonalna do log
(odwrotnie proporcjonalna do log
10
10
pz)
pz)

Żele agarozowe
Żele agarozowe
Metoda szybka, prosta, powszechnie
Metoda szybka, prosta, powszechnie
stosowana. Zdolność rozdzielcza zależy od
stosowana. Zdolność rozdzielcza zależy od
wielkości porów w żelu , których rozmiary
wielkości porów w żelu , których rozmiary
są zdeterminowane przez stężenie
są zdeterminowane przez stężenie
agarozy.
agarozy.
1,4-2,0 % używa się do rozdziału
1,4-2,0 % używa się do rozdziału
cząsteczek mniejszych (0,5-2,0 kb).
cząsteczek mniejszych (0,5-2,0 kb).
0,5-0,7 % stosuje się do rozdziału
0,5-0,7 % stosuje się do rozdziału
cząsteczek większych (10-20 kb i więcej).
cząsteczek większych (10-20 kb i więcej).

Żele akrylamidowe
Żele akrylamidowe
Powstają przez polimeryzacje monomerów
Powstają przez polimeryzacje monomerów
akrylamidu w długie łańcuchy z
akrylamidu w długie łańcuchy z
wytworzeniem wiązań poprzecznych przez
wytworzeniem wiązań poprzecznych przez
bisakrylamid.
bisakrylamid.
Gęstość sieciowania oraz rozmiary porów
można regulować poprzez odpowiedni
dobór stężenia akrylamidu i bisakrylamidu
T [%] =((AA + bis-AA)[g]/ obj.[ml])x 100

Dopełniającym parametrem jest wagowy
stosunek ilości substancji sieciującej do
sumyakrylamidu i substancji sieciującej:
C [%] = (bis-AA [g] / (AA + bis-AA) [g]) x 100
Ze wzrostem wartości T maleje średni
rozmiar porów. Natomiast minimalny rozmiar
porów, przy zadanej wartości T, uzyskuje się
dla wartości C = 5%. Powyżej i poniżej tej
wartości rozmiary porów wzrastają.

Akrylamid w postaci monomerycznej
jest bardzo silną neurotoksyną i
nawet po procesie polimeryzacji
stanowi poważne zagrożenie dla
zdrowia ze względu na pozostałości
swobodnych monomerów w objętości
żelu.

Wolnorodnikową reakcje
polimeryzacji, można zainicjować
chemicznie lub fotochemicznie. Przy
chemicznej inicjacji procesu
najczęściej stosuje się nadsiarczan
amonu (APS) lub nadsiarczan potasu
(PERS) w obecności katalizatora
N,N,N.,N.-tetrametyletylenodiaminy
(TEMED).

Fotochemiczne wyzwolenie procesu
polimeryzacji zachodzi w obecności
ryboflawiny pod działaniem
długofalowego światła UV i jest
katalizowane przez TEMED.

Żele denaturujące
Żele denaturujące
Szybkość migracji w żelu jednoniciowych
Szybkość migracji w żelu jednoniciowych
cząsteczek , zależy nie tylko od ich
cząsteczek , zależy nie tylko od ich
wielkości i ładunku ale także w dużym
wielkości i ładunku ale także w dużym
stopniu od ich struktury przestrzennej (w
stopniu od ich struktury przestrzennej (w
przypadku dwuniciowych cząsteczek DNA
przypadku dwuniciowych cząsteczek DNA
nie ma to większego znaczenia) aby
nie ma to większego znaczenia) aby
dokładnie określić ich wielkość należy
dokładnie określić ich wielkość należy
wyeliminować różnice w szybkości migracji
wyeliminować różnice w szybkości migracji
wywołane różną konformacją cząsteczki.
wywołane różną konformacją cząsteczki.
Jest to szczególnie ważne w przypadku
Jest to szczególnie ważne w przypadku
jednoniciowego RNA , które tworzy
jednoniciowego RNA , które tworzy
miejscowe struktury dwuniciowe.
miejscowe struktury dwuniciowe.

Najczęściej używanymi czynnikami
Najczęściej używanymi czynnikami
denaturującymi są : formaldehyd i
denaturującymi są : formaldehyd i
glioksal w żelach agarozowych (przy
glioksal w żelach agarozowych (przy
analizie preparatów RNA) lub
analizie preparatów RNA) lub
mocznik w żelach
mocznik w żelach
poliakrylamidowych (przy analizie
poliakrylamidowych (przy analizie
jednoniciowych fragmentów DNA).
jednoniciowych fragmentów DNA).

Ze względu na sposób umieszczenia
nośnika elektroforetycznego można
wyróżnić elektroforezę kapilarną
(ang. capillary electrophoresis),
elektroforezę pionową (ang. Vertical
electrophoresis) oraz elektroforezę
poziomą (ang. horizontal
electrophoresis).

W elektroforezie kapilarnej- HPCE (ang. high
performance capillary electrophoresis) elektrolit
wypełnia kapilaręo wewnętrznej średnicy 50 -
100 μm i długości 20-30 cm. Oba końce kapilary
zanurzone są w zasobnikach z odpowiednimi
elektrolitami. Sama kapilara wypełniona jest
swobodnym elektrolitem (elektroforeza
swobodna) lub porowatym nośnikiem
(elektroforeza na nośniku). Do obu końców
kapilary przykłada się wysokie napięcie co
skutkuje pojawieniem się we wnętrzu kapilary
pola elektrycznego o wartości natężenia około
1kV/cm i prądu rzędu 10 mA.

Przeznaczeniem elektroforezy kapilarnej są
rozdziały analityczne i mikropreparatywne. Ilość
nanoszonego materiału do analizy jest bardzo
niewielka - rzędu pojedynczych nanogramów.
Objętość aplikowanej próbki waha się zwykle w
przedziale 2-4 nl (2-4x10-9 l). Czas rozdziału
jednej próbki wynosi około 10-20 minut. Detekcja
rozdzielonych grup makrocząsteczek realizowana
jest u ujścia kapilary przy pomocy detektora o
konstrukcji zbliżonej do przepływowego detektora
stosowanego w chromatografii cieczowej.

Elektroforeza pionowa - jest
najpowszechniej stosowną techniką.
W technice tej nośnik
elektroforetyczny wypełnia szklane
rurki (elektroforeza rurkowa) lub
znajduje się pomiędzy dwoma
płytkami rozdzielonymi przekładkami
dystansowymi (ang. Spacer-
elektroforeza płytowa).
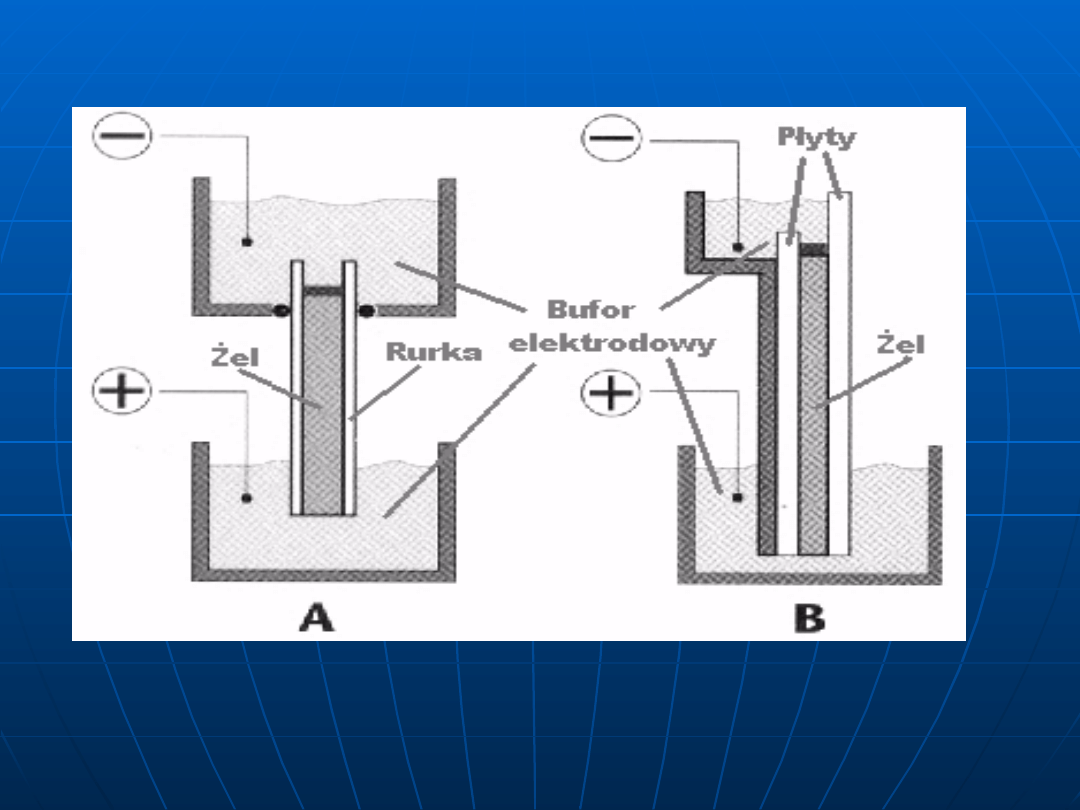

Elektroforeza pozioma - jest rodzajem
elektroforezy w którym nośnik umieszczony jest w
płaszczyźnie poziomej. Zaletą tego rozwiązania
jest brak wycieków elektrolitu oraz możliwość
łatwego odprowadzenia ciepła generowanego w
trakcie przepływu prądu.
Elektroforeza półsucha, w której bufory
elektrodowe zamknięte są w zestalonej agarozie
lub w warstwach bibuły filtracyjnej, pozwala
ponadto na znaczne ograniczenie zużycia
chemikaliów niezbędnych do przygotowania
buforów.


Elektroforeza w polu
Elektroforeza w polu
pulsacyjnym
pulsacyjnym
Stosowana do rozdzielenia dużych cząsteczek
Stosowana do rozdzielenia dużych cząsteczek
DNA - od 20 kb do 10 Mb. Można w ten sposób
DNA - od 20 kb do 10 Mb. Można w ten sposób
rozdzielić całe chromosomy np. drożdżowe. Pole
rozdzielić całe chromosomy np. drożdżowe. Pole
elektryczne jest włączane i wyłączane w krótkich
elektryczne jest włączane i wyłączane w krótkich
odstępach czasu. Kiedy pole elektryczne jest
odstępach czasu. Kiedy pole elektryczne jest
włączone cząsteczki migrują zgodnie ze swoją
włączone cząsteczki migrują zgodnie ze swoją
wielkością a gdy zostaje wyłączone mają
wielkością a gdy zostaje wyłączone mają
tendencję do relaksacji i zwijania w przypadkowe
tendencję do relaksacji i zwijania w przypadkowe
pętle. Czas wymagany do relaksacji jest wprost
pętle. Czas wymagany do relaksacji jest wprost
proporcjonalny do długości cząsteczki. Następnie
proporcjonalny do długości cząsteczki. Następnie
kierunek pola elektrycznego jest zmieniany o 90
kierunek pola elektrycznego jest zmieniany o 90
lub 180 stopni w stosunku do poprzedniego.
lub 180 stopni w stosunku do poprzedniego.
Dłuższe cząsteczki zaczynają poruszać się wolniej
Dłuższe cząsteczki zaczynają poruszać się wolniej
niż krótsze. Powtarzające się zmiany kierunku
niż krótsze. Powtarzające się zmiany kierunku
pola stopniowo powodują rozdzielenie.
pola stopniowo powodują rozdzielenie.

Detekcja
Detekcja
Barwniki inerkalujące (EtBr)
Barwniki inerkalujące (EtBr)
Barwienie srebrem
Barwienie srebrem
Autoradiografia
Autoradiografia
Detekcja laserowa
Detekcja laserowa
Immunoznakowanie
Immunoznakowanie

PCR
PCR
PCR - reakcja łańcuchowa polimerazy (ang.
polymerase chain reaction) polega na
przeprowadzeniu wielu cykli syntezy DNA z
wykorzystaniem starterów flankujących
określony odcinek DNA o długości od
kilkuset do kilku tysięcy nukleotydów. Jest to
metoda alternatywna do klonowania i może
być używana w celu amplifikacji nawet
bardzo rzadkich sekwencji w mieszaninie
ale warunkiem jest znajomość sekwencji
otaczającej powielany fragment. W praktyce
wystarczy tylko jedna cząsteczka matrycy.

DNA jest denaturowany termicznie w
DNA jest denaturowany termicznie w
temperaturze około 90°C.
temperaturze około 90°C.
Syntetyczne oligonukleotydy długości
Syntetyczne oligonukleotydy długości
mniej więcej 20 nukleotydów
mniej więcej 20 nukleotydów
komplementarne do 3` końców
komplementarne do 3` końców
kawałka DNA , którego powieleniem
kawałka DNA , którego powieleniem
jesteśmy zainteresowani , dodane są
jesteśmy zainteresowani , dodane są
w dużym nadmiarze molowym do
w dużym nadmiarze molowym do
zdenaturowanego DNA.
zdenaturowanego DNA.

Temperatura zostaje obniżona do 40-60°C.
Temperatura zostaje obniżona do 40-60°C.
DNA pozostaje zdenaturowany ponieważ
DNA pozostaje zdenaturowany ponieważ
komplementarne łańcuchy są w zbyt
komplementarne łańcuchy są w zbyt
niskiej koncentracji , w porównaniu z
niskiej koncentracji , w porównaniu z
ilością starterów , aby zrenaturować.
ilością starterów , aby zrenaturować.
Specyficzne oligonukleotydy hybrydyzują z
Specyficzne oligonukleotydy hybrydyzują z
komplementarnymi sekwencjami w DNA
komplementarnymi sekwencjami w DNA
(tzw. annealing) i służą jako startery do
(tzw. annealing) i służą jako startery do
syntezy DNA , którą prowadzi
syntezy DNA , którą prowadzi
termostabilna DNA polimeraza tzw.
termostabilna DNA polimeraza tzw.
Taq
Taq
polimeraza izolowana z bakterii
polimeraza izolowana z bakterii
Thermus
Thermus
aqaticus
aqaticus
.
.
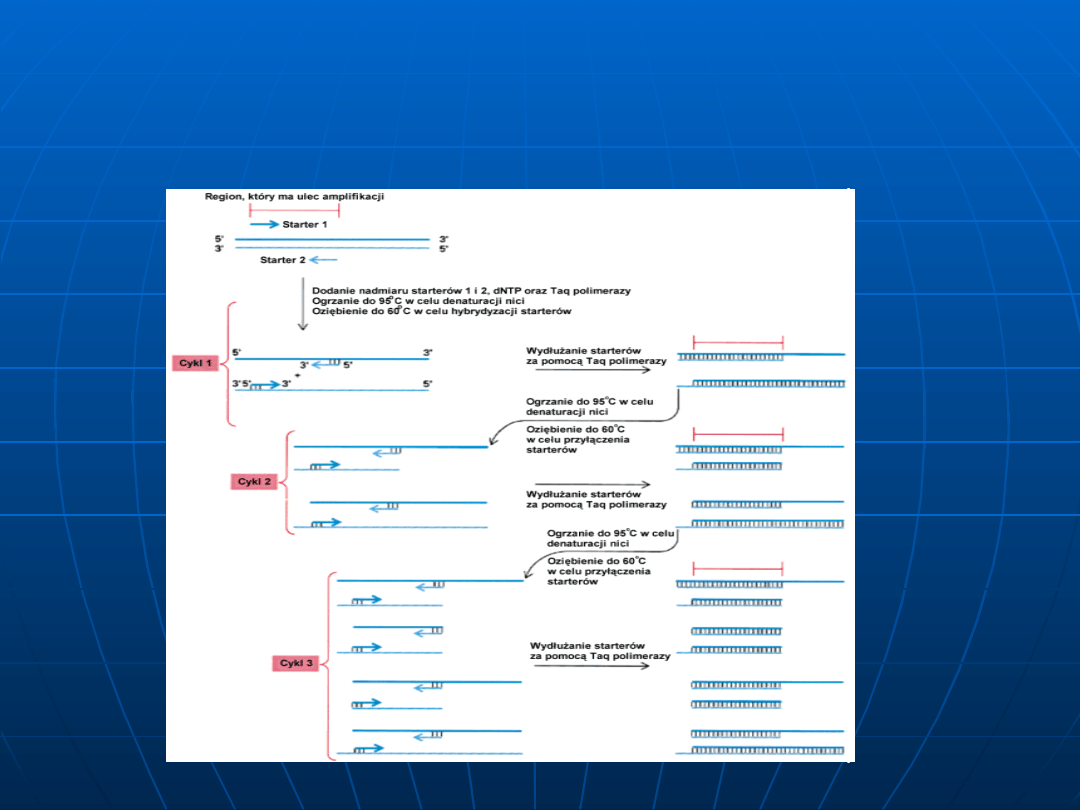

RT-PCR (reverse transcriptase PCR)
Matrycą wyjściową jest RNA, które w
procesie odwrotnej transkrypcji jest
przepisywane na komplementarne
DNA (ang. Complementary DNA,
cDNA). Następnie cDNA ulega
amplifikacji, jak w zwykłym PCR.

nested-PCR
Stosowane są 2 pary starterów:
zewnętrzna (komplementarna do końców
poszukiwanej sekwencji) i wewnętrzna
(przyłącza się przyśrodkowo w stosunku do
pierwszej pary starterów). Produkt
powstały po amplifikacji z pierwszą parą
poddaje się kolejnej rundzie z 2 parą.
Zapewnia to większą specyficzność
metody.

RAPD (random amplified polymorphic
DNA)
Metoda wykorzystywana, gdy nieznane są
sekwencje specyficzne, stosuje się krótkie
uniwersalne startery(10nt), którymi
amplifikuje się zbiór produktów, a ich
liczba i długości są zależne od sekwencji
nukleotydowej matrycy oraz warunków
reakcji.

Do każdej reakcji dodawany jest jeden
Do każdej reakcji dodawany jest jeden
rodzaj startera. Zakłada się występowanie
rodzaj startera. Zakłada się występowanie
sekwencji typu odwróconych powtórzeń w
sekwencji typu odwróconych powtórzeń w
takiej odległości od siebie w genomie ,
takiej odległości od siebie w genomie ,
która pozwoli na wydajną amplifikację czyli
która pozwoli na wydajną amplifikację czyli
300 do 1500 par zasad. Elektroforetyczny
300 do 1500 par zasad. Elektroforetyczny
obraz prążków jest charakterystyczny dla
obraz prążków jest charakterystyczny dla
osobnika. Zastosowanie; określanie
osobnika. Zastosowanie; określanie
stopnia pokrewieństwa między
stopnia pokrewieństwa między
organizmami a także m.in. w
organizmami a także m.in. w
kryminalistyce w celach identyfikacji.
kryminalistyce w celach identyfikacji.

multiplex-PCR
Równoczesne zastosowanie kilku par
starterów w mieszaninie reakcyjnej
umożliwia diagnozowanie kilku
patogenów jednocześnie. Produkty
muszą się różnić długością.

PCR
PCR
in situ
in situ
- nowa technika
- nowa technika
pozwalająca na przeprowadzenie
pozwalająca na przeprowadzenie
reakcji w tkance bez naruszania jej
reakcji w tkance bez naruszania jej
struktury np. na preparacie
struktury np. na preparacie
mikroskopowym.
mikroskopowym.

PCR-RFLP-połączenie PCR z analizą
restrykcyjną. Produkty amplifikacji
poddaje się działaniu restryktazy (1
lub więcej) i porównuje wzór
powstałych prążków.

Real-time PCR (PCR w czasie
rzeczywistym)- metoda ilościowa
pozwalająca na obserwowanie
amplifikacji w czasie rzeczywistym.
Wraz z przybywaniem produktów PCR
wzrasta intensywność fluorescencji
rejestrowanej przez detektor.
Odzwierciedlone jest to w postaci
wykresu na monitorze komputera.

Sekwencjonowanie
Sekwencjonowanie
Metoda, dzięki której możliwe jest
Metoda, dzięki której możliwe jest
ustalenie sekwencji czyli określenie
ustalenie sekwencji czyli określenie
kolejności nukleotydów. Jeden z
kolejności nukleotydów. Jeden z
etapów analizy genu. Na podstawie
etapów analizy genu. Na podstawie
sekwencji istnieje możliwość
sekwencji istnieje możliwość
przewidzenia kolejności
przewidzenia kolejności
aminokwasów w białku , które jest
aminokwasów w białku , które jest
kodowane przez dany gen.
kodowane przez dany gen.

Chemiczna - metoda
Chemiczna - metoda
Maxama-Gilberta
Maxama-Gilberta
Polega na degradacji wyznakowanych na 5`
Polega na degradacji wyznakowanych na 5`
końcu cząsteczek DNA związkami chemicznymi
końcu cząsteczek DNA związkami chemicznymi
przecinającymi specyficznie wiązania
przecinającymi specyficznie wiązania
fosfodiestrowe za nukleotydem odpowiadającym
fosfodiestrowe za nukleotydem odpowiadającym
określonej zasadzie azotowej. Wynikiem reakcji
określonej zasadzie azotowej. Wynikiem reakcji
jest zbiór fragmentów DNA o różnej długości co
jest zbiór fragmentów DNA o różnej długości co
spowodowane jest takim doborem warunków , że
spowodowane jest takim doborem warunków , że
w poszczególnych cząsteczkach przecinane jest
w poszczególnych cząsteczkach przecinane jest
tylko jedno lub dwa wiązania. Fragmenty z
tylko jedno lub dwa wiązania. Fragmenty z
czterech niezależnych reakcji (dGTP , dGTP i
czterech niezależnych reakcji (dGTP , dGTP i
dATP , dCTP , dCTP i dTTP) rozdziela się
dATP , dCTP , dCTP i dTTP) rozdziela się
elektroforetycznie po czym poddaje
elektroforetycznie po czym poddaje
autoradiografii. Prążki na autoradiogramie
autoradiografii. Prążki na autoradiogramie
odpowiadają fragmentom DNA posiadającym na
odpowiadają fragmentom DNA posiadającym na
końcu 5` znakowany fosfor a na końcu 3`
końcu 5` znakowany fosfor a na końcu 3`
określoną zasadę.
określoną zasadę.
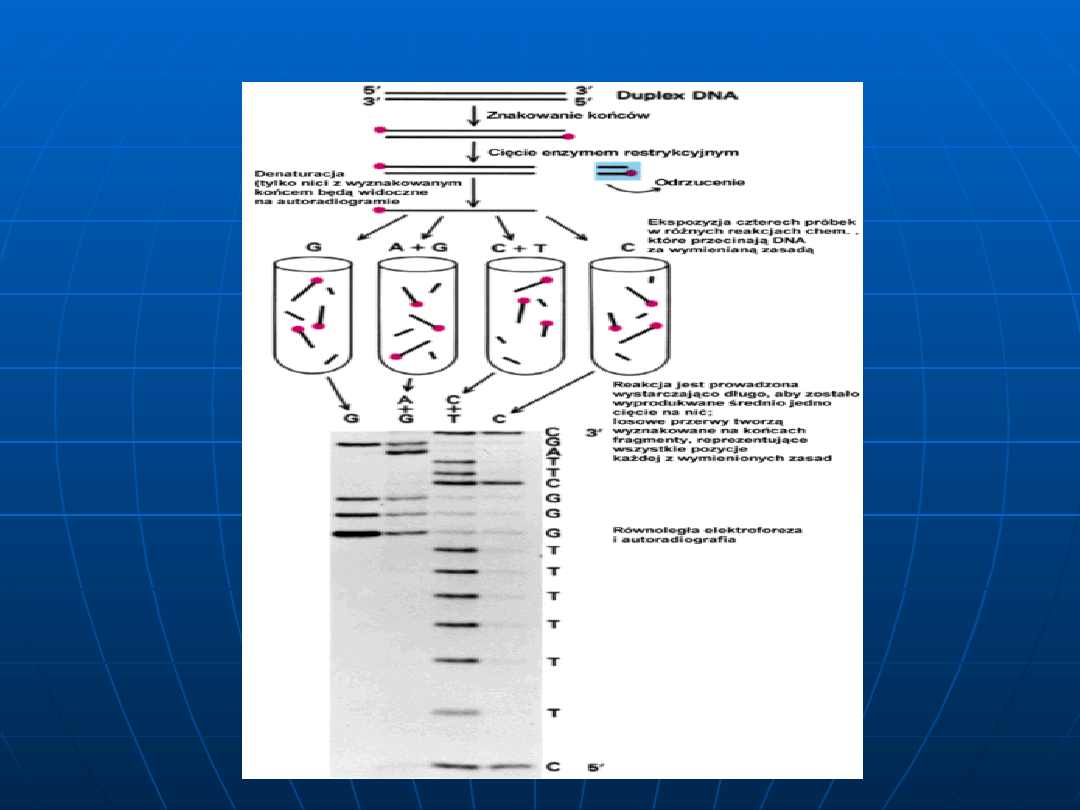

Enzymatyczna - metoda
Enzymatyczna - metoda
Sangera
Sangera
Na jednoniciowej matrycy syntetyzuje się
Na jednoniciowej matrycy syntetyzuje się
in vitro
in vitro
DNA. Synteza przebiega od radioaktywnego ,
DNA. Synteza przebiega od radioaktywnego ,
oligonukleotydowego startera komplementarnego
oligonukleotydowego startera komplementarnego
do 3` końca matrycy. Reakcję wykonuje się
do 3` końca matrycy. Reakcję wykonuje się
równolegle w czterech probówkach , w których
równolegle w czterech probówkach , w których
oprócz kompletu deoksytrifosforanów
oprócz kompletu deoksytrifosforanów
nukleotydów (dATP , dCTP , dTTP , dGTP) dodana
nukleotydów (dATP , dCTP , dTTP , dGTP) dodana
jest niewielka ilość jednego z nich w postaci
jest niewielka ilość jednego z nich w postaci
dideoksytrifosforanu nukleotydu , co uniemożliwia
dideoksytrifosforanu nukleotydu , co uniemożliwia
kontynuowanie reakcji w momencie włączenia
kontynuowanie reakcji w momencie włączenia
takiego nukleotydu w syntetyzowany łańcuch ,
takiego nukleotydu w syntetyzowany łańcuch ,
ponieważ brakuje wtedy grupy hydroksylowej na
ponieważ brakuje wtedy grupy hydroksylowej na
3` końcu.
3` końcu.
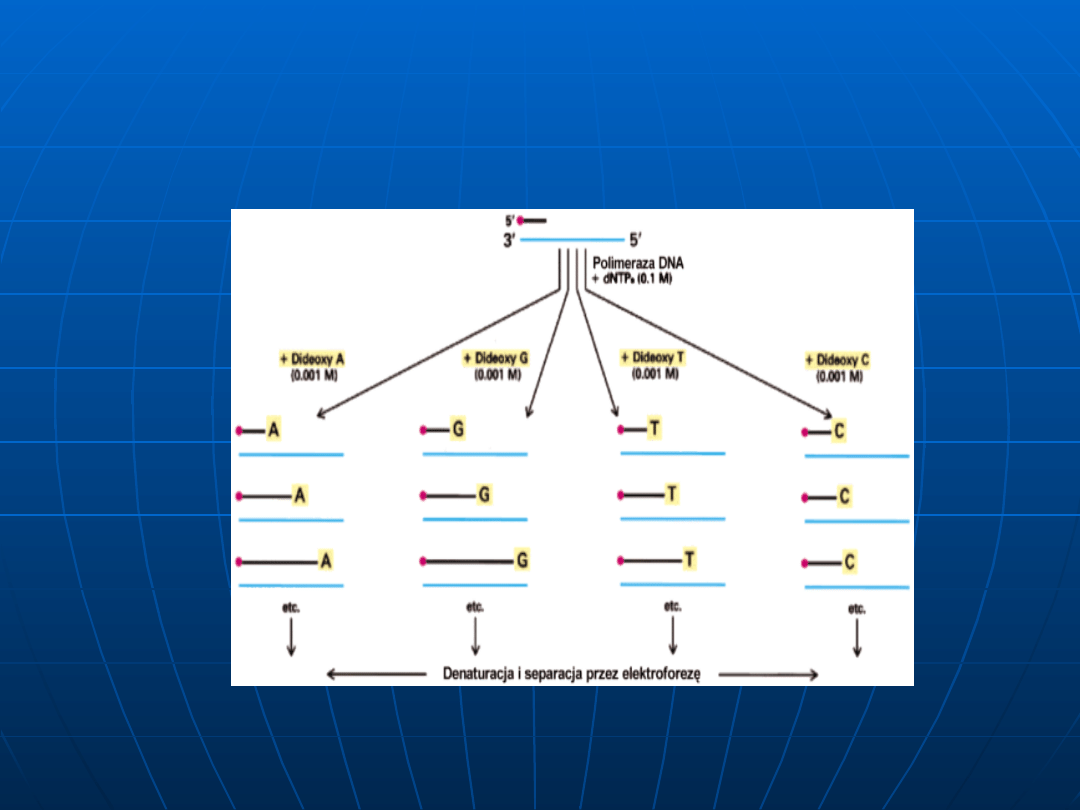

Obecnie coraz częściej znakowanie przy
Obecnie coraz częściej znakowanie przy
użyciu radioizotopów zostaje zastąpione
użyciu radioizotopów zostaje zastąpione
przez wykorzystanie znaczników
przez wykorzystanie znaczników
fluorescencyjnych.
fluorescencyjnych.
Znaczniki te mogą podobnie jak izotopy
Znaczniki te mogą podobnie jak izotopy
występować na ddNTP lub starterach. W
występować na ddNTP lub starterach. W
przypadku zastosowania tego wariantu
przypadku zastosowania tego wariantu
dalsze etapy procedury są identyczne, jak
dalsze etapy procedury są identyczne, jak
w klasycznej metodzie dideoksy. Pojawia
w klasycznej metodzie dideoksy. Pojawia
się tu jednak pewna znacząca różnica, a
się tu jednak pewna znacząca różnica, a
mianowicie w przypadku użycia starterów
mianowicie w przypadku użycia starterów
różnobarwnych, podczas elektroforezy
różnobarwnych, podczas elektroforezy
korzysta się tylko z jednej ścieżki.
korzysta się tylko z jednej ścieżki.

Wykrywanie mutacji
Wykrywanie mutacji
Bezpośrednie wykrywanie mutacji
jest najbardziej swoistą metodą
wykrywania zaburzeń w obrębie
genu. Umożliwia rozpoznanie
nosicielstwa mutacji niemal ze 100%
pewnością.

Pośrednie wykrywanie mutacji jest
metodą o nieco mniejszej swoistości
pozwala natomiast na potwierdzenie
lub wykluczenie nosicielstwa mutacji
w wielu przypadkach, w których nie
można wykryć zmian bezpośrednio w
genach.

Wykrywanie mutacji
Wykrywanie mutacji
nieznanych
nieznanych
SSCP - badanie zmian konformacji
jednoniciowego DNA
Jednoniciowe DNA w roztworze wykazuje
określoną strukturę drugorzędową.
Struktura ta zależy od rodzaju i sekwencji
zasad tworzących nić DNA. Mutacje
punktowe, delecje i insercje powodują
zmiany struktury drugorzędowej, która
wpływa na szybkość poruszania się nici w
trakcie elektroforezy na niedenaturujących
żelach poliakrylamidowych. Nici normalne i
zmutowane wykazują odmienną ruchliwość
elekroforetyczną

Niedogodności SSCP to przede wszystkim
konieczność analizowania produktów PCR
nie dłuższych niż 200-300 pz (par zasad),
bowiem przy większej długości produktów
spada znacząco czułość wykrywania
mutacji. Dlatego by ocenić sekwencję
kodującą dużych genów trzeba wykonać
wiele reakcji PCR, np. gen BRCA1 należy
analizować poprzez badanie 40 różnych
produktów amplifikacji.
Czułość SSCP w wykrywaniu mutacji nie
przekracza 80%

HET - analiza
heterodupleksów
Sekwencje niezmienione oraz sekwencje z
mutacją są obecne w reakcji PCR (jako
matryce)
Produktami tej reakcji są cztery różne
dwuniciowe fragmenty DNA.
Dwa homodupleksy (struktury dwuniciowe
w pełni komplementarne). Dwa inne to
heterodupleksy zawierające miejsca
niesparowane (mismatch).

Heterodupleksy ze zmianą co
najmniej jednej zasady mogą
wykazywać inną w porównaniu do
homodupleksów ruchliwość podczas
elektroforezy na zwykłym
poliakrylamidowym żelu.
Czułość 90%

CMC - chemiczne rozszczepianie
niesparowań heterodupleksów
Niesparowane w heterodupleksach zasady
C i T ulegają chemicznej modyfikacji,
odpowiednio z hydroksyloaminą i
czterotlenkiem osmu. Miejsca wiązania
tych substancji są cięte z użyciem
piperydny. Pocięte fragmenty DNA są
rozdzielane elektroforetycznie.
Wykrywanie mutacji w odcinkach DNA
długości 1-2 kpz.

DGGE - elektroforeza z
gradientem czynnika
denaturującego
W trakcie elektroforezy dwuniciowego DNA w żelu
o wzrastającym stężeniu związku denaturującego,
niektóre fragmenty DNA ulegają rozdzieleniu na
pojedyncze nici (denaturacja) przy niższym, a
inne fragmenty przy wyższym stężeniu
formamidu. W momencie rozejścia się na
pojedyncze nici ich przemieszczanie w żelu
zostaje gwałtownie przyhamowane. Moment
denaturacji DNA zależy od jego budowy (składu
zasad i długości).
Fragmenty zawierające mutacje „zatrzymują się”
w żelu na ogół przy innym stężeniu czynnika
denaturującego aniżeli prawidłowe.
Ponad 90% czułość wykrywania mutacji

Dziękuję za uwagę
Dziękuję za uwagę

Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
- Slide 44
- Slide 45
- Slide 46
- Slide 47
- Slide 48
- Slide 49
- Slide 50
- Slide 51
- Slide 52
- Slide 53
- Slide 54
- Slide 55
- Slide 56
- Slide 57
- Slide 58
- Slide 59
- Slide 60
Wyszukiwarka
Podobne podstrony:
2 Techniki badawcze stosowane w badaniach spozycia żywnościid 20830 ppt
Diagnostyka laboratoryjna stosowana w rehabilitacji klinicznej Fizjoterapia studia magisterskie II s
1 Infrastruktura, technika i technologia procesów logistyczid 8534 ppt
Materiałoznawstwo i Techniki Wytwarzania Plan Laboratoriów
Ciekawe techniki plastyczne stosowane w pracy z dziećmi przedszkolnymi, różności ale przydatne
Techniki plastyczne stosowane w przedszkolu
04a Komórkowe i molekularne podłoże zapaleńid 5382 ppt
Metody i techniki badan stosowanych w psychologii rozwojowej
Leki stosowane w zakażeniach bakteryjnych ppt
Technika grupowa stosowana przez kuratorów sądowych i funkcjonariuszy organów ścigania, B.W, krymin
Techniki molekularne 2009
Leki stosowane na kaca ppt
Techniki molekularne
Laboratoria z techniki pomiarowej, Cw2LO1, LABORATORIUM PODSTAW BUDOWY URZĄDZEŃ TRANSPORTOWYCH
spraw fm(1), WAT- Elektronika i Telekomunikacja, Semestr V, Technika Emisji i Odbioru, laboratoria,
więcej podobnych podstron