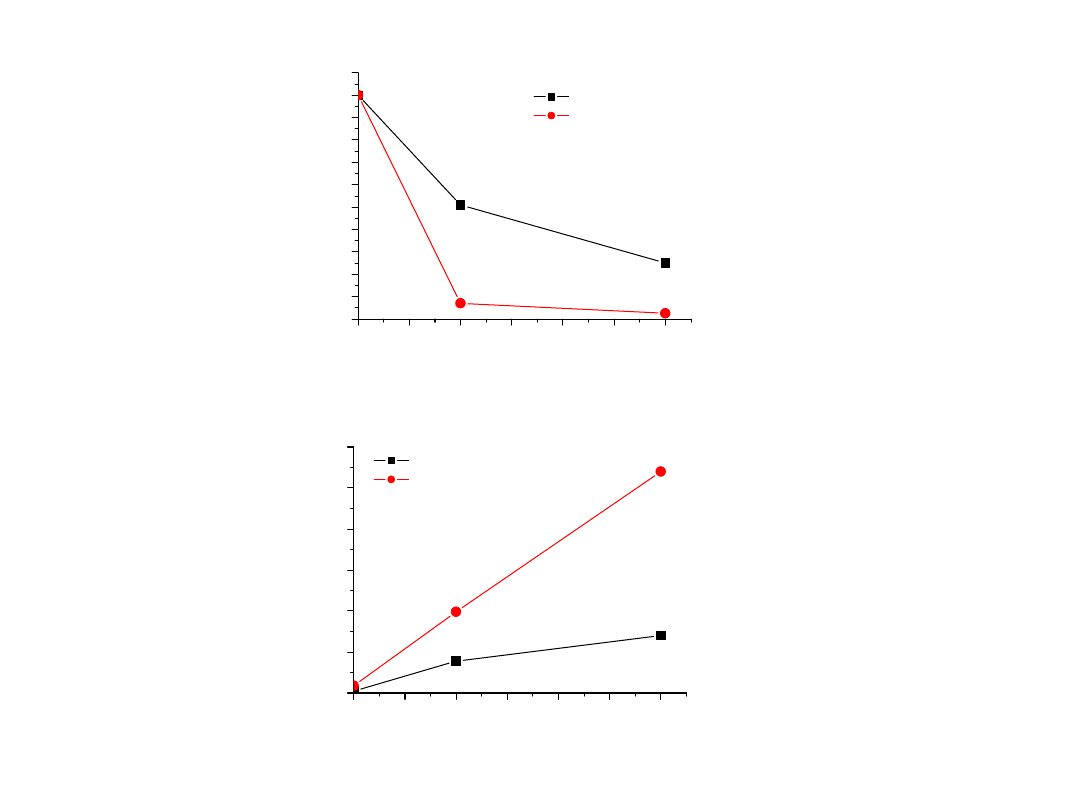
0.0
0.5
1.0
1.5
2.0
2.5
3.0
0
10
20
30
40
50
60
70
80
90
100
110
%
p
rz
ez
yc
ia
DMS (mM)
DMS
DMS+NaOH
0.0
0.5
1.0
1.5
2.0
2.5
3.0
0
50
100
150
200
250
300
M
F
x1
0
-4
DMS (mM)
DMS
DMS+NaOH

Naprawa DNA
Przez odwrócenie –
bezbłędna
1.Fotoliaza
(dimery pirymidynowe)
2.Transferaza O
6
-MeG
3.AlkB
(3MeC, 1MeA)
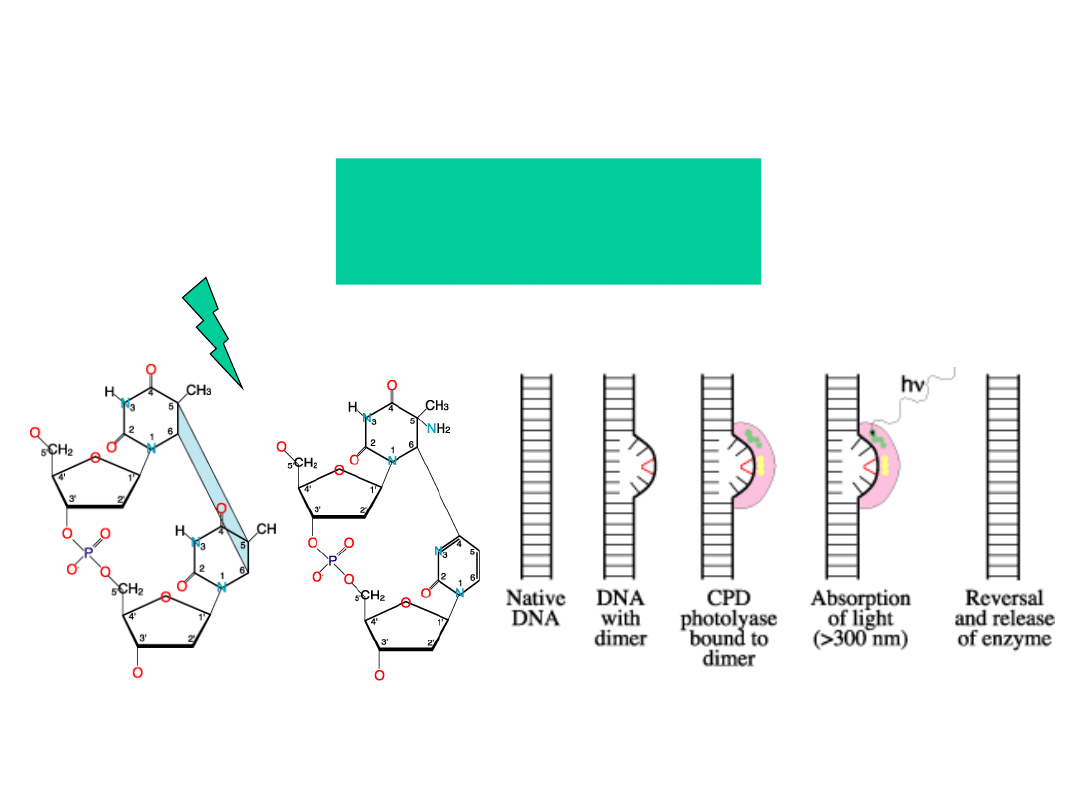
Bezpośrednie usuwanie uszkodzeń
DNA bez naruszania integralności
podwójnej helisy
Fotoreaktywac
ja
fotoliazy
Dimer
pirymidyno
wy (CPD)
6,4-fotoprodukt
UV

Bezpośrednie usuwanie uszkodzeń DNA
bez naruszania integralności podwójnej
helisy
Odmienne typy fotoliazy mogą usuwać specyficznie tylko
jeden typ uszkodzeń. E. coli posiada enzym rozrywający
podwójne wiązanie w CPD.
Fotoliaza E. coli
jest monomerycznym białkiem o masie
cząsteczkowej ok. 60 kDa, zawierającym chromofor – (5,10-
metenylo-tetrahydrofolilo)poliglutaminian (MTHF)
ulegający wzbudzeniu przez światło widzialne o długości
300 nm i rozrywające pierścień cyklobutanowy CPD.
10 – 20 cząsteczek na komórkę E.coli
Enzym powolny
Fotoliazy
– obecne u bakterii, niższych eukariontów, w
roślinach, ale brak ich u ssaków łożyskowych
Aktywność fotoliazy zmniejsza toksyczność i
poziom mutacji indukowanych światłem UV
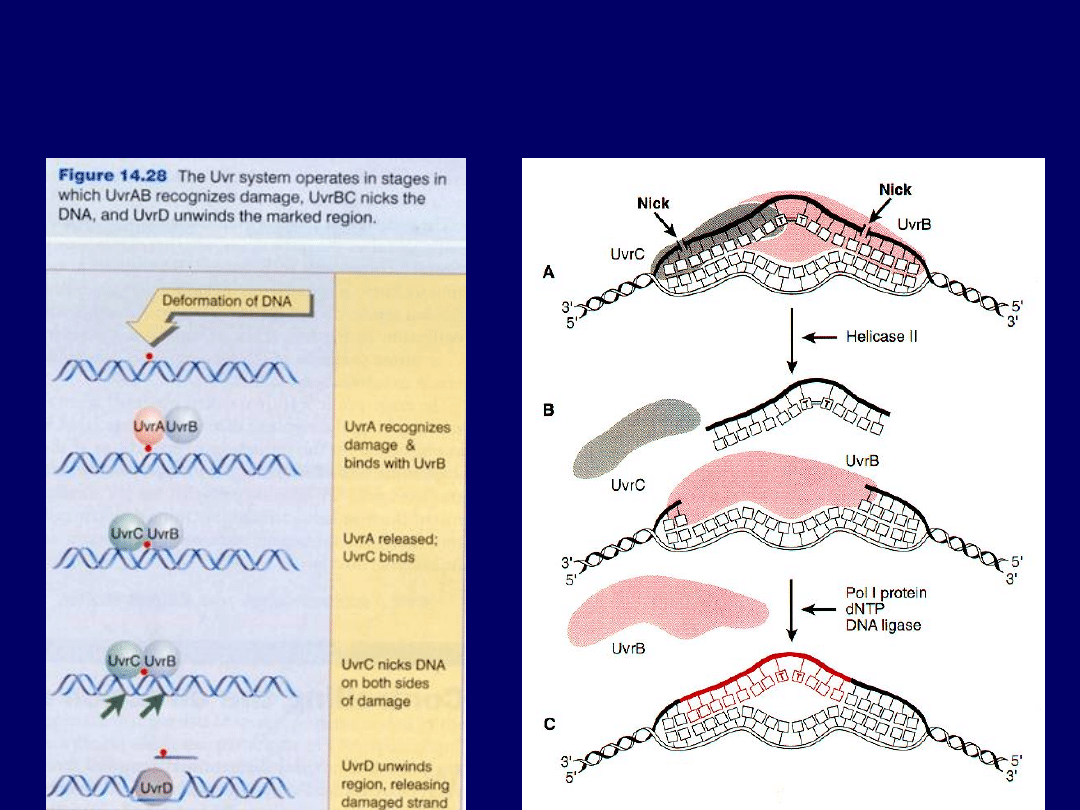
NER u bakterii
Wycinane są 12-13 nukleotydowe fragmenty DNA
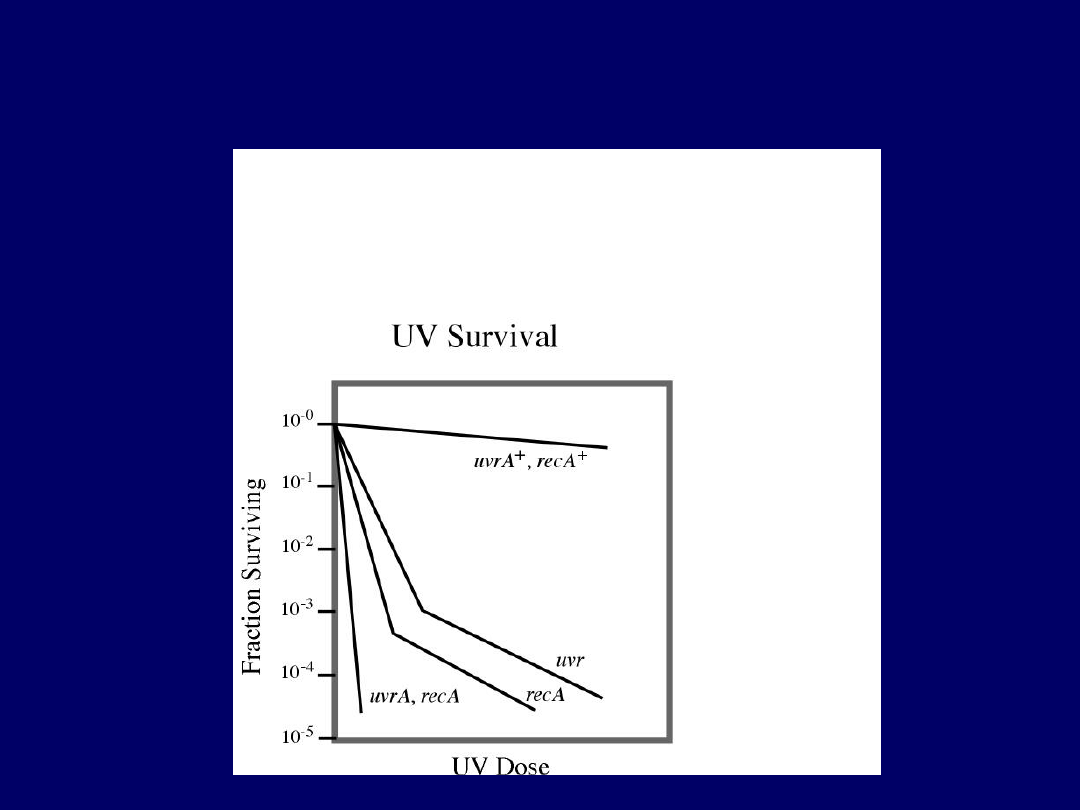
Mutanty uvr są nadwrażliwe na
promieniowanie UV
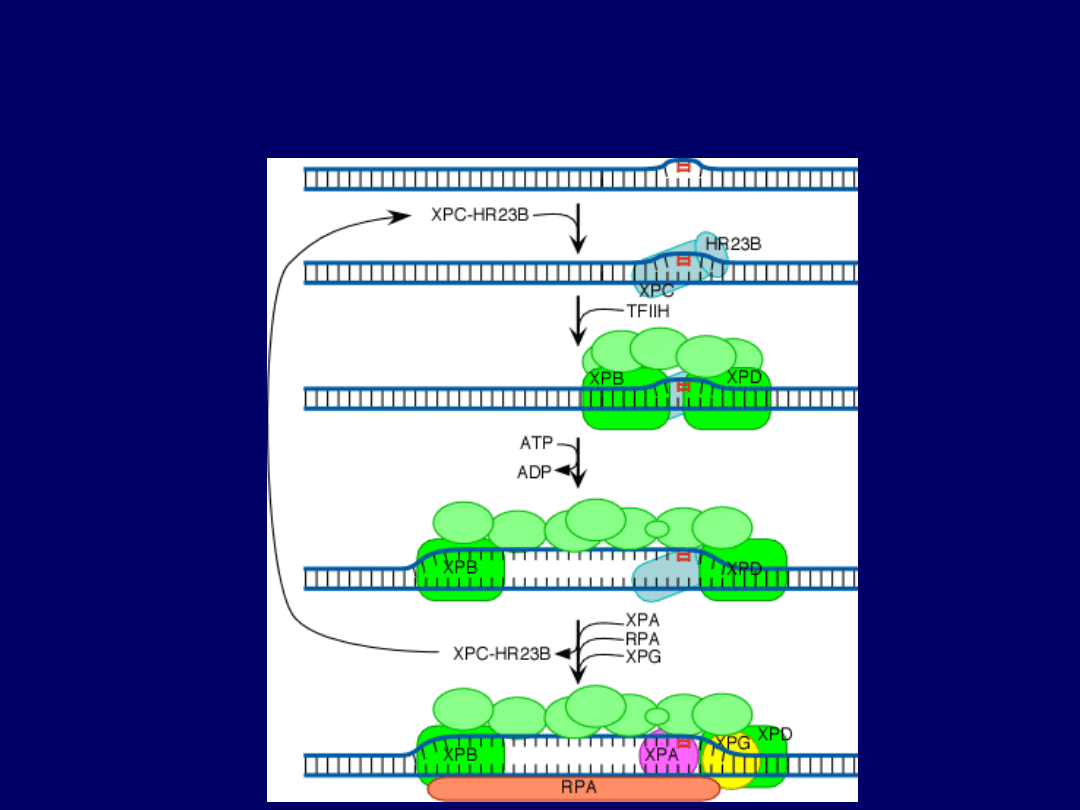
NER u ssaków
Wycinane są 25 – 27 nukleotydowe fragmenty
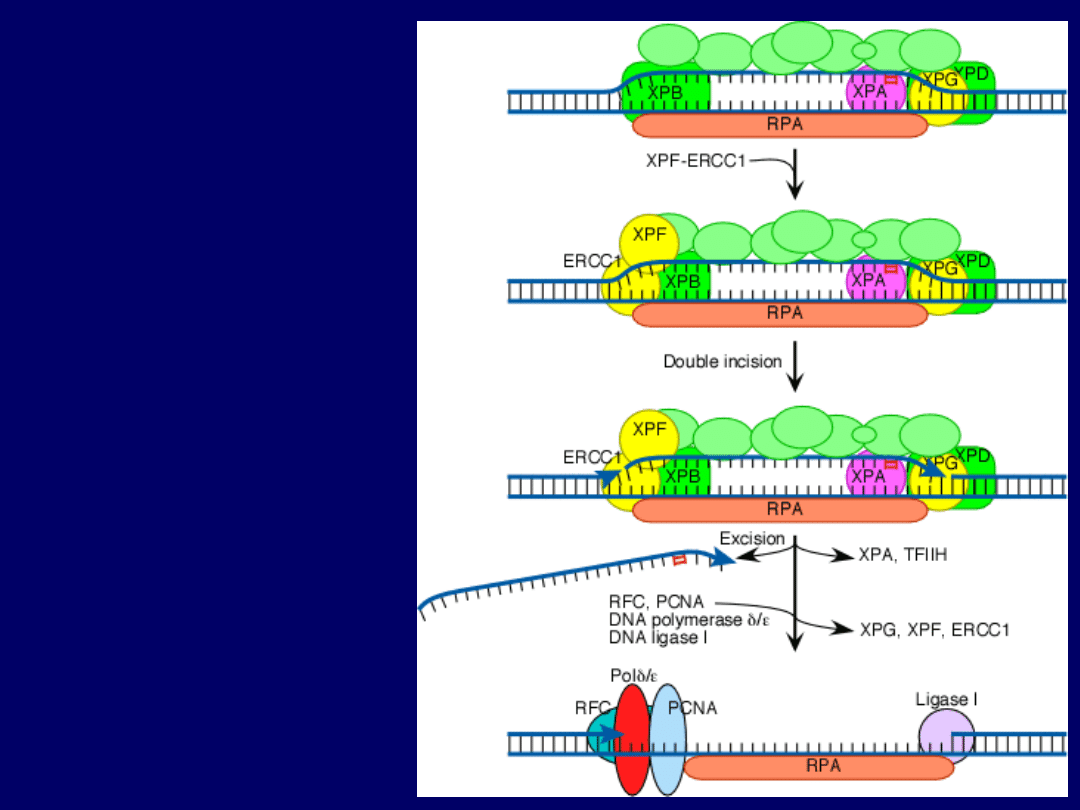
NER
u
ssaków
c.d.
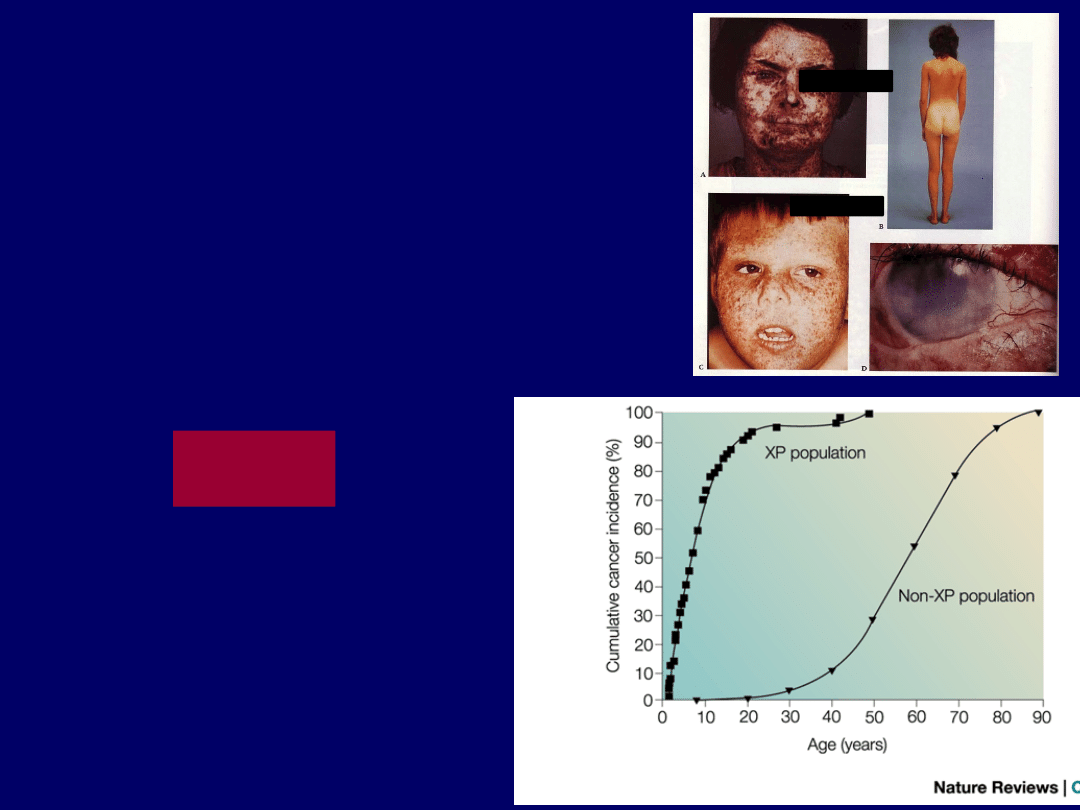
Xeroderma pigmentosum
(XP)
Nadwrażliwość na
promieniowanie
słoneczne;
wysoka
zapadalność na raka
skóry; defekt w GGR
Usuwa
uszkodzenia
DNA z całego
genomu
GGR

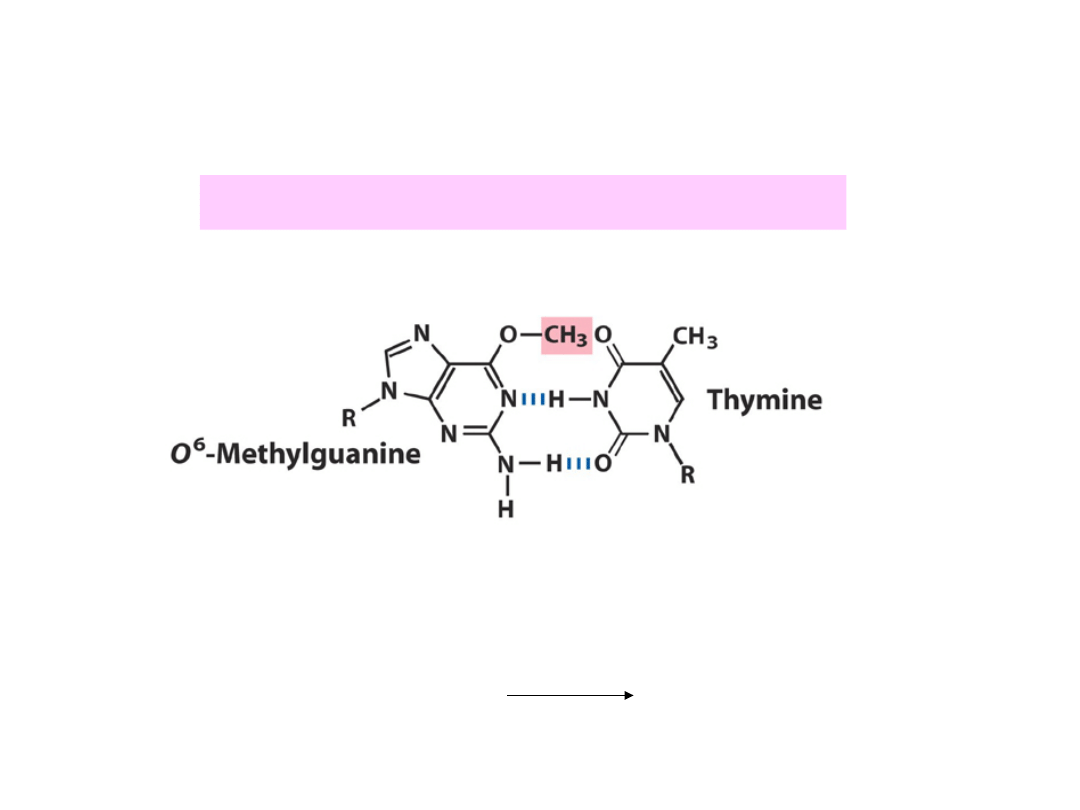
Demetylacja przez O
6
-alkiloguanylo-
alkilotransferazę (Ada – u E.coli, AGT -
ssaki)
O
6
MeG
G
AG
T
Bezpośrednie usuwanie uszkodzeń DNA bez
naruszania integralności podwójnej helisy
Usuwanie grup alkilowych
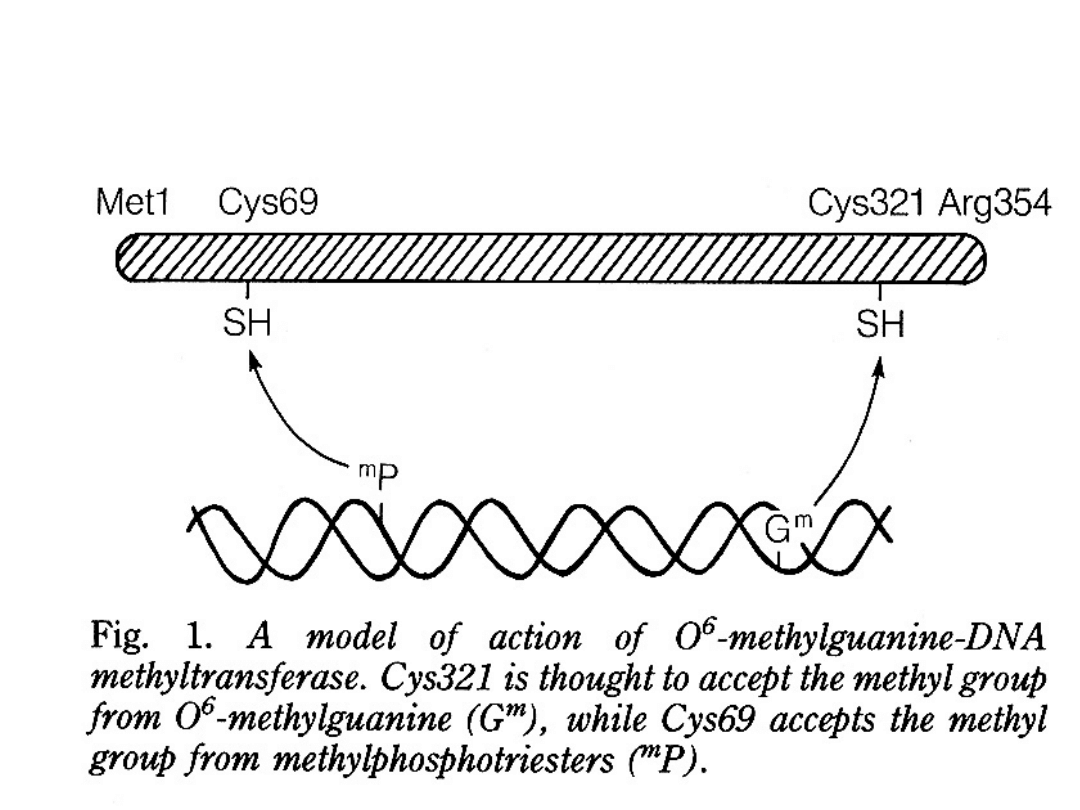
AGT i Ada
– białka samobójcze; ulegają
inaktywacji po przeniesieniu grup metylowych z
DNA na grupy –SH Cys (Cys69 i 321 w białku
Ada)
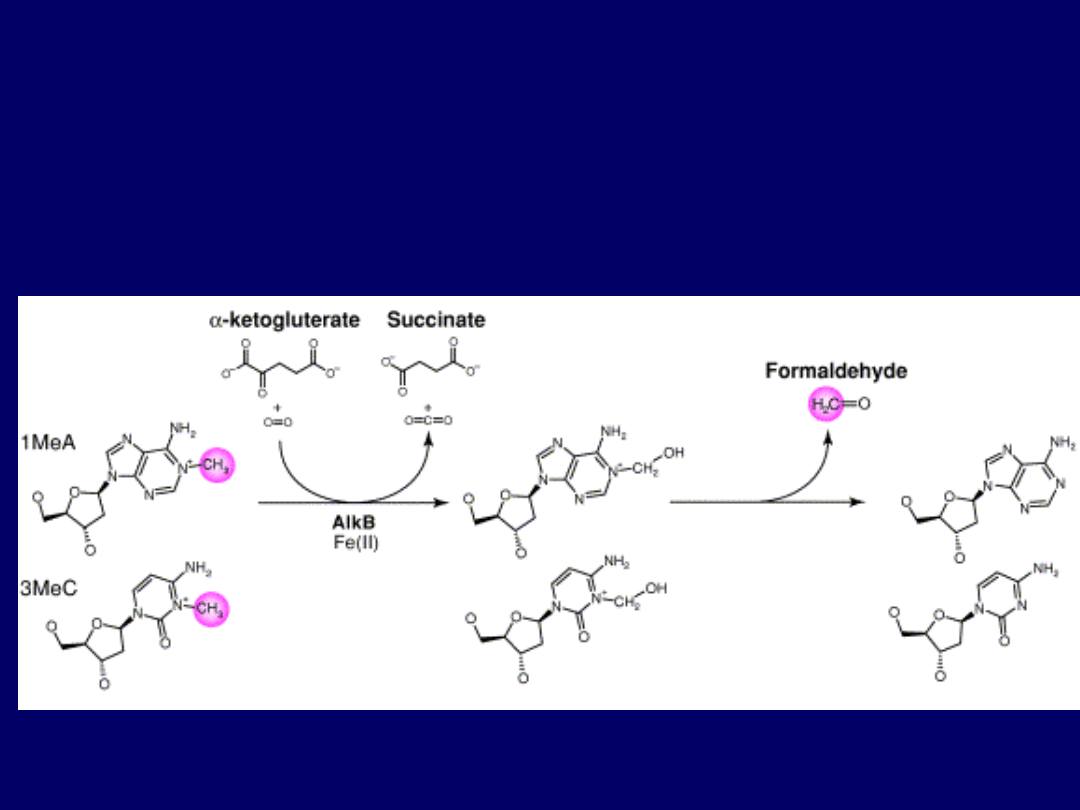
Bezpośrednie usuwanie uszkodzeń
DNA bez naruszania integralności
podwójnej helisy
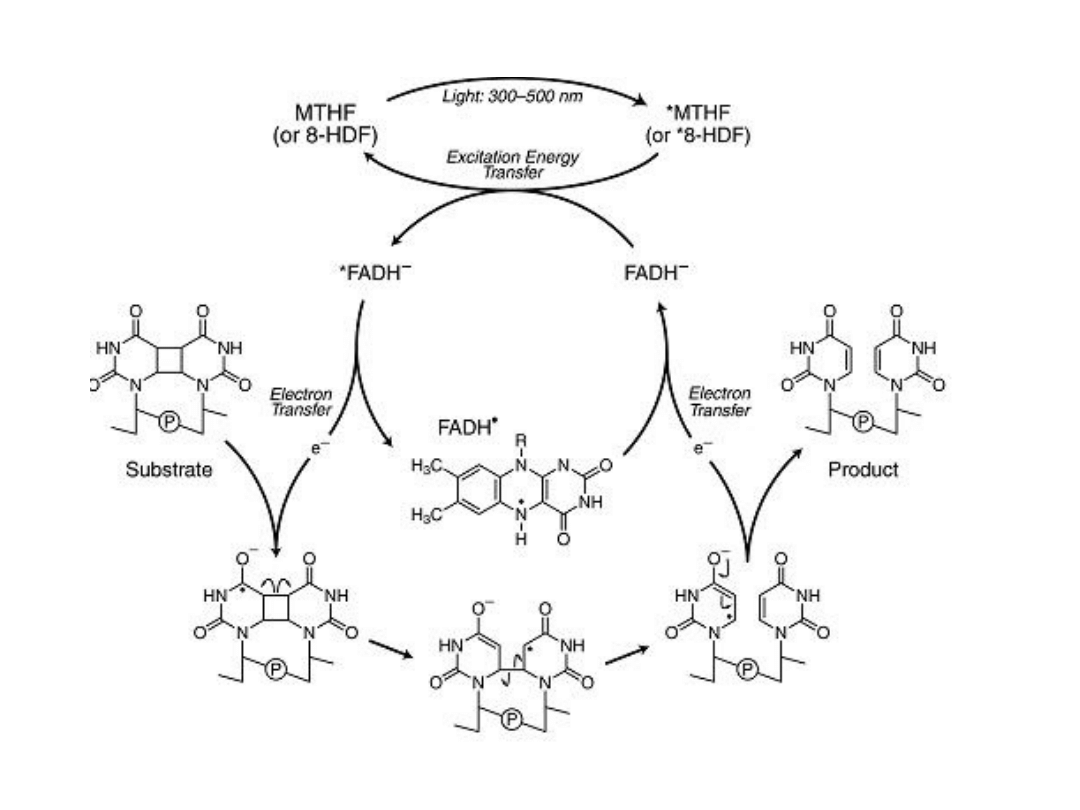
oksydacyjna naprawa zmetylowanych zasad
DNA – białko AlkB
AlkB naprawia
ssDNA.
1-meA & 3-
meC
są substratami
AlkB
AlkB należy do rodziny
dioksygenaz
ketoglutaranu
zależnych od Fe
2
+
AlkB wymaga Fe(II) jako
kofaktora, a także -
ketoglutaranu i O
2
jako
kosubstratów
Mechaniz
m reakcji
AlkB
Prof.Sedgwick’s group, Nature 2002
Prof.Seeberg’s group, Nature 2002
Bezpośrednie usuwanie uszkodzeń
DNA bez naruszania integralności
podwójnej helisy
oksydacyjna naprawa zmetylowanych
zasad DNA – białko AlkB
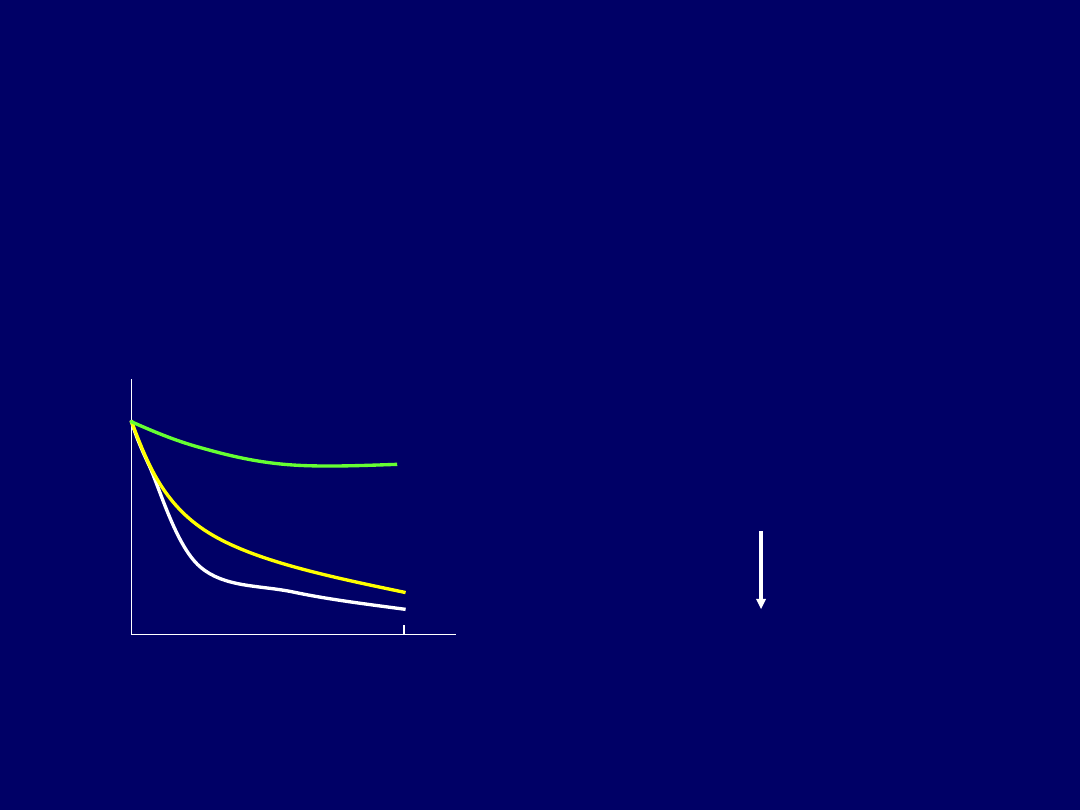
W komórkach człowieka – 8 homologów AlkB
ABH2 –
specyficzny względem dsDNA; naprawia
głównie
1MeA
ABH3 –
specyficzny względem ssDNA oraz RNA;
naprawia
głównie
3MeC
AlkB
1MeA
Czas (godz)
30
ABH2
-
ABH3
-
podstawowy
Akumulacja
1MeA
z
wiekiem u myszy
ABH2
–
, ale nie
u ABH3
–
Endogenna metylacja
DNA
Kinetyka naprawy 1MeA w
komórkach myszy typu
podstawowego, ABH2
-
i
ABH3
-

Każda grupa robi jedno doświadczenie
1. AB 1157 – naświetlamy UV 3 min
2. BH 200 (AB1157 uvrA)
-
- UV 3 sek
3. BH 200 - UV 6 sek
Szczepy są zawieszone w MgSO
4
Liczenie wyników poprzedniego ćwiczenia:
Wyniki wpisywać do Tabelki
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
Wyszukiwarka
Podobne podstrony:
2012 wprow cw 7id 27734 ppt
2012 wprow cw 9id 27735 ppt
2012 wprow cw 10
2012 wprow cw 8
2012 wprow cw 6
2012 wprow cw 4
2012 10 03 Wprow Seminarium MPid 28102 ppt
13 04 2012 TEST KOŃCOWY GASTROLOGIAid 14559 ppt
KPA 2012 13 (ćw dr AK)
13Strategie konstruowania kwestionariuszy osobowości i etapy tworzenia testu 5id 15139 ppt
16 luty Czy i jak możliwe jest poznanie Boga(cw 3)id 16775 ppt
2012 UJS ekonomia 0id 27731 ppt
1 POMOC CW ,2id 19156 ppt
2012 10 31 HESid 27655 ppt
2 Wybrane Zagadnienia Statyki Płynów 5id 19734 ppt
008 wykład nr 5id 2445 ppt
więcej podobnych podstron