
Bakteryjne wektory plazmidowe
- Koliste cząsteczki DNA wykorzystywane do wprowadzania
fragmentów DNA do bakterii, replikujące się niezależnie od
replikacji chromosomów,
- W obecności antybiotyków, które hamują syntezę białka i
replikację chromosomu bakteryjnego, same ulegają replikacji,
- Zapewniają powielanie wprowadzonego fragmentu DNA,
- Nie są niezbędne dla gospodarza, ale mogą zwiększyć jego
szanse przeżycia w pewnych warunkach,
- Wektory ekspresyjne dodatkowo pozwalają na ekspresję
genów zawartych we wprowadzanych fragmentach DNA i
produkcję białek
- Cechy kodowane przez plazmidy: oporność na antybiotyki,
jony metali ciężkich i działanie promieniowania UV,
wytwarzanie toksyn, zdolność do metabolizmu takich
związków jak kamfora i toluen
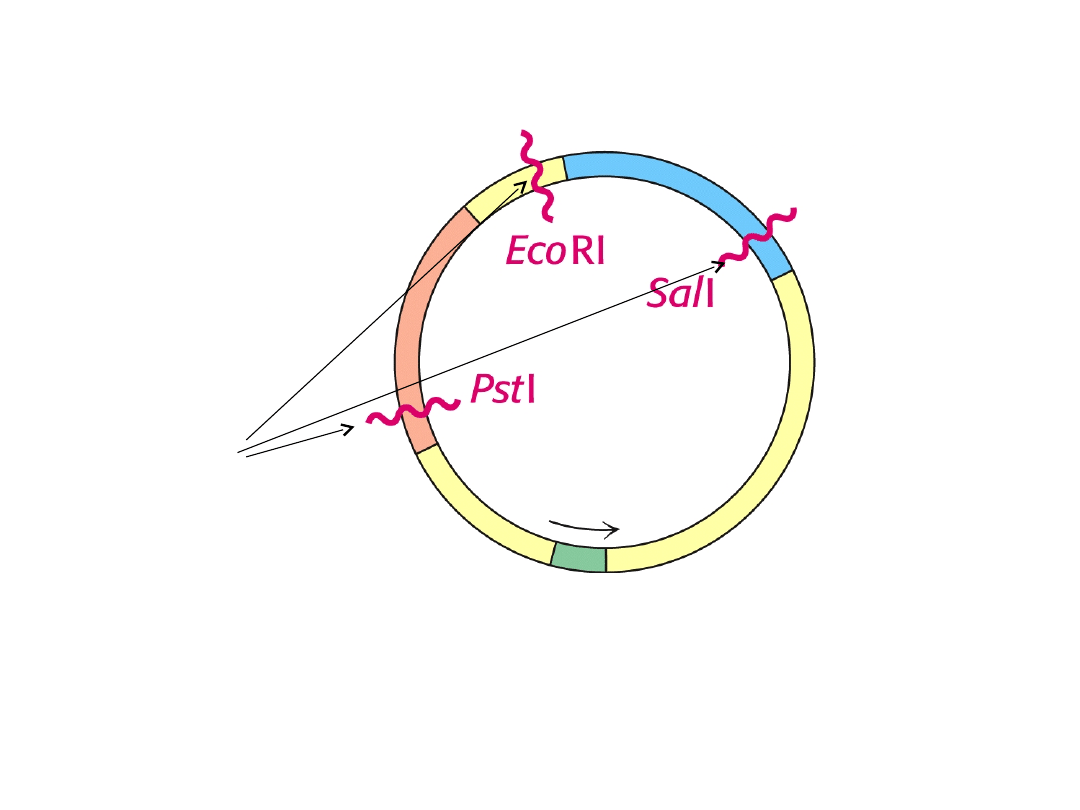
Gen oporności
na tetracyklinę
Gen oporności
na ampicylinę
Inicjacja replikacji – ori
Plazmid pBR322
Miejsca trawienia
przez enzymy
restrykcyjne

Składniki wektora bakteryjnego
- Sekwencja odpowiedzialna za inicjację replikacji, tzw.
sekwencja ori. Od tej sekwencji zależy specyficzność wektora, czyli
to w jakich komórkach może ulegać replikacji.
- Geny markerowe, czyli geny odpowiedzialne za łatwo
wyróżnialne cechy fenotypowe pozwalające odróżnić komórki, które
uległy transformacji od tych, które nie zostały stransformowane.
- Geny warunkujące oporność na antybiotyk:
- gen bla lub amp
r
kodujący enzym -laktamazę, która rozkłada
antybiotyki penicylinowe, np. ampicylinę. Ampicylina hamuje
enzymy uczestniczące w syntezie ściany komórkowej bakterii.
- gen tetA (tet
r
), który koduje pompę transbłonową zdolną do
usunięcia z komórki antybiotyku tetracykliny. Tetracyklina wiąże się
z podjednostką 30S rybosomów i hamuje translokację.
- gen Cm
r
(cat) – koduje białko, które w obecności acetyloCoA
katalizuje przemianę chloramfenikolu do pochodnych niezdolnych
do wiązania się z rybosomami. Chloramfenikol wiąże się z
podjednostką 50S rybosomów i hamuje biosyntezę białek.
- Geny warunkujące zdolność do syntezy łatwo oznaczalnego
enzymu: np. lacZ – gen kodujący - galaktozydazę.
- Miejsca rozpoznawane i trawione przez enzymy
restrykcyjne (polilinker). W miejscu polilinkera można wbudować
obcy DNA, co nie zaburza procesu replikacji.

Rekombinowany DNA
Nieistniejąca w naturze cząsteczka DNA
powstała poprzez połączenie fragmentów
różnych DNA

Trawienie DNA i enzymy restrykcyjne
Enzymy restrykcyjne czynnościowo są bakteryjnym
"układem immunologicznym" - chronią komórki bakterii przed
zakażeniem wirusowym poprzez trawienie materiału
genetycznego wirusa. Terminologia enzymów restrykcyjnych jest
odzwierciedleniem łacińskiej nazwy bakterii - pierwsza litera
pochodzi od rodzaju, a dwie następne - od gatunku, np. enzym
Eco
RI pochodzi z bakterii pałeczki okrężnicy (
E
scherichia
co
li).
Enzymy restrykcyjne rozcinają DNA mniej lub bardziej
swoiście, rozpoznając przed trawieniem określoną sekwencję
nukleotydów. Rozpoznawany odcinek może być różnej długości
(od 4 do 33 nukleotydów). DNA może zostać przecięty równo lub z
przesunięciem miejsca cięcia na obu łańcuchach o jeden, a nawet
pięć nukleotydów. W przypadku równego cięcia mówimy o
"tępych" końcach, a w przypadku nierównego cięcia o "lepkich"
końcach rozciętego łańcucha DNA.
Mechanizmem ochronnym przed trawieniem własnego
materiału genetycznego jest metylacja własnego DNA.

Nukleazy
Enzymy trawiące DNA i/lub RNA:
endonukleazy
egzonukleazy
Enzymy restrykcyjne
Specyficzne endonukleazy trawiące DNA
Np. enzym EcoRI z Escherichia coli (E. coli)
5’-GAATTC-3’
3’-CTTAAG-5’
Sekwencja palindromowa
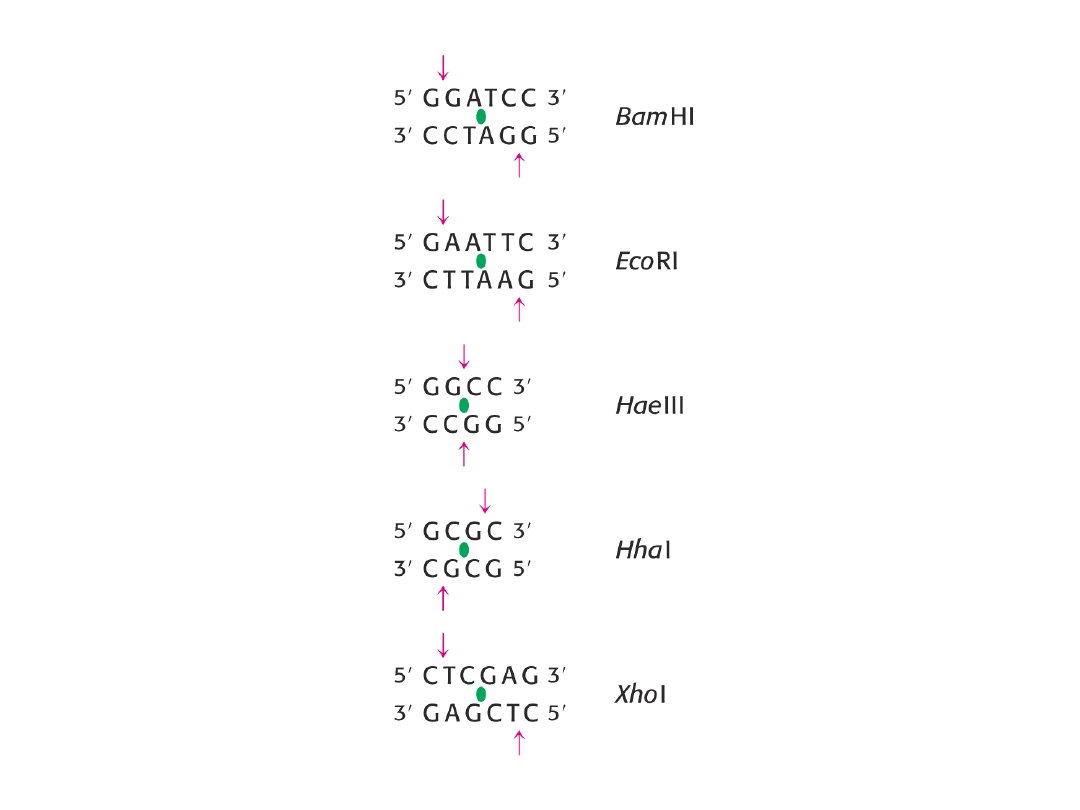
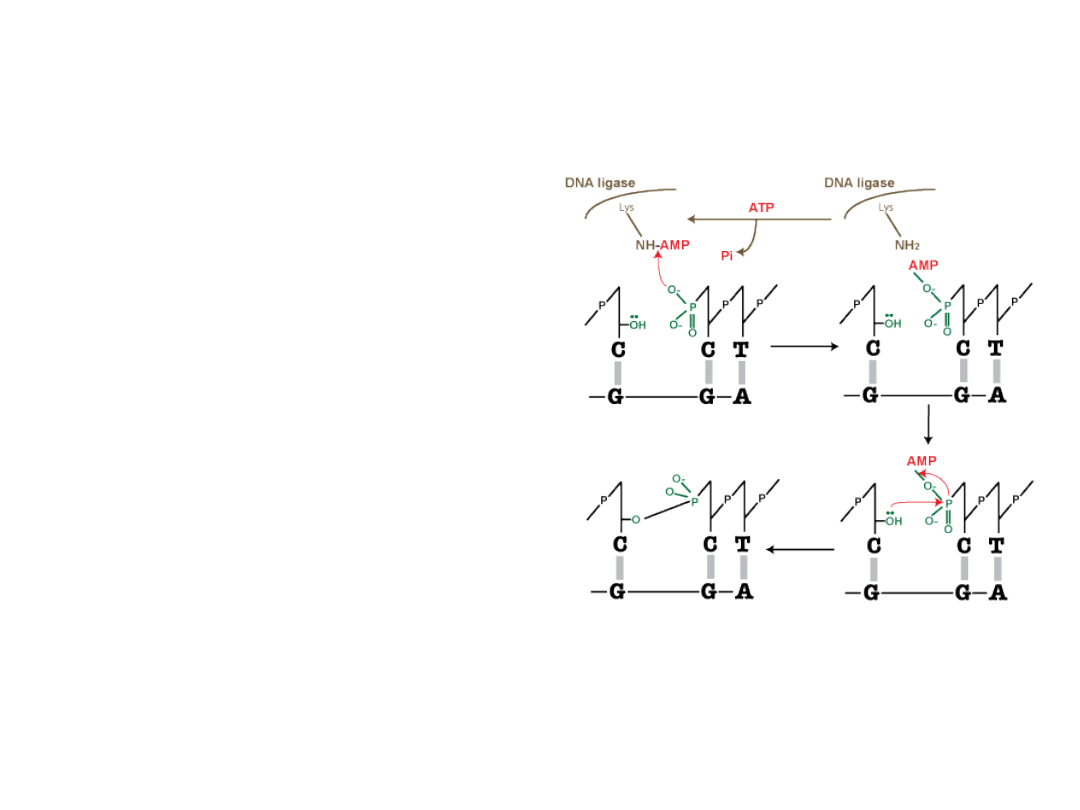
Ligacja DNA i ligazy
• Ligacja to łączenie fragmentów
DNA wiązaniami kowalencyjnymi.
W reakcji tej powstaje wiązanie
fosfodiestrowe pomiędzy grupą
hydroksylową jednego nukleotydu
(3’) a resztą fosforanową
drugiego nukleotydu (5’).
• Ligaza DNA – enzym, który
naprawia pęknięcia w jednej z
nici dwuniciowego DNA, na której
końcu 5’ znajduje się grupa
fosforanowa.
• Ligaza potrzebuje czynnika
adenylującego, aby zaktywować
grupę fosforanową i upodatnić ją
na atak grupy 3’ OH (np. ligaza
bakteriofaga T4 wykorzystuje do
tego ATP).
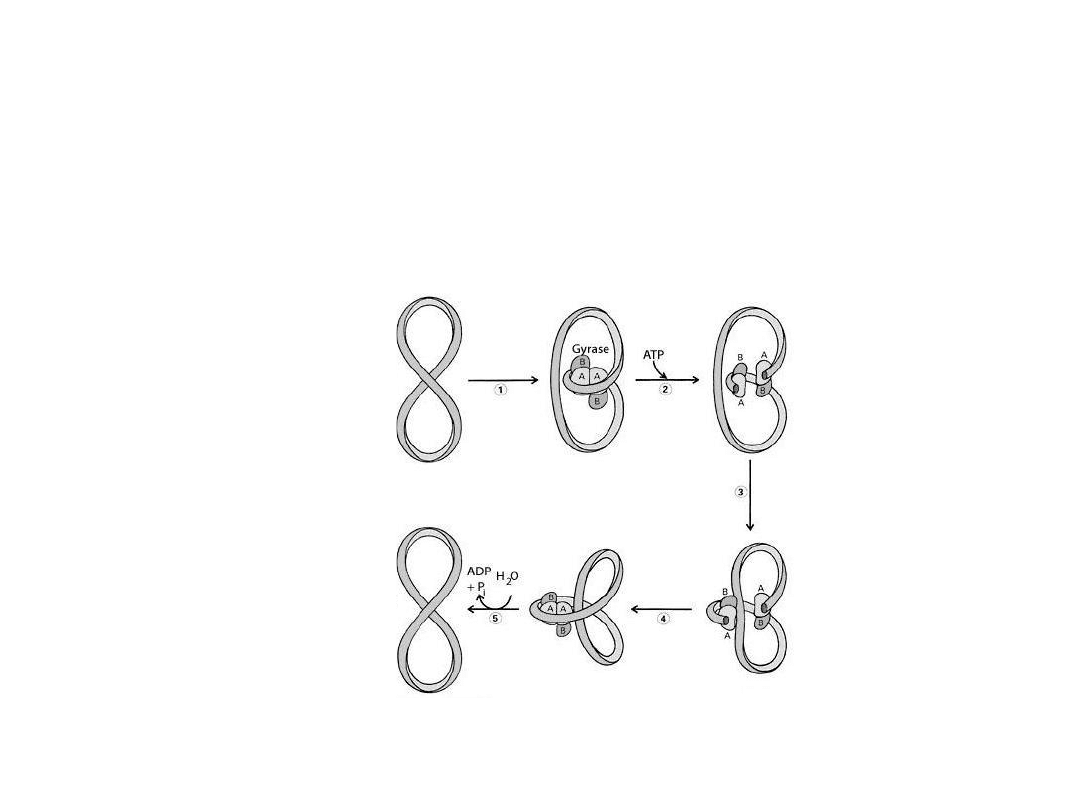
Topoizomerazy to enzymy konwertujące energię chemiczną
pochodzącą z ATP w energię torsyjnego napięcia cząsteczki o
superhelikalnej strukturze. In vivo topoizomerazy rozplatają
podwójną helisę DNA, udostępniając w ten sposób matrycę dla
enzymów replikacyjnych lub transkrypcyjnych.
DNA z 1
pozytywnym
skrętem
DNA z 1
negatywnym
skrętem
Ligacja DNA i topoizomerazy

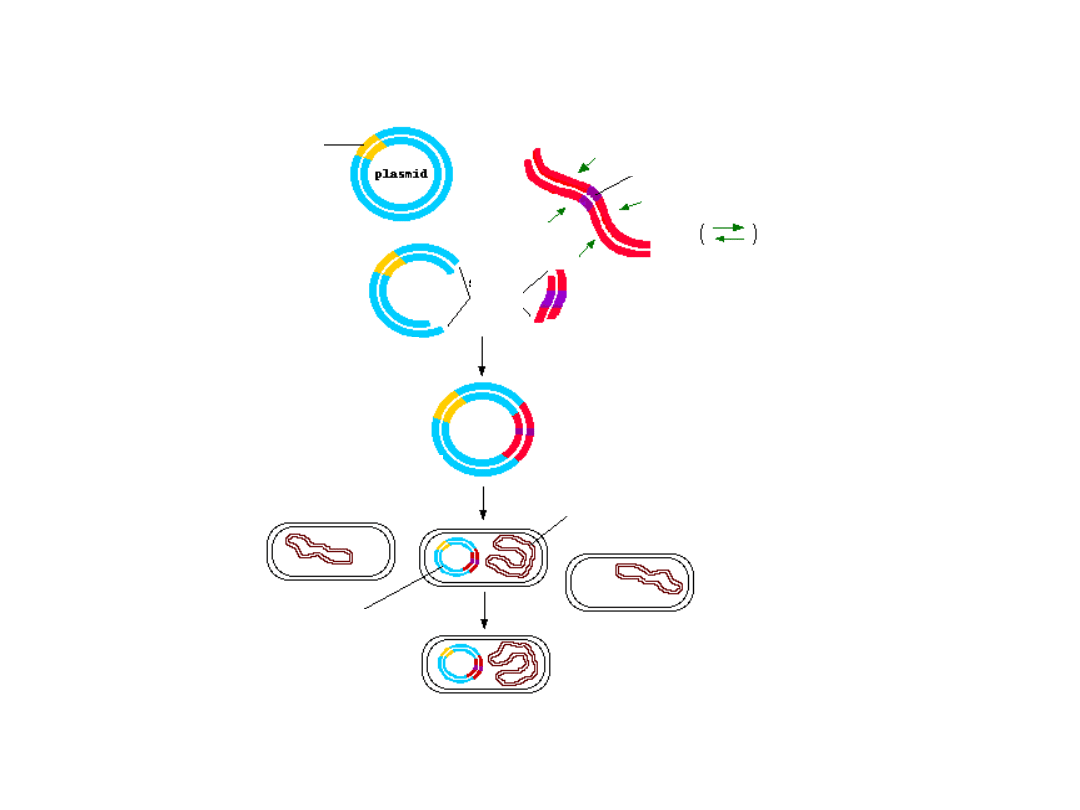
Klonowanie genu to replikacja (powielanie)
wyizolowanego fragmentu DNA
Klonowanie DNA to sposób na amplifikację (powielenie)
wybranego fragmentu DNA in vivo. Plazmid zapewniający
oporność na niesprzyjające warunki środowiskowe rozcina się
enzymem restrykcyjnym lub enzymami restrykcyjnymi i tym
samym wycina się fragment DNA, który ma ulec powieleniu. Po
zmieszaniu produktów cięcia i połączeniu ich enzymem ligującym
(ligazą) uzyskuje się plazmid z wklonowanym obcym fragmentem
DNA. Następnie dodaje się ten plazmid do hodowli bakterii w
warunkach, w których mogą przeżyć tylko te komórki, które
pobiorą plazmid. Po wyhodowaniu ogromnej biomasy
drobnoustrojów izoluje się z nich DNA plazmidowy, a z niego
wycina się enzymami restrykcyjnymi interesujący nas,
sklonowany fragment DNA.
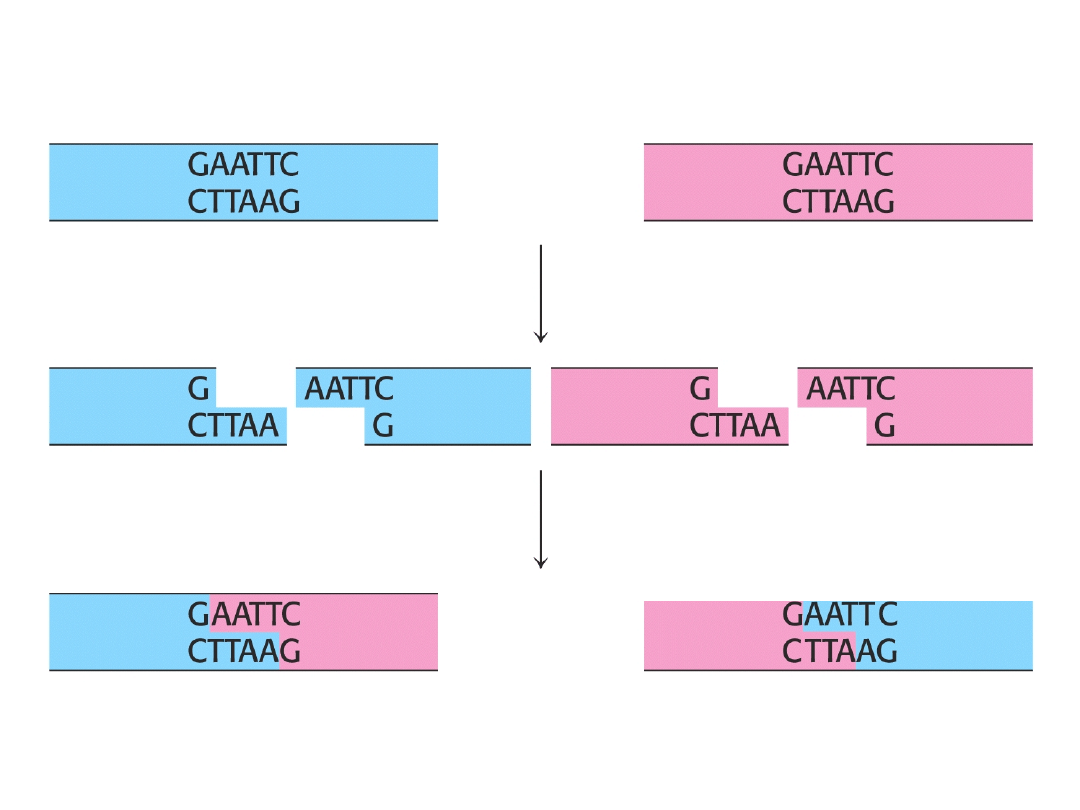
Trawienie DNA
enzymem EcoRI
Łączenie łańcucha DNA ligazą
DNA
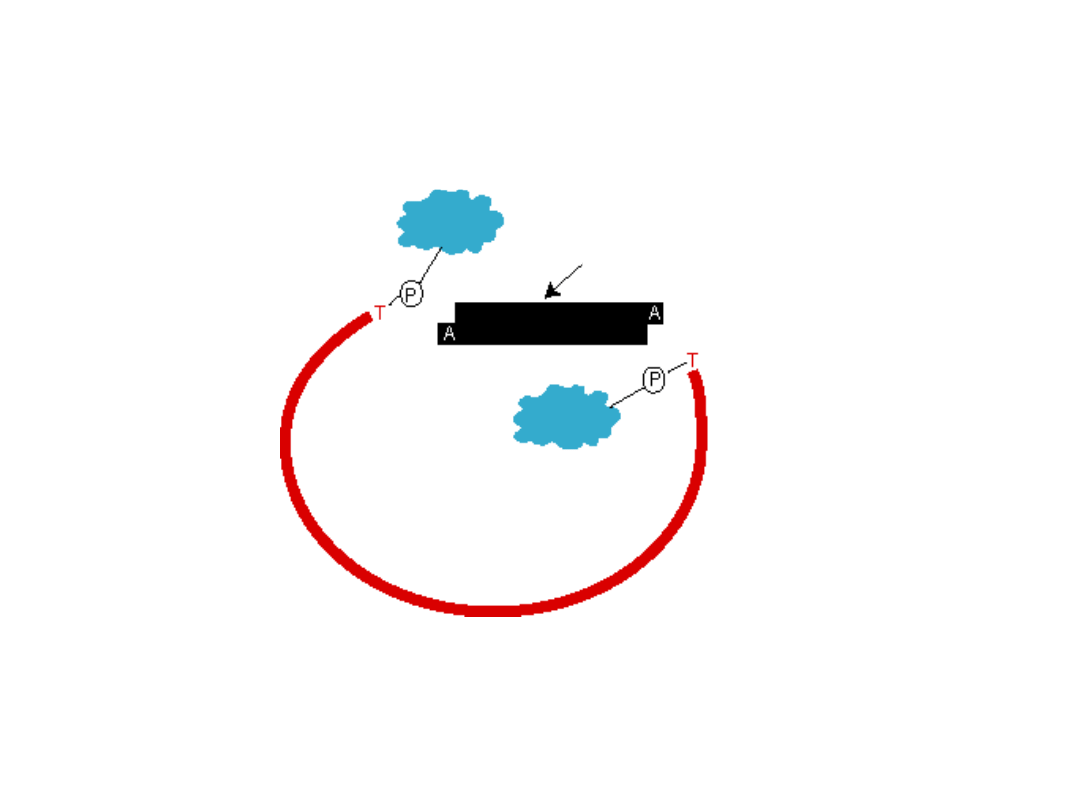
topoizomeraza
topoizomeraza
fragment
DNA
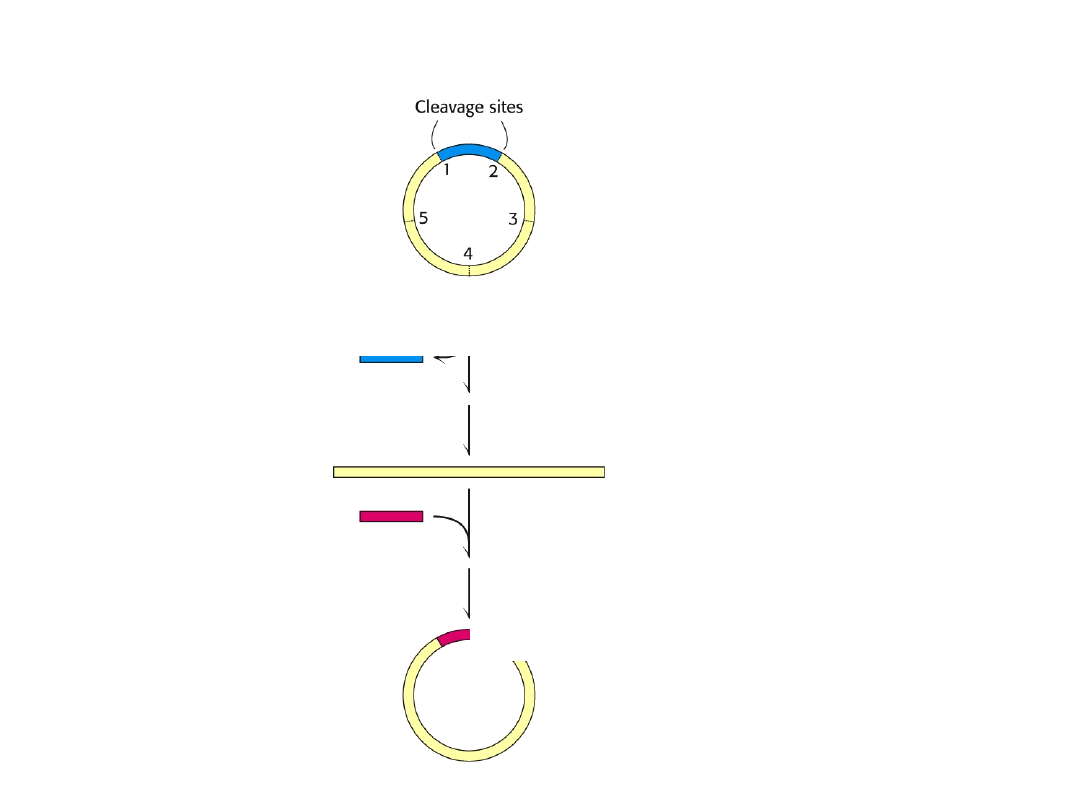
Miejsca cięcia enzymami
restrykcyjnymi w obrębie
polilinkera
Plazmid z
genem 1
(niebieski)
Trawienie enzymami
restrykcyjnymi,
Oczyszczanie
liniowego plazmidu
Ligacja oczyszczonego plazmidu
z DNA kodującym
gen 2
(czerwony)
,
Oczyszczanie uzyskanego
kolistego plazmidu
Plazmid z nowym genem
Gen oporności
na antybiotyk
Plazmid
rekombinowany Chromosom
bakteryjny
DNA genomowy
z genem docelowym
Miejsca cięcia
enzymu(ów)
restrykcyjnego(ych)
Ligacja
Plazmi
d
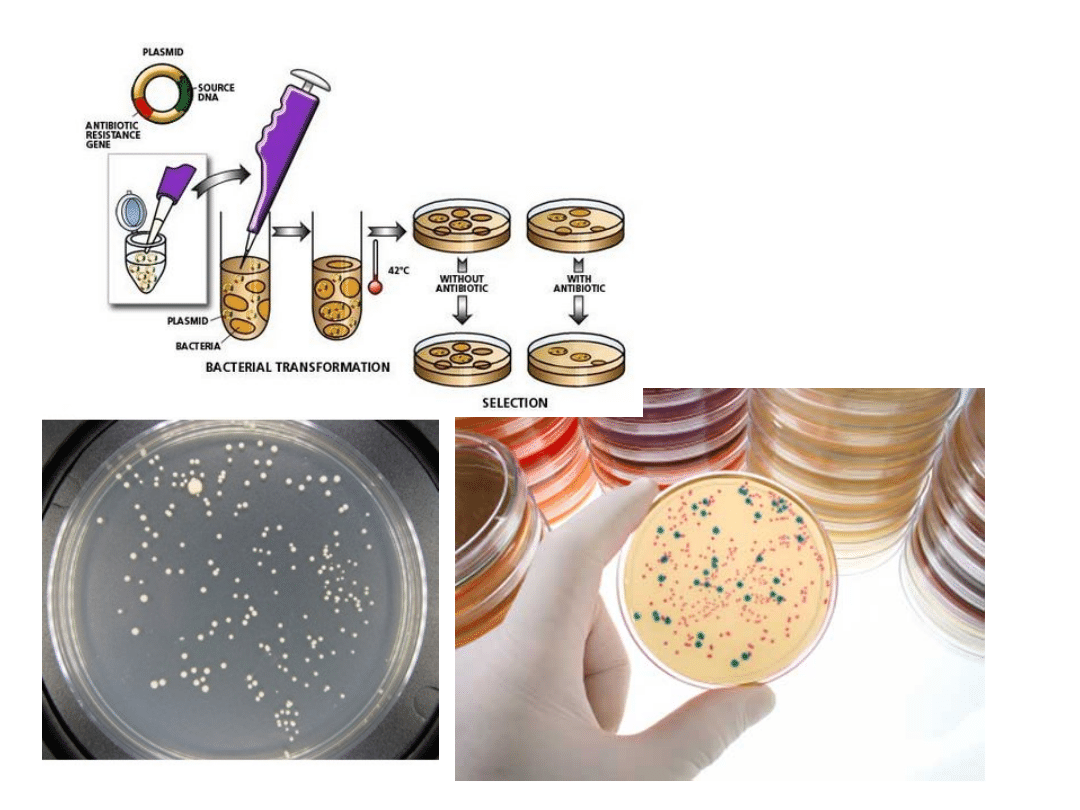
Selekcja
klonów
zawierających
skonstruowany
nowy plazmid
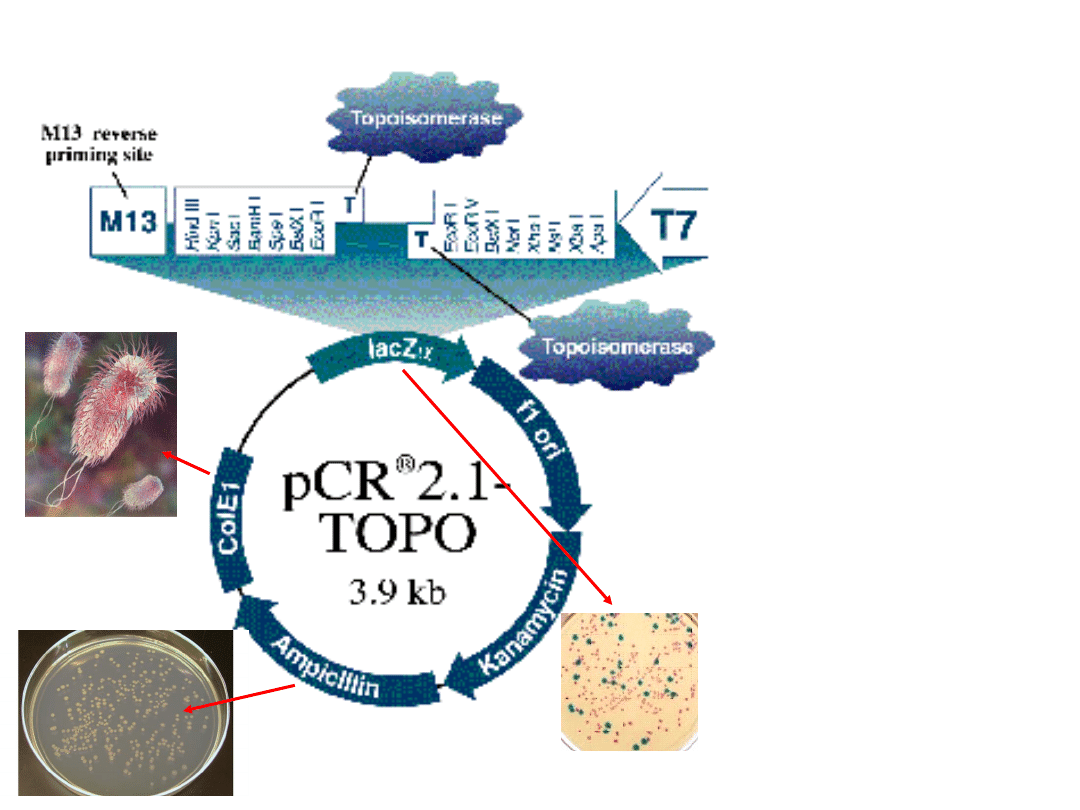
Selekcja klonów
zawierających
skonstruowany
nowy plazmid z
wykorzystaniem
-galaktozydazy
kodowanej przez
gen lacZ –
hydrolizuje
laktozę do
glukozy i
galaktozy lub
syntetyczne
substraty, np. X-
Gal
(galaktopiranozy
d z dołączonym
niebieskim
barwnikiem)

Film
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
Wyszukiwarka
Podobne podstrony:
Biol Mol wyklad 9
Biol Mol wyklad 8
Biol Mol wyklad 3
Biol Mol wyklad 9
Wykład biol mol ze Strzałką nr 2
Wykład biol mol ze Strzałką 2013, far, III rok IV sem, biologia molekularna, wykłady
seminaria biol mol onkogeneza, Płyta farmacja Poznań, III rok, Biologia molekularna, 2009, sem 6
biol mol
Test z biol.mol 2011, UG, MOLEKUŁY, biologia molekularna
pytania biol mol moje(2)
Pod Biol Mol i Biotech cw1, Studia
biol.medyczna, wykład - Ogólna budowa komórki, Ogólna budowa komórki
biol mol zadanie domowe3
biol mol kol 2 2
biol mol chyba to samo
biol mol 2014 e
prez biol.mol, prez
ĆWICZENIE II biol mol, far, III rok IV sem, biologia molekularna, II
biol kom wyklad 2103, Chemia środków bioaktywnych (umcs), BIOLOGIA KOMÓRKI
więcej podobnych podstron