
Mikromacierz DNA-
To płytka szklana lub plastikowa z naniesionymi w
regularnych pozycjach mikroskopowej wielkości
polami (ang. spots), zawierającymi różniące się od
siebie sekwencją fragmenty DNA. Fragmenty te są
sondami, które wykrywają przez hybrydyzację
komplementarne do siebie cząsteczki DNA lub RNA.

Zastosowanie mikromacierzy DNA:
Analiza jednonukleotydowych polimorfizmów DNA,
Określenie zmian w poziomie ekspresji genów,
Wykrywanie nowych transkryptów,
Określanie rejonów oddziaływań białko-DNA,
W medycynie,
W farmaceutyce,
W diagnostyce chorób zakaźnych.

Mikromacierze cDNA
Macierze
oligonukleotydowe, zwane
„chipami DNA”
Technologie
mikromacierzy DNA:

Mikromacierze cDNA
W mikromacierzach cDNA rolę sond odgrywają
fragmenty cDNA , zwykle o długości 0,5-2,0 kbp,
będące najczęściej produktami amplifikacji PCR,
uzyskanymi z bibliotek cDNA lub zbioru klonów, w
reakcji ze starterami specyficznymi dla wektora
lub genu. W mikromacierzach cDNA jeden gen na
płytce jest reprezentowany przez jeden rodzaj
sond naniesionych w formie kropelki – „spotu”, w
ściśle określonym miejscu , zdefiniowanym
współrzędnymi. Typowa wielkość średnicy spotów
wynosi 100-150 μm.

Zalety stosowania
mikromacierzy cDNA
niekonieczna znajomość sekwencji całego
genomu,
niski koszt eksperymentu,
możliwa równoległa analiza dwóch próbek
(badanej i kontrolnej) na jednej macierzy,
prosta procedura znakowania i odmywania,
duża elastyczność,
możliwość własnoręcznego zaprojektowania i
przygotowania mikromacierzy,

Wady stosowania
mikromacierzy cDNA:
prawdopodobieństwo wystąpienia hybrydyzacji
krzyżowej,
niezbędna matryca biologiczna do syntezy sond,
czasochłonne przygotowanie sond,
więcej możliwości wprowadzenia błędów (PCR),
problemy z powtarzalnością,
dwuniciowe sondy (konieczność denaturacji,
kompetycja wiązania).

Mikromacierze cDNA
dzielimy na:
Mikromacierze cDNA drukowane na
mikropłytkach szklanych
Mikromacierze cDNA drukowane na
filtrach nylonowych

W mikromacierzach
oligonukleotydowych sondy
stanowią krótkie oligonukleotydy,
zazwyczaj o długości 10-25 bp,
otrzymane na drodze syntezy
fotolitograficznej in situ na
powierzchni płytki. W chipach DNA
jeden gen jest reprezentowany
przez kilkanaście par różnych
oligonukleotydów. W tym przypadku
wielkość średnicy „spotów” mieści
się w granicach 2-20μm. W
zależności od ilości spotów
występujących na powierzchni,
wyróżnia się mikromacierze
oligonukleotydowe wysokiej (>10
tys.), średniej (1-10tys.) i niskiej
gęstości.
Macierze
oligonukleotydowe

Zalety stosowania
mikromacierzy
oligonukleotydowych
:
wysoka specyficzność,
prosta synteza (chemiczna) sond i ich
oczyszczanie,
duża powtarzalność,
jednoniciowe sondy (zbędna denaturacja,
ograniczenie tworzenia struktur drugorzędowych),
dostępność wielu komercyjnych mikromacierzy,
zwłaszcza dla znanych genomów (organizmów
modelowych).

Wady stosowania
mikromacierzy
oligonukleotydowych
:
wysoki koszt,
trudności w znalezieniu unikatowych sekwencji w
genomie,
duży wpływ składu nukleotydowego sond na proces
hybrydyzacji,
konieczna znajomość sekwencji docelowych,
skomplikowany proces projektowania sond
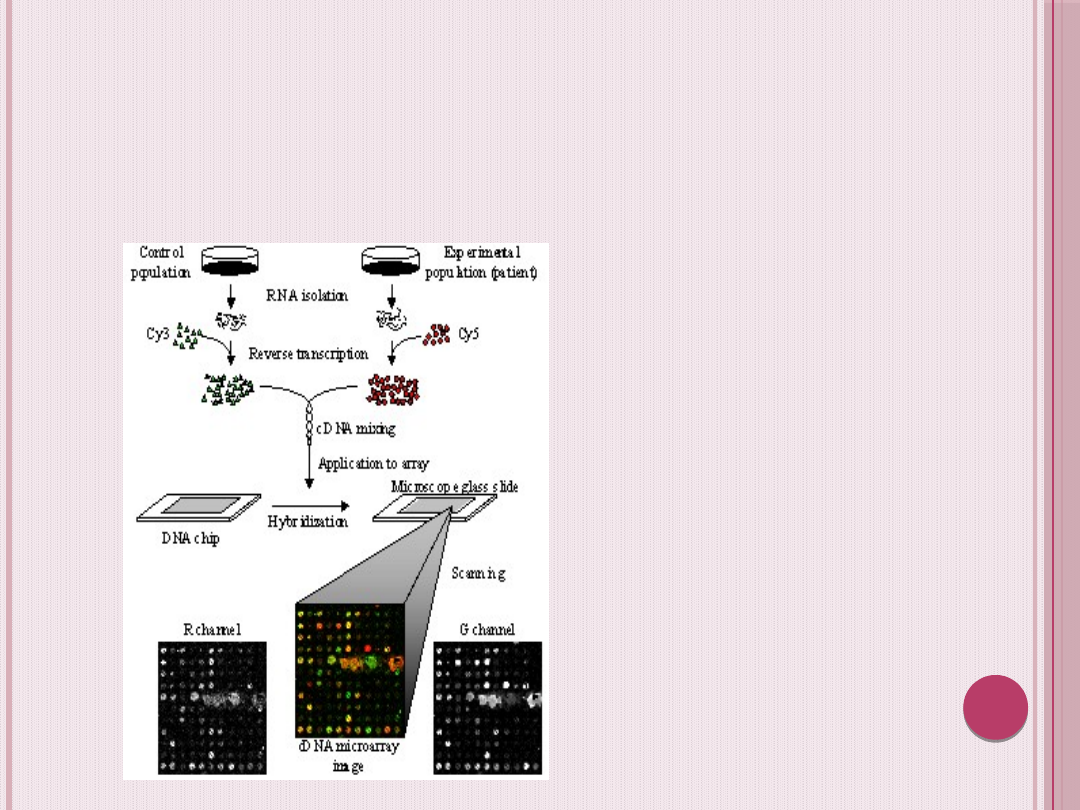
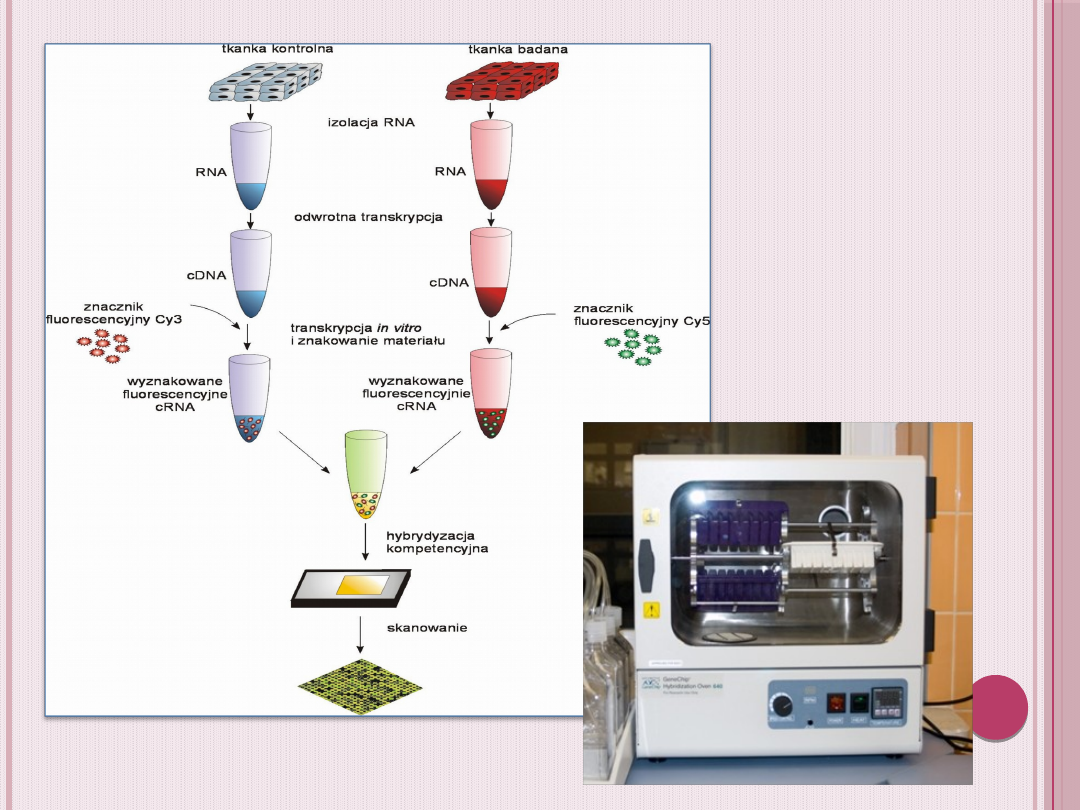
Eksperyment
mikromacierzowy:
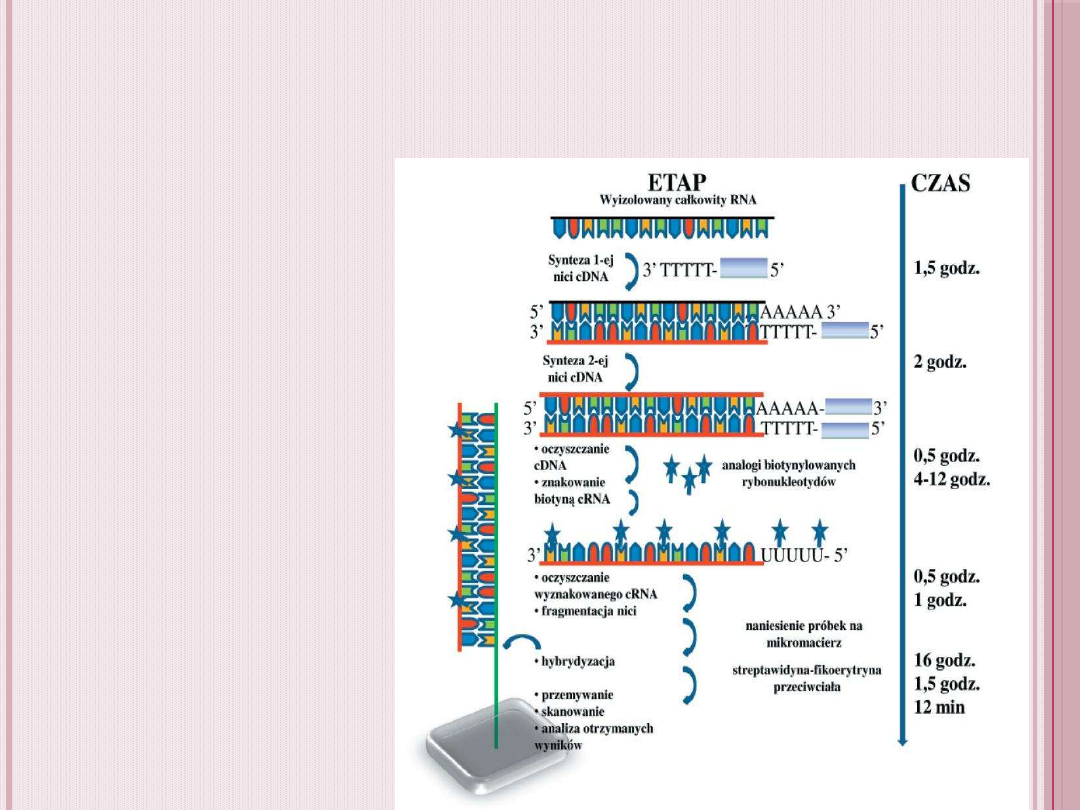
Przygotowanie materiału do
hybrydyzacji:
syntetyzowanie
cDNA w reakcji
odwrotnej
transkrypcji
powstanie cRNA w
wyniku reakcji
transkrypcji in vitro
wprowadzenie
zmodyfikowanych
nukleotydów z
dołączonymi
znacznikami
fluorescencyjnymi
fragmentacja i
hybrydyzacja z płytką
mikromacierzową
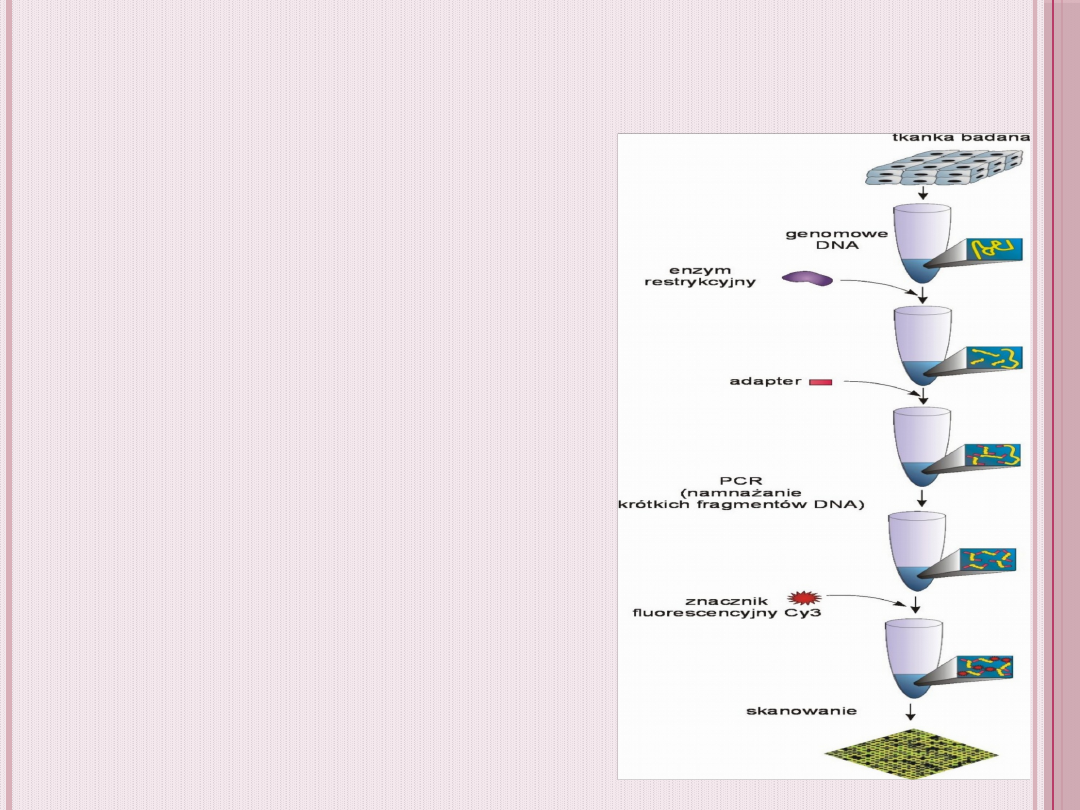
Znakowanie mikromacierzy:
Metoda bezpośrednia polegająca na
wprowadzeniu nukleotydów dUTP lub DTP
i:
• jednego znacznika fluoroscencyjnego
• dwóch różnych znaczników
fluorescencyjnych
Metoda bezpośrednia polegająca na
fragmentacji badanego materiału i
znakowaniu jego końców (Metoda ta
wykazuje największą efektywność
znakowania dla jednoniciowego DNA,
można ją jednak wykorzystać również
do znakowania dwuniciowego DNA oraz
RNA.)
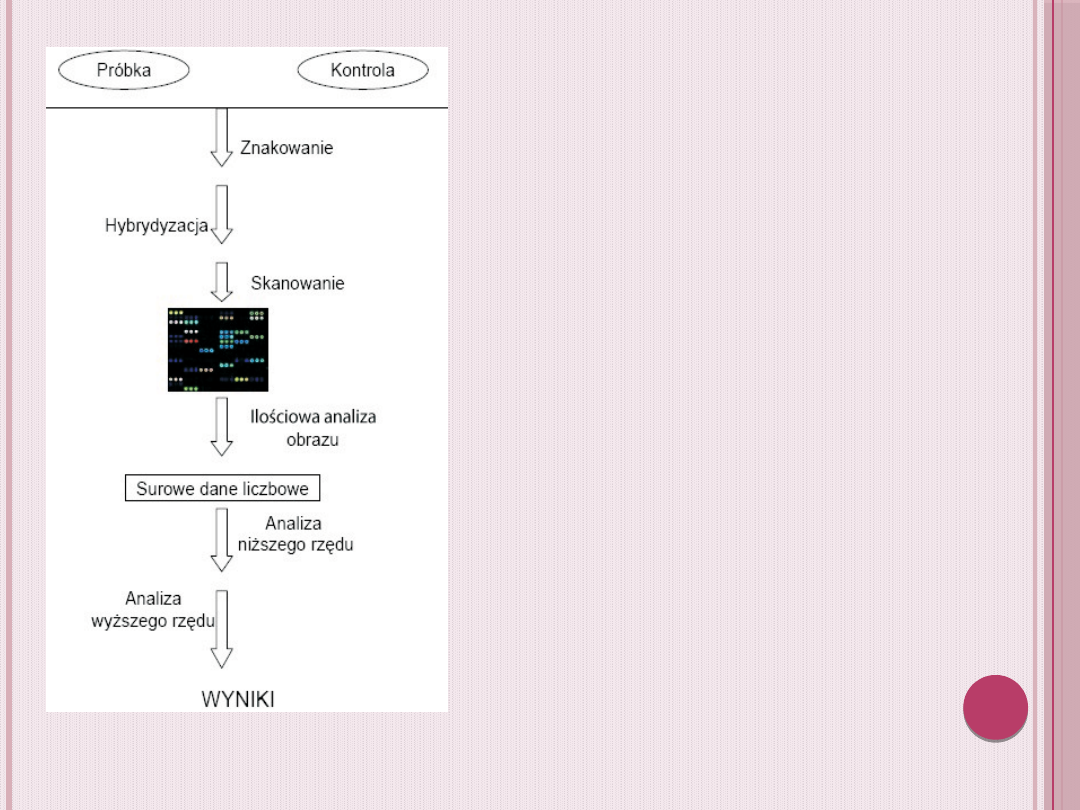
Opis eksperymentu
mikromacierzowego:
Badany RNA poddaje się przepisaniu
na cDNA (odwrotna transkrypcja),
następnie fluorescencyjnie znakuje,
hybrydyzuje z DNA
immobilizowanym na płytce po czym
następuje skanowanie i zbieranie
danych. W technice tej wykorzystuje
się dwa barwniki fluorescencyjne,
najczęściej są to Cy3 i Cy5. Każdym z
barwników znakuje się DNA z innej
puli, dzięki czemu na jednej płytce
można porównywać ze sobą dwie
tkanki i analizować różnice w
ekspresji poszczególnych genów.
Informacje o hybrydyzacji uzyskuje
się za pomocą skanera optycznego
zapisującego fluorescencję dla
każdego punktu mikromacierzy.

Na każdym z etapów sprawdza się jakość mikromacierzy wykluczając
takie, które mają poważne defekty techniczne. Następny etap zwany jest
filtrowaniem genów i ma na celu wybranie tzw. genów różnicujących,
których ekspresja zmienia się istotnie w badanych warunkach, oraz
odrzucenie tych, które nie dają żadnego sygnału. Na podstawie tak
zredukowanego zestawu genów prowadzi się analizy wyższego rzędu,
poszukując grup genów o podobnym/odmiennym profilu ekspresji. Na
zakończenie uzyskane wyniki poddawane są interpretacji biologicznej,
polegającej na powiązaniu obserwowanych zmian w poziomie ekspresji
genów z fizjologicznymi bądź patologicznymi procesami zachodzącymi w
badanym organizmie.
W kolejnym etapie
każdemu punktowi
przyporządkowana zostaje
liczba określająca
natężenie fluorescencji.
Uzyskane w ten sposób
„surowe” dane liczbowe
poddawane są najpierw
normalizacji lokalnej (w
obrębie pojedynczej
płytki), a następnie
globalnej
(w obrębie wszystkich
mikromacierzy
składających się na
eksperyment).

KONIEC
Przygotowała:
Monika Wrońska.
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
Wyszukiwarka
Podobne podstrony:
Met. izol. oczysz.DNA dla studentów, Biologia molekularna
Genetyka ogólna - pytania na egzamin, Genetyka, DNA, biologia molekularna, techniki
Ekstrakcja DNA, 1 rok, Biologia molekularna, ćw 1-2
Prelekcja 10 - cz 1 - Mutacje genowe (punktowe) i polimorfizmy DNA, Medycyna, Biologia molekularna Ś
IZOL.DNA Z ŻELI, Biotechnologia notatki, Genetyka - biologia molekularna
Biologia molekularna DNA
stez molowe DNA, biologia molekularna umed
prezentacja z boicudow, Płyta farmacja Poznań, III rok, Biologia molekularna, 2009, sem 6
Apoptoza, Genetyka, DNA, biologia molekularna, techniki
Struktura DNA, 1 rok, Biologia molekularna, ćw 1-2
wykład - Polimeraza DNA, Biotechnologia notatki, Genetyka - biologia molekularna
MUTACJA, Genetyka, DNA, biologia molekularna, techniki
BUDOWA KWASU DNA, Prezentacje Biologiczne PPT, DNA-budowa i funkcje
IZOLACJA DNA ROŚLINNEGO METODĄ MIKRO C, Biotechnologia notatki, Genetyka - biologia molekularna
Izolacja DNA, Biologia molekularna
11. Charakterystyka mitochondrialnego DNA-ok, Biologia II, Biologia molekularna
biblioteki DNA, biologia molekularna
Replikacja DNA, biologia molekularna
więcej podobnych podstron