
Czynniki, które decydują o wyborze metody diagnostycznej
Właściwości próbki
Charakterystyka celu molekularnego
Czułośd i specyficznośd metody
Powtarzalnośd
Potencjał różnicujący metody
Stopieo trudności metody (doświadczenia personelu)
Zaplecze badawcze (sprzęt jakim dysponujemy)
Łatwośd interpretacji wyników
Czas wymagany do analizy
Całkowite koszty stosowanej metody
Koszty pojedynczej próbki
Wybór metody diagnostycznej zależy od poruszanego problemu
Wykrywanie
Identyfikacja
Typowanie(różnicowanie)
Badania taksonomiczne; filogeneza
TECHNIKI NIEOPARTE NA AMPLIFIKACJI KWASÓW NUKLEINOWYCH
Analiza profili plazmidowych (ang. PPA, Plasmid Profile Analysis)
Po raz pierwszy została zastosowana w badaniach epidemiologicznych w roku 1988
Uważa się ze szczepy izogeniczne (należące do tej samej grupy) maja identyczny zestaw plazmidów
Technika prosta, ale ograniczona do szczepów posiadających plazmidy, szczepy bezplazmidowe są nietypowalne
Komórki niektórych gatunków bakterii, nawet szczepów, nabywają i tracą plazmidy spontanicznie
REAP - Restriction Enzyme Analysis of Plasmid
W przypadku dużych plazmidów (100-150 kpz) ze względu na ich niską rozdzielczośd w tradycyjnej elektroforezie stosuje się trawienie
DNA plazmidowego komercyjnymi restryktazami, co pozawala na różnicowanie szczepów na podstawie różnorodności profili restrykcyjnych -
REAP.
Liczba i wielkośd uzyskanych fragmentów DNA determinowana jest przez długośd sekwencji rozpoznania dla określonego enzymu
restrykcyjnego oraz charakter trawionego DNA (% składu par zasad GC).
Statystycznie w przypadku DNA z zawartością par GC około 50% 4-nukleotydowa sekwencja rozpoznania powinna występowad co 256
pz, 6-nukleotydowa co 4 kpz, a sekwencja 8-nukleotydowa co 65 kpz.
Zastosowanie
Analiza szpitalnych izolatów K.pneumoniae (zawierają co najmniej 3 plazmidy)
Badanie rozprzestrzeniania się lekooporności kodowanej plazmidowo
Przydatna w śledzeniu zakażeo, których przyczyna są bakterie z rodzaju Staphylococcus
Wady
Szybki transfer DNA na drodze koniugacji wykazuje podobne profile u różnych bakterii
Mała uniwersalnośd
Brak możliwości badania interakcji pomiędzy szczepami
METODY OPARTE NA HYBRYDYZACJI
Metoda hybrydyzacji wykorzystuje zjawisko tworzenia dwuniciowych kompleksów pomiędzy komplementarnymi, jednoniciowymi
odcinkami kwasów nukleinowych.
Metoda ta umożliwia lokalizację określonych sekwencji w długiej cząsteczce DNA, co pozwala na porównywanie podobieostwa różnych
fragmentów kwasów nukleinowych.
Stabilnośd dwuniciowego kompleksu jest tym większa im dłuższe odcinki wykazują komplementarnośd nukleotydów.
W metodzie hybrydyzacji mogą byd porównywane fragmenty kwasów nukleinowych, pochodzące zarówno od tego samego jak i
różnych organizmów.
Hybrydyzacja może mied miejsce zarówno pomiędzy dwoma
Cząsteczkami DNA
DNA i RNA
Hybrydyzacja
1. Hybrydyzacja standardowa: sonda wyznakowana w roztworze, niewyznakowany target związany z fazą stałą
2. Reverse nucleic hybridization assay - znakowany target w roztworze, nieznakowana sonda związana z podłożem stałym
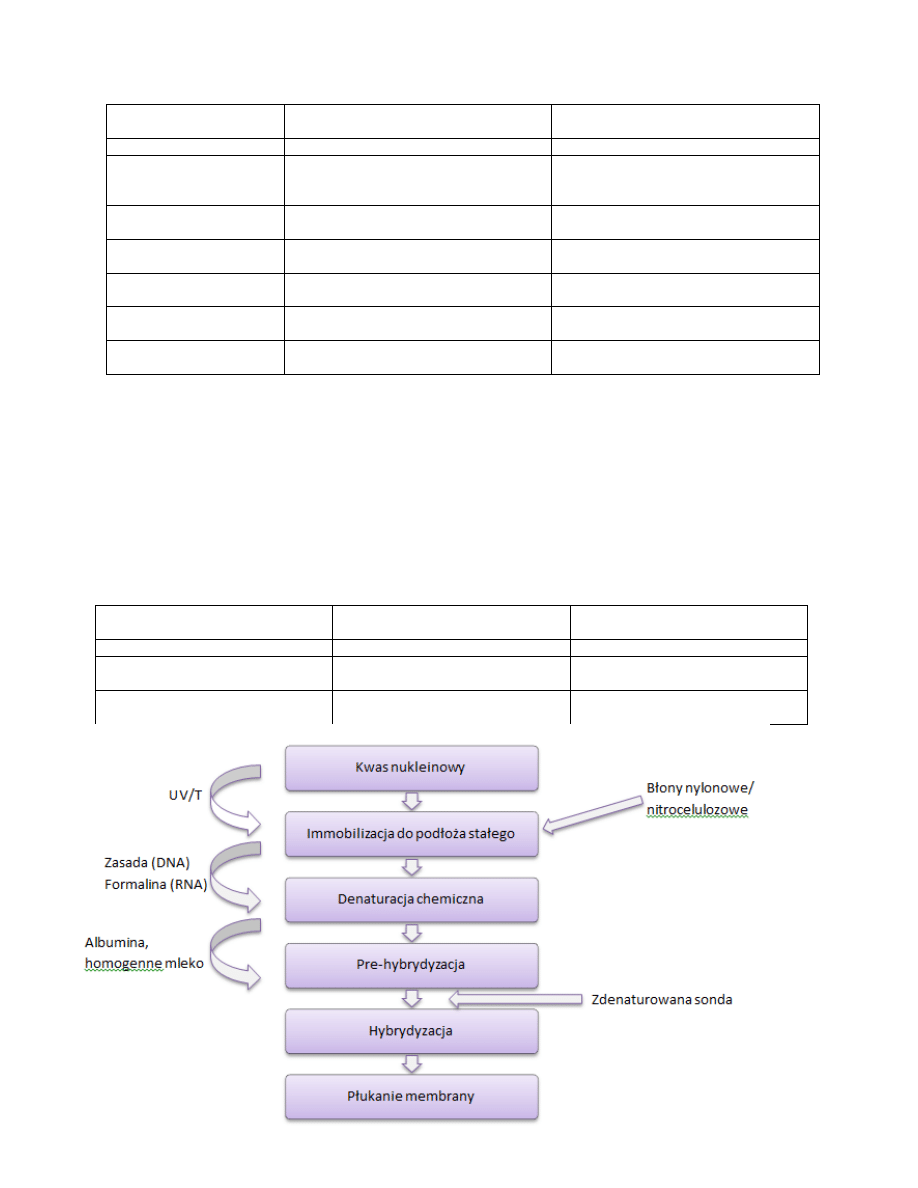
STANDARDOWE TECHNIKI HYBRYDYZACYJNE
Techniki "blottingu"
DOT – kropka
SPLOT – plamka, punkcik
SLOT – szczelina, otwór
Southern blotting
Northern blotting
Reakcje hybrydyzacji blotting są wysoce swoiste, ich zasada polega na zastosowaniu jednoniciowego kwasu nukleinowego (DNA, RNA)- sondy,
który komplementarnie wiąże się z DNA lub RNA znajdującym się w próbce badanej.
TECHNIKI HYBRYDYZACYJNE OPARTE O ODWROTNĄ HYBRYDYZACJĘ
Rodzaj hybrydyzacji
Znakowany target w roztworze
Niewyznakowana sonda na podłożu
stałym
Odwrotny dot-blot
DNA
Oligonukleotydy związane z membraną
Mikromacierze cDNA
DNA
DNA mechanicznie związane do szkiełka
mikroskopowego
Mikromacierze oligonukleotydowe
(Chipy DNA)
DNA
Oligonukleotydy syntezowane na
szkiełku
Rodzaj hybrydyzacji
Znakowana sonda w roztworze
Niewyznakowany target związany z fazą
stałą
Dot – blot
DNA lub RNA, często oligonukleotydy
DNA czy RNA związane z membraną
Southern blot
Dowolne sondy
Często genomowe DNA, trawione enzymem
restrykcyjnym, rozdzielone elektroforetycznie
i przeniesione na membranę (transfer)
Northern blot
Dowolne sondy
RNA rozdzielone elektroforetycznie, transfer
na membranę
Hybrydyzacja do
chromosomu in situ
Wyznakowane genomowe DNA
DNA chromosomu lizowanych komórek na
szkiełku mikroskopowym
Hybrydyzacja do tkanki in
situ
Wyznakowana antysensowna sonda rRNA lub
oligonukleotydy
RNA wewnątrz komórek budujących tkanki
związane ze szkiełkiem mikroskopowym
Hybrydyzacja do kolonii in
situ
Dowolne sondy
Komórki – kolonie przeniesione na agar,
potem membranę
Hybrydyzacja do łysinki in
situ
Dowolne sondy
Łysinki z agaru przeniesione na membranę
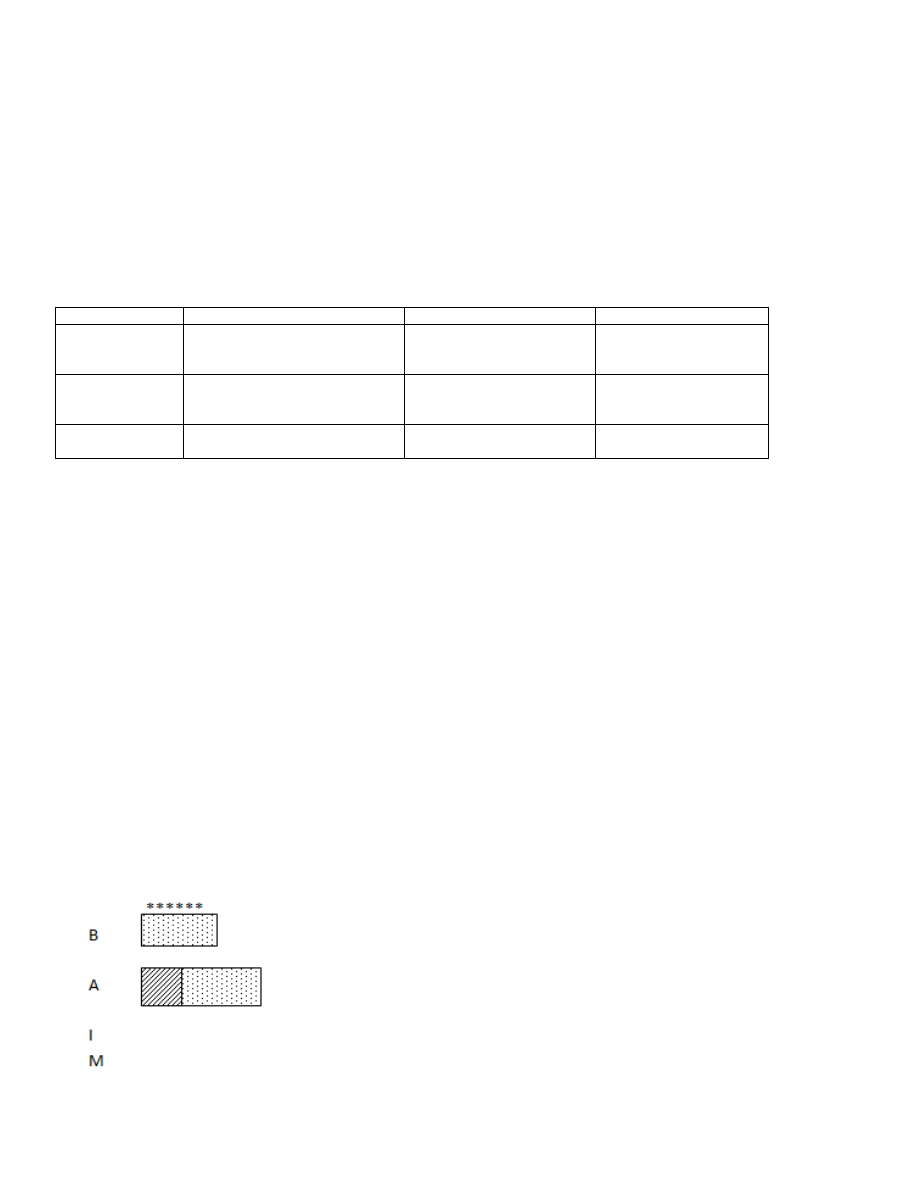
We wszystkich wariantach metody hybrydyzacji jeden z elementów uczestniczący w procesie pełni rolę sondy molekularnej o określonej
sekwencji nukleotydów i uzyskiwanej na drodze
Klonowania
Syntezy chemicznej
Sondy genetyczne:
Sondy oligonukleotydowe
Sondy polinukleotydowe
Sondy jednego locus
Sondy wielu loci
Środowisko hybrydyzacji ma większy wpływ na krótsze sondy niż na sondy dłuższe.
Charakterystyka sond i ich wielkośd
Typ
DNA
RNA
Oligonukleotydy
Źródło pochodzenia
DNA komórkowe - klonowanie lub
PCR
Transkrypcja insertu po
klonowaniu do odpowiedniego
wektora
Synteza chemiczna
Charakterystyka
dsDNA od 0,1 do kilkuset kpz dla
konwencjonalnych klonów DNA 0,1
do 20 i > kpz dla produktów PCR
ssDNA; długośd do kilku tysięcy
zasad
ssDNA; 15-50 nt
Znakowanie
Synteza DNA przez polimerazę DNA
Transkrypcja z klonowanego
DNA
Wyznakowane kooce przez
kinazę polinukleotydową
Sonda hybrydyzuje do
DNA przytwierdzonego na filtrze
DNA preparatów cytologicznych
DNA preparatów histologicznych
Sonda wiąże się tylko do fragmentów DNA, które zawierają sekwencje komplementarne do sekwencji sondy.
Do zidentyfikowania powstałych kompleksów sondę znakuje się
Izotopowo (audioradiografia)
W reakcjach barwnych
Chemilumiscencyjnie
Znakowanie sond DNA i RNA
Znakowanie nowych nici podczas syntezy in vitro DNA/RNA
o
Przemieszczanie pęknięd (nick-translation)
o
Znakowanie metoda wydłużania startera (ang. random primed labeling)
o
Za pośrednictwem PCR
o
Fotoznakowanie
o
Znakowanie RNA w systemie transkrypcji "in vitro"
Znakowanie 5’
o
Z użyciem alkalicznej fosfatazy
o
Metoda wymiany 5'-koncowego fosforanu
Znakowanie kooców 3'
o
Z użyciem terminalnej transferazy dla krótkich oligonukleotydów
Hybrydyzacja kanapkowa
A – sonda molekularna
B – sonda znakowana
Hybryd (A-B) przyłączony do identyfikowanego kwasu
nukleinowego
M - membrana
I - identyfikowany kwas nukleinowy
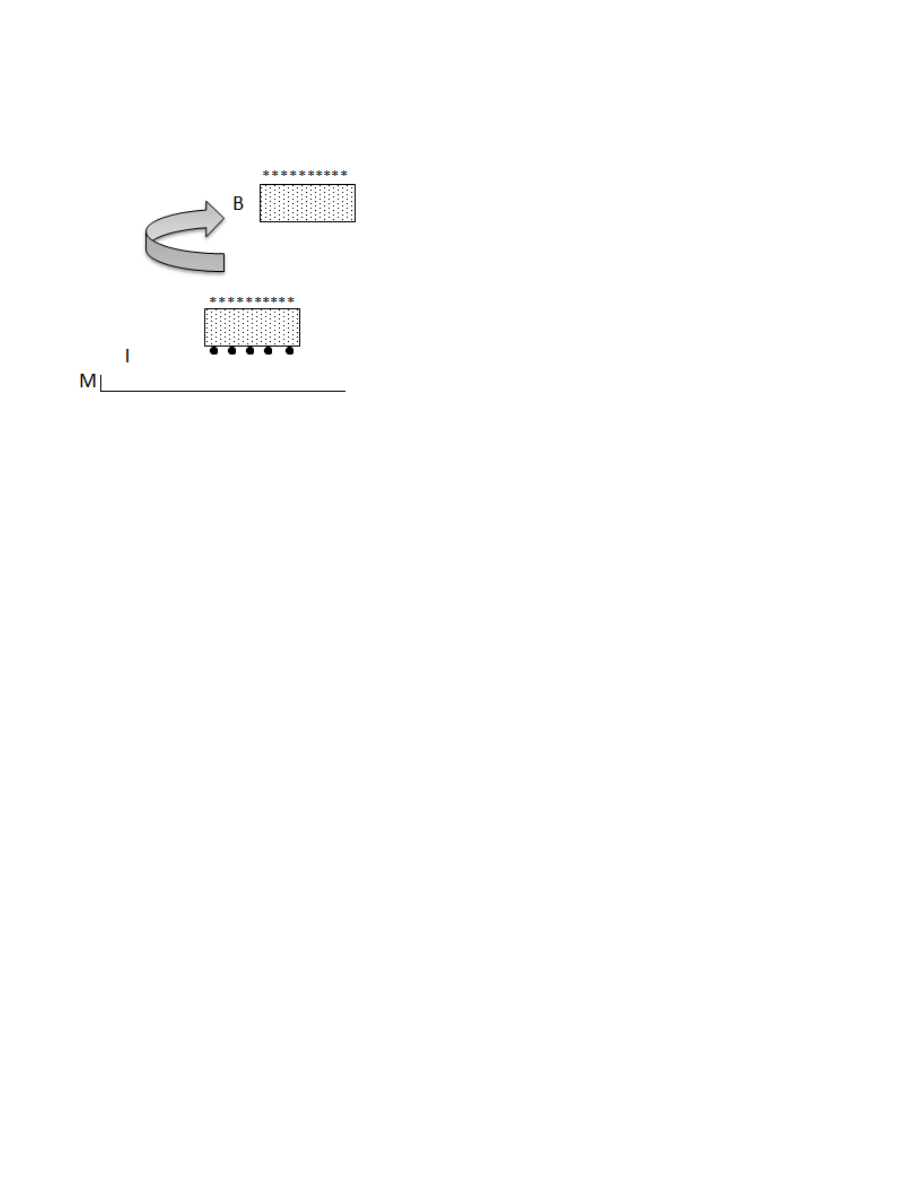
Hybrydyzacja w wyniku konkurencji między sondami
A – sonda nieznakowana
B – sonda znakowana
Z - Hybryd (A-B)
M - membrana
I - identyfikowany kwas nukleinowy
Zalety sond molekularnych stosowanych w hybrydyzacji
Stabilnośd w wysokich temperaturach i wysokim pH (są bardziej stabilne niż białka)
Opornośd na działanie rozpuszczalników organicznych i różnych związków chemicznych (duże znaczenie przy przygotowywaniu próbek
do analiz – oczyszczanie komórek i tkanek z płynów ustrojowych)
Wysoka specyficznośd (większa niż przeciwciał)
Bardzo wysoka czułośd, nawet do 1 μg kwasów nukleinowych odpowiada do 10
6
komórek bakteryjnych czy cząsteczek wirusa
Znajomośd układu sekwencji nukleotydów, do której hybrydyzuje sonda nie jest konieczna
Hybrydyzacja ze strawionym DNA enzymami restrykcyjnymi pozwala na identyfikację zmian sekwencji DNA
Zastosowanie technik hybrydyzacyjnych
1. Wykrywanie specyficznych fragmentów DNA do analizy porównawczej różnych szczepów bakteryjnych
W diagnostyce chorób genetycznych
2. Do ustalenia sekwencji ulegających ekspresji (northern hybrydyzacja)
3. Hybrydyzacja „zoo” do ustalania pokrewieostwa międzygatunkowego
4. Hybrydyzacja „in situ”
Do określania miejsc genów na chromosomie (mapowanie)
Do wizualizacji komórek, w których dochodzi do syntezy określonego rodzaju mRNA
5. Hybrydyzacja „in situ” FISH
Polimorfizm genetyczny
1. Występowanie w populacji więcej niż jednej wersji allelu z częstością większą niż wynikająca z ogólnej częstości mutacji
2. Występowanie najczęstszego allelu w danym locus z częstością mniejszą niż 99%
3. Modyfikacje sekwencji genu, które są akceptowane przez organizmy (zmiana taka zostaje utrwalona i funkcjonuje jako oddzielny
genotyp)
Allel polimorficzny może rozprzestrzeniad się w populacji
Często nie znamy jego wpływu…
Polimorfizm genetyczny jest przyczyną zmienności wewnątrzkomórkowej
Występuje bardzo często w rejonach niekodujących DNA
Skąd się biorą allele?
Błędy replikacji
Mutacje (spontaniczne; indukowane przez środowiskowe czynniki mutagenne: UV, promieniowanie jonizujące, policykliczne
węglowodany aromatyczne, leki) -> powstawanie genów nieaktywnych funkcjonalnie – pseudogenów lub nowych aktywnych genow
Rodzaje polimorfizmów
Mutacje obejmujące większe fragmenty genomu, np. zmiany krotności krótkich tandemowych powtórzeo w sekwencjach
powtarzających się mikrosatelitarnych i minisatelitarnych (STRP)
Polimorfizm długich fragmentów restrykcyjnych (RFLP)

Technika Southern-blott
do analizowania specyficznych sekwencji DNA w mieszaninie genomowego DNA w różnych sekwencjach, m.in. będących wynikiem polimorfizmu
długich fragmentów restrykcyjnych
VNTR – Jest to szczególny rodzaj polimorfizmu długości fragmentów restrykcyjnych (VNTR, variable number of tandem repeat)
VNTR-insercja wielu kopii (długośd 10-100bp) w tandemie tzw. sekwencji mini satelitarnych pomiędzy dwa miejsca restrykcyjne
Nawet rodzeostwo ma różne allele genu niosącego sekwencje minisatelitarne
„DNA fingerprints”
pokazuje identycznośd genetyczną (VNTR) bliźniąt jednojajowych (monozygotycznych); technika hybrydyzacji Southerna
Znaczenie polimorfizmów dla zdrowia może się ujawniad pod wpływem czynników zewnętrznych, tj.
o
Kancerogeny chemiczne (m.in. palenie papierosów)
o
UV
o
Czynniki dietetyczne (antyoksydanty, flawonoidy, witaminy antyoksydacyjne, nienasycone kwasy tłuszczowe)
Znaczenie polimorfizmów genetycznych
o
Badanie polimorfizmu jest przydatne w diagnostyce prenatalnej chorób genetycznych (nosiciele heterozygotyczni chorób)
o
Badanie polimorfizmu może służyd wykrywaniu chorób o wysokim lub niskim ryzyku chorób wieku dojrzałego: …
o
Przydatne w kryminalistyce
Zastosowanie technik opierających się o hybrydyzację w diagnostyce
Test paskowy - opiera się na hybrydyzacji DNA-DNA. Sonda zawiera częśd reporterową i częśd wychwytującą interesujący nas target. Sonda
wychwytująca posiada ogon poli-dA, który hybryduje z oligonukleotydem poli-dT związanym z paskiem. Stosowana do identyfikacji
mikroorganizmów np. Mycobacterium tuberculosis
Obecnie jako sondy genetyczne stosowane są
Fragmenty kwasów nukleinowych (DNA,RNA) izolowanych z komórek lub uzyskiwanych w wyniku reakcji PCR
Oligonukleotydy syntezowane chemicznie
Charakterystyczne fragmenty genomowego DNA (sekwencje …)
rRNA, tRNA
Do wykrywania sekwencji kwasu nukleinowego w komórce lub odpowiednim rejonie chromosomu stosowana jest hybrydyzacja in situ
Fluorescencyjna FISH
Chromogenna CISH
Porównawcza hybrydyzacja genomowa – fiber FISH
Hybrydyzacja in situ (ISH)
Technika ISH pozwala na specyficzne rozpoznawanie sekwencji DNA lub RNA w tkankach lub pojedynczych komórkach.
Technikę tę można stosowad na
Tkankach mrożeniowych
Tkankach parafinowych
Preparatach cytologicznych
Cytospinach (preparaty szpiku kostnego)
ISH znalazła zastosowanie w diagnostyce medycznej w następujących dziedzinach
Wirusologia
Wykrywanie patogenów (bakterie, grzyby, parazyty)
Onkologia
Endokrynologia
-Ocenę preparatów hybrydyzacji in situ przeprowadza się w mikroskopie świetlnym, uwzględniając intensywnośd reakcji histochemicznej.
-Stosując metodę ISH wykorzystując komplementarne sondy (DNA lub RNA) można wykrywad kwasy nukleinowe wirusów i prowirusów w
preparatach tkankowych.
-Dzięki bardzo wysokiej czułości pozwalającej na wykrywanie stosunkowo krótkich fragmentów genomu, możliwe jest wykrycie pojedynczych
prowirusów w komórce.
Najczęściej stosuje się sondy wykrywające kwasy nukleinowe wirusów
HPV – wirus brodawczaka ludzkiego (ang. human papilloma virus)
HSV – wirus opryszczki (ang. herpes simplex virus)
Metoda polega na hybrydyzacji znakowanej sondy z utrwalonym preparatem z tkanki.
Sondy, które nie związały się z komplementarnymi sekwencjami są z łatwością odmywane z tkanki.
Sonda jest wyznakowana
Fluoroforem, gdzie detekcja odbywa się przez obserwację mikroskopową w obecności sekcji emitujących, po wzbudzeniu światło
charakterystyczne dla danego fluoroforu
Haptenem, gdzie możliwe są różne barwienia – od enzymatycznego, gdzie enzym sprzężony z przeciwciałem rozpoznającym antygen
przekształca substrat w nierozpuszczalny barwny produkt

Chemicznie, gdzie np. złoto koloidalne sprzężone z przeciwciałem powoduje wytrącenie się srebra. Ten typ detekcji pozwalający na np.
wykrycie roli niektórych wirusów w nowotworzeniu
ISH (hybrydyzacja in situ)w wykrywalności patogenów pozwala na identyfikację
Bakterii
Mycobacterii
Grzybów
Parazytów
W tego typu analizach najczęściej stosowaną sondą jest DNA natomiast celem molekularnym rRNA.
!! Dlaczego?
rRNA – świadczy o obecności komórek żywych, dających efekt np. w postaci zachorowania (w przypadku identyfikacji DNA dotyczy również
komórek martwych), odpowiedzi immunologicznej;
w komórkach żywych rRNA występuje znacznie więcej kopii niż DNA (kopii genów) -> większa czułośd;
w komórkach występuje duża ilośd kopii rRNA do ilości kopii genów.
-Dzięki tej technice wykrywa się obecnośd Helicobacter pylori w badaniach histopatologicznych fragmentów układu pokarmowego.
-Można wykryd Chlamydia w różnych stanach chorobowych izolowane z układu oddechowego i płciowego.
-Opracowano również protokół identyfikacji Lagionelli pneumophila izolowanej z przewodu oddechowego wykorzystujący ISH.
Dzięki ISH stosunkowo łatwo odróżnid patogenne grzyby krwi tj.
Candida albicans
C.glabrata
C.krusei
C.parapsilosis
Test został opracowany w oparciu o użycie czterech różnych sond równocześnie.
Poprawna identyfikacja genetyczna drożdży jest bardzo istotna, bo C.glabrata i C.krusei są oporne na leki (np. fukonazol) używana w zwalczaniu
C.albicans.
Technika ta pozwala również na lokalizację genów w chromosomach lub ich amplifikację. Powszechnie stosuje się ją obecnie do wykrywania
protoonkogenów HER-2/neu czy też topoizomerazy I i II α.
W przypadku lokalizacji określonych genów w technice ISH i FISH stosuje się trzy rodzaje sond molekularnych
Specyficzne komplementarne do określonego regionu chromosomu, mają zastosowanie w identyfikacji rearanżacji submikroskopowych
onkogenów (HER-2/neu) i unikatowych sekwencji telomerowych
α-satelitarne specyficzne do sekwencji repetytywnych na chromosomie; tzw. sondy telomerowe i centromerowe
Malujące: pokrywają cały chromosom lub jego ramiona; stosowane głównie w identyfikacji chromosomów markerowych, translokacji
złożonych i addycji niewiadomego pochodzenia
FISH (hybrydyzacja fluorescencyjna)
Jednym ze sposobów znakowania sond jest stosowanie barwników fluorescencyjnych (fluorochromów), które w koocowym etapie umożliwiają
detekcję z wykorzystaniem mikroskopu fluorescencyjnego
Zastosowanie FISH w genetyce medycznej
Badania podstawowe
Analiza aberracji chromosomowych
Mapowanie genów i sekwencji DNA
Detekcja genów ulegających amplifikacji
Badanie ekspresji genów
Ewolucjonizm porównawczy
Genetyka kliniczna
Diagnostyka submikroskopowych aberracji chromosomowych
Identyfikacja złożonych aberracji struktury chromosomów
Identyfikacja dodatkowego materiału genetycznego
Identyfikacja chromosomów markerowych
1S0zybka diagnostyka aneuploidii chromosomowych
Wyszukiwarka
Podobne podstrony:
więcej podobnych podstron