
Mateusz Sturgulewski
gr. 3
Izolacja kwasów nukleinowych z materiału ludzkiego
Izolacja DNA genomowego z krwi metodą fenol-chloroform:
200 µl krwi wykorzystano z wcześniej rozporcjonowanej próbki 200 µl w 1,5 ml probówce typu Eppendorf.
Liza komórek została przeprowadzona metodą z użyciem 5x stężonego buforu NH
4
Cl. Jony NH
4
+
, a także K
+
(również znajdujące się w buforze dzięki obecności 10 mM KHCO
3
)
neutralizują ujemny ładunek błony
komórkowej co doprowadza do jej destabilizacji i lizy komórek znajdujących się we krwi (głównie
erytrocytów). Tris –HCl utrzymuje fizjologiczne pH, tak aby nie doszło do denaturacji genomowego DNA
podczas lizy. Następnie komórki inkubowano przez 5 minut w temperaturze pokojowej i zwirowano według
protokołu.
1x stężony bufor TAE przygotowano z 50x stężonego roztworu stock. Frakcję jądrową przemyto buforem PBS i
wirowano, następnie osad zawieszono w 750 µl buforu TAE – tak przygotowana próbka jest gotowa do
zamrożenia i może byd wykorzystana w późniejszych analizach.
Następnie dokonano ekstrakcji mieszaniną fenol:chloroform. Jest to standardowa procedura oczyszczania
DNA z zanieczyszczeo białkowych. Po kilkuminutowej inkubacji DNA przechodzi do fazy górnej – wodnej.
Natomiast zanieczyszczenia białkowe przechodzą do fazy dolnej – fazy fenol:choloroform. Podczas dwiczeo
inkubacja z mieszaniną trwała 10 minut, następnie rozdzielono fazy poprzez łagodne wirowanie.
Rozdzielono obie fazy i fazę wodną ponownie ekstrahowano mieszaniną fenol:choloroform. Fazy ponownie
rozdzielono poprzez wirowanie w warunkach podanych w protokole.
Dodano mieszaninę octanu amonu i izopropanolu (odpowiednio 0,1 objętości i 1 objętośd) do ponownie
zebranej fazy wodnej w celu wytrącenia genomowego DNA. Precypitację prowadzono przez 20 minut.
Odwirowano w warunkach podanych w sprawozdaniu.
Próbkę DNA zagęszczono dodając 75% etanol. Etap ten pozwala również na pozbycie się soli oraz wolnych
nukleotydów z próbki. Po odwirowaniu i wysuszeniu osad zawieszono w buforze TAE.
Izolacja ludzkiego DNA genomowego z krwi obwodowej (Blood Mini Kit, A&A Biotechnology)
100 µl krwi było już rozporcjonowane przed rozpoczęciem dwiczeo. Dodano 200 µl uniwersalnego buforu
lizującego LT którego zadaniem była liza komórek – uwolnienie DNA genomowego. Do reakcji dodano również
20 µl Proteinazy K – proteinaza miała za zadanie oczyszczenie preparatu z białek oraz ochronę DNA przed
degradacją (niszczenie DNAz).
Próbkę wirowano i naniesiono na kolumnę, dodano następnie 500 µl roztworu płuczącego A1 (roztwór ten
ma za zadanie wypłukanie niespecyficznie związanych związków z kolumny); próbkę zwirowano, przeniesiono
kolumnę do nowej probówki 2 ml i ponowiono płukanie 400 µl roztworu A1. Zwirowano wg protokołu.
Przeniesiono kolumnę do probówki 1,5 ml.
Dokonano elucji genomowego DNA z kolumny przy pomocy buforu Tris – HCL o pH 8,5. Bufor ten zmienia
warunki panujące na złożu – DNA traci powinowactwo do złoża na kolumnie i wypływa z eluentem. Próbkę
następnie inkubowano w temperaturze pokojowej przez 5 minut i zwirowano. DNA zachowano do dalszej
analizy.
Obserwacje i wyniki:
Analiza Nano-drop:
izolacja FC: 238,2 ng/µl 260/280 nm: 1,54 260/230 nm: 29,78
izolacja KIT: 6,6 ng/µl 260/280 nm: 0,58 260/230 nm: 0,37
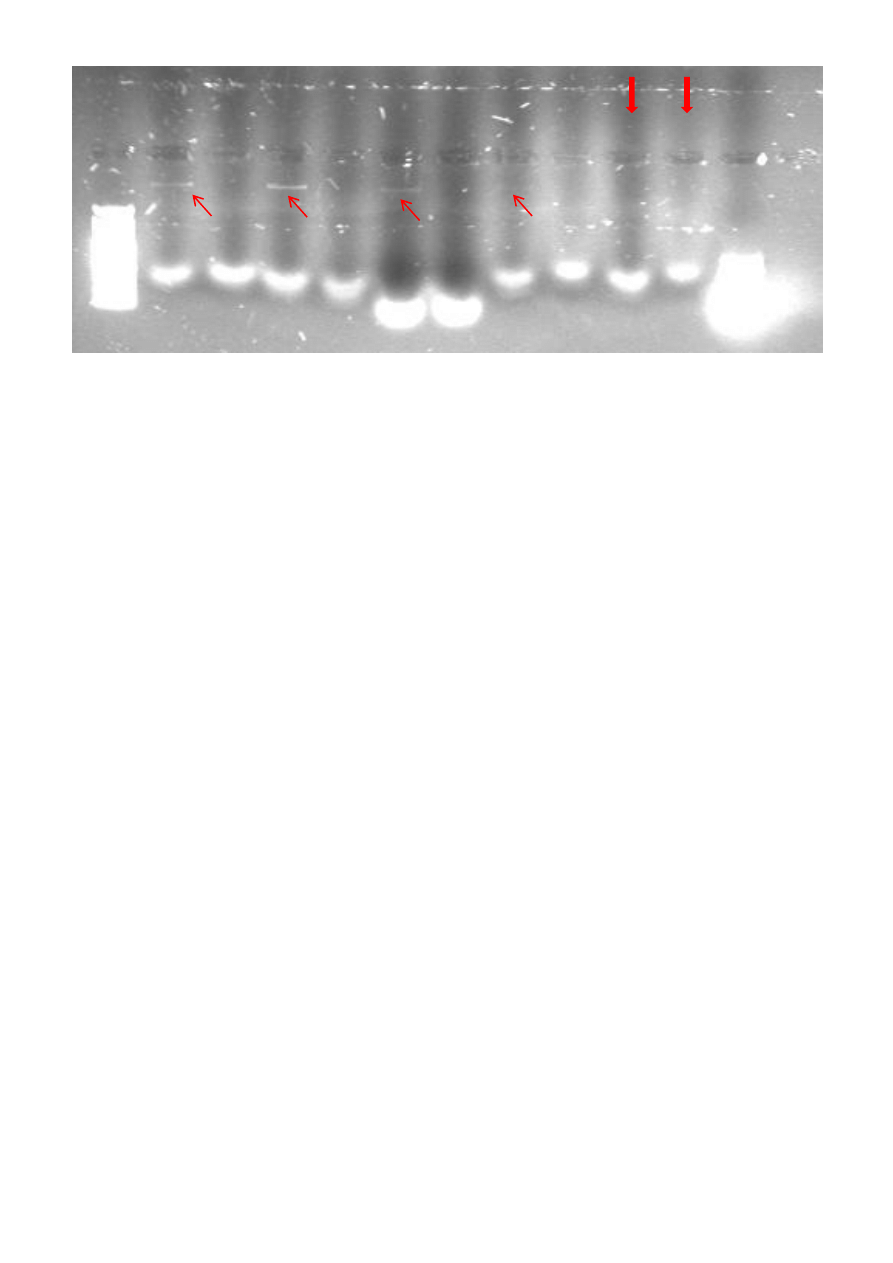
Analiza elektroforetyczna (rozdział prowadzono w 1% żelu agarozowym przy 100 V przez 30 minut, na żel
nałożono po 9 µl DNA w 4 µl buforu obciążającego, zajęto 10 i 11 studzienkę w kolejności):
M
FC
KIT
M
5 µl
5 µl
DNA
Wnioski:
W oparciu o wyniki metody Nano-drop izolacja DNA genomowego metodą fenol:choloroform okazała się byd bardzo
skuteczna. Osiągnięto preparat o znacznej czystości wg stosunku absorbancji DNA do zanieczyszczeo białkowych
(260/280 nm) oraz innych drobnocząsteczkowych zanieczyszczeo (260/230 nm). Niestety, elektroforeza agarozowa
nie ujawniła obecności DNA w tym przypadku. Możliwe iż ilośd DNA naniesiona na żel była niewystarczająca do
prawidłowej detekcji lub obecne zanieczyszczenia białkowe (np. DNAzy) zdegradowały DNA.
Izolacja DNA gotowym zestawem Blood Mini Kit nie powiodła się. Możliwe iż zestaw nie jest odpowiednio
wystandaryzowany do analizy małej objętości krwi (100 µl). Kolejną możliwością może byd zły stan buforów lub
kolumny w zestawie – DNA mogło wcale nie związad się ze złożem lub związad się częściowo i wyeluowad już na
etapie płukania.
Wyszukiwarka
Podobne podstrony:
więcej podobnych podstron