
Identyfikacja bakterii na
podstawie właściwości
biochemicznych
dr Magdalena Rzewuska

Każdy szczep bakteryjny cechuje się zdolnością do
przeprowadzania określonych reakcji biochemicznych
(co związane jest z wytwarzaniem odpowiednich
enzymów) – ma charakterystyczny profil aktywności
biochemicznej.
Poszczególne rodzaje i gatunki bakterii mają różne
profile aktywności biochemicznej.
Zatem badając aktywność biochemiczną
wyizolowanego szczepu bakteryjnego można go
zidentyfikować do rodzaju lub też do gatunku na
podstawie uzyskanego profilu reakcji.

W badaniach tych (tzw. próbach biochemicznych) oznacza się
różne właściwości bakterii, m.in.:
• Zdolność do wykorzystywania cytrynianu jako jedynego źródła
węgla (podłoże Simmonsa)
• Rozkład związków azotowych np.:
-
próba na wytwarzanie żelatynazy
-
próba na wytwarzanie kazeinazy
-
próba na wytwarzanie ureazy
(podłoże Christensena)
-
próba na wytwarzanie indolu z tryptofanu
-
próba na wytwarzanie siarkowodoru
(podczas rozkładu aminokwasów zawierających siarkę)
-
próba na zdolność redukcji azotanów do azotynów
-
próba na dezaminację tryptofanu (lub innych aminokwasów)
-
próby na dekarboksylację różnych aminokwasów

• Rozkład węglowodanów np.:
-
zdolność do fermentacji różnych cukrów
-
zdolność do wytwarzania acetoiny z glukozy
(próba Voges-Proskauera - VP)
-
zdolność do kwaśnego rozkładu glukozy
(próba z czerwienią metylową – MR)
• Zdolność do wytwarzania różnych enzymów np.:
-
próba na wytwarzanie katalazy
-
próba na wytwarzanie oksydazy cytochromowej
-
próba na wytwarzanie fosfatazy alkalicznej
-
próba na wytwarzanie różnych lipaz

Oznaczenia właściwości biochemicznych można
wykonywać metodami klasycznymi lub przy użyciu
mikrotestów (metody automatyczne).
Przykład oznaczeń metodami klasycznymi – tzw. szereg biochemiczny
do identyfikacji pałeczek Enterobacteriaceae

Przykładem mikrotestów mogą być systemy identyfikacji
bakterii firmy bioMeriéux np.:
API 20 E, ID 32 E, rapid ID 32 E - dla bakterii z rodziny
Enterobacteriaceae
API 20 NE -
dla gramujemnych pałeczek niefermentujących
API 20 A - dla bakterii beztlenowych
API 20 STREP i rapid ID 32 STREP -
dla paciorkowców
API STAPH i ID 32 STAPH -
dla gronkowców
API Coryne -
dla maczugowców
API ZYM -
do dodatkowego oznaczania aktywności
enzymatycznej różnych bakterii.
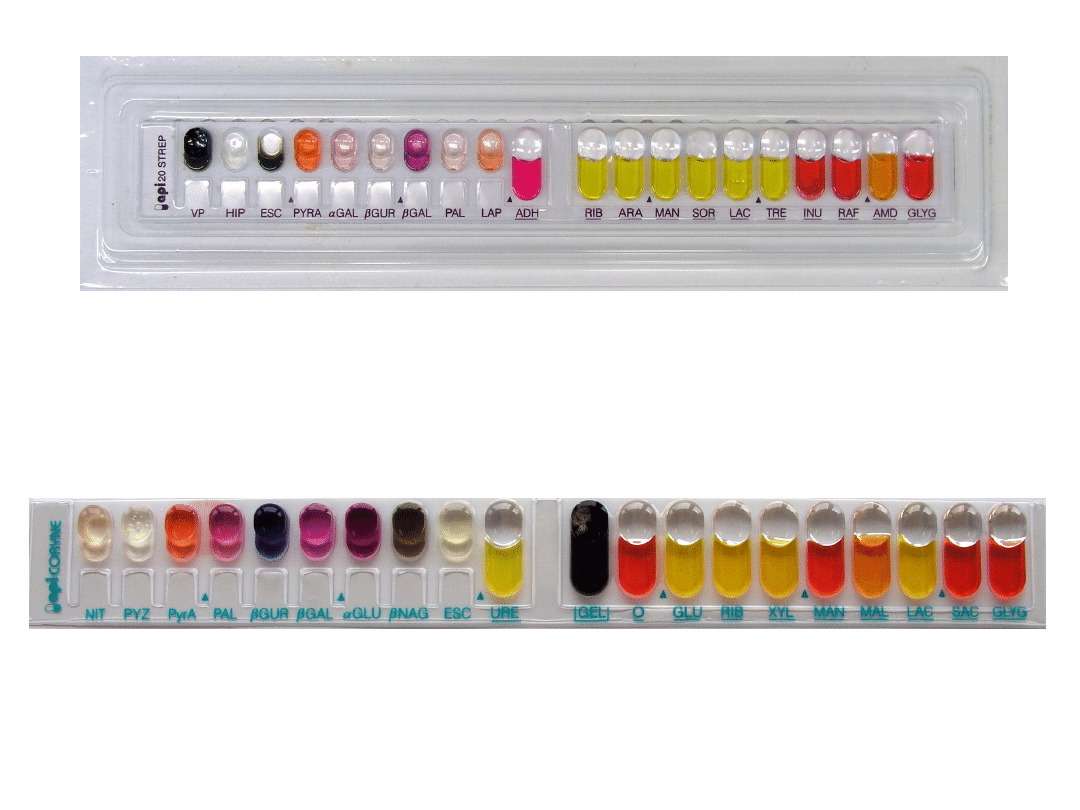
Przykład oznaczenia przy użyciu API STREP
Przykład oznaczenia przy użyciu API CORYNE
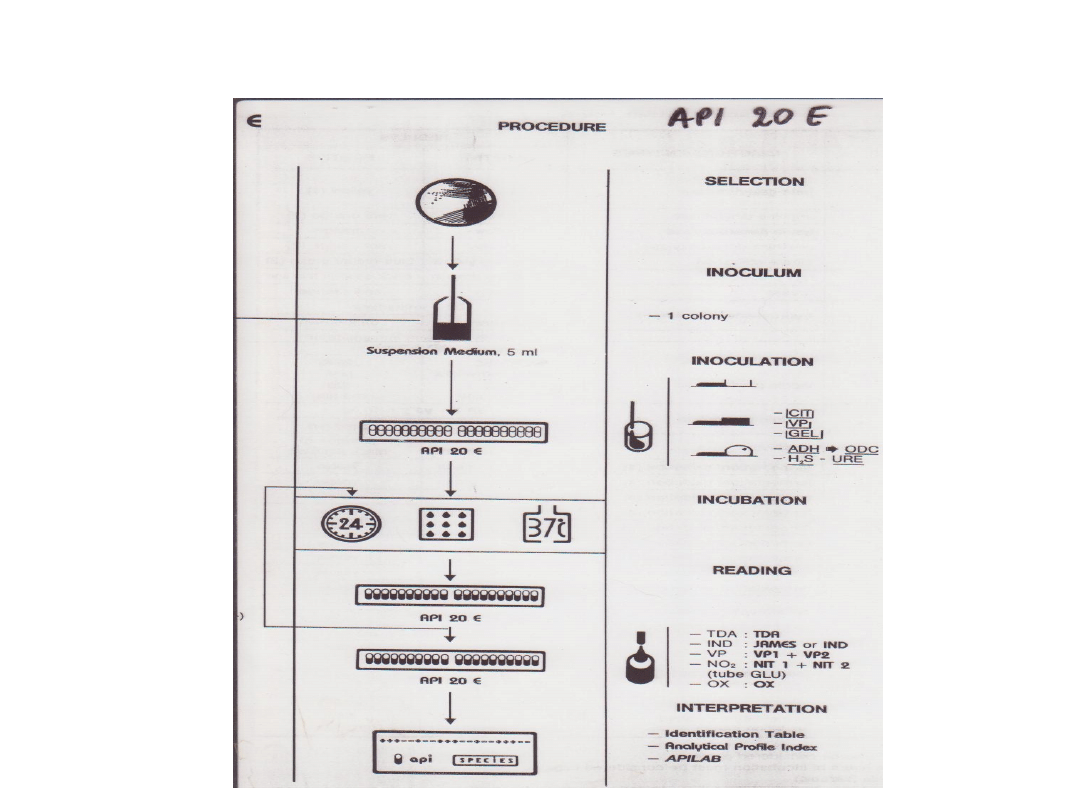
Sposób wykonania testu API 20E
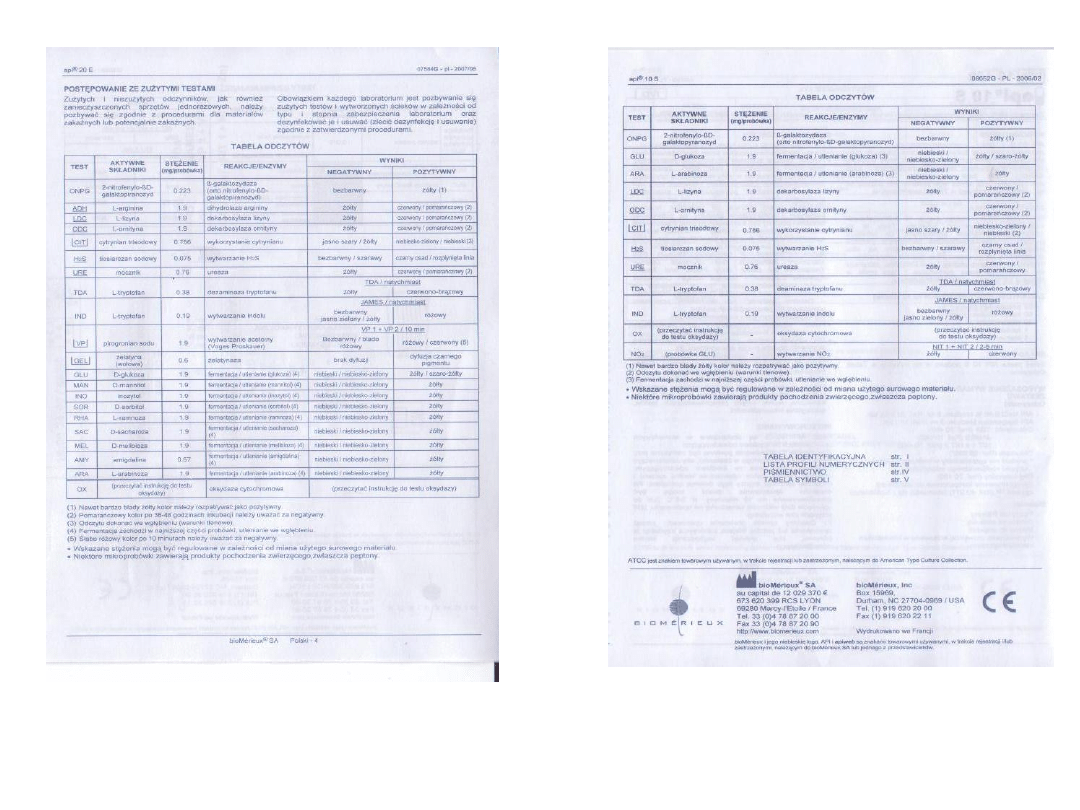
Tabela odczytu API 20E
Tabela odczytu API 10S


Wyszukiwarka
Podobne podstrony:
BIOCHEMIA KRWI, Weterynaria
mikrobiologia tabelka identyfikacja szczepów bakteryjnych
Kolokwium z bakteriiuu, weterynaria 3 rok WROC, semestr 5, Mikrobiologia Wroc
biochemia - enzymy, Weterynaria Lublin, Weterynaria 1, Biochemia
Cechy+bakterii, Weterynaria Lublin, Weterynaria 1, Mikrobiologia
Biochemia widzenia, Weterynaria, Biochemia
Zastosowanie bakterii, weterynaria
bakterie(1), weterynaria, choroby ptaków, choroby drobiu, ściągi kury, ściągi
normy biochemia krakvet, Weterynaria
pytania z koła z bakterii, Weterynaria Lublin, Weterynaria 1, Mikrobiologia
7 30 marca 2011 Identyfikacja bakterii biochemiczna i serologiczna
7 Wybrane metody biochemiczne i serologiczne identyfikacji bakterii chorobotwórczych
PAKIET WETA ZIMA 20142014, weterynaria uwm II rok, biochemia
Rodzina Brucellaceae, Medycyna Weterynaryjna, II Rok, MIKROBIOLOGIA, bakteriologia
więcej podobnych podstron