
1
4. Proces translacji
· Budowa mRNA i kod genetyczny
· Budowa tRNA i aktywacja aminokwasów
· Struktura rybosomów
· Etapy translacji
· Regulacja procesu translacji
· Inhibitory translacji
· Posttranslacyjna modyfikacja białek
5. Biosynteza białka w mitochondriach
1. Struktura i organizacja chromatyny
Kwasy nukleinowe są polimerami złożonymi z wielu nukleotydów monofosforanowych.
Struktura pierwszorzędowa to zespolenie nukleotydów wiązaniami fosfodiestrowymi. Reszta
fosforanowa połączona wiązaniem estrowym w miejscu 5’ jednego nukleotydu wytwarza wiązanie
estrowe z węglem 3’ drugiego nukleotydu. W efekcie powstaje długa nić zbudowana z przemiennie
występujących ryboz (deoksyryboz) i reszt fosforanowych; z nukleotydem 3’ końcowym (wolna grupa
OH) i 5’ końcowym z resztą fosforanową nie tworzącą wiązania. Sekwencję nukleotydową zapisuje się
od końca 5’ do 3’.
DNA jest stały i charakterystyczny dla danego gatunku. Zasady biorące udział w tworzeniu to
Adenina, Tymina, Guanina i Cytozyna.
DNA prokariota jest czasteczką kolistą tworzacą jeden chromosom bez końców 3’ i 5’. Odporny na
działanie egzonukleaz. Bakterie posiadają też pozachromosomalny, plazmidowy DNA.
DNA eukariota jest cząsteczką liniową połączoną z histonami. Oprócz niego znajduje się tez DNA
staelitarny - mitochondrialny lub plastydowy DNA.
Struktura drugorzędowa – struktura utrzymywana przez wiązania wodorowe. Temperatura
denaturacji wynosi 83-100*C i wzrasta wraz ze wzrostem zawartości par C:G. Łańcuchy są ułożne
przeciwrównolegle i tworzą podwójną helisę, w której wnętrzu znajdują się zasady purynowe i
pirymidynowe.
Forma B: Na jeden skok spirali przypada 10 par nukleotydowych, kolejne zwoje są oddalone o 3,4nm,
a odległość sąsiadujących zasad wynosi 0,34nm. W obrębie helisy występuje rowek mniejszy i rowek
większy i są wyścielone atomami – donorami lub akceptorami wodoru w procesie wytwarzania
wiązań wodorowych z białkami wiążącymi DNA.
Forma A: bardziej zwarta niż B – 11 nukleotydów w 1 zwoju, a odległośc zwojów = 2,6nm
Forma Z: 12 nukleotydów w jednym zwoju i odległość zwodjów = 3,7nm.
Punkt topnienia – taka temperatura przy której połowa wiązań wodorowych ulega rozpadowi.
Efekt hiperchromowy – zasady purynowe i pirymidynowe absorbują światło ultrafioletowe. Przy
denaturacji nastepuje wzrost absorpcji światła ultrafioletowego – zasady zostają odsłonięte.
Chromatyna i nukleosom.
DNA eukariota jest podzielona na chromosomy. U człowieka znajduje się 46 chromosomów. DNA jest
połączony z białkami, które tworzą kompleksy nukleoproteidowe zwane chromatyną jądrową.
Głównymi składnikami są Histony, które SA bogate w aminokwasy zasadowe – arginine i lizynę, co
nadaje im charakter polikationowy. Wiążą się z polianionowym szkieletem DNA tworząc obojętne
nukleoproteiny.
Histony bez względu na źródło gatunkowe dzielą się na 5 typów – H1 bogate w lizynę, H2A, H2B, H3,
H4 bogate w argininę. Są bardoz konserwatywne, różnią się niewielką ilością aminokwasów między
gatunkami.
Histon H1 jest większy od pozostałych i bardziej zasadowy, wykazuje różnce gatunkowe i nawet
tkankowe – posttranslacyjna modyfikacja.
Histony mają obojętny rdzeń i zasadowe domeny wiążące się z DNA. Domeny niepolarne i końce
karboksylowe umożliwiają łączenie się histonów ze sobą.
Oprócz histonów znajdują się też białka niehistonowe w znikomej ilości bardzo zmienne gatunkowo i
narządowo.

2
Nukleosom składa się z grona białkowego owiniętego DNA. Rdzeń Nukleosom składa się z 8 histonów
– po 2 każdego typu H2A, H2B, H3 i H4. Środek zbudowany jest z (H3)2-(H4)2, a na obu stronach
walca znadują się dimery H2A i H2B. Nić owijająca się 1 i 3/4 obrotu ma długość 146 nukleotydów.
Chromatosom – Nukleosom uzupełniony o dodatkowy fragment zwoju (+1/4 obrotu 166 zasad
łącznie) i histon H1
DNA łącznikowy uczestniczy w spinaniu nukleosomów
Solenoid – struktura utrzymywana przez histony H1 i prawdopodobnie białka niehistonowe. Na jeden
obrót solenoidu przypada 6 nukleosomów. Stuktura ta dominuje w jądrze interfazowym. Podczas
replikacji histony nie odłączają się od DNA, pozostają związane ze starą nicią a do nowej przyłączają
się nowe.
Gen – Odcinek DNA odpowiedzialny za przekazywanie określonej cechy gatunkowej, zwykle przez
kodowanie biosyntezy białka lub RNA
Genom – zespół genów. U eukariota genom jest poodzielany intergenami (sekwencjami
niekodującymi). Kodujące sekwencje – eksony są przedzielone intronami.
Białka wiążące się z DNA – są to białka, które uczestniczą w odtwarzaniu Dna przy replikacji, w
biosyntezie RNA na matrycy DNA, np. polimeraza DNA, polimeraza RNA itp. Część białek wiąże się
mało swoiście np. histony, Związanie białek z zasadami DNA wymaga kontaktu białka z jednym lub
dwoma rowkami. Rowek mniejszy pozwala odróżnić parę A-T od C-G, ale nie pozwala na rozpoznanie
która zasada znajduje się w której nici. Tylko rowek większy pozwala na rozpoznanie nici.
Motywy białek są poznane – są to helisa-skręt-helisa, palec cynkowy, zamki leucynowe i domena
zasadowa. Białka wiążą się z DNA wiązaniami niekowalencyjnymi – wodorowymi, hydrofobowymi,
elektrostatycznymi.
Helisa-skręt-helisa – zbudowana z dwóch helis oddzielonych skretem. Skręt ma konformację skrętu
β, który ustawia jedną z helis na powierzchni białka, tak że dopasowuje się do rowka większego DNA
– jest to helisa rozpoznająca. Cała domena składa się z około 20 aa.
Palec cynkowy –12 aminokwasów tworzy strukturę a-helisy i b-kartki, znajdują się tu 2 reszty
Cysteiny i 2 histydyny z umieszczonym centralnie atomem Zn, który oddziaływuje z Cys i His. Helisa
oddziaływuje z rowkiem, a jej ustawienie jest zależne od b-karki i atomu Zn, który utrzymuje
strukturę stabilną.
2. Replikacja DNA
Substratami są deoksyrybonukleotydy tri fosforanowe. Do syntezy DNA jest potrzebny jest primer
złożony z 2-10 nukleotydów syntezowany przez prymazę. Primer może być zbudowany z rybo
nukleotydów lub rybo i deoksyrybonukleotydów. Niektóre prymaty mają właściwości helikazy
(rozplata łańcuch DNA). Starterem może być oligonukleotyd z rozpadu DNA lub pojedynczy
deoksyrybonukleotyd (wirusy).
Replikacja zaczyna się w miejscu ORI, specjalne białka wiążą sekwencje dsDNA z sekwencjami DNA.
Powstaje kompleks 250pzDNA i multimetrów białka doprowadzając do miejscowego rozplecenia
dwupasmowej helisy bogatej w ary A-T. helikaza rozplata DNA zużywając ATP. Utrzymanie struktury
jednoniciowej umożliwiają białka A-RPA (a u prokariota SSB – single strand binding proteins. )
Primer jest syntezowany przez prymazę. Przyłącza się polimeraza DNA, która syntezuje DNA tylko w
kierunku 5’-3’. Ponieważ nici DNA są antyrównoległe polimerazy działają asymetrycznie. Na paśmie
wiodącym DNA jest syntezowany bez przerwy, na paśmie opóźnionym jest syntezowany jako krótkie
fragmenty Okazaki. Kompleks helikazy i prymaty nazwano primosomem. Kompleks ten otwiera
strukturę DNA i syntezuje fragmenty Okazaki.
Polimerazy DNA
Prokariota
Polimeraza I – usuwa startery i uzupełnia niewypełnione przerwy; zawiera aktywności –
polimerazowe, 3’-5’ egzonukleazowe i 5’-3’ egzonukleazowe.
Polimeraza II – nie uczestniczy w replikacji, bierze udział w naprawie uszkodzeń DNA
Polimeraza III – umiejscawia się w widełkach replikacyjnych i syntezuje nić DNA wykorzystują starter.
Posiada też właściwość 3’-5’nukleazową (usuwa 1 nukleotyd), co pozwala na zamianę źle
wprowadzonego nukleotydu.

3
Eukariota
Polimeraza α – wypełnia przerwy i syntezuje pasmo opóźnione, ma właściwości prymazy
Polimeraza ε – Wyłapuje błędy i naprawia DNA
Polimeraza β – Naprawia DNA
Polimeraza γ – syntezuje mitochondrialne DNA
Polimeraza δ – reprodukowanie i synteza pasma prowadzącego
Każda z nici u eukariota jest replikowana oddzielnie, przez odrębne kompleksy enzymów. Połączenie
nici po wycinaniu primerów i syntezie DNA w lukach jest katalizowane przez ligazę.
Telomery – Przy podziałach komórek pozostaje problem dotyczący początkowego startera. Po
wycięciu nie może zostać zsyntezowany brakujący fragment DNA. Rozwiązaniem jest telomeraza,
która zawiera matrycę do biosyntezy nici bogatej w guaninę. Zachodzi zjawisko odwrotnej
tranksypcji, gdyż telomeraza jest odwrotną transkryptazą. Przenosi informację z RNA zawartego w
enzymie na sekwencję nukleotydową telomeru na nowo powstałej nici DNA.
Topoizomerazy – przecinają 1 lub 2 nici pozwalając na relaksację i rozplecenie zwojów. Współpracują
z helikazą.
Naprawa DNA – organizmu eukariotyczne posiadają białka, które umożliwiają przejście z jednej fazy
cyklu komórkowego do następnej. Są to cykliny, które aktywują kinazy. Pojawienie się cyklin i ich
odpowiednie stężenie umożliwia przejście punktu restrykcyjnego i przejście do następnej fazy.
Naprawa niesparowanych zasad – swoiste białka szukają niesparowanych zasad, np. przez złe
wstawienie – zamiana C na T, lub przez wprowadzenie zbyt dużej ilości nukleotydów – utworzona
pętla. Białka używając zmetylowanej adeniny w sekwencji GATC oznaczają miejsca mutacji. Następnie
endonukleaza GATC przecina pasmo ze zmetylowaną adeniną, egzonukleaza trawi fragment
zawierający mutację. Później wstawione zostają prawidłowe nukleotydy.
Naprawa przez wycięcie zasady – depurynacja DNA zachodzi spontanicznie dzięki termolabilności
wiązania N-glikozydowego. Zasady Cytozyny i Adeniny mogą ulegać deaminacji z wytworzeniem
uracylu i hipoksantyny. Swoiste N-glikozylazy wyszukują błędne zasady i usuwają je. Usunięta zasada
oznacza miejsce mutacji i pozwala endonukleazom purynowym/pirymidynowym na wycięcie cukru
pozbawionego zasady. Polimeraza DNA b wstawia odpowiedni nukleotyd na puste miejsce.
Naprawa przez wycięcie nukleotydu – uzywany do naprawy uszkodzonych rejonów DNA o długości
np. 30pz spowodowanego przez promieniowanie UV. Następuje hydroliza dwóch wiązań
fosfodiestrowych przez specyficzną egzonukleazę. Usunięty fragment jest zastąpiony przez nowy
dzięki polimerazie d i e.
Naprawa pęknięć – białko Ku i DNA-zależna kinaza protein i helikaza powoduje zbliżenie, rozplecenie,
uszeregowanie i sparowanie nici, a następnie zachodzi łączenie przez ligazę.
3. transkrypcja
Biosynteza RNA to transkrypcja. Informacja zapisana w DNA jest przepisywana na sekwencję
nukleotydową RNA. Organizmy prokariotyczne mają polimerazę RNA składającą się z 5 podjednostek
– 2α – wiążących białka regulatorowe, β – tworzącej wiązania fosfodiestrowe, β’ wiążącej matrycę
DNA i sigma70 – rozpoznającej promotor i inicjującej transkrypcję.
Jednostka transkrypcji to fragment DNA od promotora do terminatora.
Produkt RNA który jest efektem transkrypcji to pierwotny transkrypt. U prokariota może to być kilka
genów, u eukariota jest to tylko jeden gen.
Koniec 5’ pierwotnego tran skryptu i RNA dojrzałego są identyczne i to od końca 5’ zachodzi
translacja. Miejsce startu transkrypcji odpowiada nukleotydowi końca 5’mRNA i oznacza się go
pozycją +1.
Pierwotny transkrypt generowany jest przez polimerazę RNA II i jest szybko opatrzony „czapeczką” 7-
metyloguanozylotrifosforanem, która jest przyczepiona do końca 5’. Chroni to mRNA przed
egzonukleazami 5’-3’. Od strony 3’ przyłączany jest ogon Poli-A przez polimerazę poliA.
Inicjacja tworzenia czasteczki RNA przy jej końcu 5’następuje z oddzieleniem czasteczki sigma.
Promotor PROKARIOTA – od miejscu transkrypcji d odległości około -40pz znajduje się promotor z 2
rogionami charakterystycznymi: -35 TGTTGACA gdzie przyłacza się polimeraza RNA – kompleks
zamknięty; -10 TATAAT który umozliwia oddzielenie od siebie nici DNA – kompleks otwarty.

4
Promotor EUKARIOTA – Sekwencje sygnalne umiesczone SA w proksymalnym regionie do promotora
– jedna określa czasteczce DNA miejsce gdzie ma się zaczac transkrypcja a druga jak często ma
zachodzić. Znajdują się tu jeszcze kasetki GC i CAAT, które wiążą odpowiednie białka.
W pozycji -32 TATAAAAG [podobna do sekwencji TATA] – wiąże TBP – tata binding protein, które
wiąże czynniki asocjujące z TBP (TAF TBP-associated factors).
Gdy nie ma kasety TATA polimeraza RNA II kieruje się do promotora dzięki sekwencji inicjacyjnej. –
transkrypcja zaczyna się od odpowiedniego miejsca – w okolicy -3 --- +5
Sekwencje wzmacniające lub tłumiące transkrypcję. Położone nawet w odległych miejscach w górę i
dół od genu. Sekwencje te wiążą białka, które oddziaływają z kompleksem transkrypcyjnym.
Kompleks transkrypcyjny eukariota – polimerazy RNA I, II i III nie rozpoznają sekwencji
promotorowych od innych regionów i potrzebne są białka, ko aktywatory, które pomagają regulować
interakcję z DNA i inicjację transkrypcji. TFIID,A,B,Poli II,F,E,H,J
TFIID wiąże się z TATA, bo zawiera TBP i jest jedynym czynnikiem wiążącym się z DNA. Związanie z
TATA powoduje zgięcie DNA o 100* i ułątwia integrację białek towarzyszących.
TFIIA – umożliwia kompleksowi odpowiedź na aktywatory.
Polimeraza RNA II posiada na końcu karboksylowym 7-aminokwasowy fragment powtarzający się 52
razy i jest to miejsce działania kinaz
Terminacja Eukariota – przez sekwencję spinki do włosów – pojawiają się palindromy i zasady łączą
się ze sobą na zasadzie komplementarności powodując oddysocjowanie kompleksu.
Istnieje drugi sposób germinacji – sekwencja Poli U – która zmniejsza powinowactwo kompleksu do
nici RNA.
Terminacja Eukariota – sygnały terminacyjne prawdopodobnie pojawiaja się w dół od sekwencji
kodujących. Prawdopodobnie sekwencją terminacyjną jest AAUAAA, która powoduje przeciecie
transkryptu.
Transkrypcja tRNA – przez polimeraze RNA III, syntezuje tRNA i rRNA o stałej sedymentacji 5S.
Transkrypt jest zazwyczaj o wiele dłuższy i zawiera kilka czasteczek, więc potrzebne są mechanizmy
powodujące dojrzewanie RNA. Cząsteczki ulegają przekształceniom nukleolitycznym przez swoistą
klasę rybonukleaz. Cząsteczki tRNA mają bardzo blisko części odpowiedzialnej za antykodon
wstawiony intron. Introny są przepisywane, a nastepnie usuwane, a cała czasteczka musi zostac
odpowiednio złozona, aby była aktywna. Następnie do tRNA dołączona zostaje końcówka CCA przy
końcu 3’ przez transferazę nukleotydową. Miejsce to przyłącza aminokwas.
TRANSKRYPCJA rRNA – przez polimerazę RNA I. Syntezuje pre rRNA, które jest cięte na 5,8S; 18S i
28S rRNA.
Eksony są poprzedzielane intronami, które ulegają transkrypcji, ale nie kodują białek. Są one usuwane
z transkryptu, a eksony są składane i łączone zanim cząsteczka RNA pojawi się w cytoplazmie.
Dojrzewanie mRNA zachodzi pod wpływem SPLICEOSOMÓW – kompleksów białkowych. Nacięcie w
miejscu połączenia końca 5’ eksonu i intronu. Wolny koniec 5’ tworzy pętle lub strukturę lassa i
przyłącza się wiązaniem fosfodiestrowym 5’-2’ z aktywnym A. Drugie przecięcie zachodzi w miejscu
połączenia intronu z eksonem ze strony 3’. W efekcie trans estryfikacji połączone zostają końce
eksonów, a intron zostaje usunięty i zhydrolizowany.
Alternatywne składanie – nie ma z góry narzuconej kolejności zdarzeń w łączeniu eksonów, istnieje
wiele miejsc alternatywnej terminacji i alternatywnej poliadenylacji.
· Regulacja procesu transkrypcji
· Inhibitory transkrypcji
4. Translacja
Kod genetyczny jest zdegenerowany – liczne kodony odpowiadają temu samemu aminokwasowi 61
kodonów sensownych i 20 aminokwasów. Na ogół 3 nukleotyd w kodonie jest mniej ważny niż
pozostałe dwa.
Kod genetyczny jest jednoznaczny – dany kodon koduje tylko jeden dany aminokwasów.
Kod genetyczny jest nienakładający się – odczytanie kodu nie obejmuje nakładania się ramki
odczytu. Dany nukleotyd należy tylko do jednego kodonu.
5
Kod genetyczny jest bezprzestankowy – odczyt zaczyna się w danym miejscu i nie następują
przerwy w odczycie, aż do momentu osiągnięcia kodonu STOP
Kod genetyczny jest uniwersalny – Taki sam wśród różnych gatunków.
Aktywacja aminokwasów i budowa tRNA
Aminokwas + enzym + ATP Enzym-AMP-aminokwas + PPi [Syntetaza aminoacylo-tRNA]
Enzym-AMP-aminokwas + tRNA AMP + Enzym + tRNA-aminokwas [Syntetaza aminoacylo-tRNA]
Aminokwas zostaje dołączony do pozycji 3’-hydroksylowej
adenozyny tRNA. Jest to wiązanie estrowe.
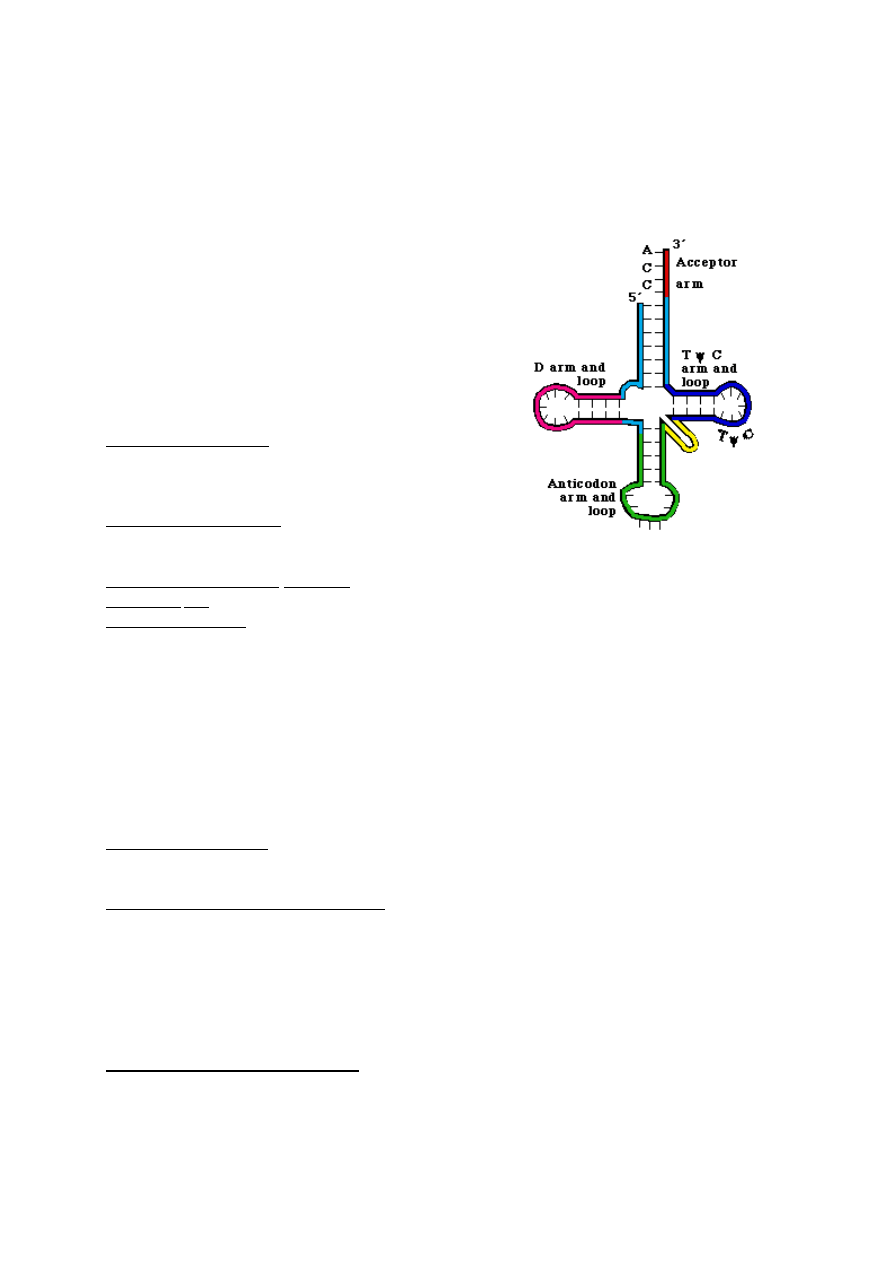
Cząsteczka tRNA składa się zkoło 75 nukleotydów. Cząsteczki
tRNA służą jako łączniki do translacji informacji zawartej w
sekwencji nukleotydów. W komórce istnieje co najmniej 20
tRNA odpowiadającym 20 aa. Każda swoista czasteczka tRNA
ma różną sekwencje nukleotydową to jednak jako klasa
czasteczek mają wspólne cechy. Komplementarność zasad
powoduje sfałdowanie tRNA w kształt liścia koniczyny.
Cząsteczka tRNA posiada 4 ramiona:
Ramię akceptorowe 7PZ – składa się z szypuły utworzonej z
zasad kończących się sekwencją CCA (5’-3’). Aminokwasy
swoją grupą karboksylową wiążą się z grupą 3’ OH
adenozyny.
Ramię antykodonowe 5PZ – Przy końcu szypuły o
sparowanych zasadach rozpoznaje tryplet nukleotydów, czyli
kodon matrycy mRNA, ma ono sekwencję nukleotydów komplementarnych do mRNA.
Ramię D (po lewej stronie) 3 lub 4PZ – nazwane dzięki obecności dihydrourydyny
Ramię TψC 5PZ –sekwencja T, pseudourydyna, C.
RAMIĘ DODATKOWE – zmienna cecha tRNA i stanowi podstawę do klasyfikacji tRNA.
Klasa 1 tRNA ma dodatkowe ramię długości 3-5ps; Klasa 2 ma ramię długości 13-21pz.
Budowa rRNA – rybosom jest nukleoproteidową strukturą cytoplazmatyczną, która syntezuje białka
na matrycach mRNA. Na rybosomach czasteczki mRNA i tRNA współdziałają w procesie translacji.
W czasie syntezy białka z cząsteczką mRNA są zasocjowane liczne rybosomy i taki kompleks nazywa
się polirybosomem.
Rybosom składa się z dwóch podjednostek i ma stałą sedymentacji 80S.
Duża podjednostka 60S składa się z 28S rRNA, 5,8S rRNA i 5S rRNA i około 50 łańcuchów
polipeptydowych.
Mała podjednostka 40S składa się z 18S rRNA i około 30 łańcuchów polipeptydowych.
Inicjacja
Dysocjacja rybosomalna – do podjednostki 40S przyłączają się eIF-3 i eIF-1A, co sprzyja rozpadowi
rybosomy 80S na podjednostki 40S i 60S. Podobne działanie ma przyłączenie czynnika eIF-3A do
podjednostki 60S
Tworzenie reinicjacyjnego kompleksu 43S – związanie GTP przez eIF-2, ten kompleks wiąże się z
met-tRNA. Met-tRNA wiąże się z mRNA z kodonem inicjacyjnym AUG. Są dwa tRNA dla metioniny, z
czego jeden określa metioninę dla AUG (kodon inicjacyjny) a drugi tRNA dla metioniny wewnątrz
łańcucha. Kompleks met-tRNA + eIF-2 + GTP łączy się z podjednostką 40S i czynnikami eIF-3 i 1A.
eIF2 – jest zbudowany z podjednostek α i β, gdzie podjednostka a jest fosforyzowana na Serynie
51przez trzy różne kinazy, które są aktywowane gdy nie ma wystarczającej energii w komórce i
wydatek energetyczny byłby szkodliwy dla komórki. Ufosforylowana podjednostka a deaktywuje
podjednostkę b. Zapobiega to tworzeniu kompleksu reinicjacyjnego 43S i białko nie powstaje.
Torzenie inicjacyjnego kompleksu 48S – Czapeczka metyloguanozylotrifosforanowa ułatwia
wiązanie mRNA do preinicjacyjnego kompleksu 43S. Białko eIF-4F składa się z białek eIF-4E,G,A i
wiąże się do czapeczki przez białko eIF-4E. Następnie eIF-4A i B wiązą i redukuję rugorzędową
strukturę kompleksu w końcu 5’ mRNA dzięki aktywności ATPazy i ATP-zależnej helikazy.
6
eIF-3 jest białkiem które wiąże się do komponentu 4G białka 4F i wiąże ten kompleks do
podjednostki 40S.
Sekwencja Kozaka – otaczająca AUG i precyzyjnie określająca jego położenie.
Utworzenie kompleksu inicjacyjnego 80S – związanie podjednostki rybosomalnej 60S z kompleksem
inicjacyjnym 40S obejmuje hydrolizę GTP związanego z eIF-2 przez eIF-5. Reakcja ta uwalnia
czynniki inicjacyjne związane z kompleksem 40S i asocjację podjednostek 40S i 60S z wytworzeniem
rybosomu 80S.
Elongacja
Wiązanie aminoacylo-tRNA do miejsca A – w kompletnym rybo somie 80S miejsce A jest wolne.
Aminoacytlo-tRNA musi rozpoznać kodon w miejscu A aby zostać przyłączonym. Wymaga to
czynnika elongacyjnego eEF-1α, który powoduje utworzenie kompleksu z GTP i umożliwia wejście
aminoacylo-tRNA i zajęcie miejsca A z uwolnieniem eEF- 1 α-GDP
Tworzenie wiązania peptydowego – grupa α-aminowa nowego aminoacylo-tRNA wiąże się z
estryfikowaną grupą karboksylową peptydylo-tRNA dzięki peptydylotransferazie. Łańcuch
polipeptydowy przeniesiony zostaje na tRNA w miejscu A. tRNA w miejscu P odłącza się.
Translokacja – Czynnik elongacyjny eEF-2 z GTP jest odpowiedzialny za translokację nowo
utworzonego peptydylo-tRNA z miejsca A na miejsce P i następuje rozpad GTP GDP + Pi.
Terminacja – gdy pojawia się kodon STOP UUA, UAG, UGA. W komórce zaczynają działać czynniki
uwalniające, które są zdolne do rozpoznania kodonu STOP. Czynnik uwalniający z połączeniu z GTp i
transferazą peptydylową powoduje hydrolizę wiązania między peptydem a tRNA.
Bilans – zapotrzebowanie energetyczne na 1 wiązanie peptydowe
Aktywacja aminokwasów: ATP AMP = 2 wiązania bogato energetyczne
Wejście aminoacylo-tRNA: GTP GDP = 1 wiązanie bogato energetyczne
Translokacja kompleksu: GTP GDP = 1 wiązanie bogato energetyczne
Posttranslacyjna modyfikacja białek
Deformylacja metioniny – u prokariota zawsze zaczyna się translacja od formylometioniny, która
nie wystepuje w dojrzałym białku. Deformaylaza powoduje deformylację metioniny. Czasem cała
metionina zostaje usunięta.
Często białko jest syntezowane jako czasteczka prekursorowa np. insulina syntezowana jako pro
hormon, który fałduje się umożliwiając wytworzenie mostków S-S, następnie swoista proteaza
usuwa segment łączący dwa łańcuchy tworząc funkcjonalną cząsteczkę insuliny.
Niektóre białka są syntezowane jako pro proteiny, które muszą ulec modyfikacji obejmujących
usunięcie reszt aminokwasowych z końca aminowego.
Hydroksylacja 3 cząsteczki pro kolagenu układają się podłużnie i swoiste enzymy przeprowadzają
modyfikację reszt aminowych - hydroksylacja i oksydacja. Wytwarzają się wiązania poprzeczne.
Białka mogą być acetylowane, fosforyzowane i glikozylowane.
Glikozylacja – wiązania O-glikozydowe z grupą OH hydroksylizyny (wiązanie Gal lub Gal-Glc) pod
działaniem galaktozylotransferazy lub glukozylotransferazy. Dawcą cukru jest UDP-galaktoza lub
UDP-glukoza.
Wiązanie O-glikozydowe z grupą OH seryny lub treoniny. Wiązanie N-acetylogalaktozaminy, a
następnie kolejnych cukrów. Dawcami są UDP-N-acetylogalaktozamina, GDP-fukoza, GDP-mannoza,
UDP-N-acetyloglukozoamina, a transferazami: fukozylotransgeraza, mannozylotransferaza, N-
acetyloglukozoaminylotransferaza. Kwas sjalowy (N-acetyloneuraminowy NANA) występuje w
połaczeniu z CMP jako CMP-NANA,a transferaza to sjalilotransferaza.
Dołączanie reszt cukrowych trwa do momentu gdy nie będzie żadnej glikozylotransferazy, która
będzie w stanie obsłużyć dany łańcuch cukrowy.
Wiązanie N-glikozydowe – łaczą się z NH2 asparaginy. W wytwarzaniu wiązania pośredniczy
fosforan dolicholu. Fosforan dolicholu na cytosolowej powierzchni RE służy jako akceptor dla N-
acetyloglukozoaminy. Dawcą cukru jest UDP-NAcGlc. Przyłącza się kolejna cząstka NAcGlc,
następnie przyłącza kilka cząstek Mannozy i glukozy.

7
Fosforylacja – grup OH łańcuchów bocznych seryny, treoniny i tyrozyny. Dawcą reszty fosforanowej
jest ATP. Reakcje katalizowane są przez kinazy białkowe.
Metylacja – Miejscem wiązania grupy metylowej jest azot w łańcuchu bocznym argininy, lizyny lub
histydyny. Dawcą grupy metlowej jest S-adenozylometionina. Najczęściej etylowane są histony.
Jodowanie – reszt tyrozyny w tyreoglobulinie
Karboksylacja – Niektóre reszty kwasu glutaminowego zawarte w białkach krzepnięcia krwi ulegają
gamma-karboksylacji, grupa karboksylowa jest zdolna wiązać jony Ca2+ potrzebne w krzepnięciu.
Mostki di siarczkowe –między resztami cysteiny, stabilizują białko.
Inhibitory translacji – antybiotyki, toksyny i analogi aminokwasowe
ANTYBIOTYKI
Streptomycyna – wiąże się z podjednostką 30S rybosomy prokariotycznego i hamuje inicjację oraz
powoduje błędne odczytywanie kodu aminokwasowego
Tetracykliny – wiążą się z podjednostką 30S, hamują wiązanie aminoacylo-tRNA
Chloramfenikol – wiąże się z podjednostką 50S rybosomy prokariotycznego i hamuje transferazę
peptydylową, uniemożliwia to tworzenie wiązań peptydowych
Erytromycyna – wiąże się z podjednostką 50S i hamuje translokację
Puromycyna – wykazuje podobieństwo do końcowego fragmentu aminoacylo-tRNA, wiąże się z
miejscem A rybosomu, przez swoją grupę aminową wiąże się z grupą karboksylową peptydu i
powstaje peptydylopuromycyna. Nie podlega ona translokacji ani elongacji
Cykloheksimid – wiąże się z podjednostką 60S komórek eukariotycznych i hamuje transferazę
peptydylową
Aktynomycyna D – wiąże się z DNA, który przestaje być matrycą do syntezy mRNA.
TOKSYNY
Toksyna błonicza – białko bakteryjne wytwarzane przez maczugowca błonicy. Hamuje elongację u
eukariota. Hamuje translokazę. Działa poprzez ADP-rybozylację translokazy
POCHODNE AMINOKWASÓW
Wskotek niedoskonałości kodu aminokwasowego mogą wbudowywać się analogi aminokwasów.
Kod nie odróżnia naturalnych aminokwasów od ich analogów.
Fluorofenyloalanina – zamiast fenyloalaniny
etionina – w miejsce metioniny
fluoroprolina i 3,4-dehydroprolina – zamiast proliny
Białka z analogami nie mogą pełnić funkcji, są rozpoznawane rpzez enzymy proteolityczne i
trawione.
· Regulacja procesu translacji
· Inhibitory translacji
· Posttranslacyjna modyfikacja białek
Wyszukiwarka
Podobne podstrony:
BW13 KWASY NUKLEINOWE id 95709 Nieznany
3 Kwasy nukleinowe id 33782 Nieznany
BW13 KWASY NUKLEINOWE id 95709 Nieznany
KWASY KARBOKSYLOWE id 256009 Nieznany
kwasy karbo id 256008 Nieznany
kwasy karboksylowe 1 id 256027 Nieznany
KWASY DIKARBOKSYLOWE 2 id 25598 Nieznany
KWASY KARBOKSYLOWE 3 id 256015 Nieznany
KWASY KARBOKSYLOWE id 256009 Nieznany
kwasy karbo id 256008 Nieznany
kwasy karboksylowe 1 id 256027 Nieznany
kwasy id 255953 Nieznany
kwasy nukleinowe
Abolicja podatkowa id 50334 Nieznany (2)
4 LIDER MENEDZER id 37733 Nieznany (2)
katechezy MB id 233498 Nieznany
metro sciaga id 296943 Nieznany
więcej podobnych podstron