
Przebieg i regulacja
procesu translacji
Autorzy:
Marta Karczewska
Ewelina Kłodowska
Edyta Korsak

Spis treści:
Pojęcie translacji
Przebieg translacji:
1. Aktywacja
2. Inicjacja
3. Elongacja
4. Terminacja
Regulacja translacji

Translacja
Translacja (z łac. Translatio - tłumaczenie) to proces syntezy
łańcucha polipeptydowego białek na matrycy mRNA. W jego
wyniku dochodzi do ostatecznego przetłumaczenia informacji
genetycznej zawartej pierwotnie w kodzie genetycznym DNA na
konkretną strukturę białka, zależną od uszeregowania
aminokwasów w łańcuchu polipeptydowym.
Translacja jest drugim (po transkrypcji) procesem w biosyntezie
białka. Powstawanie łańcucha polipeptydowego sterowane jest
przez sekwencję mRNA. Translacja odbywa się w cytoplazmie lub
na błonach szorstkiej siateczki wewnątrzplazmatycznej. Proces
ten jest katalizowany przez rybosom obejmujący podjednostkami
przesuwającą się nić mRNA. Rybosomy składają się z dwóch
podjednostek, większej i mniejszej, które są zbudowane z białek i
rRNA, a funkcję katalityczną pełnią enzymy (rybozymy) zawarte
w dużej podjednostce rybosomu. Translacja na jednej cząsteczce
mRNA może być prowadzona przez wiele rybosomów
równocześnie. Taki kompleks mRNA związanego z wieloma
rybosomami nazywa się polisomem lub polirybosomem.
Aktywacja
Zanim aminokwas może być włączony do formującego się łańcucha polipeptydowego
musi być najpierw aktywowany. W proces włączony jest zarówno tRNA jak i
specyficzny enzym, określany jako synteza aminoacylo-tRNA.
Aktywacja aminokwasu i utworzenie aminoacylo-tRNA odbywa się w drodze
dwustopniowej reakcji, która jest katalizowana przez enzym syntetazę aminoacylo-
tRNA. Wymieniony enzym, mający dwa centra aktywne, w sposób bezbłędny
rozpoznaje zarówno łańcuch boczny aminokwasu, jak i strukturę odpowiedniego tRNA.
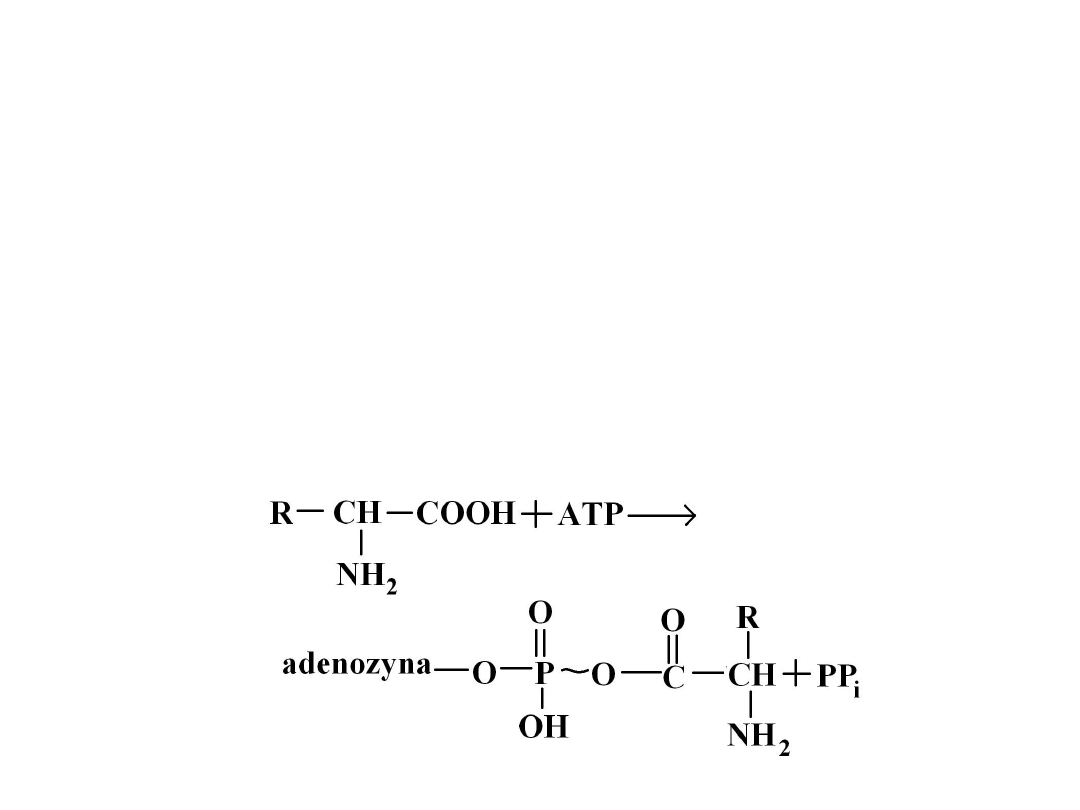
Pierwszy etap reakcji polega na aktywowaniu aminokwasów, czyli przeniesieniu ich na
wyższy poziom energetyczny. Proces ten dokonuje się z udziałem ATP jako źródła
energii oraz odpowiedniej syntetazy aminoacylo-tRNA. Enzym ten aktywuje
aminokwas przez przyłączenie AMP do grupy karboksylowej aminokwasu
pochodzącego z cząsteczki ATP i odłączenie difosforanu:
Aktywacja
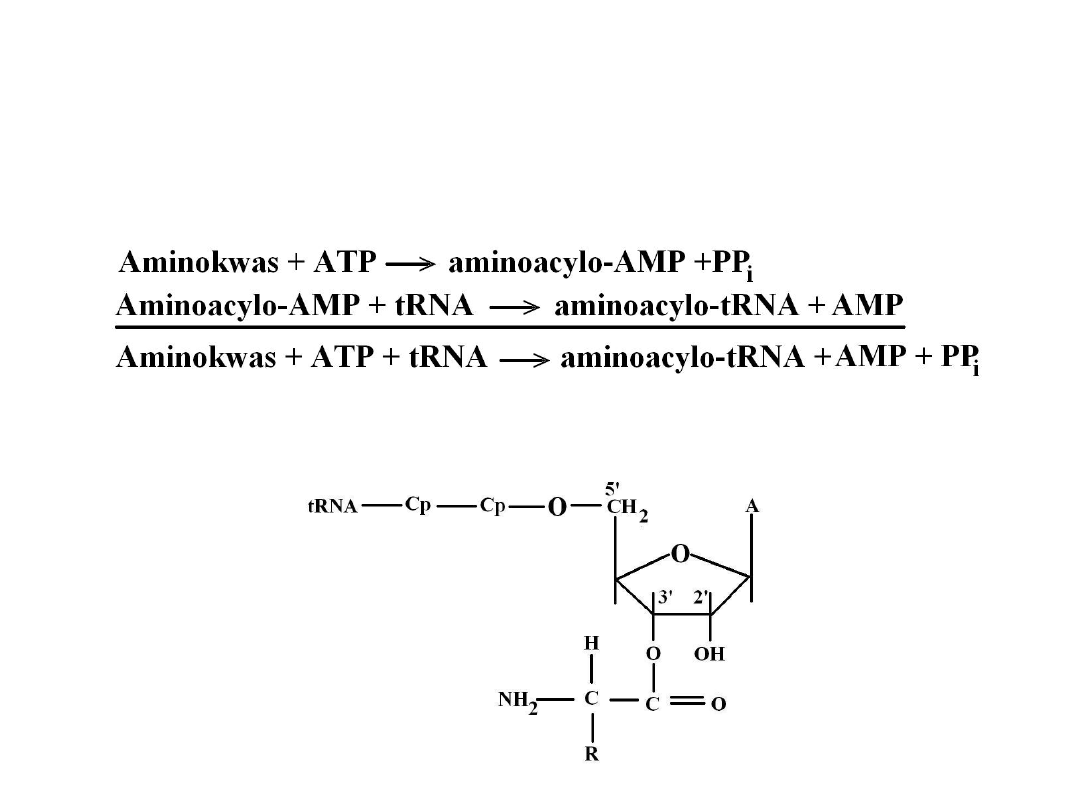
Aktywowany aminokwas pozostaje związany z cząsteczką syntetazy. W drugim
etapie reakcji ten sam enzym, tj. syntetaza, katalizuje przeniesienie grupy
aminoacylowej (R-CH2NH2-CO-) z aminoacylo-AMP na odpowiedni rodzaj tRNA.
Do utowrzenia aminoacylo-tRNA zostaje więc wykorzystana energia
zmagazynowana w dwóch wyspkoenergetycznych wiązaniach fosforanowych.
Aktywacja
Grupa aminoacylowa przyłącza się do końcowego nukleotydu adeninowego
tRNA, z najdującego się w tryplecie CCA, tj. na końcu 3’ ramienia
aminokwasowego (akceptorowego) tRNA, wiążąc się estrowo z 3’ grupa
hydroksylową rybozy. Zaaktywowane i związane z tRNA aminokwasy są
przenoszone do formującego się polirybosomu, gdzie ustawiaja się w takiej
kolejności, jak to wyznaczają sekwencje trójek nukleotydowych (kodonów)
w mrNA oraz odpowiadające im antykodony w tRNA.

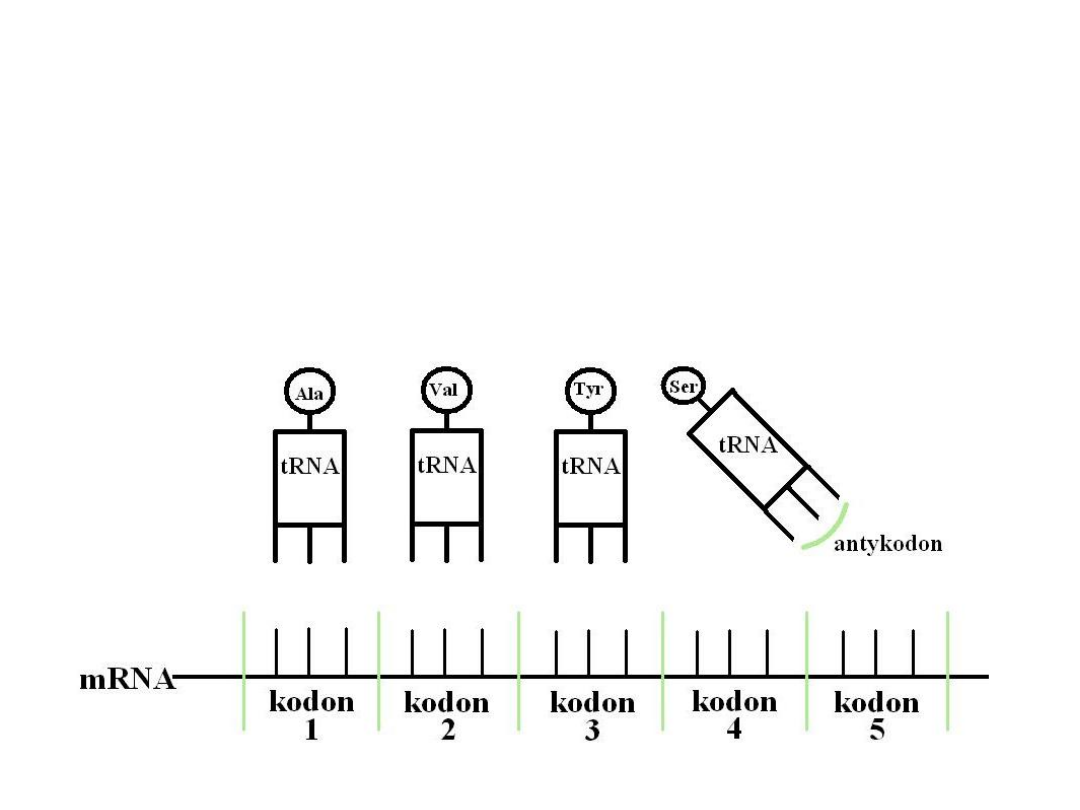
Inicjacja (rozpoczęcie)
Zapoczątkowanie formowania się łańcucha polipeptydowego polega
na wytworzeniu tzw. Kompleksu inicjacyjnego, który składa się z
rybosomu, mRNA i inicjatorowego tRNA, czyli N-formylometionylo-
TRNA (u organizmów prokariotycznych). U organizmów
eukariotycznych funkcję inicjatorowego tRNA pełni metionylo-tRNA.
W powstawaniu tego kompleksu uczestniczą także białkowe czynniki
inicjacyjne – IF, u eukariotów – eIF oraz GTP (i ATP).
Podczas inicjacji rybosom i inicjatorowy tRNA muszą właściwie
rozpoznać miejsce na nici mRNA, w którym powinna się rozpocząć
biosynteza białka, co warunkuje dalszy prawidłowy odczyt. Na tym
etapie działają też liczne mechanizmy regulacyjne biosyntezy białka.
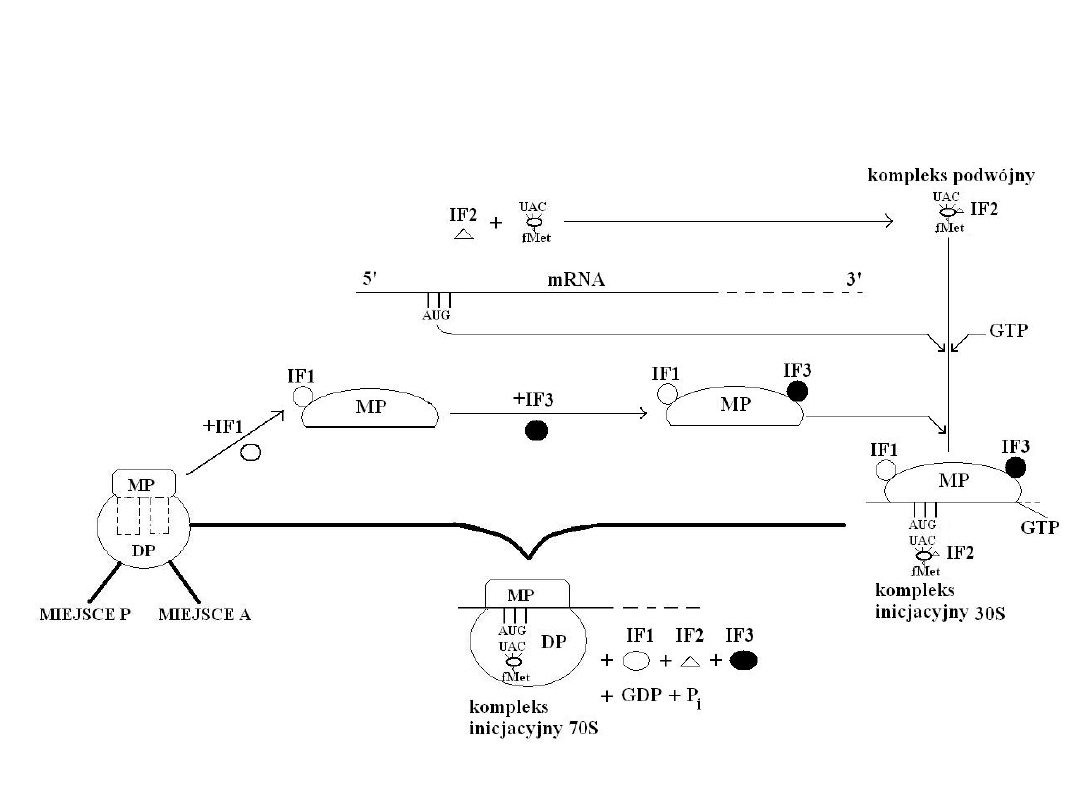
W bakteryjnej inicjacji pierwszą reakcją jest dysocjacja rybosomu na
dwie oddzielne podjednostki – małą (MP) i dużą (DP). Dysocjacja jest
symulowana przez czynnik inicjacji – 1 (IF1), który łączy się
bezpośrednio z małą podjednostką rybosomu. Odłączona mała
podjednostka jest stabilizowana przez przyłączenie IF3, który działa
jako „czynnik antyasocjacyjny” zapobiegający ponownemu
połączeniu się małej podjednostki z dużą.
Inicjacja
Inicjacja
Mała
podjednostka
(30S)
jest
teraz
przygotowana do następnego etapu, w którym
mRNA i inicjatorowy tRNA w połączniu z
aminokwasem (fMet) są przyłączane do
kompleksu. Inicjatorowy tRNA oddziałuje z IF2,
tworzac
tzw.
Kompleks
podwójny.
Dla
porównania u eukariotów występuje kompleks
potrójny.
IF2
stymuluje
wiązanie
się
inicjatorowego
tRNA
z
miejscem
P
(peptydowym). Kompleks IF2-inicjacyjny tRNA
oraz mRNA zostają następnie przyłączone do
małej podjednostki w taki sposób, że kodon
inicjujący
AUG
cząsteczki
mRNA
jest
umiejscowiony w środku miejsca P na małej
podjednostce.
Pierwszym
aminokwasem
łańcucha
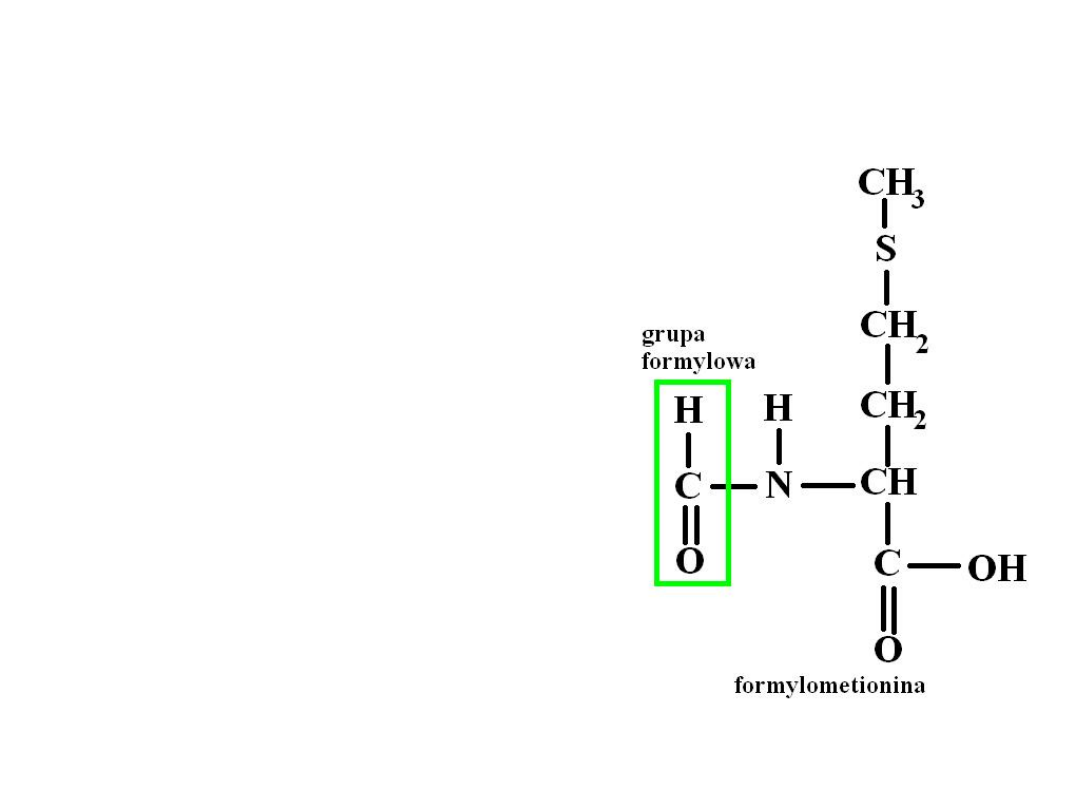
polipeptydowego jest metionina (Met). Jednak
u prokariotów niesiona przez inicjatorowy tRNA
jest zmieniana do formylometioniny, przez
dodanie grupy formylowej, która blokuje grupę
aminową aminokwasu.

Inicjacja
Końcową reakcją w inicjacji
bakteryjnej jest
przyłączenie dużej
podjednostki (50S). Po
przyłączeniu dużej
podjednostki wszystkie
czynniki inicjacyjne
odłączaja się od rybosomu.
Uwolnieniu IF2 towarzyszy
hydroliza GTP. Utworzony
kompleks inicjacyjny
zawiera rybosom, mRNA i
fMet-tRNA, z inicjatorowym
tRNA przyłączonym w
miejscu P rybosomu,
naprzeciwko kodonu
inicjującego AUG.

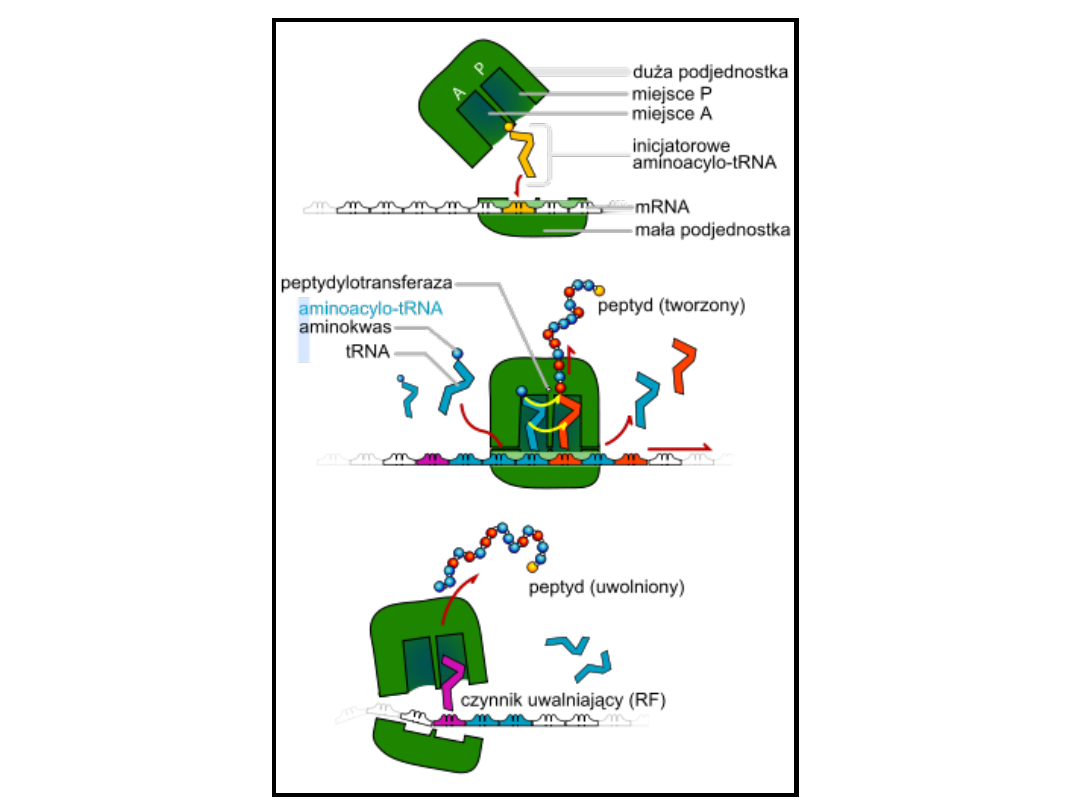
Elongacja
Elogancja jest etapem translacji, na którym kolejne aminokwasy przyłączają się do
rosnącego łańcucha polipeptydowego. Przebiega ona w zasadzie tak samo u bakterii i
eukariontów.
W początkowej fazie elogancji właściwy aminoacylo-tRNA rozpoznaje kodon mRNA (w
miejscu A) i łączy się z nim na zasadzie komplementarności par zasad przez swój
antykodon. Łączenie to wymaga udziału kilku białek zwanych czynnikami elongacyjnymi.
Wymaga ono także energii pochodzącej z guanozyno-trifosforanu (GTP), przenośnika
energii podobnego do ATP. Grupa aminowa aminokwasu nowo przyłączonego (w miejscu
A) leży obok grupy karboksylowej wcześniej przyłączonego aminokwasu (w miejscu P).
W kolejnej fazie elongacji tworzy się wiązanie peptydowe między grupą aminową nowo
przyłączonego aminokwasu (zajmującego miejsce P) a karboksylową aminokwasu
przyłączonego wcześniej. W ten sposób wcześniej przyłączony aminokwas odłącza się od
swojego tRNA i przyłącza się do aminoacylo-tRNA nowo przyłączanego (zajmującego
miejsce A). Jest to reakcja samoistna, nie wymagająca dodatkowej energii, ponieważ do
utworzenia aminoacylo-tRNA zostałą użyta energia z ATP. Utworzenie wiązania
peptydowego wymaga jednak działania enzymy transferazy peptydowej.
W ostatniej fazie elogancji, określanej jako translokacja (przemieszczenie), rybosom
przesuwa się o jeden kodon w kierunku 3’ cząsteczki mRNA. Dzięki temu kodon mRNA
określający kolejny aminokwas ustawia się w wolnym miejscu (A). Translokacja wymaga
energii, któej dostarcza GTP.
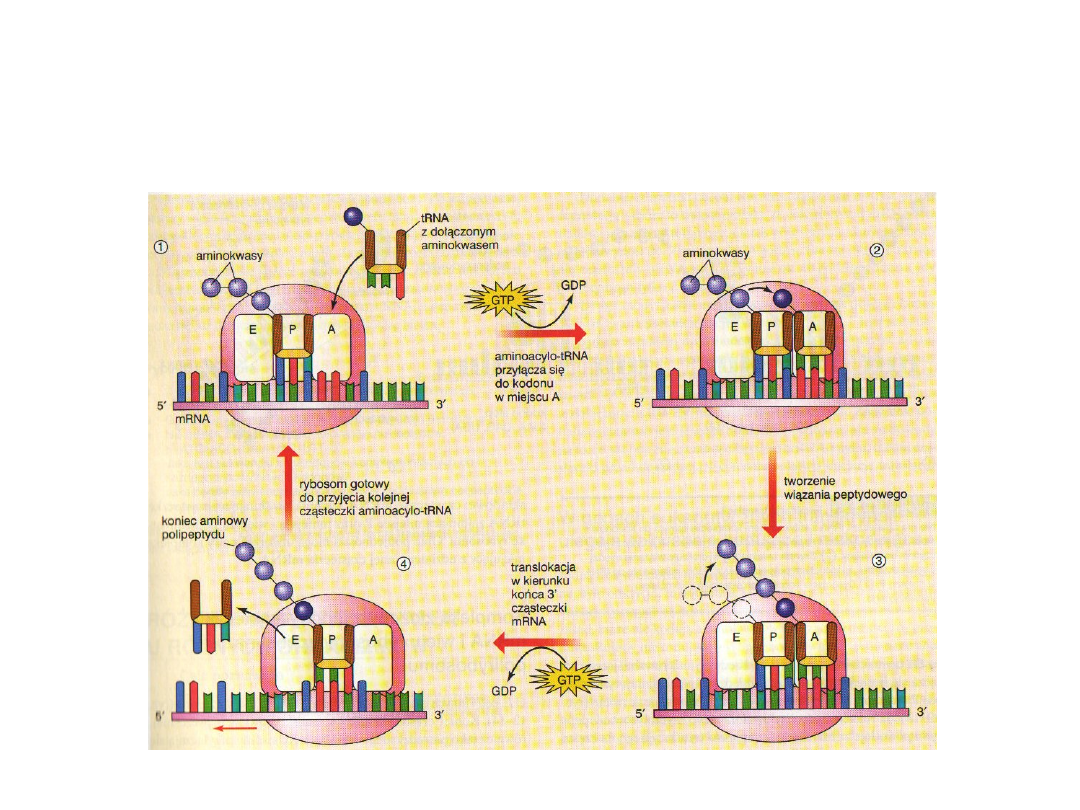
Elongacja
Jeden cykl elongacji podczas translacji
Elongacja
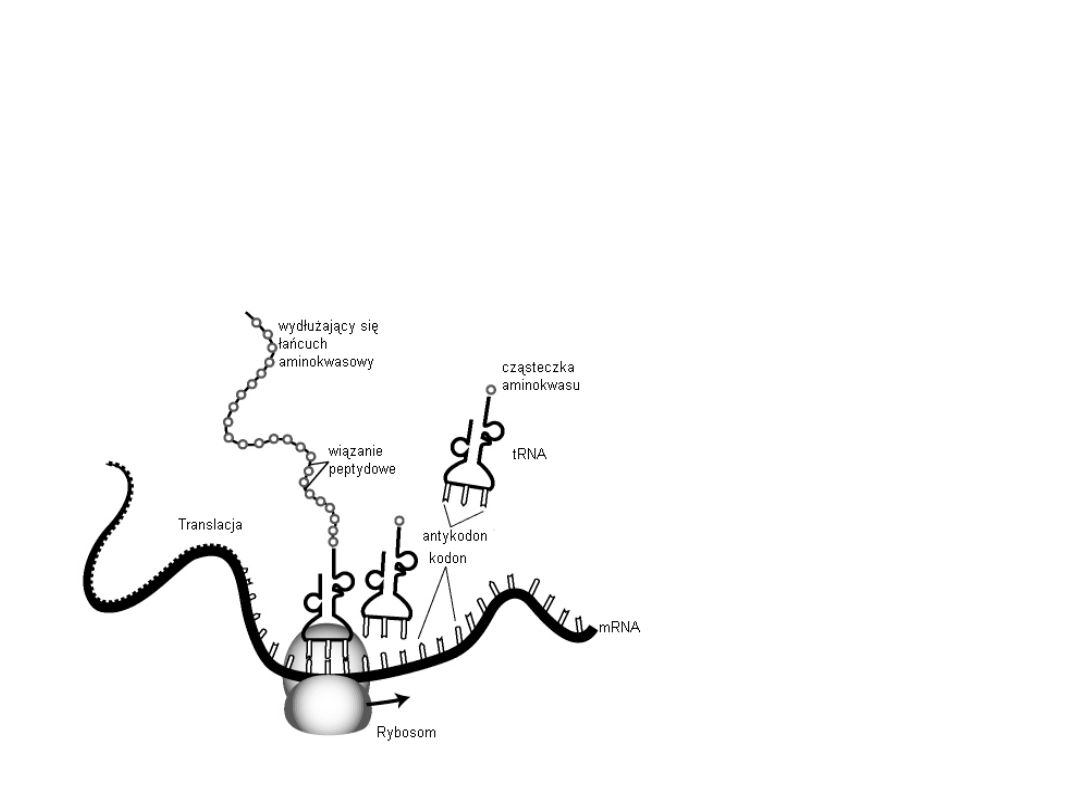
Tak więc, wydłużanie się łańcucha polipeptydowego polega na przyłączaniu kolejnych
aminokwasów na jego C-końcu. Elongację łańcucha polipeptydowego umożliwiają tzw.
czynniki elongacyjne odpowiedzialne za dostarczanie aminoacylo-tRNA, oraz za
translokację tj. przeniesienie z m-ca A na m-ce P odbywającą się kosztem energii z GTP.
Schemat ogólny
przebiegu etapu
elongacji
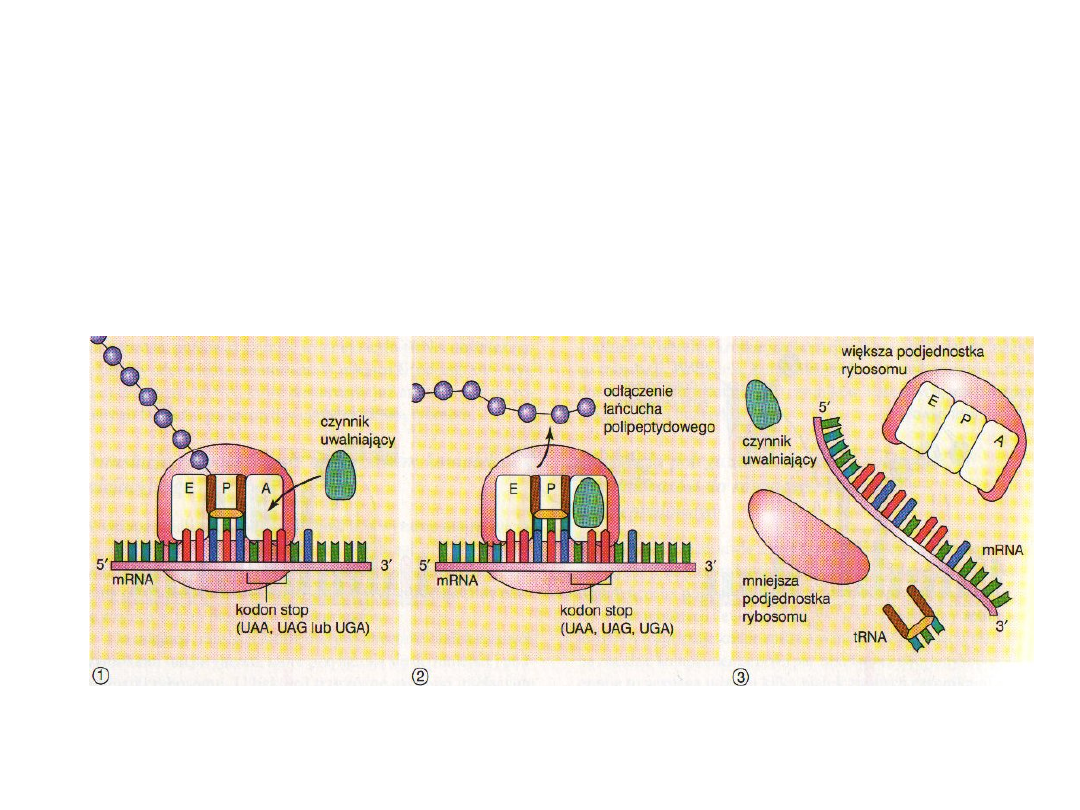
Terminacja
Ostatnim etapem translacji jest terminacja, kiedy syntezę łańcucha polipeptydowego kończą
czynniki uwalniające, czyli białka rozpoznające kodon stop na końcu sekwencji kodującej ( z
kodonem tym nie łączy się żadna cząsteczka tRNA, więc jest dostępny dla czynników
uwalniających). Kiedy czynniki uwalniające przyłączą się w miejscu A, wiązanie między tRNA,
(zajmującym miejsce P), a ostatnim aminokwasem w łańcuchu zostaje zerwane. Ta reakcja
hydrolizy uwalnia nowo zsyntetyzowany peptyd i powoduje rozdzielenie kompleksu
translacyjnego na części składowe: cząsteczkę mRNA, czynnik uwalniający, cząsteczkę tRNA
zajmującą miejsce P oraz mniejszą i większą podjednostkę rybosomu. Obie podjednostki
mogą utworzyć nowy kompleks inicjujący z udziałem innej cząsteczki mRNA.
Terminacja translacji
U Prokaryota za proces terminacji translacji odpowiedzialne są dwa czynniki — RF1 i
RF2. Czynnik RF1 rozpoznaje kodony UAA i UAG, a RF2 rozpoznaje kodony UAA i UGA

Regulacja translacji
W komórkach eukariotycznych transkrypcja i translacja są procesami bardziej złożonymi niż
w komórkach prokariotycznych - podstawowe mechanizmy transkrypcji i translacji są bardzo
podobne we wszystkich organizmach, różnią się jednak pewnymi istotnymi cechami, w
szczególności dotyczącymi właściwościami cząsteczek mRNA.
Bakteryjne mRNA wykorzystywane są do translacji natychmiast po syntezie , bez
poprzedzającej obróbki natomiast cząsteczki eukariotycznego mRNA podlegają
specyficznym i daleko idącym MODYFIKACJOM I PREZKSZTAŁCENIOM POTRANSKRYPCYJNYM.
W komórkach eukariotycznych procesy wyżej wymienione są ściśle oddzielone.
Chromosomy eukariotyczne zawarte są w jądrze komórkowym, natomiast synteza białka
odbywa się w cytoplazmie. Cząsteczki mRNA muszą być zatem przetransportowane przez
otoczkę jądrową do cytoplazmy, by mogły ulec translacji. W dodatku pierwotny transkrypt,
zanim będzie sie nadawał do transportu i translacji jeszcze w jadrze musi być poddanym
pewnym modyfikacjom

Regulacja translacji
Modyfikacja cząsteczki eukariotycznej RNA rozpoczyna sie gdy transkrypt liczy około
20 -30 nukleotydów. W tym momencie specyficzny enzymy dołączają do końca
5łancucha mRNA "czapeczkę„ czapeczka ma postać nietypowego nukleotydu 7-
metyloguanylowego, tj.guanozynomonofosforanu, w którym do jednego z atomów
azotu zasady(guaniny) dołączana jest grupa metylowa.
Niezwykłość czapeczki polega również na tym, ze stanowi zmodyfikowany nukleotyd
przyłączony jest w "w tył" tj. do końca 5 cząsteczki mMRNA, zamiast zwykłego
wiązania fosforanowego 5-3 utworzone zostaje wiązanie 5-5 miedzy reszta5
fosforanowa czapeczki a reszta 5 fosforanowa na końcu cząsteczki mRNA. RYBOSOMY
NIE MOGĄ ŁACZYC SIE Z MATRYCA KTORA NIE MA CZAPECZKI. Dołączenie czapeczki
może również chronić RNA przed degradacja przez niektóre enzymy i stanowić ,
przynajmniej częściowo, przyczynę znacznie większej stabilności mRNA
eukariotycznych w porównaniu z mRNA prokariotycznych.
Okres półtrwania eukariotycznych mRNA wynosi od 30minut do 24godzin natomiast w
komórkach ssaków średnio 10godz , podczas gdy w komórkach bakterii tylko 2minuty
2modyfikacja: eukariotycznego mRNA dotyczy końca 3czasteczki w pobliżu końca
cząsteczki 3 występuje sekwencja zasad, która stanowi sygnał do dodawania
fragmentu złożonego nukleotydów adeninowych zwanego poli -A. W ciągu około
minuty po ukończeniu syntezy transkryptu enzymy znajdujące sie w jadrze rozpoznają
sygnał poli –a i przecina w tym miejscu cząsteczkę mRNA. Następnie do końca 3
dołączanych zostaje 100 do 250 nukleotydów adeninowych.

Regulacja translacji
Regulacja translacji:
*miejsce regulacji potranskrypcyjnej u prokariontów - potranskrypcyjne mechanizmy kontrolne mogą działać na
rożnych etapach ekspresji genu.
Kontrola translacji zapewnia regulację szybkości, z jaka cząsteczki poszczególnych mRNA ulegają translacji.
Ponieważ okres trwania cząsteczki mRNA w komórce bakterii jest bardzo krotki cząsteczka, która ulega szybkiej
translacji , może dostarczyć więcej cząsteczkę białka niż. ta , której translacja przebiega powoli. U E. coli
szybkość translacji niektórych czatseczke mRNA jest 1000razy większa niż innych. Wynika to przede wszystkim
z różnicy w szybkości z jaka rybosomy mogą przyłączyć sie do RNA i inicjować translacje.
Kontrola potranslacyjna wykorzystywana jest głownie jako "przełącznik „ który aktywuje bądź dezaktywuje jeden
lub więcej istniejących enzymów. Systemy te umożliwiają komórce natychmiastowa odpowiedz na zmiany
wewnątrzkomórkowego stężenia podstawowych związków , takich jak aminokwasy, poprzez szybkie
dostosowanie aktywności odpowiednich enzymów. Powszechnie występującym mechanizmem dzięki, któremu
szybkość powstawania produktów w szlakach metabolicznych zostaje dostosowana do wymogów komórki, jest
hamowanie(inhibicja) przez "sprzężenie zwrotne„ -produkt końcowy przyłącza sie do końca miejsca
allosterycznego pierwszego enzymu szlaku powdujac jego czasowa dezaktywacje. Wyłączenie pierwszego
enzymu pozbawia substratów wszystkie pozostałe enzymy szlaku. Hamowanie przez sprzężenie zwrotne służy
precyzyjne regulacji aktywności istniejących enzymów w szlakach metabolicznych.
Podobnie obserwujemy komórek eukariotycznych. Oprócz tego wiele białek eukariotycznych ulega po syntezie
intensywnej modyfikacji. w przypadku "obróbki proteolitycznej" syntetyzowane w postaci nieaktywnych
prekursorów białka przekształcane są w formy aktywne przez usuniecie fragmentu łańcucha polipeptydowego.
poziom niektórych białek może być częściowo regulowanych za pomocą "wybiorczej degradacji", która
zapewnia utrzymywanie stałego poziomu białka w komórce, odwracalne zmiany aktywności enzymu maga być
powodowane przez „modyfikacje chemiczne „ polegające na dodawaniu lub osuwaniu grup funkcyjnych.
Powszechnym sposobem modyfikowania aktywności enzymów i innych białek jest dodawanie lub usuwanie
reszt fosforanowych, tego rodzaju modyfikacje pozwalają komórkom szybko reagować na zmiany środowiska i
warunków pokarmowych.
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
Wyszukiwarka
Podobne podstrony:
Przebieg i regulacja procesu translacji
32 Proces i regulacja procesow translacji
BIOCHEMIA Przebieg procesu translacji
3 Charakterystyka i przebieg procesu translacji
BIOCHEMIA Przebieg procesu translacji
1 hormonalna regulacja procesów życiowych
Hormonalna regulacja procesów rozwojowych, BIOMEDYKA
Regulacja Procesów Fizjologicznych
Organizacja i przebieg przykładowego procesu produkcji, różne
Regulacja procesĂłw syntezy i wydzielania hormonĂłw tarczycy, Patomorfologia
kontrola i regulacja procesów życiowych GRUPA2 wer 2007, sprawdziany, gim1
kontrola i regulacja procesów życiowych, sprawdziany, gim1
Przebieg projektowania procesu technologicznego
kontrola i regulacja procesów życiowych GRUPA2, sprawdziany, gim1
37 REGULACJA PROCESOW ZYCIOWYCH RO
2 Biosynteza FA przebieg, regulacja
kontrola i regulacja procesów życiowych wer. 2007, sprawdziany, gim1
więcej podobnych podstron