
Odwrotne transkryptazy
Odwrotna transkryptaza (rewertaza) jest to polimeraza
DNA zależna od RNA.
Posiada 3 rodzaje aktywności:
1. Aktywność polimerazy DNA zależnej od RNA.
2. Aktywność rybonukleazową .
3. Aktywność polimerazy DNA zależnej od DNA.
Za jej odkrycie w 1975 roku przyznano nagrodę Nobla.
Rewertaza występuje u retrowirusów (np. HIV) i niektórych
wirusów DNA (Hepadnawirusy).
Odwrotna transkryptaza kodowana jest przez
retrotranspozony (specyficzna cząsteczka RNA)
występujące w genomie organizmów eukariotycznych.
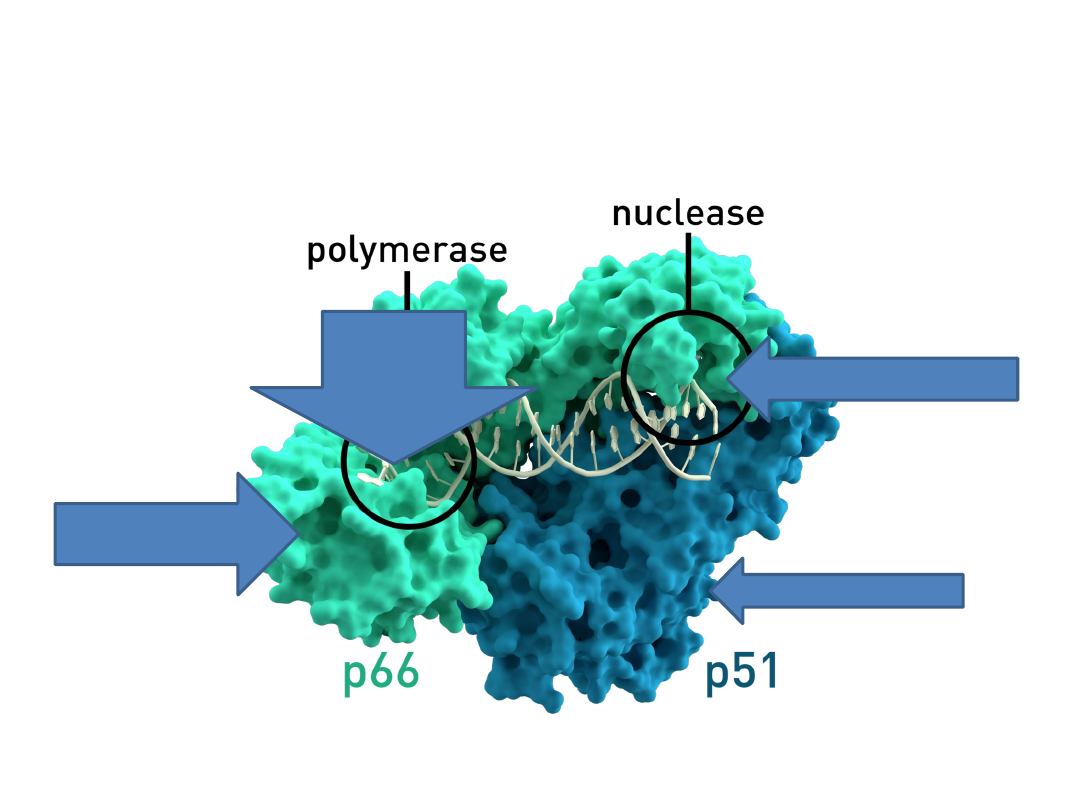
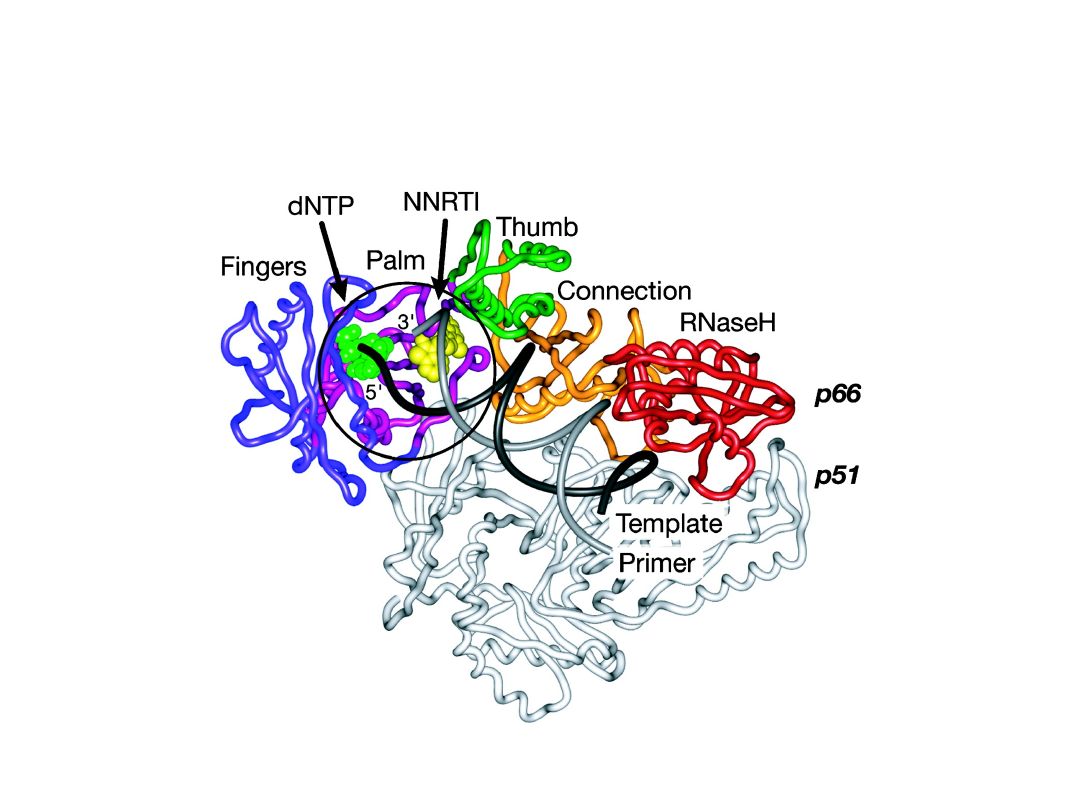
Struktura krystalograficzna
odwrotnej transkryptazy HIV-1
Centrum
aktywne
Podjednostka
Centrum aktywne
Podjednostka

Odwrotne transkryptazy służące
w RT- PCR:
RT HIV-1
Telomeraza
RT M-MLV

Inhibitor RT HIV-1
Inhibitorem RT HIV-1 jest azydotymina,
zapobiega ona replikacji i wstrzymuje syntezę
łańcucha DNA.
Jest jednym ze składników koktajlu inhibitorów
używanych w terapii wielolekowej AIDS.
Inhibitor RT HIV-1

Telomeraza
• Telomeraza- enzym o aktywności polimerazy DNA
zależnej od RNA. Jego zadaniem jest dobudowanie 3’
końcowego odcinka DNA na nici opóźnionej (
synteza
telomerów
). W ten sposób zabezpiecza strukturę DNA
przed skracaniem się.
• Telomeraza jest wyjątkowo aktywna podczas rozwoju
embrionalnego. Jej ekspresja w komórkach somatycznych
obniża się jednak w parę tygodni po narodzinach.
• Z jej mutacjami związane są takie choroby, jak: wrodzona
dyskeratoza, anemia plastyczna, idiopatyczne włóknienie
płuc. Schorzenia te są wynikiem występowania zbyt
krótkich i dysfunkcyjnych telomerów.
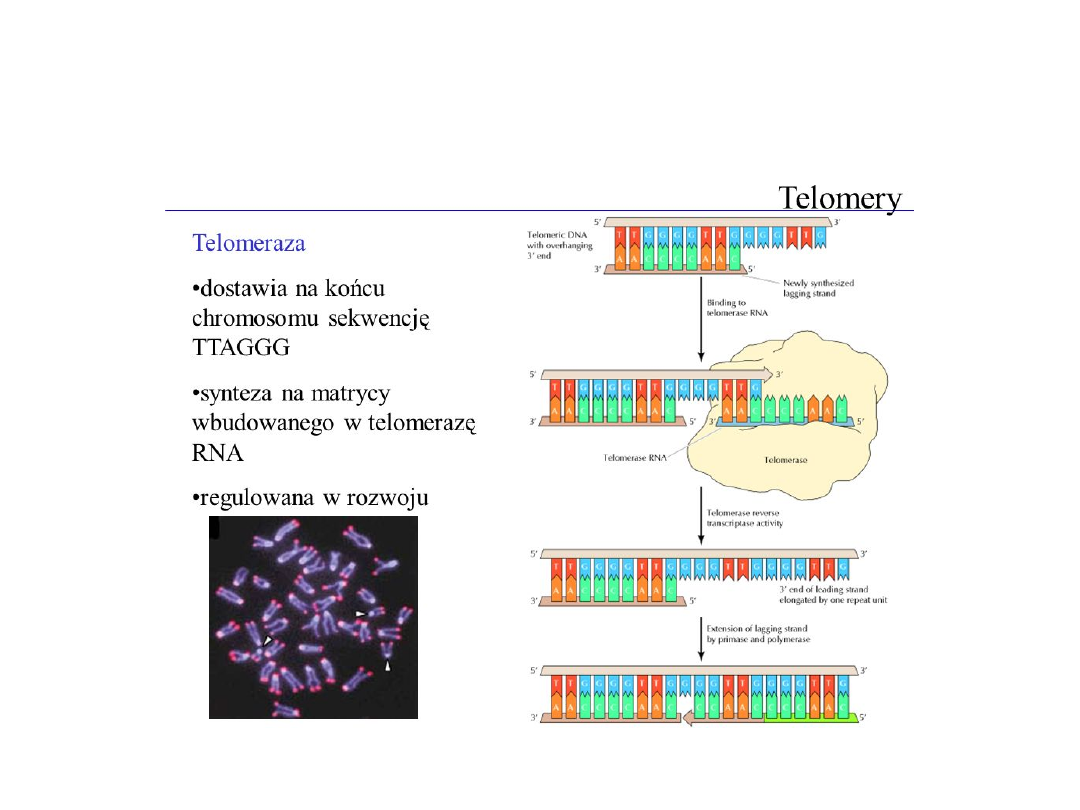
Telomeraza

RT M-MLV
RT M-MLV (Moloney murine leukemia virus)
monomeryczny enzym wyizolowany z mysiego
wirusa leukemii (białaczka spowodowana np.
działaniem retrowirusów).
jest rekombinowaną polimerazą DNA , która
syntezuje nowy łańcuch DNA z pojedynczej nici
RNA, DNA, bądź hybrydy DNA- RNA (dwuniciowa
cząsteczka kwasu nukleinowego DNA + RNA
połączone na zasadzie komplementarności).
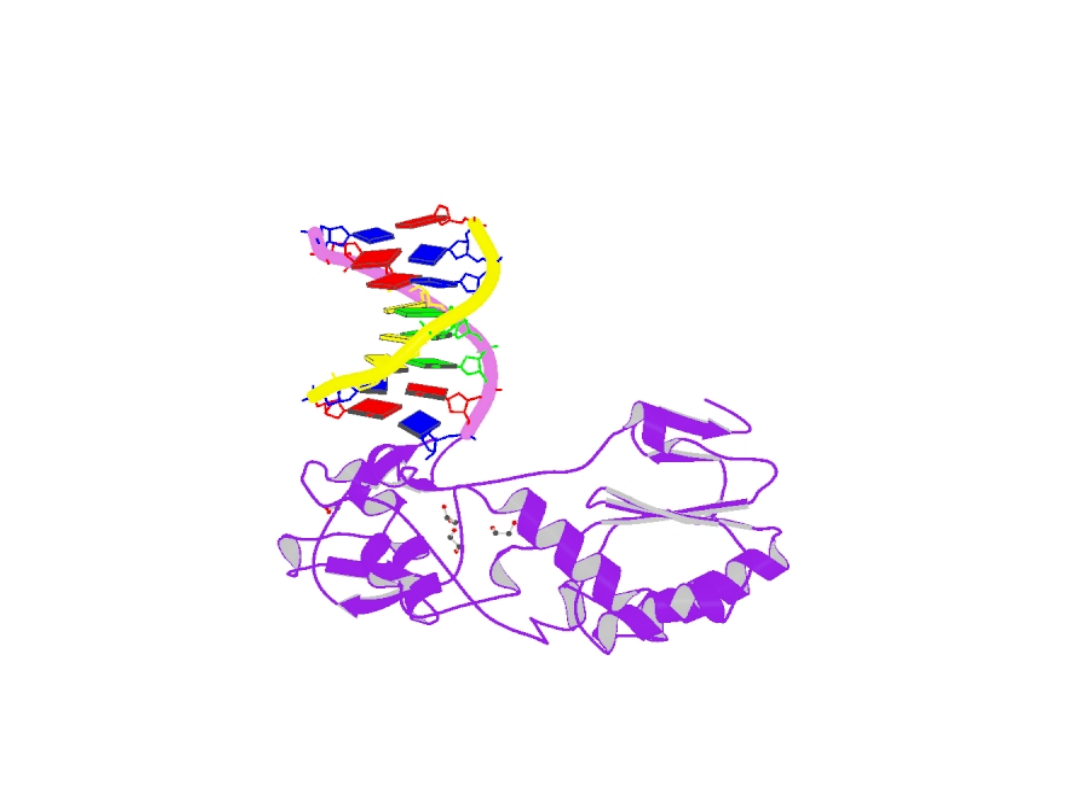
RT M-MLV

Real-time PCR
Reakcja łańcuchowa polimerazy DNA z analizą
ilości produktu w czasie rzeczywistym (ang. real-
time PCR); czuła metoda analityczna stosowana
w genetyce.
Główne cechy real time PCR:
a. umożliwia jednoczesne namnażanie DNA oraz
monitorowanie ilości produktów powstających
podczas kolejnych cykli (amplikonów),
b. czułość
c. metoda jest znacznie mniej czaso- i
pracochłonna,
d. szeroki zakres oznaczanych stężeń oraz wysoka
powtarzalność uzyskiwanych wyników
e. wysoki koszt analizy i drogi sprzęt

W skład mieszaniny reakcyjnej
standardowej reakcji PCR wchodzą:
• oligonukleotydowe primery
• nukleotydy będące substratami
• bufor
zapewniający
odpowiednie
środowisko reakcji
• jony magnezu
• termostabilna
polimeraza
DNA:
polimeraza Taq (Thermus aquaticus),
Pfu DNA polimeraza (Pyrococcus furiosus).
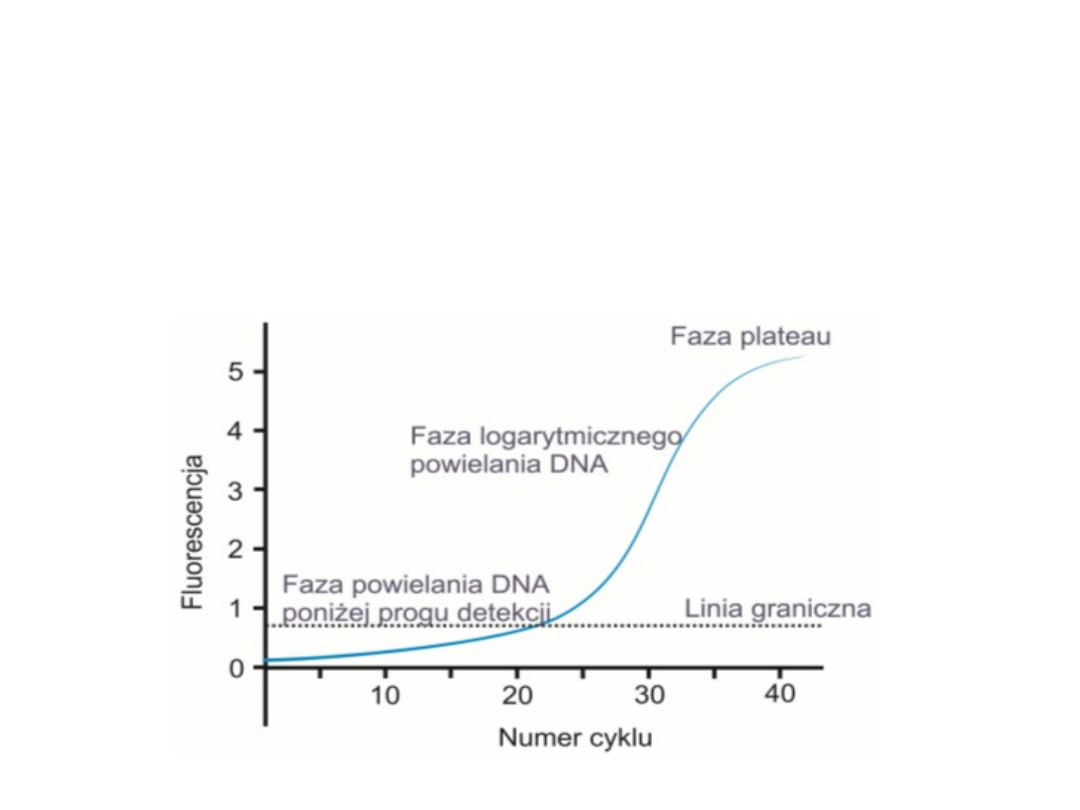
Fluorescencja
• Dzięki zjawisku fluorescencji real- time PCR pozwala
śledzić proces namnażania się DNA w czasie rzeczywistym i
tym samym oznaczyć ilości produktu w fazie jego
wykładniczego wzrostu.
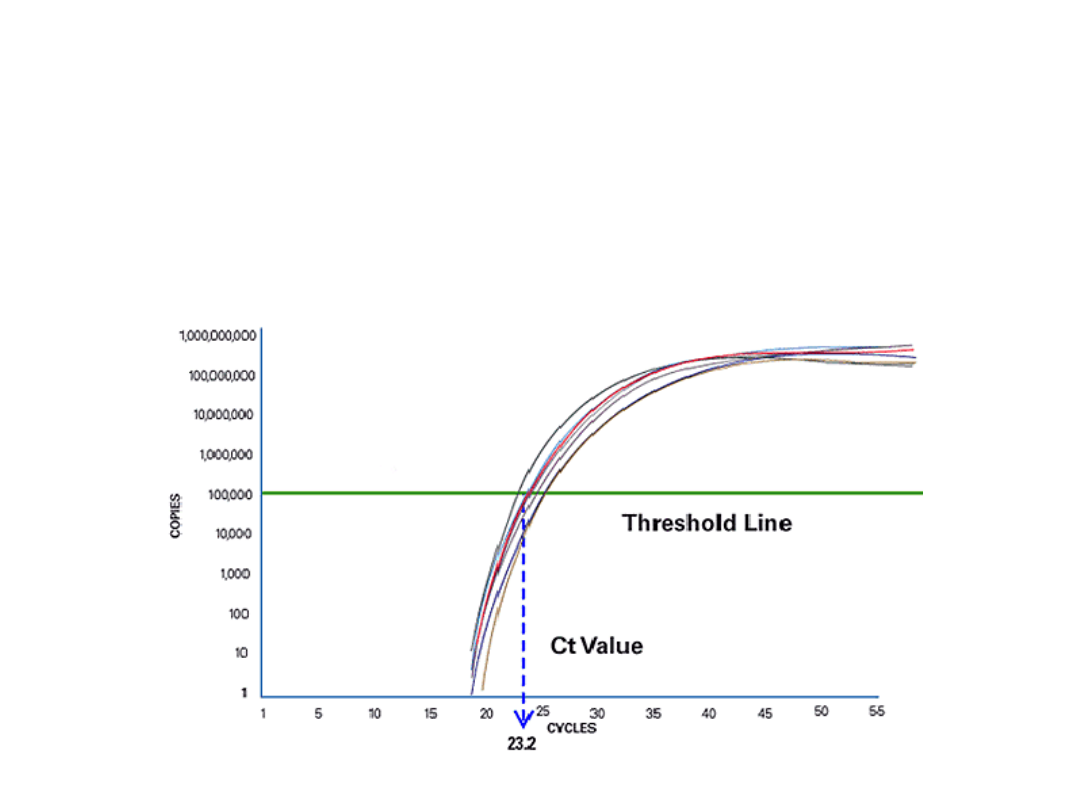
Wartość C
t
• Cykl, w którym sygnał fluorescencji osiągnął wartość
graniczną pozwalającą ma jego wykrycie (ang. threshold
cycle C
t
). Od cyklu progowego rozpoczyna się wczesna
faza logarytmiczna PCR.
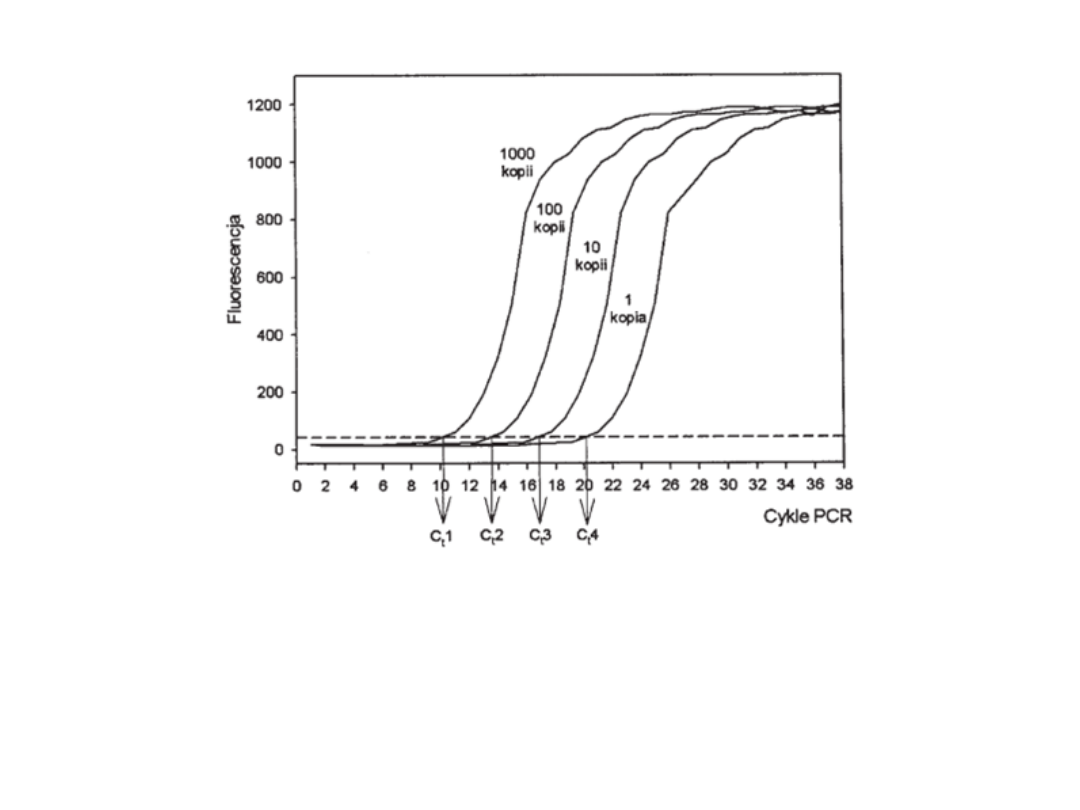
Im więcej kopii powielanej sekwencji obecnych jest
w próbie w momencie rozpoczęcia reakcji, tym
mniej
cykli
potrzeba,
aby
intensywność
fluorescencji przekroczyła poziom tła.

Znaczniki do
fluorescencji:
specyficzne (zależne od sekwencji
nukleotydów), np. wygaszacze i
reportery w sondach TaqMan (wyg.:
TAMARA rep.: 6-
karboksyfluoresceina) i typu „spinka
do włosów”.
niespecyficzne (niezależne od
sekwencji nukleotydów; wykorzystują
barwniki wiążące się z DNA) np.
SYBR Green I, bromek etydyny

Metody specyficzne
• z użyciem barwników specjalnie zaprojektowanych i
zsyntetyzowanych dla danej sekwencji. Zasada ich
działania opiera się na rezonansowym transferze
energii fluorescencji (FRET) pomiędzy cząsteczką
donora i wygaszacza.
1. Liniowe sondy oligonukleotydowe/ sondy sąsiadujące
(ang. dual hybridisation probes/adjacent probes).
2. Sondy ulegające reakcji hydrolizy, czyli fluorescencja
z
wykorzystaniem
aktywności
5’-nukleazowej
polimerazy (np. TaqMan).
3. Sondy o strukturze spinki do włosów (ang. hairpin
oligoprobes).
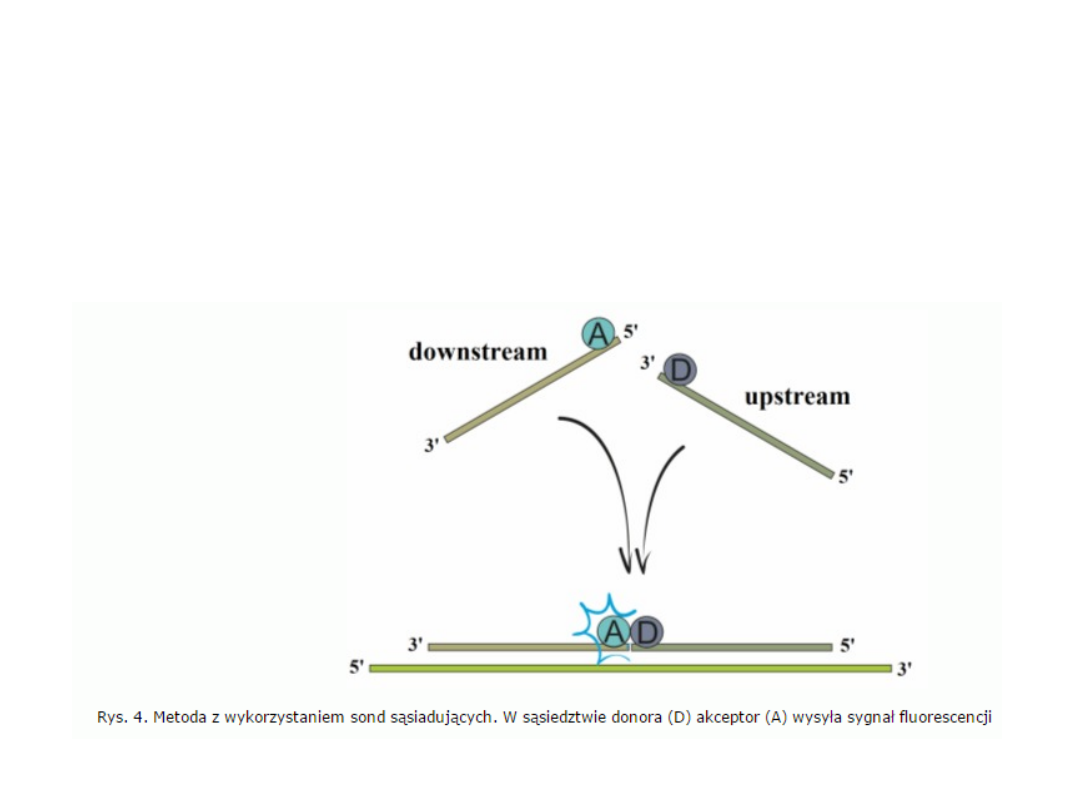
Sondy liniowe
• Metoda z użyciem pary fluorescencyjnych sond
oligonukleotydowych, emitujących sygnał jedynie
wtedy, gdy zbliżają się do siebie w wyniku
przyłączenia się do matrycy DNA.

Sonda TaqMan
• Jest to krótki odcinek DNA wyznakowany cząsteczką
fluorescencyjną na końcu 5’ oraz związkiem wygaszającym
na końcu 3’. Gdy znajdują się one w bliskim sąsiedztwie,
tzn. obie są przyłączone do tego samego oligonukleotydu,
wygaszacz pochłania sygnał emitowany przez reporter.
Podczas amplifikacji oligonukleotyd ulega rozdzieleniu
dzięki aktywności 5’ egzonukleazowej polimerazy DNA
Taq. Reporter i wygaszacz zostają rozdzielone i w ten
sposób zostaje uwolniony sygnał fluorescencji.
Najpowszechniejsze wygaszacze:
• TAMARA, DABCYL, BHQ.
Najpopularniejsze reportery:
• FAM, VIC, NED oraz pochodne fluoresceiny (np. izotiocyjanian
fluoresceiny, 6-karboksyfluoresceina)
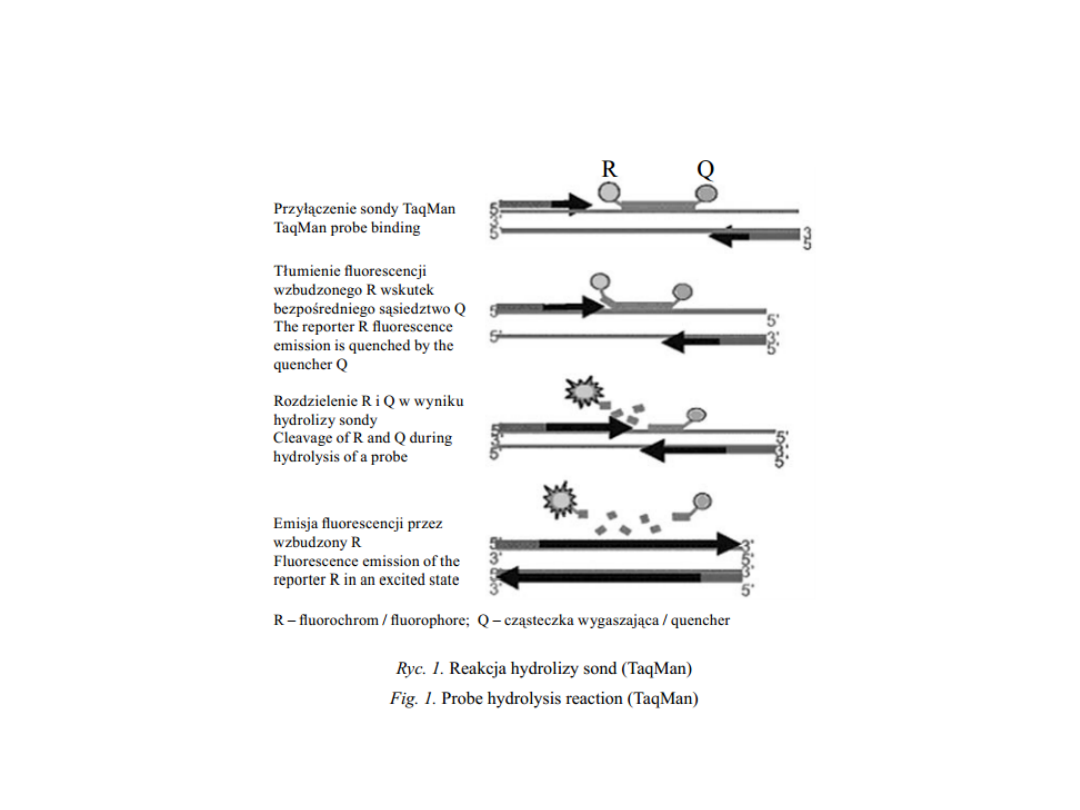
Sonda TaqMan

Sonda TaqMan
Zalety:
• wysoka specyficzność,
• wykrywają ewentualne mutacje punktowe
zachodzące podczas powielania prób.
Wady:
• wysoki koszt metody.

Sondy typu „spinka do włosów”
Dzięki
obecności
w
tych
sondach
komplementarnych
sekwencji
wewnętrznych,
ich
drugorzędowa
struktura
przybiera
charakterystyczną
postać spinki do włosów, która jest
niszczona podczas amplifikacji. Powoduje
to rozdzielenie reportera i wygaszacza, a
zatem wyzwolenie sygnału fluorescencji,
który wcześniej był rozpraszany w postaci
ciepła.

Molecular Beacon
jako
pierwsza
w
rt-PCR
wykorzystuje
pojedynczą sondę o kształcie spinki do
włosów, której część osiowa (stem) zawiera
komplementarne do siebie odcinki, a pętla
(loop) – sekwencje komplementarne do
sekwencji DNA.
Z sondą są związane dwie cząsteczki –
fluorochrom znajdujący się na końcu 5’ i
wygaszacz zlokalizowany na przeciwległym
końcu.

Istota MB
• Zamknięta struktura tej sondy utrzymuje
fluorochrom i wygaszacz w bliskim sąsiedztwie,
co powoduje, że przy odpowiedniej długości fali
świetlnej nie dochodzi do emisji światła. Dzieje się
tak dlatego, że wygaszacz, który znajduje się w
bezpośredniej odległości od wzbudzonego
fluorochromu przechwytuje jego energię,
uniemożliwiając emisję.
• Sonda ulega rozpleceniu w przypadku napotkania
sekwencji w pełni komplementarnej do sekwencji
wewnętrznej sondy. Następuje wówczas oddzielenie
reportera i wygaszacza oraz emisja fluorescencji.
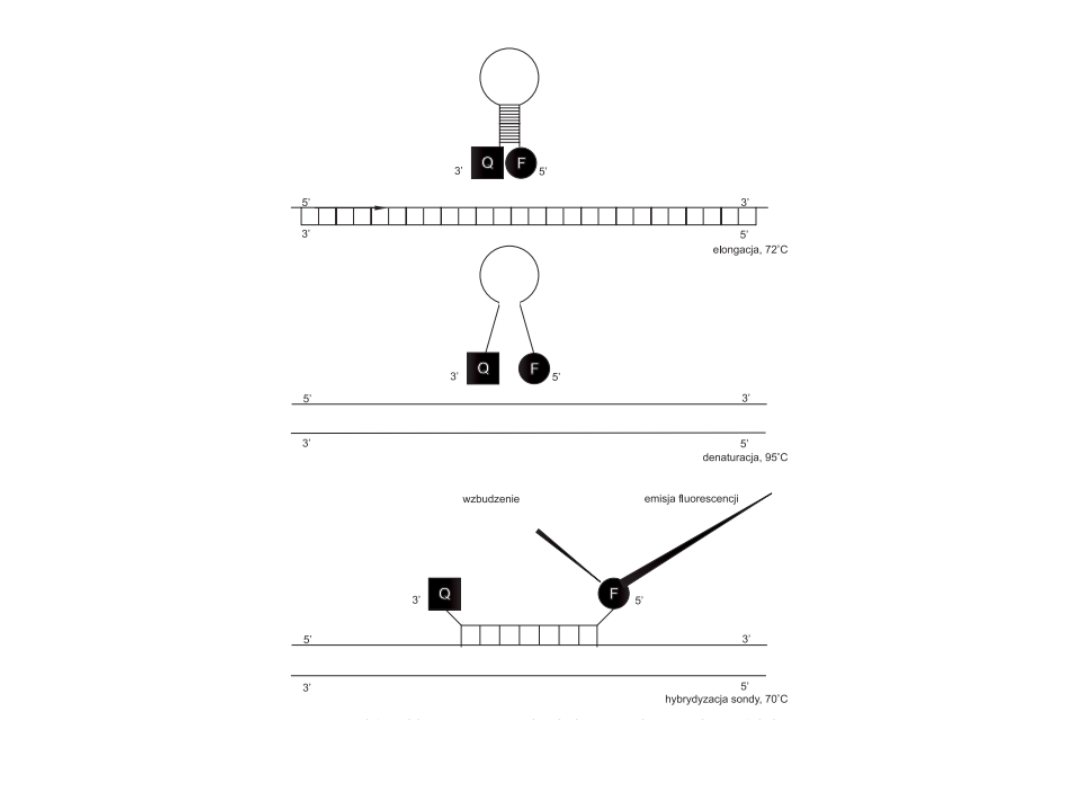

Molecular Beacon
Zalety:
• jedno z najczulszych narzędzi służących do
wykrywania mutacji, ponieważ kompleks sonda-
matryca musi być termodynamicznie stabilniejszy
niż struktura spinki do włosów,
• duża specyficzność względem powielanego
fragmentu.
Wady:
• występowanie niedopasowań (ang. mismatch)
pomiędzy sekwencją sondy, a powielanym
fragmentem. Prowadzi ono do destabilizacji
dupleksu sonda-matryca.

Fluorochrom SYBR Green I –
metoda niespecyficzna
• najczęściej stosowany niespecyficzny
barwnik,
• najprostszy i najbardziej ekonomiczny
sposób
na
detekcję
i
ilościowe
oznaczenie produktów reakcji rt-PCR,
• metoda wykorzystywana jest do analizy
liczby
kopii
cząsteczek
cDNA
przepisanego z RNA, gdzie dsDNA
będzie produktem reakcji PCR.
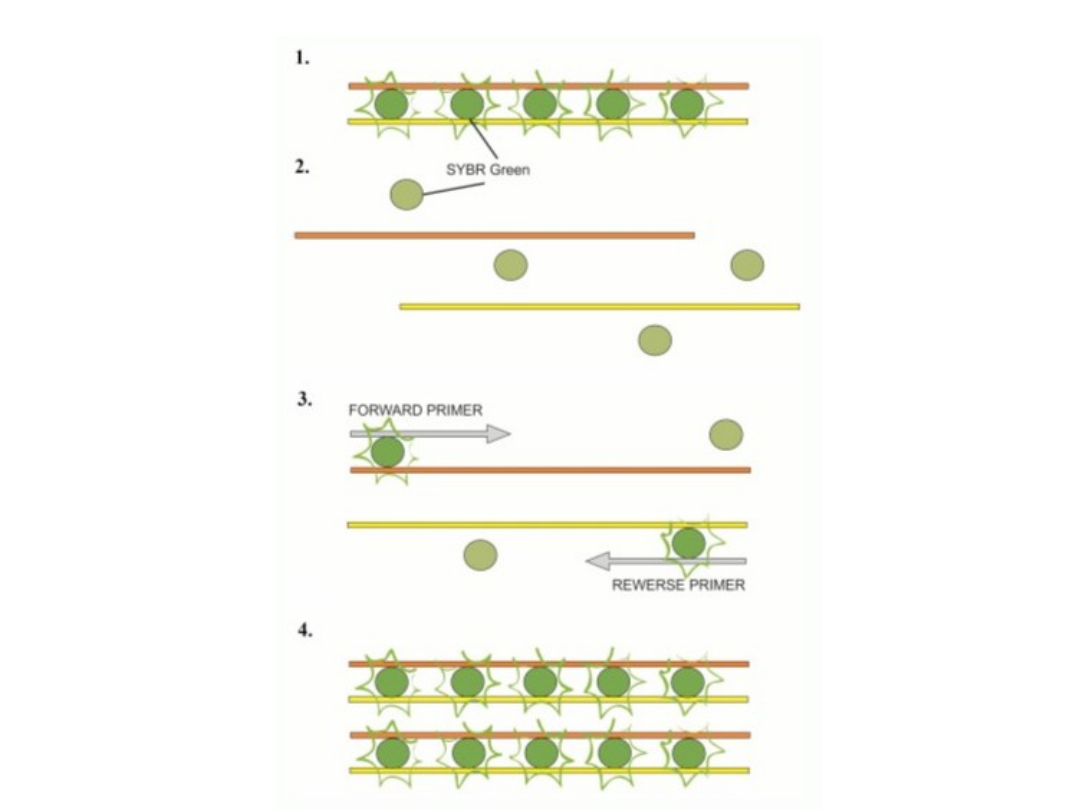

SYBR Green I
Zalety:
• nie wymaga stosowania skomplikowanej
sondy, co znacznie obniża koszty.
Wady:
• bardziej podatna na błędy. Podobnie jak
bromek etydyny, barwnik ten, interkaluje
do każdej dwuniciowej cząsteczki DNA, w
tym także do dimerów, starterów oraz
niespecyficznych produktów reakcji.

Real-time PCR -
podsumowanie
• Real-time PCR umożliwia precyzyjne
rozróżnienie i oznaczenie sekwencji kwasów
nukleinowych nawet w bardzo małej próbce.
• Może być wykorzystywana do wykonywania
analiz na obecność obcych fragmentów
DNA.
• Znajduje szerokie zastosowanie, m.in. do:
wykrywania znanych mutacji, wykrywania
patogenów
(wirusy,
bakterie,
grzyby),
badania poziomu ekspresji genów.

Źródła
•
http://www.pfb.info.pl/files/kwartalnik/1_2008/stud
zinska-tyburski.pdf
•
http://www.pum.edu.pl/__data/assets/pdf_file/00
09/13320/3528_1.pdf
•
http://www.e-biotechnologia.pl/Artykuly/real-time-
pcr
•
•
http://www.izoo.krakow.pl/czasopisma/wiadzoot/
2011/1/art18_WZ_2011_1.pdf
Document Outline
- Odwrotne transkryptazy
- Struktura krystalograficzna odwrotnej transkryptazy HIV-1
- Odwrotne transkryptazy służące w RT- PCR:
- Inhibitor RT HIV-1
- Inhibitor RT HIV-1
- Telomeraza
- Telomeraza
- RT M-MLV
- RT M-MLV
- Real-time PCR
- Slide 11
- Fluorescencja
- Wartość Ct
- Slide 14
- Znaczniki do fluorescencji:
- Metody specyficzne
- Sondy liniowe
- Sonda TaqMan
- Sonda TaqMan
- Sonda TaqMan
- Sondy typu „spinka do włosów”
- Molecular Beacon
- Istota MB
- Slide 24
- Molecular Beacon
- Fluorochrom SYBR Green I – metoda niespecyficzna
- Slide 27
- SYBR Green I
- Real-time PCR - podsumowanie
- Źródła
Wyszukiwarka
Podobne podstrony:
Real time pcr, pomoce naukowe, biotechnologia
REAL TIME PCR czarus1
REAL TIME PCR genotypowanie
Podstawy techniki real time PCR
ODWROTNA TRANSKRYPCJA RT PCR
2002%20 %20June%20 %209USMVS%20real%20time
REAL TIME
Embedded Linux Ready For Real Time Montavista
Continuous real time data protection and disaster recovery
Nukleotydopodobne inhibitory odwrotnej transkryptazy
Odwrotna transkryptaza
5 wirusy odwrotnie transkrybujace i ich wlasnosci onkogenne
2002%20 %20June%20 %209USMVS%20real%20time
Zero hour, Real time Computer Virus Defense through Collaborative Filtering
Revealing the Form and Function of Self Injurious Thoughts and Behaviours A Real Time Ecological As
A Model for Detecting the Existence of Unknown Computer Viruses in Real Time
Facebook tworzenie aplikacji real time updates
[Filmmaking Technique] The virtual cinematographer a paradigm for automatic real time camera contr
więcej podobnych podstron