Izolacja DNA plazmidowego z komórek bakterii Escherichia coli.
Część teoretyczna:
Plazmidy są to autonomiczne, pozachromosomowe elementy genetyczne występujące (zwykle w postaci kolistych lub rzadziej liniowych cząsteczek DNA) u bardzo wielu organizmów prokariotycznych oraz u niektórych eukariotów. Ich najbardziej charakterystyczną cechą jest fizyczna odrębność od chromosomu gospodarza oraz zdolność do trwałego utrzymywania się (ang. maintenance) w komórce i replikowania się w niej w kontrolowany sposób.
Cechy plazmidów:
Koegzystują w komórce bakteryjnej z głównym chromosomem
Zawierają markery selekcyjne, które są genami kodującymi określone cechy fenotypowe,
Przenoszą geny, które zwykle nie są obecne na głównym chromosomie, często geny, które nie są niezbędne, np. geny oporności na antybiotyki
Mają zdolność do przenoszenia się z komórki do komórki
Mogą uczestniczyć w wymianie i rekombinacji materiału genetycznego w naturalnych warunkach
Mają powszechne zastosowanie w inżynierii genetycznej i technologii genowej.
Zdolne są do samodzielnego powielania się niezależnie od cyklu życiowego komórki, dzięki własnym miejscom inicjacji replikacji (ori, od ang. origin)
Występują w określonej liczbie kopii w komórce, im większy plazmid tym mniejsza liczba kopii
Wielkość naturalnych plazmidów waha się od 1000 do 17000 pz
Laboratoryjne często zawierają "polylinker" (MCS, ang. multiple clonning site) sekwencja DNA, gdzie znajdują się pojedyncze miejsca cięcia dla dużej ilości enzymów restrykcyjnych
POZDIAŁY PLAZMIDÓW:
Typ plazmidu |
Funkcje genów |
Przykłady |
Warunkujący oporność (R) - (ang. Resistance) |
Oporność na antybiotyk |
Rbk E. coli i innych bakterii |
Warunkujący płeć (F) - (ang. Fertility) |
Koniugacja i transfer DNA między bakteriami |
F E. coli |
kolicynogenne (Col)
|
Synteza toksyn, które zabijają inne bakterie |
Col E. coli, wytwarzanie kolicyny |
Degradacyjny |
Enzymy metabolizujące niezwykłe cząsteczki |
TOL Pseudomonas putida, metabolizm toluenu |
Wirulentny |
patogenność |
Ti Agrobacterium tumefaciens, nadaje zdolność wywoływania guzowatych narośli na korzeniach roślin dwuliściennych |
Ze względu na liczbę kopii przypadającą na komórkę:
Jednokopijne (=centromeryczne) - tylko 1 kopia na komórkę, zachowują się jak „mini chromosomy”, zawierają sekwencję cen, która często w postaci zduplikowanej jest zabójcza dla komórki
Wielokopijne (=multikopijne) - wiele kopii w komórce, zawierają region drożdżowego 2 mikronowego DNA
Ze względu na postać, w jakiej występują w komórce:
kolista, kowalencyjnie zamknięta dwuniciowa cząsteczka DNA
liniowa, dwuniciowa cząsteczka DNA
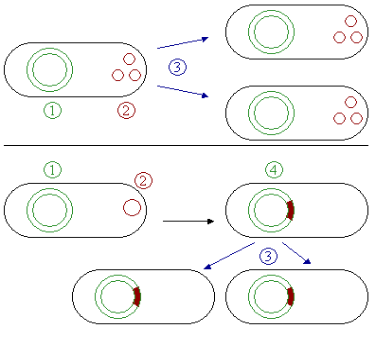
Niektóre plazmidy można nazwać EPISOMAMI. Są to takie elementy, które mogą istnieć zarówno jako autonomiczny kolisty plazmid lub jako linearna sekwencja wbudowana do chromosomu bakteryjnego.
Na bazie plazmidów naturalnych skonstruowano wiele wektorów wykorzystywanych powszechnie w biologii molekularnej:
Wektor wahadłowy, inaczej bifunkcjonalny — typ wektora, który może utrzymać się i replikować w dwóch różnych rodzajach komórek (najczęściej są to drożdże i bakterie). Warunkiem do tego, aby jakiś wektor mógł przetrwać i ulegać powielaniu w różnego rodzaju komórkach-gospodarzach jest występowanie w tym wektorze dwóch miejsc zapoczątkowania replikacji (miejsc ori), tak by polimerazy DNA obu rodzajów komórek-gospodarzy mogły odnaleźć odpowiednie sekwencje. Dzięki tym wektorom możliwe jest klonowanie genów w komórkach bakteryjnych a badanie ich ekspresji w drożdżach.
Wektory ekspresyjne: zawierają odpowiednio usytuowany silny promotor umożliwiający ekspresję na wysokim poziomie białka, którego gen znajduje się na tym wektorze. Plazmidowe wektory ekspresyjne zawierające silne, regulowane promotory. Taki silny, regulowany promotor powoduje, że w specyficznych warunkach transkrypcja jest inicjowana wiele razy na minutę. Przykładem może być promotor laktozowy. Transkrypcję wzmaga się przez dodanie do pożywki aktywatora IPTG (analog laktozy).
Wektory pochodne bakteriofagów: Do najczęściej stosowanych, wśród tej klasy, należą wektory, będące pochodnymi faga lambda. Wektory te mają pewną przewagę nad wektorami plazmidowymi. Infekcja cząstkami bakteriofagów jest dużo bardziej wydajna od transformacji bakteryjnej. Jednocześnie, mogą one pomieścić dużo więcej DNA do klonowania. W genom faga lambda można też wprowadzić mutacje punktowe, które w niektórych genach zmieniają właściwości ich produktów (np. krótkie delecje oraz niekiedy usunięcie środkowego regionu DNA odpowiedzialnego za cykl lizogenny). Z DNA faga można usunąć około 30% genomu pozostawiając jego lewe i prawe ramię zawierające wszystkie geny niezbędne w cyklu litycznym. Dzięki 12 nukleotydowym, wzajemnie komplementarnym, jednoniciowym fragmentom DNA na końcach cząsteczki DNA faga - tzw. sekwencjom cos - możliwe jest przybranie przez DNA faga w komórce bakteryjnej formy kolistej oraz zapakowanie go w białkową główkę. Również w te wektory wstawione są polilinkery, geny markerowe, silne promotory i inne sekwencje użyteczne w genetyce molekularnej.
Inne znane wektory tej klasy to na przykład pochodne faga M13 - szczególny cykl życiowy tego faga pozwala na wykorzystanie go do wytwarzania jednoniciowego DNA, jest wieć bardzo użyteczny w sekwencjonowaniu DNA lub ukierunkowanej mutagenezoe.
Kosmidy- to sztucznie wytworzone wektory używane w inżynierii genetycznej. Kosmidy tworzy się łącząc plazmidy z sekwencją cos bakteriofaga λ. Dzięki temu, tego rodzaju wektor zyskuje właściwości charakterystyczne zarówno dla plazmidu jak i dla faga. Dzięki sekwencji cos kosmidy przyjmują formę kolistą i replikują się jak plazmidy. Zrekombinowane kosmidy są pakowane w kapsydy faga λ. W zainfekowanej komórce bakterii kosmid namnaża się, jednak w przeciwieństwie do faga nie niszczy jej. Replikacja kosmidów jest bardziej czasochłonna niż replikacja plazmidów (ze względu na duże rozmiary wprowadzonego obcego DNA), a liczba kopii kosmidów jest niższa niż plazmidów w danej komórce. Dzięki kosmidom możliwe jest klonowanie długich fragmentów DNA, na co nie pozwala użycie plazmidów (kapsydy λ mogą przyjąć do 52 kpz, natomiast w kosmidach można klonować wstawki wielkości do 44 kpz.).
Wektory drożdżowe- Drożdże jako komórki gospodarzy posiadają niezaprzeczalne zalety: mają struktury komórkowe typowe dla eukariota, geny w nich klonowane mogą zawierać sekwencje intronowe, proces obróbki mRNA jest zbliżony do tego, który występuje u wyższych eukariontów, białka podlegają modyfikacjom potranslacyjnym. Klonowanie w drożdżach jest bardzo użyteczne, gdy zależy nam na uzyskaniu ekspresji badanego genu w komórce eukariotycznej i odpowiednio zmodyfikowanych białkowych produktów. W laboratoriach wykorzystuje się dwa typy tych wektorów: replikatywne - gdy ori pochodzi z chromosomów drożdży (tzw. sekwencja ars) lub episomalne - gdy ori pochodzi z naturalnie występującego u drożdży plazmidu 2μ. Poza tym wektory te posiadają geny markerowe oraz polilinkery
Wektor YAC (sztuczny chromosom drożdżowy)- Zawiera on te cechy chromosomu, które są potrzebne do jego powielania w komórkach drożdży (początek replikacji, centromer, telomery). Mogą one przyjąć bardzo duże cząsteczki DNA o długości kilkuset kpz. Indywidualne klony mogą nawet zawrzeć cały gen ssaka. Wektory te mają zastosowanie w konstruowaniu map części genomu ludzkiego poprzez identyfikację klonów zawierających sąsiadujące ze sobą regiony genomu. Podobne zastosowanie mają wektory BAC (sztuczny chromosom bakteryjny) i P1 (sztuczny chromosom).
Izolacja DNA plazmidowego
We wszystkich metodach izolacji plazmidowego DNA wykorzystuje się dwie istotne różnice pomiędzy DNA genomowym a DNA plazmidowym bakterii:
DNA genomowy jest wielokrotnie większy od DNA plazmidu i jest zakotwiczony w błonie,
podczas procedury izolacji DNA plazmidowego DNA genomowy zostaje trwale zniszczony, podczas gdy DNA plazmidowy pozostaje w postaci form CCC (ang. covalently closed circle).
Opracowano szereg metod oczyszczania plazmidowego DNA, z których każda obejmuje 3 etapy:
1. Hodowlę bakterii;
2. Lizę bakterii oraz oddzielenie DNA od „gruzu” komórkowego;
3. Oczyszczanie plazmidowego DNA.
# 1.
Plazmidy izoluje się najczęściej z hodowli płynnych, w których podłoże uzupełnione jest odpowiednim antybiotykiem (markerowy gen oporności). Wysokokopijne wektory plazmidowe (np. serii pUC), otrzymywane są z hodowli znajdującej się w późnej fazie logarytmicznej wzrostu, podczas gdy wektory nisko- i średniokopijne (np. pBR322) powinny być przed izolacją amplifikowane. Do częściowo wyrośniętej hodowli dodaje się w tym celu chloramfenikol, który selektywnie zapobiega replikacji chromosomu bakteryjnego.
# 2.
Bakterie ulegają lizie pod wpływem niejonowych lub jonowych detergentów (SDS, Sarkozyl, Triton X-100), rozpuszczalników organicznych, roztworów alkalicznych (liza alkaliczna) lub wysokiej temperatury (liza termiczna). Stosowany może być również lizozym - enzym trawiący ścianę komórkową bakterii (nie działa w pH<8.0)
# 3.
Etap oczyszczania DNA polega na oddzieleniu DNA od związanych z nim białek. Zwykle stosuje się do tego celu nasycony buforem roztwór fenolu lub jego mieszaninę z chloroformem i alkoholem izoamylowym. Białka można również usunąć przez trawienie ich proteinazami (zwykle proteinazą K). RNA, które izoluje się jednocześnie z DNA usuwa enzymatycznie, inkubując próbki z RNAzą (wolną od DNAz), przez sączenie molekularne lub wirowanie w gradiencie gęstości. Z odbiałczonych próbek, DNA wytrącany jest alkoholem etylowym lub izopropanolem w obecności octanu potasowego, sodowego lub amonowego.
Plazmidowy DNA po izolacji może przyjąć formę:
superhelisy - ccc (ang. covalently closed circular), kowalencyjnie zamknięta, kolista, dwuniciowa cząsteczka
otwartego koła - oc (ang. open circular), gdy pęka jedna z dwóch nici
liniową, gdy pękają obie nici DNA w tym samym miejscu
Izolacja plazmidowego DNA metodą lizy alkalicznej [Birnboim i Doly, 1979].
Etapy procesu:
Zawieszenie komórek w roztworze, który obniża wytrzymałość ścian komórkowych bakterii, hamuje DNazy przez chelatację jonów biwalencyjnych; niektórzy wraz z roztworem GTA dodają lizozym, trawiący ściany bakteryjne;
Zniszczenie komórek bakteryjnych - plazmid oraz pozostałe składniki komórki są uwalniane do roztworu. Jednocześnie zachodzi odwracalna denaturacja kwasów nukleinowych oraz nieodwracalna denaturacja białek.
Renaturacja plazmidowego DNA. Oddzielenie plazmidowego DNA od białek, resztek błon i chromosomu bakteryjnego.
Ad. 1
Komórki po zwirowaniu zostają zawieszone roztworze GTE (Glukoza/Tris/EDTA). Dodatkowo można stosować lizozym. Tris jest roztworu buforowym ma za zadanie utrzymać stałe pH (lekko zasadowe). EDTA jest związkiem chelatującym jony biwalencyjne (np. Ca2+, Mg2+). Wynikiem tego procesu są dwa efekty:
- zmniejszona stabilność ściany komórkowej bakterii spowodowana wiązaniem jonów Ca2+ przez EDTA; ściana komórkowa bakterii gram ujemnych składa się z jednej warstwy mureiny i otaczającej ją warstwy lipopolisacharydów, lipidów i lipoprotein; jak się wydaje, jony wapnia są potrzebne do zachowania stabilności warstwy lipopolisacharydowej;
- zahamowanie działania DNaz, przez jony magnezowe; bowiem te enzymy, jeśli byłyby aktywne mogłyby pociąć plazmidowe DNA;
Glukoza jest dodana w celu zachowania właściwego ciśnienia osmotycznego; jest to ważne szczególnie podczas stosowania lizozymu, który prowadzi do powstania protoplastów, komórek pozbawionych ściany, a przez to wrażliwych na zewnętrzne ciśnienie osmotyczne; zbyt niskie ciśnienie osmotyczne (czyli środowisko hipotoniczne) spowodowało by pobieranie wody przez komórki i ich pęknięcie; środowisko izoosmotyczne lub słabo hiperosmotyczne nie jest szkodliwe dla protoplastów.
Ad. 2
W tym etapie do zawiesiny komórek bakteryjnych dodaje się alkalicznego roztworu NaOH/SDS. W zasadowym środowisku, przy udziale SDS dochodzi do lizy komórek bakteryjnych. Uwolnione białka są denaturowane przez SDS i NaOH. Genomowy i plazmidowy DNA ulega denaturacji pod wpływem zasadowego pH. Po dodaniu NaOH/SDS w probówce znajduje się mieszanina zniszczonych błon, zdenaturowanych plazmidów, bakteryjnych chromosomów i białek, wolnych cząsteczek SDS i innych związków.
Ad. 3
Do roztworu lizatu dodawany jest octan potasu (bufor octanowy), który powoduje obniżenie pH. W efekcie jego działania plazmidowe DNA szybko renaturuje, podczas gdy chromosomowe DNA pozostaje zdenaturowane. Odmienna zdolność do renaturacji po obniżeniu pH jest istotą metody lizy alkalicznej. Pozwala na późniejsze łatwe oddzielenie DNA chromosomowego od plazmidowego. DNA genomowy wraz z białkami i fragmentami błon, wytrąca się w postaci serowatego osadu. Następnym krokiem jest wirowanie roztworu, by oddzielić plazmidowe DNA, które pozostaje rozpuszczone w supernatancie.
Po tym etapie otrzymana próbka może być dodatkowo odbiałczana mieszaniną fenol/chloroform, lub oczyszczana na minikolumnach będących fragmentem zestawów sprzedawanych przez firmy biotechnologiczne. Otrzymany, oczyszczony DNA plazmidowy można wytrącać w roztworze etanolu lub izopropanol.
Opracowanie: mgr Tomasz Bocer, dr Ewa Maciaszczyk
Część praktyczna:
Odczyt wyników transformacji komórek bakterii Escherichia coli.
Kolokwium.
Izolacja DNA plazmidowego:
Zaszczepić podłoże LB+Amp odpowiednim szczepem bakteryjnym i inkubować przez noc w 37°C z wytrząsaniem.
Hodowlę rozpipetować do probówek Eppendorfa po 1.5 ml i odwirować w temp. pokojowej przez 1 min. przy 12000 obr/min.
Osad komórek zawiesić w 100l buforu TGE (50mM Tris-HCl, 50mM glukoza, 10mM EDTA, pH 8,0).
Dodać 200 l buforu do lizy (1% SDS, 0,2 N NaOH), który należy przygotować bezpośrednio przed użyciem mieszając ze sobą równe objętości 2% SDS i 0,4 N NaOH. Zawartość mieszać delikatnie kilkukrotnie przez inwersję aż do uzyskania klarownego roztworu i inkubować do 5 min w lodzie.
Dodać 150 l 3M octanu sodu (pH 5,2), wstrząsnąć energicznie i inkubować przez 10 min w lodzie.
Odwirować w temp. pokojowej, przez 15 min przy 15000obr/min.
Supernatant przenieść ostrożnie do nowej probówki i można ponownie odwirować 5 min przy 15000 obr/min.
Supernatant przenieść do nowej probówki i dodać 850 l izopropanolu, delikatnie zamieszać.
Inkubować w temp. -20°C (zamrażarka) przez około 15 min.
Zawiesinę odwirować 15 min przy 15000 obr/min w temp. 4°C (lub temp. pokojowa).
Osad przemyć 70% EtOH (dodać l alkoholu zamieszać delikatnie), próbkę ponownie odwirować 1min przy 15000 obr/min i odciągnąć dokładnie supernatant,
Osad DNA suszyć w suszarce próżniowej przez 10 min.
Osad DNA zawiesić w 25 l wody miliQ i zamrozić w -20°C (zamrażarka) do następnych zajęć.
1
Plazmidy występujące w komórce jako wole, koliste, autonomiczne, pozachromosomowe cząsteczki DNA.
1 - chromosom główny
2 - plazmidy
3 - podział komórki
Plazmidy występujące w komórce jako koliste cząsteczki DNA mogą ulegać integracji do chromosomu głównego. Wówczas występują w postaci liniowej.
1 - chromosom główny
2 - plazmid
3 - podział komórki
4 - integracja plazmidu do chromosomu
Wyszukiwarka
Podobne podstrony:
VI-Elektroforeza DNA, Genetyka
Izolacja DNA plazmidy Genetyka Nieznany
IZOLACJA DNA ROŚLINNEGO METODĄ MIKRO C, Biotechnologia notatki, Genetyka - biologia molekularna
Ćw 2 Izolacja DNA plazmidy Genetyka
Izolacja DNA z komórek prokariotycznych i eukariotycznych
cwiczenie 8 izolacja DNA
BUDOWA CHEMICZNA DNA, Genetyka
Izolacja DNA z komorek zwierzecych
egzamin (11), pwr biotechnologia(I stopień), VI semestr, Inżynieria genetyczna - wykład, Egzamin
Etapy izolacji DNA
Izolacja DNA
egzamin (5), pwr biotechnologia(I stopień), VI semestr, Inżynieria genetyczna - wykład, Egzamin
egzamin (12), pwr biotechnologia(I stopień), VI semestr, Inżynieria genetyczna - wykład, Egzamin
egzamin (9), pwr biotechnologia(I stopień), VI semestr, Inżynieria genetyczna - wykład, Egzamin
Protokó- izolacji DNA na -wiczenia (1), analiza DNA
Izolacja DNA
egzamin (13), pwr biotechnologia(I stopień), VI semestr, Inżynieria genetyczna - wykład, Egzamin
egzamin (10), pwr biotechnologia(I stopień), VI semestr, Inżynieria genetyczna - wykład, Egzamin
egzamin (7), pwr biotechnologia(I stopień), VI semestr, Inżynieria genetyczna - wykład, Egzamin
więcej podobnych podstron