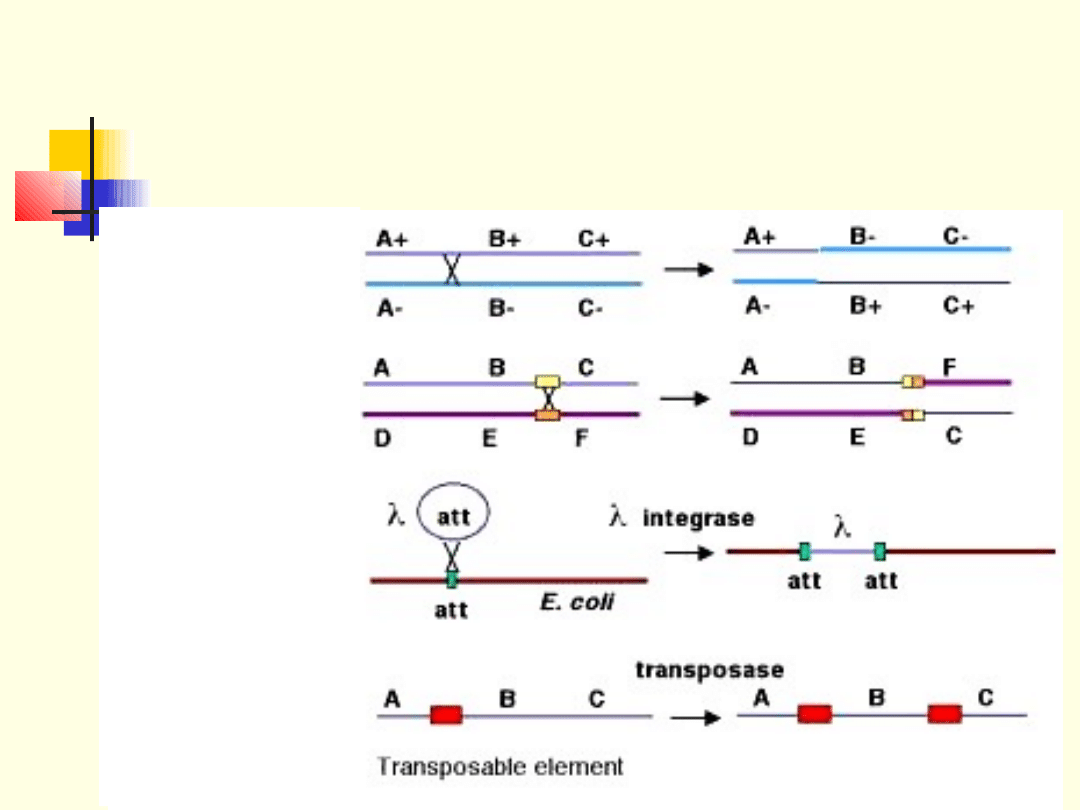
Różne rodzaje rekombinacji
Rekombinacja
homologiczna
Rekombinacja
niehomologiczna
– nieuprawniona
Rekombinacja
specyficzna
Transpozycja
replikatywna

Rekombinacja specyficzna
Zachodzi między specyficznymi parami sekwencji DNA np.
integracja faga do chromosomu
Wymagany jest przynajmniej krótki odcinek homologii (np.
attP
i
attB
u faga lambda)
Do rekombinacji swoistej niezbędna jest integraza oraz
białko IHF (
integration host factor
)
Wycięcie faga wymaga również integrazy rozpoznającej
końce genomu faga i białek Xis – enzym
eksisionaza
pomagający w wycięciu faga i IHF.
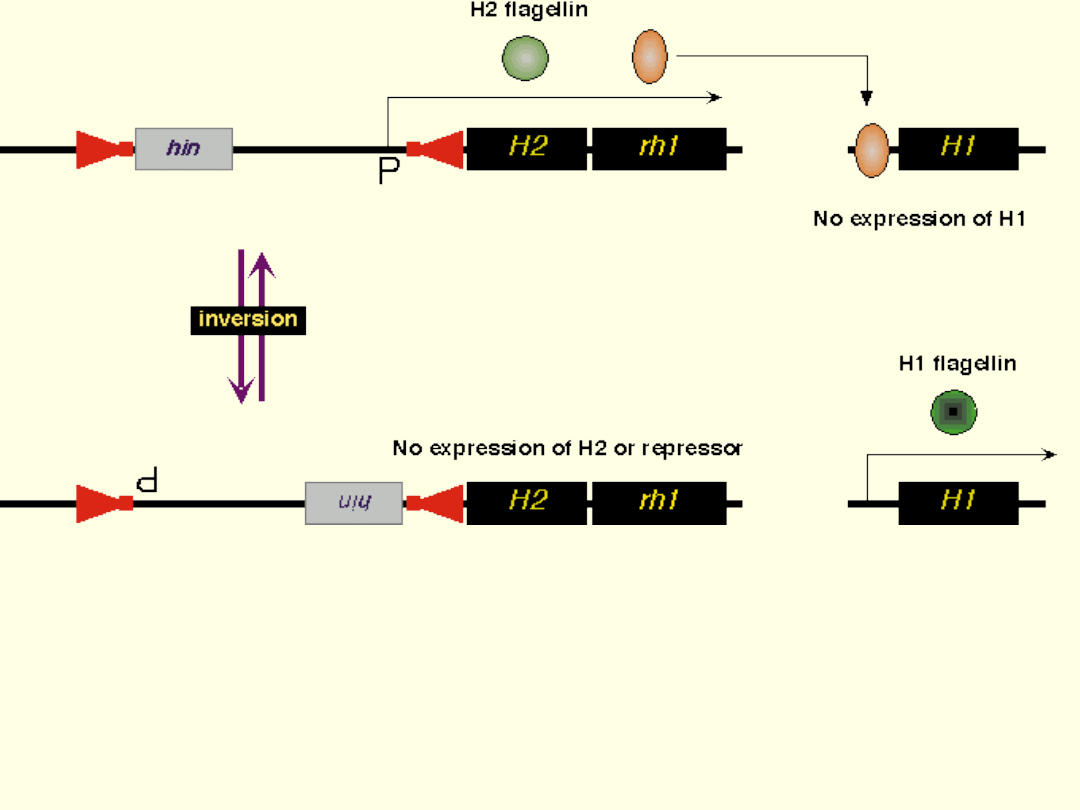
W jednej orientacji promotor operonu H2 może transkrybować operon który
zawiera dwa geny: H2 i rh1 koduje represor, który blokuje transkrypcję genu
H1. W ten sposób flagelina H2 jest syntetyzowana natomiast, H1 nie jest
syntetyzowana. Jednakże, często
rekombinaza Hin
powoduje inwersję, która
odwraca promotor o 180
o
co odsuwa promotor od operonu H2. Wtedy nie jest
syntetyzowana H2 flagelina i represor Rh1. Natomiast gen H1 jest
transkrybowany i flagelina H1 jest syntetyzowana.
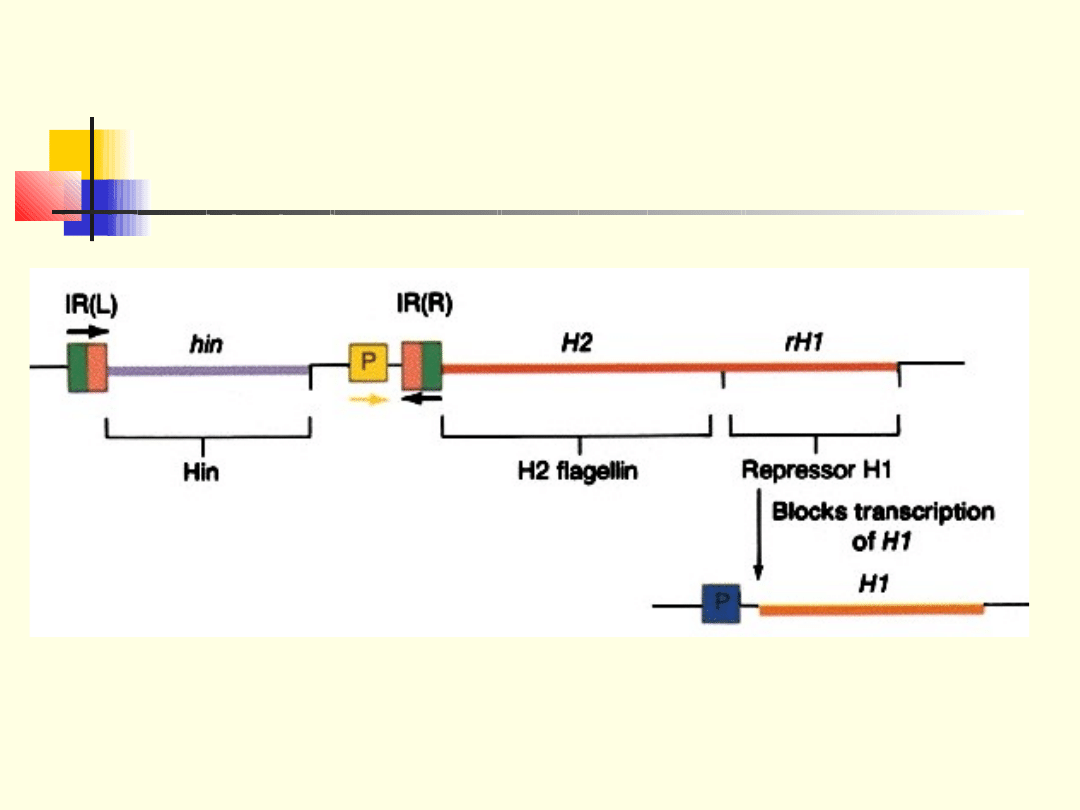
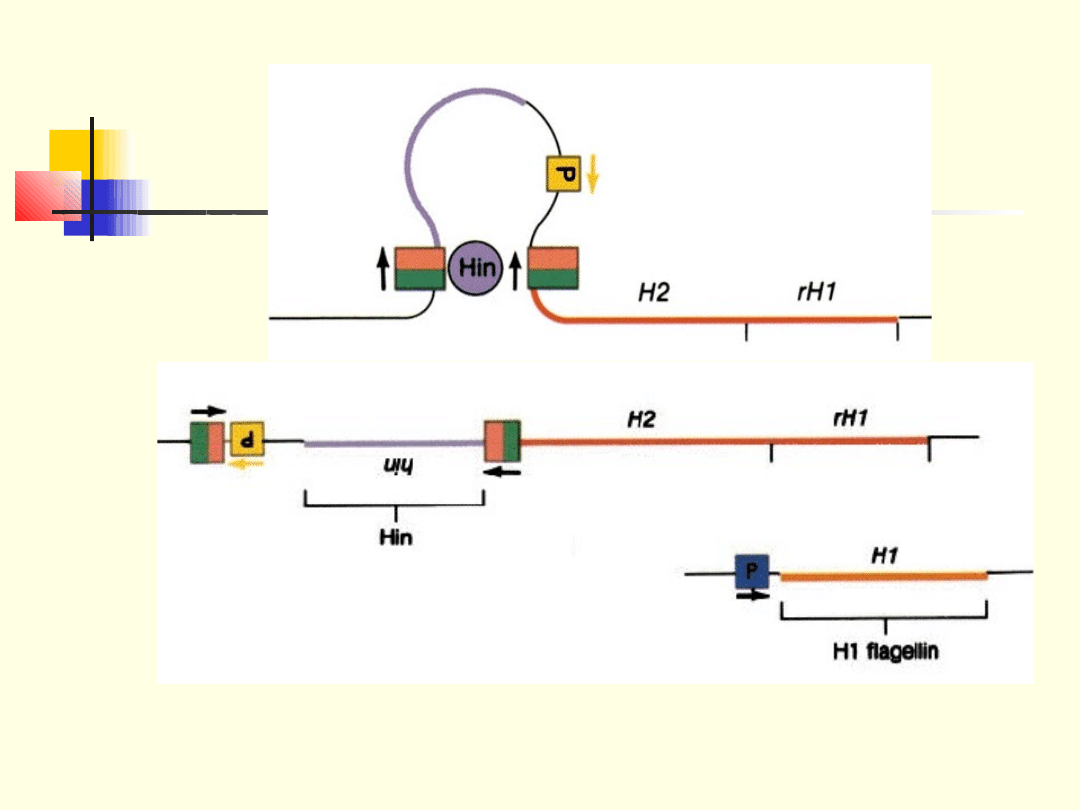
Zmienność fazowa
Salmonella
Gen
hin odpowiada za syntezę
rekombinazy Hin
, która działa
podobnie
do transposaz
, rozpoznaje końce (odwrócone powtórzenia i odwraca
odcinek DNA.
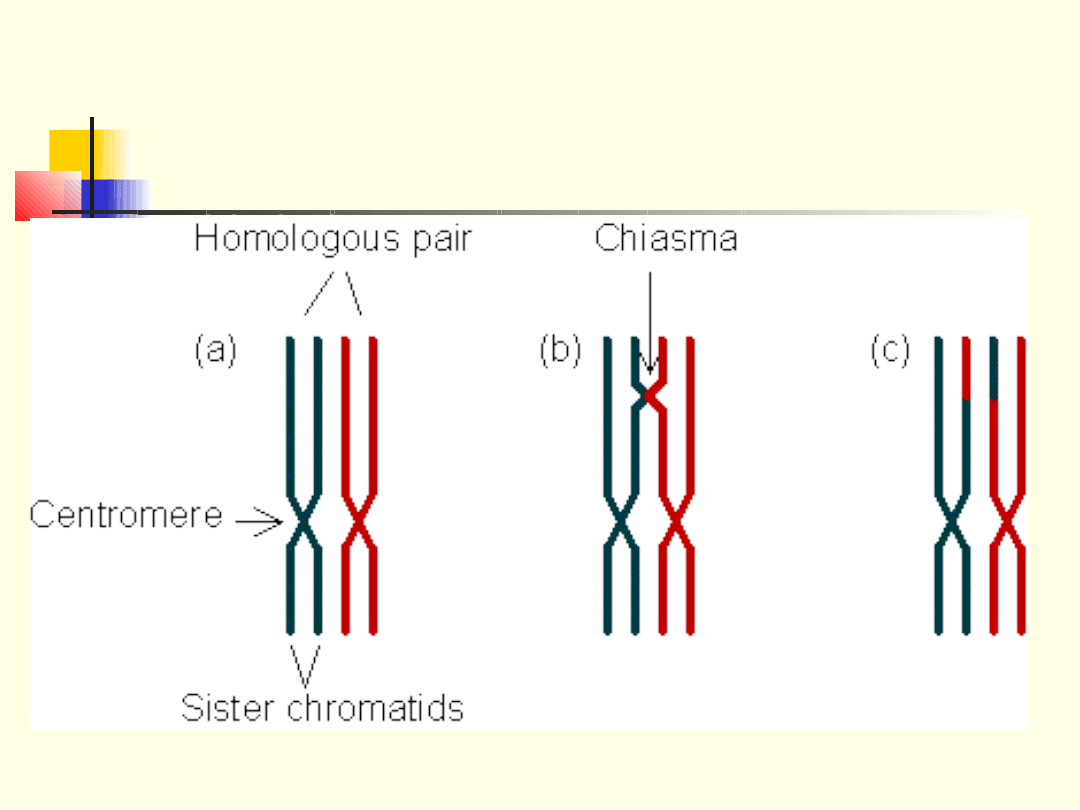
Crossing-over i rekombinacja między
homologicznymi regionami chromosomów
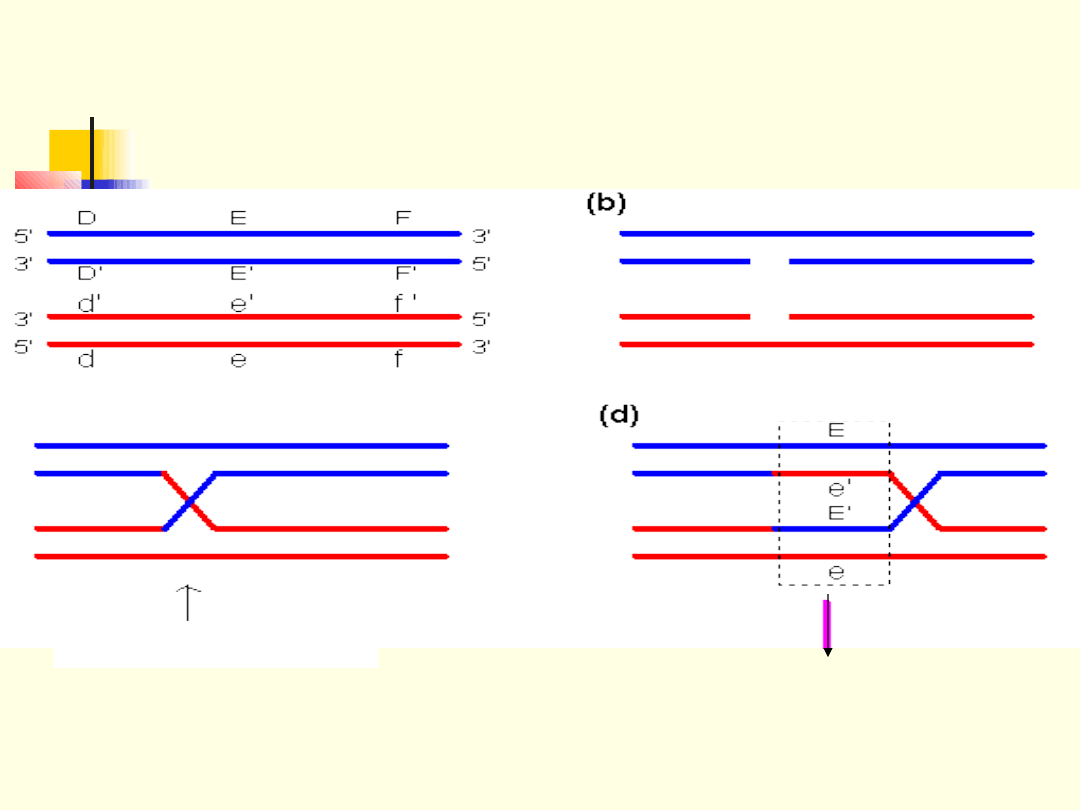
Mechanizm rekombinacji homologicznej
I
Struktura Holliday’a
Region heterodupleksu
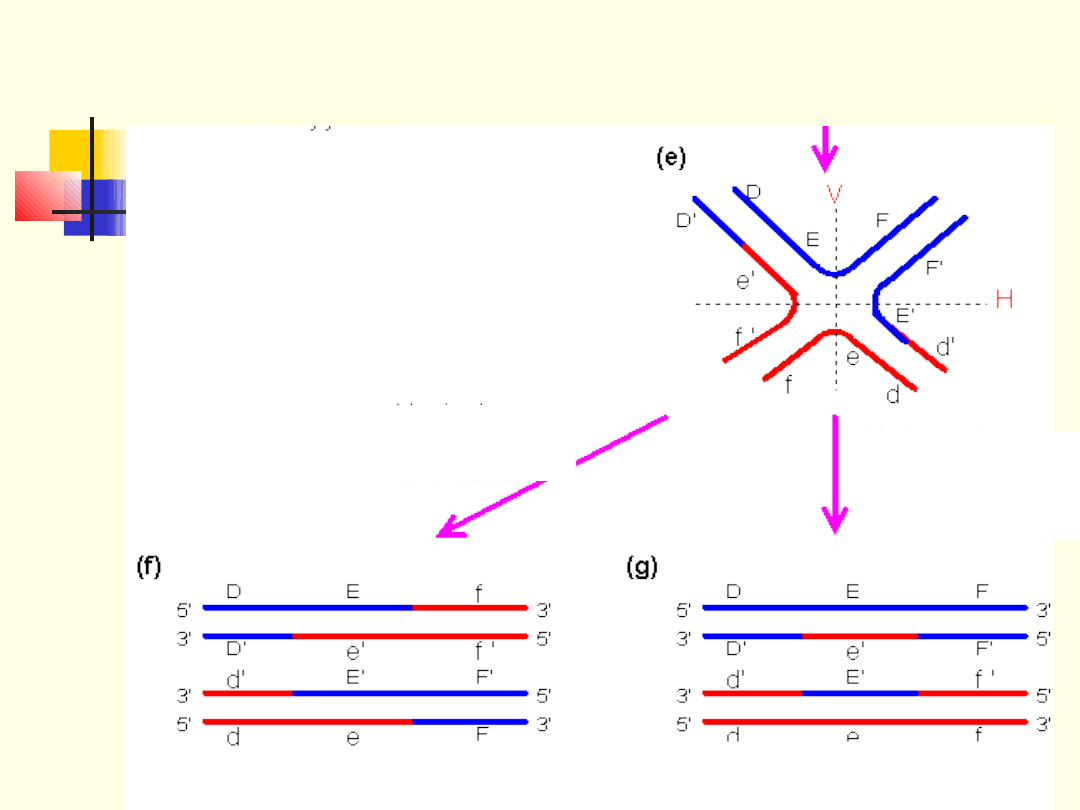
Mechanizm rekombinacji homologicznej
W zależności od tego jak zostaną
rozcięte struktury Hollidaya
powstaną rekombinanty lub nie
powstaną
Rozcięcie wzdłuż linii
V i religacja
Horyzontalne
cięcie i
religacja
Heterodupleks i rekombinanty
Heterodupleks i brak
rekombinantów

Białka w rekombinacji homologicznej
RecA
– ATP-aza promuje parowanie zasad między ssDNA i dsDNA,
odpowiada za tzw. asymilację
lub pobranie ssDNA
RecBCD
– egzonukleaza degradująca DNA (tzw. egzonukleaza V),
helikaza w obecności białka SSB
RuvA
–rozpoznaje strukturę Hollidaya i pomaga helikazie RuvB w
przeprowadzaniu procesu migracji nici DNA
RuvB
- ATP-aza dostarcza energii do poruszania się pasm, tworzy
heksamerowy pierścień naokoło dupleksu, łączy dsDNA (nie ssDNA)
RuvC
– endonukleaza, rozpoznaje i przecina strukturę Hollidaya.
Rozcina dwa z czterech pasm, w różnych płaszczyznach
Topoizomerazy
I i II
Document Outline
Wyszukiwarka
Podobne podstrony:
Culver Dart RCM 10 77
77 Nw 10 Radiotelefon przenosny
77 Nw 10 Lampa stojaca
10 1995 77 78
10 1996 75 77
77 Nw 10 Radiotelefon przenosny
10 1996 75 77
77 Nw 10 Lampa stojaca
10 1995 77 78
10 1995 77 78
10 Metody otrzymywania zwierzat transgenicznychid 10950 ppt
10 dźwigniaid 10541 ppt
wyklad 10 MNE
więcej podobnych podstron