1
Biochemia
Transkrypcja i translacja
Termostabilna
polimeraza
Tag z
Thermus
aquaticus
PCR (
p
olymerase
c
hain
r
eaction)
Etapy:
1.Denaturacja (95ºC) matrycowego,
badanego DNA, każda z
pojedynczych nici DNA staje się
matrycą do syntezy nici
komplementarnej
2.Wiązanie starterów (55ºC) startery
wiążą się na zasadzie
komplementarności z końcowymi
fragmentami nici matrycowych.
3.Polimeryzacja nukleotydów (72ºC,
polimeraza Tag), do starterów
dobudowywany jest
komplementarny łańcuch
polinukleotydowy.
4.Reakcja PCR obejmuje 25-30
takich cykli.
5.Zastosowanie: wykrywanie
mutacji, wykrywanie infekcji
wirusowych, identyfikacja osób.
2
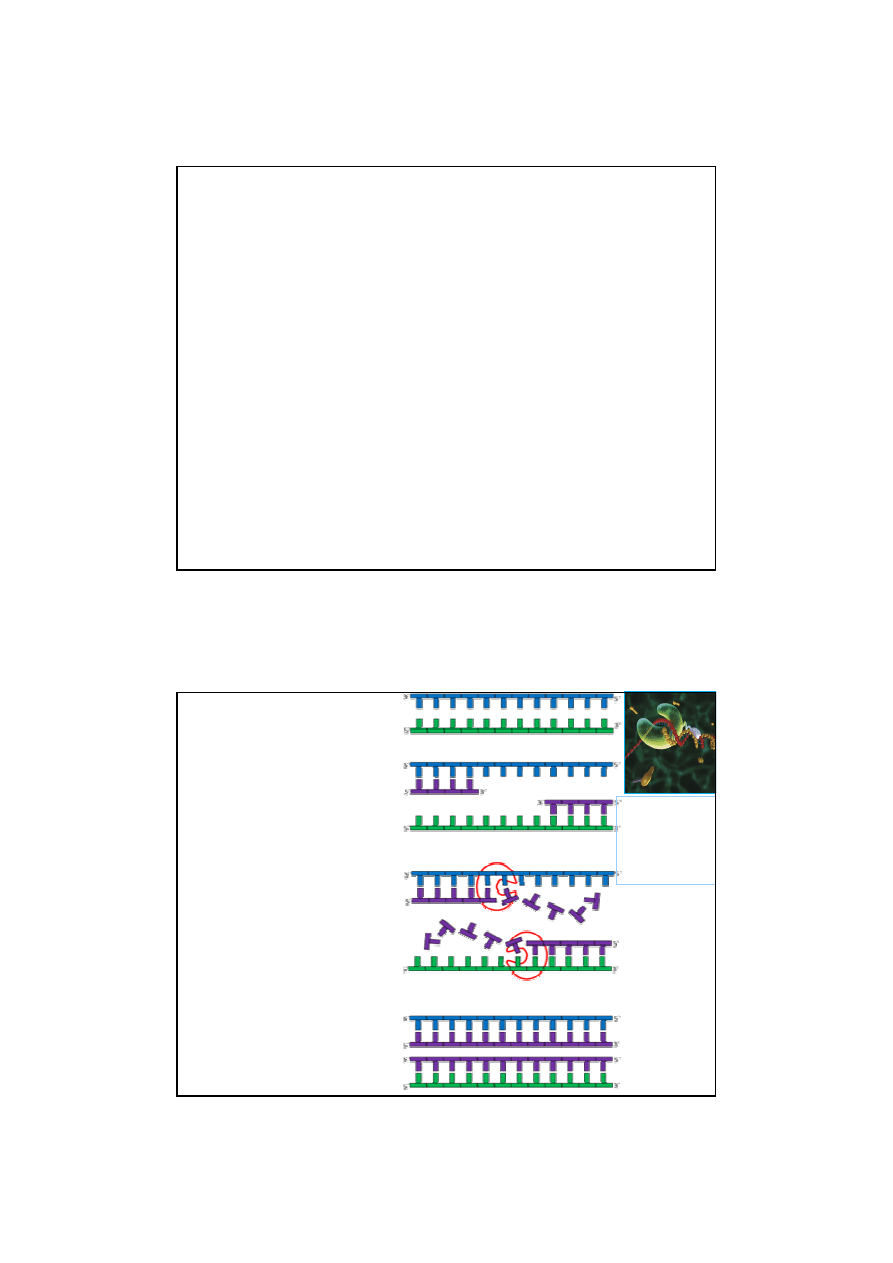
RNA ma sekwencję komplementarną do nici matrycowej i taką
samą jak sekwencja nici kodującej (w RNA zamiast T występuje
U).
Transkrypcja
-
to proces syntezy każdego typu RNA.
Polega na przepisywaniu informacji genetycznej
zawartej w sekwencji nukleotydów budujących DNA
(gen), na sekwencję nukleotydów wchodzących w skład
RNA.
GEN: odcinek DNA odpowiedzialny za wytwarzanie produktu tj. białka, rRNA,
tRNA, hnRNA, mt RNA
kolor niebieski – nić DNA kodująca (sensowna, (+))
kolor czerwony – nić DNA matrycowa (antysensowna, (-)
5'- A A T C G G C A T G C C A T G G C C T T G C G C T A - 3'
-
)
3’ T T A G C C G T A C G G T A C C G G A A C G C G A T - 5'
5'-A A U C G G C A U G C C A U G G C C U U G C G CUA - 3'
pre-mRNA
(
+
)
(
POLIMERAZY RNA
• Organizmy prokariotyczne
• Polimeraza RNA bakteryjna
składa się z czterech typów
podjednostek:
2 podjednostek α,
rdzeń
holoenzymu
podjednostek ß,ß’
podjednostek σ oraz ω.
α
- wiąże białka regulatorowe;
ß – syntetyzuje wiązania
fosfodiestrowe;
ß’ – wiążę matrycę DNA;
σ
- rozpoznaje miejsce
promotorowe oraz inicjuje
•
Organizmy eukariotyczne
1.
Polimeraza RNA I
–
zlokalizowana w jąderku,
syntetyzuje różne rodzaje
rRNA, z wyjątkiem 5S rRNA;
2.
Polimeraza RNA II
zlokalizowana w
nukleoplazmie syntetyzuje
mRNA oraz snRNA (małe
jądrowe RNA);
3.
Polimeraza RNA III
występuje
w nukleoplazmie syntetyzuje
tRNA, 5SrRNA oraz 7SRNA.
3
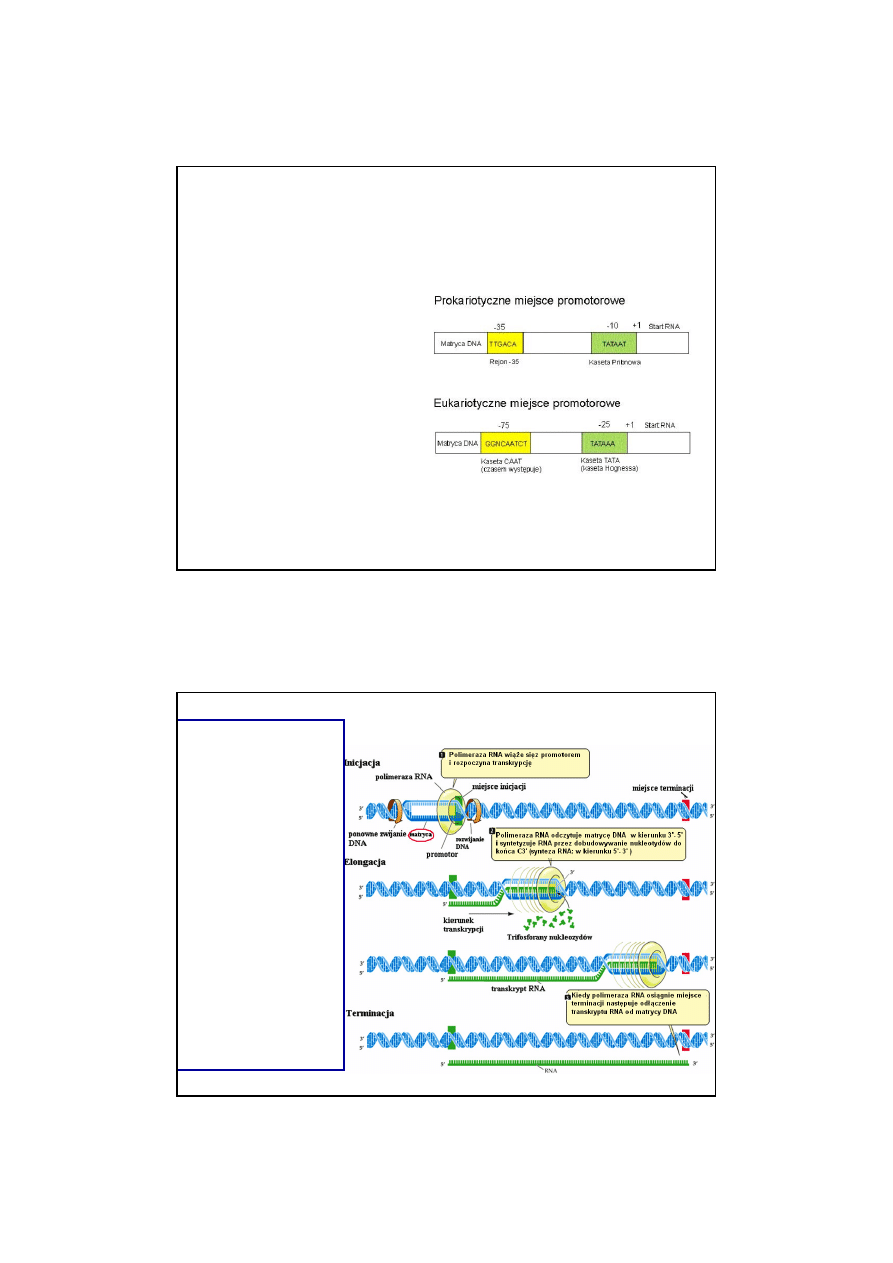
PROMOTOR
Y
1.
Promotor
jest to specyficzna
sekwencja nukleotydów w
DNA, do której przyłącza się
polimeraza RNA, aby
rozpocząć transkrypcję.
Promotor jest położony po
stronie 5’ od miejsca
początku transkrypcji i
obejmuje 40 – 60 pz,
2.
W obrębie
promotora
prokariotycznego
znajduje się
sekwencja zwana kasetą
Pribnowa ( TATAAT) i inny
element o sekwencji
TTGACA.
3.
Promotor eukariotyczny
dla
polimerazy RNA II zawiera
TRANSKRYPCJA U
BAKTERII
Transkrypcja u
eukariotów:
1. Inicjacja transkrypcji
w miejscu
promotora.
2. Transkrypcja
odbywa się na
jednej nici –
matrycowej.
3. W inicjacji
transkrypcji
uczestniczą
białkowe czynniki
transkrypcyjne (TF).
4. Synteza RNA
przebiega w
kierunku 5’→ 3’.
4
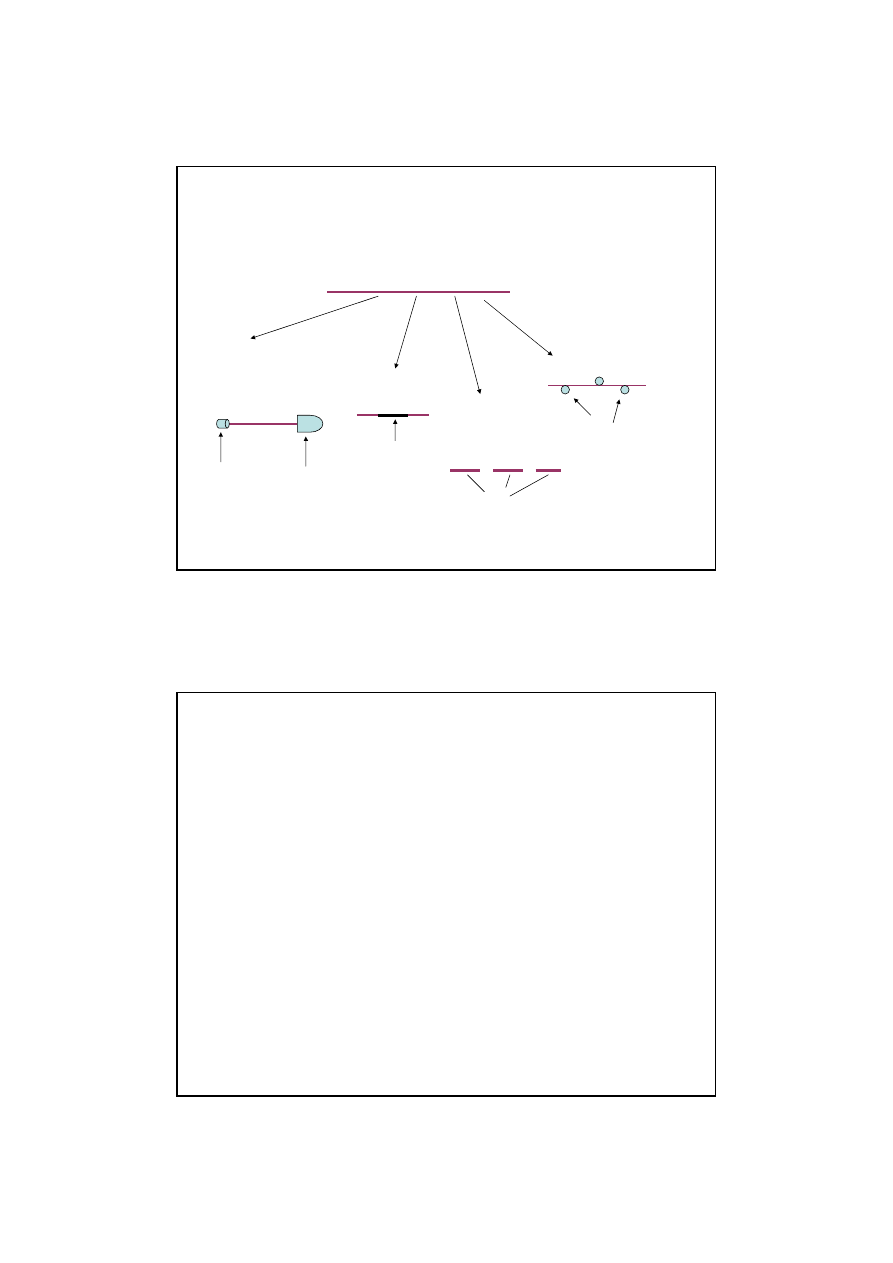
DOJRZEWANIE pre - RNA
pre – RNA ( pierwotny
transkrypt)
Modyfikacja
końców
(eukariota)
5’
3’
czapeczka
ogon poli A
Wycinanie intronów
(eukariota)
intron
dojrzałe rRNA i tRNA
Cięcie
cząsteczek
prekursorowych
(pro- i eukariota)
Modyfikacja
chemiczna ( pro- i
eukariota
)
Nowe grupy
chemiczne
KOD GENETYCZNY
1.
Kod genetyczny
to zestaw reguł określających współzależność
trójek nukleotydów (kodonów) w DNA lub mRNA z sekwencją
aminokwasów w białku.
2.
Kod genetyczny jest
tójkowy
- sekwencja nukleotydów w mRNA
jest odczytywana trójkami. Trójka nukleotydów nosi nazwę
KODONU
.
3.
Kod genetyczny jest
jednoznaczny
, prawie każda trójka
nukleotydów określa jakiś aminokwas. AUG – kodon
INICJUJĄCY(STARTOWY). Kodony UAG, UGA i UAA nie kodują
informacji, nazywane są kodonami STOP lub TERMINACYJNYMI.
4.
Kod genetyczny jest
zdegenerowany
. 64 kodony określają 20
aminokwasów, a więc jeden aminokwas jest kodowany przez kilka
różnych kodonów.
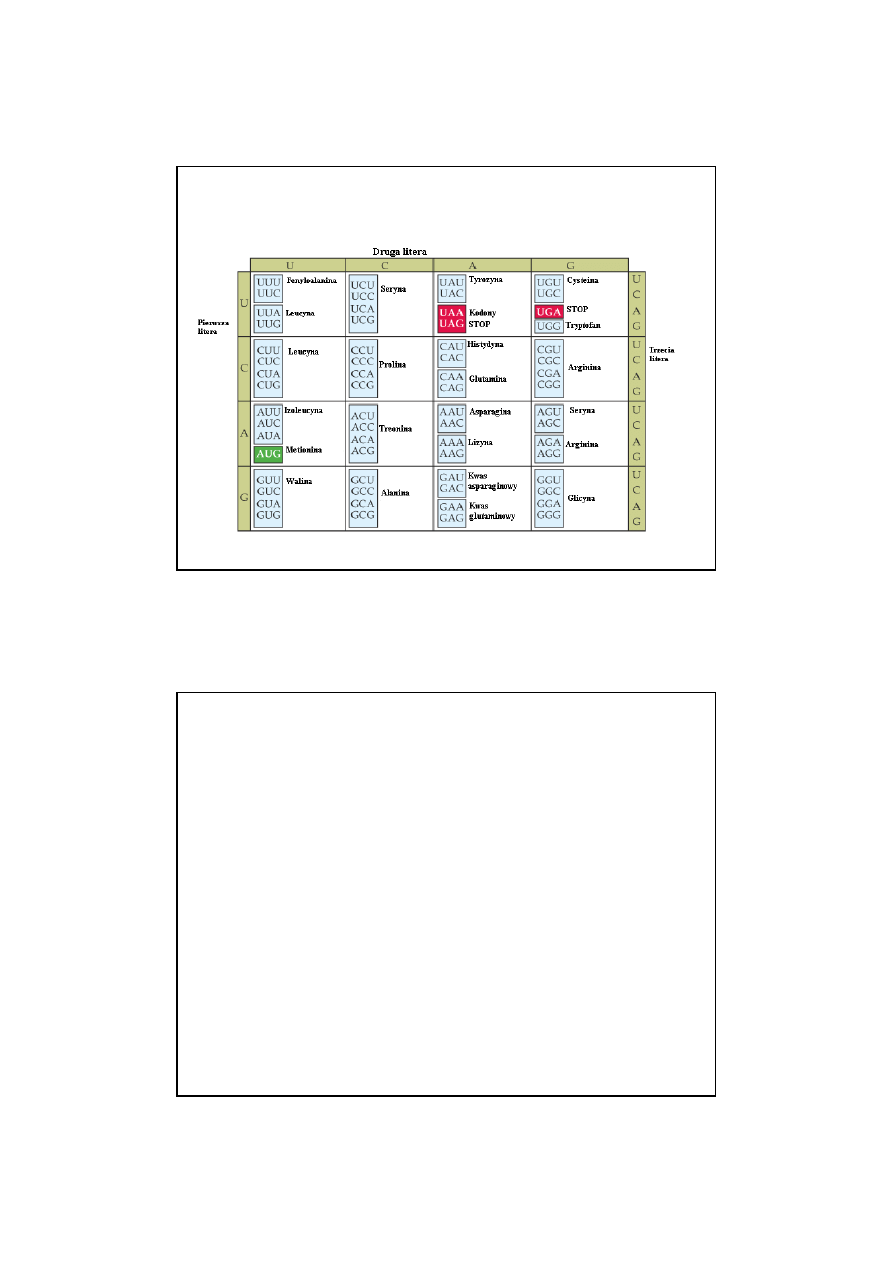
5
KOD GENETYCZNY
TRANSLACJA
1.
Translacja
jest to wieloetapowy proces enzymatyczny, w
którym sekwencja nukleotydów w cząsteczce mRNA kieruje
wbudowywaniem aminokwasów w białka, zachodzi na
rybosomach.
2.
Etapy translacji:
inicjacja
, podczas której powstaje kompleks mRNA-rybosom i
kodon inicjatorowy wiąże aminoacylo-tRNA,
elongacja
, podczas tego etapu odczytywane są kolejne
kodony, a łańcuch polipeptydowy rośnie przez dołącznie
aminokwasów do jego końca C.
terminacja
następuje gdy rybosom osiągnie kodon stop, dla
którego nie ma odpowiedniego aminoacylo-tRNA.
3.
W procesie translacji, na każdym etapie,
uczestniczą czynniki
białkowe
. Są to czynniki inicjujące (IF), czynniki elongacyjne
(EF), czynniki uwalniające (RF).

6
STRUKTURA RYBOSOMU PROKARIOTYCZNEGO
I EUKARIOTYCZNEGO
Rybosom prokariota- 70S-
zbudowany
jest z podjednostki dużej 50S i małej 30S.
W skład podjednostki 50S wchodzą:
23SrRNA, 5SrRNA oraz 34 białka.
W skład podjednostki 30S
wchodzą:16SrRNA i 21 białek.
Rybosom eukariota- 80S-
zbudowany jest z
podjednostki dużej 60S i małej 40S.
W skład podjednostki 60S wchodzą 28S rRNA,
5,8S rRNA, 5S rRNA oraz 49 białek.
W skład podjednostki 40S wchodzą 18S rRNA i
33 polipeptydy.
FORMOWANIE AMINOACYLO-tRNA
Cząsteczki adaptorowe zaangażowane w translację kodu genetycznego to:
syntetaza aminoacylo-tRNA i tRNA.
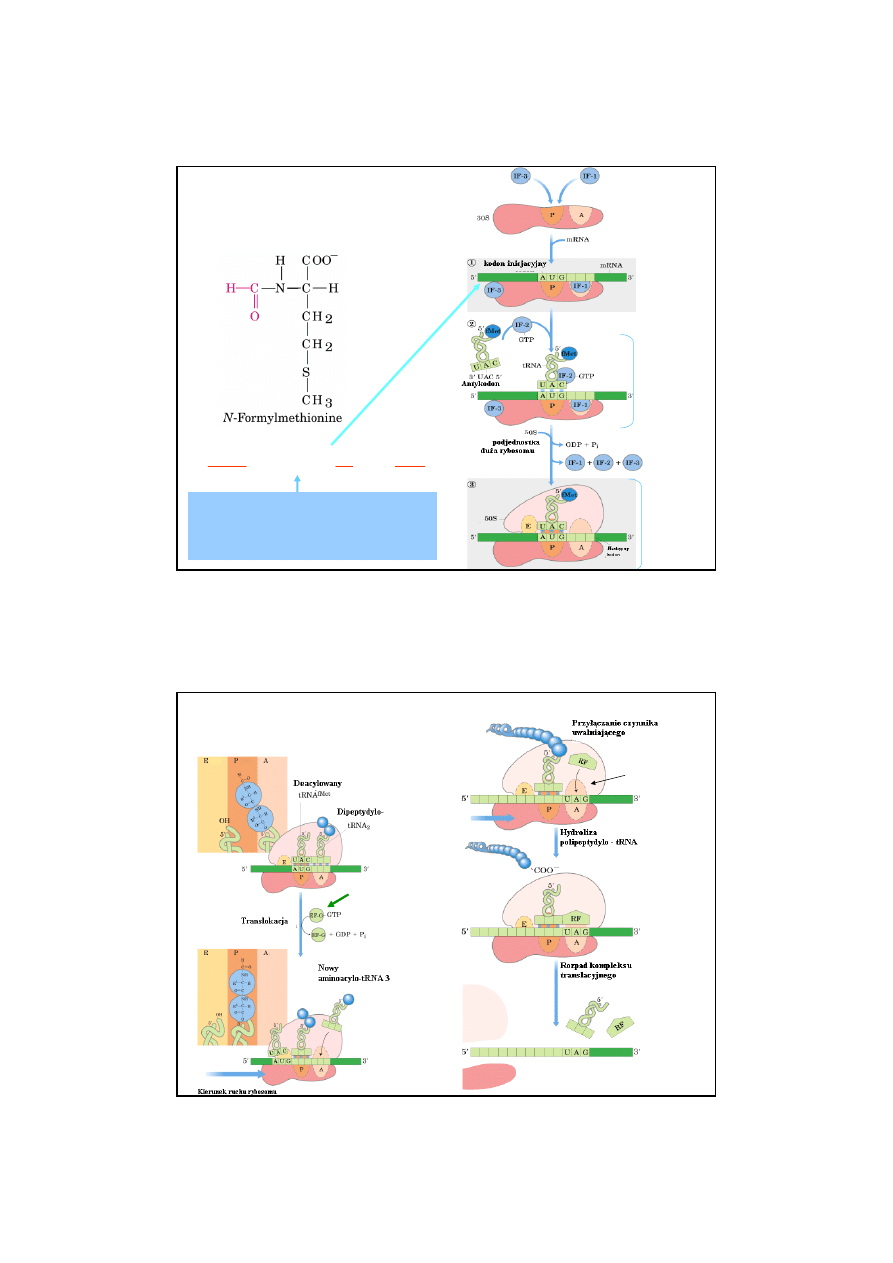
7
TRANSLACJA U
PROKARIOTA
INICJACJA
5’
AGGAGGU
AUG 3’
Sekwencja Shine-Dalgarno w mRNA
komplementarna do sekwencji końca
3’ w 16S rRNA umożliwia ustawienie
kodonu UAG w miejscu P .
Kompleks
inicjujący
30S
Kompleks
inicjujący
70S
TRANSLACJA U PROKARIOTA
ELONGACJA
TERMINACJA
RF3 +GTP
EF-G -
translokaza
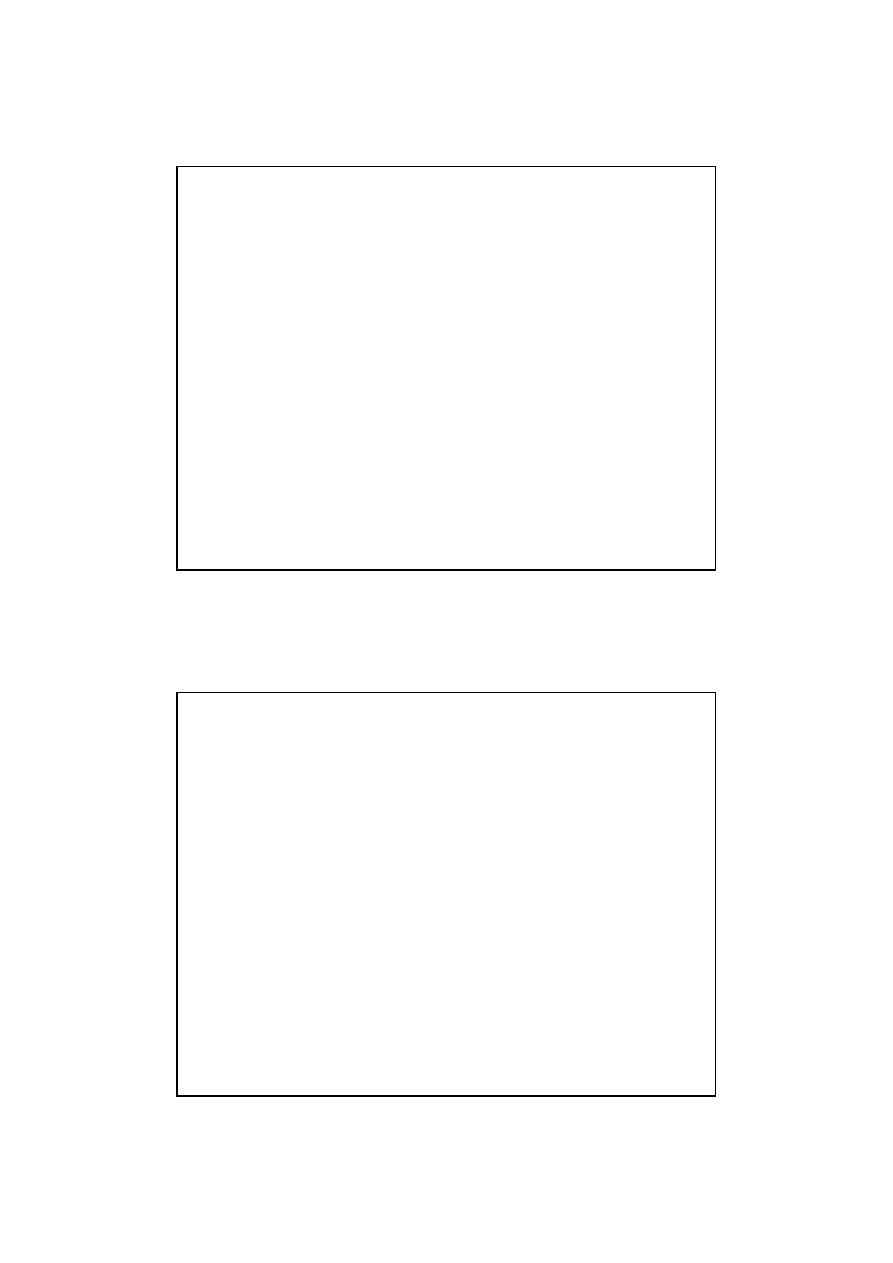
8
Translacja u eukariotów
• Każdy mRNA koduje jedno białko.
• Syntezę białka rozpoczyna Met-tRNA
i
Met
.
• Podjednostka 40S wiąże się z końcem 5’ mRNA i porusza się w
kierunku 3’ aż napotka kodon inicjujący.
• Składanie kompleksu preinicjującego: 40S + Met-tRNA
i
Met
+ eIF2 +
GTP.
• Wiązanie kompleksu preinicjującego z eIF3 i czpeczką na końcu
5’mRNA oraz białek wiążących z poli A na końcu 3’.
• Skanowanie mRNA aż zostanie zlokalizowany kodon AUG.
• Przyłączenie 70S i utworzenie kompleksu inicjującego 80S.
• Elongacja podobnie jak u prokariotów, uczestniczą eEF1α, EFIβγ i
eEF2
• U eukariotow deacylowany tRNA jest usuwany z miejsca P (brakuje
miejsca E na kompleksie translacyjnym).
• Terminacja – z udziałem czynnika uwalniającego eRF
współdziałającego z ATP.
Translacja u eukariotów
• Każdy mRNA koduje jedno białko.
• Syntezę białka rozpoczyna Met-tRNA
i
Met
.
• Podjednostka 40S wiąże się z końcem 5’ mRNA i porusza się w
kierunku 3’ aż napotka kodon inicjujący.
• Składanie kompleksu preinicjującego: 40S + Met-tRNA
i
Met
+ eIF2 +
GTP.
• Wiązanie kompleksu preinicjującego z eIF3 i czpeczką na końcu
5’mRNA oraz białek wiążących z poli A na końcu 3’.
• Skanowanie mRNA aż zostanie zlokalizowany kodon AUG.
• Przyłączenie 70S i utworzenie kompleksu inicjującego 80S.
• Elongacja podobnie jak u prokariotów, uczestniczą eEF1α, EFIβγ i
eEF2
• U eukariotow deacylowany tRNA jest usuwany z miejsca P (brakuje
miejsca E na kompleksie translacyjnym).
• Terminacja – z udziałem czynnika uwalniającego eRF
współdziałającego z ATP.
9
Energetyka biosyntezy białka
Proces
Reakcja
Ilość rozłożonych
wiązań
fosforanowych
1. Selekcja
i aktywacja
aminokwasu
Aminokwas + tRNA -> aminoacylo – tRNA
ATP -> AMP + PP
i
PP
i
+ H
2
O -> 2P
i
1
1
2. Wprowadzenie
aminoacylo-
tRNA do
rybosomu w
miejsce A
GTP + H
2
O -> GDP + P
i
1
3. Translokacja
GTP + H
2
O -> GDP + P
i
1
W sumie
4
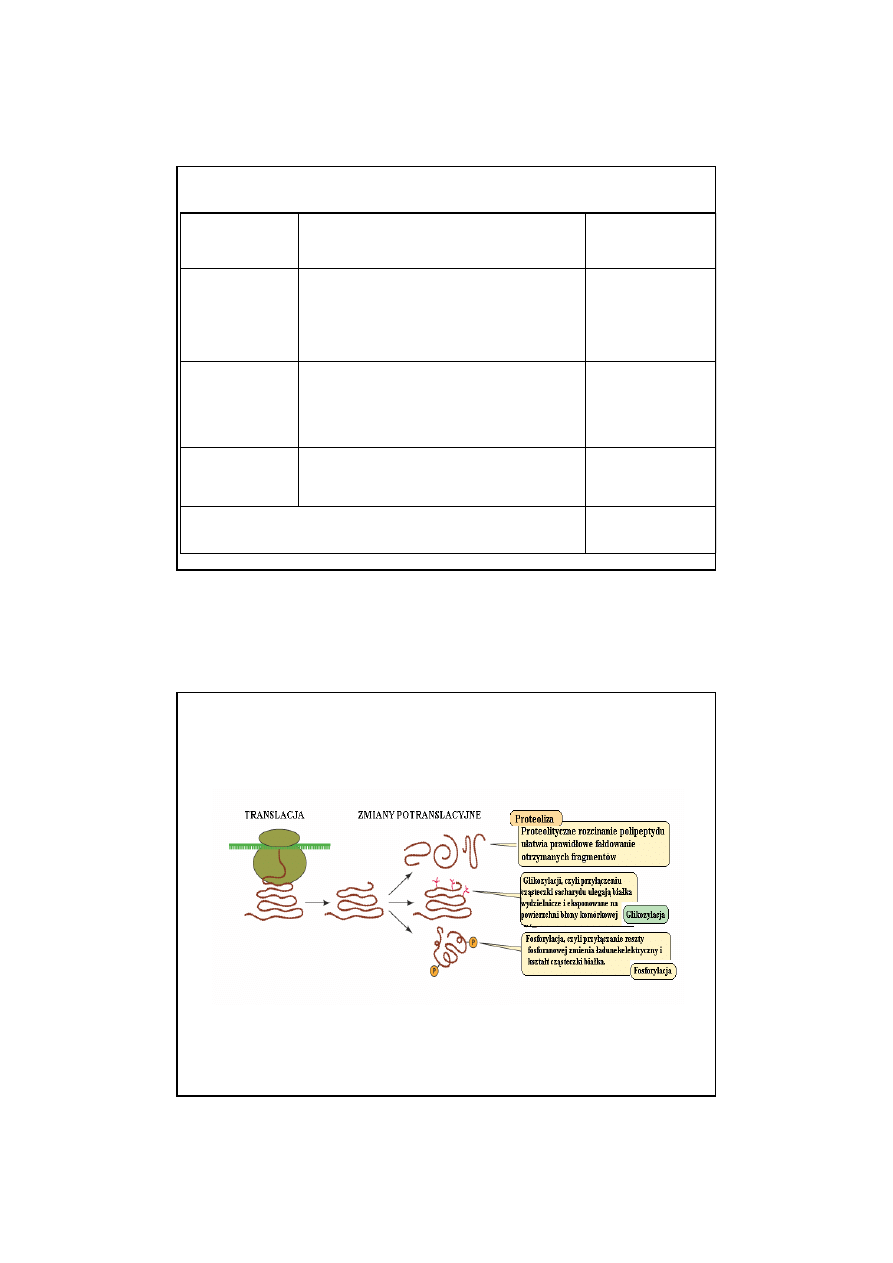
MODYFIKACJE POTRANSLACYJNE BIAŁEK
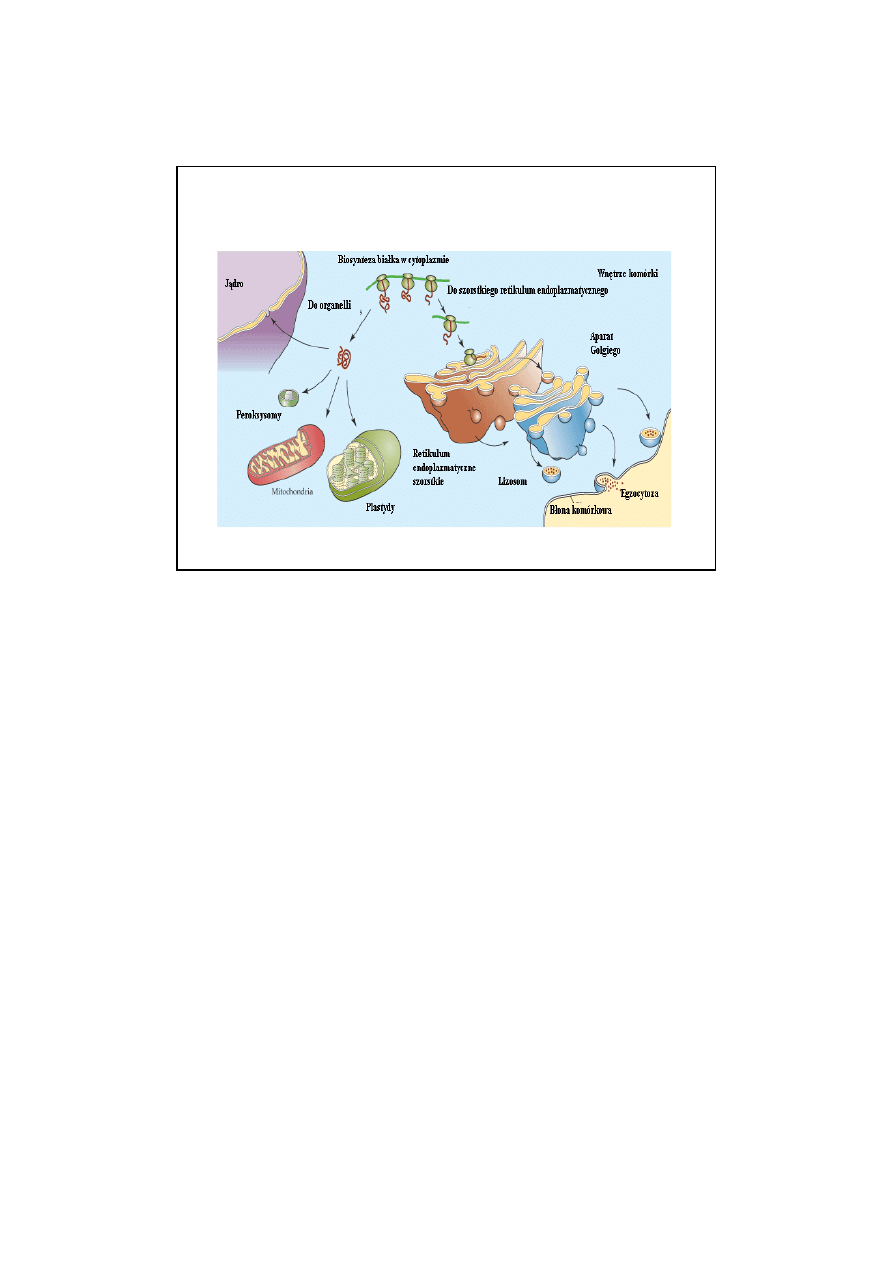
10
KIEROWANIE BIAŁEK
Wyszukiwarka
Podobne podstrony:
Ekspresja informacji genetycznej-transkrypcja i translacja, NAUKA
Procesy transkrypcji i translacji
05) Transkrypcja i translacja (wykład 5)
03 REPLIKACJA TRANSKRYPCJA TRANSLACJA
Porównanie transkrypcji i translacji u Prokaryota i Eukaryota 10
transkrypcja i translacja
Charakterystyka biosyntezy białek Transkrypcja,Translacja
Replikacja, transkrypcja, translacja (wersja dla studentów)
BIOLOGIA replikacja transkrypcja i translacja, AWF Wychowanie fizyczne, biologia, 1
Transkrypcja i translacja
Ekspresja informacji genetycznej-transkrypcja i translacja, NAUKA
03 REPLIKACJA TRANSKRYPCJA TRANSLACJA
Transkrypcja i transliteracja tekstow tatarow
kwasy nukleinowe replikacja transkrypcja translacja jak napisac
transkrypcja, Biochemia, Pomoce;)
BIOCHEMIA Przebieg procesu translacji
translacja, Biochemia, Pomoce;)
więcej podobnych podstron