8. ROLA CHROMATYNY W REGULACJI EKSPRESJI GENOW.
Kazda z komorek zywego organizmu zawiera z nielicznymi wyjatkami ten sam zestaw genow. Z drugiej strony wiadomo, ze organizmy skladaja sie niekiedy z setek roznych typow komorek, z ktorych kazda ma inna role do spelnienia. Strukturalna i funkcjonalna roznorodnosc komorek, jak rowniez roznorodnosc reakcji na bodzce srodowiskowe i fizjologiczne osiagnieta zostaje przez kontrolowana ekspresje genow zalezna od natury komorki, etapu cyklu komorkowego czy cyklu rozwojowego. Ryc. 8-1 przedstawia schematycznie rozne etapy procesu ekspresji genow w szerokim znaczeniu tego slowa, od aktywacji regulatorow transkrypcji poczawszy na syntezie funkcjonalnego bialka skonczywszy. W regulacji tej kluczowa role w komorkach eukariotow, odgrywa chromatyna a w szczegolnosci stopien jej kondensacji.
Ryc 8-1. Zintegrowany system regulacji ekspresji genow kodujacych bialka u eukariotow.
Regulatory transkrypcji. Poziom ekspresji wiekszosci genow jest regulowany przez wielką ilość czynników transkrypcyjnych zwanych regulatorami (aktywatorami i represorami), ktore wiaza sie z sekwencjami regulatorowymi w DNA znajdujacymi sie po stronie 5' miejsca w ktorym zaczyna sie transkrypcja. Różnią się one od czynników transkrypcyjnych bazowych - general transcription factors, które tworzą kompleks preinicjacyjny, którego składnikiem jest również polimeraza RNA II (patrz niżej). Ocenia sie, ze okolo 5% naszych genow koduje czynniki transkrypcyjne, co daje miare wagi tych czynnikow dla poprawnego funkcjonowania komorek. Ich aktywnosc jest z kolei regulowana przez szereg roznych sciezek regulacyjnych. I tak na przyklad czynniki transkrypcyjne, ktore reguluja ekspresje genow waznych dla cyklu komorkowego sa regulowane przez elementy sygnalizacyjne tego cyklu a czynniki, ktore moduluja enzymy metabolizmu sa czesto regulowane przez metabolity. Do elementów regulatorowych należą również sekwencje położone niekiedy w znacznej (tysiące par zasad po stronie 3' lub 5') odległości od starty transkrypcji. Nazywa się je, w zależności od funkcji) wzmacniaczami lub wyciszaczami.
Sekwencje regulatorowe wiekszosci genow eukariotow zawieraja miejsca wiazania licznych czynnikow transkrypcyjnych zapewniajacych to, ze kazdy gen moze reagowac w ramach rozmaitych sciezek sygnalizacyjnych oraz ulatwiajacych precyzyjna regulacje poziomu transkrypcji. Naogol aktywnosc czynnikow transkrypcyjnych zalezy od kontekstu tzn. moze byc modulowana przez inne bialka tworzace kompleksy z sekwencjami DNA znajdujacymi sie w poblizu. Jeden i ten sam czynnik transkrypcyjny moze indukowac transkrypcje jednego genu i wygaszac transkrypcje innego. Taka regulacja transkrypcji zalezna od kontekstu jest nazywana "kombinatoryjna". Pozwala ona reagowac komorkom eukariotycznym na zaskakujaco duza ilosc roznych bodzcow przy pomocy stosunkowo niewielkiej ilosci czynnikow transkrypcyjnych. Kontrola transkrypcji jest prostsza u prokariotow, gdzie geny jednej sciezki metabolicznej sa wspolregulowane w ramach jednej jednostki transkrypcyjnej (operonu).
Regulacja regulatorow. Regulatory transkrypcji same podlegaja roznym systemom regulacji. Jednym z podstawowych systemow jest transport pomiedzy cytoplazma a jadrem. Zachodzi on przy udziale porow jadrowych, ktore kontroluja przemieszczanie sie makromolekul. Czynniki transportujace rozrozniaja krotkie motywy aminokwasowe znajdujace sie w bialkach, ktore podlegaja transportowi przez pory. Rozroznia sie dwa typy motywow transportujacych: eksportowy, zidentyfikowany w bialkach transportowanych z jadra (nuclear export signal, NES) oraz importowy (nuclear localisation signal, NLS). Tego typu system zezwala na stosunkowo prosta kontrole wedrowki bialek. Maskowanie NES i NLS lub ich modyfikacja uniemozliwia rozpoznanie motywow przez systemy transportujace i tym samym uniemozliwia transport.
Modyfikacja czynnikow transkrypcyjnych jest niekiedy powodowana przez czasteczki sygnalizujace niezdolne do wnikniecia do komorki. Jest to sciezka regulacyjna zwana transdukcja sygnalu. Czasteczka sygnalizujaca (czesto polipeptyd) wiaze sie z receptorem obecnym na powierzchni komorki, co powoduje zmiane w konformacji tej jego czesci, ktora zlokalizowana jest wewnatrz komorki. Zmiana ta indukuje reakcje biochemiczna w komorce (na przyklad fosforylacje), ktora to reakcja aktywuje czynnik transkrypcyjny. Transdukcja sygnalu jest metoda stosowana przez komorki np. w przypadku stymulacji transkrypcji przez interferon. Niektore hormony (np glukokortykoidy) moga wnikac do wnetrza komorki i tam lacza sie z receptorami, sa transportowane do jadra gdzie kompleks hormonu z receptorem dziala jako czynnik transkrypcyjny.
Aby moc odpowiedziec szybko na zewnetrzny lub wewnetrzny bodziec wymagajacy zmiany w ekspresji genow komorka musi byc w stanie nie tylko szybko aktywowac jeden lub wiecej aktywatorow ale musi rowniez moc szybko inaktywowac inne aktywatory transkrypcyjne. Jedną z metod stosowanych w tym celu przez komorki jest system oparty na ubikwitynacji i degradacji proteolitycznej bialek, ktory jest czynny w procesie poczatkowego uaktywniania i nastepujacej w dalszym etapie degradacji czynnikow transkrypcyjnych. I tak np. stwierdzono, ze w drozdzach ubikwitynacja zwieksza aktywnosc domeny aktywujacej transkrypcje (TAD, transcription activation domain) w czynniku transkrypcyjnym VP16 wirusa herpes simplex ale rowniez "naznacza" ja w celu degradacji. Podobnie w komorkach ssakow aktywnosc proteosomu 26 S (zaangazowanego w proteolize) jest niezbedna zarowno dla uzyskania aktywnosci przez receptor estrogenu jak i dla jego pozniejszej degradacji zaleznej z kolei od obecnosci ligandu. Nastepujace po sobie aktywacja i degradacja czynnikow transkrypcyjnych zwiazanych z promotorami umozliwia szybkie dostosowanie aktywnosci genu do potrzeb komorki w zmieniajacym sie srodowisku. Jesli sygnalizowana jest koniecznosc kontynuowania transkrypcji, nowo aktywowany czynnik transkrypcyjny zastepuje ten, ktory ulegl degradacji. Natomiast, jesli sygnal kontynuowania transkrypcji zanika, czynnik zdegradowany nie jest zastepowany innym i transkrypcja ustaje. Taki model dobrze tlumaczy mechanizm dzialania glukokortykoidow u ssakow.
Ubikwityna odgrywa role i w innych fazach transkrypcji. Duza podjednostka polimerazy II RNA (RNAP II) jest ubikwitynowana podczas transkrypcji. Ubikwitynacja jest rowniez indukowana przez UV i przez uszkodzenia w DNA powodowane przez cisplatyne.
Poza ubikwitynacja szereg innych modyfykacji post-translacyjnych odgrywa wazna role w regulacji aktywnosci czynnikow transkrypcyjnych. Najlepiej zbadana z nich jest fosforylacja, co wynika z latwosci z jaka mozna ja badac. Jednak czynniki transkrypcyjne sa podatne i na inne modyfikacje takie jak acetylacja lizyny czy metylacja lizyny lub argininy.
Dwie formy wlokna chromatynowego. Dostepnosc DNA dla bialek w regionach transkrypcyjnie kompetentnych chromatyny, i jego niedostepnosc w regionach transkrypcyjnie niekompetentnych, zostaly wykazane w doswiadczeniu w ktorym traktowano chromatyne enzymami nukleolitycznymi. Okazalo sie, ze odcinki DNA odpowiadajace transkrybowanym genom oraz regionom chromosomu bezposrednio do nich przylegajacym (o dlugosci kilku do kilkudziesieciu kb) ulegaja trawieniu. Natomiast chromatyna w odcinkach chromosomu polozonych poza tym regionem istnieje w formie utrudniajacej dostep enzymow nukleolitycznych do DNA (Ryc 8-2).
Ryc. 8-2. Wrazliwosc wlokna chromatynowego na trawienie DNaza I.
Nietranskrybowane regiony chromosomow zorganizowane sa w chromatyne zbita (heterochromatyne) podczas gdy geny ulegajace transkrypcji organizuja sie w chromatyne rozproszona, (euchromatyne). Kazdy typ komorek ma wlasny jednostkowy uklad hetero- i euchromatyny i ten uklad dziedziczy sie z jednego pokolenia komorek na nastepne. Zmany struktury chromatyny odpowiadaja wiec za epigenetyczne zmiany fenotypu komorek czy organizmow, a wiec za takie zmiany, ktorym nie wywolane sa roznicami w sekwencji DNA.
Heterochromatyna od euchromatyny rozni sie konformacja DNA w nukleosomie, obecnoscia zmodyfikowanych histonow i niekiedy metylowanego DNA oraz skladem bialek niehistonowych. Stosunkowo dobrze poznano heterochromatyne drozdzy w regionie telomerow oraz w nieaktywnych transkrypcyjnie lokusach MAT (mating) zawierajacych informacje zwiazana z przelaczaniem typu koniugacyjnego (Ryc 8-3). Podstawowa role w procesie tworzenia heterochromatyny w chromosomach drozdzy odgrywaja bialka Sir3 i Sir4 (silent information regulator), ktore oddzialywuja z ogonami histonow H3 i H4 i tworzac zwarty kompleks makromolekularny chroniacy DNA przed oddzialywaniem z innymi bialkami. W heterochromatynie organizmow wyzszych natomiast znaleziono m.in. bialko HP1 (heterochromatin protein 1) pelniace podobna role jak bialka Sir.
Ryc. 8-3 Struktura heterochromatyny w drozdzach. A. heterochromatyna telomeru, B. Heterochromatyna w regionie MAT.
W euchromatynie zawierajacej sekwencje kodujace DNA, ktora jest w przyblizeniu dziesieciokrotnie bardziej wrazliwa od heterochromatyny na enzymy nukleolityczne, wyroznia sie dodatkowo regiony o jeszcze wyzszej (o kolejne dziesiec razy) wrazliwosci na nukleazy.
Regiony te o dlugosci kilkuset par zasad nazwane zostaly miejscami hiperczulymi (HS, hypersensitive sites) (Ryc. 8-4). W tych wlasnie miejscach DNA wiaze sie z bialkami regulatorowymi rozpoznajacymi specyficzne sekwencje nukleotydowe. Powstajace w wyniku takich specyficznych oddzialywan kompleksy odpowiedzialne sa za regulacje aktywnosci transkrypcyjnej sasiadujacych genow. Tak wiec obecnosc HS sygnalizuje naogol obecnosc promotora lub wzmacniacza. Niekiedy obserwuje sie kilka miejsc HS polozonych w bliskim sasiedztwie. Moze byc to oznaka, ze region ten jest regionem kontroli lokusu LCR, (locus control region) odpowiedzialnym za kontrole ekspresji jednego lub kilku sasiadujacych genow, tak jak ma to miejsce w przypadku genow betaglobiny czlowieka. (Ryc. 8-5).
Ryc. 8-5. Miejsca hiperczule w LCR czlowieka.
Struktura chromatyny regionu promotorowego. Do aktywacji transkrypcji nie wystarcza samo utworzenie kompleksu czynników transkrypcyjnych z DNA. Obecnie wiemy, że jest ona wynikiem współdziałania kilku typów białek i kompleksów białkowych.
(1) Pierwszym warunkiem utworzenia kompleksu inicjującego transkrypcje jest dostępność regionu promotorowego nici chromatynowej dla białek. Przyczynia się do tego luźna struktura chromatyny w tych regionach, która charakteryzuje się niestabilnymi nukleosomami będącymi w stanie równowagi dynamicznej z wolnymi histonami. Dzięki temu możliwe jest rozpoznanie sekwencji regulatorowych DNA przez regulatory, utworzenie kompleksu DNA - regulator i rekrutację innych białek pomocniczych.
(2) Wyodrębniono i scharakteryzowano dużą ilość regulatorów czyli czynników transkrypcyjnych, które tworzą kompleksy ze specyficznymi sekwencjami DNA w regionie promotora. Ze względu na funkcję wprowadzono podział regulatorów na aktywatory i represory.
(3) Regulatory rekrutują białka zwane koregulatorami, które nie rozpoznają sekwencji DNA lecz mają charakter enzymów. Ze względu na skutek ich działania dzielą się podobnie jak regulatory na koaktywatory i korepresory. Ze względu na typ aktywności enzymatycznej dzielimy je na dwie grupy. Do pierwszej zaliczamy enzymy modyfikujące histony z których najlepiej poznane to metylotransferazy histonów (HMT), demetylazy histonów, acetylotransferazy histonów (HAT) i deaceacetylazy histonów (HDAC), oraz kinazy histonów. Druga grupa koaktywatorów to kompleksy remodelujące chromatynę. Uzywając ATP jako źródła energii kompleksy te „otwierają” (rozluźniają) lub „zamykają” (powodują powstanie struktur zbitych) włókno chromatynowe. Koregulatory i regulatory niekiedy komunikują się ze sobą a także z elementami wzmacniaczy (enhancer) i wyciszaczy (silencer) za pośrednictwem dodatkowych składników kompleksu zwanych mediatorami.
(4) Transkrypcja genow kodujących białka dokonywana jest przez polimerazę RNA II (RNAPII), która współdziała z sześcioma czynnikami transkrypcyjnych bazowymi: TFIIA, B, D, E, F i H. Czynniki te są w zasadzie identyczne dla wszystkich genów, podczas gdy czynniki transkrypcyjne zaliczane do regulatorów wykazują się znaczną specyficzność - różne geny są regulowane przy pomocy różnych zestawów regulatorów.
Poniżej w skrócie omówiono w skrócie te cztery typy oddziaływań mających znaczenie modelujących włókno chromatynowe i w tem sposób partycypujące w procesie regulacji ekspresji genów.
Dynamika chromatyny regionu promotora. W ostatnich latach zdano sobie sprawę z tego, że dynamika chromatyny odgrywa istotną rolę w ekspresji genu. Stanem podstawowym chromatyny jest struktura zamknięta na oddziaływania z białkami w ogóle a w szczególności z białkami zaangażowanymi w ekspresję genu. Transkrypcja DNA jest możliwa dopiero po „otwarciu” (rozlużnieniu) tych struktur, co jest uwarunkowane utrzymywaniem w stanie gotowości krótkich odcinków nici chromatynowej noszących sekwencje regulatorowe (Ryc. 8-6). Komórka, w razie potrzeby, modyfikuje strukturę chromatyny rozpoczynając „otwieranie” struktur zamkniętych od tych właśnie regionów.
Ryc. 8-6 Pozycje nukleosomów w chromatynie niosącej dwa geny drożdżowe. Nukleosomy zaznaczone kolorem czerwonym wykazują wysoką dynamikę , zielone są stabilne. Strzałki pokazują regiony promotorowe.
W utzymywaniu w stanie gotowości regionów regulatorowych mają swój udział: (1) pozycjonowanie nukleosomów, (2) kontrola składu oktamerów, tak aby zawierały odpowiednie warianty histonów, (3) kontrola struktury nukleosomów przez czynniki remodelujące, (4) posttranslacyjne modyfikacje histonów i wreszcie (5) wiązanie w regionach regulatorowych odpowienich białek niehistonowych.
Ad (1) : Odcinki regulatorowe DNA są w dużej mierze pozbawione nukleosomów co jest przyczyną nadzwyczajnej wrażliwości na trawienie nukleazami segmentów nici chromatynowej, które tym odcinkom odpowiadają (miesca hiperczułe, HS). Brak nukleosomów w regionach regulatorowych jest spowodowany ich eliminacją, czemu sprzyjają różnice w powinowactwie różnych sekwencji nukleotydowych DNA do oktamerów histonów. Odcinki DNA zlokalizowane na skraju regionów regulatorowych wykazują wysokie powinowactwo do oktamerów, w odróżnieniu od odcinków położonych wewnątrz tych regionów. Tak więc sama sekwencja DNA sprzyja tworzeniu struktur otwartych chromatyny w tych kluczowych regionach chromatyny. Same różnice w powinowactwie oktamerów do DNA nie są jednak wystarczające do wyeliminowania nukleosomów.
Ryc. 8-7. Rola czynnika eskortującego Asf1 w procesach (a) składania oktameru i (b) jego dysocjacji.
Ad (2) : Do eliminowania nukleosomów z regionów regulatorowych przyczynia się też ich niestabilność spowodowana oddziaływaniem z białkami opiekuńczymi CAF1, Asf1, i HirA. Dwa pierwsze z nich biorą również udział w składaniu nukleosomów podczas replikacji DNA.
W procesie składania, po przejściu widełek replikacyjnych, czynnik CAF1 (chromatin assembly factor), który jest związany z klamrą PCNA (proliferating cell nuclear antigen), nakłada na nowosyntetyzowaną podwójną helisę DNA dwa heterodimery H3-H4. Uczestniczy w nim również białko eskortujące Asf1 (anti silencing factor). Uważa się, że jego rola polega na osłabieniu wzajemnych oddziaływań dimerów H3-H4, co zapobiega ich przedwczesnej agregacji (Ryc. 8-7a). Histony H2A i H2B są z kolei odkładane na tetramerze (H3-H4)2 przy użyciu białka opiekuńczego Nap1 (nucleosome assembly protein) w ten sposób, że heterodimery H2A-H2B flankują osadzony poprzednio tetramer. Taka budowa powoduje, że podczas rozpadu oktameru najłatwiej oddysocjowuje jeden z dimerów H2A-H2B pozostawiając heksamer (H3-H4)2(H2A-H2B) związany z DNA. Ma to np. miejsce podczas transkrypcji.
W dysocjacji składników nukleosomu udział bierze białko opiekuńcze FACT (facilitate chromatin transcription), które wiąże dimer H2A-H2B, utrudniając równocześnie jego skądinąd łatwą dysocjację na H2A i H2B. Takie „przytrzymanie” H2A-H2B sprawia, że po przejściu polimerazy RNA struktura oktameru może zostać łatwo odtworzona.
W większości przypadków do nici chromatynowej podczas replikacji wbudowywany jest podstawowy wariant histonu H3 zwany H3.1, syntetyzowany intensywnie w fazie S cyklu komórkowego. Nić chromatynowa zbudowana z oktamerów zawierających ten histon jest naogół transkrypcyjnie nieaktywna. Regiony aktywne i regulatorowe natomiast są wzbogacone w inny wariant histonu H3, mianowicie H3.3. Ten drugi wariant jest syntetyzowany konstytutywnie: zarówno w fazie S jak i poza nią. W regionach intensywnie transkrybowanych nukleosomy wzbogacane są w wariant H3.3 częściowo podczas replikacji a częściowo już po tym okresie. Zastępowanie jednego wariantu histonu innym zachodzi dzięki dynamice nukleosomów, które w regionach transkrybowanych ulegają stałemu rozpadowi i odbudowie (Ryc. 8-7b). Dynamikę tę tracą gdy transkrypcja ulaga wyhamowaniu. Chromatyna przechodzi wtedy ze stanu „otwartego” w stan „zamknięty” z wyjątkiem krótkich odcinków regulatorowych. W tych odcinkach nadal zachodzi wymiana histonów a w szczególności utrzymuje się wysoki poziom histonu H3.3 w czym bierze udział białko opiekuńcze HirA ( histon regulator A) oraz czynnik Asf1. Białko HirA pełni poza okresem replikacji taka rolę jak CAF1 podczas replikacji z tym, że odkłada na DNA dimery H3.3-H4. Nukleosomy zawierające wariant H3.3 są bardziej labilne dzięki czemu łatwiej w nich zachodzić wymiana składowych. Dodatkowym elementem ułatwiającym rozpad i rekonstrukcje oktameru w odcinkach regulatorowych i intensywnie transkrybowanych jest wariant histonu H2A.Z. Charakteryzuje się on tym, że destabilizuje strukturę nukleosomu zawierającego wariant H3.3 natomiast stabilizuje nukleosomy zawierające histon H3.1. Z tego powodu odnajduje się go również w konstytutywnej heterochromatynie centromerowej.
Ad (3) Dynamika nukleosomów polegająca na wymianie podjednostek w oktamerach histonów, na rozpadzie i rekonstrukcji nukleosomów oraz na przesuwaniu nukleosomów z jednaj pozycji na nici chromatynowej w inną wymaga dostarczania energii. Rolę tę spełniają kompleksy remodelujące (ich mechanizm działania omówiony jest niżej). W drożdżach stwierdzono, że kompleks remodelujący RSC (remodels structure of chromatin), w obecności białka opiekuńczego NAP1, usuwa nukleosomy czerpiąc energię z hydrolizy ATP. Kompleks remodelujący ISW2 natomiast przesuwa nukleosomy z pozycji energetycznie uprzywilejowanej w pozycję w której obecność nukleosomu uniemożliwia aktywację transkrypcji. Tak więc działanie kompleksów remodelujących może zależnie od sytuacji działać w kierunku aktywacji, lub represji transkrypcji.
Ad (4) Na stabilność i dynamikę nukleosomów wpływają również modyfikacje posttranslacyjne samych histonów. Jedną z takich modyfikacji jest przejściowa acetylacja lizyny H3K56 towarzysząca procesowi odkładania histonów na DNA i procesowi wymiany podjednostek oktameru w obecności białka Asf1. Lizyna ta znajduje się w pobliżu SHL (superhelix location) 6,5, w którym to miejcu DNA „opuszcza” nukleosom stając się łącznikiem (linker). Neutralizacja ładunku dodatniego lizyny H3K56 przez resztę acetylową w tej właśnie pozycji przyczynia się do destabilizuji nukleosomu. Niezależnie od tej modyfikacji histony są również modyfikowane w ogonach. (patrz niżej). Modyfikacje te mają kluczowe znaczenie jako, że stanowią one rodzaj kodu odczytywanego przez inne białka, które z kolei biorą udział w derepresji chromatyny lub odwrotnie w wyciszaniu ekspresji i konstruowaniu struktur heterochromatynowych.
Ad (5) Stabilność nukleosomu jest modyfikowana przez szereg białek niehistonowych. Wspomniano już o białkach opiekuńczych HirA, Asf1 i CAF1, które destabilizują nukleosom. Podobnie działają czynniki remoddelujące typu Brahma. Ten ostatni czynnik wiąże się z acetylowaną formą ogona histonu H4. Odwrotne działanie ma białko HP1 (heterochromatiun protein 1). Białko to posiada domenę zwaną chromodomeną (chromatin modification domain), która wykazuje powinowactwo do ogona histonu H3 metylowanego w lizynie 9 (H3K9me3).
Ryc. 8-8. Modulacja stabilności nukleosomu: (a) acetylotransferaza (HAT) destabilizuje nukleosomy przez acetylowanie ogonów. (b) HP1 wiąże się z ogonami posiadającymi metylowane lizyny H3K9me3. (c) Remodelujące białko SWR1 zastępuje dimer H2A-H2B dimerem H2A.Z-H2B, co destabilizuje nukleosomy zawierające wariant H3.3 histonu H3.
Jak czynniki transkrypcyjne regulują ekspresje genu. Podczas zmiany typu komorek zwiaznej np. z cyklem rozwojowym, niektore geny dotychczas nietranskrybowane znajdujace sie w strukturach heterochromatynowych musza ulec derepresji. W tym celu nastapic musi rozluznienie tych struktur i udostepnienie sekwencji regulatorowych w DNA dla oddzialywan z bialkami bioracymi udzial w procesie ekspresji.
Rozluznianie chromatyny wyglada troche jak problem typu: co bylo pierwsze kura czy jajko? Aby rozluznic chromatyne odpowiednie bialka musza ulec zwiazaniu z DNA, co z kolei wymaga, aby chromatyna byla rozluzniona. Jedno z rozwiazan tego paradoksu polega na wyżej wspomnianym pozycjonowaniu oraz dynamice nukleosomów w regionie promotorowym. Alternatywne rozwiązanie tego problemu polega na zdolności niektorych czynników transkrypcyjnych do rozpoznawania swoich specyficznych miejsc wiazania nawet wtedy gdy DNA jest zwiazany z oktamerem w nukleosomie.
Np. receptor glukokortykoidu GR rozpoznaje krotką sekwencje nukleotydow w DNA i wiaze sie w rowku wiekszym, po jednej stronie helisy, nawet wtedy gdy DNA jest zwiazany z histonami (Ryc 8-9). Natomiast aktywator zwany czynnikiem NF1 (nuclear factor 1) rozpoznaje dluzsze sekwencje DNA i calkowicie otacza czasteczke, tak, ze nie jest w stanie stworzyc kompleksu z DNA gdy ten jest owiniety wokol oktameru. Niekiedy dostep do DNA uwarunkowany jest precyzyjnym umiejscowieniem rozpoznawanej sekwencji w stosunku do powierzchni nukleosomu. Stwierdzono, ze pomiedzy czynnikami transkrypcyjnymi zachodzi funkcjonalna kooperacja. Na przyklad wirus MMTV posiada promotor zawierajacy pieć elementow wiazacych GR i dwa elementy NF1. GR wiaze sie do conajmniej dwoch miejsc nawet wtedy gdy DNA jest w formie nukleosomu. Po zwiazaniu sie z DNA GR rekrutuje czynniki modyfikujace chromatyne zwiekszajac tym samym dostepnosc DNA i umozliwiajac wiazanie NF1.
Aby rozluznienie zapoczatkowywane przez aktywatory transkrypcji doszlo do skutku potrzebna jest wspolpraca koaktywatorow. Z reguly, dopiero rekrutacja koaktywatorow prowadzi do lokalnego rozluznienia chromatyny umozliwiajac dostep RNAPII oraz ogolnej maszynerii transkrypcyjnej do promotora.
Obok aktywatorow w chromatynie dzialaja represory takie jak np. bialko pRB kodowane przez gen supresorowy nowotworow RB1. Jego nazwa pochodzi od tego, ze mutacje w tym genie wywoluja nowotwor dzieciecy retinoblastome. Bialko to i inne nalezace do tej samej rodziny i wykazujace podobienstwa strukturalne biora udzial w regulacji cyklu komorkowego. Ich ekspresja powoduje zahamowanie komorki w fazie G1
Koregulatory jako potencjalne koordynatory regulacji transkrypcji. Koregulatory sa rekrutowane do promotorow przez czynniki transkrypcyjne specyficzne do sekwencji DNA. Ryc. 8-10 pokazuje przykładowo działanie koregulatorów. W pierwszym przypadku receptor jadrowy RAR/RXR (retinoic acid receptor, receptor kwasu retinolowego) zwiazany z ligandem (kwasem retinolowym) rozpoznaje sekwencje regulatorową DNA, wiąże się z nią i rekrutuje koaktywatory PCAF i CBP/p300, które mają aktywność acetylotransferazy, dzięki czemu inicjują otwieranie chromatyny. Ten sam receptor jądrowy w nieobecności ligandu działa jako represor ponieważ po związaniu się z miejscem regulatorowym na DNA rekrutuje deacetylazy histonowe (HDAC 1 i 2). Aby mogła zostać zainicjowana transkrypcja konieczna jest obecność mediatora, kompleksu zlozonego z szeregu podjednostek, ktory wiaze sie z polimeraza RNA, RNAP II.
Ze wzgledu na sposob w jaki koregulatory zmieniaja strukture chromatyny mozna je podzielic na dwie grupy : pierwsza to kompleksy modyfikujace histony takie jak acetylotransferazy histonow (HAT), deacetylazy histonow (HDAC), metylo-transferazy histonow (HMT), demetylazy histonów czy kinazy histonow. Dotychczas poznane modyfikacje są wyliczone na Ryc. 8-11.
Acetylacja reszt aminokwasowych w łańcuchach bocznych lizyny i argininy w cząsteczkach histonów byla pierwszą z modyfikacji dla ktorych wykazano korelacje z kompetencja transkrypcyjna. Wynikiem acetylacji jest rozluźnienie struktury chromatyny spowodowane między innymi tym, że likwiduje ładunek dodatni grupy aminowej co powoduje osłabienie oddziaływań elektrostatycznych naładowanych dodatnio histonów z niosącym ładunki ujemne łańcuchem fosfocukrowym DNA. Ten efekt elektrostatyczny nie wchodzi jednak w grę w przypadku metylacji grup aminowych, które pozostają naładowane (Ryc. 8-12). Przez długi czas mechanizm molekularny wpływu modyfikacji histonów na ich funkcje pozostawał niewyjaśniony. Dopiero teoria kodu histonowego (patrz niżej) takie wyjaśnienie zaproponowała.
Zarówno HAT i HMT powodują modyfikację reszt aminokwasowych lizyn i arginin w "ogonach" histonów. Modyfikacje te są niezbędne dla aktywacji transkrypcji wielu genow. Odwrotnie, rekrutacja HDAC przez represory transkrypcji powoduje deacetylowanie ogonow co prowadzi najczesciej do wygaszania transkrypcji.
Zaleznosc pomiedzy modyfikacjami ogonow histonowych i ekspresja genow jest zlozona poniewaz rozne modyfikacje w roznych miejscach w ogonie wplywaja na siebie nawzajem. Po raz pierwszy zostalo to zauwazone w przypadku acetylacji i fosforylacji histonu H3 (fosforylacja H3S10ph blokuje acetylacje H3K9), ale stwierdza sie rowniez interferencje pomiedzy acetylacja i metylacja. I tak np. metylacja H3K4, która jest jedną z najlepiej poznanych modyfikacji pociągających za sobą utworzenie struktury otwartej chromatyny skutkuje utworzeniem H3K14ac natomiast wyklucza metylację lizyny H3K9. Ta ostatnia modyfikacja charakteryzuje nukleosomy chromatyny zbitej. I odwrotnie, metylacja prowadząca do utworzenia H3K9me blokuje metylację H3K4.
Skutki metylacji argininy są mniej znane, aczkolwiek wiadomo, że modyfikacja ta inerferuje z innymi modyfikacjami (patrz Ryc. 8-13c).
Ryc. 8-11. Modyfikacje posttranslacyjne w ogonach histonowych (a) rodzaje modyfikacji, (b) lizyny i argininy ogonów histonowych podlegające modyfikacji.
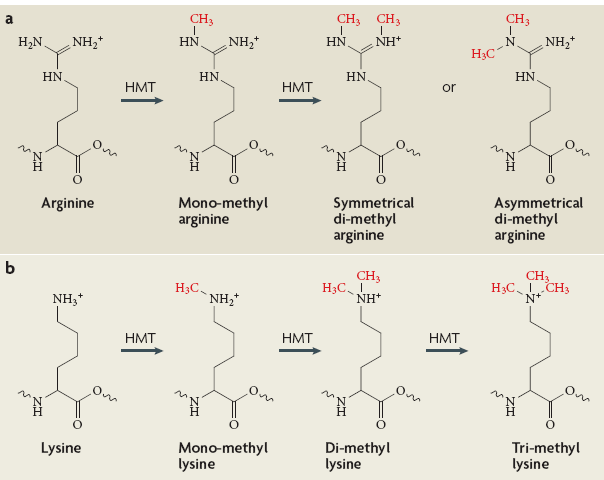
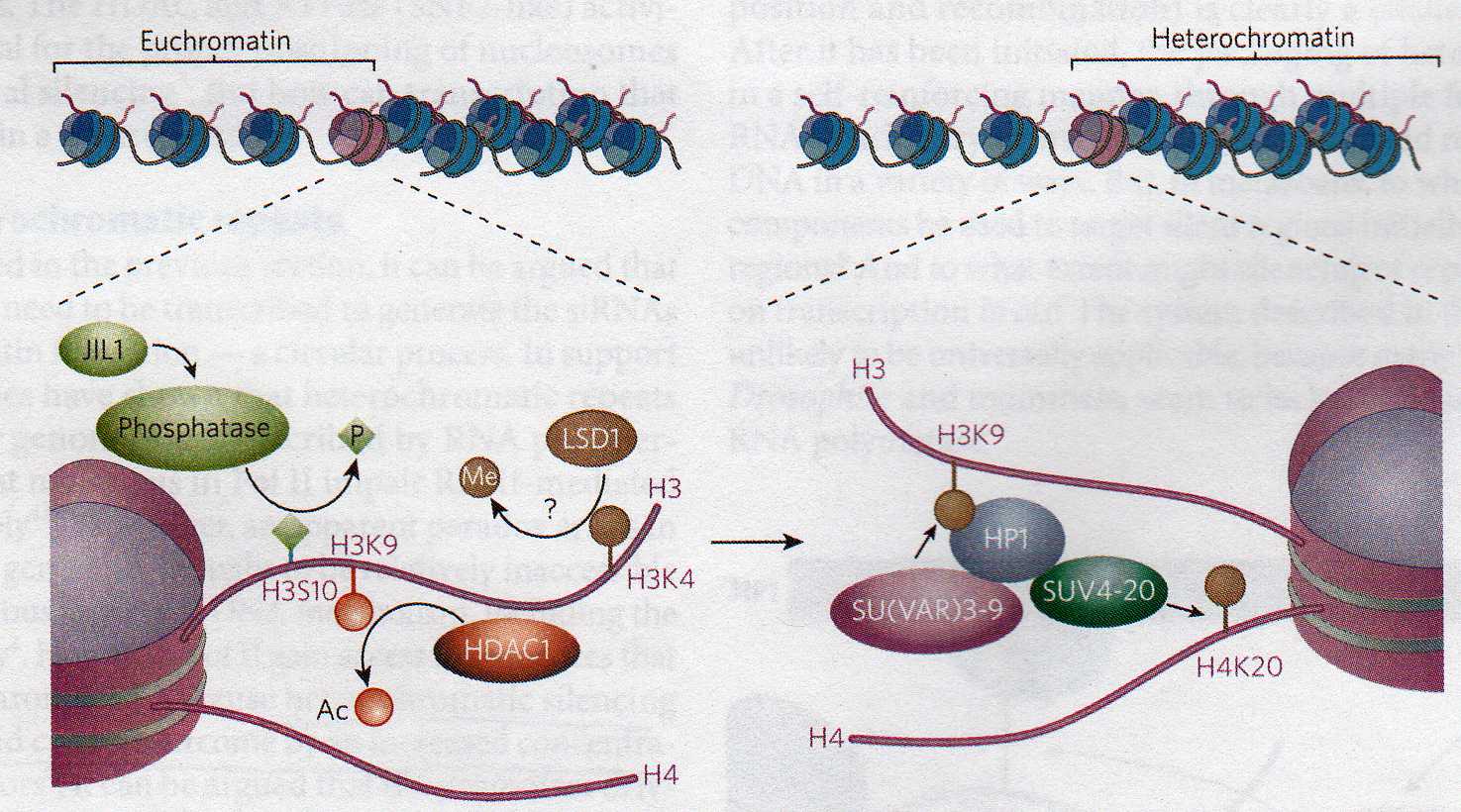
Ryc. 8-13 Modyfikacje histonowe u drosofili podczas przejścia chromatyny rozproszonej w chromatynę zbitą
Jak wspomniano wyżej modyfikacje ogonow histonowych wraz z białkami opiekuńczymi i remodelującymi regulują stabilność i dynamikę nukleosomów. Ponadto są one rozpoznawane przez niektóre białka niehistonowe, które z kolei modyfikują strukturę chromatyny. Mówi się, że modyfikacje te są elementem „kodu histonowego” : każda modyfikacja jest „odczytywana” przez odpowiednie białka, których wiązanie pociąga za sobą zmianę struktury włókna chromatynowego adekwatną do aktualnych potrzeb komórki.
Ryc. 8-14.Tabela przedstawiająca propozycję kodu histonowego. Obecnie znamy odpowiedzi na niektóre z pytań postawionych w tabeli (patrz tekst).
.
Istotnie, stwierdzono, że szereg bialek regulatorowych posiada specjalne domeny np. chromodomene (chromatin modification domain) (Fig. 8.15) czy bromodomene, ktore wiaza sie specyficznie z nicia chromatynowa zawierajaca nukleosomy w ktorych ogony histonow sa zmodyfikowane.
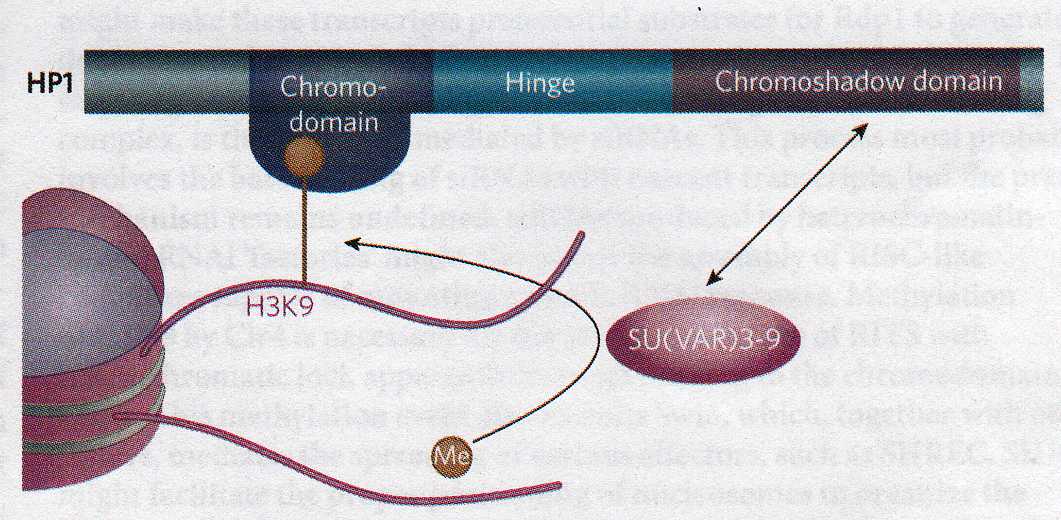
Fig. 8.15 Schemat struktury HP1 (Heterochromatin Protein 1)
Bialka posiadajace tzw "bromodomene" czyli specyficzna domene lancucha polipeptydowego o dlugosci 110 aminokwasow posiadaja powinowactwo do acetylowanej lizyny. Ich funkcją jest rozluźnianie chromatyny. Przykładem takiego białka jest komleks remodelujący Brahma. Tak więc acetylacja ogonów histonowych nie tylko pociąga za sobą zmniejszenie oddziaływania ładunków ujęmnych łańcucha fosfocukrowego DNA z dodatnimi ładunkami lizyny. Dalsze rozluźnienie jest spowodowane przez kompleks remodelujący.
"Chromodomena" (chromatin modification domain) obecna w bialkach HP1 u kregowcow i w bialkach grupy polycomb (PcG) u drosofili wiaze nukleosomy metylowane w lizynie H3K9 i H3K27 powodujac kaskadę procesów których wynikiem jest heterochromatyna. A więc np. H3K9me rekrutuje białka typu PcG (w szczególności EZH2 - enhancer of zest homologue 2), które z kolei rekrutują metylotransferazę DNA, która metyluje dinukleotydy CpG. Tak zmetylowany dinukleotyd rekrutuje z kolei białka posiadające domenę MBD (methyl CpG binding domain), które z kolei rekrutują deacetylazy histonowe HDAC. W wyniku następuje utworzenie heterochromatyny.
Innym przykładem kaskadowej reakcji związanej z modyfikacją ogonów histonowych jest reakcja wywołana przez infekcję komorek ludzkich przez wirusy. Infekcja powoduje wpierw acetylacje lizyny H3-K9 oraz fosforylacje seryny H3S10 w regionie promotora interferonu (ktory stanowi jeden z czynnikow obronnych organizmu przed inwazja wirusow). Fosforylacja H3S10 jest warunkiem niezbednym dla acetylacji H3K14, co tez wkrotce po tej fosforylacji nastepuje. Niezaleznie od tego nastepuje rowniez acetylacja H4K8. Acetylacja lizyn H3K9 i H3K14 jest warunkiem koniecznym dla rekrutacji czynnika transkrypcyjnego TFIID rozpoznajacego kasete TATA. Utworzenie kompleksu TFIID z DNA stanowi pierwszy etap w konstrukcji kompleksu preinicjacyjnego polimerazy II RNA, natomiast acetylacja H4K8 pociaga za soba rekrutacje kompleksu typu SWI/SNF rozluźniajacego chromatyne i przygotowujacego ekspresję genu. Tak wiec specyficzne odczytywanie kodu histonowego przez odpowiednie systemy modyfikacji chromatyny zapoczatkowuje kaskadowa modyfikacje (otwieranie) struktury wlokna chromatynowego na odcinku kluczowym dla obrony organizmu.
Modyfikacje ogonów histonowych współpracują z innymi elementami regulującymi ekspresję genów. Przykładem jest wplyw koaktywatora CARM1 (coactivator-associated arginine methyltransferase 1) na transkrypcje. CARM1 znajduje sie w centrum ukladu decydujacego o tym czy komorka transkrybuje geny znajdujace sie pod kontrola receptorow jadrowych czy też inną grupę genów bedacych pod kontrola czynnika transkrypcyjnego CREB (cAMP responsive element binding protein). CARM1 jest mianowicie koaktywatorem argininowej metylotransferazy (HMT), ktora metyluje ogon histonu H3. Ta metylacja powoduje indukcje genow regulowanych przez receptory jadrowe. Ale CARM1 koaktywuje rowniez metylacje argininy w innym koaktywatorze P300/CBP (CREB binding protein), ktory, jesli niemetylowany, aktywuje czynnik transkrypcyjny CREB. Metylacja P300/CBP uniemozliwia wiec aktywacje CREB. CARM1 jest wiec rownoczesnie koaktywatorem transkrypcji genow zaleznych od receptorow jadrowych i korepresorem genow zaleznych od czynnika transkrypcyjnego CREB.
Innym ciekawym przykładem współdziałania modyfikacji histonów z innymi elementami układu regulacji ekspresji genów dostarcza białko pRB (RB - retinoblastoma). Bialko to nalezace do supresorow nowotworow wiaze sie z niektorymi czynnikami transkrypcyjnymi regulujac ich aktywnosc podczas cyklu komórkowego. Uniemożliwia ono mianowicie przedwczesną, szkodliwą dla organizmu transkrypcję w fazie G1 tych genów, których produkty potrzebne są dopiero podczas replikacji w fazie S. Otóż pRB rekrutuje również bialko SUV39H1, ktore ma wlasnosc HMT metylujacej podwojnie grupe aminowa lancucha bocznego lizyny H3K9. Nukleosomy z ogonami metylowanymi w ten sposob rekrutuja bialko HP1 posiadajacą chromodomenę. W wyniku tego następuje heterochromatynizaca genow znajdujących się pod kontrolą pRB. A więc pRB uniemożliwia transkrypcję genów z jednej strony przez inhibicje czynników transkrypcyjnych a z drugiej strony przez zamknięcie chromatyny na odcinkach niosących te geny.
Druga grupa koregulatorow to kompleksy remodelujace chromatyne, ktore uzywaja jako zrodla energii ATP. Ich dzialanie polega na ulatwianiu (lub utrudnianiu) dostepu do DNA poprzez zmiane pozycji nukleosomow w obrebie promotora, lub przez całkowite ich usuwanie, przez indukowanie zmian konformacyjnych w nukleosomach, eliminację podjednostek i inicjowanie podstawienia wariantów histonowych (Ryc. 8-16). Wiekszosc z nich nalezy do jednej z dwoch rodzin : SWI/SNF oraz ISWI (Ryc. 8-17). Pierwszy z tych kompleksow odkryty zostal u drozdzy, gdzie stwierdzono, ze niektore mutanty niezdolne do fermentacji (SNF - sucrose non fermenting) mialy rownoczesnie defekt w przelaczaniu typu koniugacyjnego (SWI, switch - przełącznik). Rodzinę genów odpowiedzialnych za ten fenotyp nazwano SWI/SNF. Bialka przez nie kodowane tworza duzy kompleks zlozony w sumie z 12 podjednostek, ktory to kompleks hydrolizuje ATPi zmienia strukture chromatyny in vitro (Ryc. 8-18) w ten sposob, ze staje sie ona bardziej wrazliwa na trawienie enzymami nukleolitycznymi. Produkty trawienia przypominaja bardziej produkty trawienia DNA "nagiego" niezwiazanego z nukleosomami niz DNA obecnego we wloknie chromatynowym. Jesli chodzi o transkrypcje, to stwierdzono, ze u drozdzy mutanty swi/snf wykazuja zmniejszona ekspresje pewnych genow a zwiekszona innych. Pozwala to sądzić, ze kompleks SWI/SNF reguluje ekspresje niektorych represorow. U drozdzy wykryto rowniez drugi, homologiczny kompleks bialkowy RSC (remodel the structure of chromatin) wykazujacy podobne wlasnosci. U drosofili znaleziono homologiczny kompleks zawierajacy produkt genu Brm (brahma) a u czlowieka dwa kompleksy : hBRM (human brahma) i BRG-1 (brahma related gene).
Mutacje w genach kodujacych podjednostki kompleksow remodelujacych chromatyne moga miec bardzo rozne skutki. U myszy homozygoty ze wzgledu na delecje w genie brm, nie stwierdzono specjalnych defektow poza tym, ze byly one nieco wieksze od myszy bez delecji. Wskazuje to na zaklocenia kontroli wzrostu. Natomiast delecja genu BRG-1 jest letalna na wczesnym etapie rozwoju embrionu.
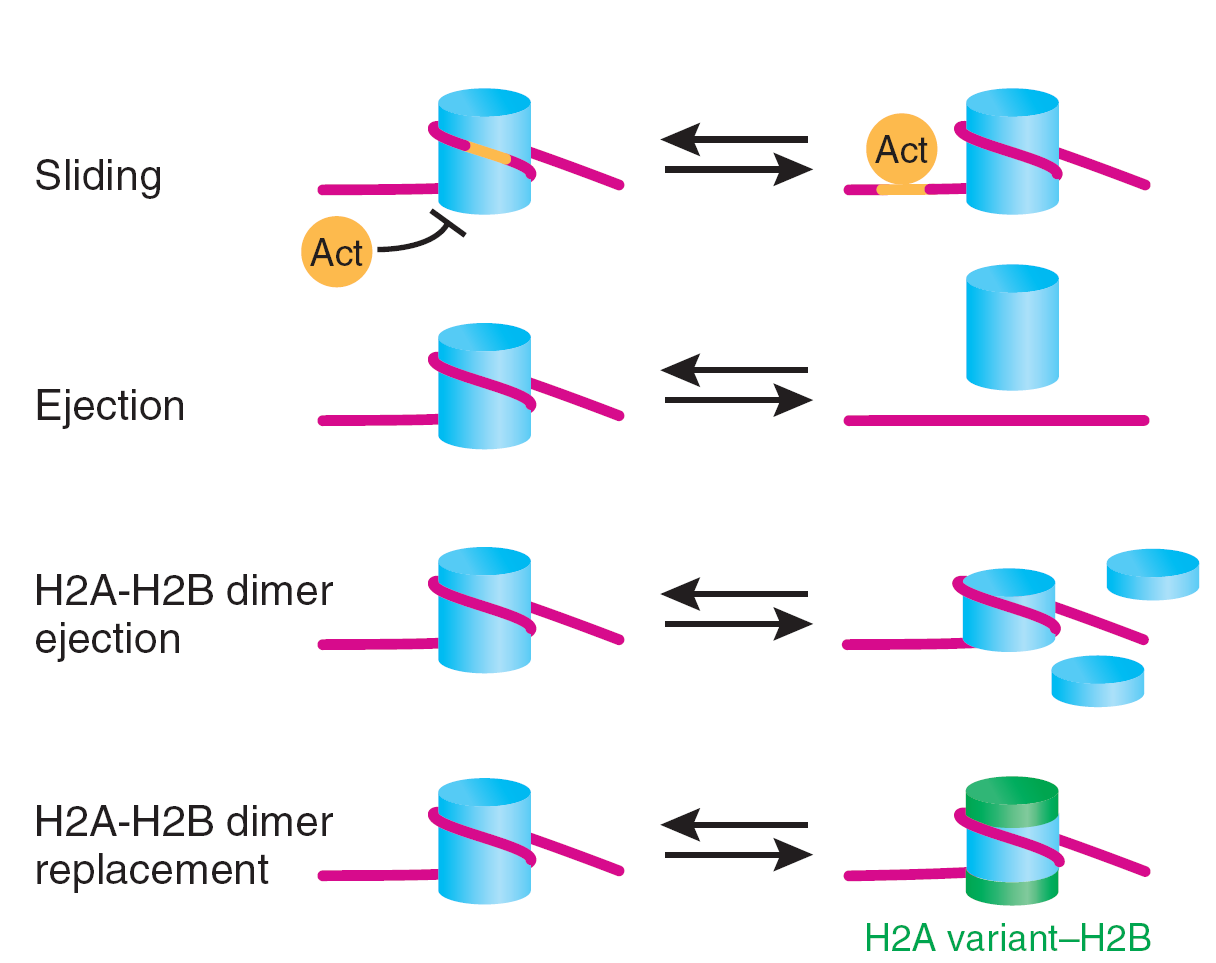
Fig 8-16. Najważiejsze reakcje katalizowane przez kompleksy remodelujące chromatynę.
Druga rodzina kompleksow zaangazowanych w remodelowanie chromatyny zwanych ISWI (Imitation SWI) rozni sie nieco pod wzgledem funkcji. Zamiast zwiekszac ogolna podatnosc wlokna chromatynowego na trawienie nukleolityczne kompleksy te powoduja zmiane w pozycjonowaniu nukleosomow pod wplywem ATP. Pod wplywem ich dzialania obserwuje sie wiec zmiany w odleglosciach pomiedzy nukleosomami.
Istnieje jeszcze trzeci typ kompleksow remodelujacych zawierajacy bialko Mi-2. Roznia sie one od kompleksow typu SWI/SNF i ISWI tym , ze po pierwsze w ich skladzie znajduje sie podjednostka wiazaca sie specyficznie do metylowanego DNA, a po drugie zawieraja one jako podjednostki deacetylazy histonow HDAC1 i HDAC2. Kompleks typu Mi-2 ilustruje dobrze ogolna zasade koordynacji dzialania mechanizmow komorkowych, ktore nie funkcjonuja in vivo oddzielnie. Zwykle skutki jednego wzmacniaja lub znosza dzialanie drugiego i vice versa.
Ryc. 8-18. Mechanizm molekularny pokazujący remodelowanie struktury chromatyny
Kompleks preinicjacyjny w przypadku polimerazy, ktora syntetyzuje informacyjny RNA tworzy sie w regionie nici chromatynowej uprzednio rozluznionej przez dzialanie aktywatorow i koaktywatorow (ryc 8-19) a wiec majacej charakter miejsca HS.
Kompleks ten, ktorego centralnym skladnikiem jest w przypadku transkrypcji genow kodujacych bialka RNA polimeraza II (RNAPII), zawiera szereg bazowych czynnikow transkrypcyjnych. Pierwszy z nich, TFIID, posiadajacy dwie podjednostki TBP (TATA box binding protein) i TAF (TBP Associated Factor), tworzy kompleks z DNA w miejscu kasety TATA. Kolejno dolaczaja inne czynniki bazowe TFIIA, B, E, F i H (Ryc. 8-20). Sekwencja aminokwasow w RNAPII od strony karboksylowej zawiera 52 powtorzenia sekwencji heptapeptydu TyrSerThrSerProSer (jest to domena CTD, carboxy terminal domain) ktora stanowi rodzaj platformy dla budowy kompleksu zaangazowanego w proces dojrzewania RNA. Dlugosc CTD oraz jego fosforylacja ma znaczenie dla aktywnosci enzymatycznej kompleksu. Fosforylacja ta ulega zmianie podczas transkrypcji.
Wkrotce po tym jak RNAPII inicjuje transkrypcje, powstajacy RNA jest modyfikowany przez dodanie czapeczki (ang. cap) na koncu 5'. Sluzy ona do ochrony RNA przed atakiem nukleolitycznym oraz jako miejsce wiazania bialek transportujacych dojrzaly RNA do cytoplazmy i jego translacje na polipeptyd. Proces ten jak sie okazalo, koordynuje wczesne etapy transkrypcji a w szczegolnosci przejscie od etapu inicjacji transkrypcji tzn. samego startu polimeryzacji do etapu elongacji tzn. wydluzania lancucha polinukleotydowego. Elongacja jest kontrolowana przez specjalne bialka "czynniki elongacyjne". W czasie tego procesu zachodzi czesto rowniez wycinanie intronow. Stwierdzono, ze luzna struktura promotora nie wystarcza dla uzyskania efektywnej transkrypcji. Zidentyfikowano specjalny czynnik, FACT (facilitates chromatin transcription), ktory ulatwia elongacje transkrypcji poprzez dysocjacje histonow H2A i H2B z chromatyny in vitro. Inny kompleks rozluzniajacy chromatyne poza promotorem zwany elongatorem ma funkcje HAT.
Gdy polimeraza dotrze do konca genu, transkrypcja zatrzymuje sie po napotkaniu sygnalu "terminacji". Lancuch polirybonukleotydowy zostaje wowczas przeciety i do jego konca 3' zostaje dodany odcinek poliA. Nowosyntetyzowane mRNA jest nastepnie transportowane przez otoczke jadrowa do cytoplazmy, gdzie ulega translacji. W eksporcie mRNA posrednicza bialka, ktore z jednej strony wiaza sie do mRNA a z drugiej oddzialywuja z bialkami porow w blonie jadrowej. Zlozonosc kazdego z tych etapow powoduje, ze badane sa one oddzielnie, co z kolei powoduje, ze tradycyjnie uwaza sie je za oddzielne niezwiazane z soba procesy. W ostatnich latach stalo sie jednak jasne, ze poszczegolne etapy ekspresji genow sa bardzo scisle powiazane i wplywaja jeden na drugi. Proces syntezy bialka przypomina produkcje na tasmie montazowej, gdzie element, ktory ulega przeksztalceniu na jednym etapie produkcji jest przekazywany jako substrat do nastepnego etapu w sposob ciagly, a czynniki bialkowe odpowiedzialne za kazdy z poszczegolnych etapow procesu sa funkcjonalnie a czasem i mechanicznie powiazane w kompleksy.
Metylacja zwiazana z procesem wygaszania aktywnosci transkrypcyjnej genow dotyczy nie tylko histonow ale rowniez i DNA (u ssakow metylacji ulega glownie dinukleotyd CpG, u roslin rowniez CpNpG). Te modyfikacje maja charakter epigenetyczny i sa dziedziczone z jednego pokolenia komorek na drugie (patrz rozdzial poswiecony wyspom CpG).
Izolatory. Izolatory sa to struktury rozgraniczajace funkcjonalnie domeny wlokna chromatynowego (Ryc. 8-21). Ich zadaniem jest ochraniac geny przed nieprawidlowymi sygnalami emanujacymi z otaczajacych segmentow wlokna. Rozroznic mozna dwa typy izolatorow : pierwszy znosi dzialanie wzmacniacza na promotor jesli znajduje sie pomiedzy nimi a drugi typ stanowi bariere uniemozliwiajaca postep procesu kondensacji chromatyny ktory ma czesto charakter "inwazyjny". Niektore izolatory posiadaja obydwie te funkcje.
U drozdzy w regionie HMR zidentyfikowano tRNAThr, ktory ma wlasnosci izolatora zatrzymujacego rozprzestrzenianie sie wygaszania ekspresji sasiadujacych genow przez bialka Sir.
Ryc. 8-21. Izolator uniemozliwiajacy rozprzestrzenianie sie struktury heterochromatynowej.
niebieski kompleks - kohezyna, czerwony kompleks - kondensyna, mur - izolator.
Bialka te powoduja heterochromatynizacje odcinka polozonego pomiedzy elementami sekwencji E i I flankujacymi HMR. Niezbedne dla aktywnosci izolujacej sa w tym przypadku czynniki transkrypcyjne TFIIIC i TFIIIB. Zarowno lewa jak i prawa granica regionu HMR posiadaja rowniez sekwencje wiazace kohezyny, co wskazuuje na to, ze odgrywaja one pewna role przy tworzeniu bariery. Element STAR (subtelomeric anti-silencing region) usytuowany pomiedzy subtelomerowymi sekwencjami X i Y rowniez ma wlasnosci izolatora uniemozliwiajacego rozprzestrzenianie sie heterochromatyny telomerowej w chromosomach drozdzy. Zawiera on wielokrotnie powtarzajace sie sekwencje rozpoznajace bialko o nieznanej funkcji Tbf1 i oraz inne rozpoznawane przez slaby aktywator transkrypcji Reb 1, obecny rowniez w wielu promotorach genow transkrybowanych przez polimeraze RNAPI i II oraz terminatorach polimerazy RNAPI.
U drosofili element Gypsy, ktory jest elementem powtarzajacym sie typu retrotranspozonu, zawiera elementy sekwencji rozpoznawane przez specyficzne bialko su(hw) (supressor of hairy wings). Bialko to oddzialywuje z bialkiem Mod(mdg4) tworzac funkcjonalny izolator.
W przypadku kregowcow aktywnosc izolatora jest czesto zwiazana z obecnoscia CTCF (CCCTC binding factor) oraz GAGA (bialka wiazacego sekwencje GAGA).
Rola chromatyny w ekspresji genow
-----------------------------------------------------------------------------------------------------------------
124
111
A.
B.
Ryc. 8-19 Powstawanie transkrypcyjnie aktywnej domeny chromatynowej przez rekrutacje regulatorow, koregulatorow i czynnikow transkrypcyjnych bazowych.
Ryc 8-20. Powstawanie kompleksu preinicjacyjnego.
Ryc 8-12. Struktury wielokrotnie metylowanych aminokwasów w histonach.
Ryc. 8-4. Miejsca czule i hiperczule w genach kodujacych histony u drosofili.
Ryc 8-9. Miejsca wiaznia GR w DNA zwiazanym z nukleosomem.
Ryc. 8-10. Modyfikacja chromatyny przez RAR/RXR. a) receptor kwasu retinolowego polaczony z ligandem rekrutuje koaktywator CPB/p300 i PCAF (HAT), b) Receptor bez ligandu rekrutuje Sin3a, HDAC2 i HDAC3 powodujac heterochromatynizacje regionu i represje transkrypcji
Ryc. 8-17 Struktura Kompleksów remodelujacych chromatynę.
eliminacja
poślizg
Eliminacja H2A-H2B
Podstawienie H2A-H2B wariantem H2A.Z-H2B
Wyszukiwarka
Podobne podstrony:
2009 08 08 Nostradamus Koniec świata już w tym roku
2009 08 26 215924
Elektronika Praktyczna 2009 08
FHM 2009 08 French
2009 08 Little and Big
upoważnienie - 2009.08.26, 1---Eksporty-all, 1---Eksporty---, 12---agencje-celne, 2---A.C.-Vega-Lesz
PILZ Compendium EN 2009 08
2009 08 29 213427
2009 08 26 220839
2009 08 08 Nostradamus Koniec świata już w tym roku
2009 08 26 215924
Elektronika Praktyczna 2009 08
( ) A wrogów jeszcze bliżej [NZ] (z 2009 08 31)
dimplex wgm90 grm95 fd de 2009 08 10
Elektronika Praktyczna 2009 08
2009 08 29 213427
2009 08 11 Bronią Liceum św Augustyna
2009 08 08 Euro 2012 nie odbędzie się
FHM 2009 08 French
więcej podobnych podstron