skanuj0035
Tabela 8.4. Długość nukleosomowego DNA
|
Typ komórki |
Liczba par zasad |
|
Aspergillus |
154 |
|
Neurospora |
170 |
|
Drożdże |
165 |
|
Physarum |
181 |
|
Komórki embrionalne jeżowca (gastrula) |
218 |
|
Sperma jeżowca |
241 |
|
Jajowód kury |
196 |
|
Erytrocyty kury |
207 |
|
Szpik kostny szczura |
192 |
|
Wątroba szczura |
196 |
|
Nerka szczura |
196 |
|
Komórki HeLa |
183 |
żującymi się konserwatywnością sekwencji aminokwasowej. Wskazuje to na uniwersalność struktury nukleosomowej podczas ewolucji.
Histon lU.wiąże się natomiast z łącznikowym DNA stabilizując dwa skręty DNA~wokół oktamem1nstonówv śprna on DNA";,wchodzący” i „schodzący” z cząstki rdzeniowej oddziałując z około 10 parami zasad obu „końców” DNA (rys. 8.2). Struktura zawierająca fragment DNA o długości około 166 par zasad, oktamerhistonówrdzeniowychijednąkopięhistonuHl określana jest mianem chromatosomu (tab. 8.5).
histon H1
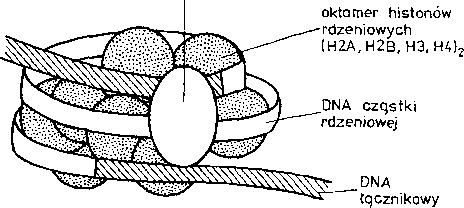
11 nm
Rys. 8.2. Schemat budowy nukleosomu (wg Stryera, 1981)
|
Nazwa |
Liczba par zasad DNA |
Oktametr (H2A, H2B, H3, H4)2 |
Histon HI |
|
Nukleosom |
-200 |
+ |
+ |
|
Chromatosom |
166 |
+ |
+ |
|
Cząstka rdzeniowa |
146 |
+ |
- |
Około 90% chromatynowego DNA zorganizowane jest w nukleosomy. Nukleosomowa struktura chromatyny występuje we wszystkich komórkach eukariotycznych. Wyjątek stanowią plemniki, w których w czasie spermatogenezy histony zastępowane są przez mniejsze i jeszcze bardziej zasadowe białka, tj. protaminy. Organizację nukleosomową wykazują również wewnątrzkomórkowe formy dwuniciowych DNA-wirusów, np. wirusa małpiego SV40. Histony w tych nukleosomach są pochodzenia eukariotycznego.
Solenoid
Strukturę wyższego rzędu, w tworzeniu której nieodzowna wydaje się obecność histonu HI określa się mianem snlenoidn (superspirali). będącego helikalną formą włókna nukleosomowego. Powstające włókno chromatynowe o średnicy około 30 nm i skoku 10-11 nm zawiera 6 nukleo-somów na skręt. Nukleosomy przypominające kształtem spłaszczone dyski ułożone są w solenoi-dzie tak, że ich powierzchnie twarzowe zorientowane są mniej więcej równolegle do długiej osi włókna. Obserwowane we włóknie 30 nm globułame jednostki (ang. superbeads) składające się z 6-14 nukleosomów wydają się powstawać w wyniku lokalnego przerwania ciągłości solenoidu i być może mają znaczenie funkcjonalne. Upakowanie nukleosomów w solenoidzie pozwala na pozorne skrócenie zawartego w nim DNA w przybliżeniu 40 razy (6-7 razy przez owinięcie DNA na oktamerze histonów i 6 razy przez superspiralne zwinięcie włókna nukleosomowego).
Doświadczenia nad rekonstytuowanymi nukleosomami wykazały, że histon HI nie wystarcza do odtworzenia włókna chromatynowego o średnicy 30 nm. Sugemje się udział również białek niehistonowych, zasadowych końców N histonów rdzenia zaangażowanych nie w interakcję histon-histon, ale prawdopodobnie w oddziaływanie histon-DNA, a także udział kationów dwu-wartościowych.
Struktury wyższego rzędu
Kolejny stopień uporządkowania strukturalnego, określany jako „model promienistych pętli”, odpowiada ułożeniu włókna o średnicy 30 nm w pętle (domeny) liczące 20-200 kpz. Rjtłe te swoją podstawą zakotwiczone są w białkowym rusztowaniu matriks jądrowej lub białek „scaffold”, stanowiącym osiową strukturę podporową chromosomów metafazowych. Białka szkieletowe (prawdopodobnie obok RNA) wydają się odpowiedzialne za ostateczną integrację materiału genetycznego, zarówno w czasie replikacji i transkrypcji, jak i w czasie mitozy i podziału komórkowego. Różnice w wewnętrznej strukturze frbryl interfazowych i metafazowych dotyczą stopnia ich upakowania. Pozorne skrócenie długości DNA w jądrach interfazowych wynosi od około 100 do 1000 razy, podczas gdy w chromosomach podziałowych — około 10 000 razy. Molekularne podstawy kondensacji chromatyny w czasie mitozy nie są ostatecznie wyjaśnione. Kontrakcja materiału genetycznego wydaje się spowodowana zmianami stężenia kationów dwu-
109
Wyszukiwarka
Podobne podstrony:
skanuj0051 Tabela 6 Analiza demograficzna badanej populacji przestępców (w liczbach
skanuj0581 Tabela 6.4. Przykład punktowej oceny atrakcyjności sektora liczba punktów w tej wersji me
skanuj0005 Tabela 2.1 Długość pręta (/„) [m] Średnica pręta (rf„)
skanuj0020 (65) 170 7. Ruch turystyczny Tabela 24. Obszary chronione w Meksyku w 1990 r. Typ obsza
84039 skanuj0020 (65) 170 7. Ruch turystyczny Tabela 24. Obszary chronione w Meksyku w 1990 r. Typ
84039 skanuj0020 (65) 170 7. Ruch turystyczny Tabela 24. Obszary chronione w Meksyku w 1990 r. Typ
skanuj0269 (3) Rozdział 10. ♦ Podstawy SQL 283 Tabela 10.1. Typy całkowitoliczbowe Typ Zakres
skanuj0015 (331) Tabela 5.2. Obiekty zakwaterowania zbiorowego według województw - liczba obiektów i
skanuj0003(1) Tabela 1.2. Zestawienie parametrów wykorzystywanych przyrządów pomiarowych. Lp. Nazw
skanuj0007(2) Tabela 1.2. Zestawienie parametrów wykorzystywanych przyrządów pomiarowych. Lp. Nazw
skanuj Typ komórki Lokalizacja w organizmie (tkance) Związek kształtu i wielkości komórki z
DHTML0019 Wprowadzenie Tabela IV. I. Wartości określające długości względne Nazwa Typ jednostki Co
więcej podobnych podstron