CCF20150312�005 (2)

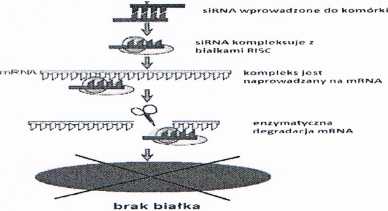
3. interferujący RNA (siRNA, smali interfering RNA) - małe cząsteczki dwuniciowego RNA (20-25 nt), które powodują wyciszanie ekspresji genów o homologicznej sekwencji (interferencja RNA, RNAi - RNA interference). Działanie przez:
• indukcję degradacji mRNA
• blokowanie translacji
X
Naprawa mutacji o charakterze trwałym Wykorzystuje:
• enzymy naprawiające błędne sparowania (mismatch repair)
• enzymy wycinające nukleotydy (excision repair)
• enzymy biorące udział w rekombinacji homologicznej
Przykład:
fragmenty DNA (400-800 pz) zawierające sekwencję homologiczną do naprawianego genu oraz prawidłową sekwencję do podstawienia (homologiczne podstawianie małych fragmentów, smali fragment homologous replacement- SFHR);
metoda SFHR w konwersji (3-globiny sierpowatej do prawidłowej w krwiotwórczych komórkach macierzystych]
4. Celowano eliminacja komórek
Precyzyjne zabijanie komórek zainfekowanych wirusem lub komórek nowotworowych
Dla nowotworów:
1. eliminacja bezpośrednia
a. wprowadzenie do komórek genu kodującego toksyczne białko (pod kontrolą promotora specyficznego dla nowotworów)
b. stosowanie genów samobójczych - przekształcających podawany systemowo prolek do toksycznego leku (np. deaminaza cytozynowa z E. coli przekształca 5-fluorocytozynę do 5-fluorouracylu; kinaza tymidynowa HSV -fosforyluje gancyklowir
c. radiouczulanie - deaminaza cytozynowa
d. geny proapoptotyczne - indukujące apoptozę (kaspazy, Bax)
2, eliminacja pośrednia
a. przez aktywację odpowiedzi immunologicznej
b. hamowanie angiogenezy
Wyszukiwarka
Podobne podstrony:
CCF20110307�006 ZADAŃ Wydatki na zakup prasy w badanej grupie osób były następujące: 20,0; 25,0; 34,
Zdjęcie139 scRNA ang smali cytoplasmic RN A - .małe cytoplazmatyczne RNA i selekcjonują białka przez
Zdjęcie136 snRNA ■ ang smali nuclear RN A - .małe ją&owe RN A ■ &
IMGD14 (2) WodaWoda - 70% masy komórek komórka baktefH 30% inne jory, małe cząsteczki (4%) łosfol ^d
CCF20120112�002 58. Jaka jest różnica między rzędem reakcji a cząsteczkowością rea
19649 PICT0008 (7) monomery— polimery j monomery - małe cząsteczki^ zdolne do łączenia się w wi
DSC00288 (10) Transport substancji w komórce □ przez błonę - małe cząsteczki
Błonowe białka transportujące - małe cząsteczki rozpuszczalne w wodzie - - — - - -
501 2 12.8. MAŁE ELEKTROWNIE WODNE 12.8. MAŁE ELEKTROWNIE WODNE Rys. 12.25. Schematy ideowe przykład
IMG?94 monomery —• polimery jmonomeiy - małe cząstecdM zdolne do łączenia się w większą j polimery -
3. Miasto przedindiistrialne Małe ośrodki miejskie (do 20 tys. mieszkańców), Miasta funkcjonowały w
Kometa., małe ciało niebieskie poruszające sią w układzie planetarnym, które na krótko pojawia sią w
więcej podobnych podstron