
PCR,
Sekwencjonowa
nie

Reakcja PCR
(Polymerase Chain Reaction)
•Najczulsza
metoda
służąca
do
wykrywania
i powielania fragmentów DNA o
określonej sekwencji.
•Pozwala na wykrycie i amplifikację
pojedynczej
kopii
interesującej
sekwencji
z preparatu DNA całkowitego.

Składniki mieszaniny
reakcyjnej
• Termostabilna polimeraza DNA
• Para syntetycznych
oligonukleotydów jako startery
syntezy DNA
• Trójfosforany deoksynukleotydów
• Kationy dwuwartościowe (Mg
2+
)
• Bufor o pH 8.3-8.8
• Kationy jednowartościowe (K
+
)
• Matryca DNA
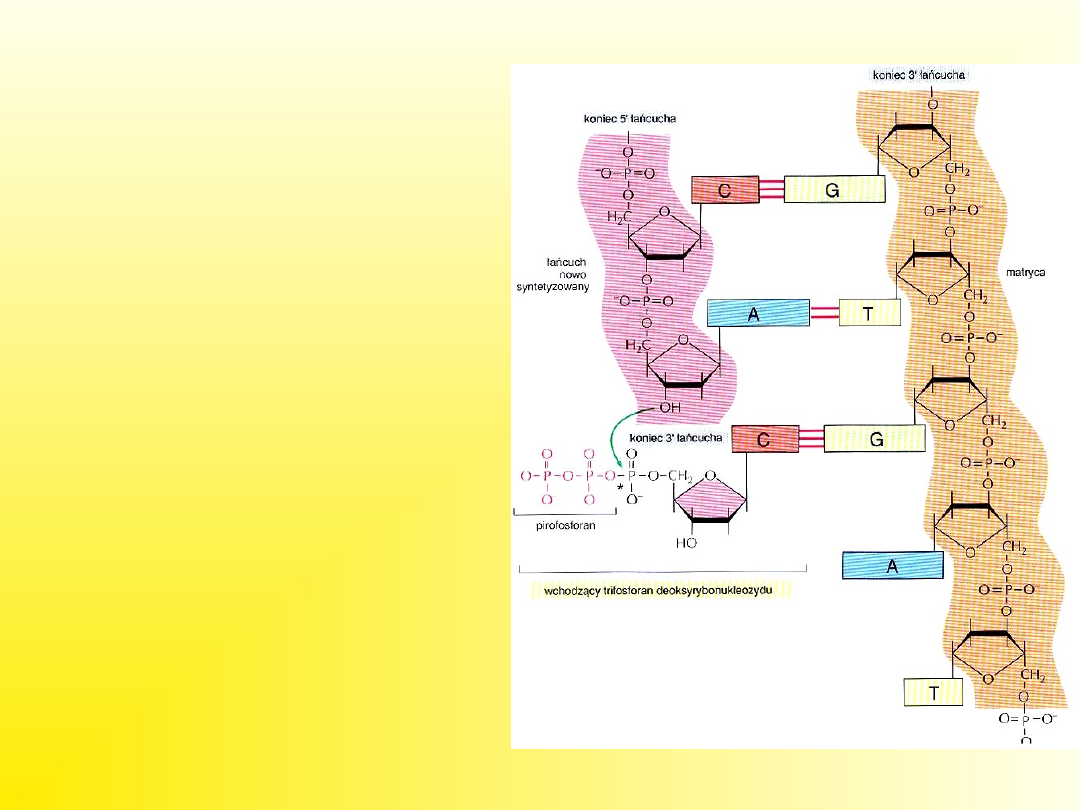
Polimeraza DNA
przyłącza
nukleotydy
do końca 3’ – tzn.
katalizuje
reakcję
wydłużania
w kierunku 5’→3’

Główne etapy reakcji PCR:
•Denaturacja wyjściowych cząsteczek
DNA
•Wiązanie starterów (annealing)
•Wydłużanie łańcucha (extension)
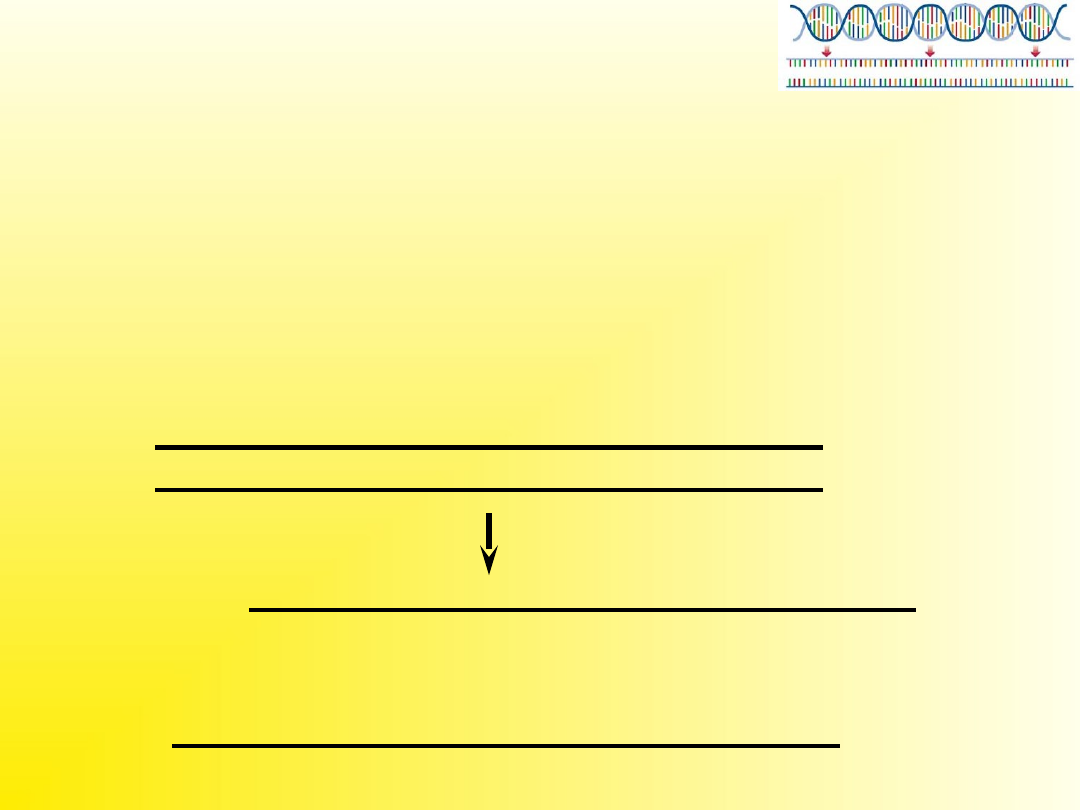
Denaturacja
Denaturacja – rozdzielenie nici DNA pod wpływem
temperatury
Temperatura denaturacji zależy od składu zasad
fragmentu który chcemy powielić i odporności
polimerazy na wysoką temperaturę.
Najpopularniejsza polimeraza używana standartowo w
PCR – Taq wytrzymuje temperaturę 94-95 stopni C.
Przy fragmentach bogatych w GC używa się polimeraz
bardziej opornych na wysoką temperaturę (np. Pwo,
Pfu).
5’
3’
5’
3’
5’
3’
5’
3’
94
0
C
Pierwszy cykl
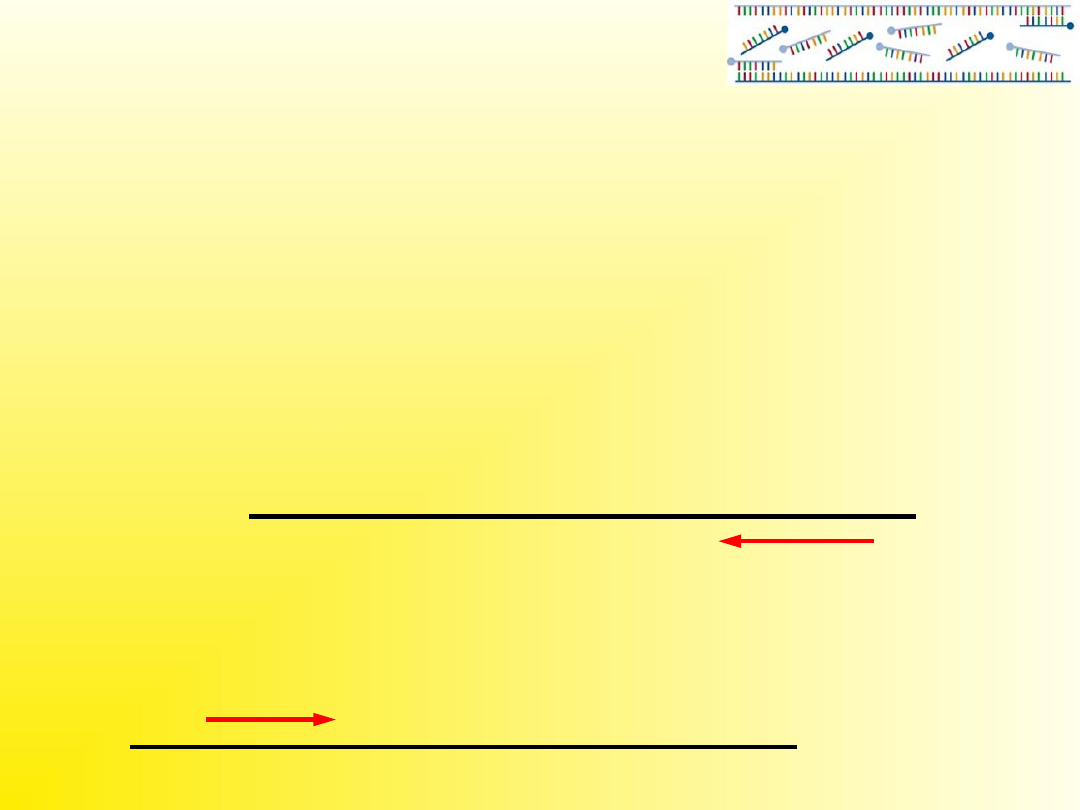
Annealing
(przyłączanie starterów)
Przyłączanie starterów (
Annealing)
Temperatura:
za wysoka – słaba wydajność
za niska - niespecyficzne powielanie
Stosuje się temperaturę około 3-5 stopni poniżej
oszacowanej temperatury topnienia.
Praktyczny wzór na temperaturę topnienia dla starterów
o długości około 20 nukleotydów:
T=2x(A+T) + 4(G+C)
5’
3’
3’
5’
5’
3’
3’
5’
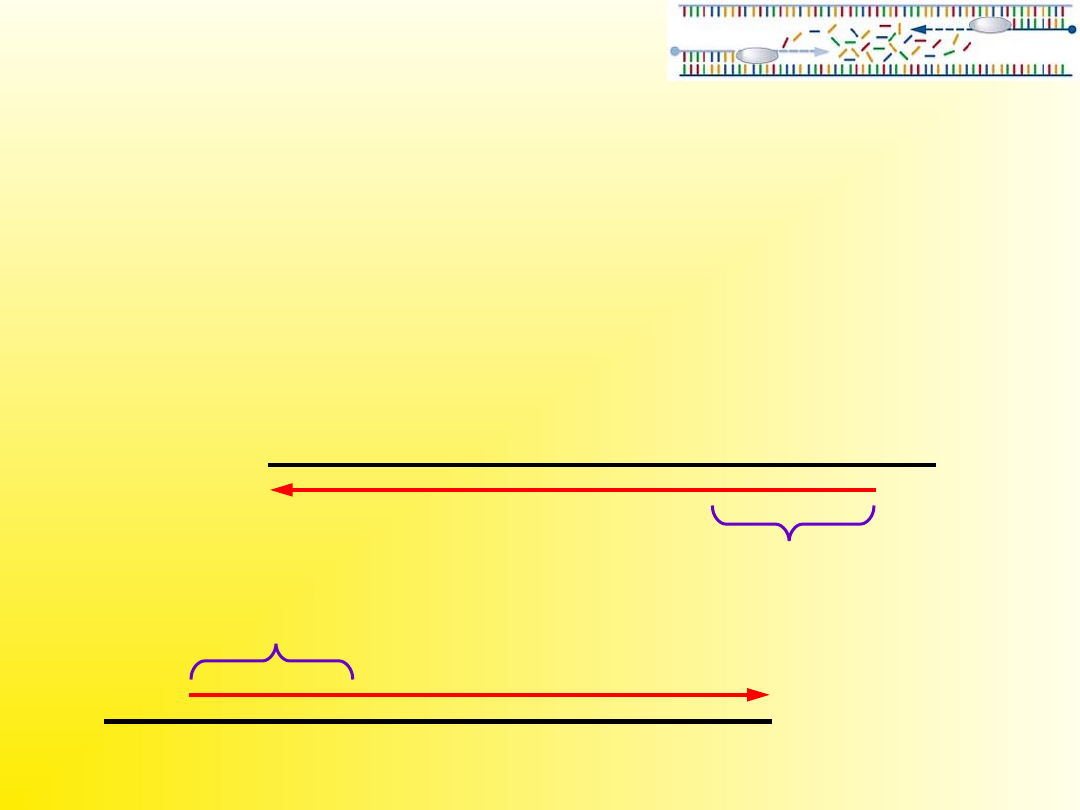
Extension (wydłużanie łańcucha)
Wydłużanie łańcucha (Extension)
Temperatura tej części cyklu zależy od właściwości polimerazy.
Dla polimerazy Taq optymalna jest temperatura 72-78 stopni.
Szybkość reakcji w optymalnych warunkach dla polimerazy Taq
wynosi około 2000 nukleotydów/min.
Do pełnej syntezy badanego fragmentu czas trwania tego etapu
wyznaczamy zakładając połowę optymalnej prędkości.
5’
3’
3’
5’
Sekwencja startera
5’
3’
5’
3’
Sekwencja startera
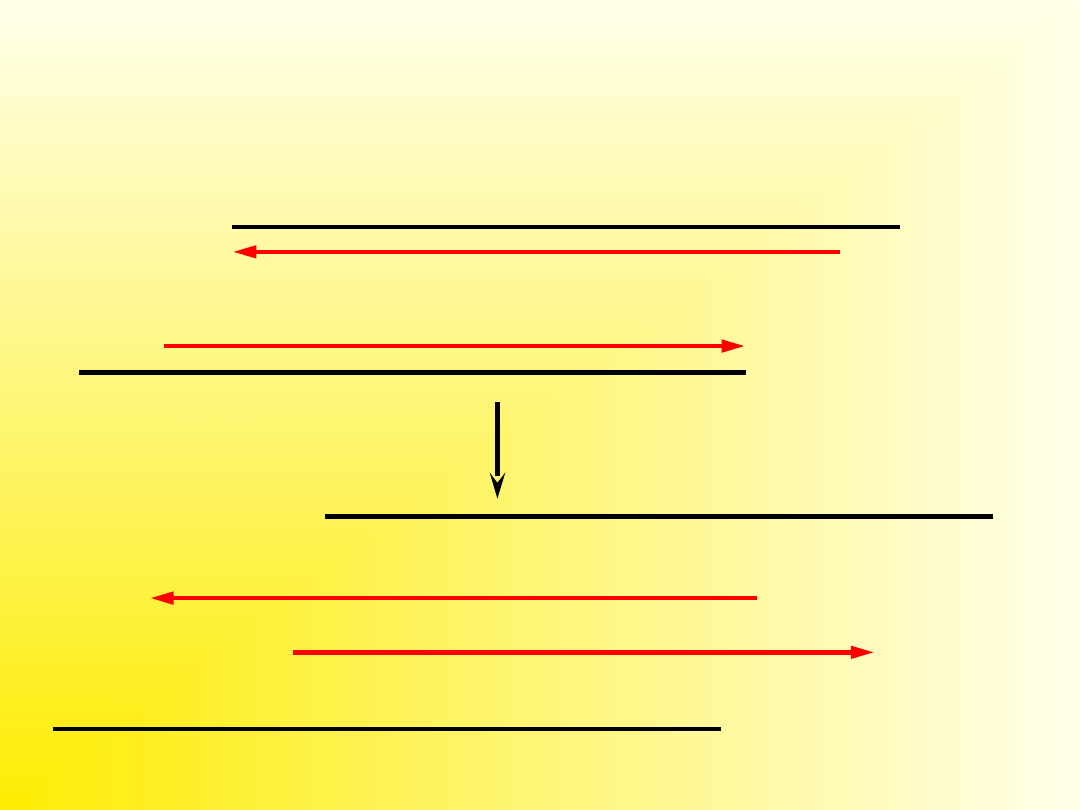
Drugi cykl - denaturacja
5’
3’
5’
3’
3’
5’
5’
3’
94
0
C
5’
3’
3’
5’
5’
3’
5’
3’
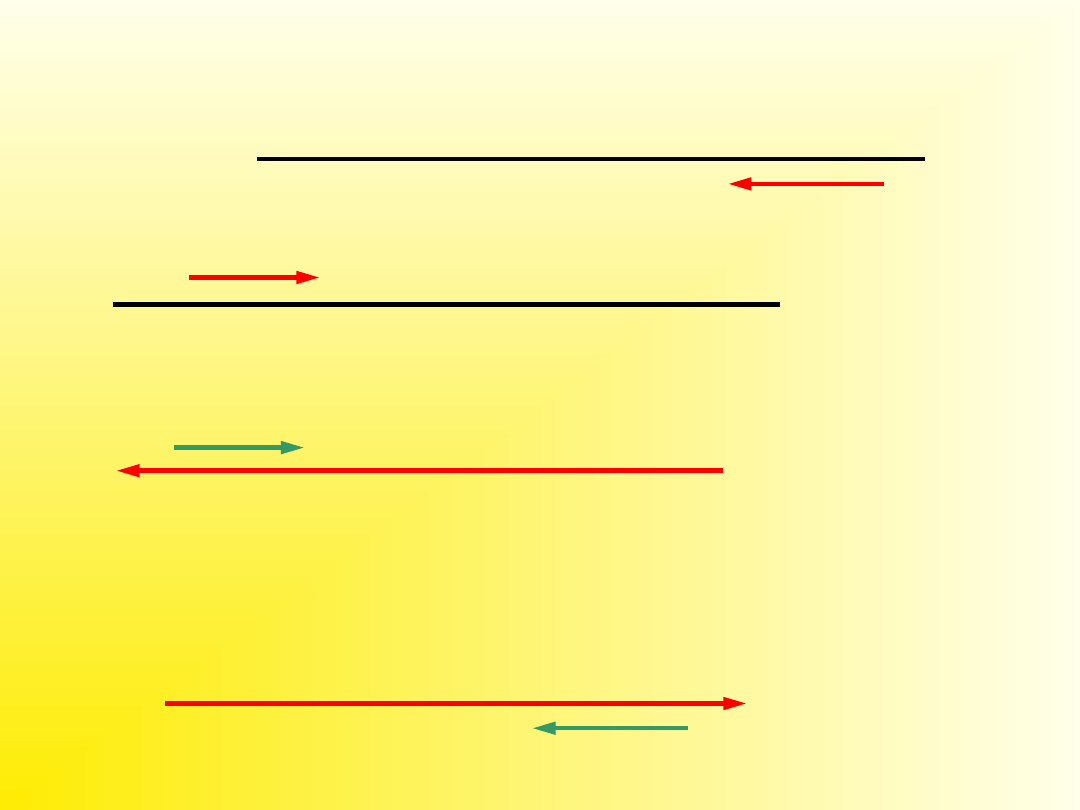
Przyłączanie starterów
5’
3’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
3’
5’
3’
5’
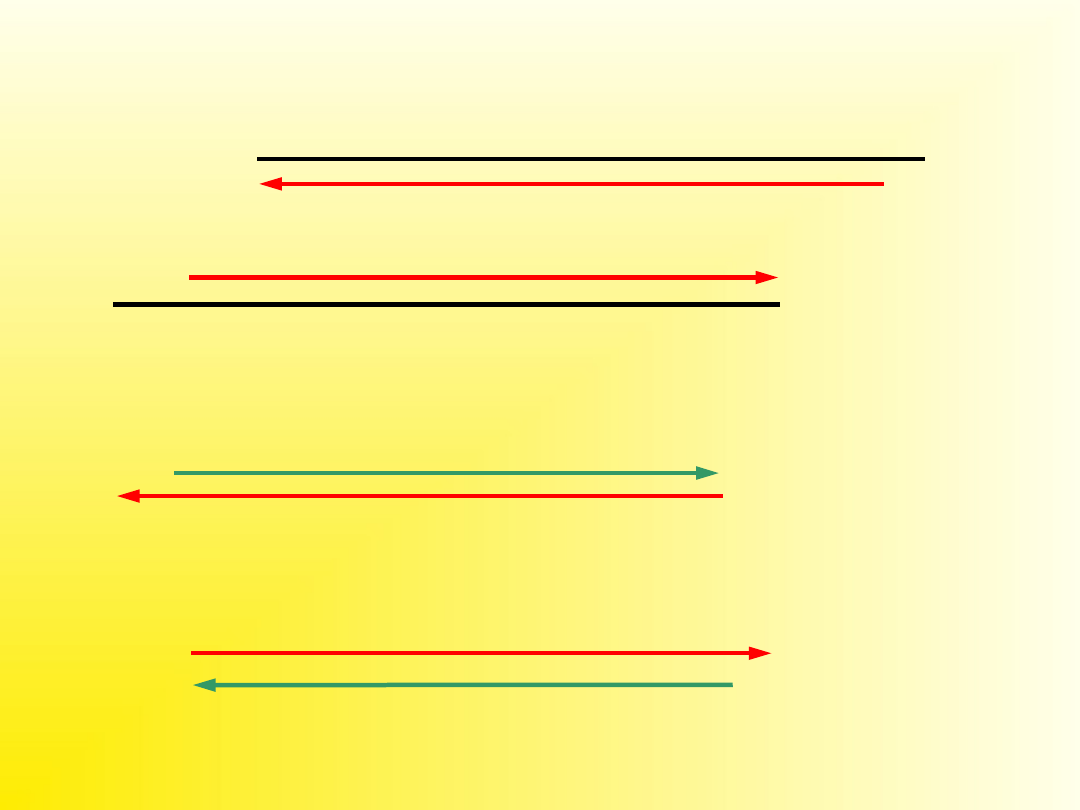
Wydłużanie nici (elongacja)
5’
3’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
3’
5’
3’
5’
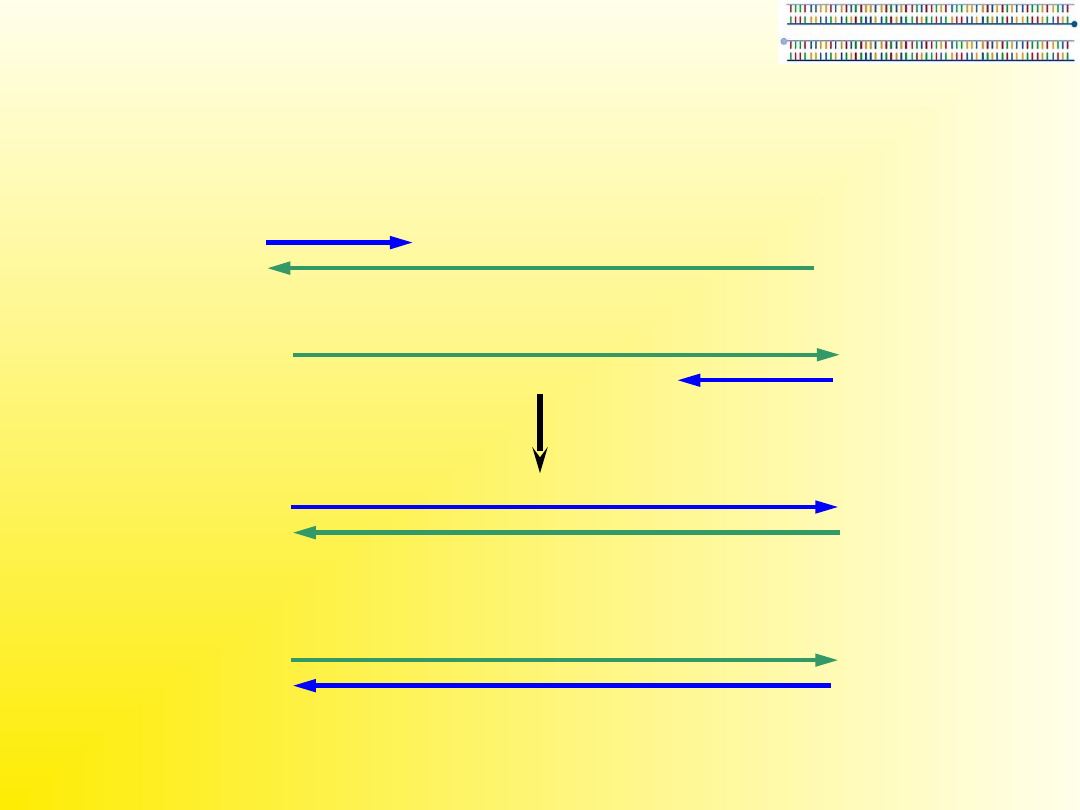
W trzecim cyklu produkowany jest
już właściwy produkt
5’
3’
3’
5’
3’
5’
3’
5’
5’
3’
3’
5’
5’
3’
3’
5’
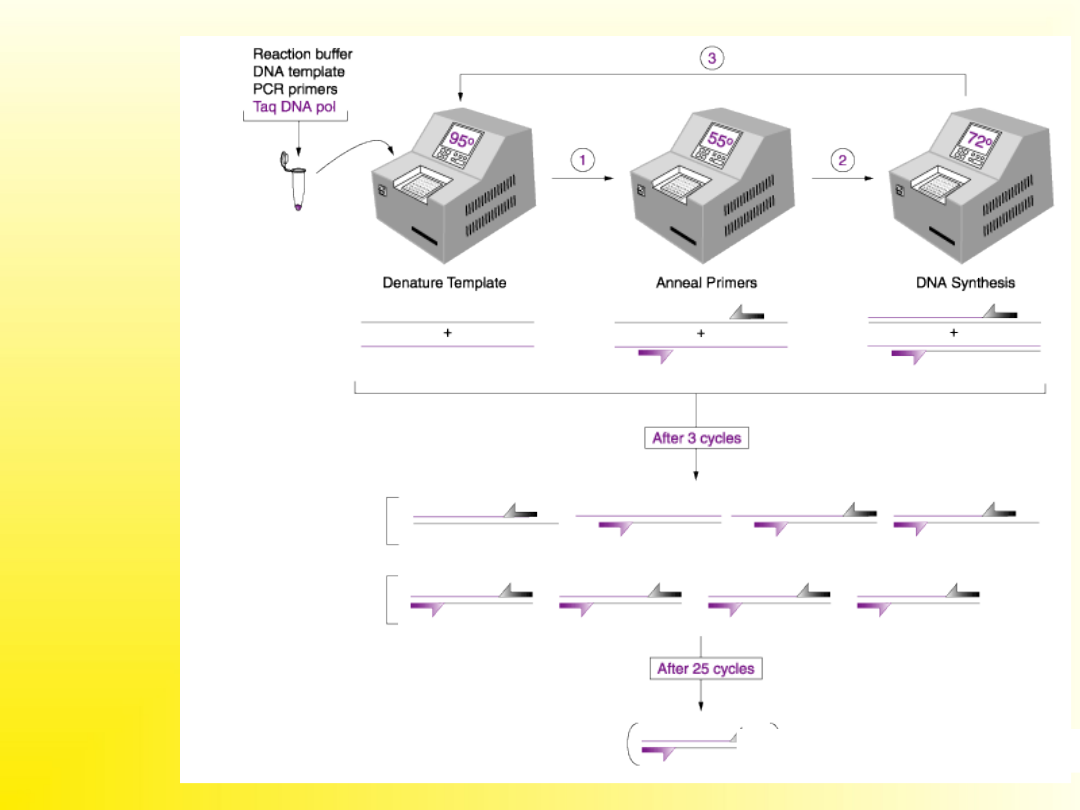
PCR
>1 000 000 cząsteczek
Wzros
t
liniow
y
Wzrost
wykładnicz
y

Termostabilne polimerazy
DNA różnią się między sobą
odpornością
na wysoką temperaturę,
dokładnością wbudowywania
nukleotydów
i prędkością reakcji
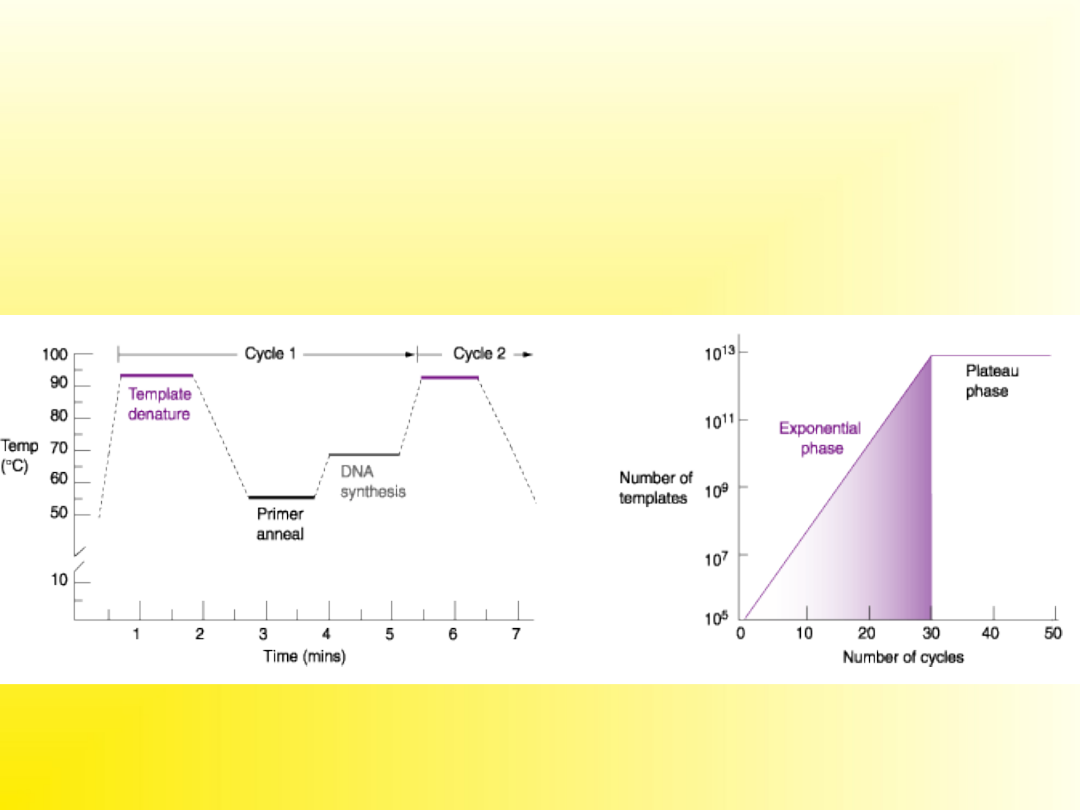
Temperatura jest najważniejszym
fizycznym czynnikiem wpływającym
na wynik w reakcji PCR

PCR
Startery
1. długość 18-25 nukleotydów.
2. skład 40-60% GC.
3. nie powinny zawierać odwróconych
powtórzeń.
4.
temperatura
topnienia
podwójnoniciowej
cząsteczki
tworzonej
przez oba startery z matrycą powinna być
podobna.
5. na końcu 3’ powinny mieć G lub C,
ale nie więcej niż jeden nukleotyd.
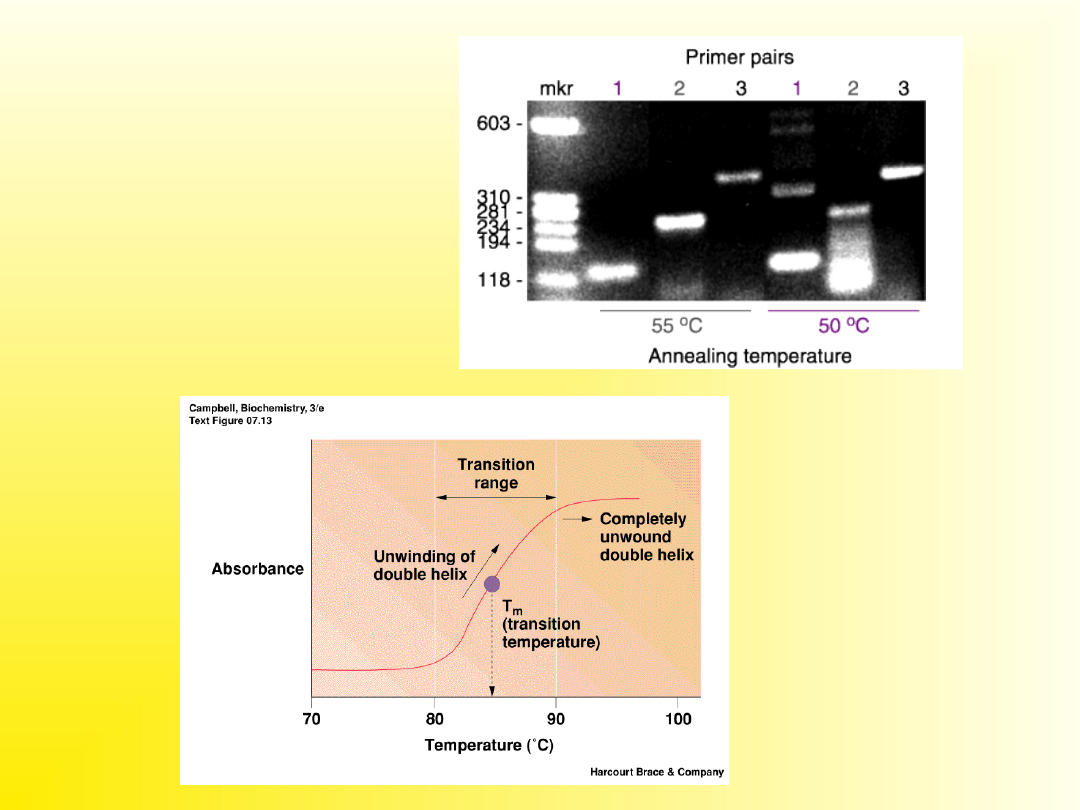
Zbyt niska
temperatura
wiązania starterów
prowadzi do
powstania
niespecyficznych
produktów PCR

Różne odmiany
metody PCR
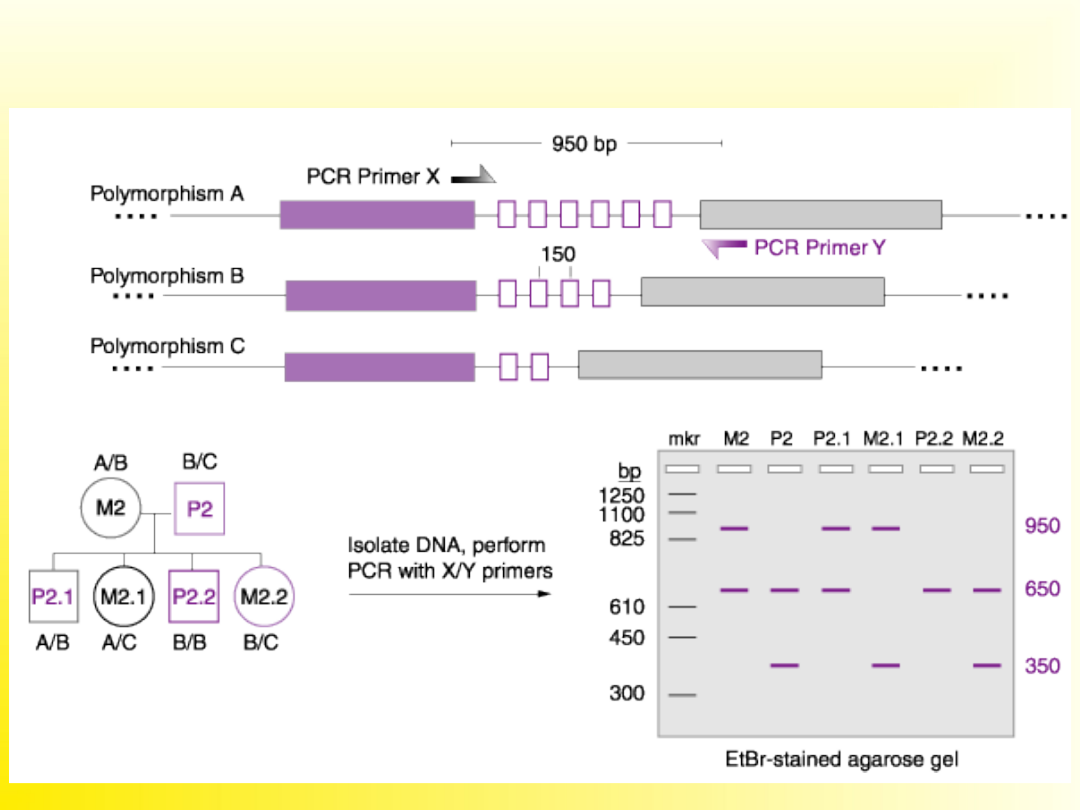
Analiza polimorfizmu powtarzalnych sekwencji DNA
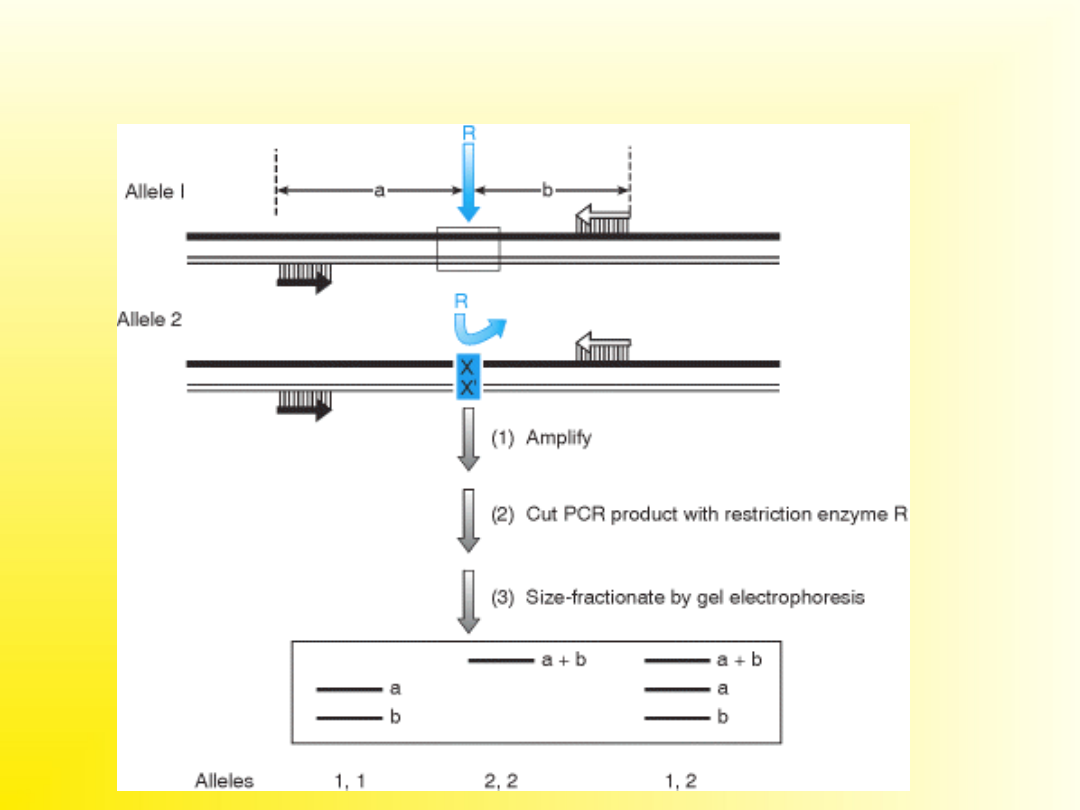
RFLP PCR – polimorfizm długości
fragmentów restrykcyjnych
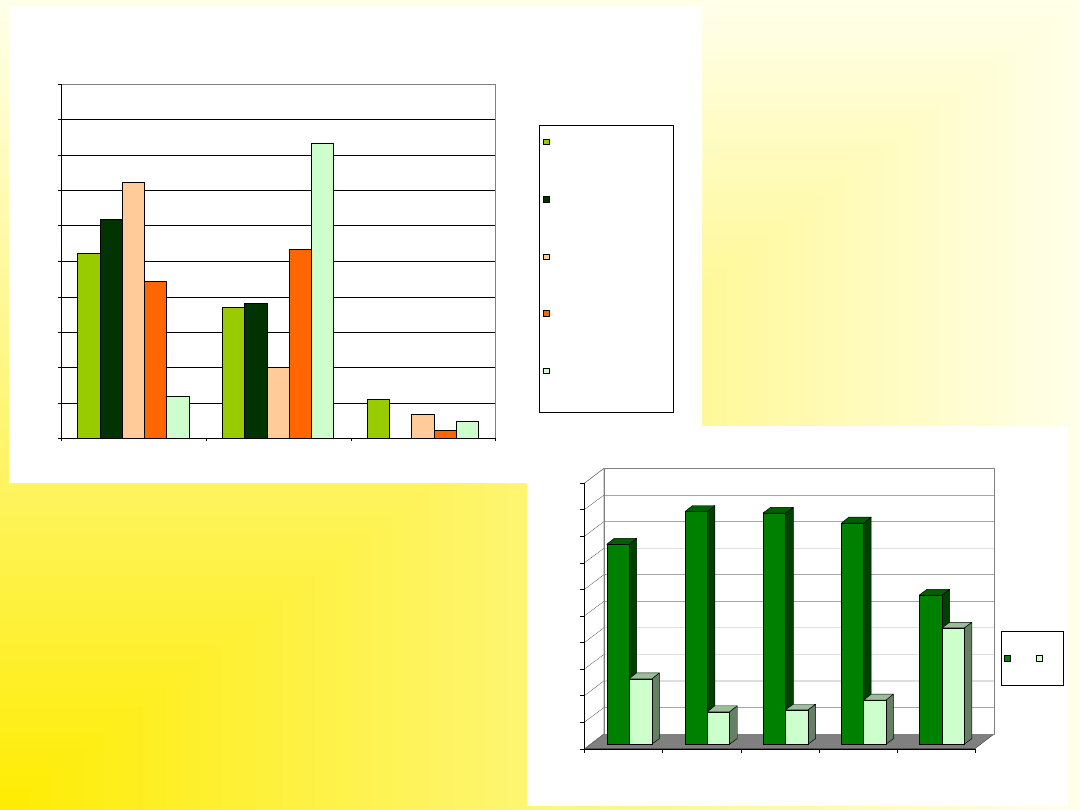
POLIMORFIZM W KODONIE 145 GENU APE1,
ROZKŁAD CZĘSTOŚCI GENOTYPÓW
0
10
20
30
40
50
60
70
80
90
100
TT(Asp/Asp)
TG(Asp/Glu)
GG(Glu/Glu)
GENOTYPY
%
B
A
D
A
N
E
J
P
O
P
U
L
A
C
JI
CHORZY Z
NOWOTWORAMI
REGIONU GŁOWY I SZYI
CHORZY Z RAKIEM PŁUC
CHORZY Z RAKIEM
SZYJ KI MACICY
CHORZY Z RAKIEM
J ELITA GRUBEGO
ZDROWI
0
10
20
30
40
50
60
70
80
90
100
CHORZY NA
NOWOTWORY
REGIONU GŁOWY I
SZYI
CHORZY NA RAKA
PŁUC
CHORZY NA RAKA
SZYJ KI MACICY
CHORZY NA
NOWOTWORY
J ELITA GRUBEGO
ZDROWI
ROZKŁAD CZĘSTOŚCI ALLELI GENU 145 APE 1
Asp
Glu
Porównanie
częstości
genotypów w
stanach
patologicznych
wskazuje na
istnienie korelacji
pomiędzy pewnymi
wariantami
genetycznymi
a chorobami
Pewne allele tego samego
genu mogą być
odpowiedzialne
za wystąpienie stanów
patologicznych lub
skłonności do określonych
patologii
A. Gdowicz 2005

„Odwrócony” PCR
Używany do powielenia fragmentu o nieznanej
sekwencji, występującego w sąsiedztwie znanej
sekwencji DNA
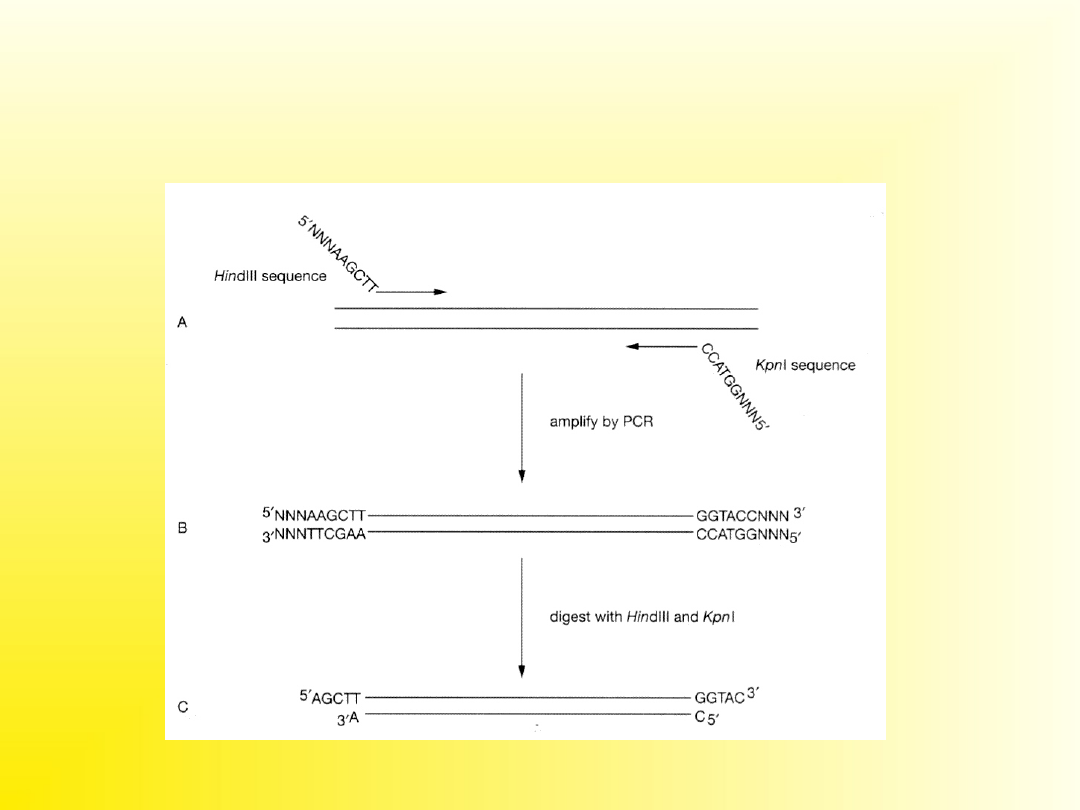
PCR jako źródło DNA do
klonowania

Polimeraza Taq dodaje często
dodatkowy nukleotyd (A) do
końca 3’
Do
klonowania
można
wykorzystać
tzw. wektory T, które posiadają lepki
koniec
–
pojedynczy
nukleotyd
tymidynowy, albo usunąć dodatkowy
nukleotyd A.
Można też użyć polimerazy DNA z faga
T4,
lub
polimerazy
Pfu,
a
następnie
wklonować wstawkę w zdefosforylowany
wektor o tępych końcach.

Multiplex PCR
• Wykorzystywana więcej niż jedna
para starterów.
• Pozwala
na
powielenie
kilku
fragmentów DNA w jednej reakcji.
• Warunki reakcji muszą być jednakowe
dla
wszystkich
par
starterów,
a
produkty
powinny
różnić
się
długością.

Hot start PCR
• Metoda
ułatwiająca
optymalizację
otrzymywania pożądanego fragmentu
DNA.
• Dopiero po ogrzaniu do temperatury
wyższej
niż
temperatura
niespecyficznego
wiązania
starterów
wszystkie
składniki
mieszaniny
reakcyjnej są łączone ze sobą.
• Obecnie taki efekt osiąga się przez
rozdzielenie części mieszaniny reakcyjnej
przy
pomocy
niskotopliwego wosku
(np. Ampliwax PCR Gems)

PCR dla szczególnie długich
fragmentów DNA (long PCR)
• Doskonała jakość matrycy DNA o długości
średnio powyżej 50 kb
• Mieszanina polimeraz o różnych
charakterystykach
• Dłuższe startery (24-34 pz)
• Wyższa temperatura topnienia dla starterów
(najlepiej 63-68
0
C)
• Minimalne czasy denaturacji (12 sekund) aby
uniknąć degradacji DNA
• Znacznie wydłużone czasy syntezy, zwiększające
się jeszcze wraz z ilością cykli

Zagnieżdżony (nested) PCR
Pierwsza para
starterów,
pierwsza reakcja
PCR
Druga para
starterów, druga
reakcja PCR
Mieszaniny polimeraz
• DyNAzyme
–
mieszanina
polimerazy Taq (duża szybkość
reakcji) oraz Tbr (duża dokładność)
• Taq Plus Long PCR System –
mieszanina polimerazy Taq oraz
Pfu (dobra procesywność)
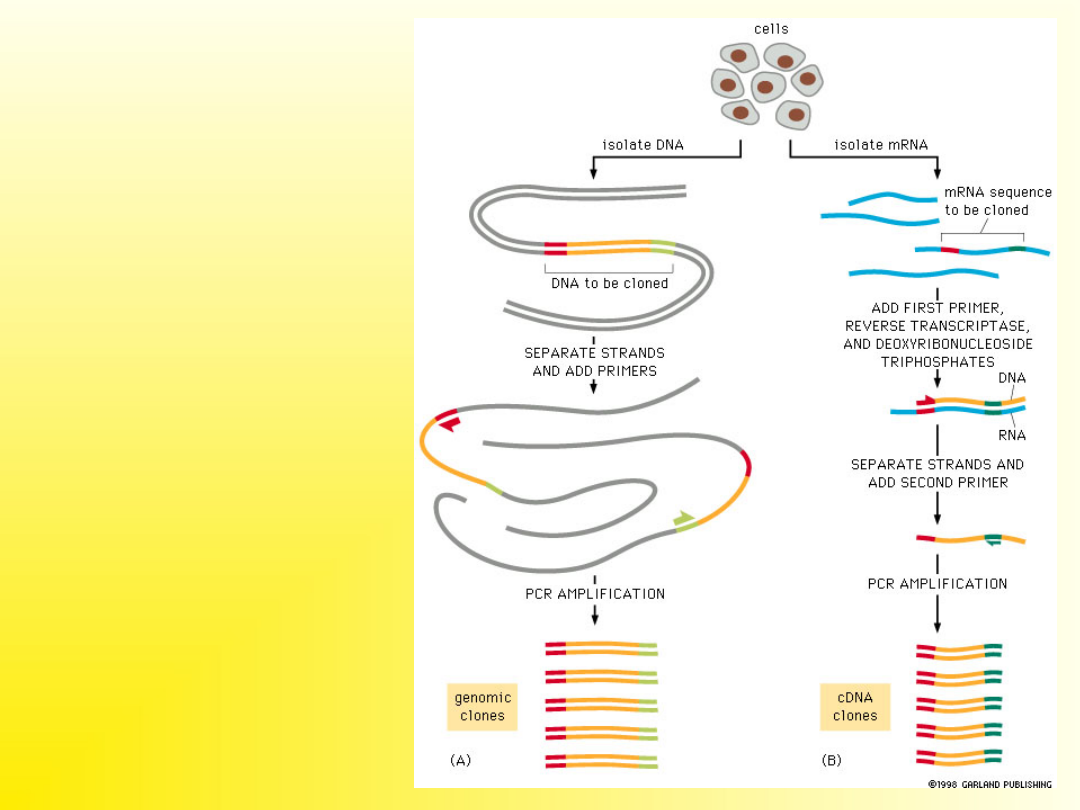
Do powielania
fragmentów
metodą PCR
można użyć
zarówno DNA
jak i RNA

Sekwencjonowanie
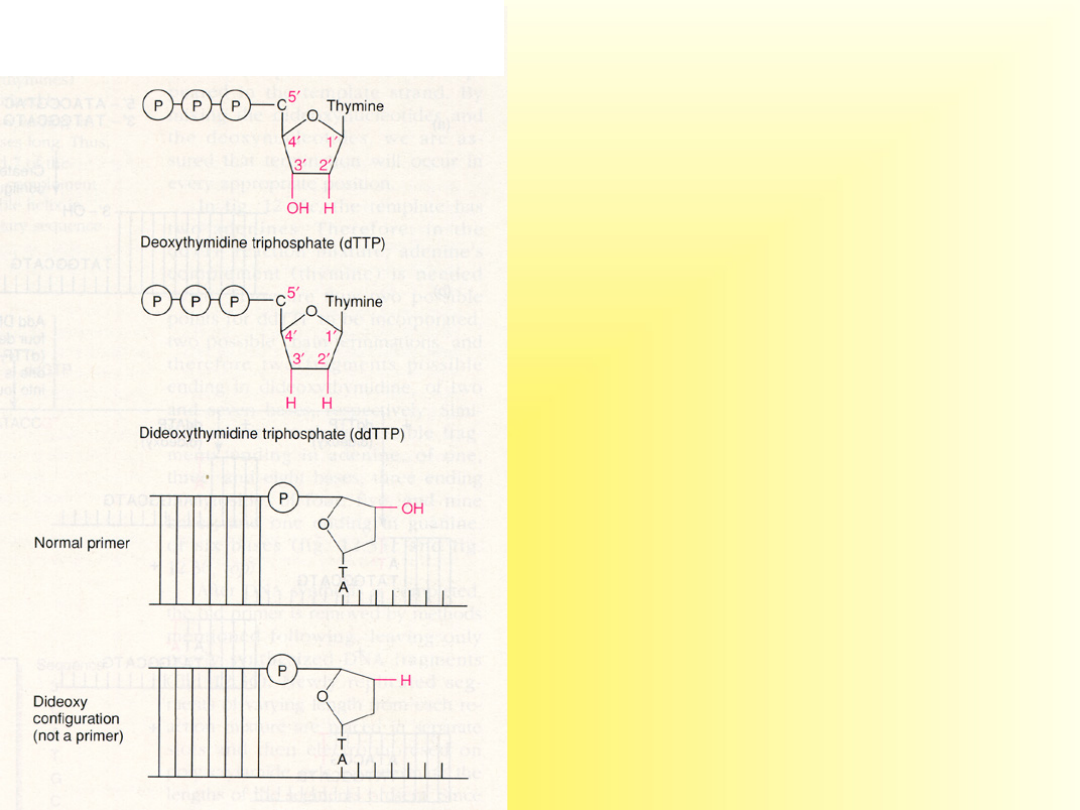
Sekwencjonowanie
DNA
(metoda dideoxy-)
• Jest enzymatyczną reakcją
używaną
w
odczytywaniu
sekwencji nukleotydowej DNA
uprzednio sklonowanego.
• Metoda oparta jest na
użyciu
czterech
zatrzymujących
reakcję
wydłużania
łańcucha
nukleotydów, które w pozycji
3’ mają H zamiast OH (di-
deoxy: ddATP, ddGTP, ddCTP,
ddTTP).
•W klasycznej metodzie każdy
nukleotyd
dodawany
jest
oddzielnie
do
czterech
prowadzonych
równolegle
reakcji replikacji DNA.
•Produkty
tych
czterech
reakcji są rozdzielane metodą
elektroforezy
w żelu poliakrylamidowym
Nukleotydy dideoxy- powodują
zakończenie replikacji DNA
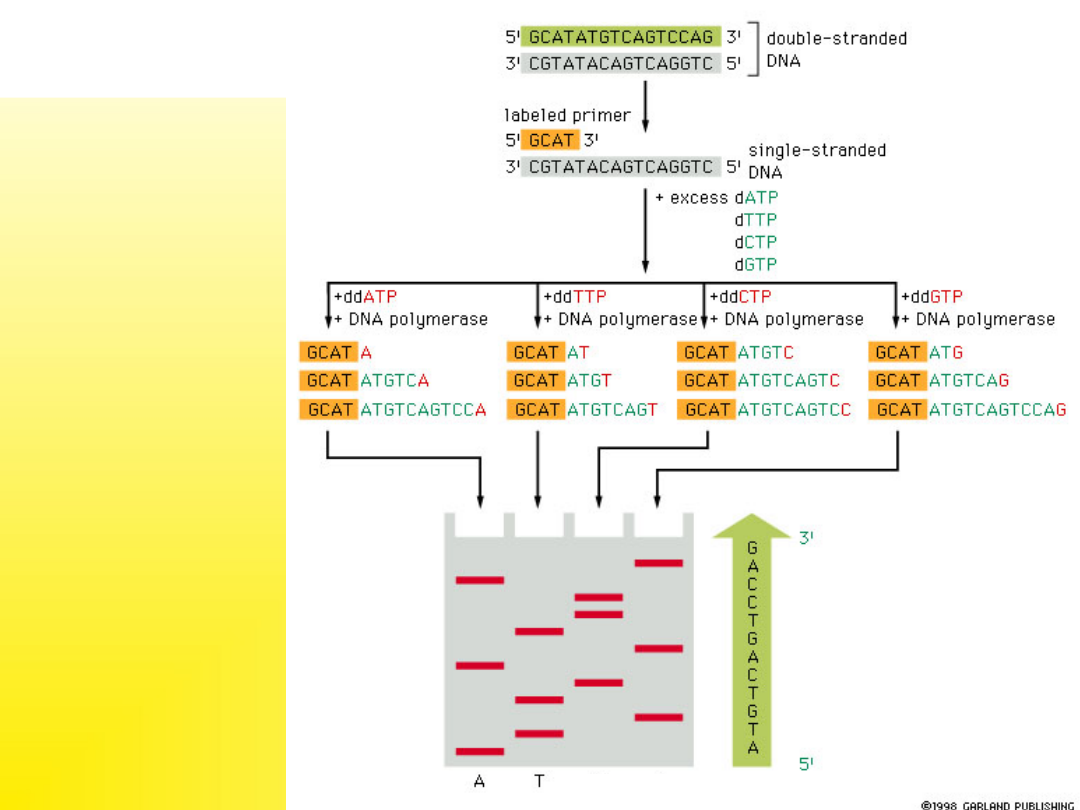
Sekwencjonowanie
DNA
C
G
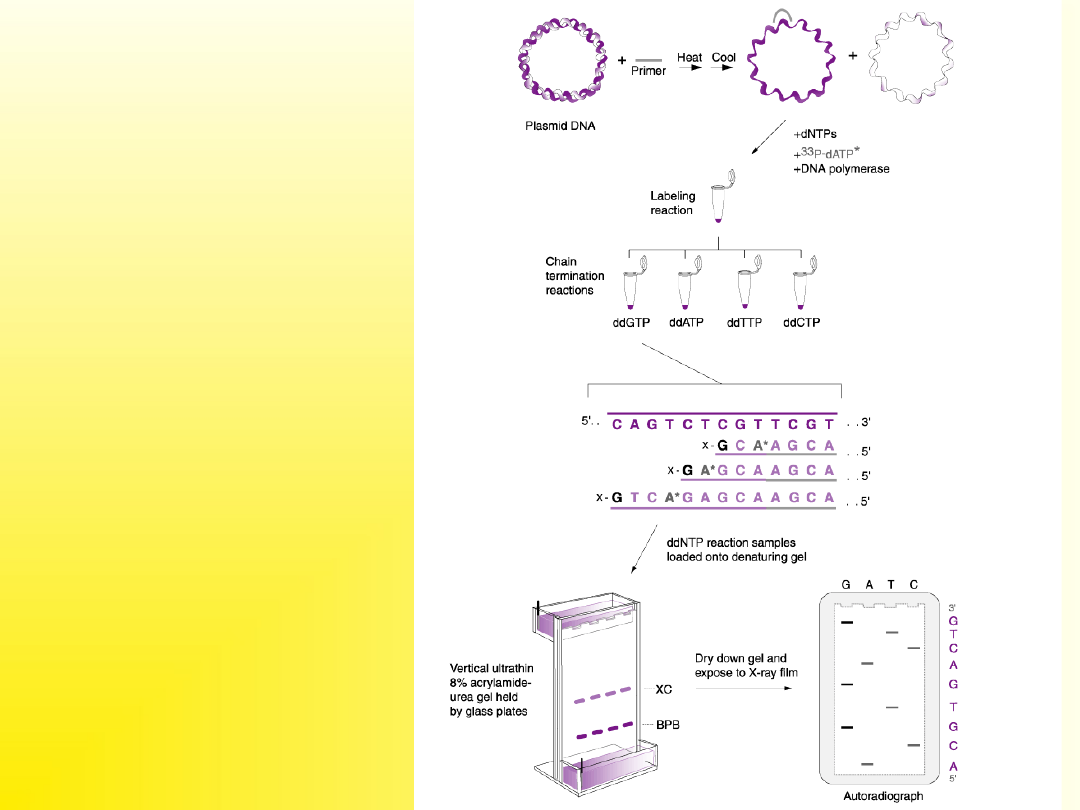
Znakowanie
sekwencjonowane
go
fragmentu
pojedynczym
nukleotydem

Znakowanie końcowego
nukleotydu (dideoxy)

Odczytywanie sekwencji DNA

Metoda chemiczna sekwencjonowania polega na
wykorzystaniu specyficznych wobec
poszczególnych zasad reakcji chemicznych, co
pozwala na identyfikację miejsca występowania
danej zasady
Zasada
Specyficzna modyfikacja
G
Metylacja N7 dimetylosiarczanem w pH 8.0
sprawia, że wiązanie C8-C9 staje się specyficznie
wrażliwe na rozkład przy pomocy zasady.
A+G
Mrówczan piperydyny w pH 2.0 osłabia wiązania
glikozydowe puryn, co powoduje depurynację
C+T
Hydrazyna otwiera pierścień pirymidynowy, który
następnie recyklizuje w formę podatną na
usunięcie
C
W obecności 1.5 M NaCl jedynie cytozyna reaguje
specyficznie z hydrazyną
A>C
1.2 N NaOH w temp. 90
0
C powoduje częste
przecinanie reszt A i rzadsze - C
Piperydyna przecina łańcuch fosfocukrowy w miejscu modyfikacji zasady
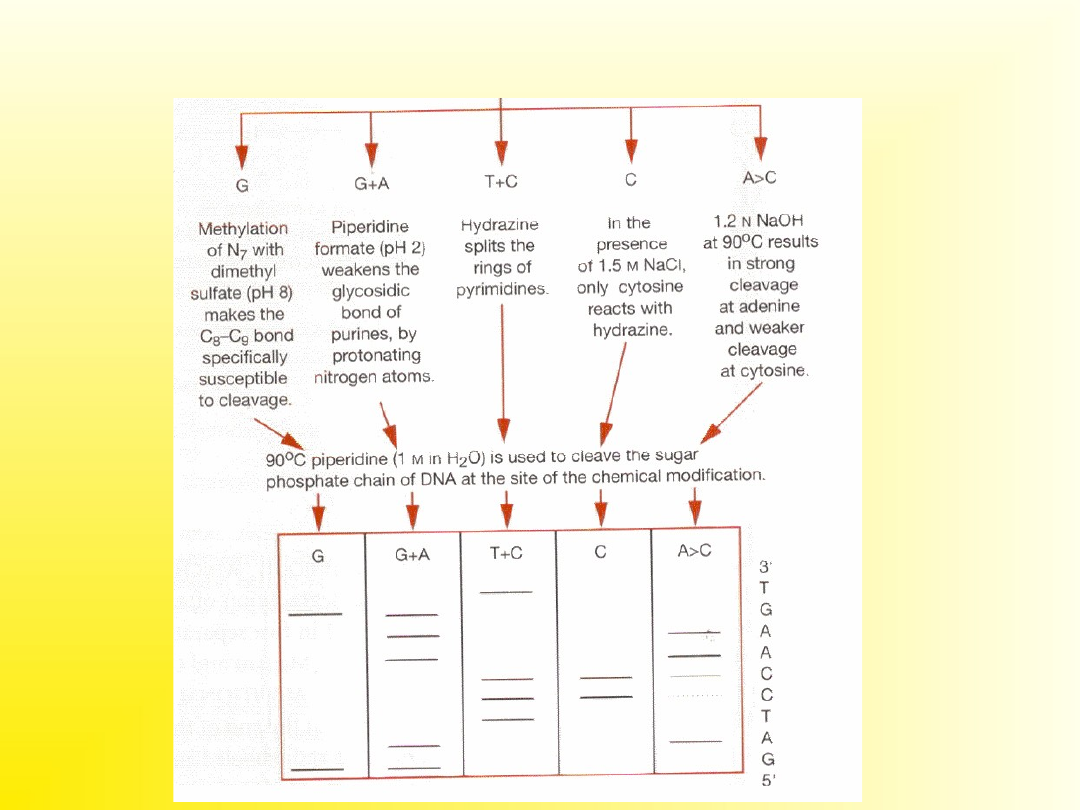
Odczytywanie sekwencji
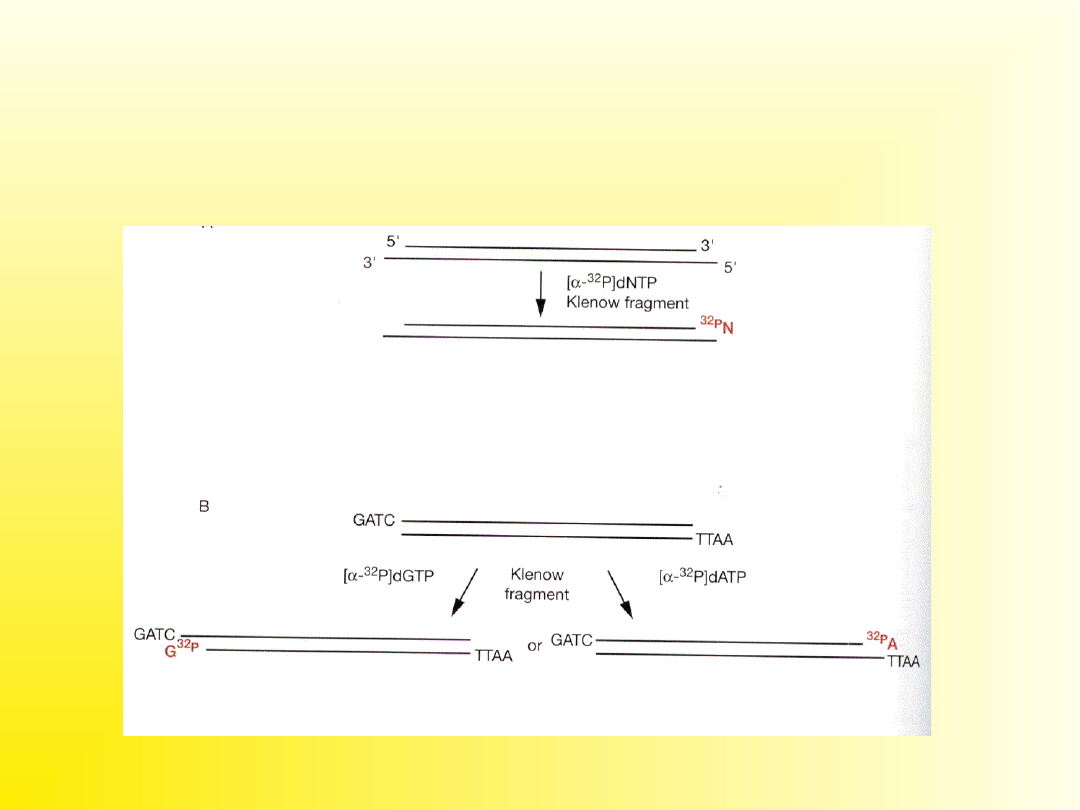
Specyficzne znakowanie jednej
nici DNA przy użyciu fragmentu
Klenowa polimerazy I
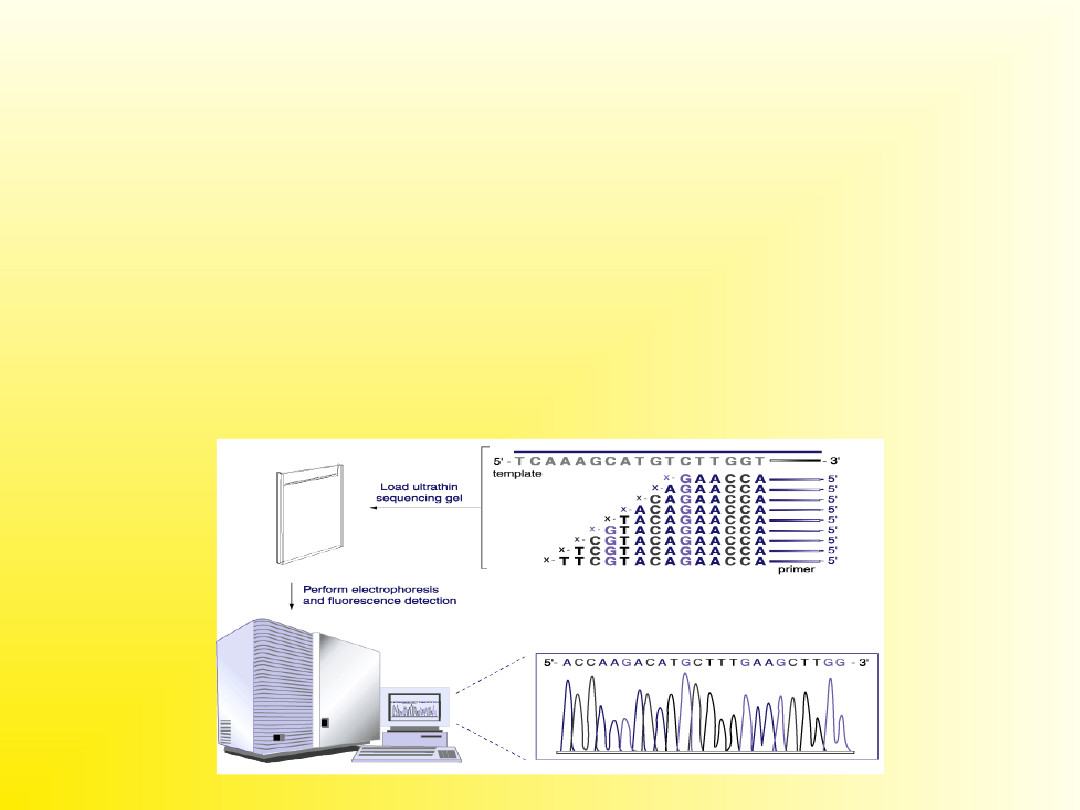
Sekwencjonowanie automatyczne
W systemie tym stosuje się nukleotydy dideoxy lub startery
znakowane czterema różnymi znacznikami fluorescencyjnymi.
Można stosować sekwencjonowanie rozpoczynające się od jednego
startera
i zatrzymujące się na znakowanych nukleotydach dideoxy.
Wszystkie cztery znakowane nukleotydy dideoxy występują wtedy
w jednej mieszaninie.
Inną możliwością jest stosowanie znakowanych starterów,
prowadzenie czterech niezależnych reakcji, a następnie rozdział na
jednej ścieżce żelu. Po reakcji mieszanina nanoszona jest na żel
poliakrylamidowy,
a elektroforeza prowadzona jest w aparacie wyposażonym w
detektor promieniowania.
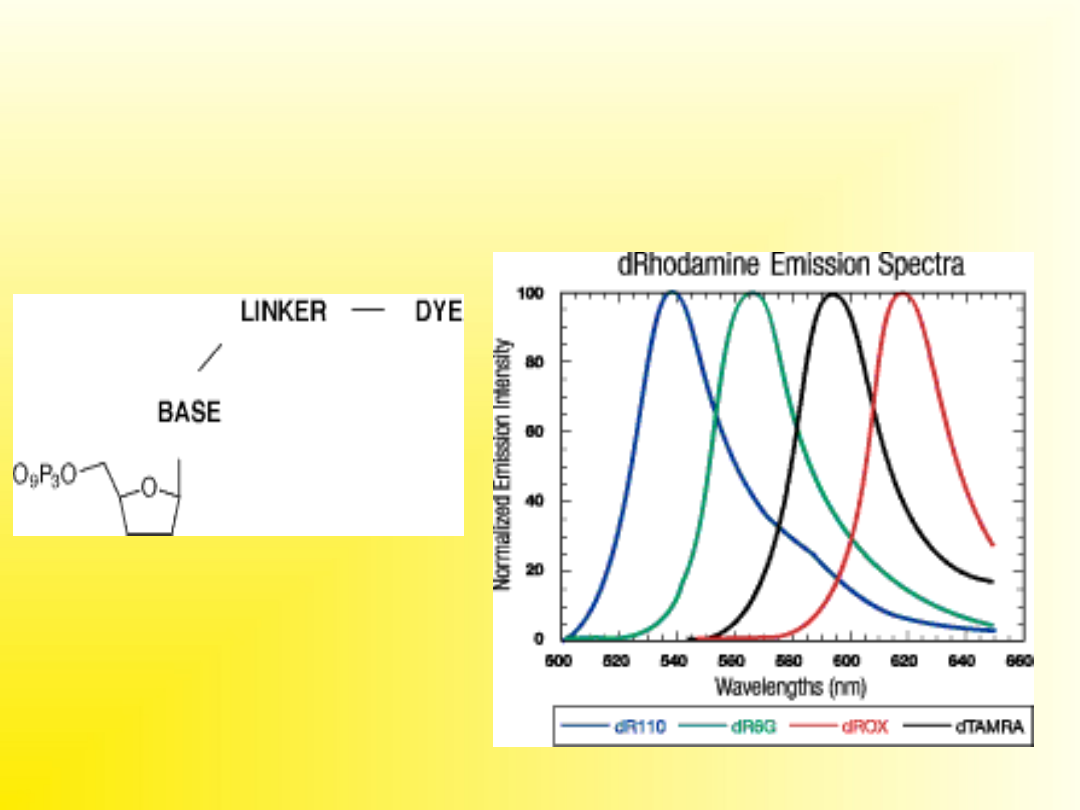
System
ABI
(Applied
Biosystems
Incorporated)
używa
czterech
znakowanych
fluorescencyjnymi
barwnikami
ddNTPs
i polimerazy Taq
(AmpliTaq).
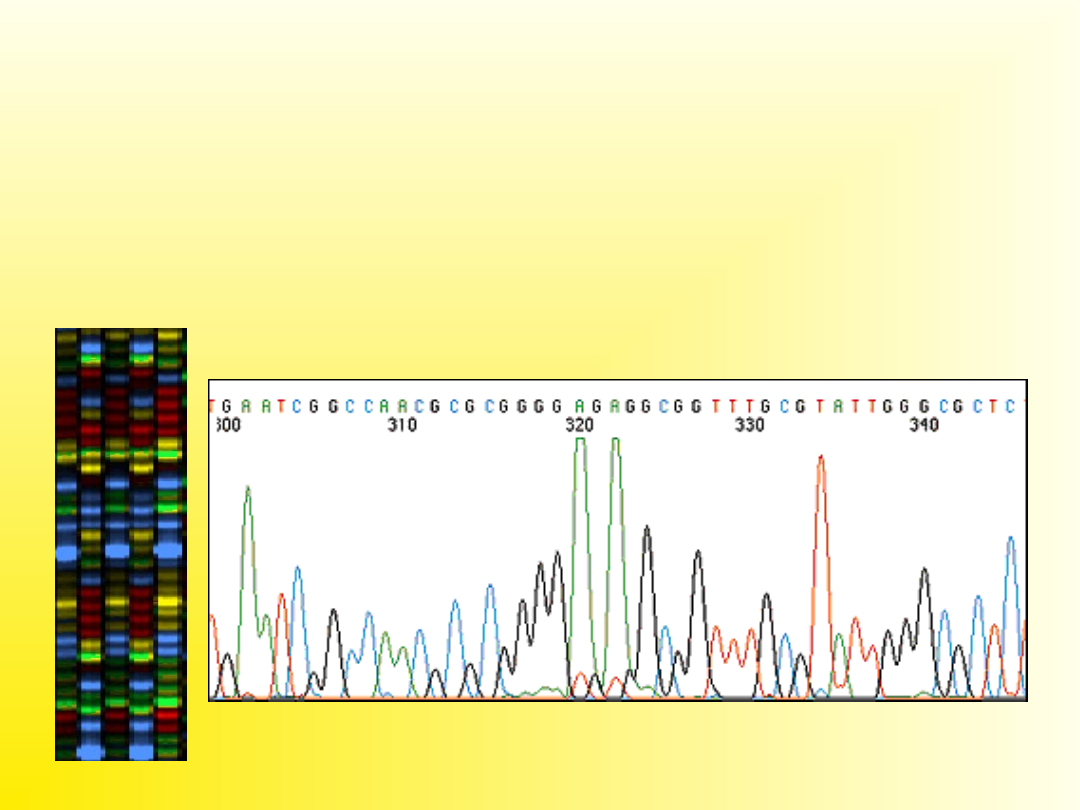
Wzbudzanie fluorochromów i detekcja emisji
mają miejsce w określonej pozycji w pobliżu
dolnej części żelu. Dane są odczytywane na
podstawie spektrum emisyjnego kolejnych pasm
mijających
detektor
w czasie elektroforezy. Dane przekazywane są
do komputera podłączonego do urządzenia
.
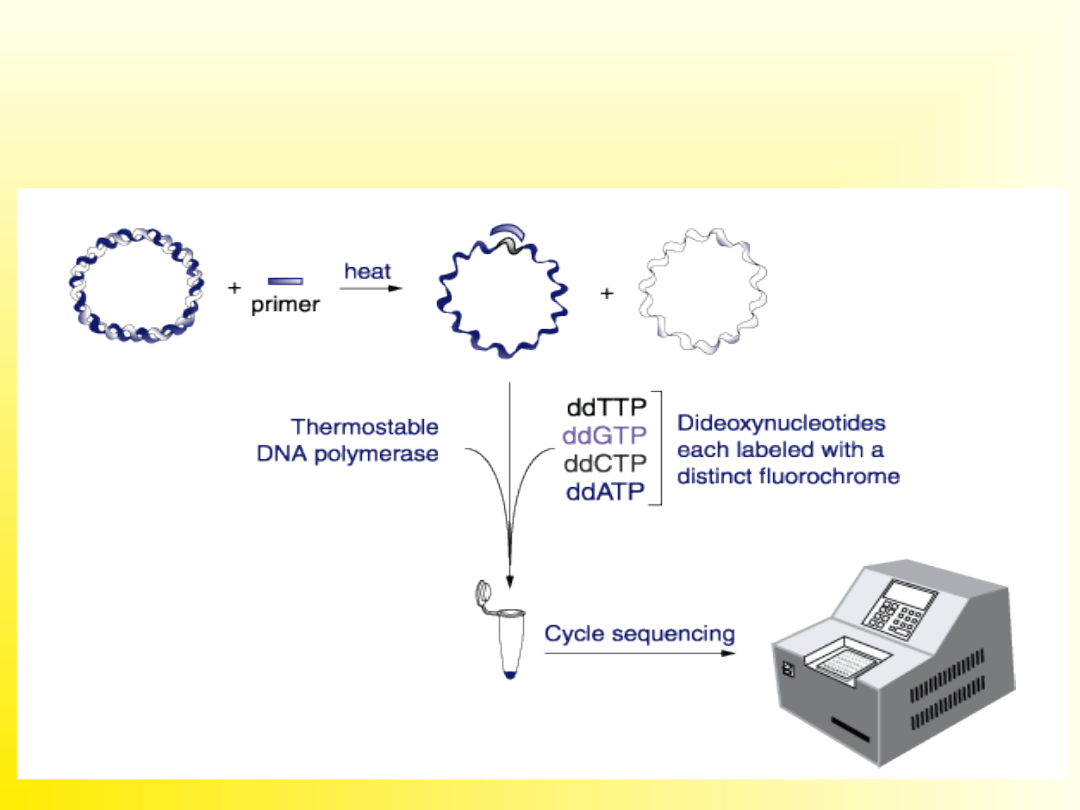
Sekwencjonowanie cykliczne
(połączenie metody PCR i
sekwencjonowania) pozwala
na liniową amplifikację sygnału

Enzymy używane
do sekwencjonowania
Sekwenaza
-
zmodyfikowana
wersja
polimerazy bakteriofaga T7, nie mająca
aktywności 3’-5’ egzonukleazy. Wydajne
włączanie
dideoksy-nukleotydów
zapewnia
obecność
tyrozyny
w pozycji 667 enzymu.
Termostabilne zmodyfikowane polimerazy
- AmpliTaqFS (fluorescent sequencing)
Taquenase
ThermoSequenase

Budowa polimeraz wykorzystywanych
w sekwencjonowaniu
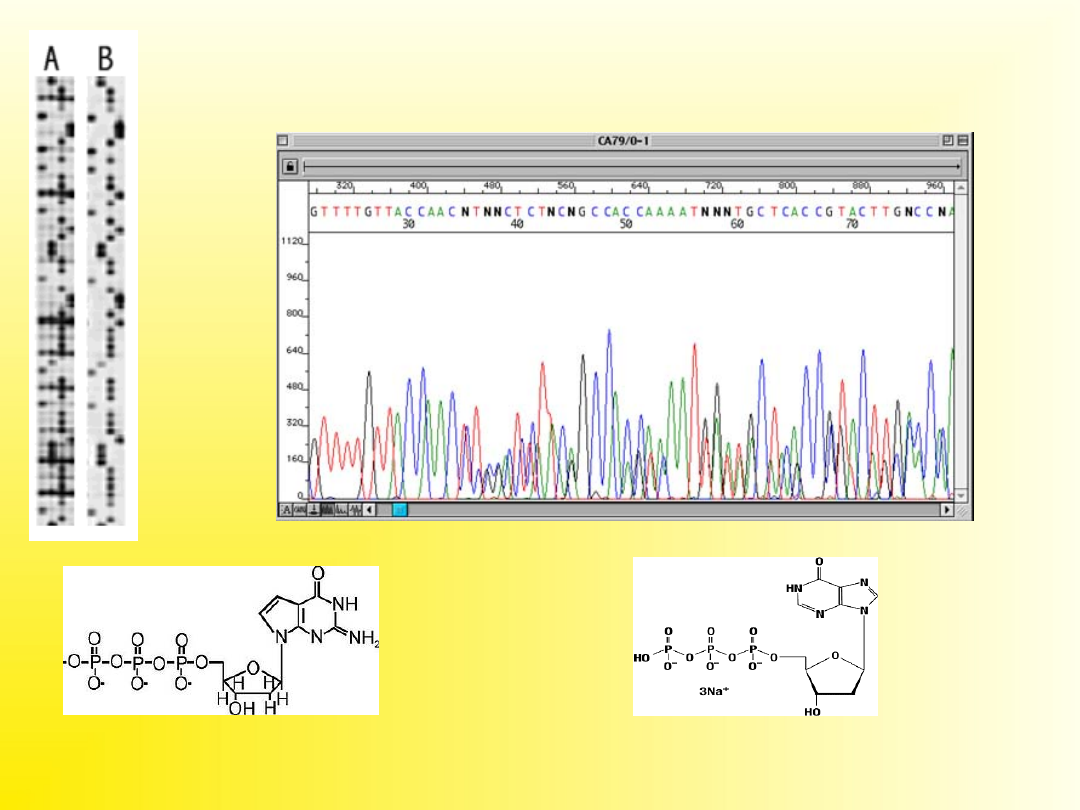
Kompresja w żelu
7-deaza-dGTP
dITP
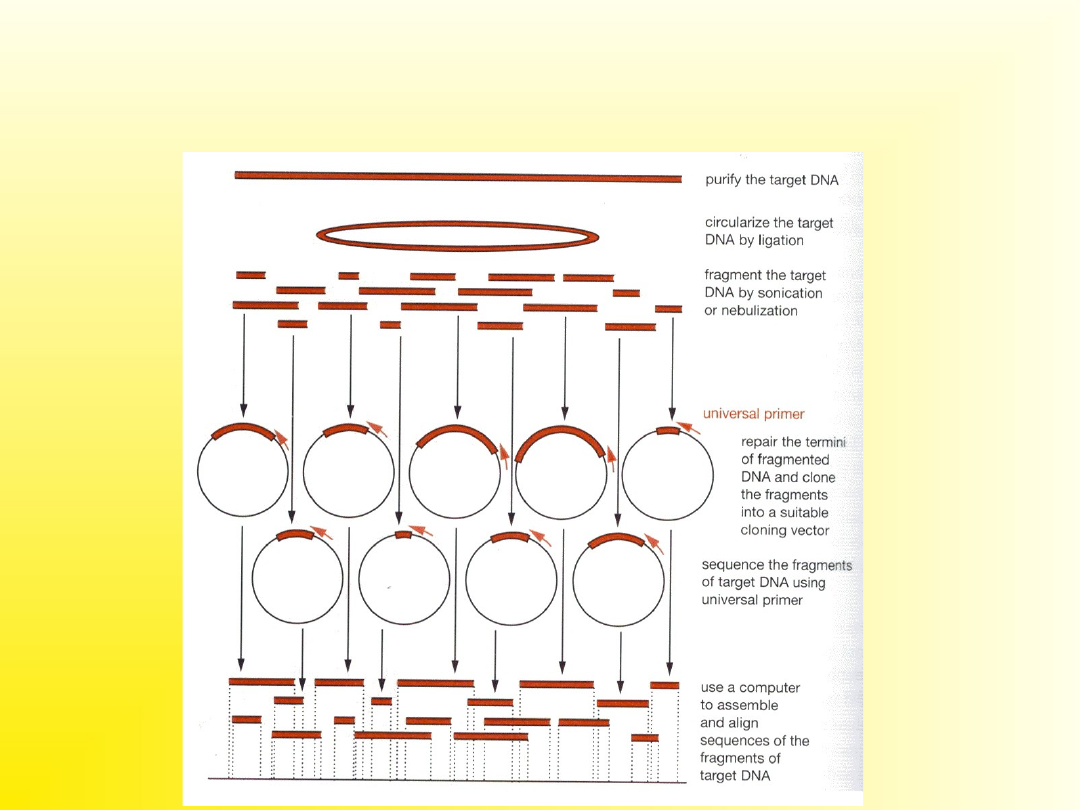
Sekwencjonowanie długich
fragmentów DNA – metoda
shotgun

Etapy
sekwencjonowania
1. Oczyszczanie DNA z wektora o dużej pojemności (kosmid,
BAC itp.)
2. Ligacja wewnątrzcząsteczkowa (cyrkularyzacja)
3. Rozrywane cząsteczek DNA na fragmenty (sonikowanie,
nebulizacja, trawienie Dnazą I w obecności Mn
2+
)
4. Naprawa końców DNA:
polimeraza faga T4
fragment Klenowa polimerazy I DNA
kinaza polinukleotydowa faga T4
5. Frakcjonowanie fragmentów DNA w żelu agarozowym
lub poliakrylamidowym
6. Ligacja z wektorem M13, transformacja bakterii
7. Izolacja poszczególnych klonów na płytkę mikrotitracyjną
8. Sekwencjonowanie przy użyciu uniwersalnego startera
9. Komputerowe składanie sekwencji we właściwej
kolejności

Na ćwiczenie 2 proszę przygotować:
1. budowa komórek prokariotycznych i
eukariotycznych.
2. zadania z rozcieńczeń roztworów
Na ćwiczenie 3:
1. budowa DNA i zasad azotowych
2. instrukcja do ćwiczeń – działanie odczynników
w izolacji DNA (wstęp teoretyczny)
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
- Slide 44
- Slide 45
- Slide 46
- Slide 47
- Slide 48
- Slide 49
Wyszukiwarka
Podobne podstrony:
W5 sII PCR i sekwencjonowanie cz 2
BMiGO Wykład 4 PCR, Sekwencjonowanie
Wyklad 4 PCR, Sekwencjonowanie
W5 sII PCR i sekwencjonowanie cz 2
W4 Metody powielania sekwencji DNA
W4 Proces wytwórczy oprogramowania
W4 2010
Statystyka SUM w4
w4 3
W4 2
W4 1
w4 skrócony
w4 orbitale molekularne hybrydyzacja
in w4
w4 Zazębienie ewolwentowe
więcej podobnych podstron