
Zagadnienie szczegółowe na egzamin z mikrobiologii:
GENETYKA BAKTERII
podpunkty: 118 -149.
118. Genetyka bakterii.
Bakteria, tak jak każdy żywy organizm, przeprowadza setki różnych reakcji w sposób
uporządkowany w czasie i przestrzeni i tym samym utrzymuje i powiela swoją strukturę.
W czasie rozwoju komórka bakteryjna zmienia nieco swoją wielkość, może zmieniać
kształt i pewne drugorzędowe właściwości fizjologiczne, może wytwarzać formy przetrwalne
i przechodzić stosunkowo mało skomplikowany, właściwy sobie cykl rozwojowy. Zmian
rozwojowych nie dziedziczy się. Dziedziczy się jednak dolność podlegania określonym dla
danego gatunku zmianom rozwojowym. W każdym pokoleniu powtarza się ten sam ciąg
przemian rozwojowych, od powstania organizmu Az do śmierci. Zmienność tego typu na ogół
łatwo odróżnić od zmienności środowiskowej, zależnej od czynników zewnętrznych.
119. Genotyp i fenotyp.
Bakterie, tak jak inne organizmy, dziedziczą nie określone cechy, ile potencjalną
zdolność do ich ujawnienia. Czy dana cecha ujawni się czy nie, decyduje często środowisko.
Fenotyp jest to zespół cech ujawniających się.
Genotyp jest to potencjalna zdolność do ujawnienia cech, przekazywana z pokolenia na
pokolenie, uwarunkowana strukturą materiału genetycznego, jakim jest DNA (wyjątkowo, u
niektórych wirusów – RNA).
120. Transformacja genetyczna bakterii Gram (+) i Gram (-) (mechanizmy tych
procesów).
Materiał genetyczny może być przenoszony z jednej bakterii do drugiej przez sam DNA.
Proces ten nazywamy transformacją genetyczną. Charakterystyczne jest to, że proces
transformacji zachodzi najłatwiej w obrębie szczepów należących do jednego gatunku.
Transformacja między gatunkowa zachodzi rzadko i przede wszystkim między gatunkami
zbliżonymi do siebie. Wydajność procesu jest też wtedy znacznie mniejsza.
Transformacja genetyczna ma dwa aspekty:
−
transformacja naturalna – związana z naturalną zdolnością komórek do pobierania
DNA (tzw. naturalny stan kompetencji); do bakterii posiadających tego typu zdolności należą
Streptococcus pneumoniae, Bacillus subtilis, Bacillus cereus, Bacillus stearothermophilus,
niektóre spośród Gram (-), Archeobacteriaceae.
−
transformacja indukowana – związana z działaniem pewnych czynników, które
stymulują możliwość pobierania materiału genetycznego; należą tu bakterie takie jak
Escherichia coli, Salmonella typhimurium, Pseudomonas aeruginosa.
Mechanizm transformacji naturalnej
W przypadku transformacji naturalnej mamy do czynienia z występowaniem w
komórce czynnika kompetencji przyłączającego się do błony komórkowej. Czynnik ten
powoduje wytwarzanie lizyn i proces transkrypcji. Autolizyna jest wytwarzanym przez
komórkę– biorcę (enzymem nadtrawiającym murenę).
W pobliżu miejsca działania autolizyny występuje białko zdolne do wiązania DNA;
występuje tu także egzonukleaza.

Pierwszy kontakt komórki z DNA ma charakter odwracalny, potem – nieodwracalny.
Wiązanie DNA może nastąpić w kilku miejscach. Bakteria dąży do „schwytania” możliwie
dużego fragmentu DNA, a gdy tak się stanie nukleaza zaczyna nadtrawiać jedną z nici. DNA
w postaci jednoniciowej (ssDNA) charakteryzuje się dużą wrażliwością na różne czynniki,
dlatego też szybko zostaje osłonięte przez białka wiążące DNA i w takiej postaci jest
wprowadzane.
W komórce może dochodzić do rekombinacji zgodnie z homologią zasad.
Wytwarzanie otoczki przez dwoinki zapalenia płuc z doświadczenia Griffitha było efektem
rekombinacji materiału genetycznego. Rekombinacja zachodzi tylko wówczas, gdy występuje
bliska 100% zgodność materiału rekombinującego się. Nabycie przez bakterię nie
spokrewnionego DNA wiąże się zwykle z degradacją pozyskanego materiału lub z jego
przetrwaniem w postaci plazmidu.
Wszystkie rodzaje bakterii, z wyjątkiem Haemophilus influenzae, zdolne są do
związania i pobrania heterologicznego DNA, choć wówczas wydajność potransformacyjnej
rekombinacji jest mała. Haemophilus influenzae wiąże i pobiera jedynie własny DNA,
ponieważ do zajścia tych procesów konieczna jest obecność sekwencji 5’-AAGTGCGGTCA-
3’ (Alę Angina Grzmotnęła Tęga Gdyż Całą Godzinę Ganiała Tomka Co jest Atrakcyjny),
która występuje w genomie tej bakterii aż w 600 kopiach, mimo że, nie powinno być więcej
niż jedna kopia.
Mechanizm transformacji indukowanej
Transformacja indukowana dotyczy komórek które naturalnie nie występują w stanie
kompetencji, a więc nie są zdolne do pobranie DNA. Aby komórki mogły przyjąć informację
genetyczną działa się na nie różnymi czynnikami i w ten sposób wprowadza w stan
kompetencji (indukuje się ten stan). Transformację indukowaną stosuje się w przypadku
sztandarowego gatunku bakterii wykorzystywanej w laboratorium, a mianowicie E. coli. W
omawianym przypadku do komórki bakteryjnej, po zastymulowaniu jej do transformacji,
wnikają zwykle koliste cząsteczki DNA – plazmidy;
E. coli często pobiera DNA lub plazmid większy od własnego DNA. Wnikanie
materiału genetycznego do wnętrza komórki następuje poprzez tzw. złącza Bayera; taki
transport zachodzi bez większego nakładu energii. Wytworzenie stanu kompetencji następuje
poprzez działanie CaCl
2
. Jony Ca
2+
znoszą ujemny ładunek lipopolisacharydów błony
zewnętrznej E. coli, co umożliwia zbliżenie się ujemnie naładowanego DNA do komórki i nie
pozwala na odpychanie materiału genetycznego.
Transformacja indukowana występuje u Staphyllococcus aureus spośród Gram (+), a
także u Salmonella typhimurium i u wspominanej już E. coli spośród Gram (-).
121. Na czym polega stan kompetencji bakterii.
Bakterie ulegają transformacji tylko w pewnych warunkach i okresach rozwojowych.
Stan, w którym bakteria daje się transformować, określa się terminem „kompetencja”. Stan
kompetencji jest związany z różnorodnymi zmianami osłon komórkowych. Kompetencja
może się rozwinąć pod wpływem jakiegoś czynnika enzymatycznego, czy też innej natury,
wytwarzanego przez same komórki podlegające transformacji. Konieczne jest też do tego
zapewnienie szczególnych warunków środowiskowych i składu podłoża. Kompetencja
komórek bakteryjnych zależy też od wielu genów i często dotyczy małej części populacji.
Niektóre bakterie nie są w stanie rozwinąć kompetencji spontanicznie. Przygotowanie
komórek do transformacji wymaga wówczas wielu zabiegów zmieniających strukturę osłon.
Do najczęściej stosowanych należy poddanie bakterii działaniu wysokich stężeń kationów

(Ca
2+
, Rb
+
), połączone z szokiem cieplnym, lub działaniu krótkich impulsów silnego pola
elektrycznego (elektrotransformacja zwana również elektroporacją).
Komórki kompetentne wiążą na swojej powierzchni dwuniciowe, stosunkowo nieduże
odcinki DNA o długości 7-9 kb, zawierające informację genetyczną równoważną najwyżej
paru genom. Podczas przejścia przez osłony komórki biorcy jedna z nici transformującego
DNA ulega degradacji. Pomiędzy fragmentem jednej nici DNA dawcy, która wniknęła do
komórki biorcy, i chromosomem biorcy może dojść do rekombinacji genetycznej.
122. Rekombinacja genetyczna.
Mutacje nie są jedynym źródłem zmienności organizmów. Znacznie większą rolę
odgrywają różne mechanizmy rekombinacji genetycznej związanej z procesem płciowym.
Proces y paraseksualne, a więc prowadzące do rekombinacji genetycznej, są obecnie znane u
bakterii jako trzy podstawowe typy: transformacji, transdukcji i koniugacji.
Rekombinacja genetyczną nazywamy utworzenie, w zasadzi z dwóch różnych genów
(lub ich części), jednej nowej struktury zawierającej zapis informacji genetycznej pochodzący
od obu rekombinujących elementów. U bakterii (i u bakteriofagów) znamy dwa zasadnicze
typy rekombinacji: 1) rekombinację ogólną i 2) tzw. rekombinację nieuprawnioną, która
dzieli się na rekombinację swoistą dla miejsca i transpozycję.
Rekombinacja ogólna
Rekombinacja ogólna, zwana inaczej homologiczną, zachodzi między
homologicznymi nićmi DNA, tj. takimi, których sekwencje nukleotydów są w zasadzie
identyczne. Nici takie mogą się, oczywiście, różnić miejscowymi podstawieniami
nukleotydów lub małymi delecjami czy inercjami, ale liczba różniących je nukleotydów
pozostaje znikoma w stosunku do ogólnej ich liczby. Rekombinacja taka jest więc możliwa,
gdy w jednej komórce znajdą się w jakiś sposób genomy lub odcinki genomów pochodzące z
różnych osobników tego samego gatunku lub gatunków blisko spokrewnionych.
U bakterii rekombinacja ogólna jest bardzo złożonym i wieloetapowym procesem i
może przebiegać według trzech podstawowych szlaków rekombinacyjnych nazywanych
szlakami RecBCD, RecE i RecF, od nazwy uczestniczących w nich białek. Szlak RecBCD
rządzi rekombinacja pokoniugacyjną i potransdukcyjną, gdy jednym z rekombinujących
partnerów jest dwuniciowy, liniowy odcinek DNA, natomiast rekombinacja według szlaku
RecF zachodzi między cząsteczkami kolistymi (jedna z nich musi mieć nacięcie lub lukę) i
pełni rolę w wymianie materiału genetycznego między współwystępującymi w komórce
plazmidami. W każdym z wyżej wymienionych szlaków rekombinacyjnych kluczową rolę
odgrywa białko RecA, kodowane przez gen recA.
Mechanizm działania białka RecA polega na rozpoznawaniu jednoniciowego DNA i
związaniu się z nim. Przyłączenie białka nadaje nici DNA zdolność do „inwazji”
dwuniciowego DNA drugiej, rekombinującej cząsteczki. Po denaturacji dupleksu, jedna z
jego nici zostaje zastąpiona nicią „inwazyjną”.
Inne białka biorące udział w poszczególnych szlakach rekombinacyjnych nacinaja i rozplatają
DNA, a tym samym stwarzają warunki do przyłączenia białka RecA. Niewątpliwie najlepiej
poznany jest kompleks białka RecBCD, tradycyjnie zwany egzonukleazą V. Kompleks ten
ma również aktywność endonukleolityczną i helikazową, a jego działanie wymaga obecności
w DNA 8-nukleotydowej sekwencji Chi. W rekombinacji biorą również udział inne nukleazy
i helikazy, białka wiążące jednoniciowy DNA, gyraza, itp.
Rekombinacja nieuprawniona

Rekombinacja ta może zachodzić między helisami DNA nie wykazującymi ogólnej
homologii, choć oczywiście może też zajść między strukturami homologicznymi.
Rekombinacja nie uprawniona swoista dla miejsca jest uwarunkowana obecnością krótkich
odcinków homologii sekwencji między rekombinującymi partnerami, transpozycja natomiast
nie wymaga homologii sekwencji. Rekombinacja zależy od aktywności różnych enzymów
typu topoizomeraz (integrazy, inwertazy, resolwazy), mających zdolność do przecięcia DNA,
przeplecenia dwóch nici i ponownej ich ligacji. Rekombinacja nieuprawniona nie zależy od
genu RecA i innych znanych bakteryjnych genów wpływających na rekombinację ogólną.
Rekombinacja nieuprawniona ma znaczenie w rearanżacji genów u bakterii i
powstawaniu duplikacji, a czasem zwielokrotnienia odcinków DNA przyczyniających się do
powiększenia genomu bakterii.
123. Eksperyment Griffith`a.
Odkrycie transformacji miało miejsce pierwotnie u pneumokoków (Streptococcus
pneumoniae – bakteria wywołująca zapalenie płuc) w 1928r. przez Griffitha, a wyjaśnione
zostało w 1944r. przez Avery`ego. Pneumokoki wytwarzają otoczki zbudowane ze śluzu
węglowodanowego. W chwili obecnej znanych jest kilkadziesiąt typów pneumokoków,
różniących się budową chemiczną śluzów otoczkowych i oznaczanych cyframi rzymskimi I,
II, III itp. Właściwość wytwarzania danego typu otoczki jest stała i przekazywana
dziedzicznie. W trakcie hodowli laboratoryjnej pneumokok może jednak utracić otoczkę i
staje się wtedy niezdolny do wywołania choroby. Szczepy z otoczką określa się literą S od
smooth – gładkie (chodzi tu o wgląd kolonii bakteryjnych), natomiast bezotoczkowe literą R,
od routh – szorstkie. Doświadczenia Griffitha na myszach: o wstrzyknięciu do krwi myszy
zabitego pneumokoka typu SIII (wytwarza otoczkę typu III) i żywego bezotoczkowego
pneumokoka typu II (wytwarzał otoczkę typu II, lecz stracił tę zdolność i przeobraził się w
szczep typu R) – mysz pada. Z padłej myszy można wyizolować żywego pneumokoka typu
III. Zatem bezotoczkowy pneumokok, który kiedyś tworzył otoczkę typu II, teraz zaczyna
wytwarzać otoczkę typu III. Bakteria nabyła więc nową cechę – zdolność do tworzenia
nowego typu otoczki, różnego od tego jaki był jej właściwy. Zjawisko to nazwano
transformacją.
124. Doświadczenie Avery`ego.
Doświadczenia Avery`ego wykazały, że czynnikiem odpowiedzialnym za
przekazywanie cech martwych komórek komórkom żywym jest DNA. Avery poddawał
wyciąg z bakterii otoczkowych działaniu różnych enzymów i sprawdzał, czy nadal zachowuje
zdolność do transformacji. Enzymy trawiące białka (enzymy proteolityczne) nie zmieniały
właściwości wyciągu – nadal następowała transformacja. Oznaczało to, że białka, wbrew
temu co wówczas sądzono nie są czynnikiem transformującym, odpowiedzialnym za
przekazywanie informacji. Dopiero potraktowanie wyciągu enzymem rozkładającym DNA
(deoksyrybonukleazą) zniszczyło zdolność do transformacji. W kolejnych doświadczeniach
Avery`emu udało się wyizolować i oczyścić wielocząsteczkowy DNA i wykazać, że tylko
DNA może transformować bakterie.
Z czasem wykazano, że transformować można liczne inne cechy, np. odporność na
antybiotyki, na fagi, zdolność do fermentacji cukrów, do syntezy aminokwasów i witamin i
in. Okazało się również, że zjawisko to można obserwować u innych bakterii: u
paciorkowców (Streptococcus), u pałeczki grypowej (Haemofilus influenzae), laseczki siennej
(Bacillus subtilis), bakterii brodawkowych (Rhizobium), sinic i innych.
125. Doświadczenie Alfreda Hershey`a i Marty Chase.
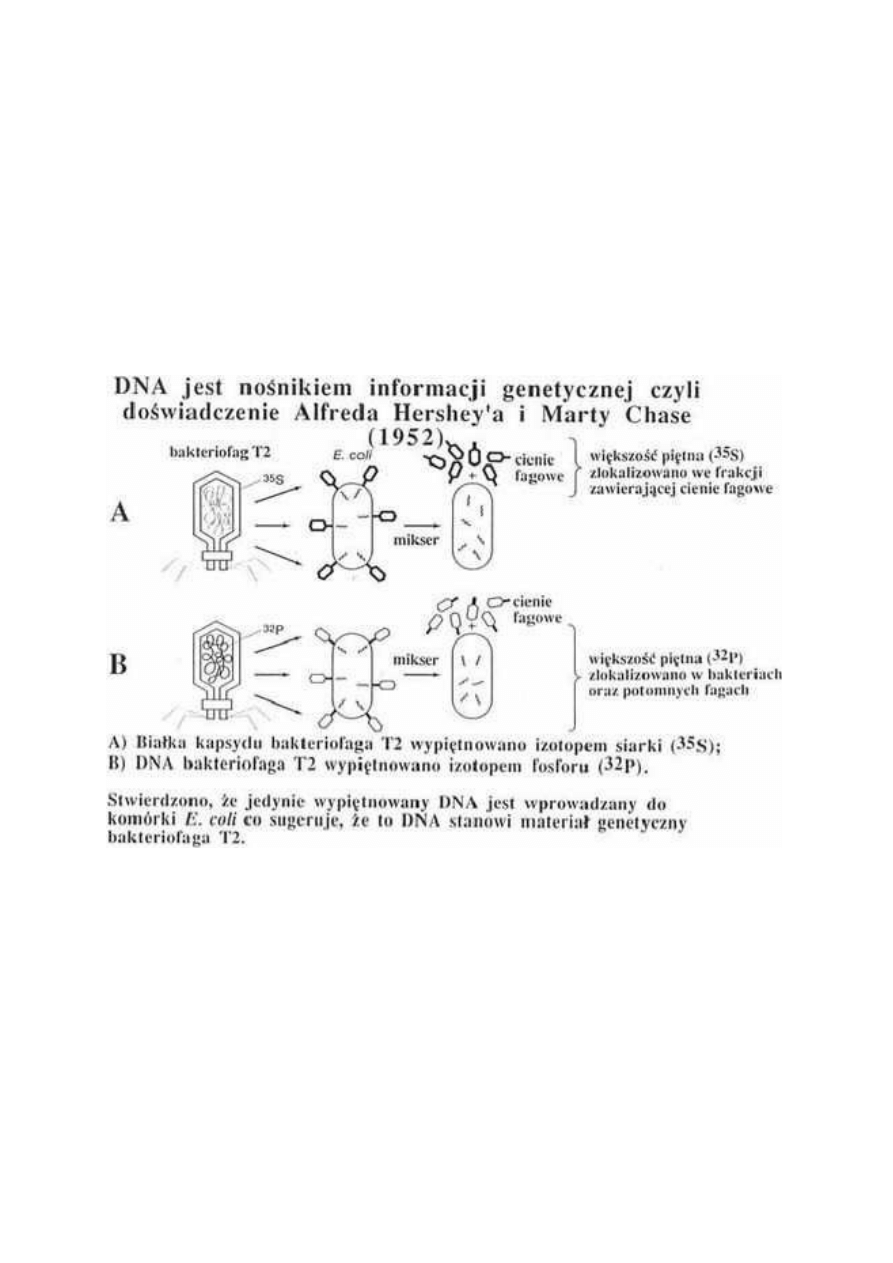
Doświadczeniem potwierdzającym udział DNA w przenoszeniu informacji
genetycznej było to, które przeprowadzili Alfred Hershey i Marta Chase.
Eksperyment polegał na znakowaniu faga T2 przy pomocy izotopów radioaktywnych.
Białkowy kapsyd zawierający siarkę jako składową aminokwasów wyznakowano przy
pomocy izotopu
35
S, zaś DNA znakowano
32
P. Tak wyznakowanymi fagami zakażono
hodowlę bakteryjną. Okazało się, że do komórki bakteryjnej dostaje się izotop
32
P a więc –
zawierający go DNA, zaś piętnowana siarka pozostaje na zewnątrz komórki bakteryjnej.
Materiał genetyczny bakteriofaga służy jako informacja genetyczna, przy pomocy której
możliwe jest składanie nowych kapsydów i powstawanie całych nowych fagów po
zapakowaniu DNA.
126. Doświadczenie Meselsona i Stahla.
Badacze ci opracowali bardzo pomysłową metodę oddzielania i rozróżniania
cząsteczek DNA. W tym celu Meselson i Stahl wyhodowali dwie kultury bakteryjne
Escherichia coli: pierwsza rozwijała się na podłożu zawierającym „typowy” izotop azotu –
14
N, druga zaś na podłożu zawierającym ciężki izotop azotu –
15
N.
Azot jest istotnym składnikiem zasad azotowych, a więc hodowane bakterie
wbudowywały odpowiednie izotopy do syntetyzowanych zasad azotowych, a później do
DNA. Należało więc oczekiwać, że DNA bakterii zawierający izotop
15
N będzie nieznacznie
cięższy, niż ten zawierający izotop
14
N. Różnice te można stwierdzić, wykorzystując metodę
wirowania w gradiencie gęstości odpowiedniego roztworu. Meselson i Stahl poddawali
wirowaniu wypreparowany DNA w roztworze doskonale rozpuszczalnej soli – chlorku cezu.
Rozpuszczalność tej soli pozwala na takie dobranie gęstości roztworu, aby była ona zbliżona
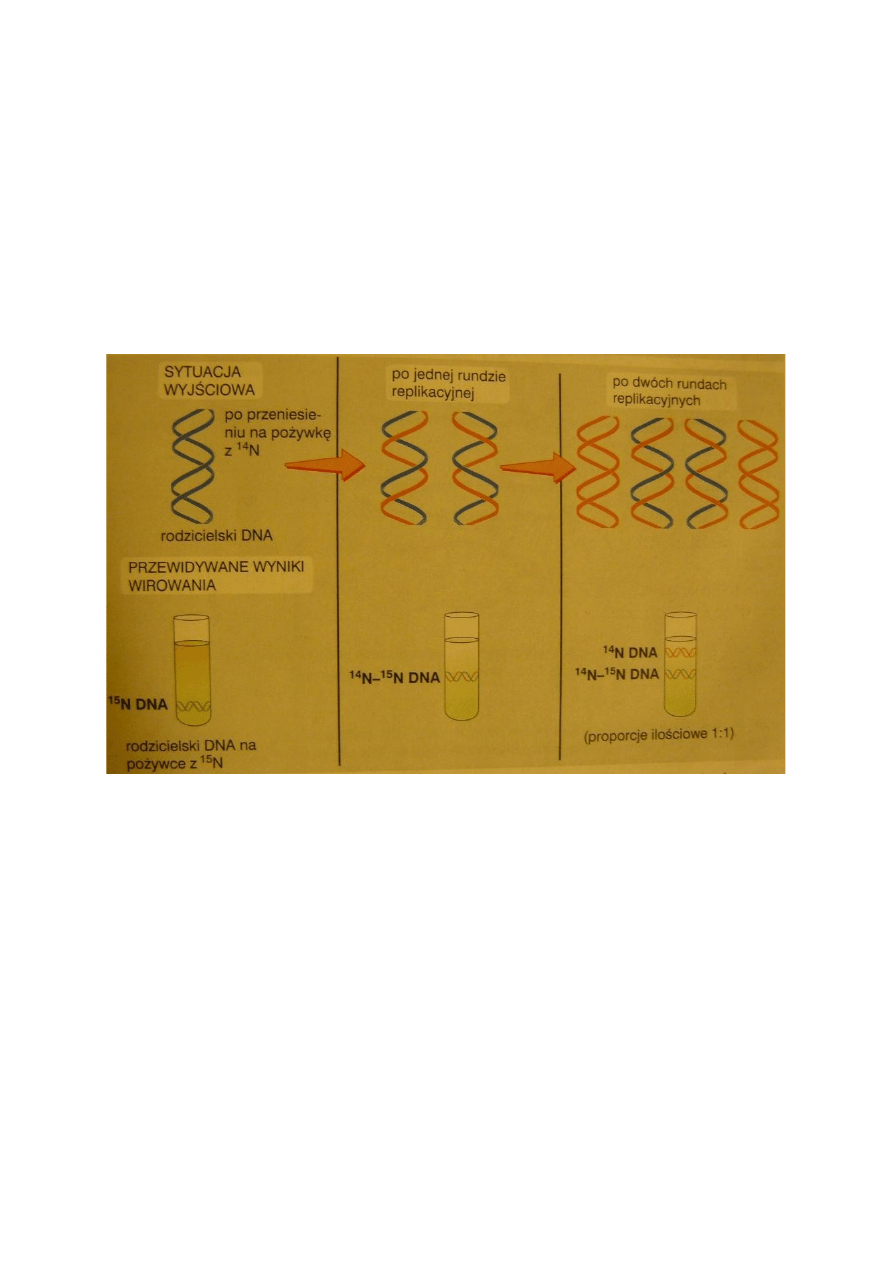
do gęstości DNA. Jeżeli umieścić specjalną probówkę w szybkiej wirówce, to długotrwałe
wirowanie spowoduje, że cząsteczki soli będą przemieszczały się w stronę dna probówki, tak
więc gęstość roztworu będzie mniejsza przy jego powierzchni i będzie rosła w stronę dna.
Podobnie DNA – lżejsze cząsteczki z „typowym” izotopem azotu ulokują się bliżej
powierzchni, natomiast te z izotopem ciężkim – bliżej dna.
Wyniki uzyskane przez Meselsona i Stahla dowodziły jednoznacznie, że replikacja
DNA ma charakter semikonserwatywny. Cały DNA powstały po jednej rundzie replikacyjnej,
ulokował się mniej więcej w środku probówki (ze względu na hybrydowy charakter miały
gęstość pośrednią). Zgodnie z tym modelem, po dwóch rundach replikacji (dwóch cyklach
podziałowych komórek E. coli), jedną połowę wytworzonego DNA stanowiła frakcja
„średnia”, a drugą część – lekki, nowy DNA.
127. Przebieg procesu koniugacji u bakterii Gram (+) i Gram (-).
Gram (+)
Plazmidy koniugujące występują także u bakterii gramdodatnich, lecz dotychczas w
żadnym z funkcjonujących u nich systemów koniugujących nie stwierdzono udziału pilusów
płciowych.
Pewne szczepu Enterococcus, potencjalni biorcy w koniugacji, wydzielają do podłoża
krótkie, siedmio- lub osmioaminokwasowe, hydrofobowe peptydy zwane feromonami
płciowymi. Feromony te są atraktantami dla szczepów niosących odpowiednie plazmidy i
stymulują tworzenie z nimi par (agregatów) koniugujących. Jedna komórka może wytworzyć
kilka różnych feromonów, lecz parę koniugującą utworzy jedynie z komórką potencjalnego
dawcy, który niesie plazmid koniugacyjny „odpowiadający” na swoisty typ feromonu
płciowego.
Feromony, wydzielane do podłoża w niewielkiej ilości, wnikają do komórek dawcy,
ale indukcja dalszych etapów procesu zachodzi dopiero wówczas, gdy ich stężenie osiągnie
pewną wartość progową, co jest możliwe jedynie w warunkach odpowiednio gęstej hodowli.
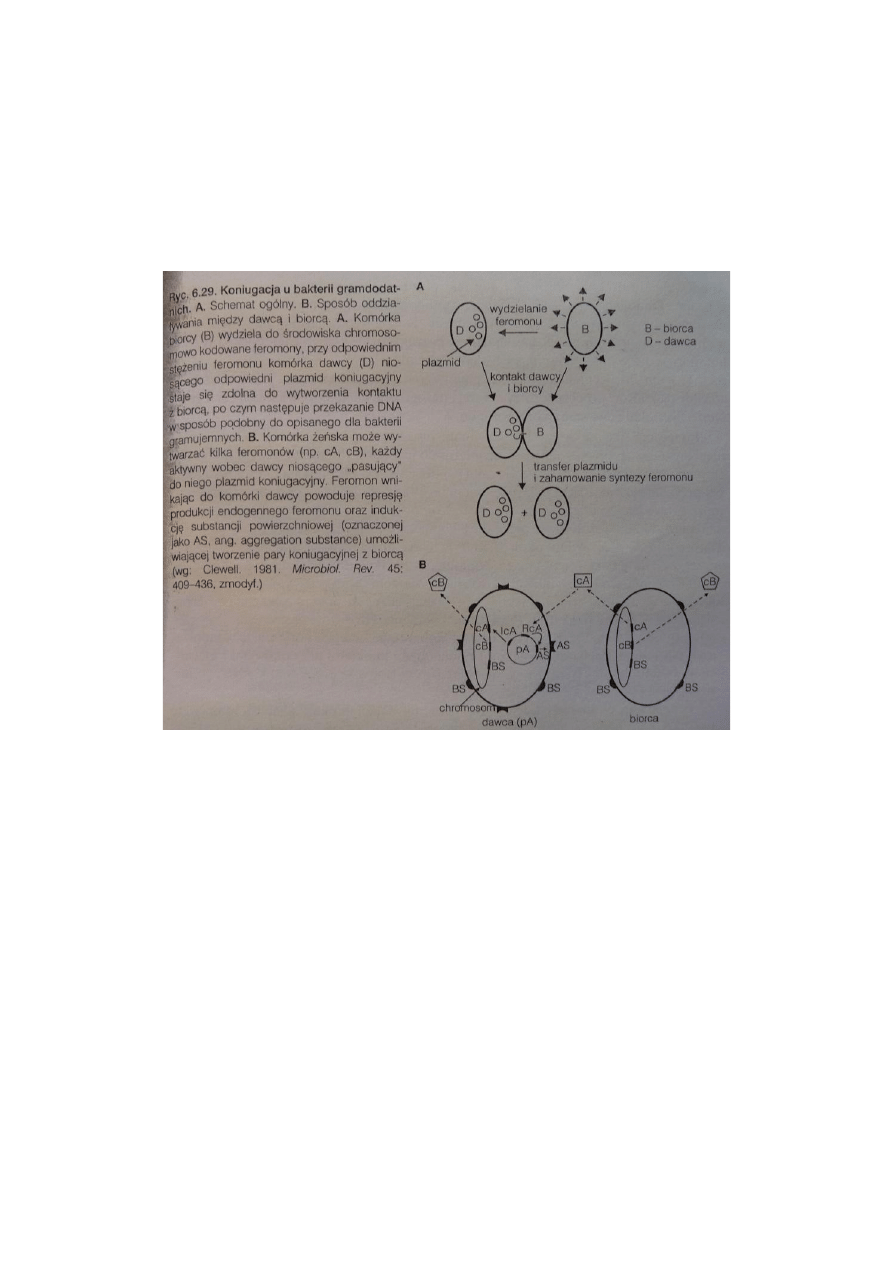
Transport feromonów do komórki potencjalnego dawcy następuje z udziałem białek
kodowanych przez plazmid. Określony feromon działa swoiście wobec „odpowiadającego”
mu plazmidu koniugacyjnego. Transkrypcja genu traE, kodującego pozytywny aktywator
pozostałych genów tra, w tym genu (asa) odpowiedzialnego za tworzenie, na powierzchni
komórki dawcy, substancji agregującej. Substancja agregująca oddziałuje z, obecną na
powierzchni biorcy (powstałą pod kontrolą genów chromosomowych), substancji wiążącą BS,
co prowadzi do tworzenia do par (agregatów) koniugacyjnych.
Gram (-)
Kontakt koniugujących bakterii możliwy jest dzięki wytwarzaniu przez komórki
męskie pili płciowych, wystających z błony zewnętrznej. Pile są strukturami bardzo cienkimi
i zwykle na zdjęciach spod mikroskopu elektronowego, dla lepszego ich uwidocznienia
bywają opłaszczone bakteriofagiem do nich powinowatym (np. fag MS-2).
Dotknięcie przez pilus komórki żeńskiej i przymocowanie się do receptora OmpA na
jej powierzchni powoduje skracanie (depolimeryzację) pilusa od jego podstawy (na
powierzchni komórki F+) i umożliwia zmniejszenie dystansu między bakteriami. Bakteria
męska silnie przyciąga żeńską, aż do osiągnięcia maksymalnego zbliżenia pozwalającego na
wytworzenie mostka cytoplazmatycznego łączącego obie komórki.
Koniugacja pomiędzy komórkami męskimi jest niemożliwa ze względu na produkcję
przez te komórki białka TraT, które wiąże się do receptora OmpA na powierzchni komórek
i uniemożliwia tym samym przyłączenie się pilusa płciowego, a w efekcie – koniugację.
Czynnik F jest niskokopijnym plazmidem (1-2 cząsteczek na komórkę) wielkości
około 100 kp. Plazmid ten zawiera operon Tra składający się z ponad 20 genów
odpowiedzialnych za „bakteryjną męskość” i do tego jeden promotor. Plazmid ma własne ori,
z którego rozpoczyna się jego transfer do komórki biorcy. Posiada też sekwencje insercyjne
umożliwiające jego włączanie do chromosomu bakterii.
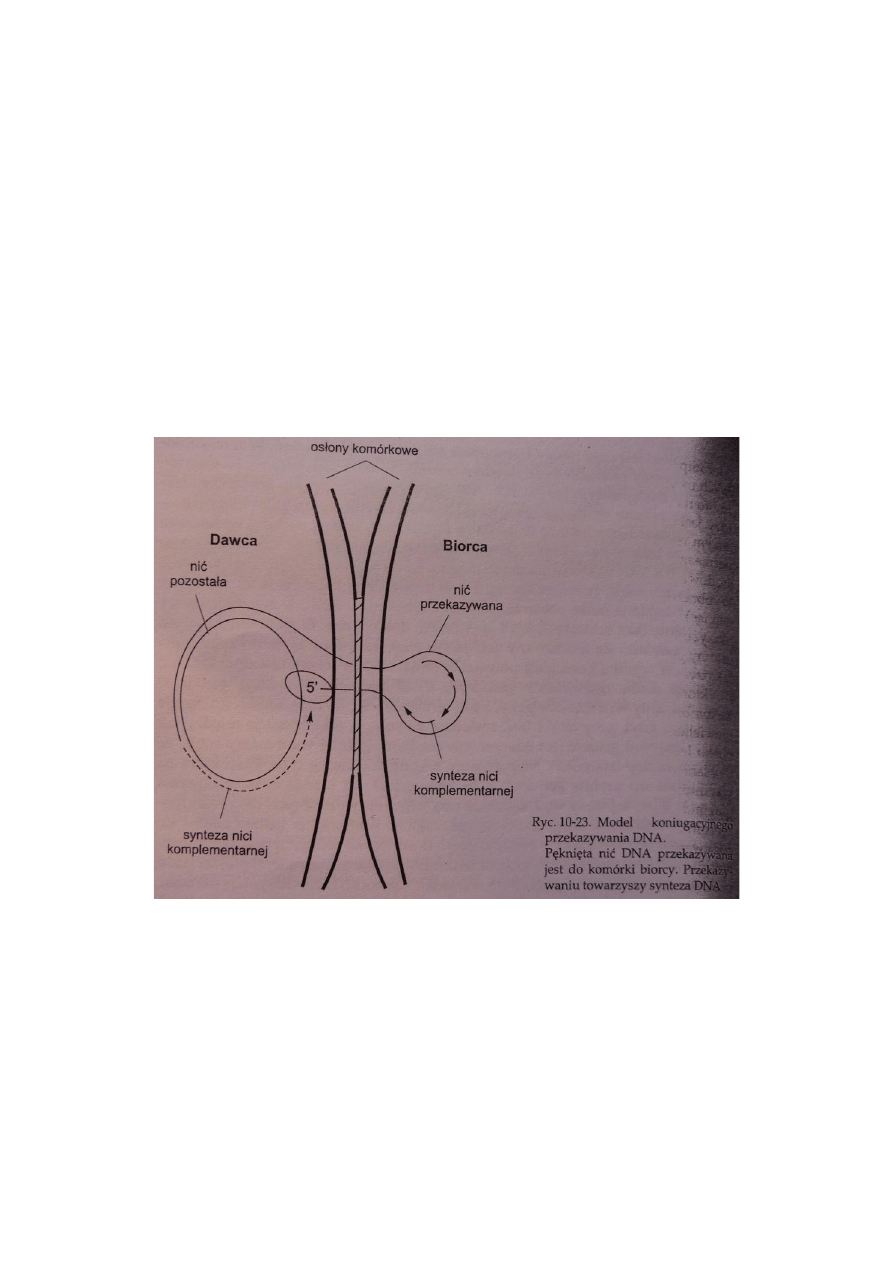
Plazmid przekazywany jest z komórki do komórki w postaci jednoniciowego DNA.
Przekazywanie rozpoczyna się od nacięcia DNA w miejscu oriT. Koniec 5’ wchodzi do
komórki biorcy; najpierw przekazywany jest rejon odpowiedzialny za replikację, potem – za
integrację, a ostatecznie – rejon tra. Plazmid ten może integrować się z chromosomem
bakteryjnym; w warunkach naturalnych jest to dosyć rzadkie zjawisko. Zwiększenie częstości
integracji może być zastymulowane poprzez działanie różnych czynników mutagennych.
Integracja jest możliwa dzięki występowaniu w chromosomie bakterii miejsc o
homologicznych do plazmidu sekwencjach.
Wnikanie nici DNA do komórki biorcy (co równoznaczne jest z koniugacją) możliwe
jest dzięki białku TraY oraz TraI. Powstający po nacięciu DNA w miejscu oriT fragment
jednoniciowy końcem 5’ wnika do komórki biorcy. Zarówno w komórce biorcy jak i dawcy
odbywa się dosyntetyzowywanie nowej, drugiej nici DNA. W komórce biorcy odbywa
się to poprzez fragmenty Okazaki, a w komórce dawcy – ma charakter ciągły. Ostatecznym
efektem transferu jest powstanie dwóch plazmidów; jeden znajduje się w komórce biorcy,
drugi – dawcy.
128. Plazmid F – istotne elementy genetyczne.
Koniugacja bakterii
Koniugację prowokują plazmidy F (fertility factor), które mogą:
- replikować się i rozchodzić do obu potomnych komórek bakterii
- syntetyzować pili (l. poj. pilus), małe białkowe rurki, które odszukują inną komórkę,
przyczepiają się do niej, a potem skracając się przyciągają komórki do siebie
- przez te rurki przechodzi kopia plazmidu od dawcy (F+) do biorcy, w której go jeszcze nie
ma (F-)
- jeżeli plazmid jest wczepiony do chromosomu bakterii dawcy to przechodząc do biorcy
przeciąga
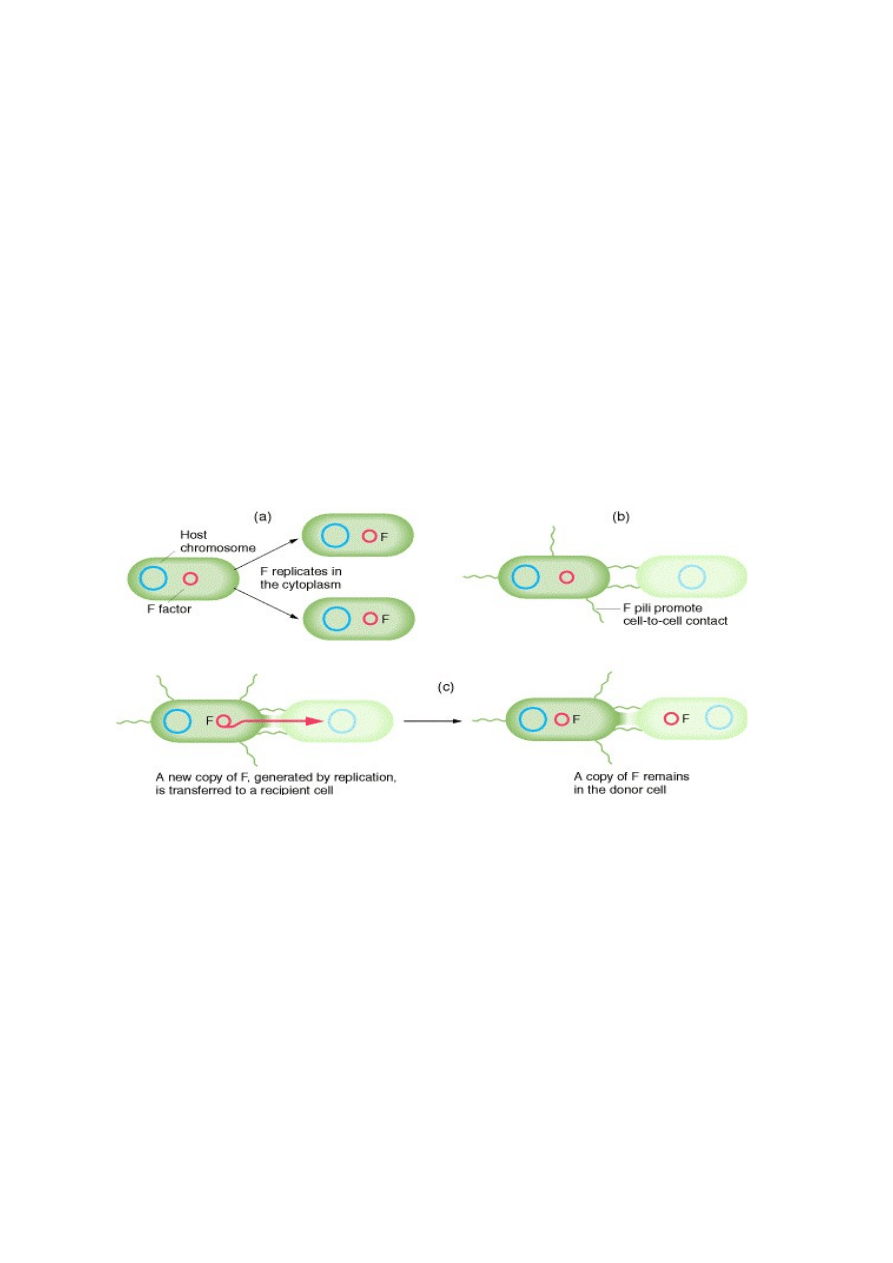
jego fragment; dawca ginie a jego geny mogą włączyć się w genom biorcy; proces taki
zachodzi
stosunkowo często, dlatego szczepy takie nazywamy Hfr (high frequency of recombination)
Ogólna charakterystyka plazmidów i bakteriofagów
Plazmid to pozachromosomowa, z reguły kolista cząsteczka DNA zdolna do samodzielnej
replikacji, która występuje u prokariota i niektórych organizmów eukariotycznych. Plazmidy
pełnia w komórkach funkcje pomocniczych chromosomów i są przenoszone w trakcie
procesu
koniugacji miedzy komórkami bakteryjnymi. Jednym z najlepiej poznanych plazmidów
jest plazmid F z E. coli. Jest to plazmid typu koniugacyjnego, ponieważ jest transferowany
z komórki donora do akceptora. Szczepy bakterii zawierają różną liczbę plazmidów. Plazmidy
ulegają replikacji niezależnie od macierzystego genomu bakterii, take w komórkach
gospodarzy należacych do innych gatunków. Chociaż plazmidy nie są one konieczne
do życia bakterii, to jednak mogą: (1) powodować oporność na antybiotyki i jony metali
ciekich, (2) umożliwiając katabolizm toksycznych związków (toluen), (3) wpływać na
chorobotwórczość
bakterii oraz na zdolność do syntezy związków oddziaływając na rozwój
innych bakterii, (4) umożliwiać interakcje z roślinami (wiązanie azotu) oraz koniugacje
bakterii. Plazmidy można wyizolować niezależnie od chromosomu bakteryjnego.
129. Operon tra.
Pary koniugujące są tworzone z udziałem, syntezowanych pod kontrolą grupy genów
tra, pilusów płciowych. Region tra plazmidu F to jeden fragment DNA o długości 33,3 kpz.
Na tym fragmencie zlokalizowanych jest glisko 40 genów pełniących różne funkcje w
procesie koniugacji. Aż kilkanaście genów tra to geny odpowiedzialne za syntezę pilusów
płciowych. Produkty trzech z nich (traA, traQ i traX) uczestniczą w syntezie białka
budulcowego pilusa – piliny. Pozostałe geny tra z tej grupy kontrolują składanie pilusa.
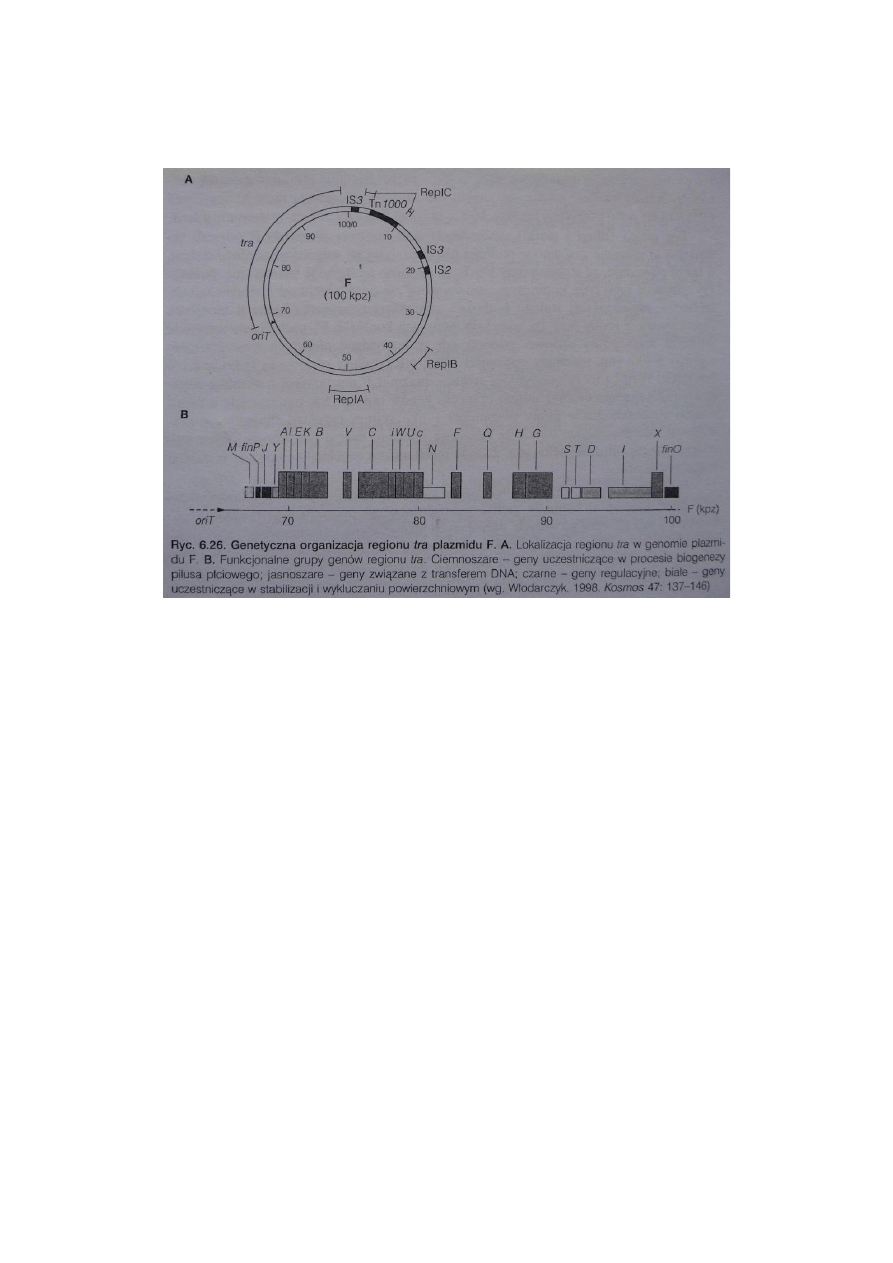
Mutacja w którymkolwiek z wymienionych genów sprawia, iż komórka nie wytwarza
funkcjonalnych pilusów.
130. Mechanizm replikacji plazmidu F.
Podczas koniugacji komórki obu koniugujących partnerów przybliżają się do siebie w
wyniku wciągania pilusa i depolimeryzacji jego podjednostek u podstawy. Powoduje to
wytworzenie ścisłego kontaktu :ściana-ściana” i uformowanie kanału koniugacyjnego. W tym
rejonie powstaje relaksosom, stabilny nukleoproteidowy kompleks (głownie białko
relaksacyjne TraI), uczestniczącego w przygotowaniu DNA do transferu do komórki biorcy.
W przypadku plazmidu F, w rejonie oriT plazmidu i kanału koniugacyjnego formuje
się stabilny kompleks nukleoproteidowy. Relaksosom do momentu utworzenia stabilnych par
koniugacyjnych funkcjonuje w stanie równowagi między nacinaniem i relegacją miejsca nic i
oriT. Po utworzeniu stabilnej pary koniugującej przekazywany jest do relaksysomu sygnał,
powodujący zmianę jego stanu z typu nacinanie/relegacja na stan gotowości do transferu
poprzez indukcję aktywności helikazowej białka TraI, które może teraz rozwijać helisę DNA
plazmidu F od miejsca nacięcia w punkcie nic. Rozwijanie helisy DNA umożliwia transfer
naciętej nici do komórki biorcy w kierunku 5` - 3`. Relaksaza (TraI), pozostaje kowalencyjnie
połączona z końcem 5` naciętej nici, podczas gdy wolny koniec 3`-OH stanowi starter syntezy
nici uzupełniającej u dawcy, zachodzącej według modelu toczącego się koła (RCR).
131. Szczepy Hfr E. coli.
Zwykle podczas koniugacji do komórki biorcy przekazywany jest jedynie plazmid. Jednak
czasem plazmid może pośredniczysz w transferze DNA chromosomu swojego gospodarza.

Czasem plazmidy koniugujące wbudowują się do chromosomu, i gdy taki plazmid podejmuje
proces transferu koniugującego, „zabiera” ze sobą cały, wielokrotnie od siebie dłuższy
chromosom, z którym jest kowalencyjnie połączony. Taki dawca z plazmidem wbudowanym
w chromosom komórki gospodarza nosi nazwę Hfr, gdyż wynikiem koniugacji w tym
systemie jest powstawanie, z wysoką częstotliwością, łatwych do wyselekcjonowania,
genetycznych rekombinatów.
132. W jaki sposób powstają szczepy Hfr.
Integracja plazmidów z chromosomem może następować według kilku różnych
mechanizmów. Jednym z najpowszechniejszych jest proces homologicznej rekombinacji
pomiędzy sekwencjami insercyjnymi (IS) obecnymi w plazmidach i chromosomach.
Rekombinacja między elementem IS2 w plazmidzie i chromosomie E. coli może prowadzić
do integracji plazmidu F. Komorka E. coli z wbudowanym w chromosom plazmidem F
(otoczonym przez dwie kopie sekwencji IS2) jest teraz szczepem Hfr. Chromosom E. coli
zawiera około 20 miejsc, które mogą stanowić podstawę tworzenia szczepów Hfr dzięki
rekombinacji homologicznej. Ponadto, włączenie plazmidu do chromosomu może być
wynikiem klasycznego zdarzenia transpozycyjnego, a ponieważ obecność elementów
transpozycyjnych w plazmidach jest dość powszechna, to ta druga droga tworzenia Hfr-ów
też jest częsta.
134. Krzyżówki bakteryjne: bakteria Hfr x bakteria żeńska (F
-
).
133. Znaczenie szczepów Hfr w genetyce bakterii.
Szczep Hfr, podobnie jak F
+
, wytwarza na powierzchni pilusy płciowe, może więc
utworzyć parę koniugującą z komórką biorcy F
-
i rozpocząć proces transferu DNA. Jednak,
podczas gdy przekazanie plazmidu F (100 kpz) trwa 2 minuty, transfer całego chromosomu
(4700 kpz) wymaga około 100 minut. Transfer zaczyna się zawsze od miejsca oriT w ten
sposób, że najpierw wprowadzany jest region wiodący plazmidu F, potem, zależnie od punktu
i orientacji połączenia plazmidu z chromosomem, DNA chromosomu, a na końcu reszta DNA
plazmidu. Przekazywanie następuje selekcyjnie, ze stała prędkością, poczynając od genów
chromosomowych zlokalizowanych blisko miejsca integracji połączenia z plazmidem. W
końcu do biorcy jest wprowadzony region tra plazmidu F i tylko w sytuacji, gdy proces nie
zostanie w sposób przypadkowy lub celowy przerwany przed upływem około 100 minut,
może nastąpić wprowadzenie do komórki biorcy całego chromosomu dawcy. Jest to sytuacja
raczej teoretyczna, w praktyce zazwyczaj następuje przedwczesne przerwanie procesu. Tak
więc największą szansę „wejścia” do komórki biorcy mają geny zlokalizowane blisko miejsca
połączenia z plazmidem. Zjawisko to jest wykorzystywane do mapowania genów na
chromosomie (oraz określania punktu początkowego wprowadzania chromosomu), poprzez
eksperymentalne porównywanie częstości pojawiania się w biorcy odpowiednich markerów
genetycznych dawcy (lub alternatywnie czasu ich wejścia do biorcy z zastosowaniem technik
koniugacji przerywanej).
134. Krzyżówki bakteryjne: bakteria męska (F
+
) x bakteria żeńska (F
-
).
W przypadku krzyżowania komórek F
-
i F
+
uzyskujemy F
+
.
Po zmieszaniu komórek F
-
i F
+
wykrywa się z bardzo małą częstością (ok. 1 na 10
5
komórek) rekombinanty będące wynikiem skrzyżowania i mające cechy obu partnerów, przy
czym z reguły przeważają cechy komórek F
-
. Zjawiskiem regularnie obserwowanym jest
natomiast przemiana bakterii F
-
w F
+
, z tym że inne cechy komórek F
-
nie podlegają żadnym

zmianom. Mamy więc tutaj do czynienia z jednokierunkowym przekazywanie cechy, w tym
przypadku cechy „bycia” F
+
.
135. Plazmid F` pierwotny i wtórny.
Czynnik F' – F zawierający geny bakteryjne, powstaje w wyniku nieprecyzyjnego
wycinania F z chromosomu bakterii, np. F'(lacZ) = F niosący gen lacZ F' może zawierać do
15% genomu E .coli.
Wbudowanie się czynnika F w chromosom bakteryjny jest odwracalne. Czynnik F
może zostać wycięty z chromosomu, co powoduje rewersję Hfr do F+. Częstość wycięcia jest
zbliżona do częstości integracji, a proces przebiega, gdy miejsce wycięcia jest identyczne z
miejscem wstawienia. W rzadkich przypadkach miejsca wycięcia i integracji nie pokrywają
się, co prowadzi do przyłączenia fragmentu DNA gospodarza do uwolnionego czynnika F.
Czynnik F z małym odcinkiem chromosomowego DNA to czynnik F'. Widoczne jest
podobieństwo w sposobie powstawania F' i fagów transdukujących.
Komórki zawierające czynnik F" to tzw. pierwotne komórki F'. DNA połączony z
czynnikiem F' może być przeniesiony z dawcy F' do biorcy F~ z 100% częstością, tak jak
czynnik F ze szczepu F+ do F . Ten sam segment chromosomowego DNA byłby przeniesiony
z Hfr do F~ z częstością wynoszącą jedynie 1%. Przeniesienie czynnika F' z pierwotnej
komórki F' do następnej komórki F~ prowadzi do powstania wtórnej komórki F', w której
fragment chromosomu bakteryjnego jest podwojony (diploidalny).
136. Mechanizm powstawania merodiplodalnych komórek E.coli.
Medodiploidalność to stan diploidalności ograniczony do niewielkiej części genomu.
Czynnik F`lac lub inny F` może być koniugacyjnie przekazany bakterii F
-
. Bakteria taka
będzie zawierać odcinek chromosomu w dwóch egzemplarzach, jeden w chromosomie, drugi
w F`lac. Na tym odcinku układ będzie więc diploidalny. Układ taki – częściowo diploidalny –
nazywamy merodiploidem.
Medodiploid jest dość trwały i utrzymuje się przez wiele pokoleń. W merodiploidach
możemy zaobserwować zjawisko komplementacji genetycznej. Zdolność Escherichia coli do
fermentacji laktozy jest zależna od dwóch genów; genu lacZ, kodującego β-galaktozydazę –
enzym rozkładający laktozę na glukozę i galaktozę, oraz genu lacY, kodującego permeazę
laktozową – białko błony rozpoznające laktozę i wychwytujące je ze środowiska. Gdy
bakteria nie ma jednego z tych enzymów, laktozy nie zużywa.
Szczep bakterii F
-
skrzyżowany z komórką męską typu F`lac, która na plazmidzie ma
czynny gen lacZ, a nieczynny lacY. W komórce, która otrzyma plazmid F`lac, będą się
znajdować się oba geny, ale jeden z nich (lacY) na chromosomie bakterii, a drugi (lacZ) na
plazmidzie. Komórka ta będzie zdolna do zużywania laktozy. Efekt ten nie będzie jednak
wynikiem rekombinacji, komplementacji: genom plazmidu będzie tu komplementował
(uzupełniał) genom bakterii, przy czym wszystkie komórki, które otrzymają plazmid, będą
fermentowały laktozę.
137. Transdukcja ogólna i specyficzna.
Transdukcją nazywamy przenoszenie materiału genetycznego pomiędzy komórkami
bakteryjnymi przez bakteriofag. Wyróżniamy dwa rodzaje transdukcji:
- niespecyficzną (ogólną) - gdy każdy odcinek DNA gospodarza może zostać przeniesiony
- specyficzną - gdy przenoszeniu ulegają tylko określone fragmenty DNA gospodarza.

138. Jakie fagi przeprowadzają te procesy.
Transdukcja specyficzna zachodzi, gdy odłączający się od chromosomu fagowy
DNA zabierze ze sobą mały fragment DNA bakteryjnego sąsiadujący z nim przed wycięciem
(niespecyficzne wycinanie się faga). Transdukcja specyficzna zachodzi np. za pośrednictwem
faga λ, który wbudowuje się do chromosomu bakteryjneg w ściśle określonym miejscu,
pomiędzy genami galE a bioA. Poprzez nieprecyzyjne wycięcie swego materiału
genetycznego z genomu lizogennej bakterii, fag może przenieść gen gal lub bio (bądź
fragment któregoś z nich).Taki kompleks DNA fagowego i fragmentu DNA gospodarza po
wycięciu się z chromosomu zachowuje się jako całość i jest w całości replikowany w
cytoplazmie bakterii, a następnie pakowany do główek faga. Jeśli taki fag zakazi nową
komórkę (biorcę),może dojść do rekombinacji pomiędzy chromosomalnym DNA gospodarza
(biorcy) a tym fragmentem DNA faga, który został zabrany z genomu dawcy. Fag λ
transdukuje (przenosi) zdolność do syntezy biotyny lub do fermentacji galaktozy.
Transdukcja ogólna jest przeprowadzana przez bakteriofag zjadliwe, takie jak P1 dla
Escherichia coli, P22 dla Salmonella, PPS1 i SP8 dla Bacillus subtilis. Średnia wielkość
przenoszonego fragmentu DNA jest równa ok. 400 p.z. Fag PPS1 przenosi fragmenty DNA
do 300 tys. p.z.
W przypadku transdukcji ogólnej wszystkie geny mają podobną szansę na
przeniesienie przez faga. Po wniknięciu faga do komórki często następuje fragmentacja jej
chromosomu na odcinki różnej długości. Przy dojrzewaniu fagów, często zamiast DNA
fagowego, do główki faga może być włączany fragment chromosomu bakteryjnego o długości
zbliżonej do długości DNA fagowego. Powstaje wtedy fag, który zamiast własnego DNA,
zawiera fragment DNA chromosomu bakterii, z której pochodzi. Pomiędzy tym fragmentem,
a chromosomem zakażonej p rzez takiego faga bakterii może dochodzić do rekombinacji.
Ponieważ włączanie fragmentu DNA do główki faga jest zdarzeniem przypadkowym, zatem
fragment ten
również jest przypadkowy, może zawierać różne geny. W transdukcji ogólnej
prawdopodobieństwo zajścia rekombinacji jednej określonej cechy jest bardzo małe, rzędu
od 10
-6
do 10
-8
na jedno zdarzenie transdukcyjne ( tzn. przez jedną cząstkę faga
transdukującego).
139.Na czym polega test fluktuacyjny Lurii i Delbrucka.
Doświadczenie Lurii i Delbrücka
W połowie XX wieku antybiotyki w badaniach mikrobiologicznych nie były jeszcze w
powszechnym użyciu. Z tego też względu wspomniani badacze do swoich doświadczeń
wykorzystali faga T1. Przygotowali oni faga w pożywce. Następnym krokiem było natarcie
płytek Petriego, zawierających pożywkę, fagami T1 w ilości 1010. Na tak sporządzone płytki
wylano około109 komórek E. coli, a więc około 1 ml hodowli bakteryjnej. Następnego dnia
na płytkach wyrosło kilkanaście hodowli bakteryjnych. Każda z wyrosłych kolonii zawierała
bakterie oporne na faga. Po wysianiu hodowli sporządzonej z komórek opornych na faga
otrzymano murawę. Taki wynik był punktem wyjścia do tzw. testu fluktuacyjnego.
Test fluktuacyjny Lurii i Delbrücka
Do testu fluktuacyjnego badacze przygotowali 20 probówek. Każda z nich była
napełniona płynną pożywką LB (około 5ml). Każdą probówkę indywidualnie zaszczepiono
jedną kolonią bakterii. Pojedynczą kolonią zainfekowano też kolbę zawierającą 100 ml
pożywki LB. Następnego dnia przygotowano płytki z podłożem LA zainfekowane fagiem. Na

każdą z płytek wysiano taką samą ilość zawiesiny bakterii z poszczególnych hodowli
znajdujących się w probówkach (cała hodowla była potomkiem jednej bakterii); w przypadku
hodowli inkubowanej w kolbie sporządzono 10 płytek. Tak przygotowane płytki wstawiono
do cieplarki; okazało się, że nastąpił wzrost kolonii na podłożach, ale ilość wyrosłych kolonii
była różna. Każda z wyrosłych kolonii była odporna na faga.
Badaczy zainteresował rozrzut statystyczny wyników. Otóż wyniki na poszczególnych
płytkach, na których posiano bakterie z poszczególnych probówek, miały ogromny rozrzut
ilości wyrosłych kolonii; były płytki na
których nie pojawiły się kolonie, a były takie, gdzie ich liczba przekraczała 100. Ilości kolonii
wyrosłych na wszystkich 10 płytkach, po wysianiu zawiesiny bakterii z kolby, były zbliżone i
wynosiły około 17.
Widzimy różnicę w rozbieżności wyników w poszczególnych wariantach
doświadczenia. Luria i Delbrück z powyższych wyników wyciągnęli wniosek, że na pewno
nie mają do czynienia z adaptacją jednorodną, bo gdyby tak było wszystkie wyniki byłyby
bardzo do siebie zbliżone. Bardziej prawdopodobne jest zjawisko adaptacji niejednorodnej
lub zjawisko spontanicznej mutacji chroniącej bakterie przed infekcją bakteriofagiem.
Najbardziej słuszna jest myśl o mutacji, ponieważ w przypadku adaptacji
niejednorodnej mielibyśmy do czynienia z podobnymi wynikami, a tak nie było. Mutacja
wydaje się być najbardziej sensownym sposobem nabycia przez bakterie odporności na faga.
Faktem popierającym to przekonanie było uzyskiwanie na podłożu z fagiem wzrostu kolonii
po wysianiu odpornych na faga komórek uzyskanych w poprzednim etapie doświadczenia.
Również wysiewanie kolonii z probówek na wiele płytek dawało powtarzalne wyniki
charakterystyczne dla poszczególnych prób, na wzór wysiewów z kolby.
Nazwa samego doświadczenia – „test fluktuacyjny” – wzięła się od niejednorodności
wyników.
Badacze przeprowadzający omawiane powyżej doświadczenie zdawali sobie
sprawę, że nie rozwiązali do końca zagadki związanej z pytaniem o podstawy mechanizmów
zmienności bakterii. Udało się im odkryć to, że bakterie się zmieniają, ale brak było
dowodów na to jak to czynić. Nadal nie wiedziano, czy komórki zmieniają się bo jest to
odpowiedź na obecność faga, czy owa zmienność jest samoistna , bezkierunkowa i nie
potrzebuje indukcji.
Odpowiedzi na nieznane udzielił Joshua Lederberg. Jego zasługi dla mikrobiologii i
ogólnie biologii są ogromne. To jemu prócz modyfikacji testu fluktuacyjnego zawdzięczamy
odkrycie płciowości bakterii i odkrycie możliwości przenoszenia informacji genetycznej
przez bakteriofagi.
140. Zmodyfikowany test fluktuacyjny przeprowadzony przez Lederberga.
Kluczem do sukcesu Lederberga był geniusz i jego czysto materialny owoc, jakim jest
stosowany dotychczas stempel welurowy, zwany również pieczęcią welurową. Stempel
Lederberga to metalowy walec, na który nakłada się kawałek weluru. Metalowy walec ma
średnicę pozwalającą na włożenie go do płytki i odciśnięcie na założonym welurze kolonii, a
następnie przeniesienie ich na nową płytkę. Welur jest materiałem posiadającym sterczące
sztywno włoski pozwalające na przenoszenie pojedynczych nawet bakterii; welur można
używać wielokrotnie dzięki temu, że można go poddawać sterylizacji.
Stemplowi Lederberga zawdzięczamy odkrycie tego, czy mutacje pojawiają się samoistnie,
czy też są indukowane warunkami środowiska. Lederberg w swoim doświadczeniu stosował,
w przeciwieństwie do Lurii i Delbrücka, nie bakteriofaga a antybiotyk – streptomycynę.
Punktem wyjścia w doświadczeniu Lederbrga była gęsta kolonia bakterii. Bakterie były
wysiewane początkowo na płytkę nie zawierającą czynnika selekcyjnego. Przygotowano też

płytki z pożywką LA zawierające streptomycynę – podłoże takie zostało nazwane
selekcyjnym. Na płytce nie zawierającej streptomycyny wyrosła murawa. Za pomocą stempla
płytka została odbita na podłożu selekcyjnym (użyto do tego trzech płytek) i na normalnej
płytce (użyto jednej płytki). Lederberg przestrzegał w stemplowaniu kierunku nanoszenia
odcisków – zgrywał odcisk ze stemplem uzyskujac powtarzalna lokalizację klonów
pochodzacych od bakterii z określonych szczepów. „Stemplowane” płytki inkubowano
w cieplarce. W efekcie inkubacji na płytkach pojawiły się kolonie bakterii – na podłożu bez
streptomycyny wzrost był obfitszy, a na płytkach ze streptomycyna wyrosło po kilkanaście
kolonii. Patrząc na lokalizację kolonii odpornych na streptomycynę, wyrosłych na podłożu
selekcyjnym, Lederberg wycinał jałowym nożykiem kolonie z odpowiednich miejsc na płytce
nie zawierającej czynnika selekcyjnego. Wyciąty fragment agaru był wrzucany do probówki i
opłukiwany w pożywce; następnie do probówki dodawano worteksu (zapobiegał namnażaniu
komórek). Pozyskane po opłukaniu z agaru komórki nie były namnażane, ale przy każdym
powtórzeniu doświadczenia ulegały rozcięczeniu.
Zawartość probówki była wysiewana ponownie na cztery płytki – jedna była z
pożywką normalną, a trzy zawierały antybiotyk. Po inkubacji postępowanie było podobne jak
w poprzednim przypadku, a więc następowało wycięcie, opłukanie i ponowne wysianie
bakterii, które nigdy nie spotkały się ze streptomycyna, a które były na nią odporne.
Przeprowadzona po raz kolejny próba dawała coraz czystsze szczepy; kolonii na płytkach
było coraz mniej (rozcięczanie hodowli), ale ich topografia na płytkach bez antybiotyku i ze
streptomycyną była łatwiejsza do określenia – kolonie te można już było na siebie nałożyć.
Ostatecznym efektem było wyhodowanie tylko tych kolonii bakteryjnych, które były oporne
na antybiotyk. Namnożenie takiej kolonii i jej wysianie dawało na płytce ze streptomycyną
murawą.
Z doświadczenia Lederberga wypływa jednoznacznie wniosek o tym, że zmienność
bakterii nie jest skutkiem zetknięcia się z czynnikiem stymulującym adaptację, ale jest
konsekwencją mutacji w genie. Potwierdzeniem tego jest istnienie bakterii odpornych na dany
czynnik (w rozpatrywanym przypadku jest nim antybiotyk) mimo braku wcześniejszego
kontaktu z tym czynnikiem.
141. Mutageneza, typy mutacji, mutageny.
Mutacja jest to nagłe pojawienie się, z małą częstotliwością, komórki zmienionej i
zdolnej do przekazywania zmienionych cech swemu potomstwu. Mutantem jest forma o
zmienionym genotypie. Proces prowadzący do powstania mutacji nazywamy mutagenezą.
Przyczyną obserwowanej zmiany jest modyfikacja w pierwszorzędowej strukturze
DNA. Może to być zmiana jednego nukleotydu przez drugi, opuszczenie nukleotydu, lub
wreszcie wstawienie w łańcuchu dodatkowego nukleotydu. Mutacje mogą polegać na
podstawieniu zasad (czyli nukleotydów), ich opuszczeniu lub dodaniu. Zdarzenie takie może
być skutkiem modyfikacji matrycy przed jej kopiowaniem (replikacją) lub może wynikać z
błędów kopiowania. Mutacje mogą też być spowodowane ubytkiem kilku lub wielu
nukleotydów, wstawieniem kilku lub wielu nukleotydów, albo odwróceniem kolejności
ułożenia zasad w sekwencji.
Typy mutacji:
- delecja - polega na utracie jednej lub kilku par
- insercja -
polegająca na wstawieniu krótkiej sekwencji
lub kilku nukleotydów
- tranzycja - zmiana prawidłowych
na inne w ramach jednej grupy zasad
azotowych (
(i na odwrót)
- transwersja -
w której zasada purynowa ulega zamianie na pirymidynową lub
odwrotnie

- substytucja - mutacji , w której dochodzi do zmiany składu
poprzez
zamiast
Mutageny:
- promieniowanie UV
- promieniowanie jonizujące
- analogi zasad
- barwniki akrydynowe
- kwas azotanowy
142. Mutanty auksotroficzne.
Mutanty auksotroficzne posiadają mutacje osłabiające ich przeżywalność; takie
mutanty nie potrafią syntetyzować któregoś z niezbędnych im do życia składników i
potrzebują dodatku tej brakującej substancji do pożywki minimalnej. Mutanty auksotroficzne
są defektywne w syntezie witamin, aminokwasów, kwasów nukleinowych lub innych
komponentów budujących komórkę.
143.Metoda otrzymywania mutantów auksotroficznych poprzez zagęszczenie
penicylinowe.
Udało się opracować metodę izolacji auksotrofów wykorzystując swego rodzaju
stagnację w ich rozwoju.
Rozpatrzmy przypadek E. coli. Prototroficzna bakteria – pałeczka okrężnicy – rośnie
na pożywce mineralnej zawierającej glukozę jako jedyne źródło węgla i dodatek soli
mineralnych. Szczep E. coli trp
-
oprócz pożywki mineralnej potrzebuje tryptofanu, bo sam nie
jest w stanie go wytworzyć.
Izolowanie szczepów bakterii prototroficznych przebiega w kilku etapach. Pierwszm z
nich jest poddanie auksotroficznych bakterii działaniu czynnika mutagennego (może to być
np. promieniuwanie UV). Bakterie wysiewamy na podłoże mineralne z dodatkiem penicyliny.
Penicylina działa na bakterie podczas ich wzrostu – hamuje rozbudowę ściany komórkowej.
Bakterie auksotroficzne, powstałe w wyniku działania mutagenu na pożywce mineralnej nie
urosną – ich rozwój będzie zatrzymany, ale jednoczeęnie nie zostaną zniszczone na skutek
działania penicyliny. Takie działanie nosi miano „zagęszczania penicylinowego”; jest to
eliminowanie prototrofów i pozostawianie nierosnących auksotrofów. Kolejnym krokiem jest
pozbycie się penicyliny z hodowli i wysianie bakterii na podłoże bogate, wzbogacone w
rozpatrywanym przypadku w tryptofan. Urosną tylko bakterie E. coli Trp
-
.
Stosując podłoża bogate różnego rodzaju możemy uzyskać różne auksotrofy; będą na
takich podłożach rosły bakterie genetycznie upośledzone pod względem syntezy konkretnego
(a w tym przypadku podanego) czynnika.
144. Mechanizmy działania mutagenów.
- barwniki akrydynowe np. proflawina
Cząsteczki tych barwników są płaskimi pierścieniowymi strukturami, wykazującymi dzięki
grupom aminowym duże powinowactwo do kwasów nukleinowych. Wiążą się one z DNA,
między innymi w ten sposób, iż układają się między również płaskimi, równolegle do siebie
leżącymi cząsteczkami zasad. Prowadzi to do odkształcenia spirali DNA, a w rezultacie do
opuszczenia lub dodania nukleotydów w czasie replikacji.

- analogi zasad
Są to pochodne normalnych zasad, tak do nich podobne stechiometrycznie, że mogą
zajmować ich miejsce. Takimi związkiem jest na przykład 5-bromouracyl, będący analogiem
tyminy, z tym że miejsce grupy metylowej zajmuje atom bromu, oraz 2-aminopuryna, analog
adeniny, różniący się od niej tym, że grupa aminowa jest w pozycji 2, nie zaś w 6. Jeśli takie
analogi znajdują się w środowisku i dostaną się do komórki, mogą być w czasie replikacji
wstawione zamiast właściwych zasad do rosnącego łańcucha polinukleotydowego.
Tautomeryzacja 5-bromouracylu, będąca zjawiskiem dość częstym, prowadzi do zmiany pary
A-T w parę G-C lub odwrotnie.
- promieniowanie UV
Promieniowanie ultrafioletowe o fali 250-260 nm są silnie pochłaniane przez kwasy
nukleinowe. Energia ich fotonów jest dostateczna, by rozbić wiązanie C-N, C-C i C=C, nie
wystarcza jednak zazwyczaj do zupełnego wybicia elektronu z cząsteczki naświetlanej, czyli
do jej jonizacji. Działanie mutagenne jest głównie związane z dimeryzają tyminy, w
mniejszym stopniu z dimeryzacją lub hydratacją cytozyny. Sąsiadujące ze sobą w łańcuchu
polinukleotydowym tyminy, cytozyny lub tyminy i cytozyny, poddane naświetlanu falami
250-275 nm, reagują ze sobą. Najczęściej powstają dimery tymin o budowie cyklobutanu.
Tymina, po dimeryzacji, nie jest zdolna do tworzenia mostków wodorowych. Skutkiem tego
jest zaburzenie replikacji, jeśli nić zawierająca dimery wykorzystywana jest jako matryca. W
takiej nowo replikowanej nici naprzeciw dimerów tyminy powstają luki. Ilość dimerów
tyminy może być znaczna. Po naświetlaniu promieniami o fali 275 nm do 20% tymin może
występować w postaci dimerów. Ilość ta zależy też oczywiście od natężenia promieniowania.
- chemiczne czynniki mutagenne
Kwas azotanowy deaminuje cytozynę i adeninę. Działa on silnie na RNA i jednoniciowy
DNA. Hydroksyloamina działa na pirymidyny, a przede wszystkim na cytozynę. Do
najefektowniejszych środków mutagennych należą związki alkilujące, tj. przyłączające do
zasad grupę alkilową (CH
3
, C
2
H
5
itd.). Są to na przykład iperyt gazowy, dimetylosulfonian
(DMS), etylometylosulfonian (EMS), związki epoksydowe, takie jak np. diepoksybutan
(DEB). Alkilowanie zmienia strukturę zasad, wskutek czego podlegają one odczepieniu od
pentozy w łańcuchu polinukleotydowym. Ponieważ najczęściej alkilacji ulegają puryny (G i
A), zatem dochodzi do ich odłączenia, czyli tzw. depurynacji. Do wolnego wiązania pentozy,
pozostającego po depurynacji, może następnie przyłączyć się dowolna zasada i często będzie
nią inna niż została oderwana.
146. Odpowiedź SOS w komórkach E. coli.
147. Rola białka RecA w odpowiedzi SOS.
Jeśli pod wpływem mutagenu powstaną bardzo liczne uszkodzenia DNA tak, iż
systemy reparacyjne nie są w stanie ich w pełni eliminować, to w DNA powstają liczne
pęknięcia i następuje fragmentacja cząsteczek DNA. W tej krytycznej sytuacji następuje
indukcja systemu, zwanego SOS.
Indukcja dotyczy kilkunastu rożnych genów biorących udział w rekombinacji i
reperacji DNA. Wśród indukowanych genów jest też gen recA. U E.coli występuje gen LexA
kodujący białko represorowe które hamuje nie tylko produkcję samego represora LexA, ale
także białka RecA i kilkunastu innych białek związanych z rekombinacją i reperacją DNA,
wiążąc się z określoną sekwencją sygnałową w obszarach promotorowych genów kodujących
te białka. Indukcja wynika z inaktywacji represora LexA na skutek proteolitycznego
przecięcia tego polipeptydu. Przecięcia dokonuje kompleks białka RecA z jednoniciowymi
fragmentami DNA, powstający w momencie zahamowania replikacji DNA i jego
zaczynającej się degradacji. W wyniku proteolizy represora następuje derepresja genu RecA i

gwałtowny wzrost ilości białka RecA w komórce. Jednocześnie indukcji podlega ekspresja
innych genów reprymowanych przez represor LexA, jak na przykład uvrA, uvrB, uvrC, uvrD
oraz innych genów o bliżej nieznanych funkcjach, jak umuC, umuD. Być może
geny umuC i umuD kodują białka, które nadają polimerazie DNA nowe właściwości
losowego włączania zasad do nowo syntetyzowanego łańcucha DNA, gdy w łańcuchu
matrycowym występuje luka czy na przykład dimery pirymidynowe.
Reakcja SOS zwiększa istotnie przeżywalność komórek po działaniu mutagenu w
wyniku wzmożonej rekombinacji, a także wypełniania luk występujących w DNA mimo
braku nie uszkodzonej matrycy. Oczywiście taki proces reperacji DNA powoduje istotny
wzrost częstości mutacji w wyniku losowego włączania zasad i kontynuacji syntezy DNA
mimo braku właściwej matrycy. Do tej pory system reperacji SOS został opisany jedynie u
bakterii.
Pojęcia dla zrozumienia tekstu:
Represor - w transkrypcji DNA to substancja spowalniająca wydajność wytwarzania mRNA.
reprymowany - hamowany
Indukcja - wyzwolenie określonego procesu w organizmie przez proces go poprzedzający.
Wprowadzenie w jakiś stan, wzbudzenie jakiegoś zjawiska.
Promotor genu – odcinek DNA, położony zazwyczaj powyżej sekwencji kodującej genu,
który zawiera sekwencje rozpoznawane przez polimerazę RNA zależną od DNA. Po
połączeniu się polimerazy RNA z promotorem rozpoczyna się transkrypcja (proces
przepisywania informacji genetycznej z DNA na RNA).
Proteoliza – rozkład białek na peptydy i aminokwasy
148. Test Ames`a, na czym polega, zastosowanie.
Ames i współpracownicy opracowali bakteriologiczną metodę pozwalającą na wykrycie
uszkodzeń DNA powodowanych przez daną substancję, co wskazuje na jej potencjalne
właściwości rakotwórcze.

149. Enzymy uczestniczące w procesie replikacji i transkrypcji u E. coli.
Transkrypcja
Inicjacja transkrypcji u E. coli polega na związaniu się
odpowiednim odcinkiem pasma matrycowego DNA - tzw.
. Polimeraza
promotora (a transkrypcja zaczyna się od nukleotydu +1).
Specyficzność wiązania zapewnia czynnik σ (sigma). Rozsunięcie nici DNA na odcinku
kilkunastu nukleotydów (czyli powstanie tzw. kompleksu otwartego) umożliwia wstawianie
(włączenie) kolejnych, odpowiednich nukleotydów. Substratami są trifosforany
rybonukleozydów (
). Transkrypcja zaczyna się od produkcji
kilku krótkich (kilka nukleotydów) transkryptów. Dopiero po oddysocjowaniu czynnika σ
może rozpocząć się kolejny etap - elongacja transkrypcji (wydłużanie RNA). Polimeraza
RNA przesuwa się systematycznie wzdłuż
DNA, rozplatając ją (na odcinku kilkunastu
) i wydłużając łańcuch RNA, przy czym nukleotydy włączane są zgodnie z
. Powyżej aktualnego miejsca syntezy powstający hybrydowy kompleks
DNA - RNA ulega rozpadowi, DNA powraca do swojej pierwotnej dwuniciowej struktury, a
łańcuch powstającego
oddziela się. Etap elongacji kończy się, gdy polimeraza RNA
dotrze do terminatora - sekwencji kończącej, wyznaczającej miejsce terminacji (zakończenia)
transkrypcji. Sekwencja taka tworzy strukturę szpilki do włosów (hairpin), która zatrzymuje
polimerazę RNA, co powoduje rozpad kompleksu enzym - DNA - RNA. Drugim
mechanizmem terminacji wykorzystywanym przez bakterie jest terminacja rho-zależna, gdzie
do terminacji transkrypcji potrzebne jest działanie czynnika rho (ρ). Transkrypt
prokariotyczny nie wymaga dalszej obróbki, a
rozpoczyna się, zanim transkrypcja
dobiegnie końca.

Replikacja
W komórkach E. coli głównym enzymem katalizującym replikację DNA jest
polimeraza III.
Polimerazy DNA, w tym polimeraza DNA III, mogą przyłączać nowy nukleotyd tylko
do końca 3`-OH w DNA. W związku z tym replikacja zachodzi zawsze w kierunku od 5` do
3`. Replikacja w E. coli zaczyna się w miejscu zwanym origin, będącym odcinkiem DNA
liczącym około 250 nukleotydów. Z odcinkiem tym związane są liczne białka inicjujące
replikację, m. in. Białko DnaA. Białko DnaA wiąże się do helisy DNA w origin replikacji, po
uprzednim przyłączeniu ATP. Z origin replikacji łączy się też enzym, helikaza DNA,
wykorzystujący energię z hydrolizy ATP do rozwijania podwójnej helisy DNA i posuwania
się wzdłuż niej, tworząc widełki replikacyjne. Kompleks helikazy z prymazą nazywany jest
prymosomem. Rozdzielone nici DNA są stabilizowane w formie jednoniciowej przez swoiste
białka zapobiegające ponownemu tworzeniu wiązań wodorowych między nimi. Niezależnie
od tego, czy replikacja jest ciągła, czy w postaci fragmentów Okazaki, najpierw musi powstać
„primer”, czyli odcinek RNA zawierający wolną grupę 3`-OH. Jest on syntetyzowany przez
enzym zwany prymazą RNA. Przy replikacji ciągłej primer powstaje tylko w miejscu origin.
Gdy synteza odbywa się w sposób nieciągły, krótki jedenastonukleotydowy odcinek RNA
(primer) poprzedza syntezę każdego kolejnego fragmentu Okazaki. Po powstaniu primera
polimeraza DNA III dołącza nukleotydy do końca 3`-OH. Po spełnieniu swojej funkcji,
primer jest usuwany, a na jego miejsce wstawiane są deoksyrybonukleotydy komplementarne
do matrycy. W reakcja tych uczestniczy polimeraza DNA I wraz z jej egzonukleolityczną
aktywnością 5` -> 3` oraz rybonukleaza H1. powstanie ostatniego wiązania fosfodiestrowego
katalizowane jest przez enzym zwany ligazą. W replikacji DNA E. coli nie bierze udziału
polimeraza DNA II.
Wyszukiwarka
Podobne podstrony:
Genetyka bakterii fermentacji mlekowej
Genetyka bakterii tekst egz 2009
GENETYKA BAKTERII- sciaga, Edukacyjnie, K, Kosmetologia, Technik usług kosmetycznych, bakteriologia
lab6wyklad Genetyka bakterii fermentacji mlekowej
GENETYKA BAKTERII, Mikrobiologia
Genetyka bakterii fermentacji mlekowej
Żymańczyk Duda, mikrobiologia, GENETYKA BAKTERII
Modyfikacje genetyczne bakterii
GENETYKA MOLEKULARNA BAKTERII I WIRUSÓW(1)
Bakteriocyny i L-antybiityki, Naukowe PL, Biotechnologia, Enzymologia, Genetyka
Genetyka i czynniki zjadliwości bakterii
IV-Transformacja bakterii, Genetyka
GENETYKA MOLEKULARNA BAKTERII I WIRUSÓW
B M GENETYKA MOLEKULARNA BAKTERII I WIRUSÓW
Choroby bakteryjne wirusowe i genetyczne
więcej podobnych podstron