
Transkrypcja i rola RNA w komórce
1. Zasada transkrypcji z DNA do RNA
2. Transkrypcja u bakterii i operon
3. RNA eukariontów
4. Regulacja transkrypcji u eukariontów
5. Usuwanie intronów
6. Alternatywne składanie egzonów
7. Degradacja RNA
8. Wyciszanie RNA
9. Transportowanie RNA
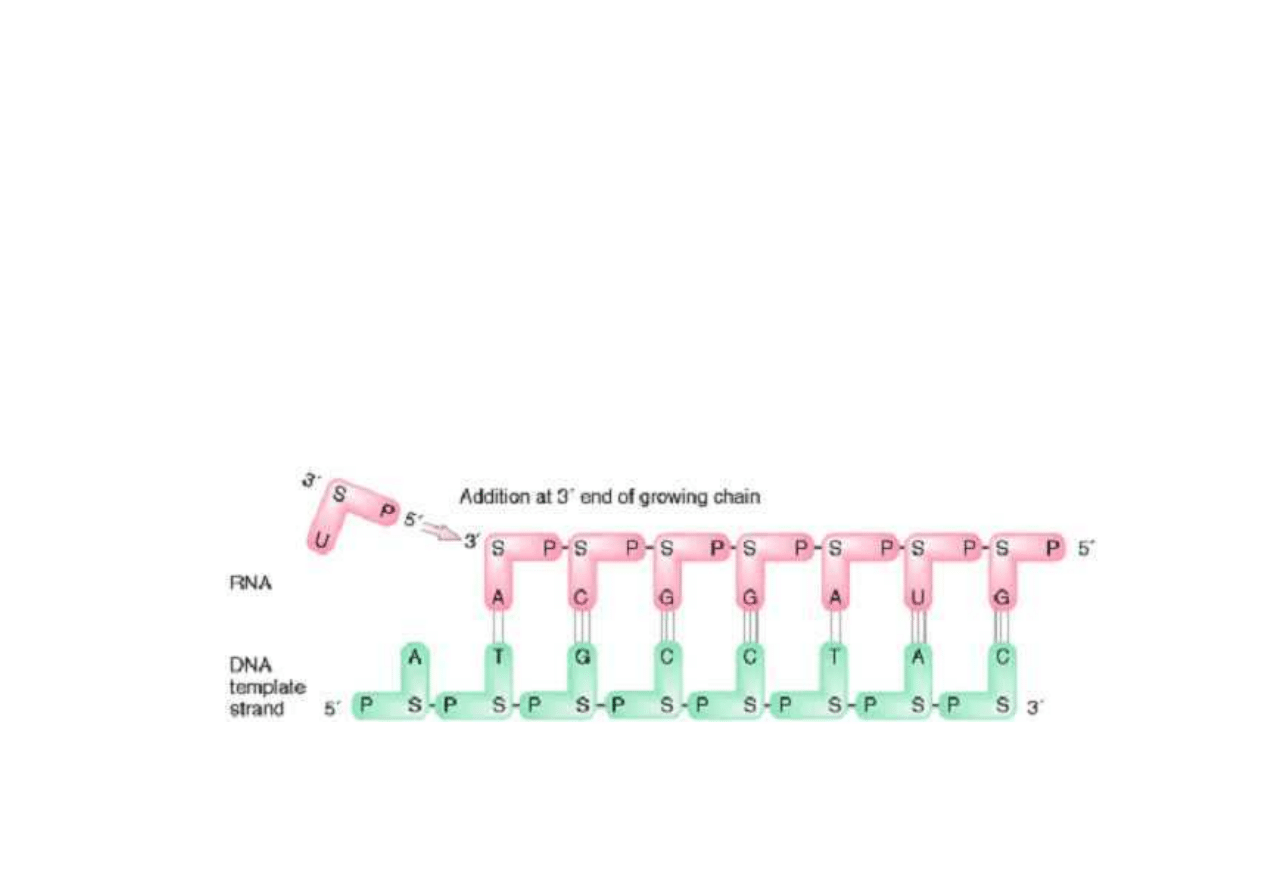
1. Zasada transkrypcji z DNA do RNA
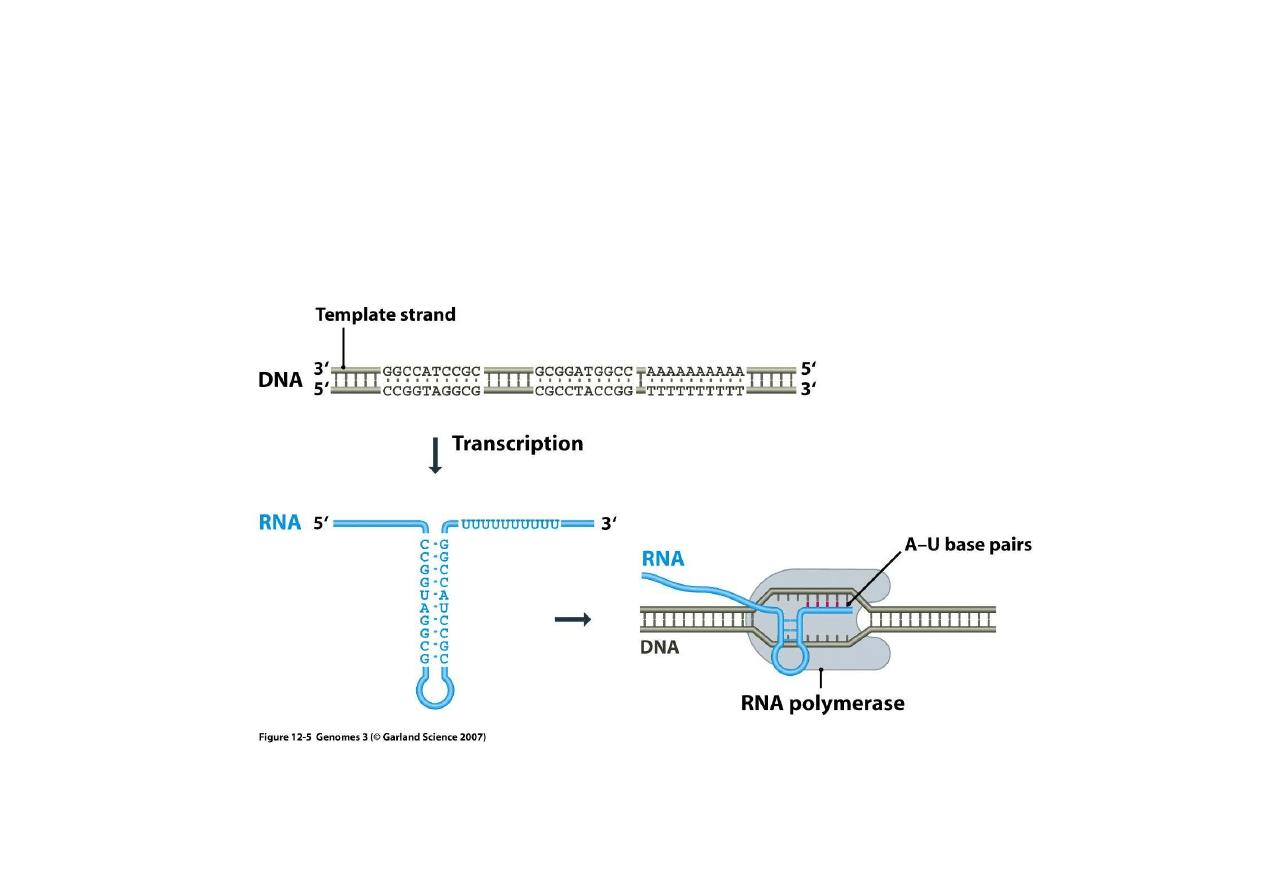
Transkrypcja, czyli syntetyzowanie mRNA na matrycy DNA, polega zawsze na
dodawaniu rybonukleotydów przez polimeraz
ę
RNA do ko
ń
ca 3’ syntetyzowanej nici
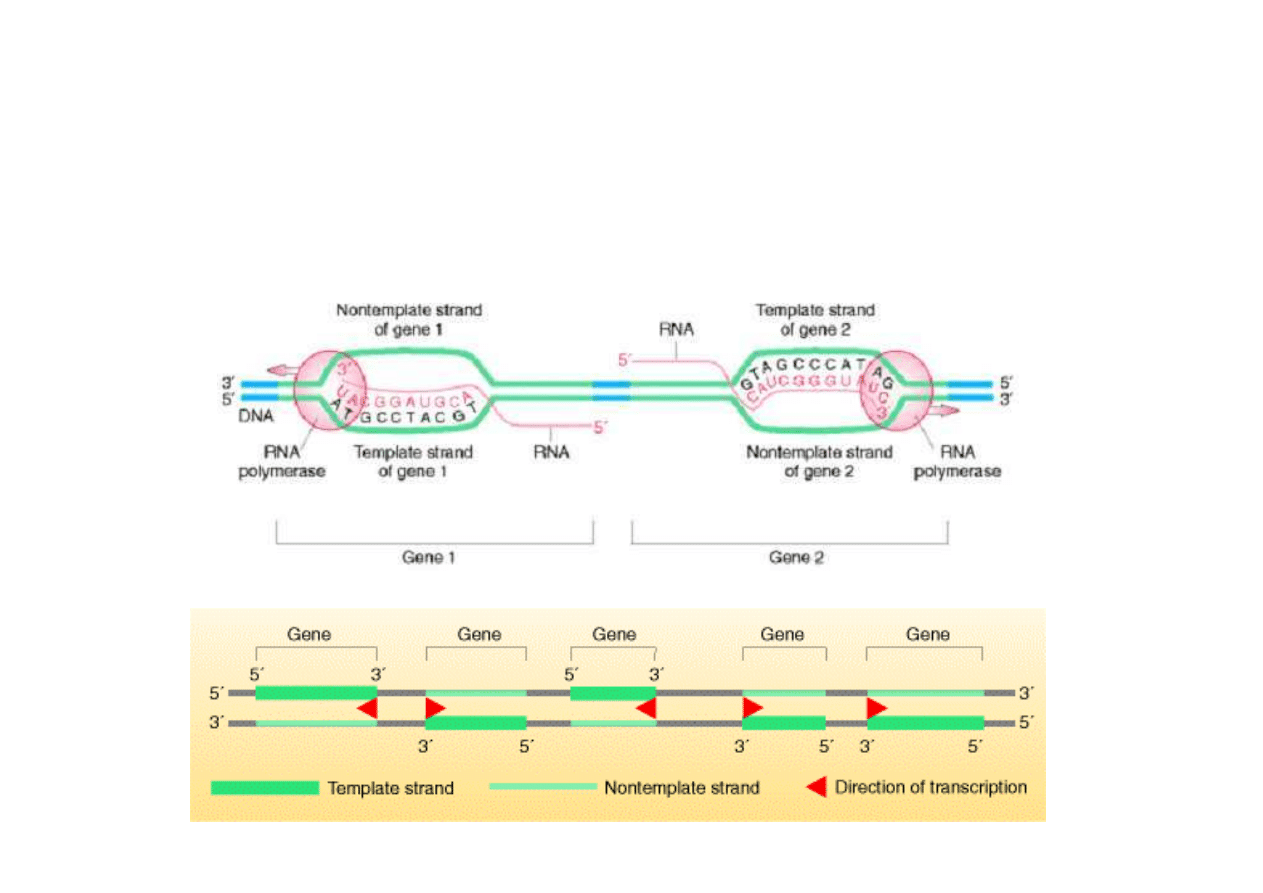
1.
Zasada transkrypcji z DNA do RNA
W czasie transkrypcji nici DNA rozdzielaj
ą
si
ę
na stosunkowo krótkim odcinku: jedna ni
ć
jest czynna jako matryca (template) a druga bierna. Nawet gdy geny le
żą
blisko siebie,
to nie zawsze ta sama ni
ć
jest czynna, co sprawia,
ż
e kierunek przesuwania si
ę
polimerazy RNA wzd
ł
u
ż
chromosomu jest ró
ż
ny w ró
ż
nych genach
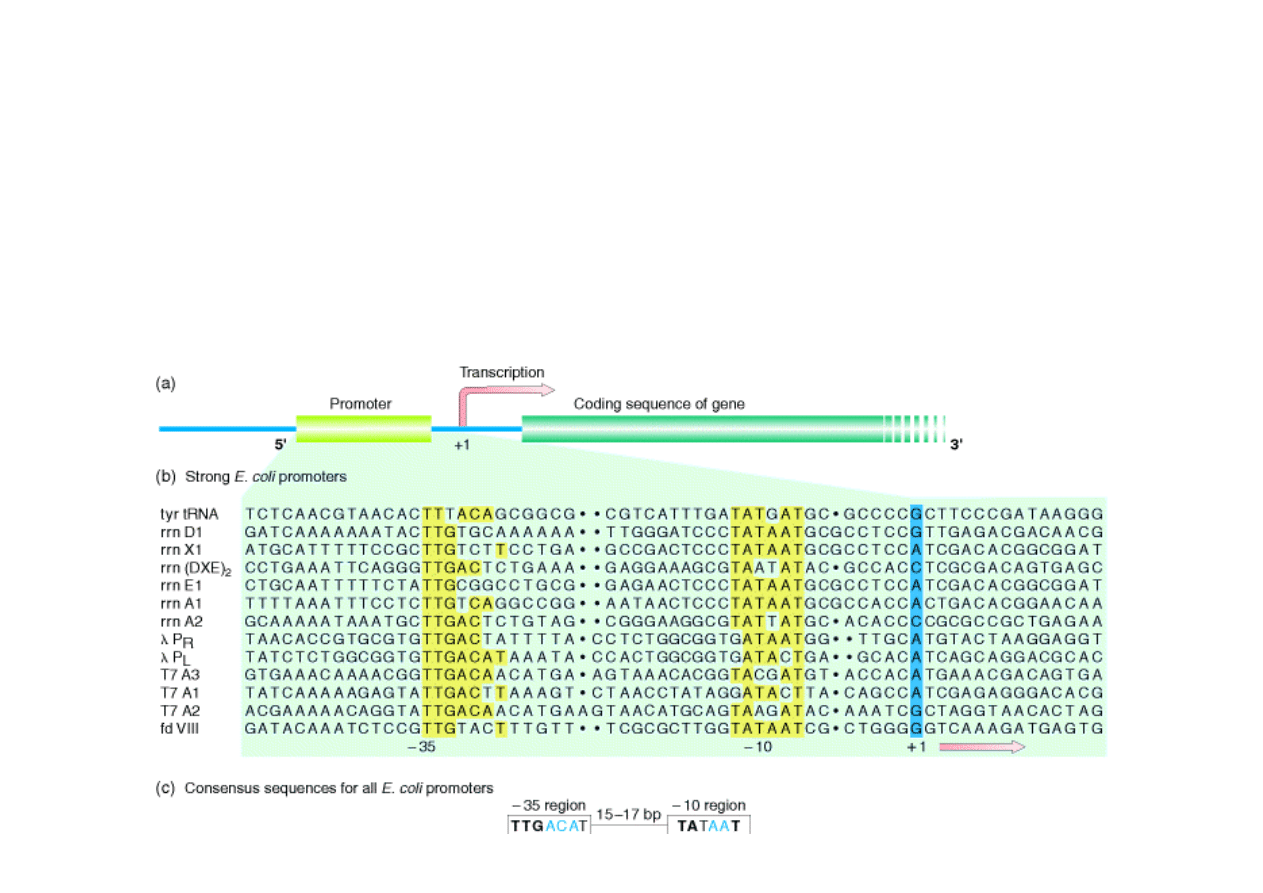
2.
Transkrypcja u bakterii -
inicjacja
Na transkrypcj
ę
sk
ł
adaj
ą
si
ę
trzy etapy:
- inicjacja: polimeraza RNA rozpoznaje promotory czyli odcinki DNA zawieraj
ą
ce
specyficzne sekwencje; u E.coli około 35 zasad przed pocz
ą
tkiem transkrypcji wyst
ę
puje
sekwencja TTGACAT, a około10 zasad przed tym pocz
ą
tkiem sekwencja TATAAT
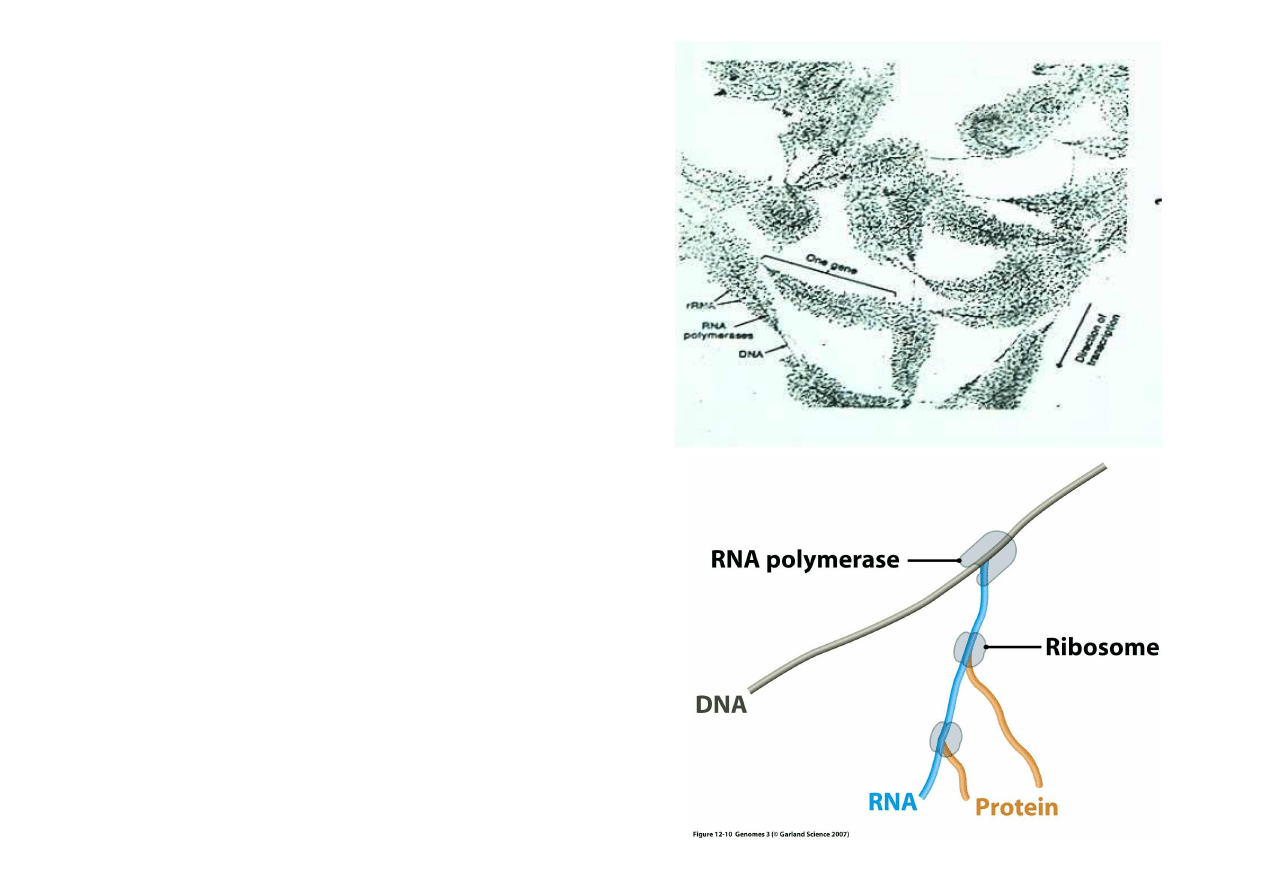
2. Transkrypcja u bakterii
- elongacja: dodawanie nukleotydów
z wykorzystaniem energii
zgromadzonej w dostarczanych do tej
reakcji nukleotydów z trójfosforanem
;
jeden gen mo
ż
e by
ć
odczytywany
przez wiele kompleksów polimerazy
RNA na raz, dlatego produkcja mRNA
mo
ż
e by
ć
obfita
... co wi
ę
cej, do nieuko
ń
czonych
ła
ń
cuchów RNA doczepiaj
ą
si
ę
rybosomy i rozpoczyna si
ę
translacja;
w ten sposób z jednego odcinka DNA
powstaje od razu wiele polipeptydów
(w genach gdzie transkrypcja jest
intensywna)
2. Transkrypcja u bakterii
- terminacja, zale
ż
y cz
ę
sto od sekwencji DNA, która le
ż
y za genem; typow
ą
przyczyn
ą
terminacji u E. coli jest taka sekwencja DNA, która
transkrybowana na mRNA powoduje,
ż
e tworzy ono p
ę
tle b
ę
d
ą
ce sygna
ł
em
dla polimerazy RNA by od
łą
czy
ć
si
ę
od DNA
2. Transkrypcja u bakterii
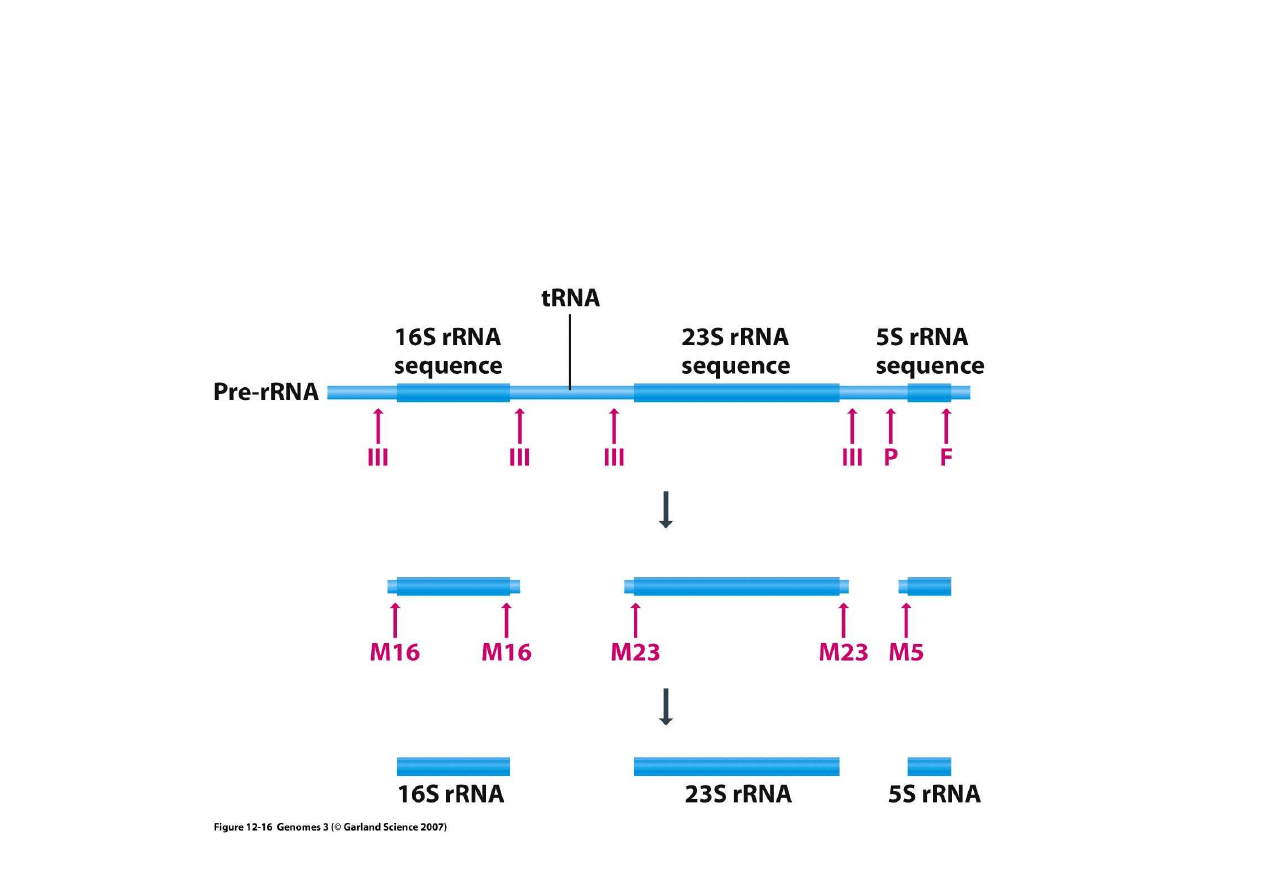
Obróbka zsyntetyzowanego bakteryjnego RNA jest mało intensywna.
Bakteryjny mRNA nie jest modyfikowany, nadaje si
ę
od razu do translacji.
Natomiast bakteryjne rRNA (wchodzi w skład rybosomów) i tRNA
(transportuj
ą
cy) wchodz
ą
w skład długich pre-RNA, które s
ą
potem ci
ę
te
przez specyficzne rybonukleazy (w przykładzie M5, M16, M23)
2. Transkrypcja u bakterii
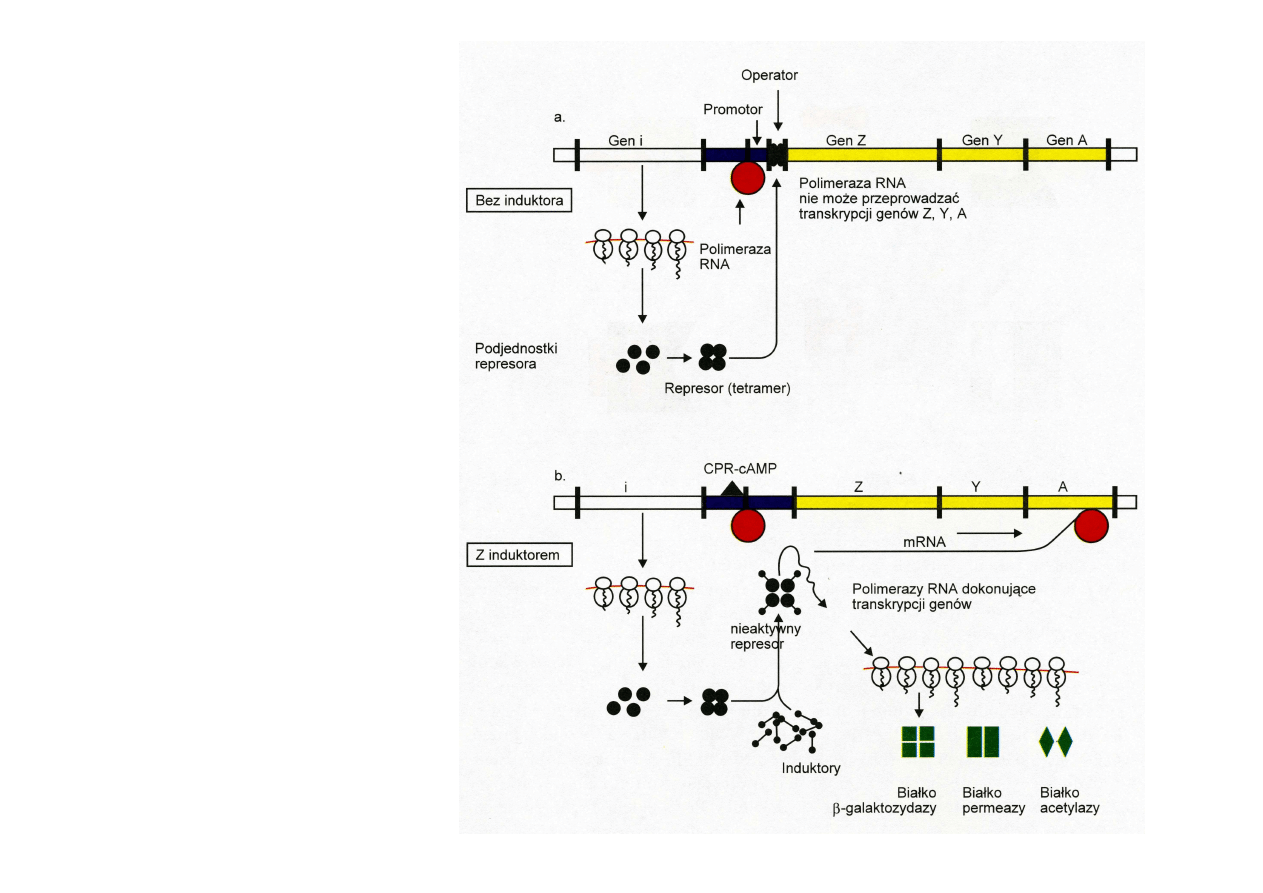
- operon.
- ekspresja stała
(konstytutywna) wyst
ę
puje
w przypadku wielu genów,
których produkty s
ą
zawsze
potrzebne w komórce (na
rycinie obok stale jest
produkowane białko
represora słu
żą
ce
normalnie do wyciszenia
operonu laktozy)
-ekspresja indukowana:
(a) operon laktozy -
regulacja negatywna –
normalnie wył
ą
czony,
obecno
ść
laktozy go
uruchamia
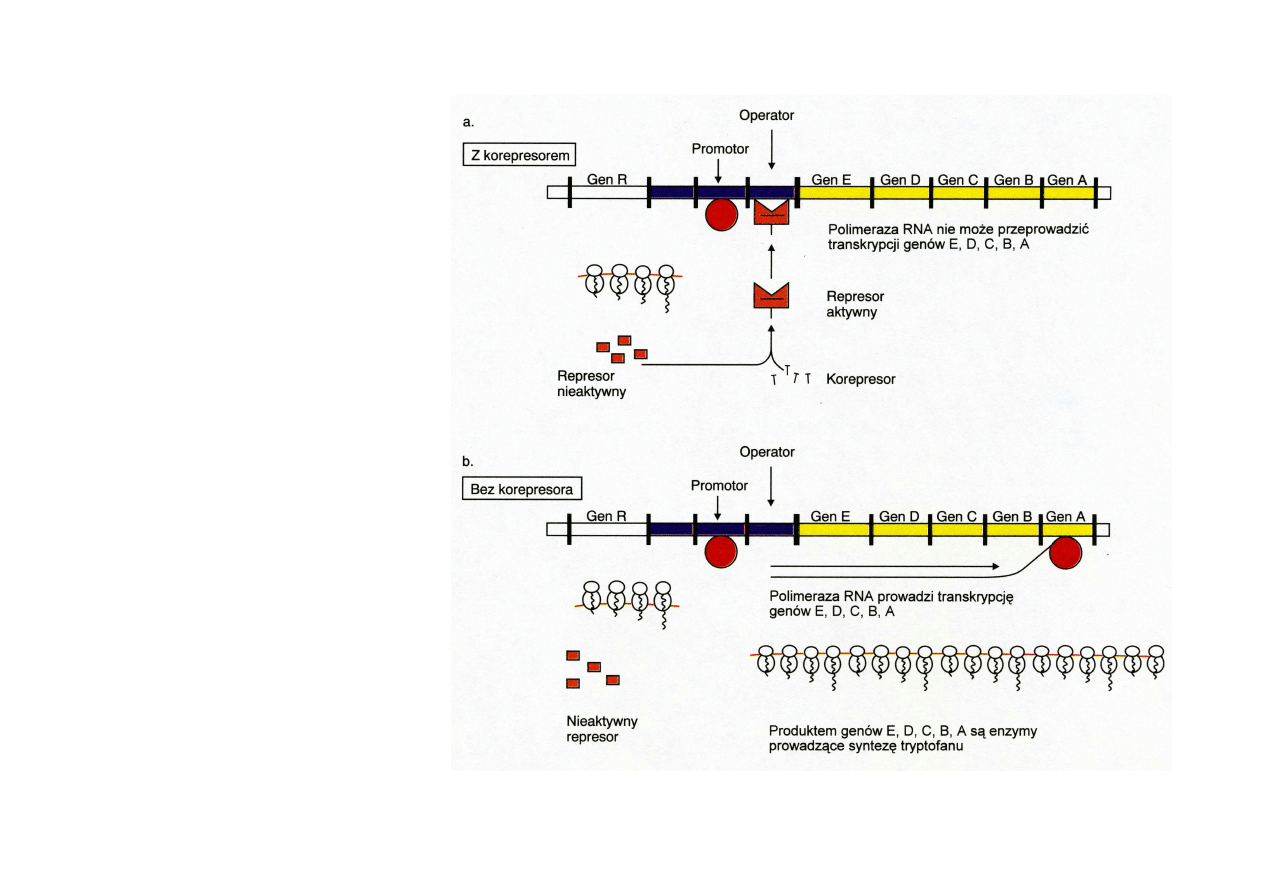
2. Transkrypcja u bakterii -
operon
- ekspresja indukowana:
operon tryptofanu - regulacja
pozytywna – normalnie
czynny, obfito
ść
tryptofanu
go unieczynnia
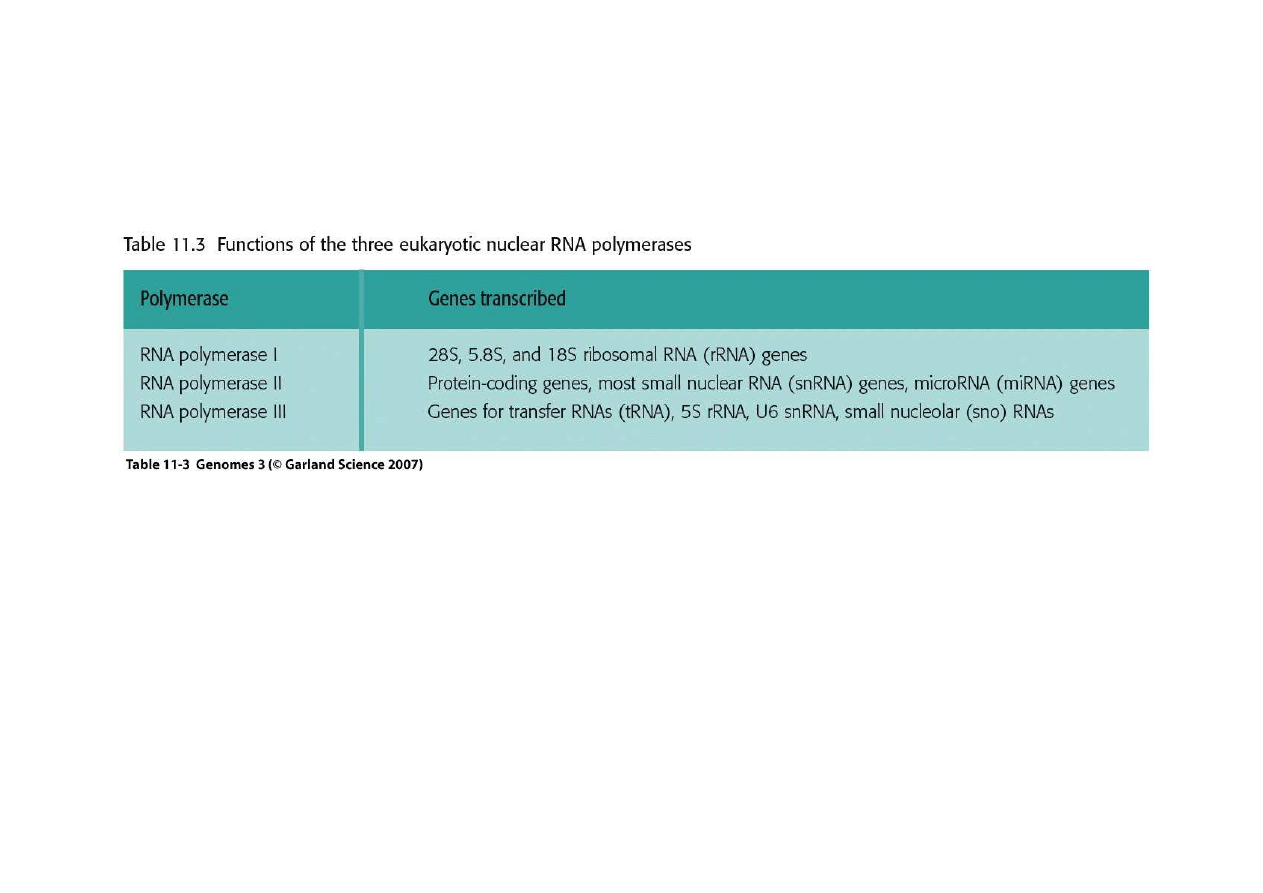
3. RNA eukariontów
W komórkach eukariontów s
ą
trzy polimerazy RNA, ka
ż
da transkrybuje inne rodzaje RNA
mRNA – transkrypty genów daj
ą
cych bialka (protein coding RNA)
tRNA – transport aminokwasów do rybosomu
rRNA – rybosomalne RNA, komponent rybosomów, stanowi 80% całego RNA w komórce
snRNA – small nuclear RNA, bior
ą
udział w przycinaniu pre-mRNA do dojrzałego mRNA
snoRNA – w j
ą
derku, uczestnicz
ą
w dojrzewaniu rRNA
miRNA – mikro RNA – małe cz
ą
steczki RNA, reguluj
ą
ekspresj
ę
indywidualnych genów
siRNA – short interfering RNA, reguluj
ą
ekspresj
ę
indywidualnych genów
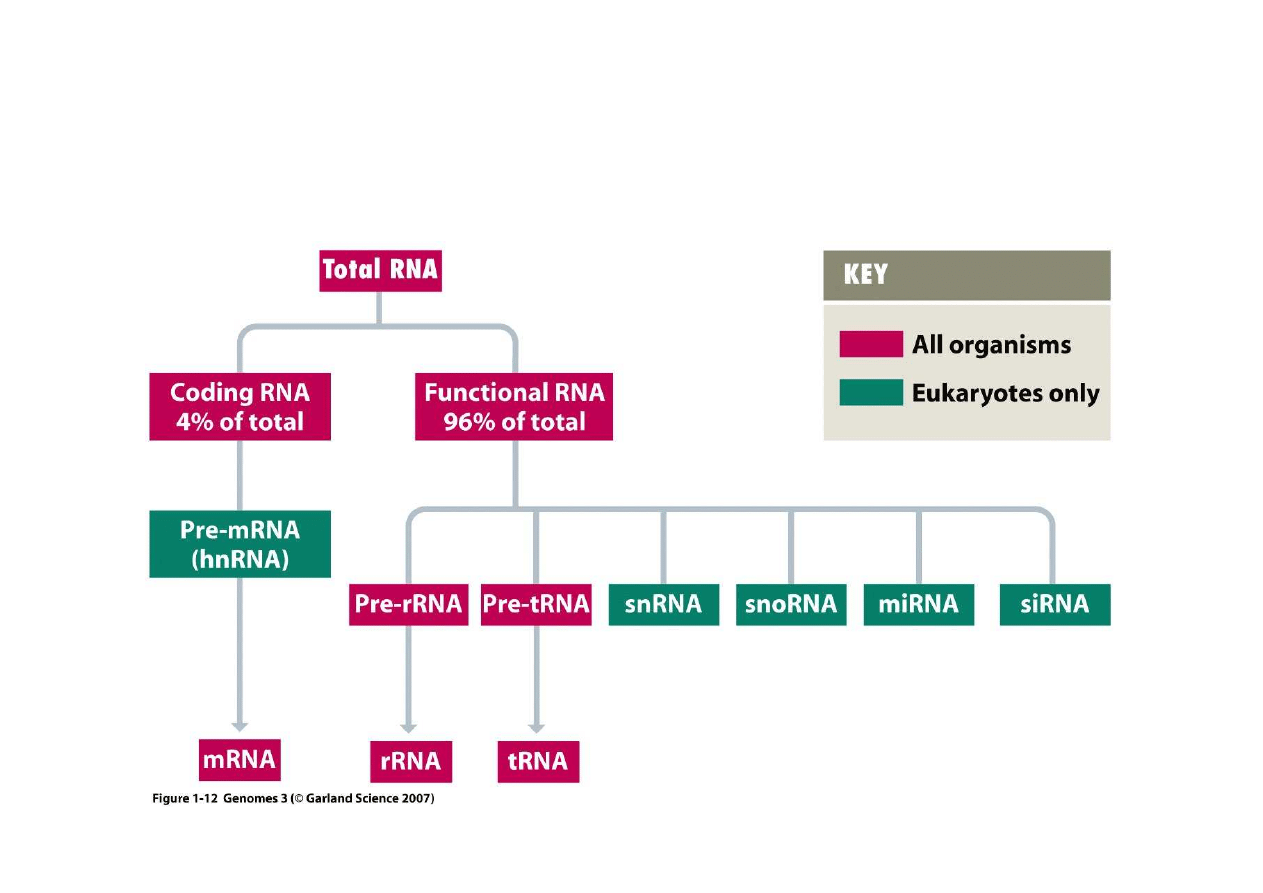
3. RNA eukariontów
mRNA, czyli informacja z genów koduj
ą
cych to bardzo mała cz
ęść
RNA, ogromna
wi
ę
kszo
ść
to RNA funkcjonuj
ą
ca w ró
ż
nych procesach (głównie translacji). RNA
eukariontów jest znacznie bardziej zró
ż
nicowane ni
ż
RNA prokariontów
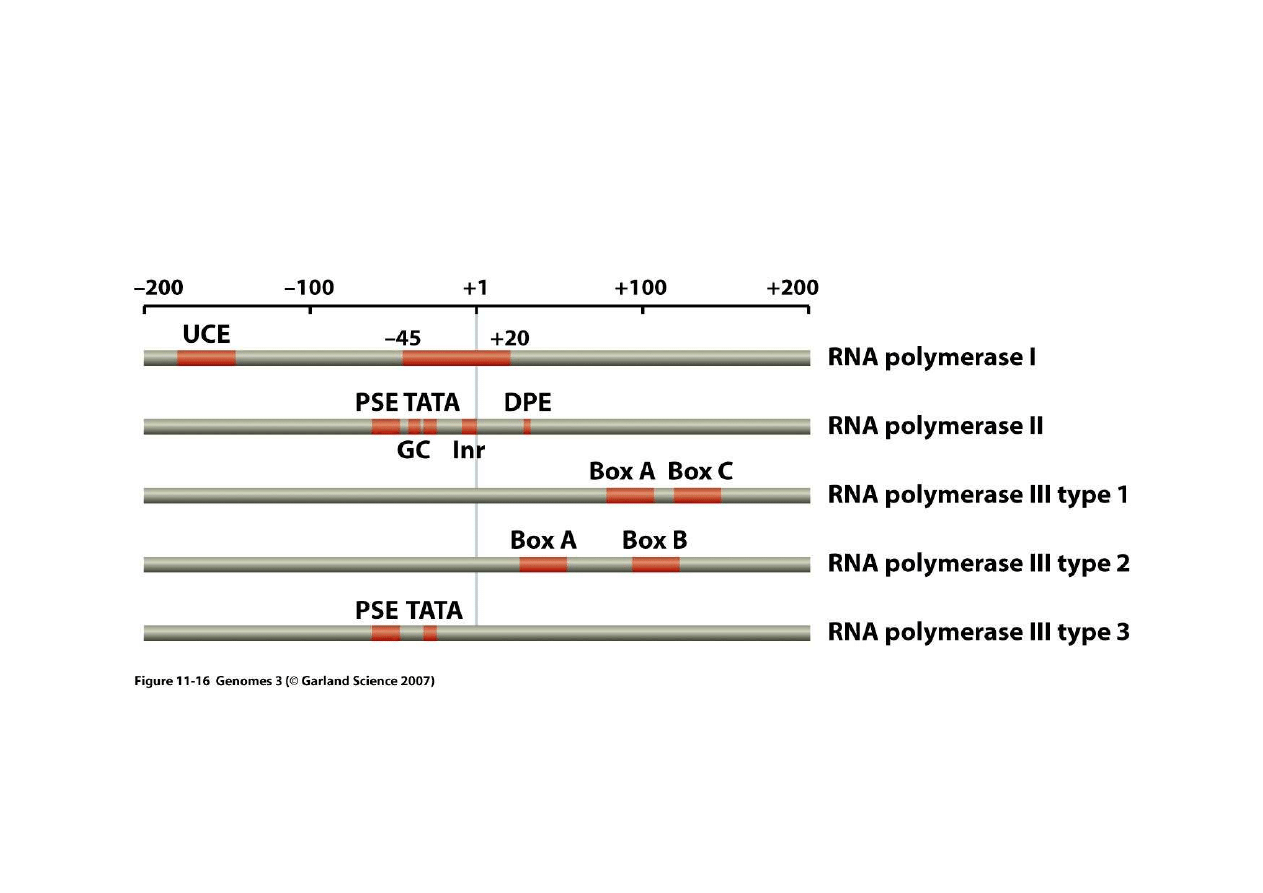
4. Regulacja transkrypcji u eukariontów
Ka
ż
da z polimeraz RNA ma swoje typowe promotory
W dalszej cz
ęś
ci wykładu zajmiemy si
ę
głównie polimeraz
ą
RNA II bo jej aktywno
ść
ma zwi
ą
zek z regulacj
ą
ekspresji genów.
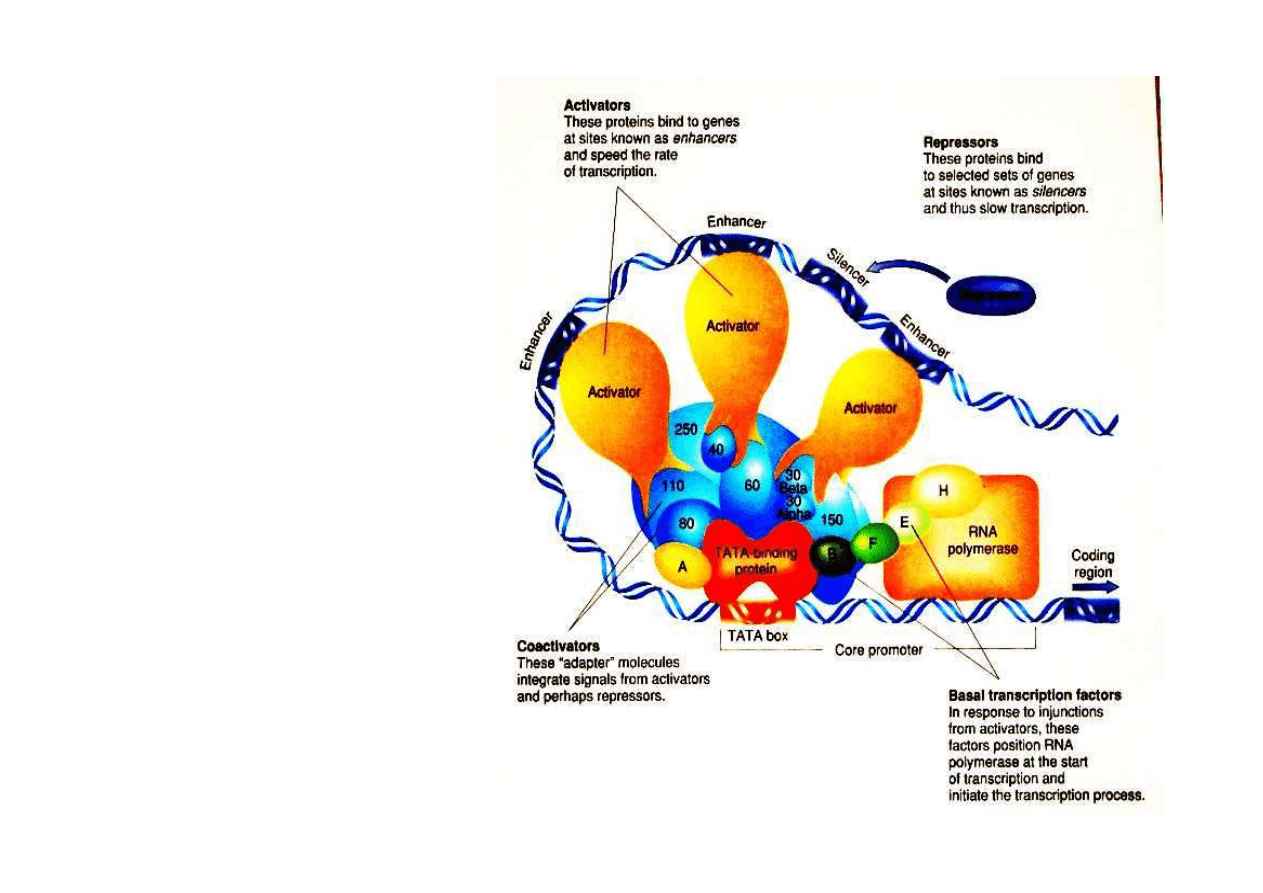
W wi
ę
kszej odleg
ł
o
ś
ci od
promotora (tysi
ą
ce bp) mog
ą
le
ż
e
ć
inne sekwencje DNA podnosz
ą
ce
(enhancers) lub obni
ż
aj
ą
ce
(silencers) prawdopodobie
ń
stwo
utworzenia kompleksu
transkrypcyjnego – s
ą
to odleg
ł
e
(distant) elementy typu cis.
W dowolnych miejscach genomu
(inne chromosomy) le
żą
geny
koduj
ą
ce bia
ł
ka, które w
łą
czaj
ą
si
ę
w kompleks transkrypcyjny – s
ą
to
bia
ł
ka regulatorowe typu trans.
Bia
ł
ka
łą
cz
ą
si
ę
z polimeraz
ą
RNA
i s
ą
oplatane nici
ą
DNA. Jest to
kompleks transkrypcyjny, którego
ł
atwo
ść
tworzenia, czyli
intensywno
ść
transkrypcji, mo
ż
e
by
ć
precyzyjnie regulowana
poprzez zmiany we wszystkich
elementach.
4. Regulacja transkrypcji u eukariontów
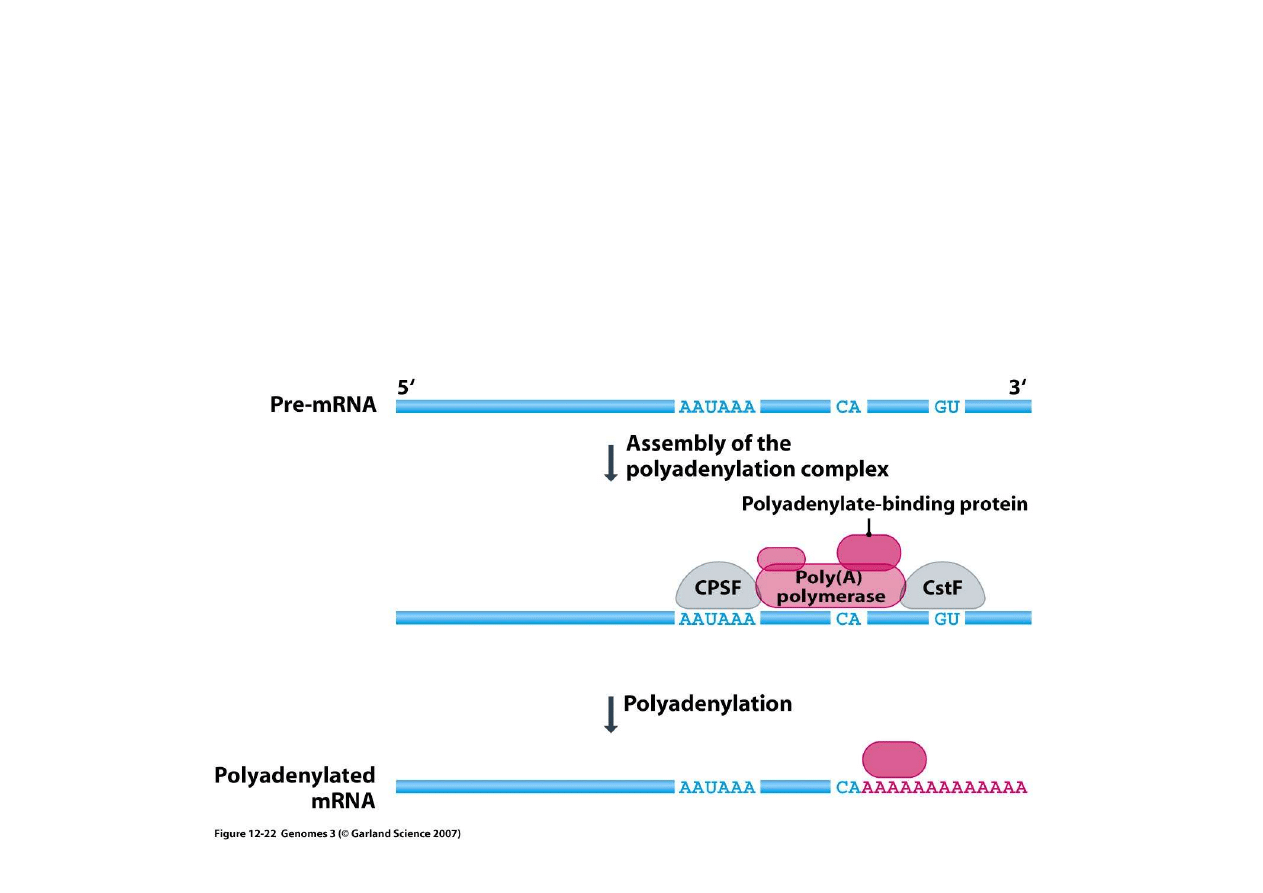
Transkrypt eukariotycznego mRNA jest modyfikowany poprzez dodanie czapeczki (cap)
i ogonka (tail).
Czapeczka to zmetylowany GTP (guanozynotrójfosforan) dodawany przed pierwszy
rybonukleotyd transkryptu (czyli na jego ko
ń
cu 5’). Nast
ę
puje to gdy transkrypt ma
zaledwie 30 nukleotydów.
W terminalnej cz
ęś
ci transkryptu znajduje si
ę
sekwencja 5’-AAUAAA-3’ (czyli w DNA jest
to 3’-TTATTT-5’). Ta sekwencja transkryptu jest sygnałem do ci
ę
cia i
dodania ci
ą
gu około 250 adenin jako ogona w odległo
ś
ci około 20 nukleotydów za
sekwencj
ą
5’-AAUAAA-3’ (czyli na ko
ń
cu 3’ transkryptu)
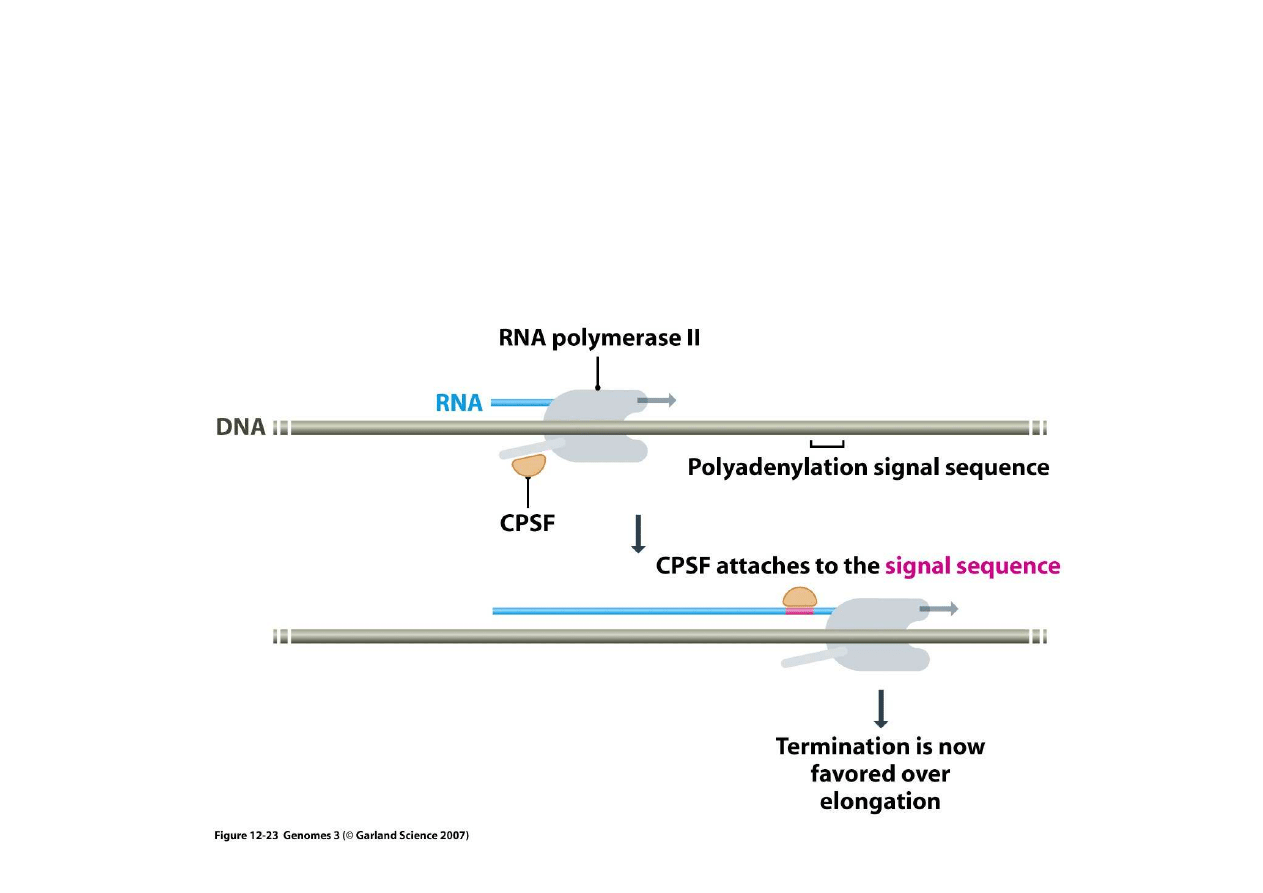
4. Regulacja transkrypcji u eukariontów
Poliadenylacja ma wpływ na terminacj
ę
transkrypcji. Do kompleksu polimerazy II
doczepione jest białko CPSF. Gdy w transkrypcie pojawi si
ę
sekwencja poli(A), CPSF
przenosi si
ę
na t
ę
sekwencj
ę
transkryptu, co jest sygnałem do rozwi
ą
zania kompleksu
polimerazy.
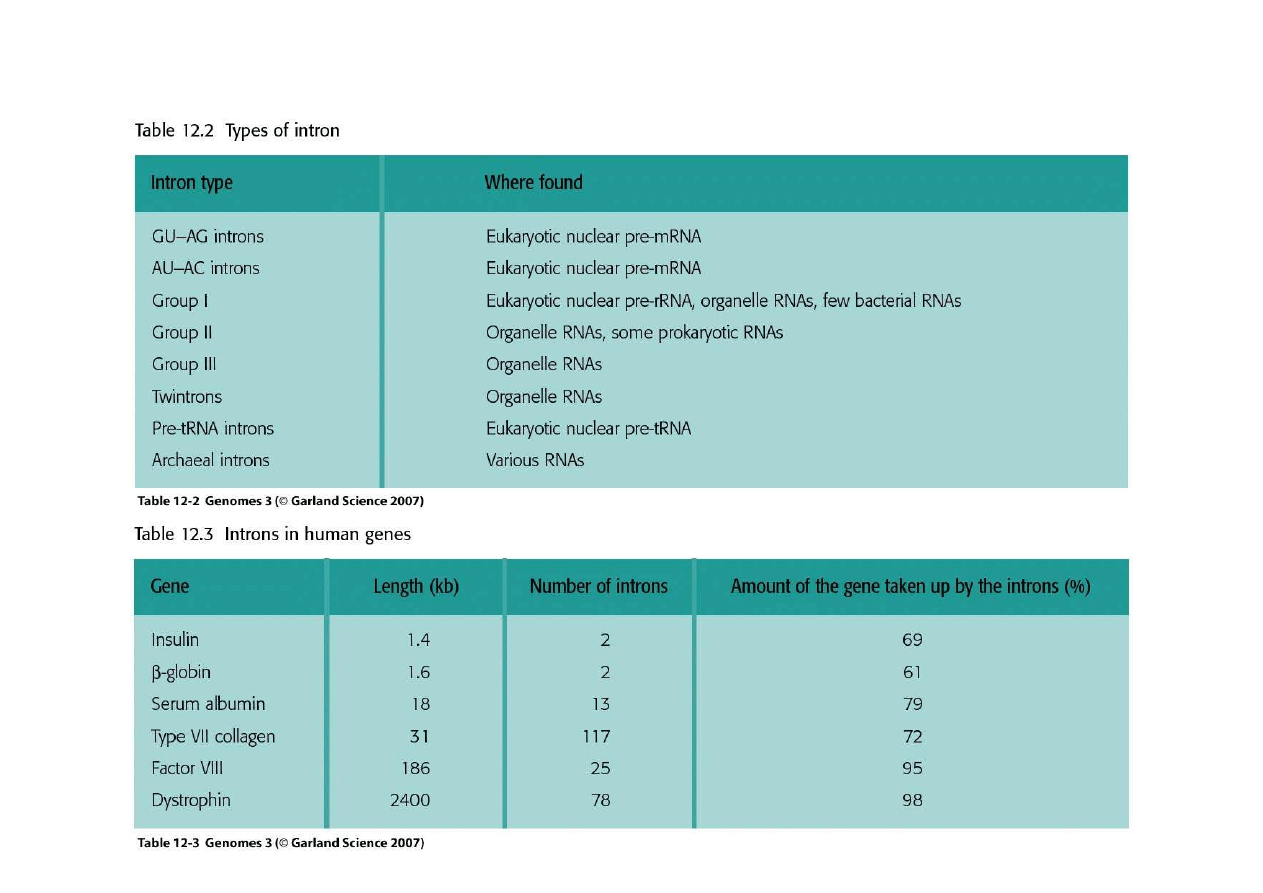
5. Usuwanie intronów
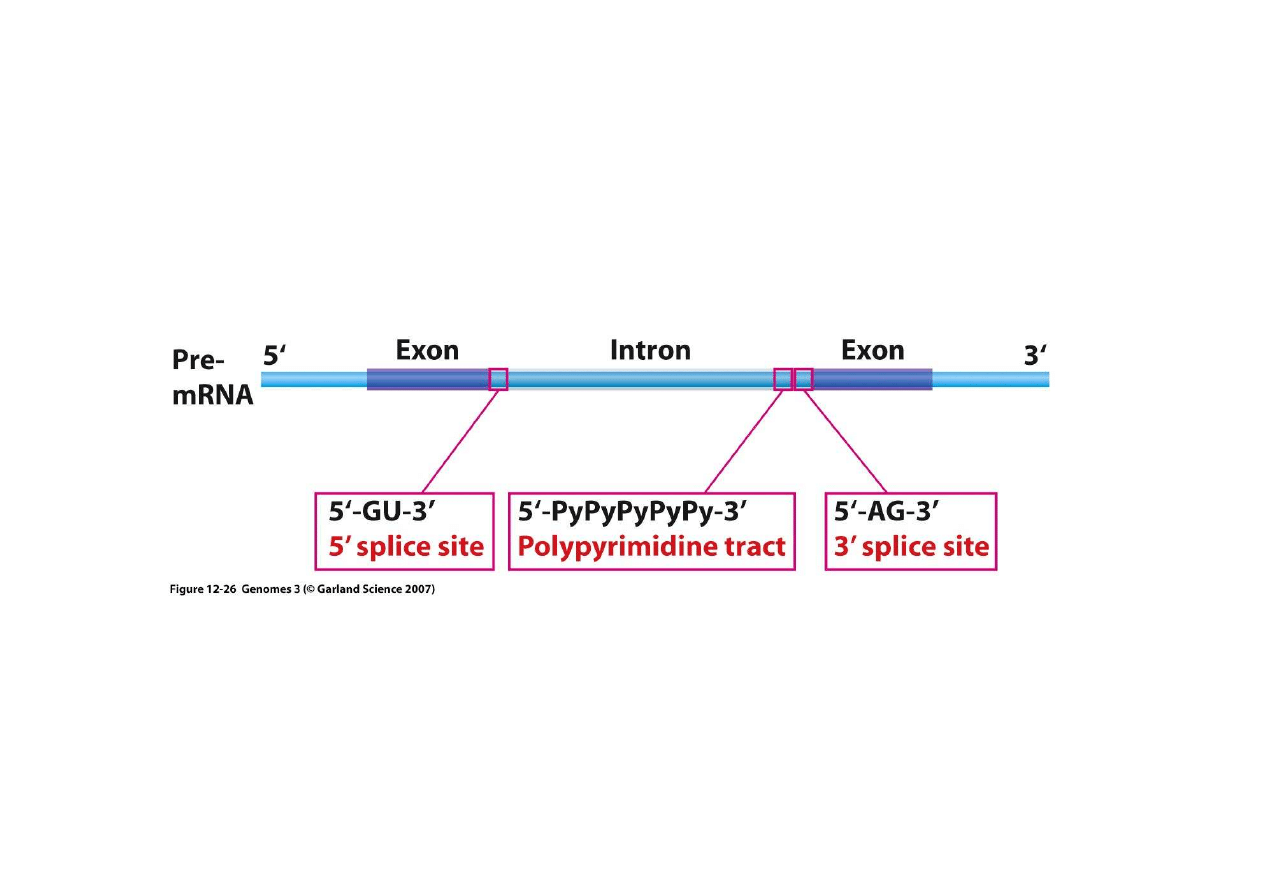
5. Usuwanie intronów
Znaczna wi
ę
kszo
ść
intronów zaczyna si
ę
od 5’-GU-3’ a ko
ń
czy na 5’-AG-3’. S
ą
to miejsca
ci
ę
cia (splice sites). U wy
ż
szych eukariontów cz
ę
sto wyst
ę
puje te
ż
odcinek
polipirymidynowy. Te sekwencje maj
ą
swoj
ą
rol
ę
w procesie usuwania intronów.
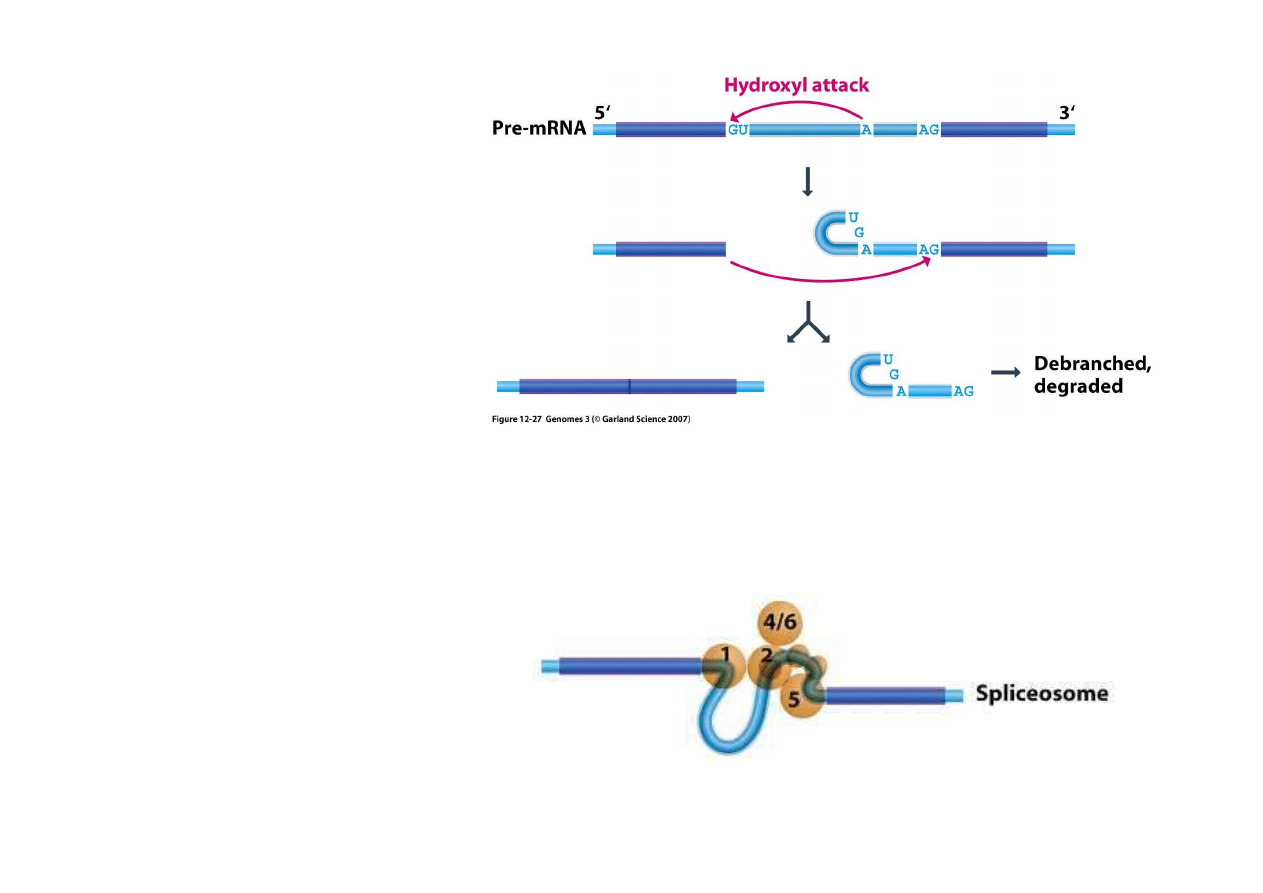
5. Usuwanie intronów
W ogólnym zarysie, wycinanie
intronów rozpoczyna si
ę
od
tego,
ż
e grupa hydroksylowa
jednej z adenin w
ś
rodku
intronu doprowadza do rozbicia
wi
ą
zania fosforanowego przed
pierwszym nukleotydem intronu
(5’-G). W drugim etapie, grupa
hydroksylowa (3’-OH) z
ko
ń
ca poprzedzaj
ą
cego egzonu
doprowadza do rozbicia
wi
ą
zania fosforanowego
za ostatni
ą
zasada intronu (G-
3’). Egzony si
ę
ł
ą
cz
ą
, intron
ulega degradacji.
Operacja ta jest katalizowana
przez zespół snRNA, z których
ka
ż
dy jest poł
ą
czony z białkiem
(tutaj oznaczone 1-6). Tworz
ą
one razem spliceosome.
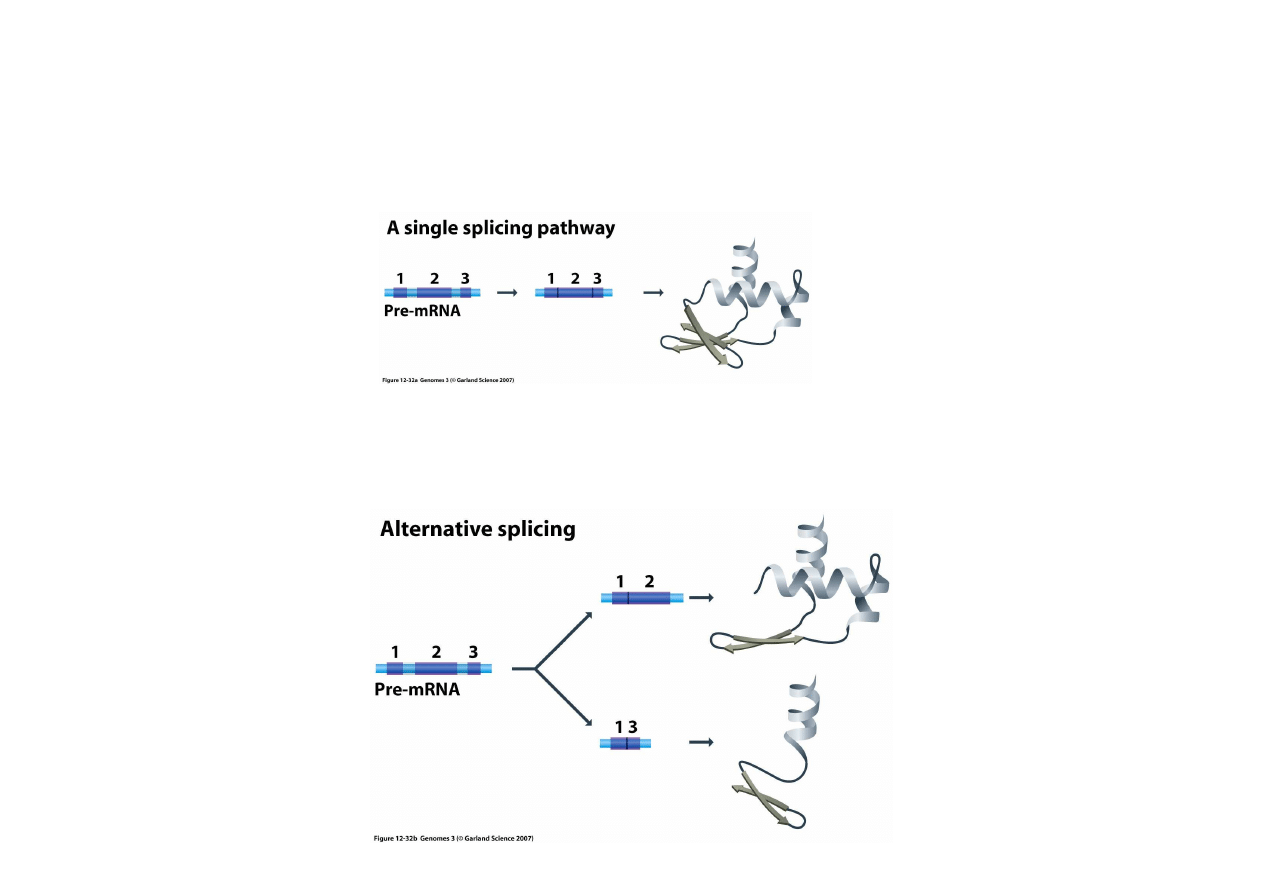
6. Aternatywne składanie egzonów (alternative spiling)
Wyci
ę
cie wszystkich egzonów prowadziłoby do powstania zawsze takiego samego
ko
ń
cowego mRNA.
U eukariontów cz
ę
ste jest jednak wycinanie niektórych egzonów przy okazji wycinania
intronów. Jest to alternatywne składanie egzonów, z jednego genu powstaj
ą
rózne mRNA!
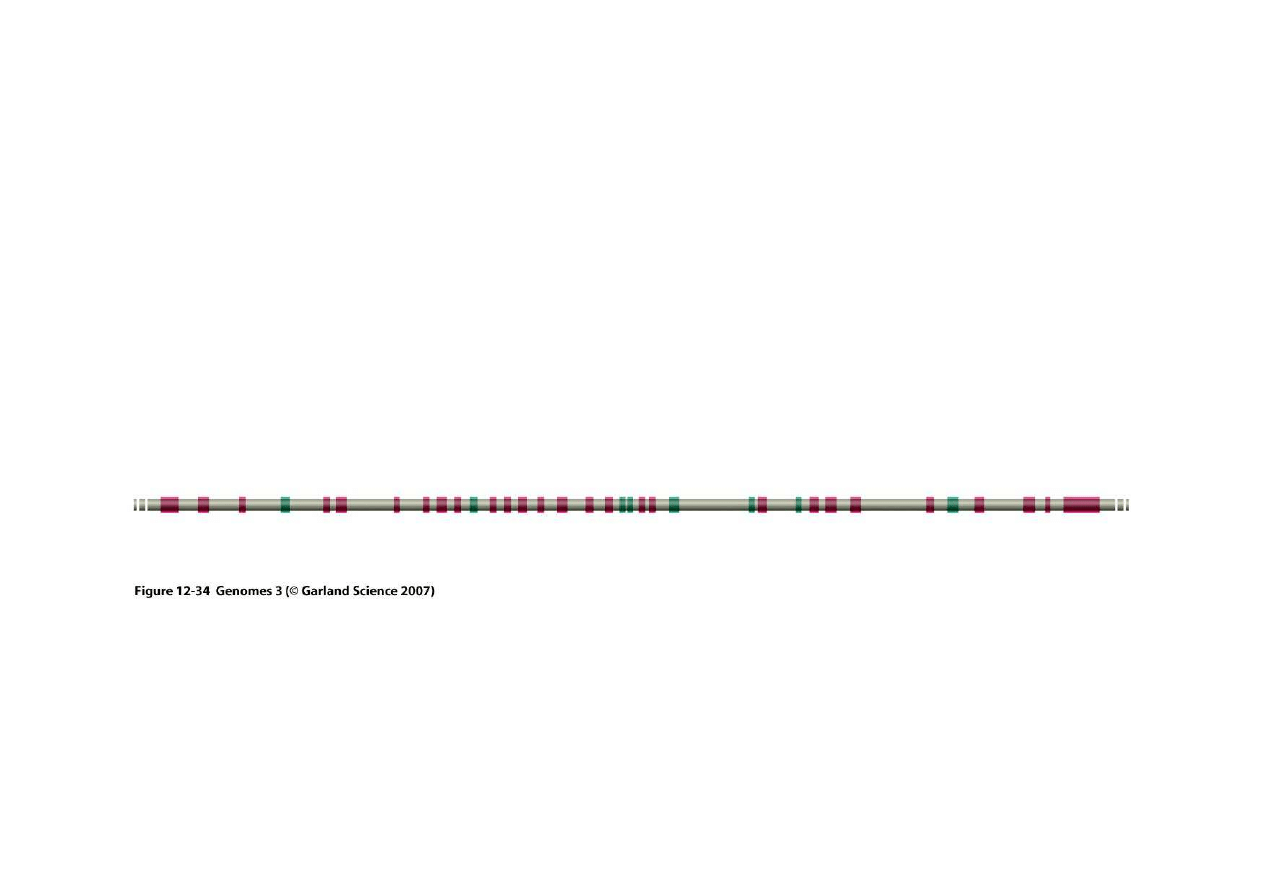
6. Aternatywne składanie egzonów
Regulacja alternatywnego składania jest słabo poznana, ale wiadomo,
ż
e jego efekty mog
ą
by
ć
wa
ż
ne funkcjonalnie. U ludzi wyst
ę
puje gen slo koduj
ą
cy białko reguluj
ą
ce przepływ
jonów potasu prze błon
ę
komórkow
ą
. Ma on 35 egzonów, z których 8 (na zielono) mog
ą
wyst
ę
powa
ć
lub nie w ró
ż
nych kombinacjach. Znanych jest około 500 alternatywnych form
tego białka.
Gen slo jest aktywny w uchu wewn
ę
trznym, w otoczeniu włosów słuchowych. Alternatywne
aktywno
ś
ci form kodowanego przez niego białka daj
ą
wra
ż
liwo
ść
na ró
ż
ne cz
ę
stotliwo
ś
ci fal
d
ź
wi
ę
kowych, od 20 do 20.000 Hz.
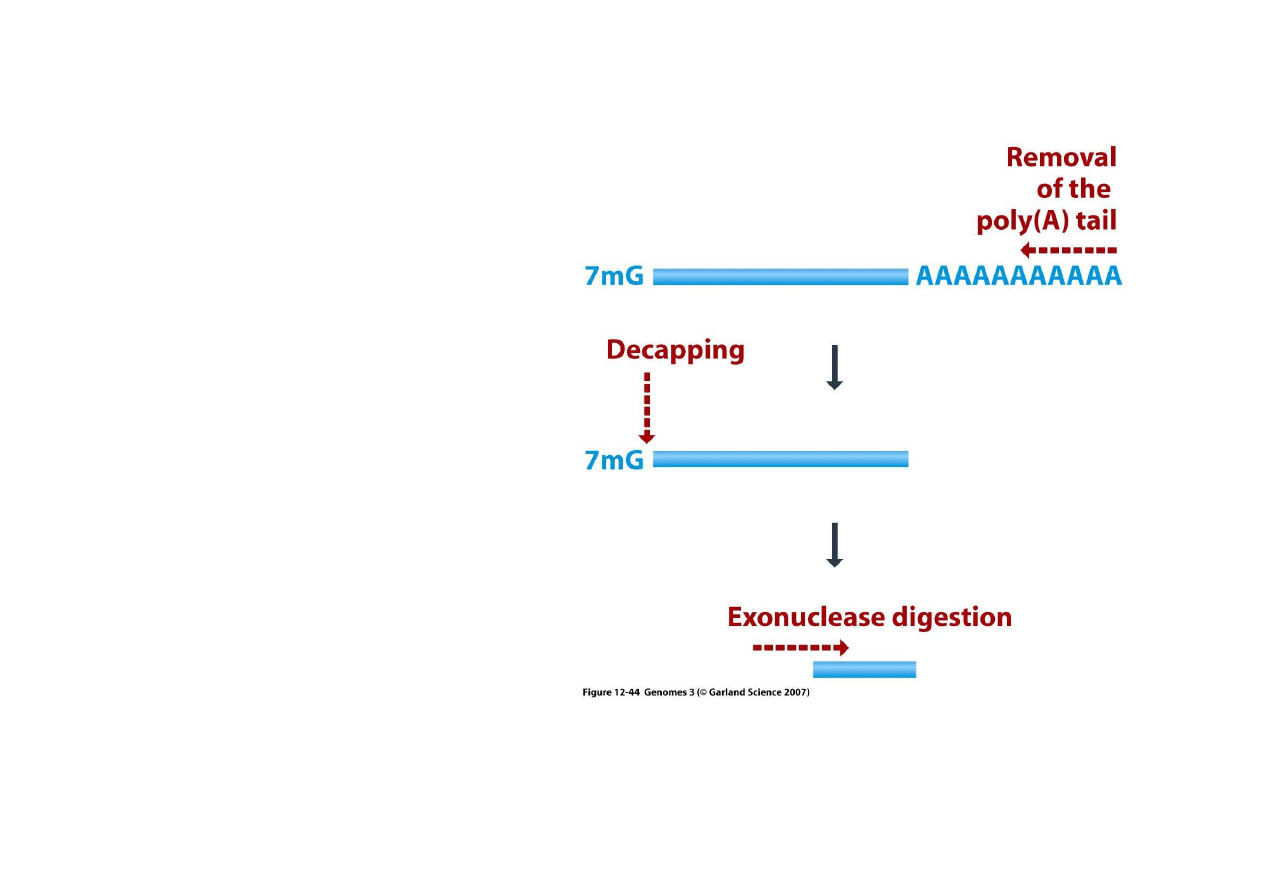
7. Degradacja RNA
Czas
ż
ycia mRNA liczy si
ę
od
minut w bakteriach do godzin u
ssaków. Usuni
ę
cie mRNA
zapobiega powstawaniu białka,
tym samym mo
ż
e by
ć
elementem
regulacji ekspresji genów. Istnieje
kilka sposobów usuwania
wewn
ą
trzkomórkowego DNA.
Jednym z lepiej poznanych
mechanizmów (deanylation
dependent decapping) wymaga by
najpierw usuni
ę
ty został ogon
poli(A) a potem czapeczka
metyloguaniny. Ogon staje si
ę
dost
ę
pny dla egzonukleaz, gdy
odł
ą
cz
ą
si
ę
białka, które normalnie
go stabilizuj
ą
. Koniec 5’ staje si
ę
dost
ę
pny dla egzonukleazy gdy
czapeczka nie zwi
ąż
e si
ę
z
białkami stymuluj
ą
cymi translacj
ę
.
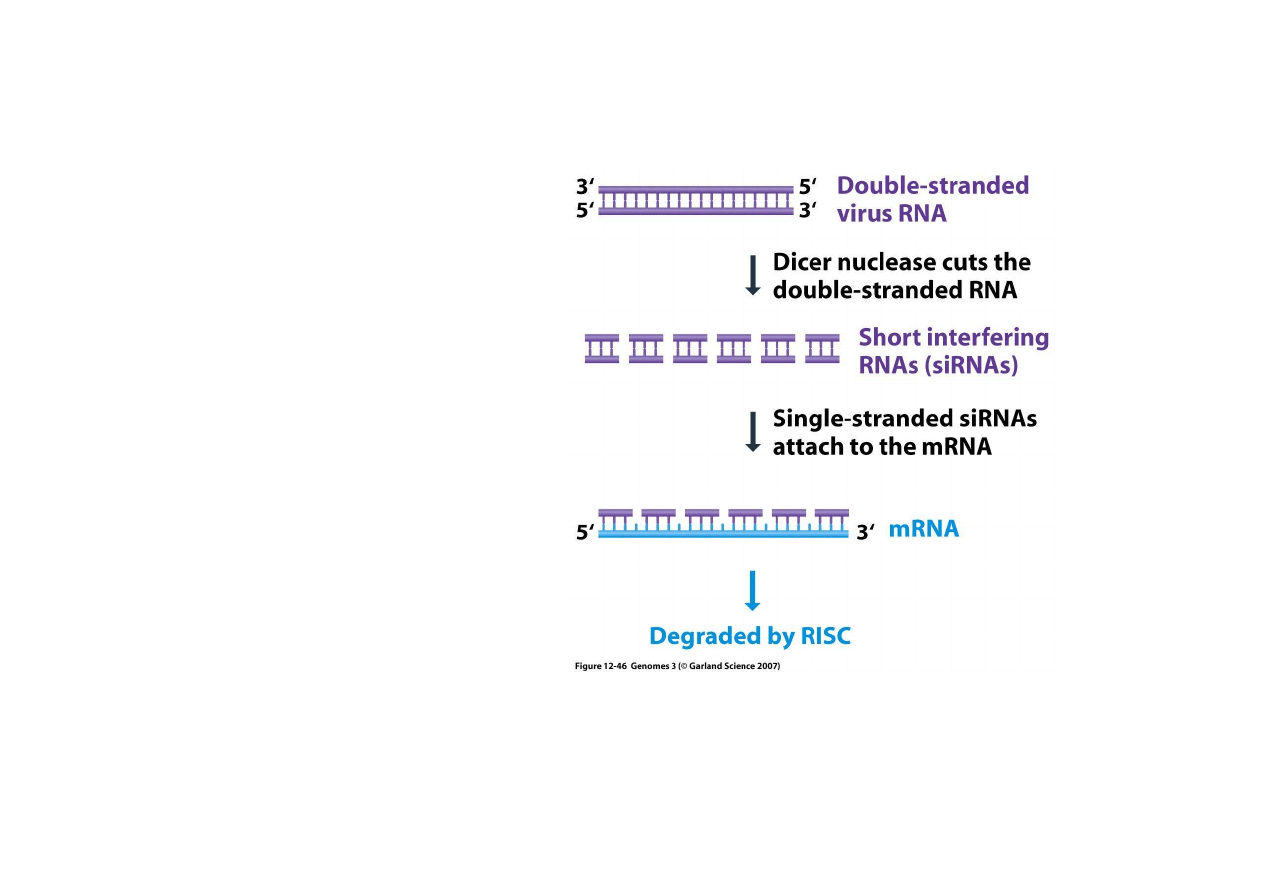
8. Wyciszanie RNA (RNA
silencing)
Eukarionty potrafia rozpozna
ć
i
usun
ąć
obce RNA z komórki,
jest to zabezpieczenie przed
np. retrowirusami. W
przeciwie
ń
stwie do mRNA, RNA
wirusów jest dwuniciowe
(cho
ć
by przej
ś
ciowo).
Rybonukleaza Dicer tnie
dwuniciowe RNA na kawałki po
21-28 nukleotydów nazywane
siRNA (short interfering RNA).
Najciekawsze jest to,
ż
e siRNA
jest rozdzielane na pojedyncze
nici i wtedy mo
ż
e odszuka
ć
komplementarne mRNA wirusa,
który zd
ąż
ył ju
ż
si
ę
namno
ż
y
ć
.
Te odcinki dwuniciowe
przywołuj
ą
kompleks białkowy
RISC, który rozkłada tak
wyznakowany RNA. Inwazja
wirusa jest powstrzymana.
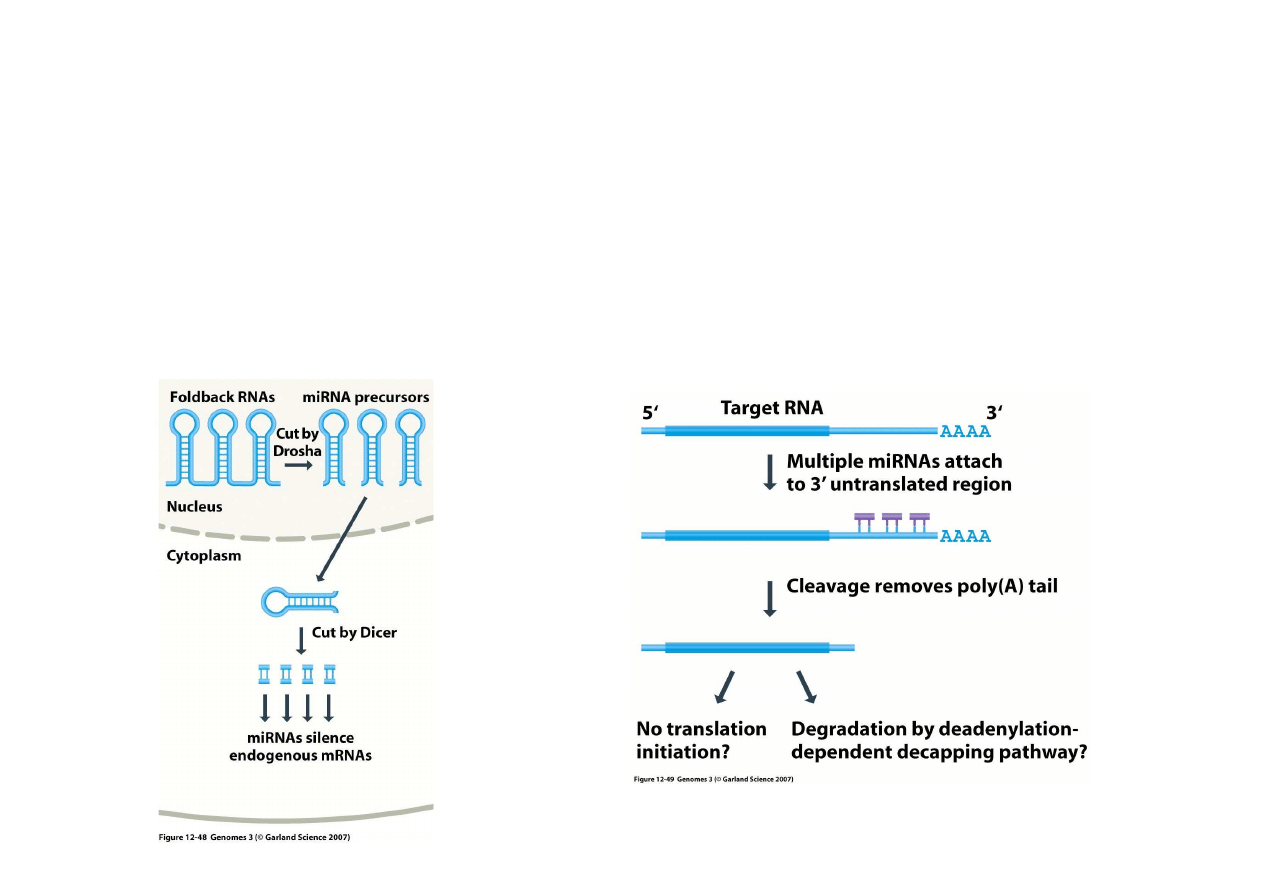
8. Wyciszanie RNA (RNA silencing)
Bardzo podobne systemy s
ą
u
ż
ywane do selektywnego usuwania własnego mRNA. W
ró
ż
nych miejscach w genomie kodowane s
ą
krótkie odcinki RNA, które daj
ą
podwójna ni
ć
po transkrypcji (bo maj
ą
odwrócone powtórzenia). Sa one ci
ę
te przez Dicer a powstałe
jednoniciowe RNA okazuje si
ę
komplementarne do mRNA komórki, np. w odcinku
terminalnym mRNA nie podlegaj
ą
cym translacji. mRNA z doł
ą
czonymi miRNA jest
usuwane z komórki. Coraz wi
ę
cej miRNA jest znajdowane w genomach eukariontów.
Nasuwa si
ę
podejrzenie,
ż
e taka regulacja ekspresji jest cz
ę
sta i wa
ż
na. Biotechnolodzy
wykorzystuj
ą
to do wyciszenia aktywno
ś
ci genów bez przeprowadzania ich delecji!
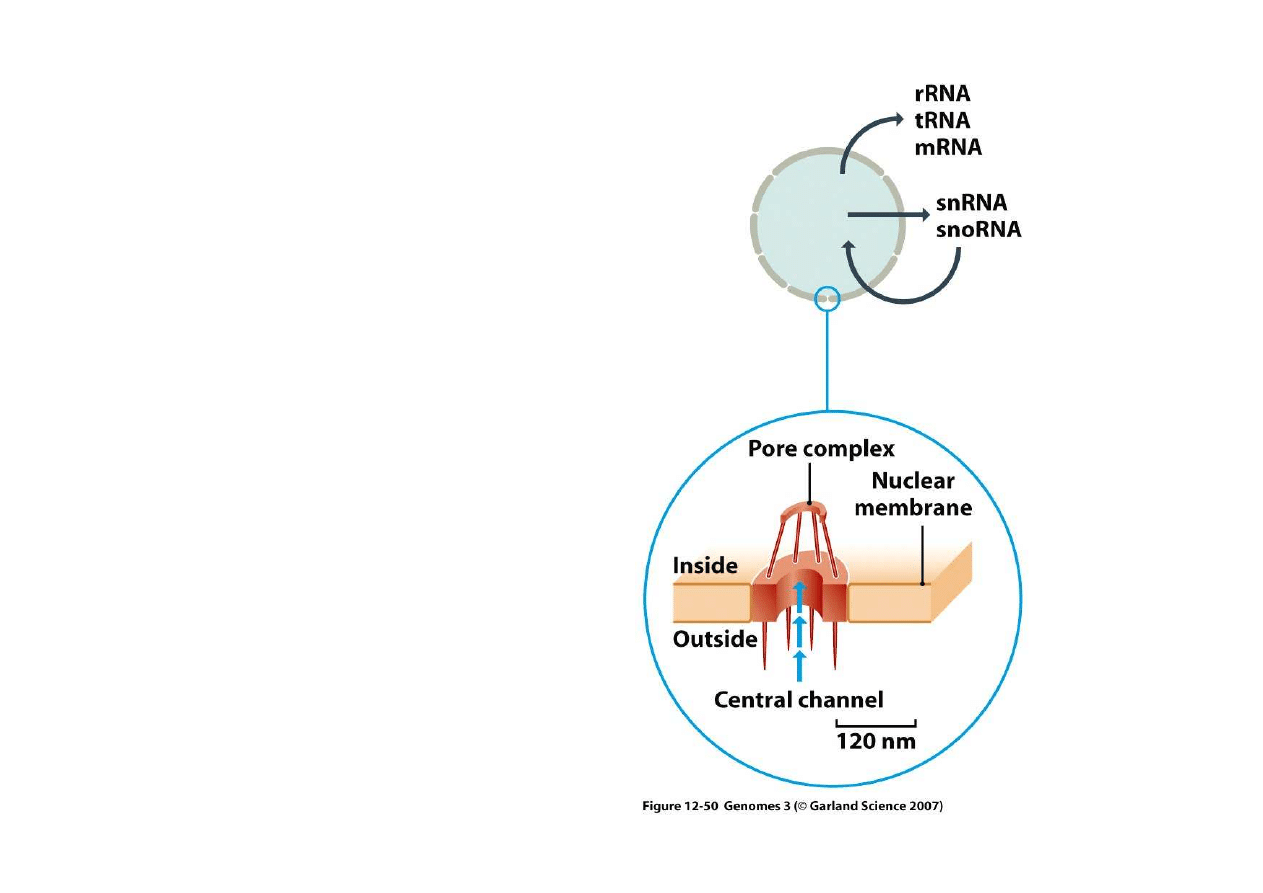
9. Transportowanie RNA
RNA wydostaje si
ę
na zewn
ą
trz
j
ą
dra na drodze aktywnego
transportu poprzez kanały w
błonie j
ą
drowej. W transporcie
uczestnicz
ą
białka karioferyny
(ro
ż
ne dla ró
ż
nych rodzajów
RNA) i zu
ż
ywana jest energia
(hydroliza GTP).
Niektóre rodzaje RNA, takie jak
snRNA i snoRNA wydostaj
ą
si
ę
na zewn
ą
trz j
ą
dra ł
ą
cz
ą
w
kompleksy z odpowiednimi
białkami i takie połaczone
cz
ą
steczki wracaj
ą
do j
ą
dra
gdzie uczestnicz
ą
w
przekształcaniu/dojrzewaniu
j
ą
drowego RNA.
Wyszukiwarka
Podobne podstrony:
transkrypcja biosynteza rna, INNE KIERUNKI, biologia
Transkrypcja-biosynteza RNA
biologia, transkrypcja biosynteza rna, TRANSKRYPCJA-BIOSYNTEZA RNA:
Cechy kodu genetycznego Budowa i rola RNA
zestaw74, Rola t-RNA w biosyntezie aminokwasow
transkrypcja biosynteza rna, INNE KIERUNKI, biologia
biologia, Replikacja DNA, Zjawisko transkrypcji to zjawisko powstawania rodzajów RNA ( t, m, r, )
POPL T 2 2 Miejsce i rola Powszechnej OPL w systemie OPL wojsk (07 03 2006)
Rodzaje i rola kwasów RNA, Notatki(1)
07. Flawonoidy ich rola w kosmetyce i terapii, Studium kosmetyczne, Chemia kosmetyczna
Rodzaje i rola kwasów RNA, Biologia
07-10 PAM-Wasza rola w procesie wstępowania, ezoteryka
rola neofitow w dziejach polski stanislaw didier transkrypt 1934r(1)
POPL T 2 2 Miejsce i rola Powszechnej OPL w systemie OPL wojsk (07 03 2006)
ROLA NEOFITÓW W DZIEJACH POLSKI STANISŁAW DIDIER TRANSKRYPT Z 1934r
42 Frakcje RNA w każdej komórce
07 Rola i cele masonerii w świecie i Kościele
więcej podobnych podstron