
BLAST
Wprowadzenie do bioinformatyki

BLAST
ang. Basic Local Alignment Search Tool
Jest to narzędzie do porównywania białek i
sekwencji nukleotydów
Wprowadzona sekwencja jest porównywana z
bazą danych
W wyniku otrzymuje się listę sekwencji
nukleotydów, białek ze statystycznie obliczonym
stopniem podobieństwa do wprowadzonej
struktury
Istnieje możliwość porównywania dwóch lub
więcej sekwencji nukleotydów
21-11-13
2
Wprowadzenie do bioinformatyki - ćw. 2

BLAST
Został zaprojektowany w 1990 roku
przez:
Eugene Myers
Stephen Altschul
Warren Gish
David J. Lipman
Webb Miller
Dostępny jest pod adresem:
http://blast.ncbi.nlm.nih.gov
21-11-13
3
Wprowadzenie do bioinformatyki - ćw. 2

BLAST
Dopuszczalne formaty danych:
Genebank
FASTA
21-11-13
4
Wprowadzenie do bioinformatyki - ćw. 2

Format FASTA
Jest to format zapisu sekwencji kw.
nukleinowych i białek
Pierwsza linijka zawiera opis sekwencji
Kolejne linijki zawierają sekwencję
21-11-13
5
Wprowadzenie do bioinformatyki - ćw. 2
>Keratyna 5, egzon 2, Homo sapiens
GTGCGGTTCCTGGAGCAGCAGAACAAGGTTCTGGACACCAAGTGGACCCTGCTGCA
GGAG
CAGGGCACCAAGACTGTGAGGCAGAACCTGGAGCCGTTGTTCGAGCAGTACATCAA
CAAC
CTCAGGAGGCAGCTGGACAGCATCGTGGGGGAACGGGGCCGCCTGGACTCAGAGC
TGAGA
AACATGCAGGACCTGGTGGAAGACTTCAAGAACAA

BLAST
Rodzaje BLAST
nucleotide BLAST (blastn) – do
porównywania sekwencji DNA
protein BLAST (blastp) – do porównywania
białek
blastx – konwertuje sekwencję nukleotydów
na sekwencję białek, sekwencja białka jest
porównywana z dostępnymi białkami w
bazie
21-11-13
6
Wprowadzenie do bioinformatyki - ćw. 2

BLAST
Rodzaje BLAST
tblastn – konwertuje sekwencje białek na
sekwencję nukleotydów, sekwencja
nukleotydów jest porównywana z bazą
tblastx – konwertuje sekwencję
nukleotydów na sekwencję białek we
wszystkich ramkach odczytu i porównuje z
bazą nukleotydów przetłumaczoną na
sekwencje białek
21-11-13
7
Wprowadzenie do bioinformatyki - ćw. 2
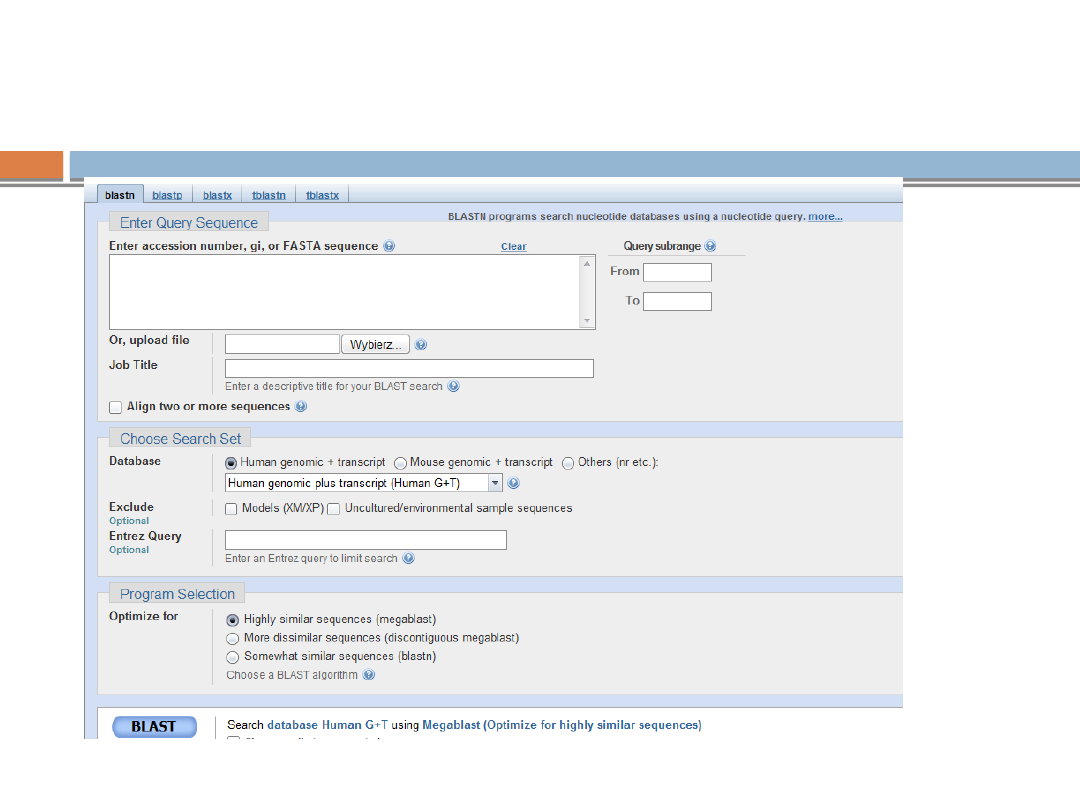
blastn
21-11-13
8
Wprowadzenie do bioinformatyki - ćw. 2
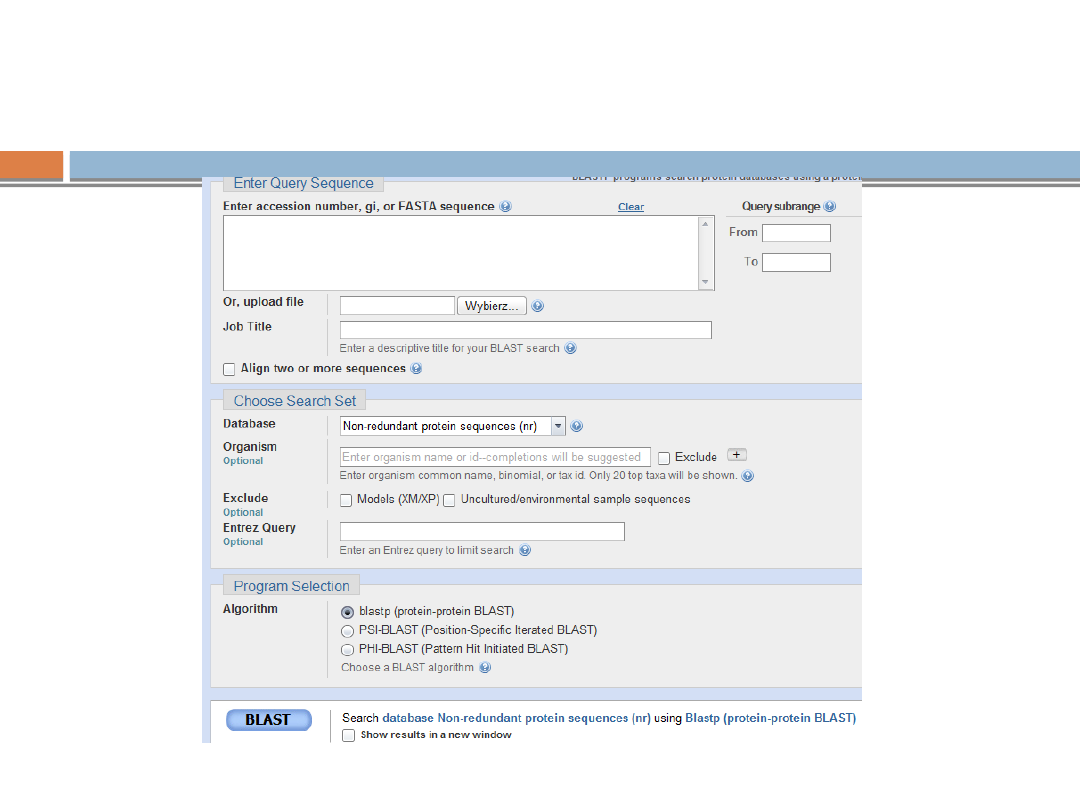
blastp
21-11-13
9
Wprowadzenie do bioinformatyki - ćw. 2
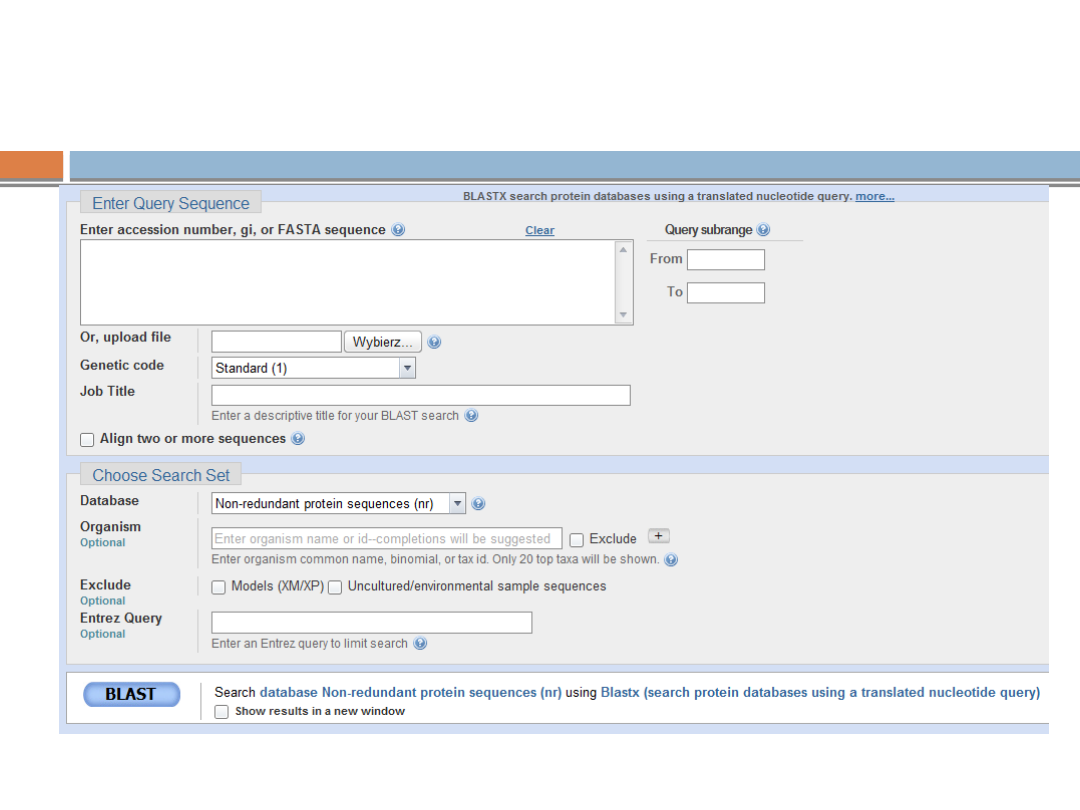
blastx
21-11-13
10
Wprowadzenie do bioinformatyki - ćw. 2
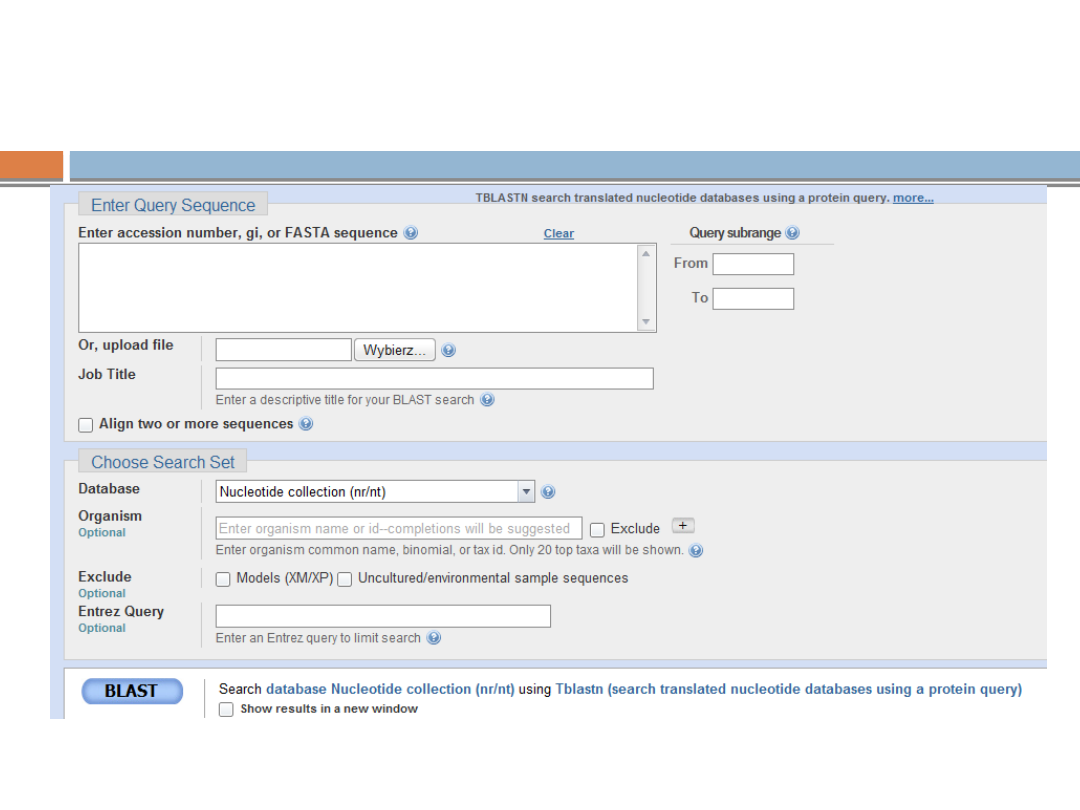
tblastn
21-11-13
11
Wprowadzenie do bioinformatyki - ćw. 2
tblastx
21-11-13
12
Wprowadzenie do bioinformatyki - ćw. 2

Ćwiczenia
1.
W programie BLAST odszukać białko
CAA43114 – podać nazwę gatunku (polska i
łacińska zwierzęcia), do jakich dwóch białek
u innych zwierząt jest homologiczny w
największym stopniu, odnaleźć
podobieństwo do enzymu występującego u
człowieka – Mieloperoksydaza
(Myeloperoxidase).
Odnaleźć informację w bazie MeSH na temat
Myeloperoxidase
21-11-13
13
Wprowadzenie do bioinformatyki - ćw. 2

Ćwiczenia
2.
Odczytać rodzaj genu i nazwę organizmu
dla sekwencji nukleotydów znajdującej
się w pliku sekw_1.txt
3.
Odnaleźć sekwencję przeciwciała
aglutyniny (Agglutinin) u ślimaka
winniczka (baza białkowa), dokonać
analizy w BLAST i wygenerować drzewo.
Odnaleźć wizualizację przeciwciała
aglutynina bez ligandów (Agglutinin With
No Ligands)
21-11-13
14
Wprowadzenie do bioinformatyki - ćw. 2

Ćwiczenia
4.
Dokonać porównania dwóch sekwencji
nukleotydów (dostępnych w plikach
sekw_2.txt i sekw_3.txt) – podać stopień
podobieństwa.
Podać nazwę białka oraz nazwy
organizmów od których pochodzi.
21-11-13
15
Wprowadzenie do bioinformatyki - ćw. 2
Document Outline
- Slide 1
- BLAST
- BLAST
- BLAST
- Format FASTA
- BLAST
- BLAST
- blastn
- blastp
- blastx
- tblastn
- tblastx
- Ćwiczenia
- Ćwiczenia
- Ćwiczenia
Wyszukiwarka
Podobne podstrony:
ODL I sem termin3 10 02 26
2006.10.02 psychometria ćw, Psychologia, Psychometria
10 26 geogr turyst ćw
PD W1 Wprowadzenie do PD(2010 10 02) 1 1
10 02 09
2003 02 26
22 12 10 02 12 55 Egz podst Ana2 B2
2011 02 26 klasa O
29 12 10 02 12 55 am2 2004 k1 grupaPS
10 02 SW Nocken Doku
OOZ, Medycyna, Pobr materiały, VI rok-2015-10-02, VI rok, Zdrowie Publiczne
2 TERMIN -10.02.2011 -A, Barbasze IMiR mibm
Siatkówka doskonalenie odbić, zagrywki 02 10 02
10 02 18 chegz popr
25 11 2009 12 10 02 0173 001
22 12 10 02 12 16 Egz popr
plan PZ stacjonarne sem6 Elektro 10 02 06
02 26 o zbiorowym zaopatrzeniu w wodę i
10 02 podstawy endokrynologi
więcej podobnych podstron