Phage display - Metoda prezentacji fagowej.
Metoda ta została uznana jako innowacyjny sposób wytwarzania białek o pożądanych właściwościach.
Jest ona techniką prezentacji peptydów lub białek po fuzji do białek płaszcza faga.
Idea
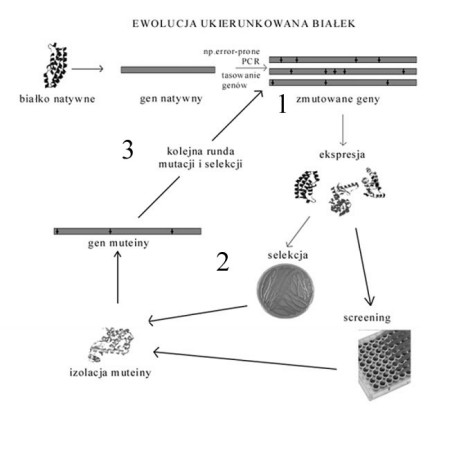
Ewolucja ukierunkowana białek jest sposobem tworzenia cząsteczek o nowych właściwościach fizykochemicznych. Nie jest to metoda, lecz sposób podejścia do problemu tworzenia nowych cząstek (zazwyczaj białek), w którym wykorzystywane są techniki biochemii, inżynierii genetycznej, oraz innych dziedzin nauk przyrodniczych. Celem jest uzyskanie biocząsteczki o określonych parametrach. Może to być białko, wirus lub każdy inny typ cząsteczek, dla których jesteśmy w stanie uzyskać strukturalną zmienność i wyszukać z nich te o pożądanych właściwościach.
Idea ewolucji ukierunkowanej białek to dwa procesy: wytworzenie zmienności oraz selekcja z pośród tej zmienności. Te dwa etapy są powtarzane w cyklach, aż do uzyskania zadowalającego rezultatu. W ogólnym przebiegu, przypomina naturalny proces ewolucji, gdzie odmienne osobniki są selekcjonowane przy udziale doboru naturalnego. Tak samo w przypadku ewolucji ukierunkowanej najlepszy wariant jest wybierany przy zadanych przez eksperymentatora warunkach, z pośród różniących się składem aminokwasowym białek. Jest to proces ukierunkowany, ponieważ to człowiek wybiera kierunek zmian.
Można to podejście zastosować także do innych, niż białkowe układów.
W etapie wytworzenia zmienności białko podlega jednocześnie namnożeniu i mutacjom. W efekcie powstaje wiele genów mających losowe mutacje punktowe w swojej sekwencji. Często, aby dodatkowo zwiększyć zmienność genów prowadzi się tzw. tasowanie egzonów (egson shuffling), w którym wiele fragmentów tego samego genu, zawierającego mutacje punktowe, są losowo łączone tworząc kompletne geny. Geny kodujące białko i zawierające losowe mutacje podlegają ekspresji, czego efektem jest powstanie biblioteki białek.
Tworzenie biblioteki białek.
- tworzenie zmienności. Poszczególne białka w bibliotece różnią się między sobą składem aminokwasowym, więc i właściwościami chemiczno-fizycznymi.
Selekcja/screening.
Z utworzonej biblioteki wybierane są białka o pożądanej cesze. Zachodzi to na drodze selekcji w mikroorganizmach (głównie bakteriach) lub tzw. 'screeningu'. Screening polega na wykorzystaniu pewnej cechy białka (np. zdolności do katalizy substratu do fluoryzującego produktu), aby na podstawie jej ilościowej oceny (np. intensywności fluorescencji, aktywności katalitycznej), móc wybrać najlepszą formę białka.
Selekcja jest podobna do dziania doboru naturalnego, przeżywają nieliczni, dostosowani do warunków środowiska. Istnieje wiele metod selekcji i screeningu, a ich różnorodność wynika między innymi z konieczności dostosowania tych procedur do wymagań konkretnego eksperymentu. Inaczej mówiąc, dla różnych białek screening/selekcja także będzie różna. Jedną z metod screeningu (przeszukiwania zmienności) jest phage display.
Kolejny etap mutagenezy.
Po przeprowadzeniu screeningu/selekcji, konieczne jest uzyskanie materiału genetycznego zawierającego informację o składzie aminokwasowym wybranego białka. Następnie, poddaje się go mutagenezie, by na jego bazie utworzyć kolejną bibliotekę białek. Ilość etapów mutagenezy i selekcji zależy od eksperymentatora.
Screening polega na wykorzystaniu fizycznych bądź chemicznych właściwości cząsteczki w celu przetestowania każdej i selekcji najlepszych wariantów. Metoda screeningu może wykorzystywać właściwości: cząsteczki; substratu lub produktu reakcji enzymatycznej do oceny aktywności katalitycznej; dołączonej cząsteczki. Można ją zastosować do oczyszczonych enzymów, lizatów komórkowych lub całych komórek. Najpoważniejszym ograniczeniem metody jest mała, w stosunku do metod selekcyjnych, ilość wariantów ocenianych. Nawet przy zastosowaniu zautomatyzowanej aparatury osiągane wielkości sprawdzonych bibliotek mieszczą się w granicach 104-106. W efekcie przeszukana przestrzeń sekwencyjna jest mniejsza niż w metodach selekcyjnych. Zaletą jest łatwiejsze stworzenie systemu, oraz możliwość przeprowadzenia oceny na mikropłytce ze studzienkami, w systemie in vitro, także przy udziale nienaturalnych substratów i/lub warunków, których nie wytrzymała by komórka (np. wysoka temperatura). Wśród metod screeningu najistotniejsze, znajdujące ogólne zastosowanie i rozwijane, to: 'phage display' i 'ribosome display'. Szczególne cechy obu systemów to możliwość selekcji białek na podstawie ich wiązania się do ligandów, oraz fizyczne połączenie pomiędzy genem, a zakodowanym przez niego białkiem. Dzięki temu, bez dodatkowej pracy, izolując białko, eksperymentator otrzymuje także gen je kodujący. Metoda 'phage display' wykorzystuje filamentarne fagi M13 zawierające materiał genetyczny i białko na powierzchni, a 'ribosome display' rybosomy z doczepionym białkiem i mRNA.
Phage display jest doskonałą techniką do wykrywania interakcji białko-białko. Jest to jedna z wielu technik do generowania cząsteczek wykorzystywanych w projektowaniu nowych leków. Bazuje ona na tym, że bardzo wiele oddziaływań białko-białko zależy od kontaktu krótkich sekwencji aminokwasów.
Dwa typy bibliotek phage display:
- nielityczna (non-lytic phage) ukazująca jednostki z M13 bakteryjnego nitkowatego faga: losowe peptydy, zaprojektowane własne sekwencje peptydów lub białek, przeciwciała (pojedyncza łańcuchowa fuzja Fv do pIII)
- lityczna (lytic phage) - oparta na bakteriofagach λ, T7 oraz T4, preferowana do uzyskiwania obrazu biblioteki cDNA (cDNA library display).
Ogólne korzyści wynikające ze stosowania phage display:
- bezpośrednie powiązanie między pokazywanym peptydem a kodowaną sekwencją. W tradycyjnych bibliotekach, jak λ-ekspresyjnych, takie powiązania nie są ukazywane.
- ogromna zaletą takich powiązań jest możliwość przeprowadzenia procesu selekcji powinowactwa bez filter screening lub microarray.
- uzyskujemy bardzo wiele różnych peptydów. Biblioteki zawierające 106 do 1010 niezależnych klonów mogą być uzyskiwane z łatwością. Mutanty replikacyjne nie są ukazywane w tej technice. Nielityczna, tetracyklinooporność możliwa jest do przekazywania i rozprzestrzeniania się jak plazmid.
Cele dla phage display:
-cząstki immobilizowane na nośnikach stałych;
-powierzchniowe białka komórkowe w warunkach in vitro;
-organo-specyficzne markery śródbłonowe komórek in vivo.
Etap decydujący o jakości przeprowadzenia metody to precyzja sporządzenia bibliotek.
Biblioteki typu phage display są używane głównie jako próba określenia od zera lokalizacji interesującego nas genu. Przypadkowe fragmenty DNA zostają umieszczone w genomie faga. Wprowadzone produkty zostają następnie uwidocznione przez fagowe genowe III białko kontrolne. Te fagi zostają następnie umieszczone w pojemniku zawierającym stałe kompleksy, specyficzne interesujące nas białka, które będziemy wyszukiwać. Po kilku oczyszczeniach, tylko fagi zawierające fragment białka z wysokim powinowactwem do ligandów pozostaną przyłączone do pojemnika, pozostałość zostanie wypłukana w trakcie czyszczeń. Tak pozostałe fagi mogą być potem wymyte i przeniesione do E. coli. Wszystkie te czynności powtarzamy tak długo, aż uzyskamy satysfakcjonujące nas, skrupulatne rezultaty. Ten specyficzny proces skriningu nazywa się panning.
Istotą metody phage display jest wprowadzenie do genomu faga filamentowego określonej sekwencji DNA, co prowadzi do ekspresji kodowanego przez nią peptydu na powierzchni faga. Punktem wyjścia do rozwoju technologii phage display była praca Smitha, który sklonował fragment sekwencji bakteryjnej endonukleazy EcoRI w środku genu kodujacego białko kapsydowe pIII faga filamentowego f1 i wykazał, że:
1) białko fuzyjne pIII*EcoRI jest wbudowywane w obręb wirionu,
2) fag częściowo zachowuje infekcyjność,
3) Eco RI prezentowane przez faga zachowuje pierwotna immunogenność,
4) fag taki może być wyizolowany spośród innych za pomocą przeciwciała wiążącego EcoRI.
W kolejnej pracy zaproponowano dwie ważne modyfikacje umożliwiające stworzenie biblioteki epitopów prezentowanych przez faga:
1) miejsce klonowania przesunięto w obręb sekwencji kodującej N-koniec pIII, co umożliwiło przywrócenie pełnej infekcyjności faga,
2) zastosowano fagi fd-tet zawierąjace segment transpozonu Tn10, kodujący oporność na tetracyklinę, co ułatwiło kontrolę propagacji fagów i umożliwiło ich mianowanie w jednostkach transdukujących oporność na tetracyklinę.
Stosujac biotynylowane przeciwciała wyizolowano reaktywnego faga spośród 108 fagów typu dzikiego.
Wkrótce potem utworzono pierwsze bakteriofagowe biblioteki randomizowanych peptydów.
Dzięki zastosowaniu bibliotek fagowych można zidentyfikować peptyd stanowiacy ligand określonej molekuły.
Selekcja obejmuje trzy główne etapy:
1) inkubacja biblioteki fagowej z daną cząsteczką
(tzw. panning),
2) usunięcie fagów niezwiązanych,
3) amplifikacja fagów wiażących określoną czasteczkę w komórkach E.coli. Sekwencję aminokwasów w peptydzie określa się dzięki sekwencjonowaniu
DNA faga.
Spośród wielu zastosowań tej techniki na uwagę zasługuje procedura uzyskiwania epitopów peptydowych (mimeotopów) identycznych strukturalnie z wybranym epitopem. Strategia zastosowania mimeotopów zamiast antygenów glikolipidowych otwiera nowe możliwości czynnej immunoterapii nowotworów. Przykładem takiego podejścia badawczego jest poszukiwanie sekwencji peptydów imitujących strukturalnie gangliozyd GD2 obecny na komórkach nerwiaka zarodkowego (neuroblastoma).
Phage display może zatem spełniać rolę metody pomocniczej przy terapii genowej lub blokowania angiogenezy nowotworowej. Patrząc z tej perspektywy technika ta budzi duże nadzieje naukowców.
Phage display jest swoistym połączeniem niskiej ceny z możliwościami technik molekularnych. Jednakże do projektowania nowych leków i pomocnych wysokozaawansowanych urządzeń i programów, wydaje się nie być optymalna. Chociaż dla zwykłego badacza jest tania i prosta w stosunku do oferowanych możliwości. Wykorzystywane w niej standardowe techniki mikrobiologiczne podobne do wszystkich w biologii molekularnej i jej kluczowe zasoby: biblioteki fagowe i klony są powielarne i niezwykle tanie po założeniu i selekcji. Godne zauważenia jest, że w pojedynczej 1,5 ml mikrowirówkowej probówce możemy umieścić kilka bilionów cząstek fagowych ukazujących miliardy różnych struktur peptydowych - z nadmiaru których różni badacze w różnych celach selekcjonują klony o wysokiej wartości.
Rysunek 1. Schemat ewolucji ukierunkowanej białek.
Legenda:
1. Tworzenie biblioteki białek
2. Screening lub selekcja molekuł
3. Kolejny etap mutagenezy
Rysunek 2. Selekcja powinowactwa ligandów z bibliotek typu phage display
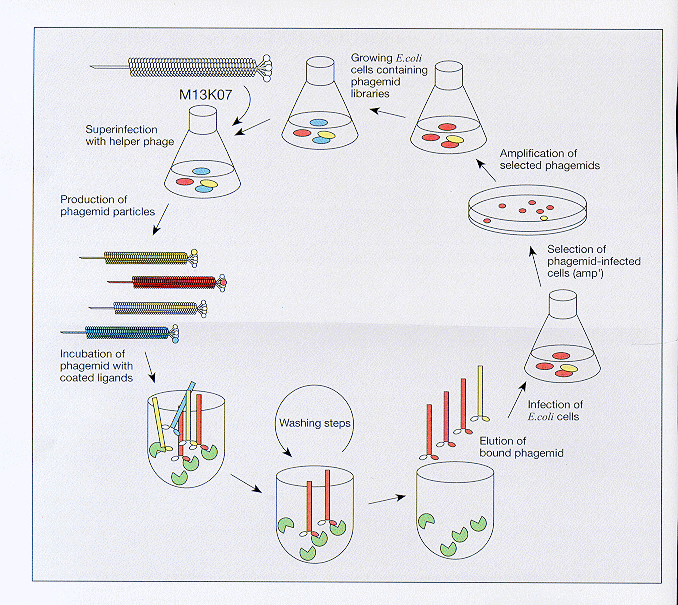
Rysunek 3. Schemat panningu
Wyszukiwarka
Podobne podstrony:
Phage display (2)
KB04 SMD DISPLAY
Plant Display
Display A Z
easy500 Text display HLP EN
Display, Imager Troubleshooting
evoluation of displacement
Add a signal strength display to an FM receiver IC
20LCD DISPLAYid 28898
Display Information
Display Cabinet 2
DisplayScripts A Z
Numerisches LED Display
PCI POSTcode display
10 Large Displacement Electrostatically Actuated Microrelay
Display Cases
diag display
Display Cabinet 3
więcej podobnych podstron