Które stwierdzenia dotyczące transkrypcji genów u E.coli są prawdziwe?
produktem może być policistronowy mRNA
faza elongacji podczas transkrypcji jest przeprowadzana przez rdzeń polimerazy
polimeraza RNA wykazuje aktywność egzonukleazową, która umożliwia jej korekcję błędów
rRNA (tzw.odwrotny RNA) jest syntetyzowany w kierunku odwrotnym do typowego 5'3', stąd jego nazwa
pre-mRNA podlega procesowi składania (slicingu) jeszcze zanim zajdzie translacja
ekspresja genów jest kontrolowana głównie na poziomie transkrypcji
Przykładami przedtranslacyjnej kontroli ekspresji genów u Eukaryota są:
alternatywny splicing pre-mRNA, dzięki któremu powstają różne cząsteczki transkryptu
rozluźnienie struktury spakowanego DNA poprzez modyfikacje białek histonowych
glikozylacja i fosforylacja polipeptydów, aby mogły się stać funkcjonalnymi białkami
selektywna degradacja cząsteczek mRNA
regulacja tworzenia się kompleksu inicjującego translację
selektywne składanie aktywnych kompleksów transkrypcyjnych na ….
Które stwierdzenia dotyczące ekspresji genów u Eukaryota są poprawne?
geny w obrębie upakowanej chromatyny są aktywne
aktywatory i represory działają poprzez zmianę szybkości tworzenia się kompleksu transkrypcyjnego
zaktywowane związanym hormonem receptory wiążą się ze specyficznymi sekwencjami…….”… elementami odpowiedzi na hormon”
rejony chromosomów, w których aktywnie zachodzi transkrypcja nazywamy „pufami” …szczególnie zwartą strukturą DNA
większość genów Eukaryota znajduje się pod kontrolą jednego aktywatora i jednego represora
w wielu białkach kontrolujących ekspresję genów, wiążących się specyficznie do….. „palca cynkowego”
Które stwierdzenia dotyczące roli białek ochronnych są poprawne?
wiążą się z rosnącymi łańcuchami polipeptydowymi
umożliwiają na zwykle niedozwolone interakcje pomiędzy cząsteczkami
są powolnie działającymi ATPazami
przykładem jest grupa białek szoku termicznego
kompleks ADP-chaoperon ma duże powinowactwo do białek rozfałdowanych
żadne z powyższych
Które z poniższych stwierdzeń dotyczących sekwencji sygnałowych są poprawne?
wewnętrzne sekwencje sygnałowe nie są odcinane po przejściu przez błonę ER
cząsteczką rozpoznającą sygnał (SRP) jest białkiem składającym się z …
uwolnienie SRP z rybosomu powoduje zahamowanie elongacji polipeptydu
SRP zapobiega przedwczesnej elongacji, a przez to fałdowaniu się białka
cykl ATP-ADP…sekwencję sygnałową z SRP i odłącza ją od …
rozfałdowane łańcuchy polipeptydowe są optymalnymi substratami do transportu przez błonę
Które stwierdzenia na temat aminoacylo-tRNA są poprawne?
wiązanie estrowe w aminoacylo-tRNA tworzone jest między grupą karboksylową aminokwasu a grupą fosforanową tRNA
dla każdego aminokwasu istnieje inny enzym katalizujący tworzenie odpowiedniego …
rodzaj aminokwasu przyłączanego do tRNA zależy od rodzaju syntetazy … jest substratem
niektóre syntetazy aminoacylo-tRNA posiadają aktywność korekcyjną … niewłaściwym tRNA
wszystkie syntetazy aminoacylo-tRNA rozpoznają właściwy tRNA rozpoznając …
aminokwas połączony z tRNA odgrywa istotną rolę w rozpoznaniu …
Które z poniższych stwierdzeń dotyczących sygnałów rozpoczynających i kończących transkrypcję są poprawne?
za prawidłowe rozpoznanie miejsca startu transkrypcji odpowiada podjednostka α polimerazy RNA
typowe promotory E.coli zawierają w obrębie -10 tzw. kasetę TATA o sekwencji zgodnej TATAA
promotory genów szoku cieplnego różnią się w sposób zasadniczy od typowych promotorów rozpoznawanych przez inne warianty odpowiedniej podjednostki polimerazy RNA
sygnałem terminacji transkrypcji jest cześć RNA o strukturze spinki do włosów przed kilkoma resztami UUU
heksameryczne białko rho jest ATPazą w obecności jednoniciowego RNA i uczestniczy w … niektórych genów E.coli
u E.coli wyspecjalizowane sygnały terminacji transkrypcji zwane atenuatorami … określonych genów do potrzeb pokarmowych komórki
Pytanie na temat syntezy białka u Prokaryota?
hydroliza GTP przez czynnik elongacyjny EF-Tu umożliwia uwolnienie …
energia potrzebna do utworzenia wiązań peptydowych … peptydylotransferazę
puromycyna powoduje przedwczesną terminację łańcucha … umożliwiając tym samym przesunięcie deacylowanego …
(brak pytania)
ponieważ transkrypcja zachodzi w jadrze komórkowym, a translacja w cytoplazmie, konieczny jest transport transkryptów z jądra do cytozolu
ponieważ wszystkie pierwotne transkrypty zawierają introny, konieczny jest splicing
pierwotne transkrypty tRNA podlegają intensywnej obróbce potranskrypcyjnej, po … transestryfikacji zbliżone mechanizmem do splicingu pre-mRNA
struktura określana mianem „kap” tworzona jest na końcu 5' większości mRNA oraz …
większość genów kodujących mRNA jest rozcinana przez endonukleazę rozpoznającą … od końca 3' przez polimerazę poli(A)
cząsteczki pre-mRNA kodujące część białek podlegają procesowi redagowania co, … transkryptu nie jest w pełni zgodna z sekwencją odpowiedniego egzonu w genie kodującym …
Kierowanie białek:
wewnętrzne sekwencje sygnałowe nie są odcinane po przejściu przez błonę
cząsteczki rozpoznające SRP to białka składające się z 7 łańcuchów polipeptydowych
uwolnienie SRP z rybosomu powoduje zakończenie elongacji
SRP zapobiega przed elongacją i fałdowaniem
rozfałdowanie łańcuchów polipeptydowych - optymalna substancja do transportu
Translacja mRNA u Prokaryota:
1 kodon AUG od końca 5' transkryptu hydrolizującego ATP
równanie reakcji: …
cząsteczka elongacyjna Ts wiąże GTP, podlega hydrolizie EF-Ts do GDP
cząsteczka elongacyjna Tu oddziałuje ze wszystkimi cząsteczkami aminoacylo-tRNA oprócz fMet-RNAf
peptydylo-tRNA może zajmować miejsce P lub A zależnie od tego, w którym cyklu komórkowym się znajduje
fMet-tRNA zajmuje miejsce P (jako jedyne)
Transkrypt o sekwencji 5' AAUUGUAA 3'
RNA 5' AAUUGUAA 3'
DNAMATRYCOWA, ANTYSENSOWNA 3' TTAACAT T 5' 5'TTACAATT 3'
Komplementarna nić do transkryptu
DNAKODUJACA, SENSOWNA 5' AATTGTAA 3'
Histony - modyfikacje potranslacyjne na końcu N'
a) ulegają acetylacji , fosforylacji , ATP-zowane , ubikwitynacji
b) wystawienie ogonów na działanie acetylaz
c) acetylacja reszt Lys w ogonach histonowych (Lys i Arg mogą być!)
d) kod histonowy - było coś o tym, że kod histonowy jest informacją czy zbiorem informacji o modyfikacjach czy coś takiego.
Operon laktozowy
a) y koduje β-galaktozydaze (z koduje beta-galaktozydazę, a y permeazę)
b) tworzy wiązanie 1,6-βglikozydowe w allolaktozie
c) katalizuje tworzenie glukozy i czegoś tam jeszcze (nieprawda, bo glukozy i galaktozy)
d) katalizuje syntezę laktozy z glukozy i galaktozy
e) łączy z kompleksem CAP-cAMP w miejscu operatorowym (to jest źle bo ten kompleks łączy się z miejscem tzw. CAP-site, a nie z operatorem)
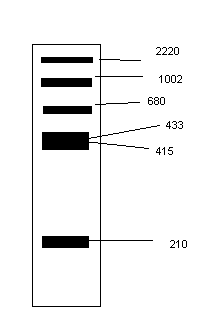
Enzymy restrykcyjne. Pocięto liniowy odcinek DNA enzymem EcoRV , obraz po elektroforezie uwidoczniono na żelu. Co możesz powiedzieć na podstawie rysunku poniżej? Żel wybarwiono bromkiem etydyny. Zwróć uwagę na słabo rozdzielone elementy o długościach 433 i 415 pz.
W żelu znajduje się bromek etydyny
Na tym odcinku DNA znajduje się 5 unikalnych miejsc restrykcyjnych rozpoznawanych przez enzym (tak, bo to był liniowy DNA i powstało 6 kawałków, czyli 5 miejsc restrykcyjnych)
Długość cząsteczki Dna wynosi 2220 pz
Najszybciej migrującym fragmentem jest fragment o długości 210 pz
Najszybciej migrującym fragmentem jest fragment o dług. 2220 pz
Żadna z powyższych nie jest prawdziwa
Metoda ta może być stosowania do produktów ekspresji metody Sangera (nie, bo w metodzie dideoksy Sangera nie stosuje się elektroforezy, tylko robi się autoradiogram, a elektroforezę stosuje się do metody Southern)
17. Represor λ
- gdzie się łączy białko cro i co robi - łączy się z OR3
- kiedy faza lityczna, kiedy lizogenna
- w swoim cyklu lizogennym ma taki okres, że powstaje hybryd DNA-RNA
- faza lizogenna jest utrzymywana dzięki działaniu represora lambda
18. Redagowanie RNA
- dodatkowa zmienność genetyczna
- zachodzi już po transkrypcji
- zachodzi przy udziale enzymów, np. polimerazy poli(A) (inny przykład to deaminaza przy apolipoproteinie B 100 i 48)
- zachodzi autokatalitycznie - raczej nie
19. Immunoprecypitacja
a) dotyczy to euchromatyny (czyli tej rozwiniętej chromatyny), tzn. że te przeciwciała zwiążą się z euchromatyną (bo tam jest fragment rozpleciony, czyli grupy acetylowe są na wierzchu)
b) przeciwciało łączy się do Lys na N' końcu
c) ukazane rejony są aktywne transkrypcyjnie
d) mają więcej etylowanych reszt
20. Represory transkrypcji u Eucaryota
a) przyłączają się do rejonów cis, a same są trans
b) mają budowę modułową
c) mają 2 rejony wiążące
d) np. deacylacja N-końców histonów
21. Które z następujących zdań określa podwójną helisę DNA typu Watson-Crick?
a) dwa polinukleotydowe łańcuchy są zwinięte wokół wspólnej osi
b) tworzy pary A-C i G-T (nie, A-T i G-C)
c) helisa ma pełny obrót o 34 st., bo każda para obraca się o 36 st. względem sąsiedniej pary zasady i oddalonego 3,4 A
d) puryny i pirymidyny znajdują się w wewnętrznej stronie helisy, a szkielet fosfodiestrowy na zewnątrz
e) skład zasad analizowany z DNA wielu organizmów pokazuje ze ilość A i T jest równe, tak jak ilość G i C
f) można zmieniać sekwencje w jednej nici niezależnie od drugiej nici
22. Wprowadzenie genu w środek wektora, który zawiera odporność na antybiotyk to:
a) prowadzi do przeniesienia odporności na leki
b) jest nazywane inaktywacją insercyjną
c) powoduje czułość komórki na antybiotyk
d) może być używany do identyfikacji bakterii zawierających wektor z fragmentem DNA
e) jest metodą niszczenia patogenicznych bakterii
23. Bardzo długi odcinek DNA z genu Eukariota ( > 100 kb)
a) może być pakowany w fag
b) może być wydzielana z chromosomów sztucznych drożdży
c) musi posiadać końce kohezyjne do klonowania
d) może być analizowany przez wędrówkę wzdłuż chromosomu
e) mogą być odseparowane przez elektroforezę w żelu poliakryloamidowym
24. Ligaza DNA
a) katalizuje tworzenie się wiązania 3'-OH i 5P
b) wymaga kofaktora NAD+ albo ATP zależnie od źródła enzymu, dostarcza energii do tworzenia wiązań fosfodiestrowych
c) mechanizm katalizowanej reakcji wymaga utworzenia związania kowalencyjnie enzym-adenylan
d) katalizuje reakcje przez mechanizm który wymaga aktywacji fosforanem DNA przez formowanie wiązania fosfobezwodnikowego z AMP
e) wymagana w replikacji DNA naprawy i rekombinacji
25. Polimeraza DNA I
Wymagana w naprawie DNA
Wymagana w replikacji
Potrzebuje primera i matrycy
Usuwa primer i wypełnia szczeliny podczas replikacji
26. Polimeraza DNA II
Potrzebuje primera z 3OH wolna i matrycy
Wymagana w naprawie DNA
Posiada aktywność nukleazową 3-5
27. Polimeraza DNA III
Wymagana w replikacji
Potrzebuje primera i matrycy
Tworzy najwięcej DNA podczas replikacji
Posiada aktywność nukleazową 3-5
28. Podjednostki polimerazy RNA :
α wiąże bialko regulatorowe
β wiąże trifosforanów rybonukleotydów
β wiąże matrycę DNA
σ odszukuje miejsca promotorowe, pomaga zainicjować syntezę i oddysocjowuje od enzymu
29. Miejsca promotorowe E.coli
a) mogą wykazywać różną wydajność transkrypcji
b) dla większości genów zawierają rożne zgodne sekwencje
c) określa stronę startu dla transkrypcji na matrycy DNA
d) mają identyczne i określone sekwencje
e) są aktywne kiedy G lub C są zamienione w ich -10 rejon w czasie mutacji (mutacja powoduje utratę aktywności)
f) te które mają sekwencje prawie zgodne z sekwencjami zgodnymi i są separowane przez 17 par zasad są bardzo wydajne
30. Bąbel transkrypcyjny
a) część polimeryzacji enzymu
b) podjednostka σ (utrata tej podjednostki, bo wtedy rdzeń silniej się wiąże do matrycy DNA)
c) w przybliżeniu 17 nukleozydów nici kodującej DNA w formie pojedynczej
d) w przybliżeniu 12 nukleozydów końce 5' linii wydłużającej się RNA
e) w przybliżeniu 12 bp helisy DNA-RNA
31. Polimeraza RNA I
Jest zlokalizowana w jąderku
Tworzy prekursory 18s, 5,8s, 28s rRNA
Syntezuje RNA w kierunku 5'3'
Jest zbudowana z kilku podjednostek
32. Polimeraza RNA II
Jest zlokalizowana w nukleoplazmie
Tworzy prekursorowy mRNA
Silnie inhibitowany przez amanitynę
Syntezuje RNA w kierunku 5'3'
Jest zbudowana z kilku podjednostek
Ma podjednostkę z powtarzalną sekwencją aminokwasów regulowaną w fosforylacji
33. Polimeraza RNA III
Jest zlokalizowana w nukleoplazmie
Tworzy prekursorowy tRNA
Tworzy 5s rRNA
Syntezuje RNA w kierunku 5'3'
Jest zbudowana z kilku podjednostek
34. I grupa splicingowa
Reakcje transestryfikacji zrywania i tworzenia wiązań
Wymaga nukleozyd guanozowy albo nukleotyd
35. II grupa splicingowa
Wymagane wiązanie 2'-5' fosfodiestrowe
Reakcje transestryfikacji zrywania i tworzenia wiązań
Spliceosom jest wymagany
Produkt pośredni o kształcie lassa
36. Splicing mRNA
Wymagane wiązanie 2'-5' fosfodiestrowe
Reakcje transestryfikacji zrywania i tworzenia wiązań
Spliceosom jest wymagany
Produkt pośredni o kształcie lassa
snRNPs są wymagane
37. Splicing eukariotyczny tRNA
Aktywności ligazy i nukleazy są potrzebne
38. Splicing Tetrahymena thermophila
a) potrzebny kofaktor GTP, GDP
b) kofaktor atakuje miejsce 5' i traci PPi z konca 5'
c) kieszeń (zawiera cząsteczki prekursorowego RNA)
d) nie potrzeba ATP
e) kofaktor atakuje miejsce 3'
39. Kompleks cAMP-CAP
a) chroni przez DNAza -87 do -47
b) po jego związaniu do atenuatora trp może być transkrypcja genów struktury (nie wiąże się do miejsc atenuatora!!)
c) represor transkrypcji (aktywator!!)
d) w operonie ora wpływa na geny struktury
e) chroni miejsca -48 do -5
f) w obecności arabinozy wpływa na geny struktury
40. Bialko C operonu ara
a) wiąże z araO1 hamując transkrypcję genu
b) wiąże araO2 aktywując geny struktury
c) w obecności cAMP-CAP geny struktury operonu i związanie arabinozowego do araO1 i araI
d) powoduje wypętlenie, gdy zwiąże się z araO2 i araI
e) wiąże się specyficznie z DNA w obecności arabinozy
41. Bakteriofag λ
a) syntetyzuje tylko represor λ w bakterii lizogenicznych
b) ssDNA = recA oddziałuje z nim także represor λ
c) zostaje uwolnione z chromosomu bakteryjnego z kompleksu rexDNA
d) gdy nic nie atakuje, zmienia to w fazie lizogenicznej jest on obecny z uwagi na represor, który ulega autoregulacji
e) profag (DNA) jest także lizogenicznej, gdy nie aktywuje zmiany bo represor λ …
f) białko cro wiąże się do or3 i wtedy hamuje represor (gen c1)
g) białko cro związane z or1 to hamuje syntezę represora
42. Operon arabinozowy
a) konstutywna synteza białka C
b) w obecności cAMP-CAP i arabinozy nie da się zapętlić
c) cAMP-CAP i arabiznoza może przeprowadzać transkrypcje genów struktury, nieznacznie obniżają szybkość
d)może syntezować arabinozę przy wykorzystaniu białek powstałych z białek struktury
43. Odcinek liderowy mRNA
a) w obrębie genu trpD
b) transkrypcja operonu trp kontrolowana atenuatorem
c) krótki transkrypt liderowy kończy sekwencja poli(U)
d) reszty trp obecne obok siebie
e) niedobór trp bo brak tryptofanylo-tRNA rybosom zatrzymuje na kodujących trp
f) rybosom zatrzymuje tak, że transkrypcja poniżej atenuatora
g) koduje krotki peptyd testowy z 2 resztami trp
44. tRNA
a) antykodon i miejsce wiązania aminokwasu na przeciwległym końcu
b) zawiera od 70-90 nukleotydów
c) duża cześć jest sparowana
d) CCA na końcu 3'
e) wiele zmodyfikowanych nukleotydów
f) zawiera ACC, gdzie przyłączone aminokwasy (CCA!!)
g) zbudowany z helikalnych końców tworzących literę U
45. Translacja mRNA u Eukariota
a) formylometionylo-tRNA rozpoczyna łańcuch
b) kodon AUG wybierany przez parowanie rejonu mRNA
c) mała podjednostka powyżej miejsca transkrypcji
d) kończy czynnik uwalniający (u prokariotów one hydrolizują GTP)
e) biorą udział białka wiążące z 5' mRNA i inne czynniki inicjujące
f) może być regulowana przez kinazy białkowe
46. Translacja mRNA u Prokaryota
a) mRNA policistronowe
b) miejscem translacji jest kodom met AUG za sekwencją bogatą w puryny komplementarną do 16s tRNA
c) czynniki elongacyjne EF-Ts i EF-Tu
d) formylometionylo-tRNA powstaje w reakcji
e) EF-Ts wiąże nukleotyd podlegający hydrolizie
f) Tu oddziałuje z tRNA oprócz fMet-tRNA
47. Translacja
a) aminokwasy dodawane do N-końca
b) aminokwasy aktywowane przez przyłączenia do tRNA
c) rozpoczęcie na kodonie przez specyficzne pre-tRNA i kodonu
d) wiązanie peptydowe aminoacylo-tRNA miejsce A i reszta peptydylowa miejsca P
48. Dostępne mutanty:
i+ typ dziki
ic konstytutywna mutacja
is niewrażliwy na atak
o+ sekwencja dzikiego typu
oc mutacja konstytutywna
z+ sekwencja dzika
mutacja konstytutywna = nieregulowana, prowadzi do konstytutywnej aktywności
ic o+ z+
sekwencja nietypowa (nieregulowana) nie ma represora, bo substancja nieaktywna, -, niewrażliwa na laktozę = konstytutywna ekspresja zachodzi
is o+ z+
niewrażliwa na laktozę, bo ona nie jest związana, -, laktoza jest obecna i tak (jest represja bo nie ma laktozy lub IPTG który …… represor)
i+ oc z+
ic o+ z+
represor nierozróżnialny, typ dziki
is o+ z+
typ dziki, bo z dominujący, brak syntezy genu
ic nie jest dominujący względem i+
1,2,3 ich synteza nie zależy od laktozy
2 nie zależy od IPTG
4 ma fenotyp typu dzikiego, lambda represor w układzie trans
49. Inicjacja translacji u Prokaryota
a) aminoacylo-tRNA wiąże się do miejsca P
b) IF2 oddziałuje z podjednostka dużą
c) IF2 zdolny do hydrolizy GTP, dzięki czemu dostarcza energii do translacji
d) IF1 i IF3 dostarczają aminoacylo-tRNA do rybosomu
e) tRNA komplementarny do dużego segmentu
50. Syntetaza aminoacylo-tRNA
a) estry aminokwasów syntetyzowane na końcu 3' tRNA
b) reakcja katalizowana I etapowa (nie, bo II etapowa!!)
c) produkt przejściowy którego grupa karboksylowa tworzy wiązanie bezwodnikowe z ortofosforanem
d) większość syntetaz ma właściwości korekcyjne
e) wszystkie syntetazy rozpoznają tRNA oddziałując z pętlą antykodonu
f) potrzebne ATP
51. Histony
a) 140 par zasad otacza 8 białek histonowych
b) białka aktywatorami transkrypcji podjednostek
c) replikacja eukariotycznego chromosomu to kilka widełek
d) białka kodowane przez pojedyncze kopie genów
e) sekwencje powtórzeniowe to znaczny % DNA eukariotycznego
f) silnie zasadowe białka, ładunek `+'
g) białka histonowe H1, H2A, H2B, H3, H4
h) H1 rdzeń nukleosomu
i) geny kodujące histony wielokrotnie powtórzone
j) mRNA histonowe ulega adenylacji
k) histony nie maja intronów
l) histony wiążą się z nicią wiodącą
52. Białka zaangażowane w regulacje transkrypcji
- muszą aktywować polimerazę DNA (może!)
- muszą aktywować polimerze RNA (może!)
- muszą mieć zdolność wchodzenia do jąderka
- mogą się wiązać do DNA w białkach pęcherzyka
- musi wiązać się do histonów (może!)
+ zdolność wchodzenia do jądra (ale tylko u Eukariota)
52. Kontrola ekspresji genów
+ alternatywny splicing
+ sekwencje doprowadzające mRNA
+ rozluźnienie struktur upakowanych
+ regulacja tworzy kompleks inicjujący transkrypcję
53. Kontrola ekspresji genów u Eukariota
+ geny w rozluźnionej chromatydzie nieaktywne, zanim nie zostaną zaktywowane
- eukariotyczna polimeraza RNA samodzielnie transkrybuje mRNA
+ aktywator i represor działają przez zmiany tworzenia kompleksu inicjującego
- większość genów znajduje się pod kontrolą jednego aktywatora i represora (nie bo jest ich wiele!!)
+ pufy aktywują transkrypcje, luźna struktura
54. Telomerazy
+ nie uzupełniają nici opóźnionej
+ odwrotna transkryptaza 5'3'
+ RNA jest matrycą (nie starterem)
+ syntezuje nic w kierunku 5'3'
+ wymaga matrycy
+ syntezuje nić bogatą w G
- mają aktywność polimerazy RNA
55. Represor λ
- wiąże jako monomer
- ma miejsce wiązania ara
+ wiąże specyficznie do sekwencji DNA rozpoznawanych przez białko cro
+ rożne powinowactwo do miejsc OR
- rożne powinowactwo przez białko lex A (nie bo rex A!!)
56. Splicing alternatywny
+ segregacje selektywne RNA
+ regulacja kompleksu inicjującego translację
+ przetasowanie ekspresji genów
57. β-galaktozydaza
+ obecna w rożnych stężeniach zależnie od źródła węgla używanego do wzrostu
+ jest produkowana z jednostki genu zwanej operonem
+ hydrolizuje wiązanie 1,4-β oligosacharydy laktozy i tworzy galaktozę i glukozę
+ tworzy wiązanie 1,6-β oligosacharydu allolaktozy
- jest allosterycznie aktywowana przez niemetaboliczny składnik IPTG (izopropylotiogalaktozyd)
+ jej poziom wzrasta w koordynacji z permeazą galaktozydową i acetylofransferazą tiogalaktozydową
58. Operon laktozowy lac
i - koduje białko, które zakłóca aktywność polimerazy RNA
- koduje białko wiążące allolaktozę
- jest genem regulatorowym
- koduje represor lac
p - jest promotorem lac operonu
- zawiera specyficzne sekwencje wiązania dla polimerazy RNA
- zawiera miejsce wiązania dla kompleksu cAMP-CAP
o - jest operonem lac
- zawiera specyficzne sekwencje wiązania dla represora lac
z - koduje bialsko wiążące allolaktozę
y i a - koduje permeazę galaktozydową
- koduje represor lac
miejsce wiążące CAP - zawiera miejsce wiązania dla kompleksu cAMP-CAP
59. Bakteriofag λ w fazie litycznej
+ syntetyzuje 3 rożne klasy mRNA, które są wyznaczane poprzez czas po infekcji ich pojawienia
+ niosą programowaną syntezę białek potrzebnych do replikacji genów i produkcji ich strukturalnych komponentów
+ tworzą produkty genów N i Q, które działają jako białka regulacji pozytywnej, prowadzące sekwencyjną λ- kodującego białka
- początkowo produkuje dwie cząsteczki jądra, pełni rolę inhibitora syntezy λ represora, a inną gra rolę końca transkrypcji
+ N i Q zapobiegają końcu transkrypcji
+ N powoduje produkcję białka niezbędnego do replikacji DNA faga i rekombinacji
+ Q potrzebne, gdy są transkrybowane geny białka główki i ogonka wirusa oraz białka konieczne do lizy komórki gospodarza
60. Operon Trp
+ kontrola ilości policistronowego mRNA tworzącego na poziomie inicjacji transkrypcji
+ kontrola ilości policistronowego mRNA tworzącego na poziomie terminacji transkrypcji (atenuator sygnał terminacji)
+ sekwencja i skoordynowana produkcja 5 enzymów metabolizmu tryptofanowego z pojedynczej nici RNA
- sekwencja i skoordynowana produkcja 5 enzymów metabolizmu tryptofanowego z 5 rożnych mRNA, produkowanych w różnych stężeniach
+ produkcja transkryptów rożnych rozmiarów zależna od poziomu tryptofanu w komórce
61. RNA liderowy operonu trp
+ mutacja delecji w DNA kodującym 3' koniec RNA daje powód do wzrostu poziomu biosyntezy enzymów biosentyzowanych przekształcających trp
+ krótkie otwarcie ramki odczytu zawierającej kodon trp , wśród innych istnieje wewnątrz literowego RNA
+ liderowy RNA koduje peptyd którego zdolność syntezy monitoruje poziom trp-tRNA w komórce
- liderowy RNA zawiera wiązanie rybosomowe Shine-Dalgarno (nie, bo są dwie sekwencje!)
+ liderowy RNA może tworzyć dwie alternatywne i wzajemnie wykluczające się drugorzędowe struktury
+ struktura literowego RNA In vivo zależy od pozycji rybosomy translacyjnego go
62. lac represor
- wiąże sekwencje DNA z rejonami o podwójnej symetrii
- przeszkadza w funkcjonowaniu polimerazy RNA przez związanie z DNA
λ represor
- wiąże sekwencje DNA z rejonami o podwójnej symetrii
- przeszkadza w funkcjonowaniu polimerazy RNA przez związanie z DNA
- efekty regulatorowe są antagonizowane białkiem rho
- inaktywowane przez recA
- reguluje własną syntezę
- współdziała z powtarzalną sekwencją wariantów z ich operonami
- stymuluje inicjację transkrypcji
- bierze udział jako pozytywny i negatywny regulator
trp represor
- wiąże sekwencje DNA z rejonami o podwójnej symetrii
- przeszkadza w funkcjonowaniu polimerazy RNA przez związanie z DNA
kompleks cAMP-CAP
- wiąże sekwencje DNA z rejonami o podwójnej symetrii
- stymuluje transkrypcje różnych operonów katabolicznych
- stężenie zależy od poziomu glukozy
- stymuluje inicjację transkrypcji
białko araC
- reguluje własną syntezę
- bierze udział jako pozytywny i negatywny regulator
- przeszkadza w funkcjonowaniu polimerazy RNA przez związanie z DNA
- stymuluje inicjację transkrypcji
białka N i Q λ
- stymuluje transkrypcje przez stłumienie terminacji transkrypcji
atenuator operonu trp
- wymaga syntezy białka do funkcjonowania
- zależy od ściśliwości powiązania transkrypcji i translacji
- oddziałuje przez poziom aminoacyloacylo-tRNA w komórce
- wymaga rybosomów
bialka rybosomalne
- wymaga rybosomów
- kontrolowany przez represje translacyjna
- reguluje własną syntezę
- oddziałuje przez poziom aminoacyloacylo-tRNA w komórce
rRNA i tRNA
- wymaga rybosomów
- oddziałuje przez poziom aminoacyloacylo-tRNA w komórce
- wymaga PPPP
rekombinowany hin
- przekształca orientację promotora w stosunku do represora genu
- łamie i rozdziela wiązania fosfodiestrowe DNA
- wiąże sekwencje DNA z rejonami o podwójnej symetrii
Wyszukiwarka
Podobne podstrony:
5475
5475
M 5475 Leather jacket
5475
L 5475 Leather jacket
5475
więcej podobnych podstron