skanuj0031

6
6
9
7 8
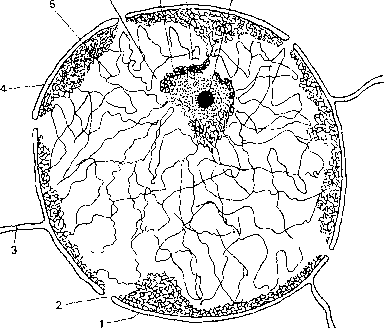
Rys. 8.1. Budowa jądra komórkowego.
1 — otoczka jądrowa, 2 — pory otoczki jądrowej, 3 — kanał siateczki s'ródplazmatycznej, 4 — fibiyle chromatyny skondensowanej, 5 — fibiyle chromatyny luźnej, 6 — kariolimfa, 7 — chromatyna jąderkowa, 8 — ziarniste składniki jąderka, 9 — włókniste składniki jąderka
Skład chemiczny chromatyny
DNA
Zawartość DNA w przeliczeniu na genom, tj. podstawowy (monoploidałny) zespół chromosomów organizmu eukariotycznego, jest znacznie wyższa niż u organizmu prokariotycznego i waha się w znacznych granicach. Nie jest ona skorelowana z liczbą chromosomów, lecz z ich łączną długością (tab. 8.1).
Tabela 8.1. Zawartość DNA na genom oraz liczba chromosomów u różnych gatunków
|
Gatunek |
Ploidalność |
DNA (pg) |
Liczba chromosomów |
|
Saccharomyces cerevisiae (drożdże) |
n |
0,025 |
17 |
|
Drosophila melanogaster (muszka owocowa) |
2n |
0,241 |
8 |
|
Rana pipiens (żaba) |
2n |
15,600 |
26 |
|
Triturus viridescens (traszka) |
2n |
98,000 |
22 |
|
Człowiek |
2n |
5,060 |
46 |
Liczba cząsteczek DNA wynosi tyle, ile liczba chromosomów. Za obecnością tylko jednej cząsteczki DNA w każdym chromosomie (chromatydzie), tj. za koncepcją jednopasmowej budowy chromosomów, przemawiają następujące argumenty zaczerpnięte z dowodów genetycznych i biochemicznych:

— analiza wyników rekombinacji, w świetle której każdy chromosom zachowuje się tak, jak pojedyncza cząsteczka DNA;
— mutacje punktowe (czyli genowe) polegające na drobnych modyfikacjach w strukturze DNA dają w konsekwencji zmiany w strukturze pierwszorzędowej kodowanych przez nie białek;
— obecność w DNA plemnika myszy znacznej liczby genów w pojedynczych kopiach;
— sposób wyznakowania chromosomów radioaktywną tymidyną po 2 cyklach replikacji DNA: wyznakowanie chromosomów jest zgodne z semikonserwatywnąreplikacjąDNA(p. s. 120);
— obecność jednej cząsteczki DNA w jednej chromatydzie wykazana w badaniach określających masę cząsteczkową DNA metodą wirowania.
Za koncepcją przeciwną, tj. za wielopasmowością chromosomów, przemawiały wyniki badań cytologicznych u gatunku z dużymi chromosomami, wykazujące występowanie w chromosomach anafazowych (chromatydach) 2, 4 lub więcej linearnych podjednostek zwiniętych wokół siebie. Obrazy takie mogą być artefaktem utrwalania lub artefaktem optycznym. Wymiany subchromatydowe, uważane za czołowy dowód świadczący o wielopasmowości chromosomów, mogą być interpretowane jako wynik lokalnej denaturacji DNA powodującej rozdzielenie obu nici polideoksyrybonukleotydowych. Nie jest wykluczone, że u niektórych gatunków nastąpiła politenizacja, tj. zwiększenie liczby cząsteczek DNA w chromosomie. Zjawisko takie stanowiłoby jednak wyjątek od reguły jednopasmowości chromosomów.
Zawartość podstawowa (2C) DNA zwiększa się w miarę osiągania wyższego szczebla rozwoju ewolucyjnego tylko oAProkaryota do niższych Eukaryota, tj. do owadów w świecie zwierząt i do grzybów włącznie w szeregu ewolucyjnym roślin (tab. 8.1). W skład DNA wchodzą tzw. sekwencje unikatowe oraz sekwencje powtarzalne. Brak zgodności między szczeblem ewolucji a zawartością podstawową DNA u wyższych organizmów zwierzęcych i roślinnych jest spowodowany obecnością powtarzalnych sekwencji DNA, których ilość w znacznej mierze decyduje
0 zawartości 2C DNA, ponieważ u organizmów wyższych stanowią one od 40 do 90% całkowitego DNA. Sekwencje unikatowe, tj. kodujące białka występują w zasadzie w liczbie 2 na genom diploidalny; rzadko jest ich więcej, jak np. geny kodujące histony, keratyny oraz białka zapasowe u roślin. Zawierają one odcinki kodujące poszczególne fragmenty cząsteczek białek, tzw. eksony oraz rozdzielające je, nie ulegające translacji — introny (p. rozdz. 10). Do sekwencji powtarzalnych zalicza się geny kodujące RNA (tylko niektóre z nich zawierają introny) nie ulegające translacji. tj. rRNA. tRNA i 5SJRNA (p. rozdz. 10) oraz tzy...satelitarny DNA nie transkrybowany in vivo; pewnym sekwencjom powtarzalnym przypisywany jest udział w regulacji ekspresji genów. Geny kodujące rRNA występują u zwierząt w liczbie od 200 (Drosophila melanogaster) do 440 (człowiek); u roślin okrytozalążkowych liczba kopii rDNA jest znacznie większa i wynosi od około 2 000 (len) do około 14 000 (cebula) w genomie diploidalnym. U żaby Xenopus laevis geny kodujące 5S RNA są powtórzone w 24 000 kopii (po 700 par nukleotydów), stanowiąc 0,5% całkowitego DNA, zaś geny kodujące tRNA — w 8 000 kopii (po 800 par nukleotydów), co wynosi 0,0115% całkowitego DNA. W genach kodujących rRNA, 5S RNA i tRNA tylko ich część ulega transkrypcji i dalszemu dojrzewaniu. U Xenopus laevis długość nietranskrybowanego odcinka (tzw. przerywnika) stanowi od 10% długości genu tRNA do 50% genu rRNA. Satelitarny DNA wykazuje inną lokalizację przy wirowaniu preparatów DNA w gradiencie stężeń CsCl niż główne pasmo DNA (stąd nazwa tej frakcji). Zawartość DNA satelitarnego stanowi stałą cechę genomu i wynosi np. 0,5% całkowitego DNA u człowieka, 10% u myszy, około 20% u melona
1 aż 30% u kraba. U niektórych gatunków roślin wyższych nie wykryto satelitarnego DNA. Istotną cechą satelitarnego DNA jest największa—w porównaniu z innymi frakcjami DNA — szybkość renaturacji (tj. ponownego łączenia w dwuniciowe struktury cząsteczek DNA zdenaturowanych,
101
Wyszukiwarka
Podobne podstrony:
PODSTAWY HISTOLOGII I CYTOLOGII Ewolucja komórki. Ogólna budowa jądra komórkowego, Poziomy organizac
BUDOWA JĄDRA KOMÓRKOWEGO, JEGO BUDOWA I ROLA W KOMÓRCE. Jądro komórkowe jest główną organellą
Ogólna budowa jądra komórkowego nucłear envelopc nucteolus nuclear
Budowa jadra komórkowego H jądro komórkowe w komórce roślinnej ma zwykle postać drobnego ciałka o
Spis treści >Budowa jądra atomowego >Siły jądrowe > Defekt masy >
CCF20100206�037 2. Analiza budowy i funkcji składników komórki czynnościowego komórki otoczka jądrow
skanuj0005 Prometafaza Na początku prometafazy otoczka jądrowa ulega fragmentacji. Zapoczątkowuje ją
PB041117 Budowa jądra interfazowego Dwa główne składniki: 1. otoczka jądrowa: 2.
skanuj0012 WYKŁAD 5 Budowa i funkcje ośrodkowego układu nerwowego c.d.: kresoraózgowie - jądra r>
skanuj0014 (93) Budowa oka gałka oczna ciało szkliste neurony komórki światłoczułe siatkówka tRyc.
IMGd56 JĄDRO KOMORKOWI*; • Otoczone podwójną błoną jądrową z Pory „furtki"
Jadro komórkowe-jest otoczone otoczką jądrową (podwójną błoną), mającą bezpośrednie połączenie z
więcej podobnych podstron