
2009-01-08
1
Bioinformatyka
– wykład 8
2.XII.2008
białkowa
bioinformatyka
strukturalna
krzysztof_pawlowski@sggw.pl

2009-01-08
2
Lecture
outline, Dec. 6th
•
protein structures
–
why?
•
protein structures
–
geometry and
physics
•
covalent
modifications
•
globular
proteins
vs
transmembrane and
fibrous
proteins
•
protein topology, disordered
regions, structural
domains

2009-01-08
3
Plan wykładu
•
struktury
białek
–
dlaczego?
•
struktury
białek
–
geometria i fizyka
•
modyfikacje kowalencyjne
•
białka globularne a białka
transmembranowe i włókniste
•
regiony nieuporządkowane

2009-01-08
4
Struktury białek –
dlaczego
warto je znać
i rozumieć
a
zrozumienie lub przewidywanie funkcji
a
planowanie modulowania funkcji
– np. projektowanie leków (drug design)
a
projektowanie modyfikacji struktury bądź funkcji
(inżynieria białkowa - protein engineering
)
a
niektórzy uważają, że białka są ładne i ciekawe
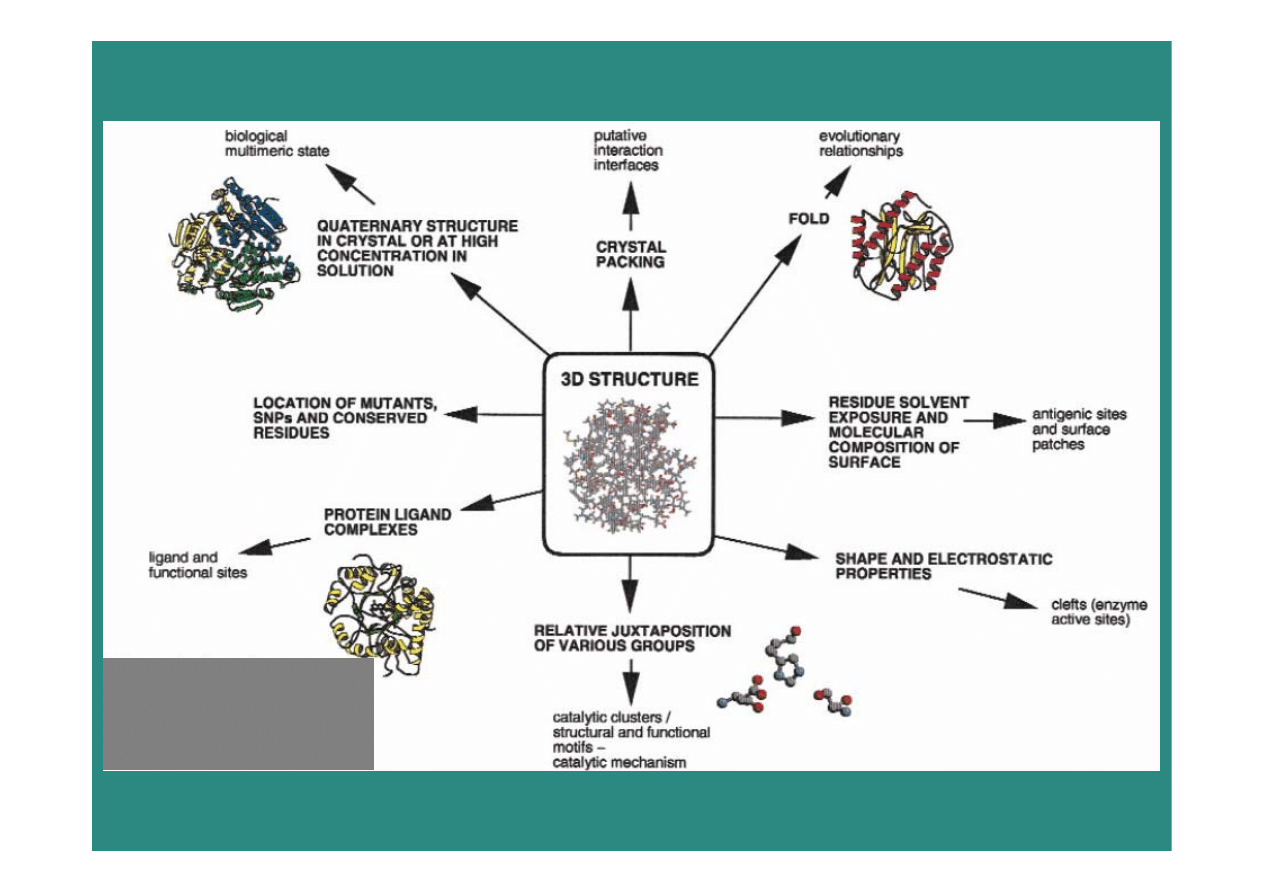
2009-01-08
5
Thornton
Nat Struct
Biol. 2000; 7 Suppl:991-4.

2009-01-08
6
Plan wykładu
•
struktury
białek
–
dlaczego?
•
struktury
białek
–
geometria i fizyka
•
modyfikacje kowalencyjne
•
białka globularne a białka
transmembranowe i włókniste
•
regiony nieuporządkowane

2009-01-08
7
Christian Anfinsen:
w danym środowisku struktura
trójwymiarowa białka jest w pełni
zdeterminowana przez jego sekwencję
aminokwasową
i odpowiada minimum
energii swobodnej
Anfinsen, C.B.,
Principles that govern
the folding of protein chains.
Science, 1973. 181: p. 223-30.
2009-01-08
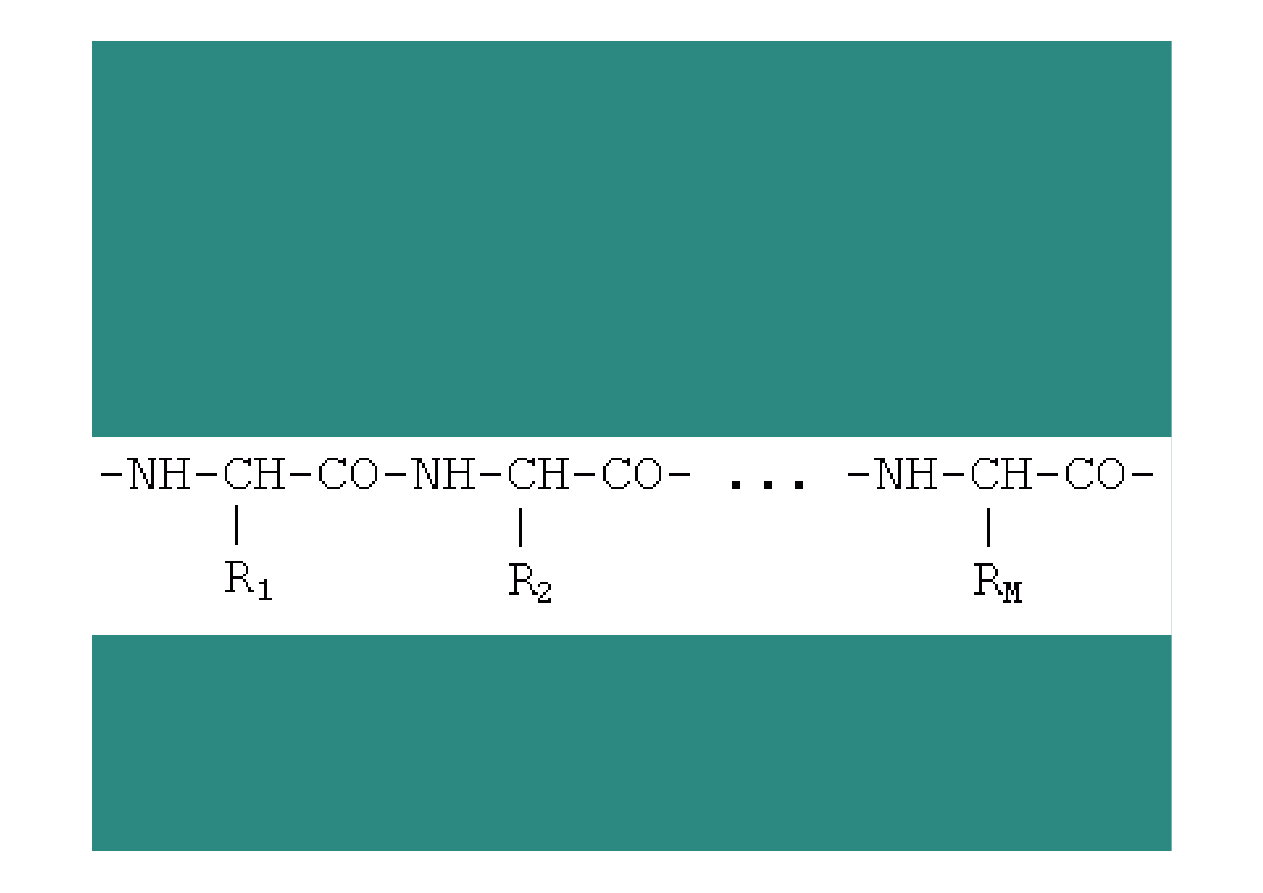
8
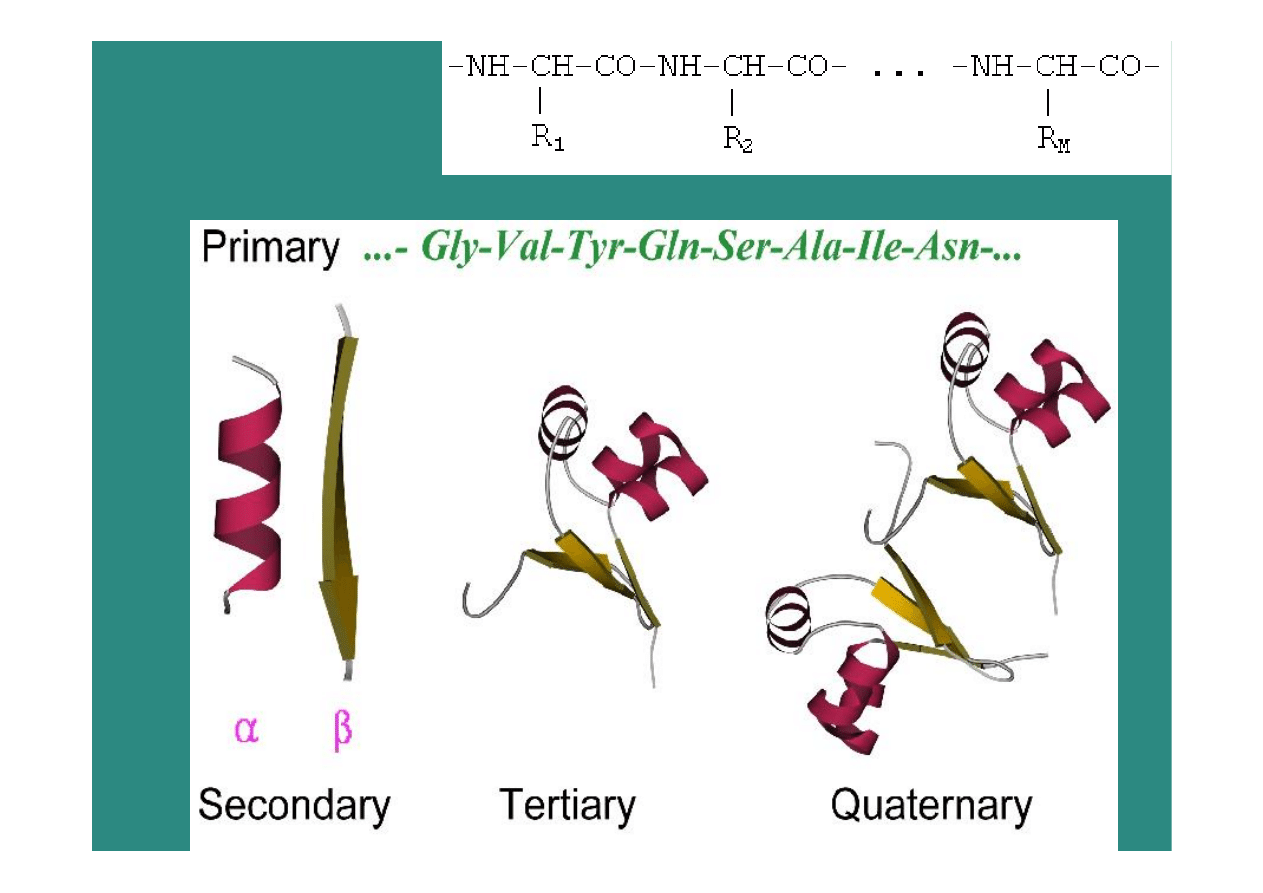
Łańcuch
białkowy
:
Regularny
łańcuch główny (main
chain),
Kodowane przez geny łańcuchy boczne
(side chains)
~ 20
100
sekwencji
~ 3
100
konformacji
2009-01-08
9
Protein chain
Covalent bond
lengths:
0.9 –
1.8 Å
Covalent bond
angles:
109
o
– 120
o
Atom radii:
1 –
2 Å
Peptide
bond
2009-01-08
10
Protein chain
Covalent bond
lengths:
0.9 –
1.8 Å
Covalent bond
angles:
109
o
– 120
o
Atom radii:
1 –
2 Å
Amino-acid
residue
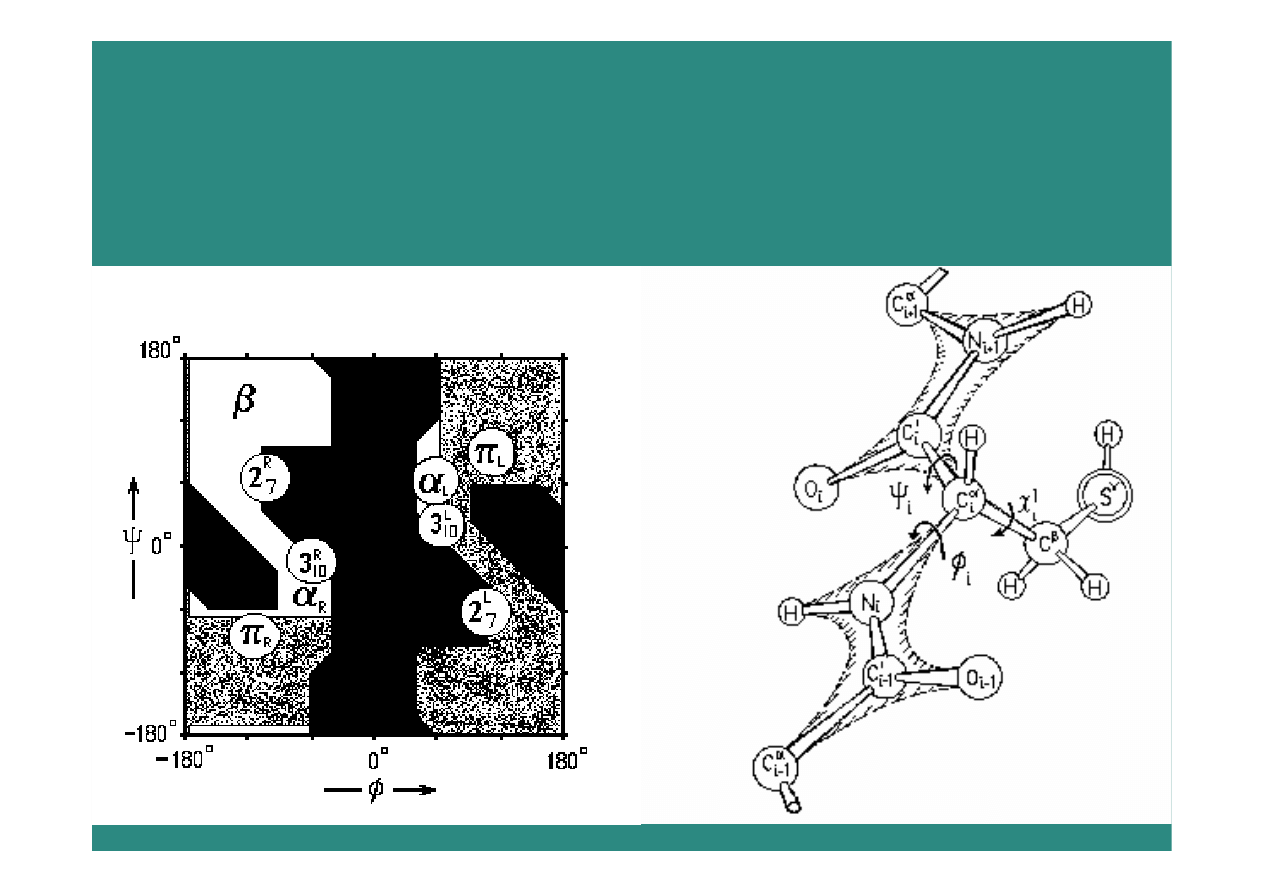
2009-01-08
11
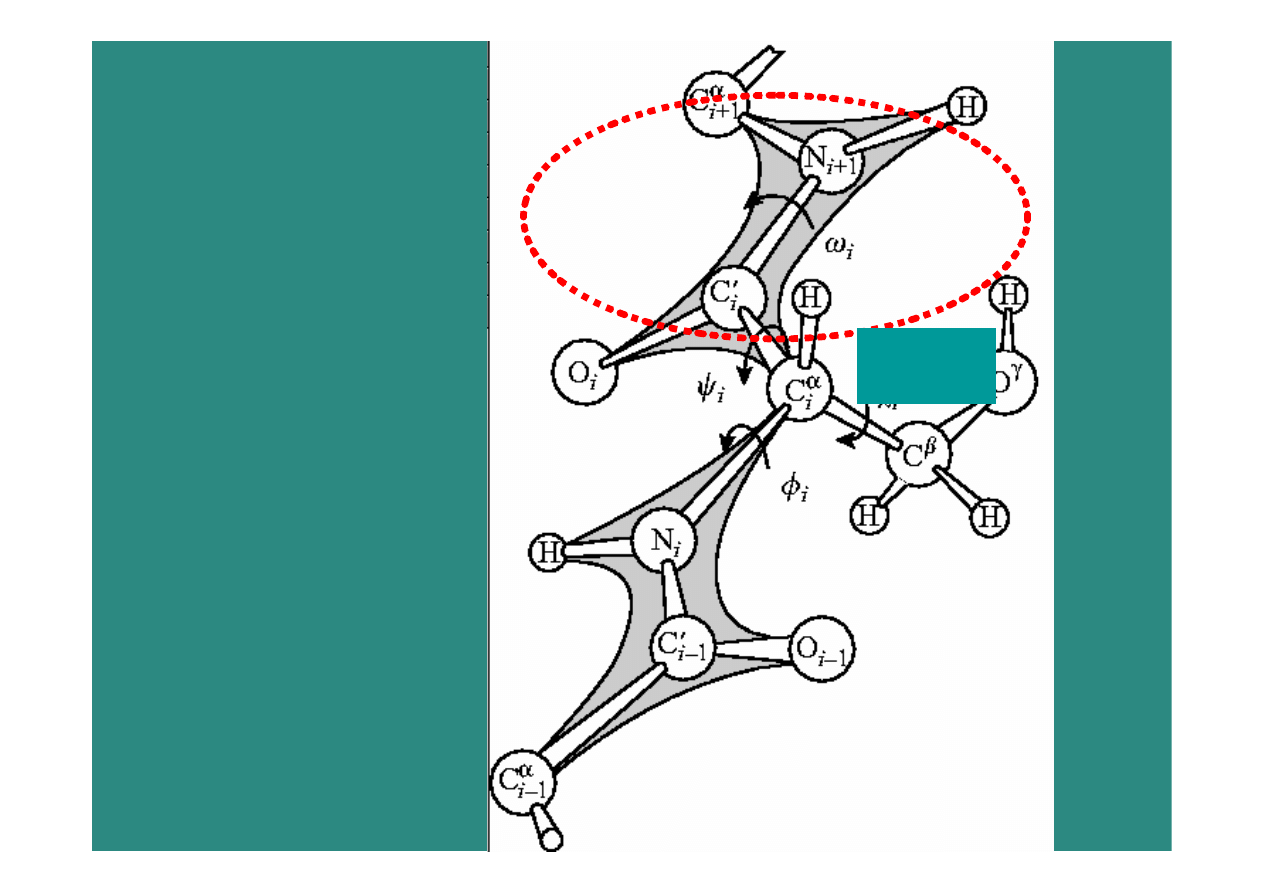
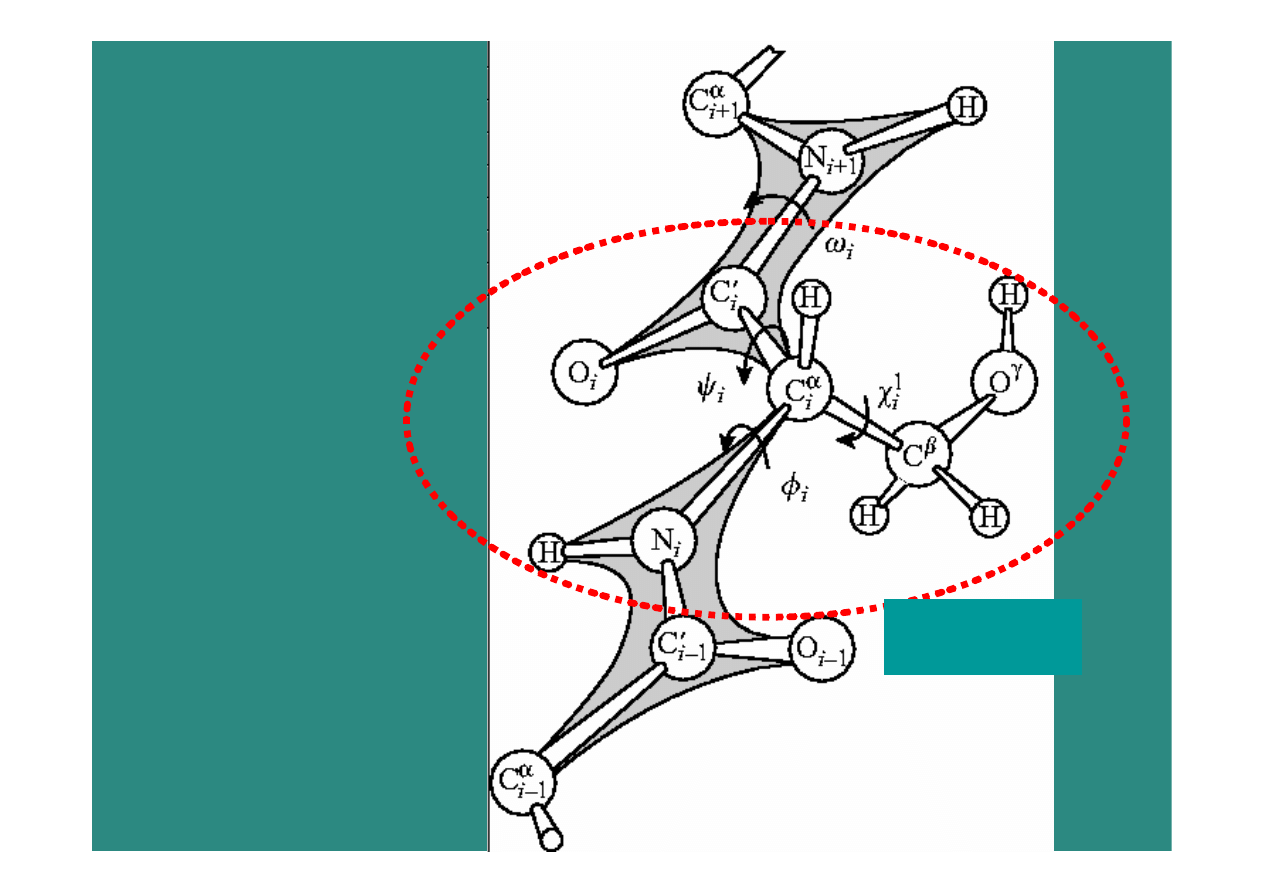
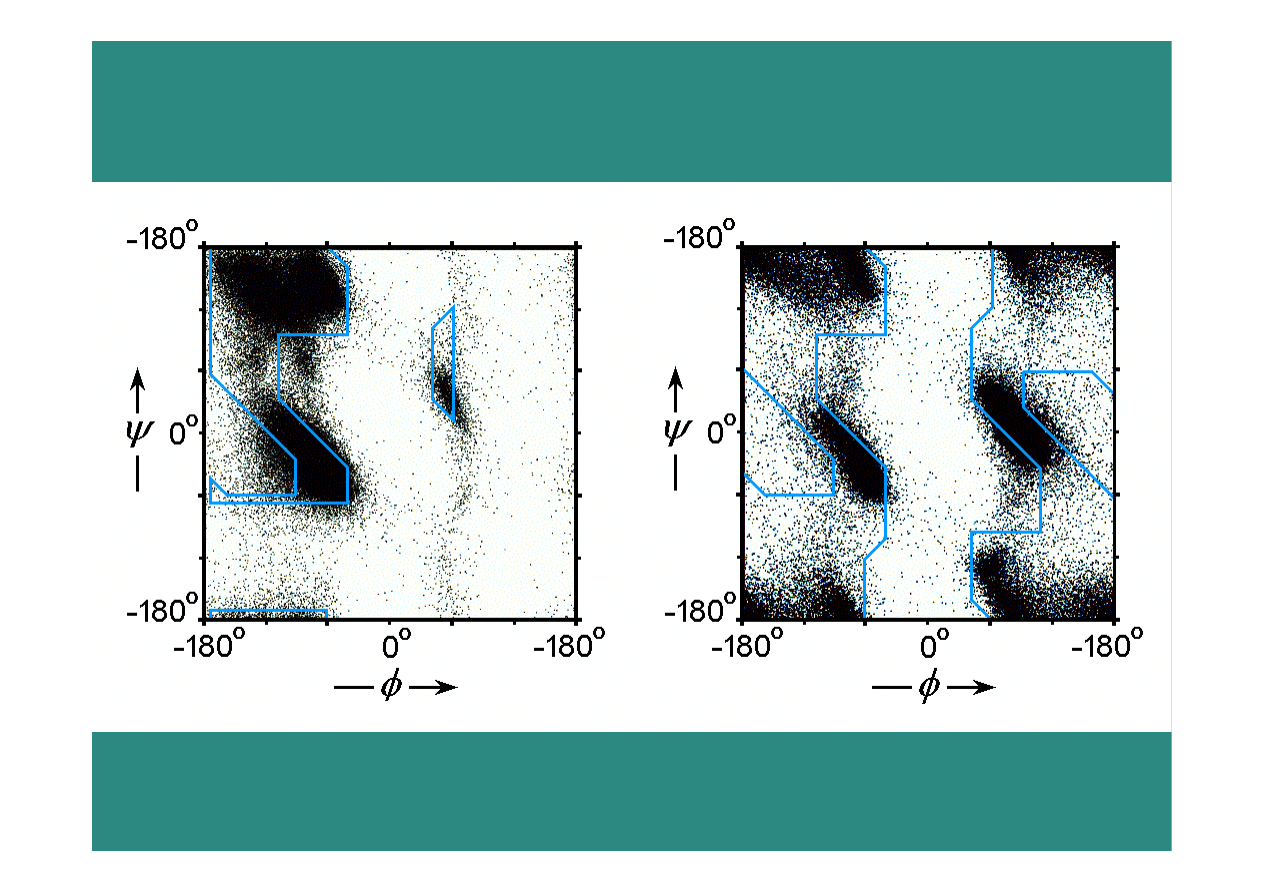
Wykres Ramachandrana
dozwolone obszary kątów
φ, ψ
2009-01-08
12
ALA, etc. GLY
ALA, etc. GLY

2009-01-08
13
Zapis struktury białka:
Współrzędne wewnętrzne -
reszty
aminokwasowe
φ1, ψ1
φ2, ψ2
φ3, ψ3
...
Współrzędne kartezjańskie –
atomy
x1, y1, z1
x2, y2, z2
…

2009-01-08
14
Wiązania wodorowe
•
„likelihood
of
finding
an
unsatisfied
hydrogen
bond
in
a protein is
insignificant„
Protein Sci. 2005;14:1911
•
Problem definicji
–
wykrywania wiązania wodorowego
w znanych strukturach
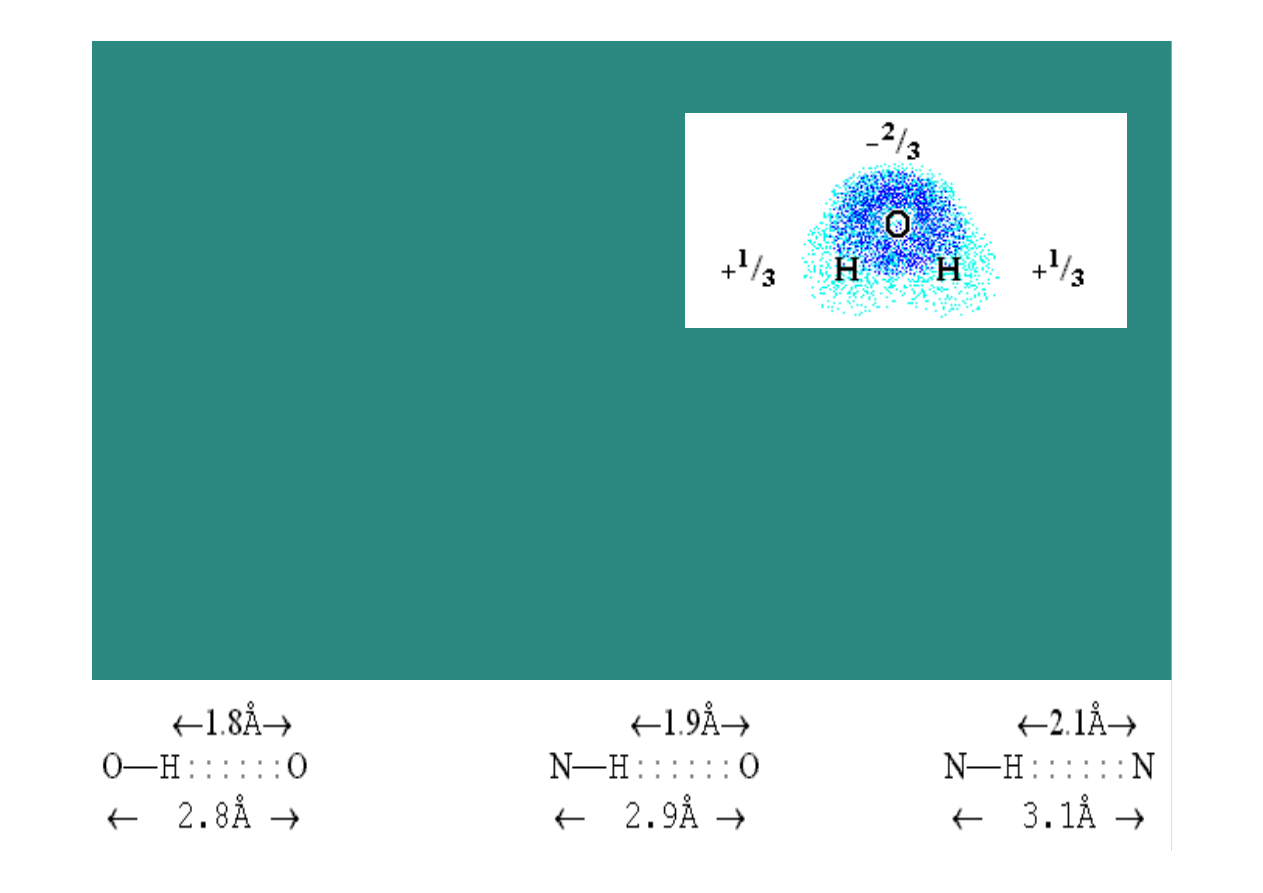
2009-01-08
15
cząsteczka wody
Wiązania wodorowe
Oddziaływanie dipol-dipol
Energia wiązania wodorowego w białku
– rzędu 2
kcal/mol
(w wodzie
5 kcal/mol
)
wiązania białko-białko oraz białko-woda
2009-01-08
16
Wiązania wodorowe
•
Donor: H w grupach OH, NH, NH2
(NH -
łańcuch główny)
•
Akceptor: O, N (wolne pary elektronowe).
(CO -
łańcuch główny)
•
Łańcuchy boczne –
np. Ser, Tyr
(często
mogą
być
akceptorami oraz donorami
•
WODA
2009-01-08
17

2009-01-08
18
2009-01-08
19
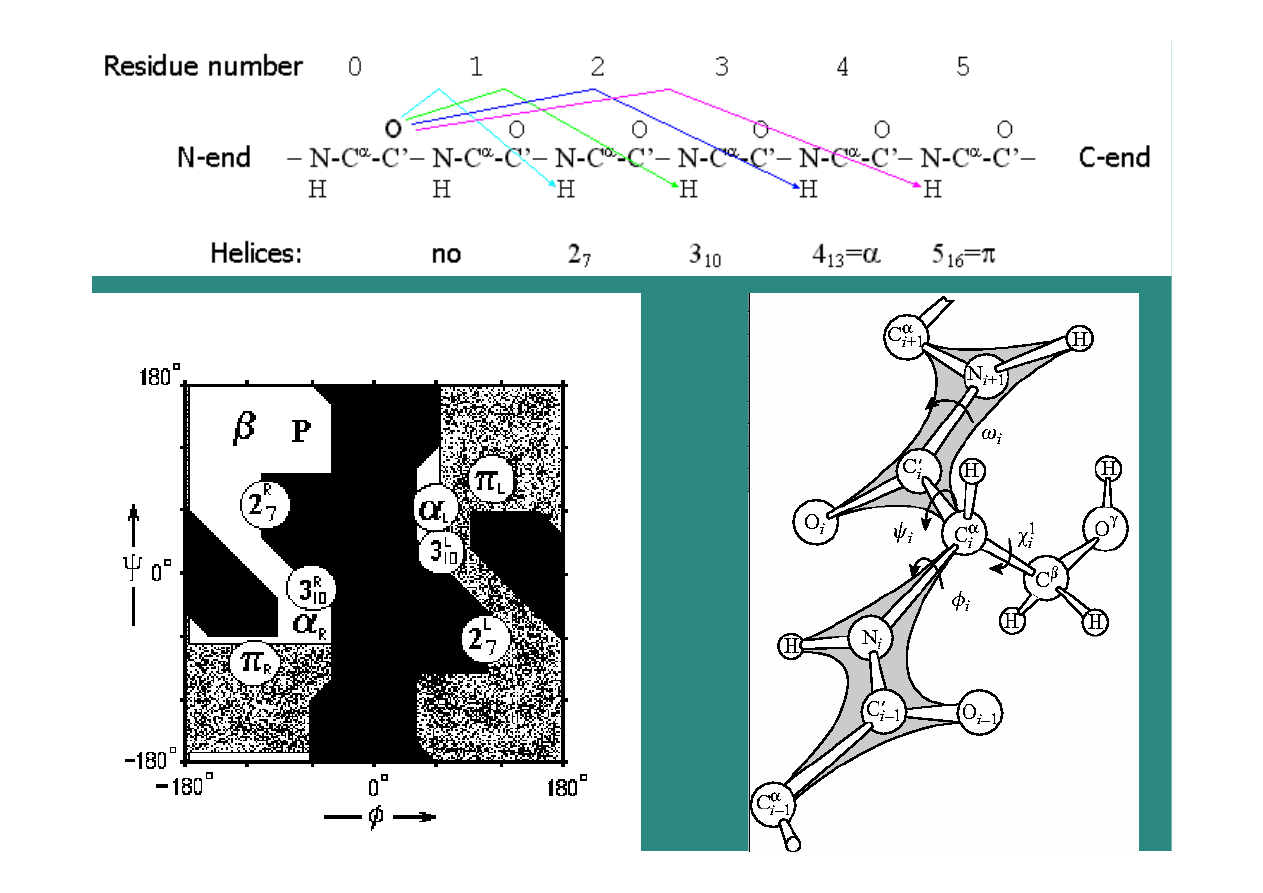
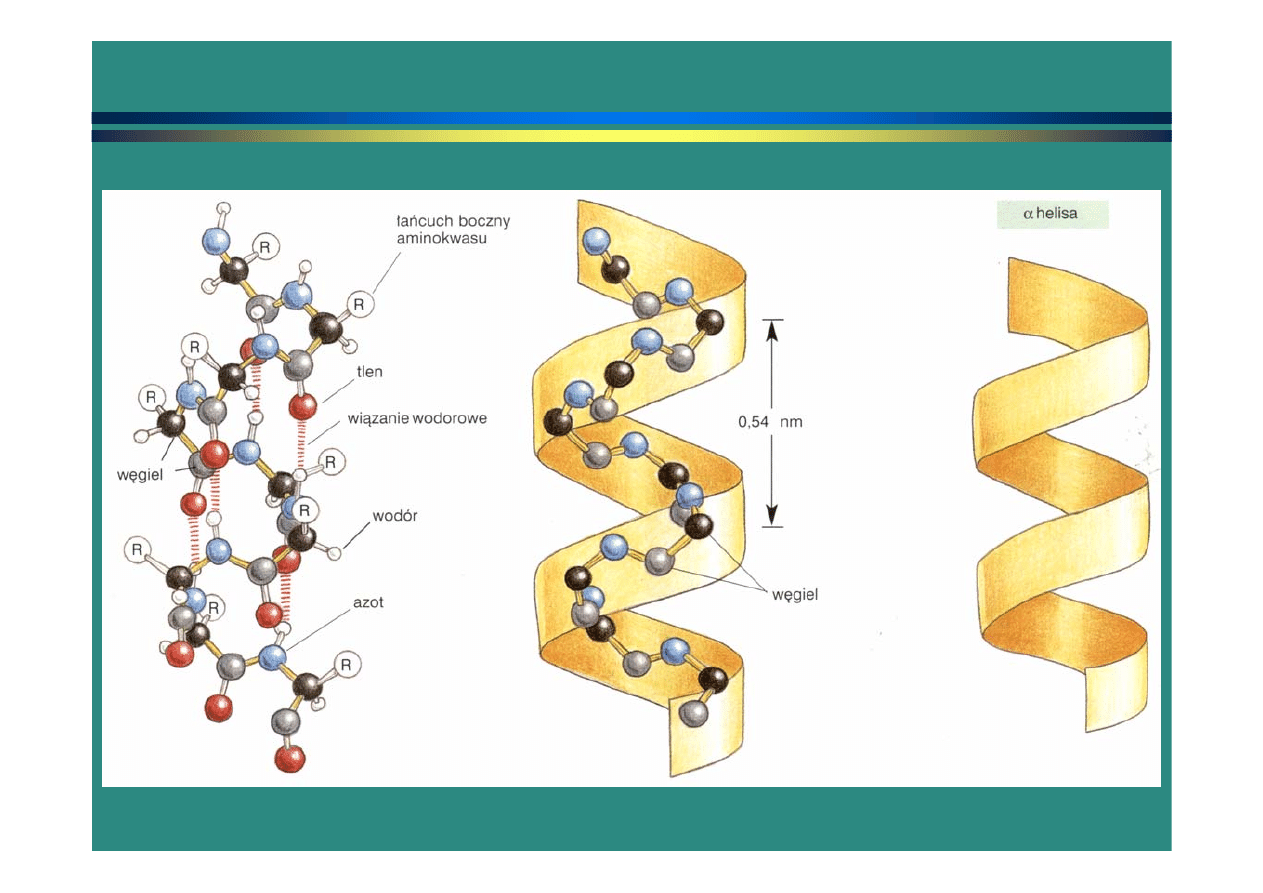
Struktury
drugorzędowe:
α
helisa
(
α
helix) -
stabilizowana
wiązaniami
wodorowymi
w helisie
Struktury
drugorzędowe
β
struktura
(
β
sheet) -
stabilizowana
wiązaniami
wodorowymi
z
inną
β
strukturą; układy
równoległe
i antyrównoległe
zwój
(coil, random coil) -
pozostałe struktury
zwrot
β
(
β
turn, reverse turn, harpin
bend)
pętla
(loop) -
łączy
inne
struktury
2009-01-08
20
Łańcuch
białkowy

2009-01-08
21
Plan wykładu
•
struktury
białek
–
dlaczego?
•
struktury
białek
–
geometria i fizyka
•
modyfikacje kowalencyjne
•
białka globularne a białka
transmembranowe i włókniste
•
regiony nieuporządkowane

2009-01-08
22
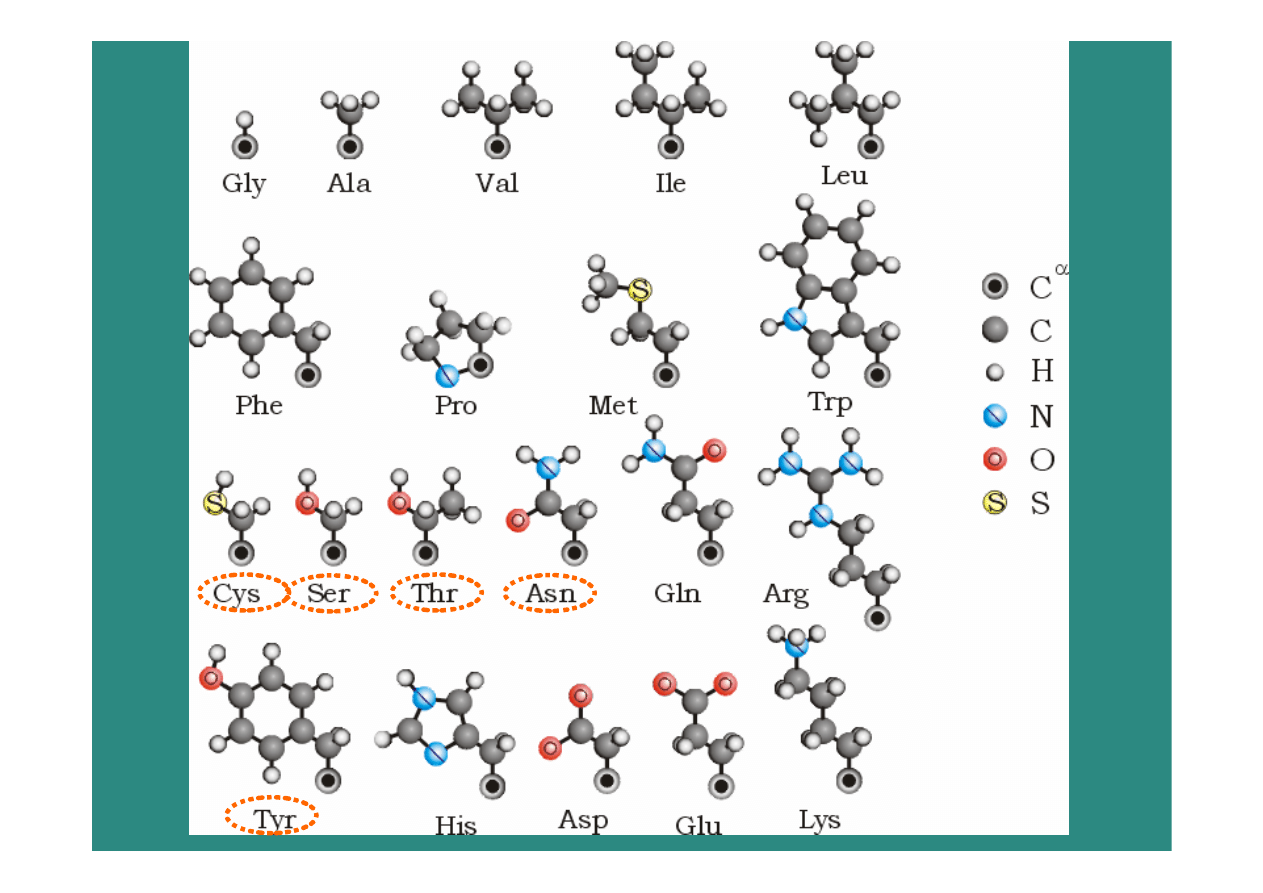
Modyfikacje posttranslacyjne
•
Cięcia łańcucha białkowego (proteoliza)
•
Glikozylacja, ...
•
Modyfikacje końców (acetylacja, ...)
•
Modyfikacje łańcuchów bocznych
(wiązania dwusiarczkowe, fosforylacja, ...)
•
Wiązanie kofaktorów, jonów, …
•
Efektywnie alfabet aminokwasowy
się
powiększa
20 N
2009-01-08
23
Side
Side
chains
chains
2009-01-08
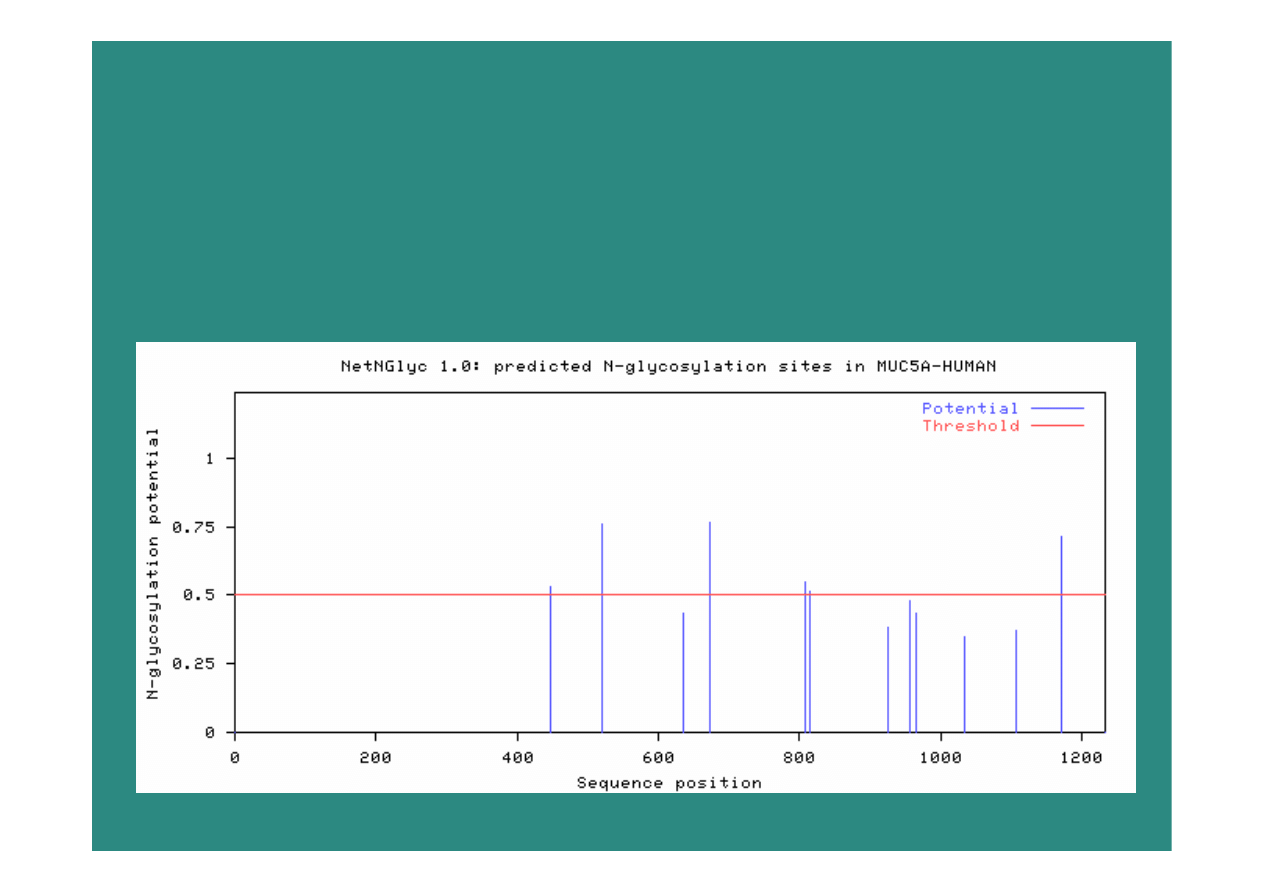
24
Przewidywanie glikozylacji
–
sieci neuronowe, www.cbs.dtu.dk
2009-01-08
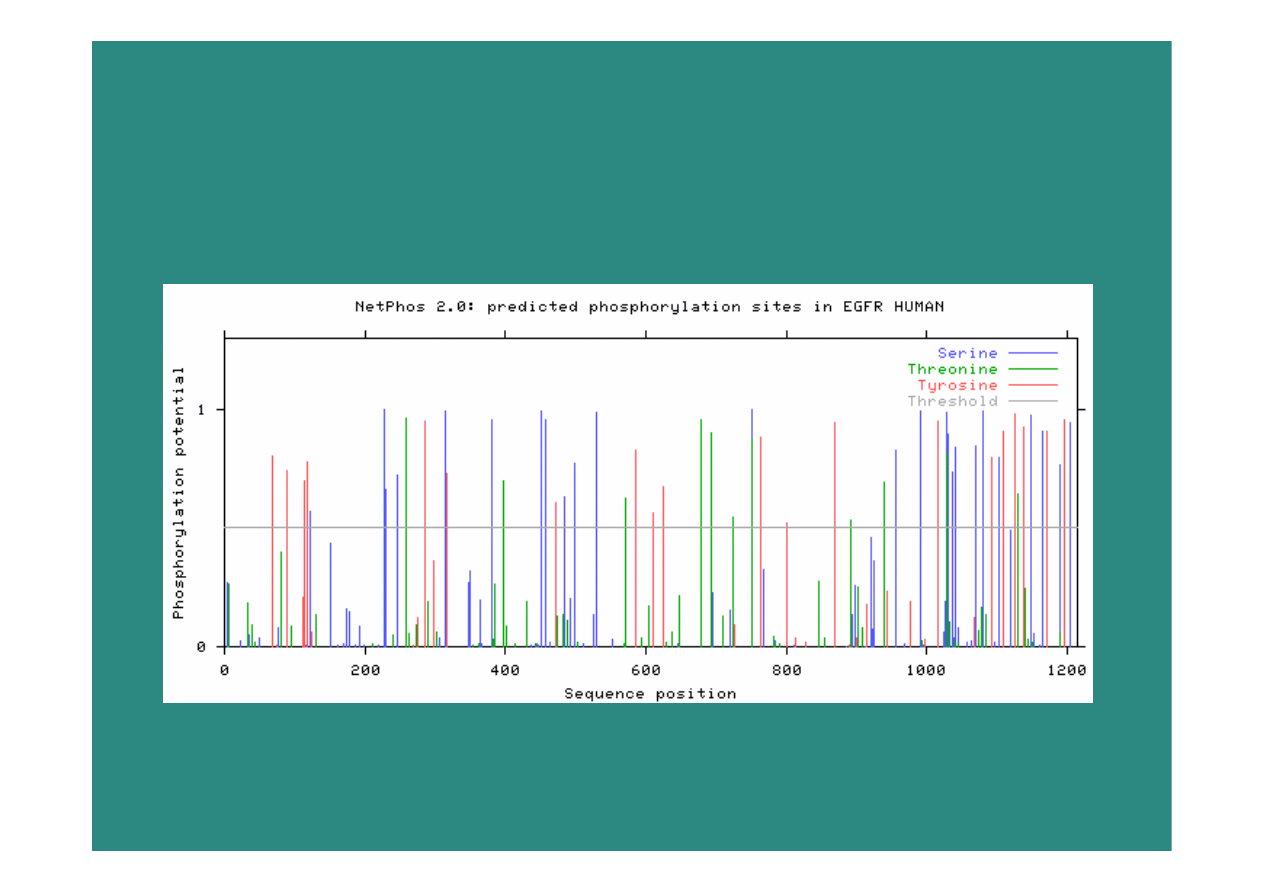
25
Przewidywanie fosforylacji
–
sieci neuronowe, www.cbs.dtu.dk
2009-01-08
26
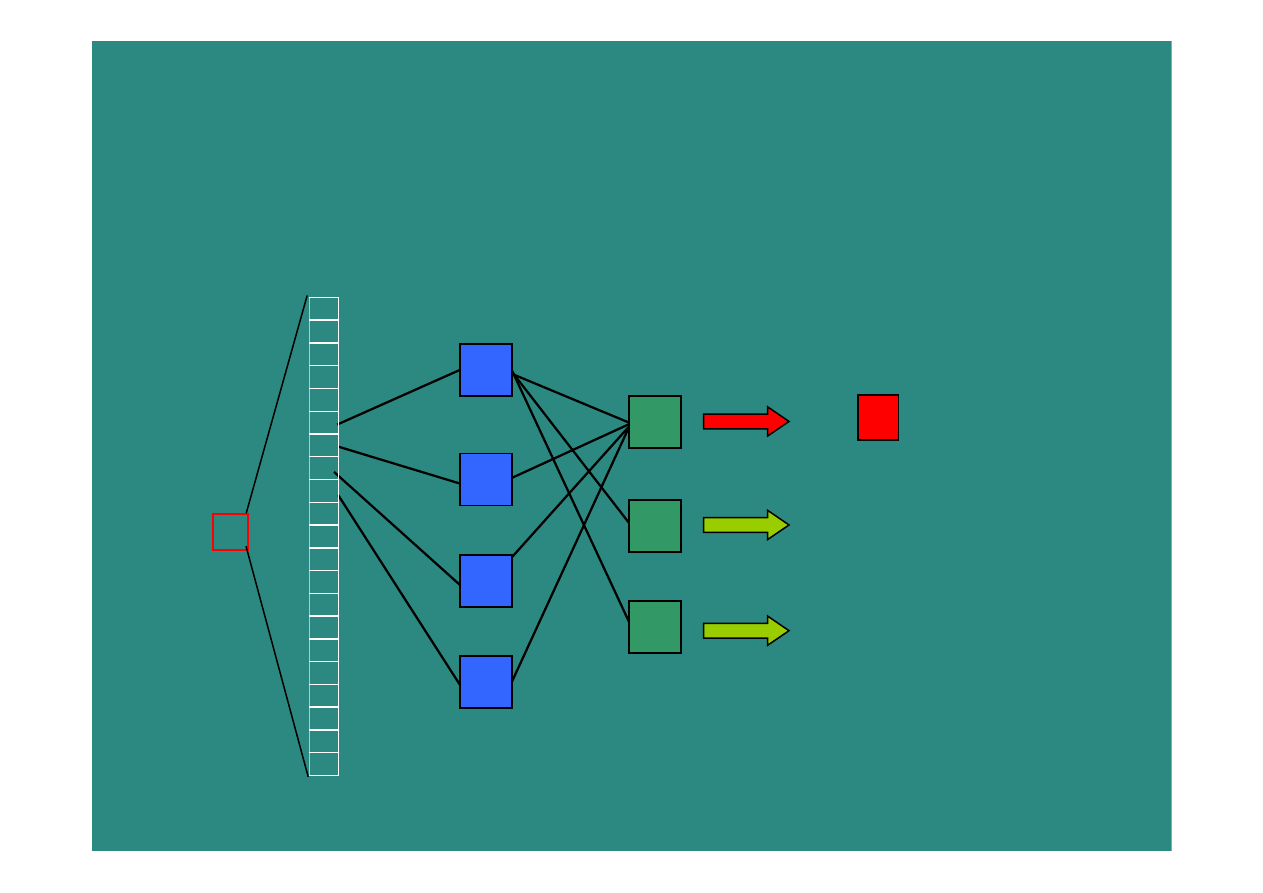
Sieci
neuronowe
-
sekwencja
jest analizowana
zachodzącymi
oknami
(13-17
aminokwasów); na
wejściu
podawana
jest sekwencja
w oknie; przewidywana
jest
struktura
dla
aminokwasu
centralnego; uwzględniane
są
oddziaływania
aminokwasów
na
siebie
przy
określaniu
struktury
-
analiza
w kontekście; sieć
jest
uczona
na
sekwencjach
o znanej
strukturze, podczas
uczenia
określane
są
wagi,
które
są
później
nadawane
sygnałom; przewiduje
struktury
2D i regiony
hydrofobowe
L
S
W
T
K
C
Y
A
V
S
G
A
P
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
α
β
coil
1
0
0
warstwa
ukryta
warstwa
wyjściowa
przewidywana
struktura
α
warstwa
wejściowa
-
1 jedn. wej. dla
każdego
aa
w oknie; informacje
o innych
aa, właściwości, profil
sekw
encj
a
w
ej
ści
owa
w
okni
e
2009-01-08
27
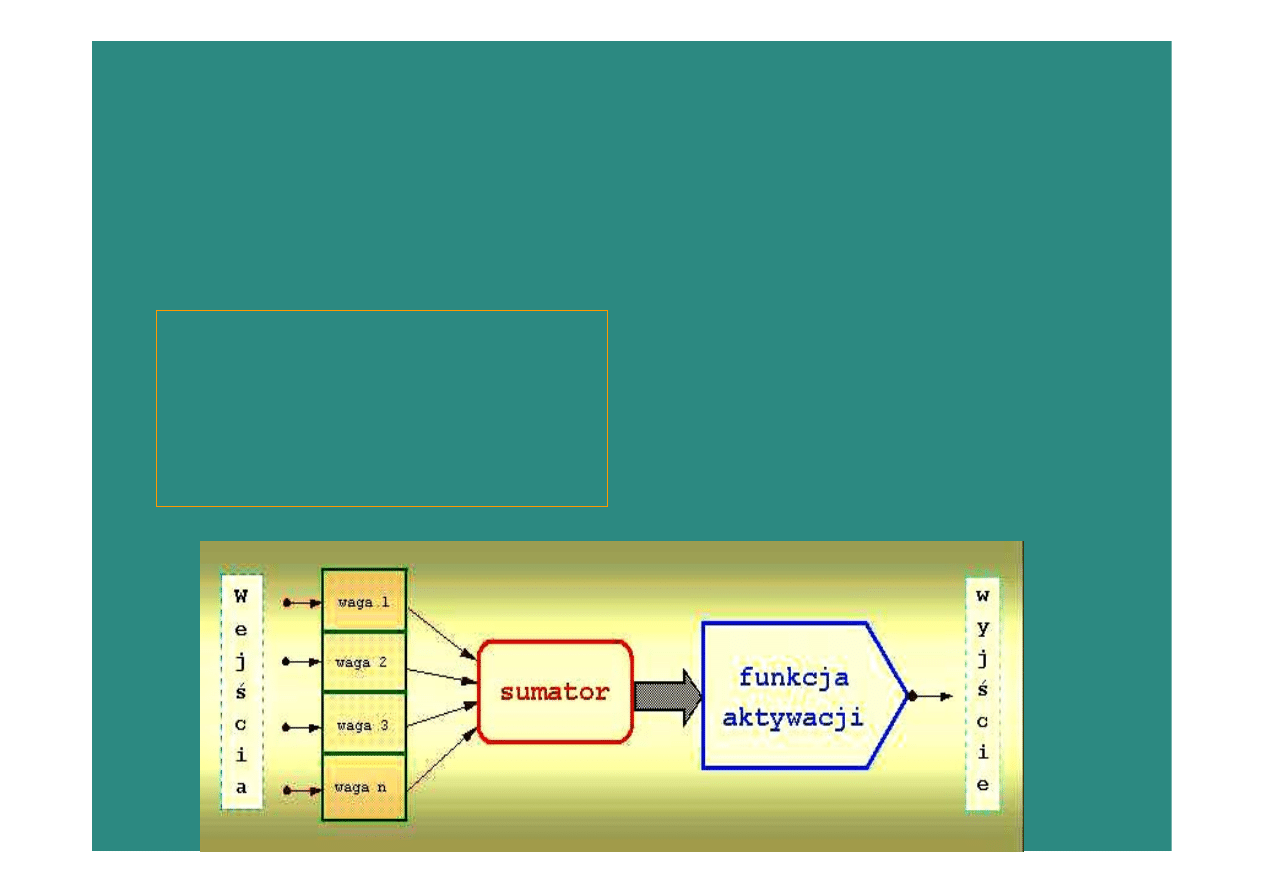
•
Istota działania
sztucznego neuronu:
Sumowanie sygnałów wejściowych z
odpowiednią
wagą
i poddanie sumy funkcji
aktywacji
)
(
1
j
N
j
ij
i
x
W
f
y
∑
=
=
Xj
–
sygnał
wejściowy
Wij –
współczynniki wagowe
–
wagi synaptyczne przy
ujemnych wagach neuron
przekazuje sygnał
gaszący,
przy dodatnich -
pobudzający

2009-01-08
28
Modyfikacje lokalne
a długozasięgowe
•
Przewidywanie oddziaływań
długozasięgowych
znacznie trudniejsze
•
Parowanie struktur beta
•
Parowanie cystein
w mostkach
dwusiarczkowych

2009-01-08
29
Modyfikacje sekwencyjnie
specyficzne a niespecyficzne
•
Rzadsze modyfikacje,
np. glutationylacja
bądź
nitrozylacja
cystein,
mogą
być
trudniejsze do przewidzenia
na podstawie lokalnej sekwencji

2009-01-08
30
Plan wykładu
•
struktury
białek
–
dlaczego?
•
struktury
białek
–
geometria i fizyka
•
modyfikacje kowalencyjne
•
białka globularne a białka
transmembranowe i włókniste
•
regiony nieuporządkowane

2009-01-08
31
Białka
globularne
Białka włókniste
Białka
transmembranowe

2009-01-08
32
Białka wielodomenowe
•
Znaczna część
białek u eukariontów
to białka wielodomenowe, niekiedy
zawierające regiony transmembranowe
oraz domeny globularne

2009-01-08
33
Białka transmembranowe
•
Kanały jonowe
•
Transportery
•
Receptory (7TM, RTK, …)
•
Proteazy
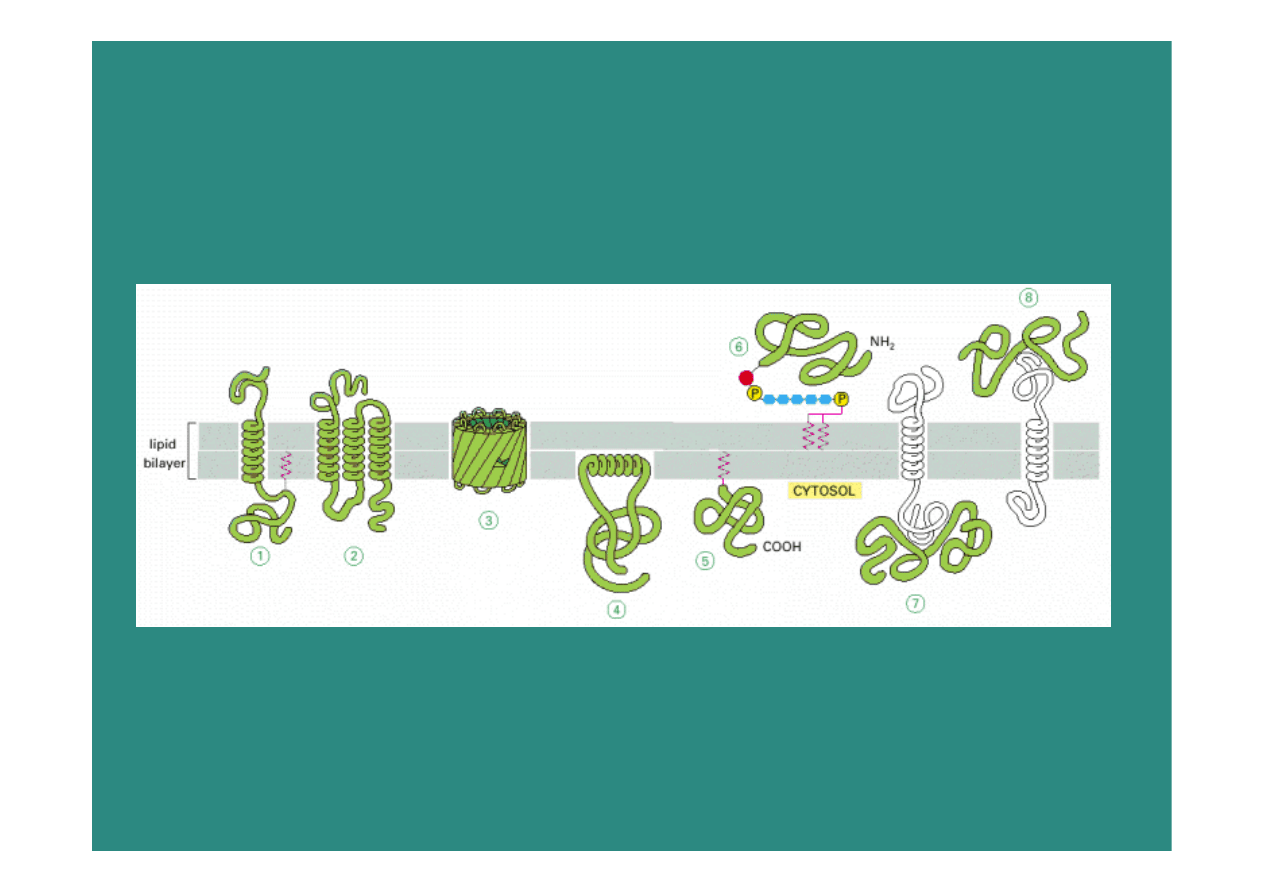
2009-01-08
34
Białka transmembranowe
2009-01-08
35
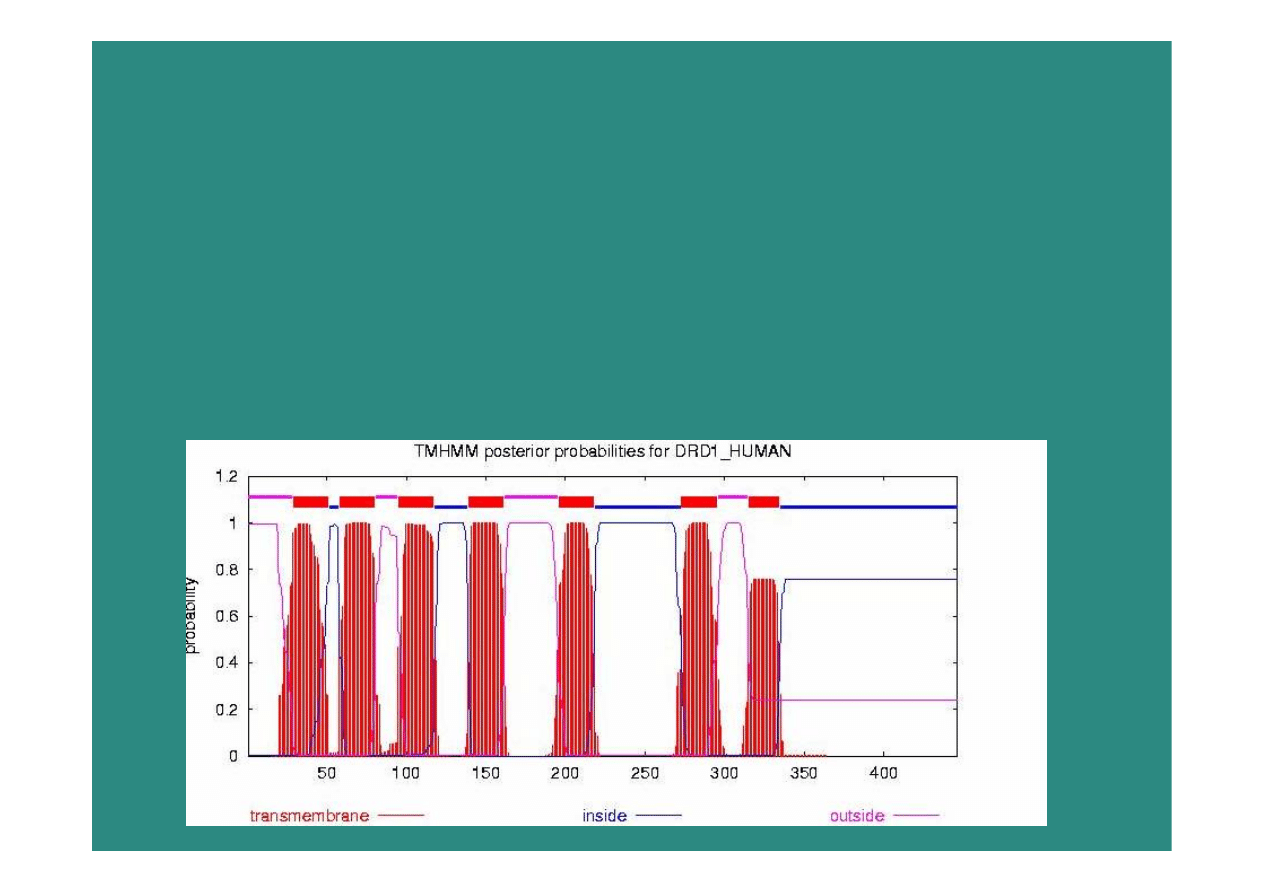
przewidywanie topologii
transmembranowej
ludzki receptor dopaminy
–
HMM, www.cbs.dtu.dk
2009-01-08
36
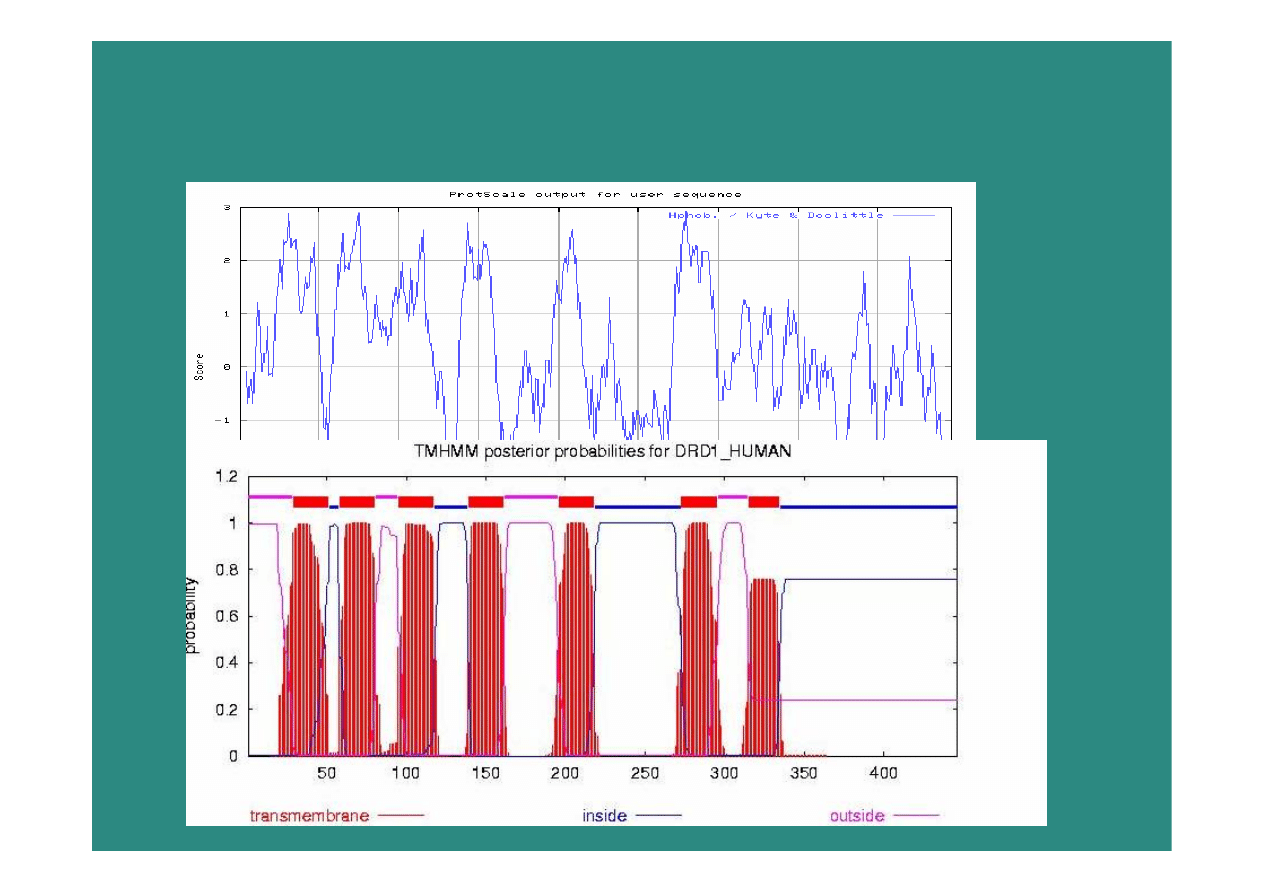
przewidywanie topologii
transmembranowej
ludzki receptor dopaminy
–
HMM, www.cbs.dtu.dk
2009-01-08
37
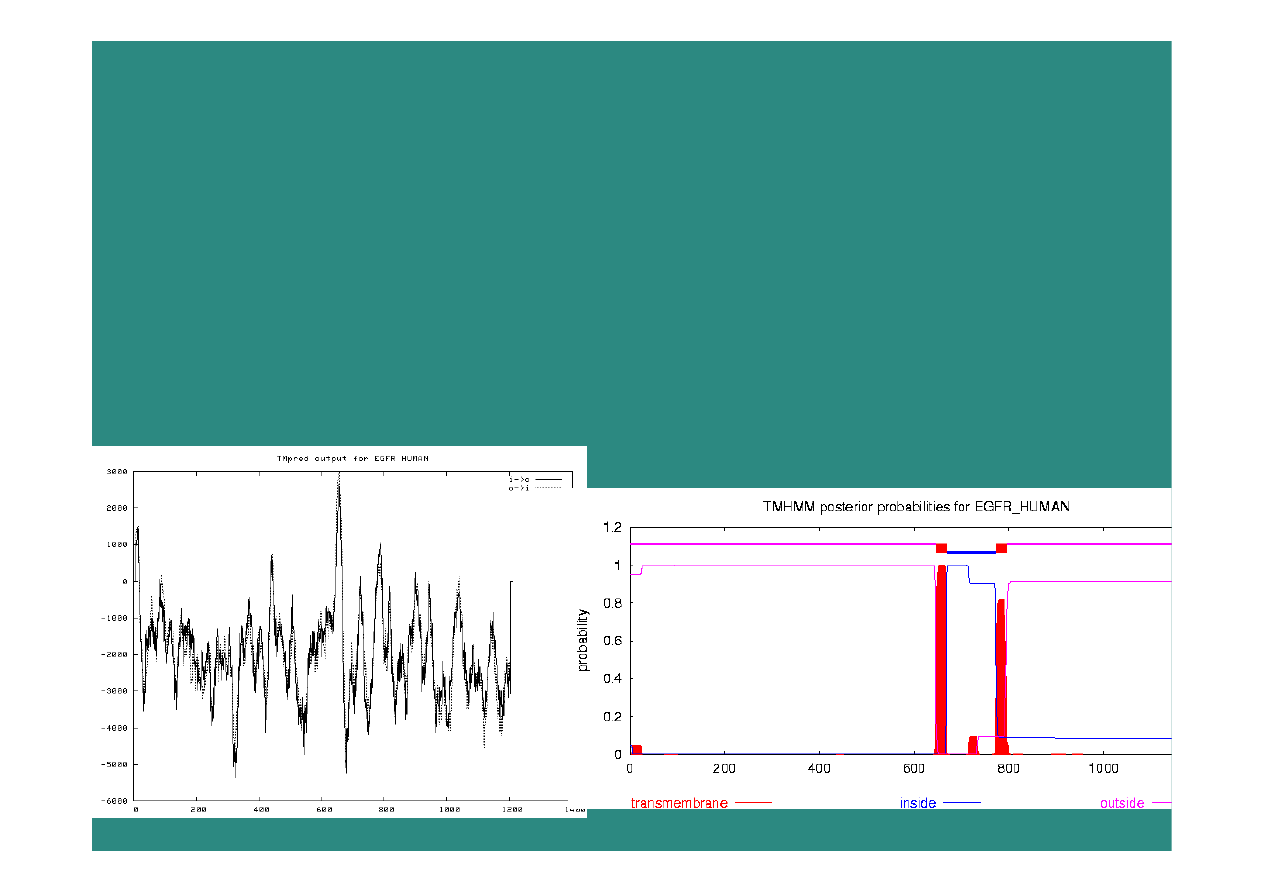
Pamiętajmy o błędach!
2009-01-08
38
Pamiętajmy o błędach!

2009-01-08
39
Plan wykładu
•
struktury
białek
–
dlaczego?
•
struktury
białek
–
geometria i fizyka
•
modyfikacje kowalencyjne
•
białka globularne a białka
transmembranowe i włókniste
•
regiony nieuporządkowane

2009-01-08
40
Regiony nieuporządkowane –
disordered
regions
•
trudna definicja
•
trudne do przewidzenia
•
nie zawsze tożsame z pętlami
•
nie zawsze tożsame z regionami o niskiej
specyficzności
•
ważne biologicznie
•
sprzężenie zwijania białka i wiązania
•
duże znaczenie praktyczne

2009-01-08
41
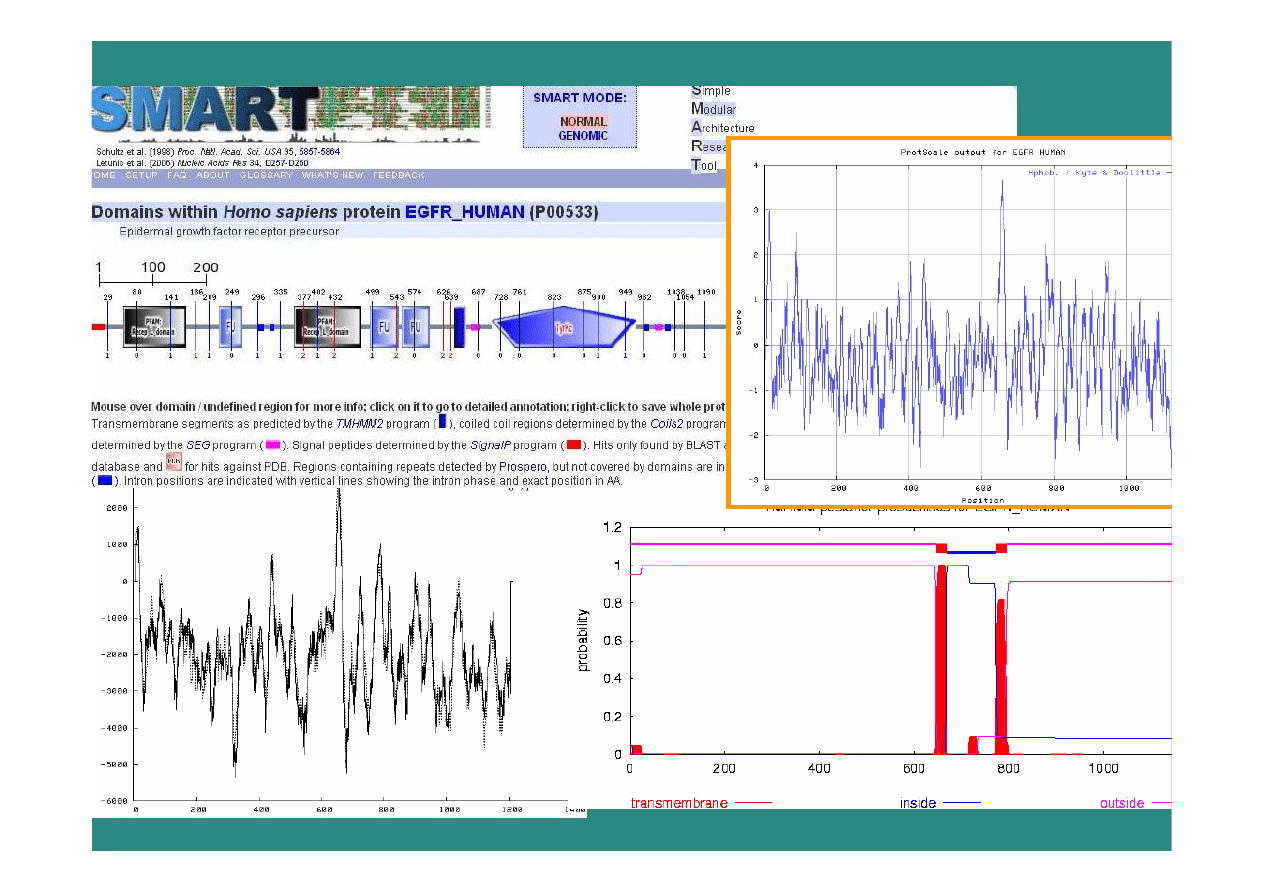
Regiony nieuporządkowane –
gdzie?
•
pętle / zwoje
•
”gorące pętle”
(wg czynników temperatury ze struktur
krystalograficznych)
•
obszary o brakujących współrzędnych
(w strukturach krystalograficznych i NMR)
przewidywanie –
np. sieci neuronowe
2009-01-08
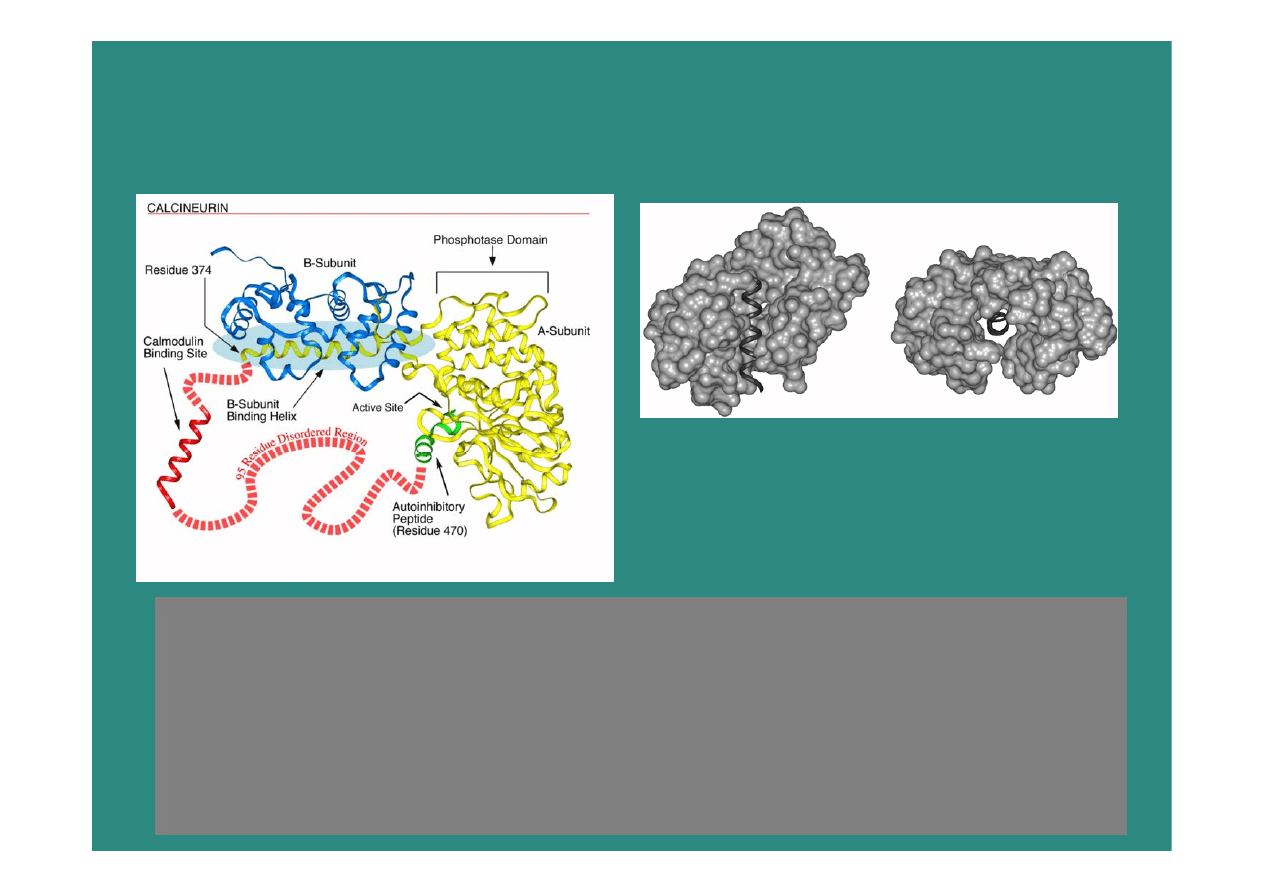
42
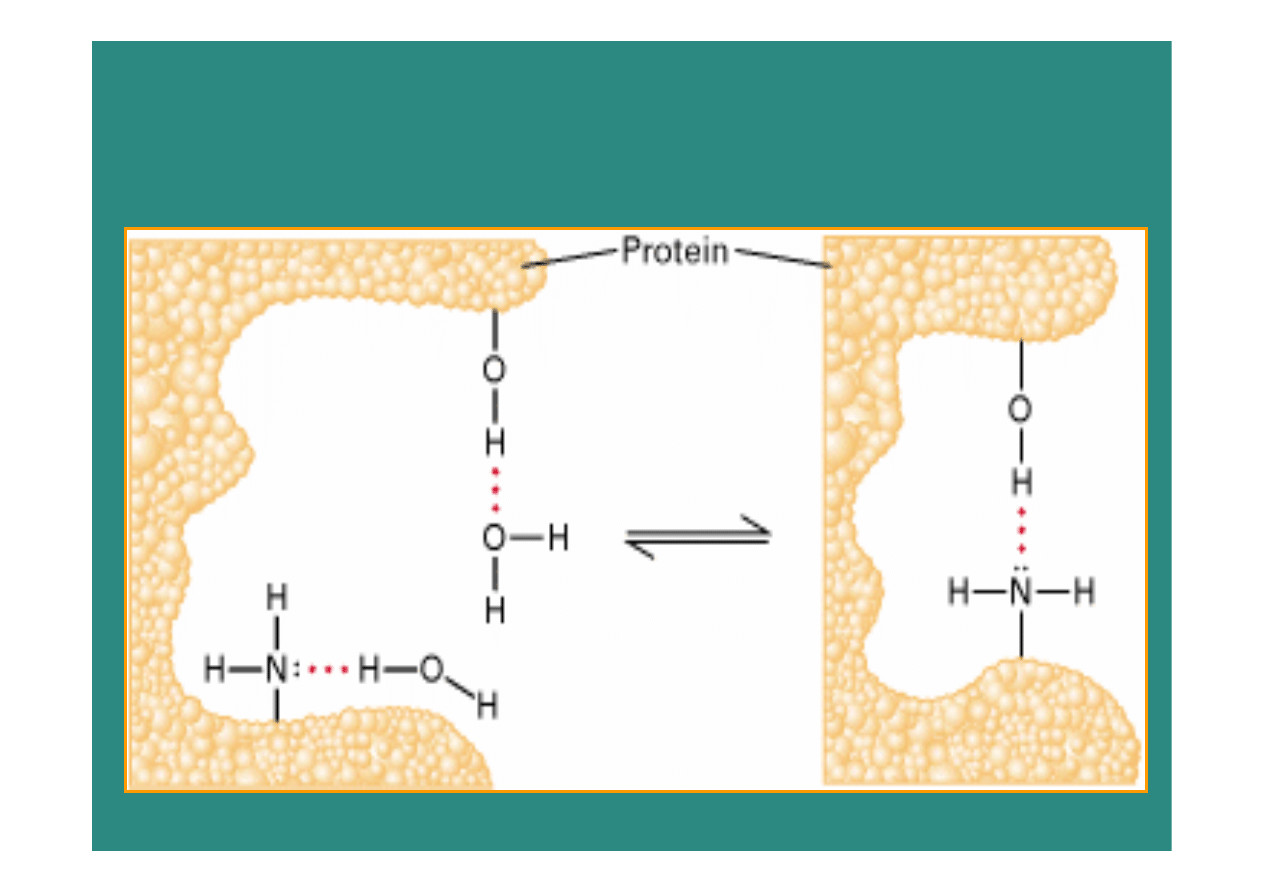
Kalcyneuryna
•
extremely sensitive to protease digestion:
a disordered ensemble;
•
confirmed in X-ray diffraction structure by missing coordinates
•
disorder likely to be essential to provide calmodulin
(right) with space needed
to completely surround its target helix
...the existence and commonness of proteins with intrinsic disorder
call for
a reassessment of the structure-function paradigm...
(Wright and Dyson )
2009-01-08
43
Nieporządek –
przewidywarka
komercyjna
2009-01-08
44
Nieporządek –
przewidywarka
http://dis.embl.de
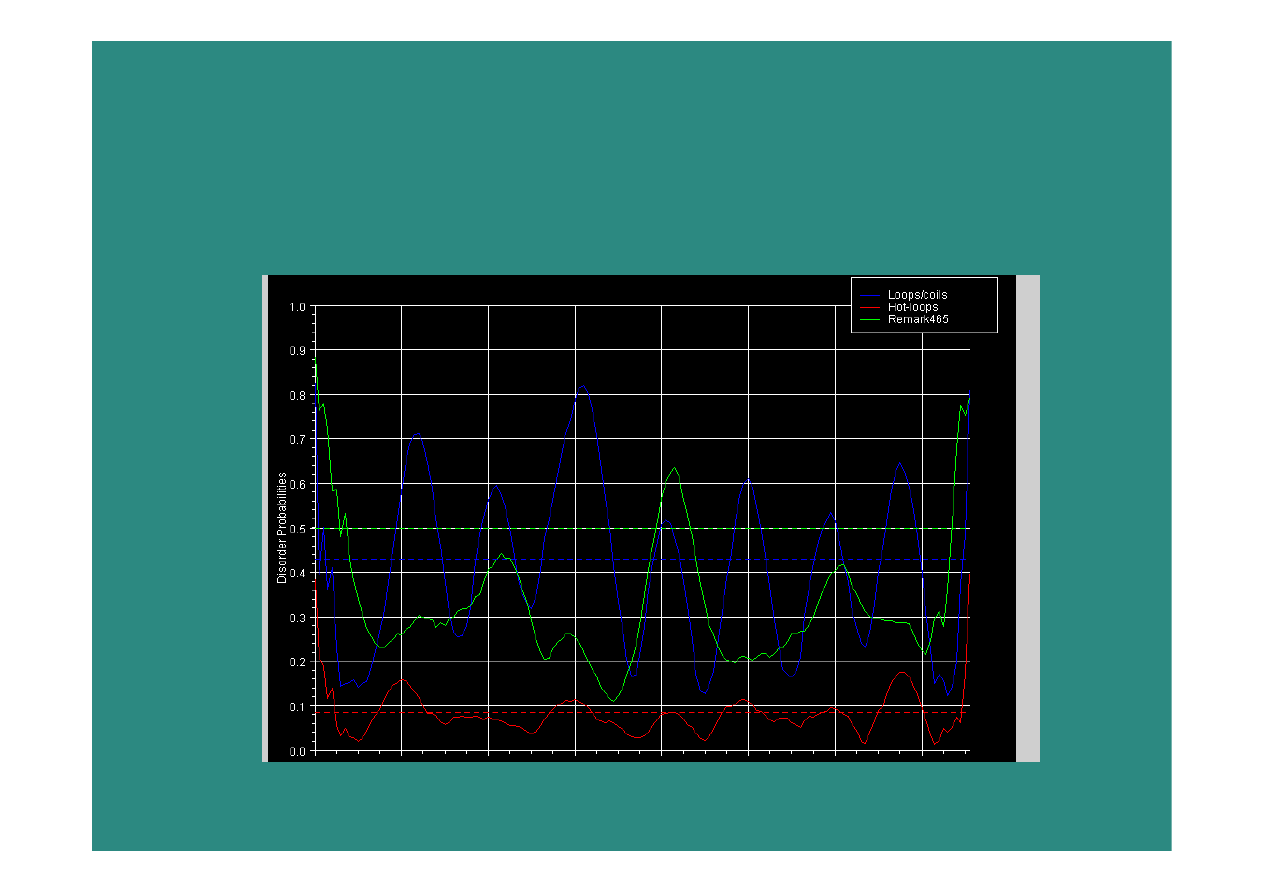
2009-01-08
45
Nieporządek w kalmodulinie

2009-01-08
46
Granice dokładności
przewidywań
strukturalnych
•
Ograniczone zestawy danych do „uczenia”
algorytmów
•
Niejednoznaczność
definicji przedmiotu
przewidywań
–
np. struktura II-rzędowa
•
Warto łączyć
różne przewidywania –
pamiętać
o kontekście. Np. fosforylacja-
wewnątrz komórki; glikozylacja
–
na
zewnątrz
•
Interpretacja
Document Outline
- Bioinformatyka – wykład 8 2.XII.2008 białkowa bioinformatyka strukturalna
- Lecture outline, Dec. 6th
- Plan wykładu
- Struktury białek – dlaczego warto je znać i rozumieć
- Slide Number 5
- Plan wykładu
- Slide Number 7
- Slide Number 8
- Slide Number 9
- Slide Number 10
- Wykres Ramachandrana dozwolone obszary kątów f, y
- Slide Number 12
- Zapis struktury białka:
- Wiązania wodorowe
- Slide Number 15
- Wiązania wodorowe
- Slide Number 17
- Slide Number 18
- Slide Number 19
- Slide Number 20
- Plan wykładu
- Modyfikacje posttranslacyjne
- Slide Number 23
- Przewidywanie glikozylacji – sieci neuronowe, www.cbs.dtu.dk
- Przewidywanie fosforylacji – sieci neuronowe, www.cbs.dtu.dk
- Slide Number 26
- Slide Number 27
- Modyfikacje lokalne a długozasięgowe
- Modyfikacje sekwencyjnie specyficzne a niespecyficzne
- Plan wykładu
- Slide Number 31
- Białka wielodomenowe
- Białka transmembranowe
- Slide Number 34
- przewidywanie topologii transmembranowej ludzki receptor dopaminy – HMM, www.cbs.dtu.dk
- przewidywanie topologii transmembranowej ludzki receptor dopaminy – HMM, www.cbs.dtu.dk
- Pamiętajmy o błędach!
- Pamiętajmy o błędach!
- Plan wykładu
- Regiony nieuporządkowane – disordered regions
- Regiony nieuporządkowane – gdzie?
- Kalcyneuryna
- Nieporządek – przewidywarka komercyjna
- Nieporządek – przewidywarka http://dis.embl.de
- Nieporządek w kalmodulinie
- Granice dokładności przewidywań strukturalnych
Wyszukiwarka
Podobne podstrony:
bioinformatyka w13 2008 9 web
bioinformatyka w2 2008 web
bioinformatyka w9 2008 web
bioinformatyka w6 2008 web
bioinformatyka w11 2008 web
bioinformatyka w4 2008 web
bioinformatyka w10 2008 web
bioinformatyka w12 2008 9 web
bioinformatyka w3 2008 web
bioinformatyka w7 2008 web
bioinformatyka w1 2008 web
bioinformatyka w5 2008 web
bioinformatyka w13 2008 9 web
bioinformatyka w2 2008 web
bioinformatyka w9 2008 web
więcej podobnych podstron