
2009-01-09
1
Bioinformatyka
– wykład 9, 9.XII.2008
białkowa
bioinformatyka strukturalna
–
c.d.
krzysztof_pawlowski@sggw.pl

2009-01-09
2
Plan wykładu
•
sposoby przedstawienia struktur
białkowych
•
powierzchnia białka
•
programy do obrazowania struktur
białkowych.
•
analiza i porównywanie struktur białek
2009-01-09
3
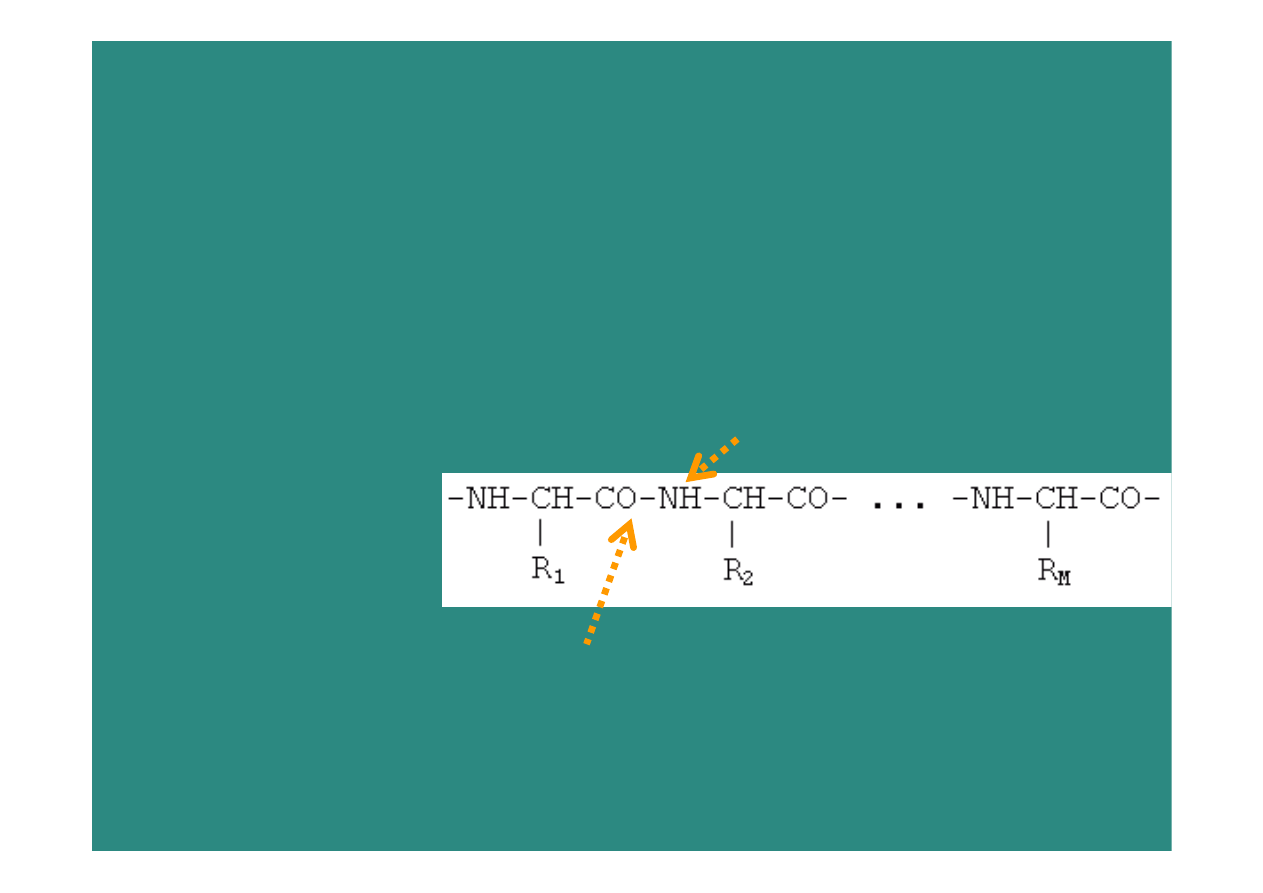
Wiązania wodorowe...
... a struktury drugorzędowe
Łańcuch
białkowy
H-bond
donor
H-bond
acceptor
2009-01-09
4
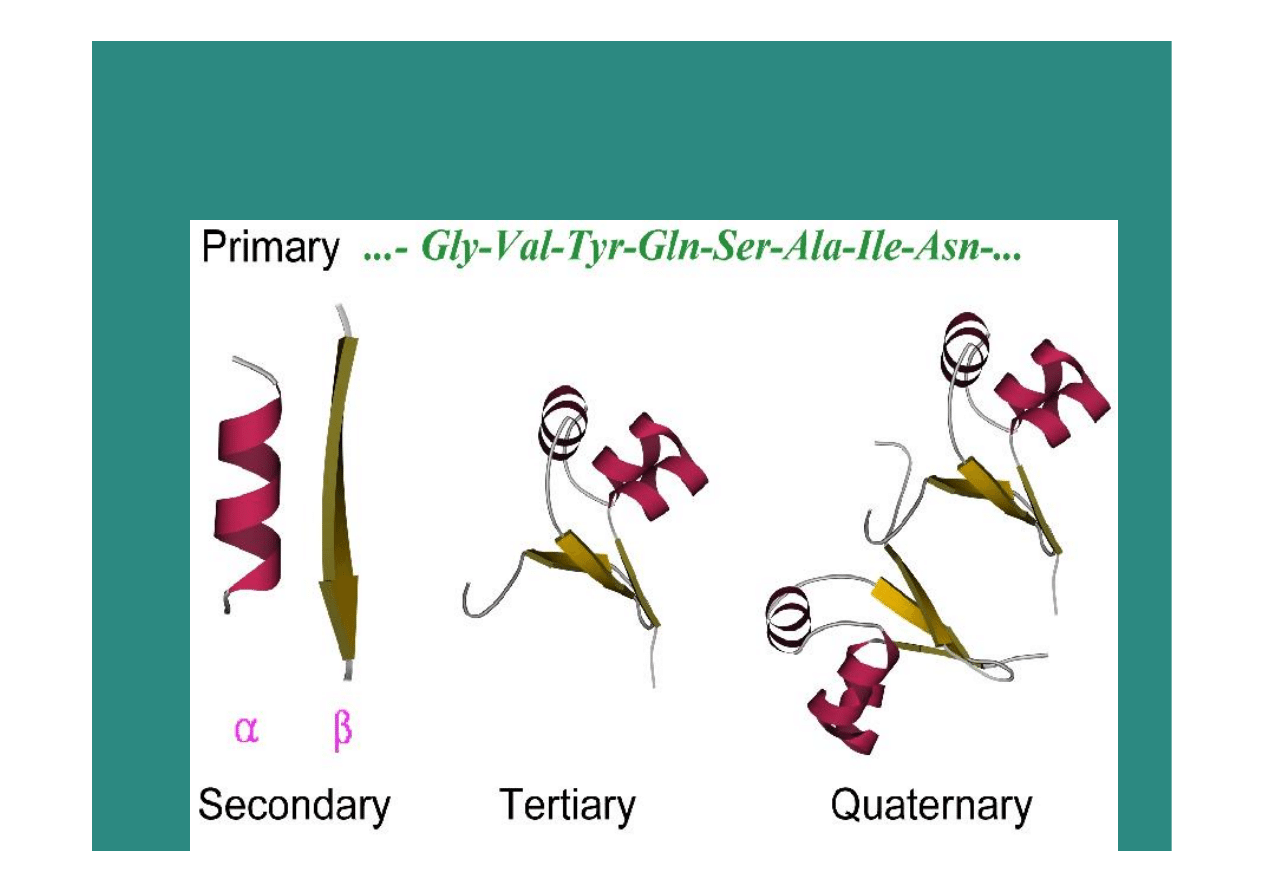
Poziomy organizacji
struktury białka

2009-01-09
5
Granice dokładności
przewidywań
strukturalnych
•
Ograniczone zestawy danych do „uczenia”
algorytmów
•
Niejednoznaczność
definicji przedmiotu
przewidywań
–
np. struktura II-rzędowa
•
Warto łączyć
różne przewidywania –
pamiętać
o kontekście. Np. fosforylacja-
wewnątrz komórki; glikozylacja
–
na
zewnątrz
•
Interpretacja

2009-01-09
6
Plan wykładu
•
sposoby przedstawienia struktur
białkowych
•
powierzchnia białka
•
programy do obrazowania struktur
białkowych.
•
analiza i porównywanie struktur białek

2009-01-09
7
Struktura białka –
jak wygląda?
a
all-atom representation
a
ribbon representation
a
surface representation
Jak warto sobie wyobrażać
strukturę
białka?

2009-01-09
8
Kształt –
fold. C
α
Struktura łańcucha głównego

2009-01-09
9
Kształt –
fold.
Wiązania wodorowe łańcucha głównego

2009-01-09
10
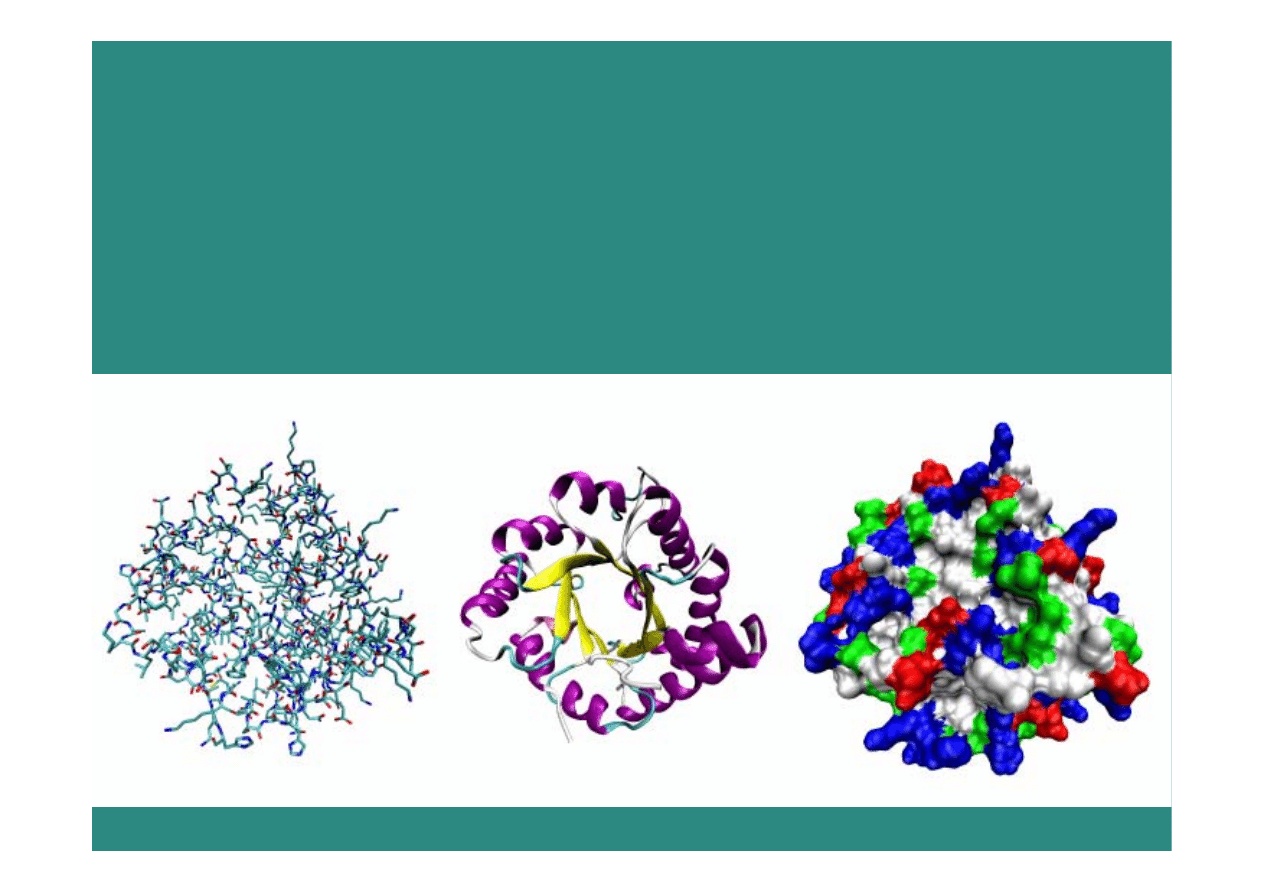
Szczegóły atomowe, np. miejsca aktywne,
dokowanie ligandów
2009-01-09
11
Wybór przedstawienia białka
zależy od zadawanych pytań

2009-01-09
12
Plan wykładu
•
sposoby przedstawienia struktur
białkowych
•
powierzchnia białka
•
programy do obrazowania struktur
białkowych
•
analiza i porównywanie struktur białek
2009-01-09
13
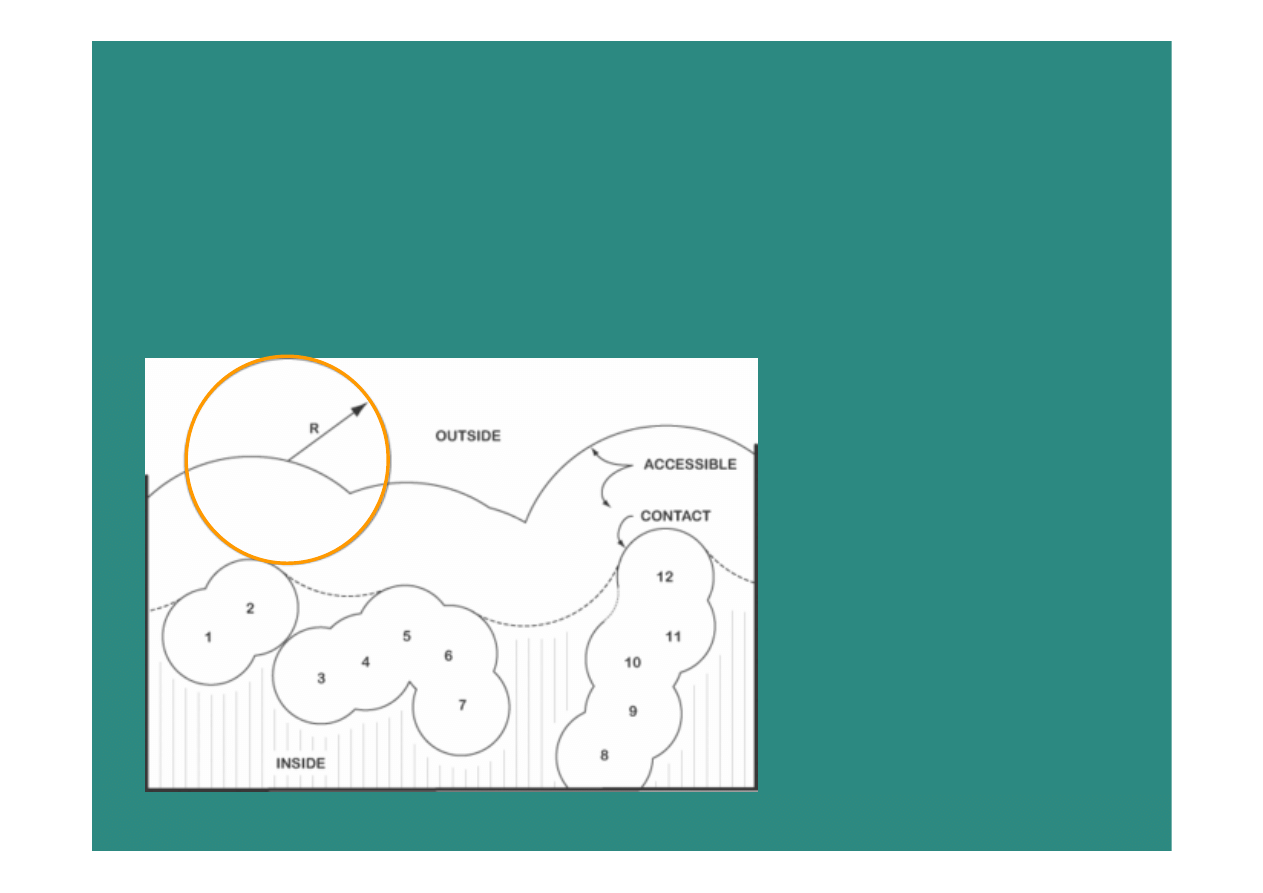
Powierzchnia białka
•
Powierzchnia molekularna
(Connolly‘ego)
•
Powierzchnia dostępna dla rozpuszczalnika
(Richardsa)
protein
solvent

2009-01-09
14
Plan wykładu
•
sposoby przedstawienia struktur
białkowych
•
powierzchnia białka
•
programy do obrazowania struktur
białkowych.
•
analiza i porównywanie struktur białek

2009-01-09
15
Przykładowe programy do
obrazowania struktur białkowych
•
Rasmol
-
klasyka
•
PDB website> Jmol, WebMol
•
Swiss-PDBviewer
•
Komercyjne –
Sybyl
(Tripos), InsightII
(Accelrys), Schrödinger
•
dziesiątki innych...

2009-01-09
16
Istotne cechy programów
do obrazowania struktur
•
Szybkość
operacji –
np. obroty
•
Jakość
obrazu –
np. powierzchnie
•
Operacje na wielu strukturach
•
Operacje na sekwencjach
•
Połączenie z bazami danych
•
Połączenie z programami do modelowania
struktur

2009-01-09
17
Plan wykładu
•
sposoby przedstawienia struktur
białkowych
•
powierzchnia białka
•
programy do obrazowania struktur
białkowych
•
analiza i porównywanie struktur białek
2009-01-09
18
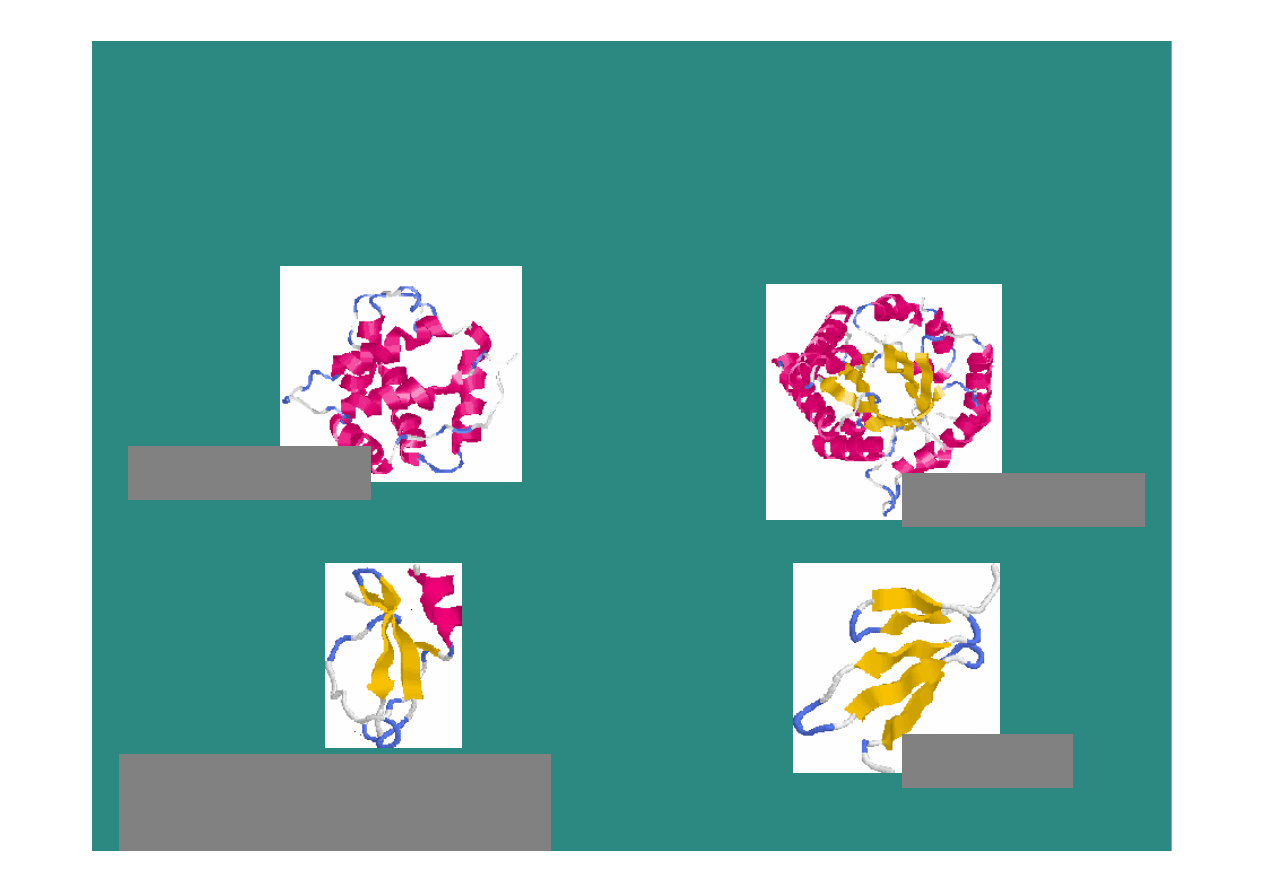
Klasy struktur białkowych
few secondary
structure elements
all-alpha
alpha/beta
all-beta

2009-01-09
19
Popularne systemy klasyfikacji
struktur białkowych
•
SCOP -
visual
inspection
& automated
methods, A. Murzin
http://scop.mrc-lmb.cam.ac.uk/scop/
•
CATH -
semi-automatic,
http://www.cathdb.info
•
FSSP –
automated
(DALI)
http://ekhidna.biocenter.helsinki.fi/dali

2009-01-09
20
Porównywanie struktur
•
Porównanie dwóch modeli tej samej
struktury
•
Porównanie dwóch bliskich homologów
•
Porównanie białek o podobieństwie
odległym (w najlepszym razie)
a)
dopasowanie sekwencji oczywiste
b)
dopasowanie sekwencji niejednoznaczne
2009-01-09
21
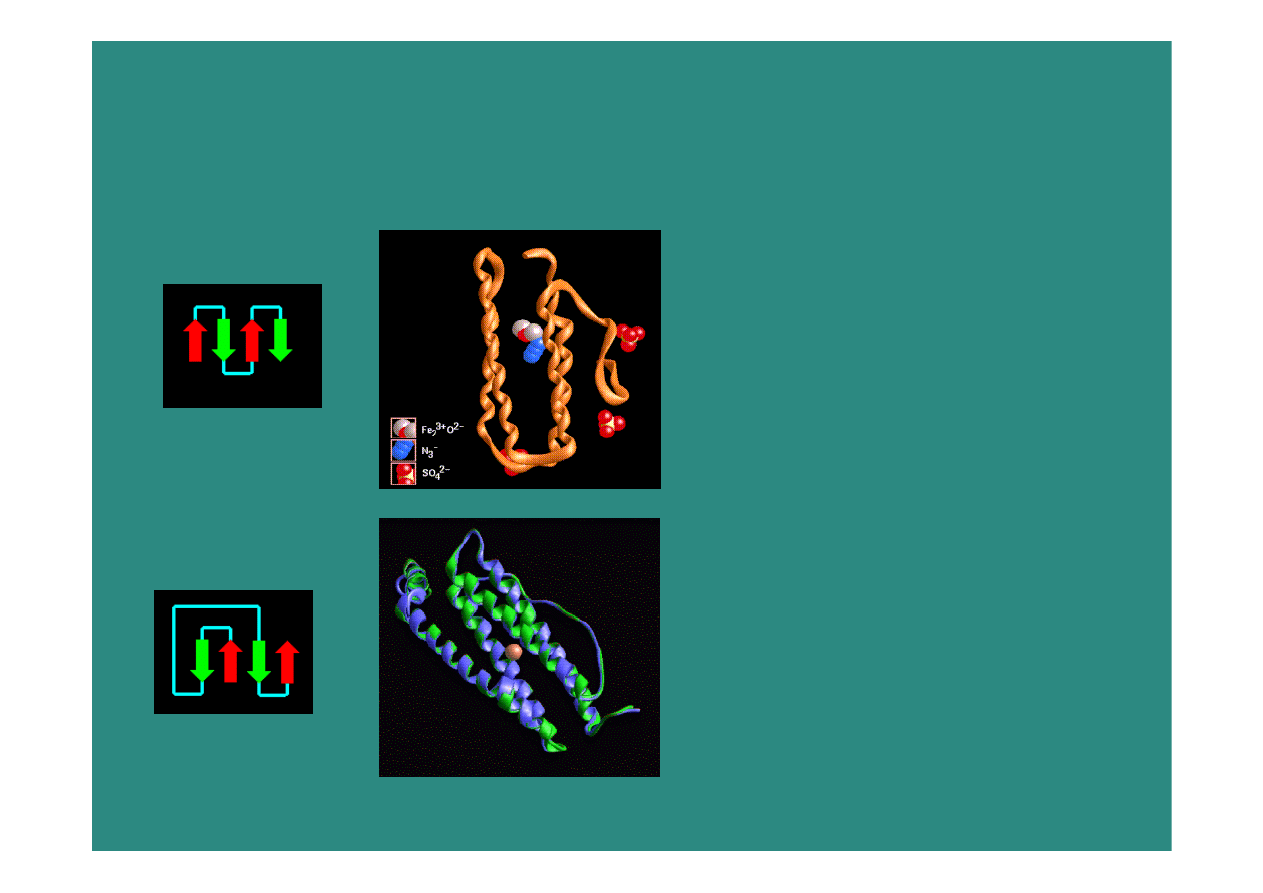
Kształt (fold) a topologia
myohemeyrthrin
ferritin

2009-01-09
22
Dopasowanie struktur białkowych
Dopasowanie (alignment) strukturalne
-
struktura 3D
domeny jednego białka jest nakładana na
strukturę
domeny drugiego białka tak, aby średnia odległość
między odpowiednimi
atomami struktur była możliwie jak
najmniejsza;
•
Podobieństwo strukturalne mogą
wykazywać
białka, które
nie wykazują
podobieństwa sekwencji.
•
Podobieństwo strukturalne może, ale nie musi świadczyć
o związkach ewolucyjnych.
•
Podobieństwo strukturalne może, ale nie musi świadczyć
o podobieństwie funkcji
2009-01-09
23
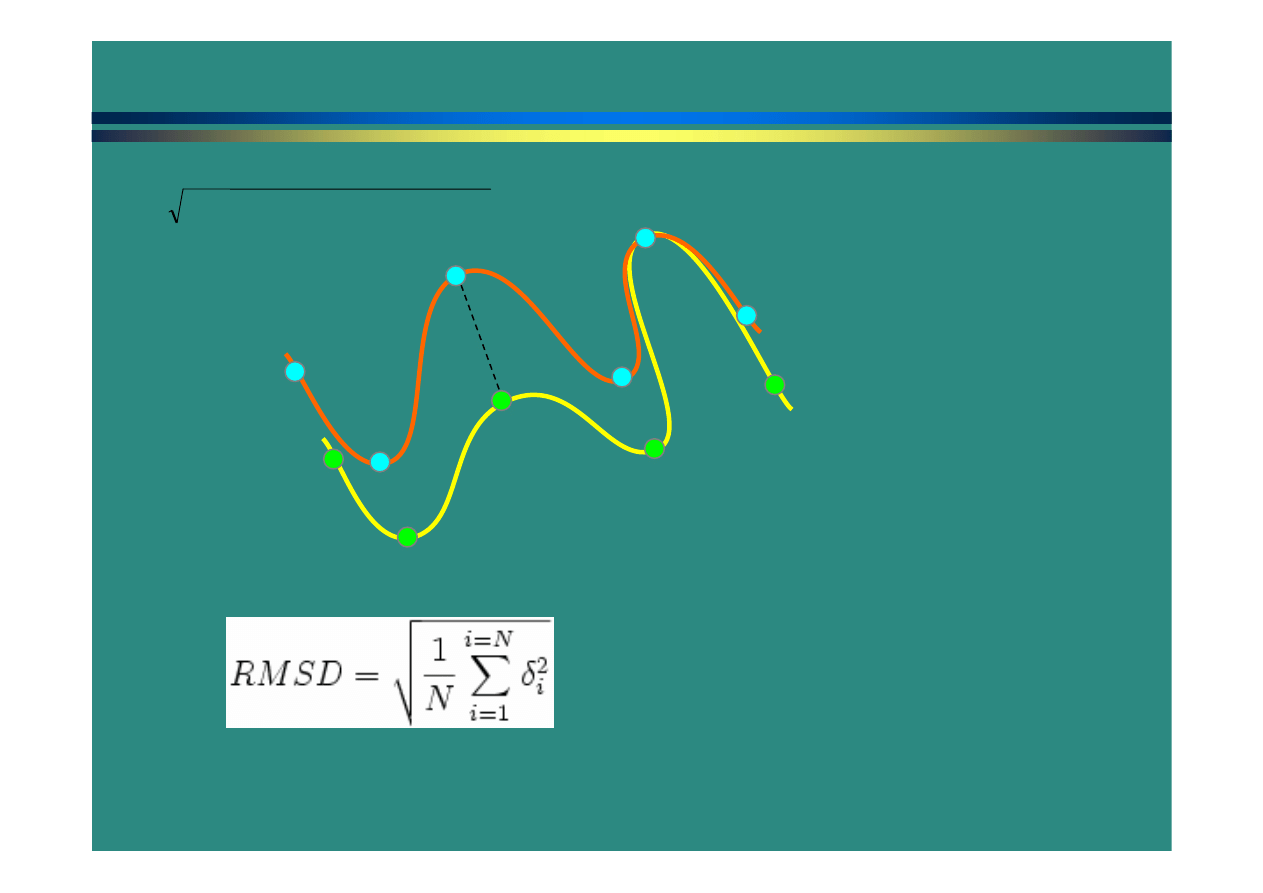
Dopasowanie strukturalne (alignment)
d
iJ
=
odległość:
(x
i
- x
J
)
2
+ (y
i
- y
J
)
2
+ (z
i
- z
J
)
2
b
c
d
e
f
α
β
γ
δ
ε
ζ
Root mean square deviation
-
zwykle C
α
2009-01-09
24
podobnie jak identyczność
sekwencji,
RMSD ma sens tylko dla określonego
dopasowania i regionu sekwencji
2009-01-09
25
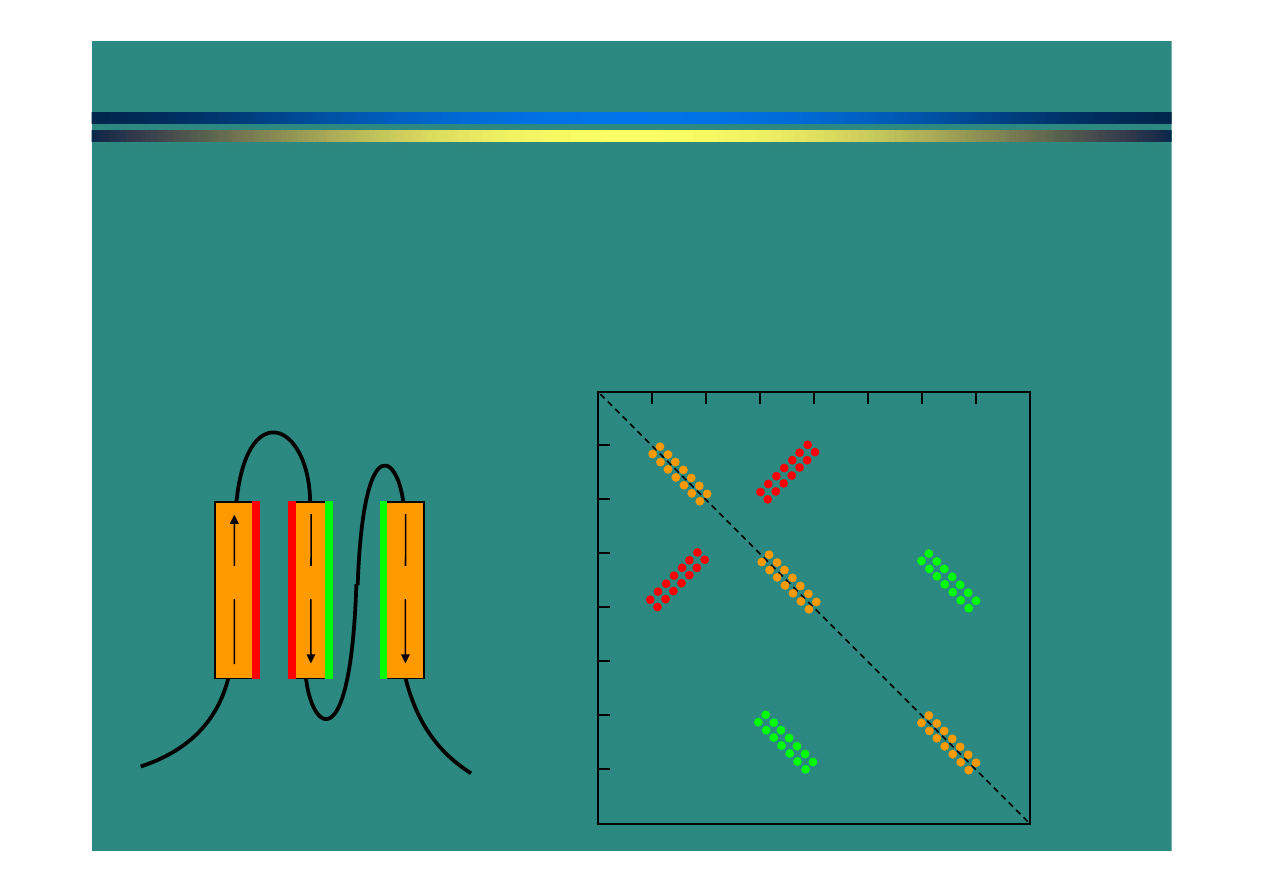
Macierz odległości / macierz kontaktów
Macierz kontaktów –
uproszczona (0 albo 1) macierz odległosci.
reszty pozostajace
”w kontakcie”, (np. < 5
A).
białka posiadają
podobną
strukturę, ->
macierze są
podobne
10 40
0
10
30
20
60
20
30
40
50
60
70
70
0
10
20
30
40
50
60
70
a
b
c
a
b
c
80
80

2009-01-09
26
porównanie struktur -
programy
DALI
-
Distance Alignment Tool; dopasowanie macierzy
odległości
VAST
-
Vector alignment Search Tool; dopasowanie
wektorowe; wektory
opisujące
struktury
drugorzędowe
FATCAT
-
Flexible Alignment allowing Twists
LGA
–
lokalna i globalna optymalizacja RMSD
(longest
continuous
segments
& global
distance
test)
Wybór metody porównania struktur zależy od problemu
Document Outline
- Bioinformatyka – wykład 9, 9.XII.2008 białkowa bioinformatyka strukturalna – c.d.
- Plan wykładu
- Wiązania wodorowe...
- Poziomy organizacji struktury białka
- Granice dokładności przewidywań strukturalnych
- Plan wykładu
- Struktura białka – jak wygląda?
- Slajd numer 8
- Slajd numer 9
- Slajd numer 10
- Wybór przedstawienia białka zależy od zadawanych pytań
- Plan wykładu
- Powierzchnia białka
- Plan wykładu
- Przykładowe programy do obrazowania struktur białkowych
- Istotne cechy programów do obrazowania struktur
- Plan wykładu
- Slajd numer 18
- Popularne systemy klasyfikacji struktur białkowych
- Porównywanie struktur
- Kształt (fold) a topologia
- Slajd numer 22
- Slajd numer 23
- Slajd numer 24
- Slajd numer 25
- Slajd numer 26
Wyszukiwarka
Podobne podstrony:
bioinformatyka w13 2008 9 web
bioinformatyka w2 2008 web
bioinformatyka w6 2008 web
bioinformatyka w11 2008 web
bioinformatyka w4 2008 web
bioinformatyka w10 2008 web
bioinformatyka w12 2008 9 web
bioinformatyka w3 2008 web
bioinformatyka w7 2008 web
bioinformatyka w1 2008 web
bioinformatyka w8 2008 web
bioinformatyka w5 2008 web
bioinformatyka w13 2008 9 web
bioinformatyka w2 2008 web
więcej podobnych podstron