Laboratorium Modelowania Biosystemów Modele stochastyczne
Prowadzący: dr inż. Zbigniew Starosolski
1. WPROWADZENIE
1) Model w wykładniczym czasem podziału
W tym modelu przyjmujemy, w odróżnieniu od deterministycznego modelu wzrostu malthusiańskiego, iż
czas podziału nie jest stały, lecz fluktuuje wokół pewnej wartości średniej. W związku z tym do opisu modelu
niezbędne jest określenie rozkładu czasów podziału. Jedną z możliwości jest np. rozkład wykładniczy, którego
funkcja gęstości opisana jest wzorem :
f(t) = λe
-λt
dla t≥0 przy czym λ = 1/T
c
gdzie T
c
jest średnią wartością czasu podziału
Zmianę liczebności populacji możemy opisać jako różnicę strumieni dopływającego (+) oraz odpływającego (-) :
dN = ( X
+
- X
-
)dt
przy czym
X
-
= aN (gdzie a = λ)
oraz przyjmujemy podział na 2 komórki potomne:
X+ = 2X
otrzymujemy więc :
dN/dt = aN
gdzie N jest średnią liczebnością populacji
2) Modele kompartmentalne – stochastyczne
Mamy układ składający się z k równań różniczkowych zapisanych w postaci:
k
i
t
N
t
N
N
i
i
i
i
i
1
),
(
)
(
1
1
każde i-te równanie możemy zamodelować wykorzystując rachunek operatorowy jako:
1
i
i
s
1
i
X
i
X
i
N
i
N
1
i
N
+
-
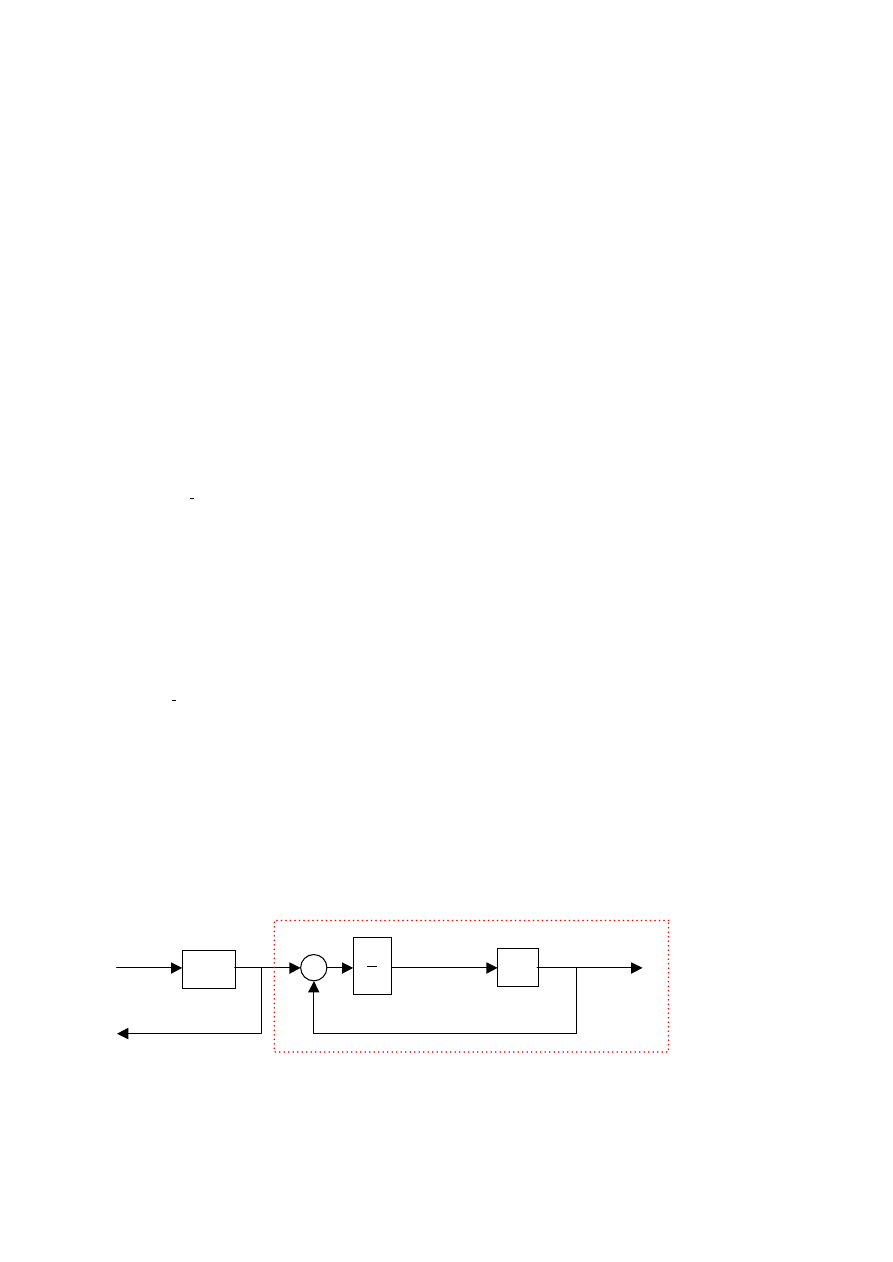
po przekształceniu (części schematu zaznaczoną czerwoną linią punktową) możemy uprościć do bloku postaci
(inercja I rzędu, o stałej czasowej
i
i
1
)
Układ równań różniczkowych wraz z równaniem granicznym wiążącym populacje można zapisać następująco jako:
k
i
t
N
t
N
N
t
N
t
N
N
i
i
i
i
i
k
k
2
),
(
)
(
)
(
)
(
2
1
1
1
1
1
co można zapisać za pomocą schematu, którego analizowanie opiera się na obserwacji przepływów i stanów
kompartmentów:
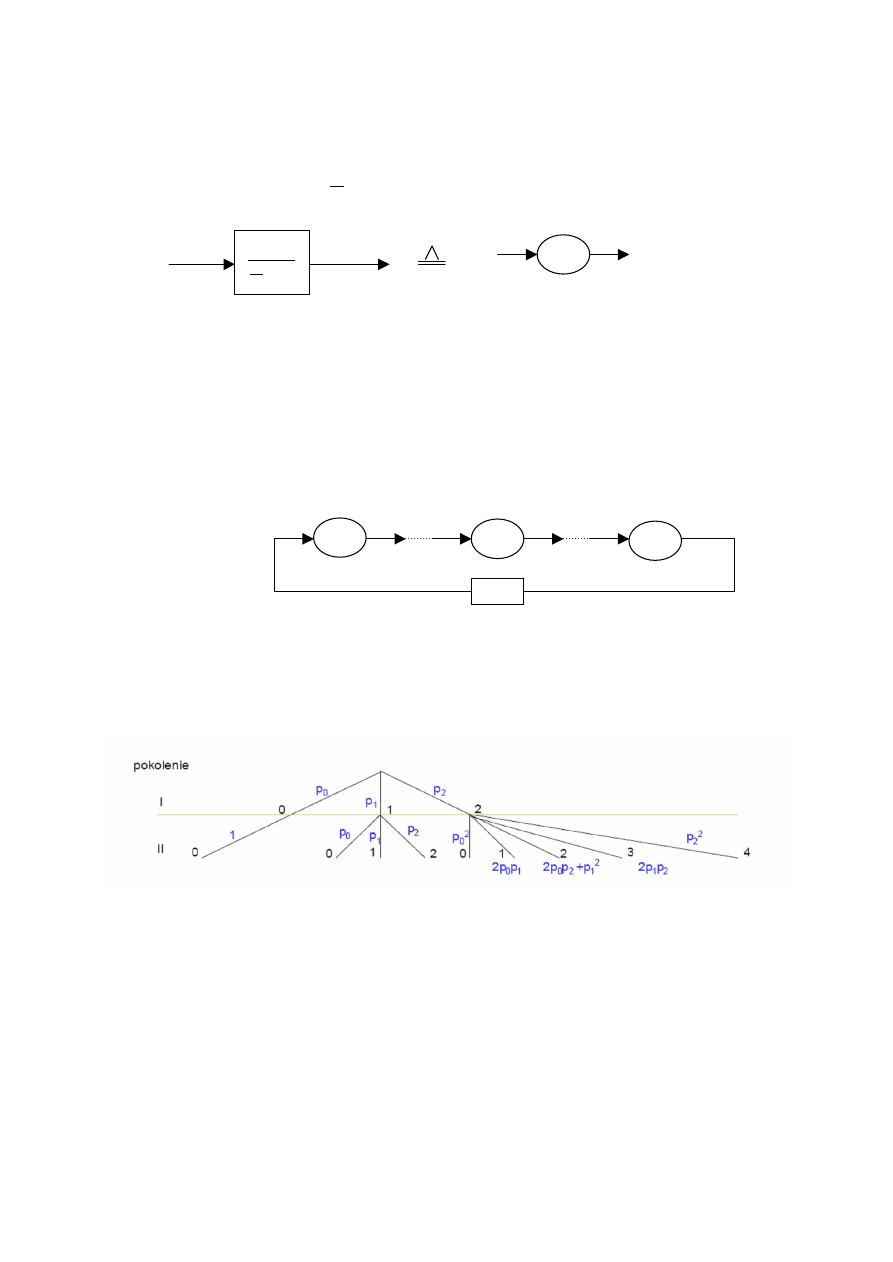
3) Proces gałązkowy
Model ten jest przydatny do badania populacji komórek, w sytuacji, gdy komórka ma różną ilość potomków lub gdy
podział nie oznacza śmierci komórki macierzystej. Załóżmy, iż komórka może mieć 0, 1 lub 2 potomków, jako p0,
p1 i p2 oznaczmy prawdopodobieństwa tych zdarzeń (p
0
+p
1
+p
2
=1). Poniższy rysunek prezentuje tzw. „drzewo”
procesu dla dwóch pokoleń:
zatem w prosty sposób można zapisać prawdopodobieństwa ilości osobników w danej generacji p(X
k
):
X
k
– liczba osobników k-tej generacji
p(X0=1)= 1
p(X1=0)= p
0
p(X1=1)= p
1
p(X1=2)= p
2
p(X2=0)= p
0
+ p
0
p
1
+ p
2
p
02
p(X2=1)= p
0
+ 2p
2
p
0
p
1
p(X2=2)= p
1
p
2
+ p
2
(2p
0
p
2
+ p
12
)
p(X2=3)= 2p
22
p
1
p(X2=4)= p
23
Jeśli potraktujemy X
k
jako zmienną losową możemy wyznaczyć jej wartość oczekiwaną ( E(X
1
)=µ ).
1
1
1
s
i
i
X
i
X
i
i
X
i
X
1
i
k
2
i
X
i
X

2. PROGRAM ĆWICZENIA
Zadanie 1:
Dla modelu z wykładniczym czasem podziału, mając daną wielkość początkową populacji N0, określić średnią
wielkość populacji po zadanym czasie. Czasy podziału wygenerować generatorem o rozkładzie wykładniczym.
Zadanie 2:
Dla populacji komórkowej k=10 zbadaj zależność zachowania się modelu kompartmentalnego od przebiegu funkcji
czasów podziałów. Dla wybranego przez prowadzącego typu przebiegu funkcji czasów podziału dla k=2 zbadaj
stabilność układu. Wykreśl przykładowe przebiegi.
Zadanie 3:
Stosując proces gałązkowy, mając daną wielkość początkową populacji N0, wyznaczyć wartość oczekiwaną
populacji w zadanym pokoleniu. Wyjaśnić jaki los czeka populacje w zależności od wielkości wartości oczekiwanej
w jednym pokoleniu (µ).
3 PYTANIA SPRAWDZAJĄCE
Podaj definicje i własności : proces stochastyczny, proces deterministyczny, zmienna losowa, bilans masy, rozkład
normalny, rozkład wykładniczy, wartość średnia, funkcja tworząca, funkcja prawdopodobieństwa, gęstość funkcji
prawdopodobieństwa, wartość własna macierzy, wyznacznik macierzy, warunki konieczne i wystarczające
stabilności asymptotycznej układu
Wyszukiwarka
Podobne podstrony:
7 4 2 Lab Install AllinOne id 4 Nieznany (2)
Lab 05 Obliczenia w C id 257534 Nieznany
CCNA4 lab 1 1 4a pl id 109119 Nieznany
CCNA4 lab 1 1 4b pl id 109120 Nieznany
Lab KN cw 5 id 258468 Nieznany
Lab 06 Instrukcje sterujace id Nieznany
5 6 3 Lab Registry Backup id 40 Nieznany (2)
lab pwsp 05 id 258618 Nieznany
GNS3 Lab Workbook v0 2 id 19267 Nieznany
ES lab uklad zaplonowy id 16347 Nieznany
lab 02 php id 258739 Nieznany
Lab technologii cw 4 id 258645 Nieznany
Modele dynamiczne id 305054 Nieznany
Procesy stochast id 393917 Nieznany
modele rynkowe1 id 305129 Nieznany
Lab technologii cw 6 id 258649 Nieznany
Lab ME MS instrukcja 2012 E id Nieznany
lab 4 Narzedzia scierne id 257 Nieznany
IMiR lab 2014 15 id 211868 Nieznany
więcej podobnych podstron