
Uniwersytet Gdański, Wydział Biologii
Katedra Biologii Molekularnej
Przedmiot: Biologia Molekularna z Biotechnologią
Biologia II rok
========================================================
Ćwiczenie 1
Sekwencjonowanie DNA
Prowadzący: mgr Michalina Filipiak i mgr Karolina Sawiuk
Sekwencjonowanie jest techniką umożliwiającą określenie kolejności nukleotydów w
analizowanym fragmencie DNA. Przełom nad pracami przyniósł rok 1977, w którym
opublikowano dwie metody:
A) chemicznej degradacji DNA, znanej jako metoda Maxama i Gilberta
B) kontrolowanej terminacji replikacji (zwana również metodą dideoksy) Sangera i
Coulsona
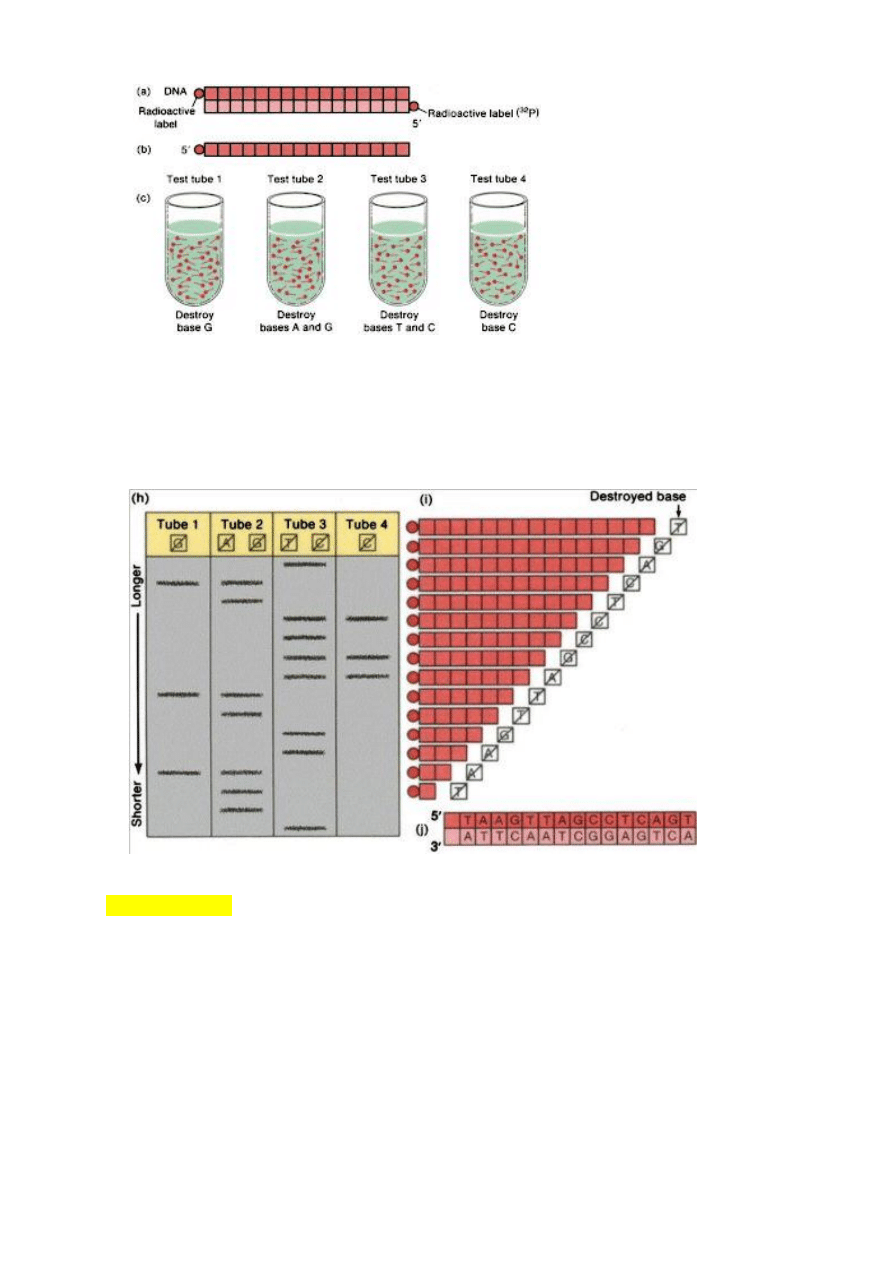
A) Metoda opracowana przez Maxama i Gilberta, polega na degradacji wyznakowanych
na końcu 3’ lub 5’ cząsteczek DNA odczynnikami atakującymi specyficznie wiązania
fosfodiestrowe za nukleotydem odpowiadającym odpowiedniej zasadzie azotowej.
Warunki reakcji są tak ustalone, że w poszczególnych cząsteczkach zostaje przecięte
jedno lub dwa wiązania, przez co otrzymuje się zbiór fragmentów DNA o różnej
długości. Równolegle w czterech probówkach prowadzi się reakcje modyfikacji dla
nukleotydów: G, A + G, T + C i C. Stosuje się tutaj reakcje dla A + G, T + C,
ponieważ uzyskanie specyficznej degradacji w pozycji A i T jest bardzo trudne.
Uzyskane fragmenty rozdzielane są w żelach poliakrylamidowych i poddawane
autoradiografii. Na autoradiogramie widoczne są tylko prążki odpowiadające
fragmentom zaczynającym się od wyznakowanego końca 3’ lub 5’, ułożone w postaci
drabinki, w której każdy kolejny szczebel odpowiada fragmentom różniącym się od
otaczających go o jeden nukleotyd.
Miejsce cięcia
Związki chemiczne
G
siarczan dimetylu + piperydyna
G + A
siarczan dimetylu + piperydyna +
C
hydrazyna/chlorek sodu + piperydyna
C + T
hydrazyna + piperydyna
Piperydyna powoduje degradację wiązań
N-glikozydowych i fosfodiestrowych, co wykorzystywane
jest w metodzie sekwencjonowania DNA
według Maxama-Gilberta.
Autoradiogram- obraz różnych frakcji DNA w formie ciemnych prążków na wywołanej
kliszy. Najmniejszym cząsteczkom odpowiadają prążki położone najdalej od jednej z
krawędzi kliszy (odpowiadającej miejscu naniesienia próbek na żel).
B) Drugą, częściej obecnie stosowaną, metodę sekwencjonowania DNA opracował
zespół F. Sangera. Metoda ta polega na analizie produktów syntezy in vitro DNA na
jednoniciowej matrycy, począwszy od przygotowanego oligonukleotydowego startera,
komplementarnego do końca 3’ matrycy. Równolegle stosuje się cztery różne
mieszaniny reakcyjne, przy czym każda zawiera tylko jeden z czterech 2’,3’-
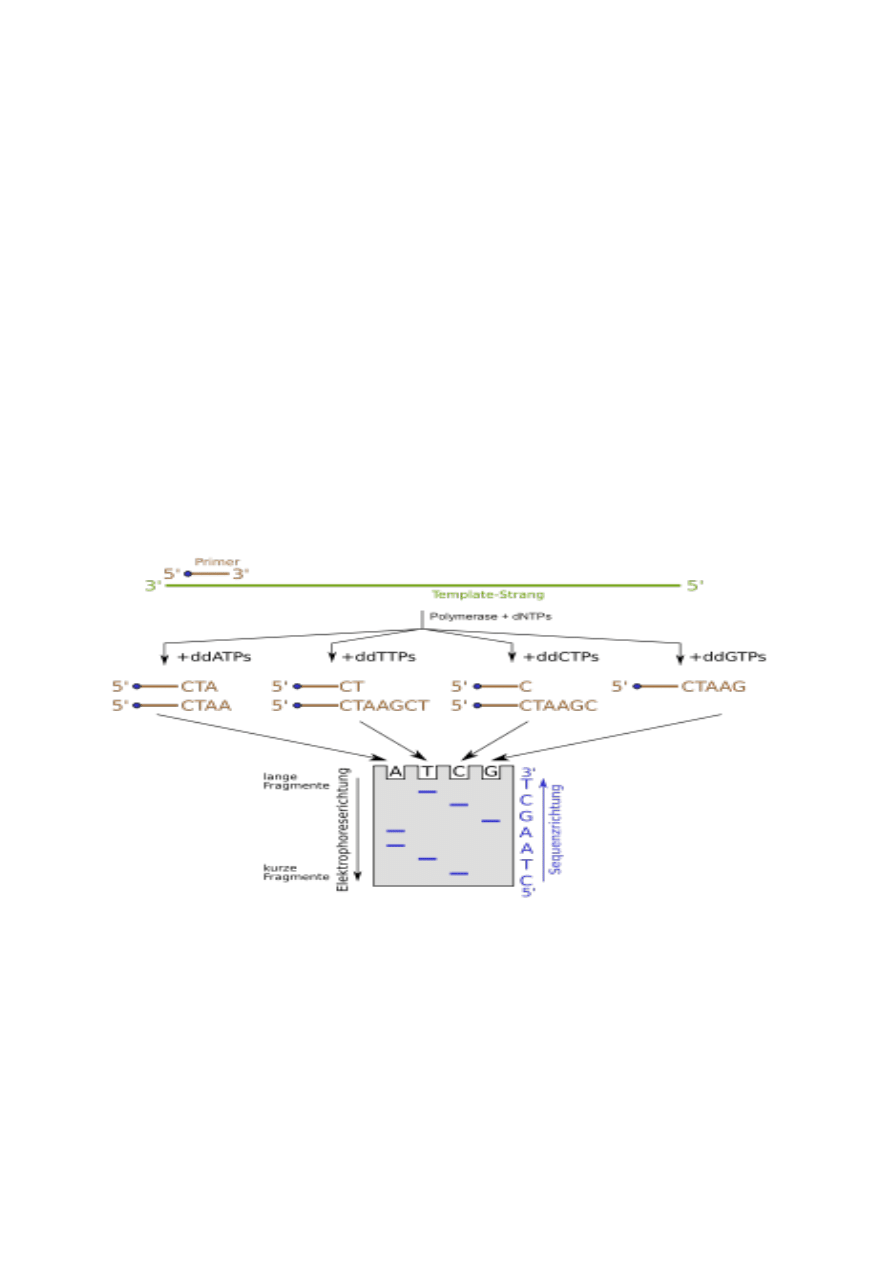
dideoksynukleotydów. Gdy 2’,3’-dideoksynukleotyd zostanie wbudowany w
cząsteczkę DNA (w miejsce odpowiedniego nukleotydu) następuje terminacja syntezy
w pozycji, w której został włączony do rosnącej nici. Cztery zbiory fragmenów o
zakończonym łańcuchu poddaje się następnie elektroforezie i odczytuje sekwencję
zasad DNA z czterech ścieżek na autoradiogramie.
Skład mieszaniny reakcyjnej:
ta sama matryca
starter
mieszanina deoksyrybonukleotydów (dATP, dTTP, dCTP, dGTP)
bufor
polimeraza DNA
jeden z ddNTP, będących analogami dNTP (odpowiednio: ddATP, ddTTP, ddCTP,
ddGTP)
Dideoksynukleotydy to zmodyfikowane nukleotydy, pozbawione grupy hydroksylowej w
pozycji 3' deoksyrybozy. Zmiana ta uniemożliwia dalszą syntezę nici DNA. Co więcej,
zatrzymanie wydłużania łańcucha nukleotydowego następuje zawsze w pozycji zajmowanej
przez ten sam dideoksynukleotyd.
Nowoczesne sekwencjonowanie metodą Sangera (metoda dideoksy pochodnych)
wykorzystuje dideoksynukleotydy wyznakowane barwnikami fluorescencyjnymi. Reakcja
przypomina standardową reakcję PCR z matrycą (zazwyczaj dsDNA) i polimerazą DNA,

jednak wykorzystywany jest tylko jeden starter komplementarny do sekwencji powyżej
fragmentu ulegającego sekwencjonowaniu. Dodatkowo poza mieszaniną trójfosforanów
deoksyrybonukleotydów (dNTPs), w niewielkim stężeniu występują również trójfosforany
dideoksyrybonukleotydów (ddNTPs), gdzie każdy wyznakowany jest innym barwnikiem
fluorescencyjnym. W takiej reakcji, starter zostaje wydlużony do czasu kiedy wbudowany
zostanie ddNTP. W tym momencie dalsze wydłużanie jest niemożliwe i fragment DNA o
określonej długości „wyznakowany” zostaje barwnikiem związanym z danym ddNTP. W
jednej probówce, po określonej liczbie cykli powstaje mieszanina jednoniciowych
fragmentów DNA o różnej długości i każdy zakończony jest ddNTP. Ta mieszanina
rozdzielana jest przy pomocy elektroforezy w kapilarze. Fluorescencja sczytywana jest przy
użyciu lasera do wzbudzenia i rejestratora. Każdy barwnik użyty do znakowania ddNTP
emituje światło o innej długości fali (kolorze). Program obsługującey urządzenie, każdemu
odczytowi przypisuje odpowiedni nukleotyd i generuje chromatogram (obrazek poniżej), na
którym odczyt długości fali przedstawiony jest w postaci kolorowej linii (przykładowo: A –
zielony, T – czerwony, C – niebieski, G – czarny).
Przykładowe wyniki sekwencjonowania z różnych matryc DNA (Homo sapiens i Saccharomyces cerevisiae)
Nowoczesne metody sekwencjonowania (Next Generation Sequencing)
Rozwój metod sekwencjonowania kwasów nukleinowych dąży w kierunku wysokiej
przepustowości (high throughput). Pozwala to na jednoczesną analizę wielu krótkich
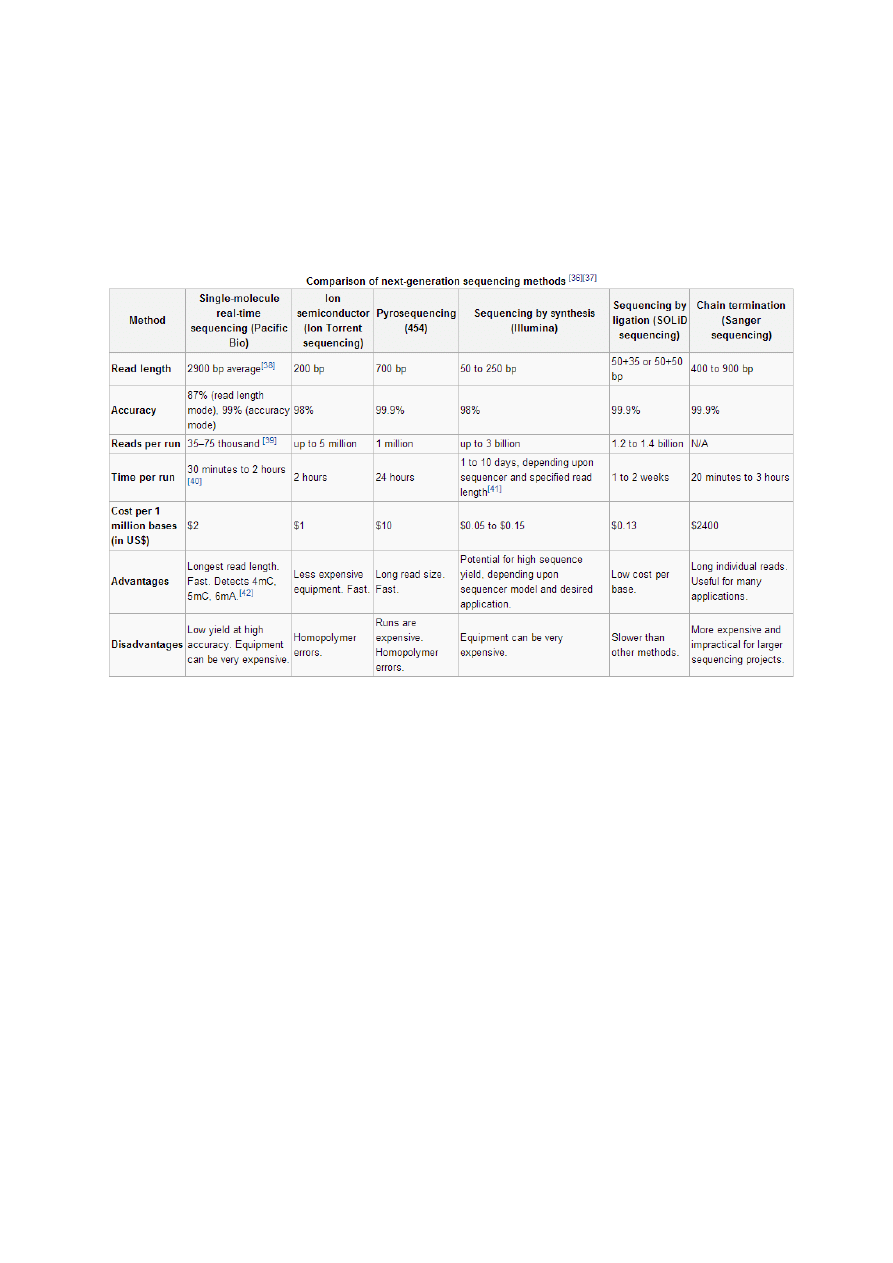
sekwencji i zestawienie ich przy użyciu bioinformatyki. Trendy w unowocześnianiu
sekwencjonowania można opisać słowami: szybciej, więcej, taniej. W poniższej tabeli
przedstawione jest zestawienie zalet i wad nowoczesnych metod sekwencjonowania (tzw.
Next Generation Sequencing, NGS). Poza metodami sekwencjonowania DNA, w ostatnich
latach wynaleziono metode bezpośredniego sekwencjonowania RNA (Direct RNA
Sequencing).
Tabela przedstawiająca podsumowanie nowoczesnych metod wykorzystywanych do sekwencjonowania
DNA.
Sekwencjonowanie pojedyńczej cząsteczki w czasie rzeczywistym – SMRT (Single-
molecule real-time sequencing)
Wykorzystuje sekwencjonowanie przez syntezę. Reakcja przeprowadzana jest w studzienkach
w których na dnie przymocowana chemicznie jest polimeraza DNA. Wyznakowane
fluorescencyjnie nukleotydy DNA występują w mieszaninie reakcyjnej. Konstrukcja
studzienki pozwala na odczyt fluorescencji jedynie na dnie studzienki (poziomie polimerazy
DNA). Podczas syntezy, barwnik fluorescencyjny jest uwalniany z łańcucha DNA.
Odczyt fluorescencji w pozycji polimerazy pozwala na identyfikacje nukleotydy, który jest
aktualnie wbudowywany do syntetyzowanego łańcucha DNA.
Sekwencjonowanie z użyciem jonowego pólprzewodnika – (Ion semiconductor
sequencing)
Jest to technika wynaleziona i opracowana przez firmę Ion Torrent Systems Inc. Metoda
wykorzystuje standardowa chemię reakcji PCR. Podstawą jest detekcja jonu wodorowego

uwalnianego podczas syntezy DNA. Studzienka reakcyjna wypelniona jest mieszaniną
zawierającą jeden typ nukleotydu w danym momencie, polimerazą DNA i matrycowym
fragmentem DNA (który ulega sekwencjonowaniu). Po każdym cyklu mieszanina zmienia się
na nową z innym typem nukleotydu (np. 1 cykl - dATP, 2 cykl - dTTP, 3 cykl - dCTP, 4 cykl
- dGTP). Detekcja uwolnionego jonu wodorowego oznacza, że w danych warunkach (tzn.
kiedy w reakcji był okreslony nukleotyd) doszło do syntezy DNA.
Pirosekwencjonowanie (Pyrosequencing, 454 sequencing)
Metoda wykorzystuje standardową amplifikację za pomocą PCR (podobnie jak wyżej) z
mieszaniną reakcyjną, która również zawiera pojedyńczy nukleotyd. Należy zaznaczyć, że
mimo kolejności opisu w tym skrypcie, pirosekwencjonowanie jest starszą metodą niż
opisana metoda Ion Torrent Systems. Wykorzystywany jest starter który jest wydłużany w
kolejnych cyklach, o ile w mieszaninie znajduje się nukleotyd komplementarny do matrycy.
Podczas syntezy uwalniany jest fosforan organiczny, który pośrednio umożliwia
wygenerowanie światła przez enzym lucyferazę. Detekcja światła oznacza, że w danych
warunkach zaszła synteza DNA.
Sekwencjonowanie na platformie Illumina (Solexa) – Illumina (Solexa) sequencing
Metoda wykorzystuje odwracalnie zmodyfikowane nukleotydy, jednak wszystkie występują
w jednej mieszaninie. Modyfikacja zawiera barwnik fluorescencyjny umożliwiający detekcje
nukleotydy, jak również zapobiega dalszej elongacji. Po etapie syntezy, czyli addycji
nukleotydu, fluorescencja jest sczytywana, a następnie wykorzysuje się enzymy do usunięcia
modyfikacji i odpłukuje pozostałości reakcji. Podczas kolejnego cyklu dodany zostaje nowy
nukleotyd, odczytana zostaje fluorescencja i dochodzi do recyklingu (usunięcia) modyfikacji.
Wybrane, opisane powyżej metody są w pełni zautomatyzowane. W niektórych zachodzi
jedynie potrzeba uprzedniego przygotowania „biblioteki” np. poprzez ligację odpowiednich
adapterów na końcach fragmentów DNA. Należy również dodać, że zarówno wygenerowanie
danych, jak również ich dalsza analiza często wymaga zaawansowanych narzędzi
bioinformatycznych. Zautomatyzowane metody odczytu sekwencji pozwoliło na szybki
postęp w dziedzinie sekwencjonowania całych genomów. W 1995 poznano pierwsze genomy
bakterii (Haemophilus influenzae i Mycoplasma genitalium), w 1997 genom pierwszego
eukarionta - drożdży piekarskich (Saccharomyces cerevisiae), w 1998 genom pierwszego
organizmu wielokomórkowego nicienia Caenorhabditis elegans, w 2000 genom muszki
owocowej (Drosophila melanogaster). W lutym 2001 niezależnie Human Genome Project i
Celera Genomics opublikowali sekwencję ok. 3 miliardów nukleotydów genomu ludzkiego
(tj. ok. 90% genomu ludzkiego). W roku 2003 opublikowano dokument stwierdzający

zakończenie sekwencjonowania 99% genomu ludzkiego. W dzisiejszych czasach
sekwencjonowanie nie ogranicza się jedynie do analizy genomu. Pośrednio, poprzez analizę
cDNA, sekwencjonowaniu ulega transkryptom (sekwencja ulegająca transkrypcji na RNA)
organizmu lub komórki. Pozwala to m.in. na analizę ekspresji genu. Techniki
sekwencjonowania sa rozwijane i modyfikowane na bieżąco, tak że najnowsze osiągniecia w
tej dziedzinie pozwalają na analizę transkryptomu pojedyńczej komórki.
Dlaczego badania nad ludzkim genomem są tak ważne?
PERSPEKTYWY WYKORZYSTANIA
1.Rozwój bioinformatyki- dział informatyki zajmujący się badaniem DNA
2. Opracowanie mikromacierzy. Płytka szklana lub plastikowa z naniesionymi w regularnych
pozycjach mikroskopowej wielkości polami, zawierającymi różniące się od siebie sekwencją
fragmenty DNA. Fragmenty te są sondami, które wykrywają przez hybrydyzację
komplementarne do siebie cząsteczki DNA lub RNA.
3. Rozwój biologii ewolucyjnej- możliwość porównywania genomu ludzkiego z genomami
innych organizmów.
4. Rozpoznanie, jakie geny są odpowiedzialne za powstawanie, powiązanie i utrzymanie
systemu nerwowego- leczenie chorób neurologicznych.
5. Leczenie nowotworów.
Nowotwory są najczęściej spowodowane akumulacją kilku mutacji genowych, które aktywują
onkogeny. Zidentyfikowanie do tej pory ponad 100 onkogenów obrazuje zmiany powodujące
rozwój nowotworu. Niektóre nowotwory, choć wizualnie podobne, mają inny rozkład
ekspresji genów- poznanie ułatwia wybranie terapii.
6. Wprowadzenie metody „fingerprinting”. Metodę stosuje się w diagnostyce chorób
dziedzicznych (np. hemofilii, anemii sierpowatej, choroby Alzheimera, choroby
Huntingtona), w kryminalistyce do identyfikacji przestępców na podstawie pozostawionych
przez nich śladów biologicznych.
7. Rozwój badań nad uzależnieniami od narkotyków- kokaina wpływa na transportery
dopaminy, które różnią się u różnych ludzi.

ZNAKOWANIE KWASÓW NUKLEINOWYCH
W pracach biologii molekularnej często zachodzi konieczność używania znakowanych
cząsteczek kwasów nukleinowych, czyli takich, które zawierają wbudowany radioaktywny
izotop lub znacznik nieradioaktywny, np. biotynę. Znakowany DNA lub RNA
wykorzystywany jest w takich technikach jak: testy hybrydyzacji, primer extension, foot-
printing, EMSA, RNA protection, sekwencjonowanie i wiele innych.
1. Najczęściej wykorzystywanymi znacznikami kwasów nukleinowych są radioaktywne
izotopy
32
P i
35
S,
3
H oraz związki nieradioaktywne, takie jak: biotyna, digoksygenina,
fluoresceina, rodamina, kumaryna. Wybór znacznika zależy głównie od czułości i
rozdzielczości testu, do którego zostanie użyty znakowany DNA, a także od trwałości
znacznika i bezpieczeństwa pracy.
2. Detekcja znaczników.
a. Znaczniki radioaktywne. Identyfikacja opiera się na detekcji promieniowania β.
Promieniowanie to można zlokalizować, wykonując ekspozycję sygnału do na błonie
radiologicznej lub z wykorzystaniem specjalistycznego sprzętu (licznik
scyntylacyjny).
b. Znaczniki nieradioaktywne. Obecność tych znaczników można zidentyfikować na
podstawie ich aktywności własnej (bezpośrednie) lub aktywności enzymów
połączonych ze znacznikiem (pośrednie)
- bezpośrednie. Związki wbudowane do DNA, takie jak rodamina, kumaryna,
fluoresceina, charakteryzują się zdolnością do emisji światła o określonej długości
fali. Ich detekcja odbywa się przy pomocy mikroskopu fluorescencyjnego.
- pośrednie. Oparte są na technikach enzymatycznych. Znaczniki rozpoznawane są
przez specyficzne przeciwciała (ew. streptawidynę w przypadku biotyny) sprzężone z
enzymami takimi jak alkaliczna fosfataza, peroksydaza. Enzymy te po przyłączeniu
się do znacznika katalizują reakcje, które prowadzą do luminescencji lub powstania
barwnych produktów.

Przebieg ćwiczenia:
Omówienie metod sekwencjonowania.
Odczytywanie sekwencji z autoradiogramu.
Rozpoznawanie polimorfizmów na chromatogramie (wyniki minisekwencjonowania).
Zagadnienia do przygotowania
Etapy reakcji sekwencjonowania.
Modyfikacje nukleotydów stosowane w metodzie Maxama i Gilberta.
Modyfikacje klasycznych metod sekwencjonowania oraz alternatywne techniki.
Znaczniki fluorescencyjne oraz pierwiastki radioaktywne najczęściej stosowane w
znakowaniu starterów lub dNTPs.
Na czy polega strategia „shotgun sequencing”(sekwencjonowanie losowe).
Sposoby uzyskania jednoniciowego DNA w przypadku sekwencjonowania metodą terminacji
łańcucha.
Literatura obowiązkowa
1. Sanger F., Nicklen S., Coulson A. R. (1977) DNA sequencing with chain-terminating
inhibitors. Proc Natl Acad Sci U S A, 74(12): 5463–5467.
2. Venter J. Craig et al.(2001), The Sequence of the Human Genome, Science 291, 1304,
1304-1351,
3. Ivo Glynne Gut, Clin Transl Oncol (2013) 15:879–881
4. Brown T. A. Genomy. Wydawnictwo Naukowe PWN Warszawa 2003
5. Genetyka molekularna, Praca zbiorowa pod redakcją Piotra Węgleńskiego.
Wydawnictwo Naukowe PWN Warszawa 2000
Wyszukiwarka
Podobne podstrony:
9 ćwiczenie 2014
Plan cwiczen V X 2014
Ćwiczenia, Instrukcja do ćwiczenia 4, Układy sekwencyjne - przerzutniki asynchroniczne i synchronicz
Ćwiczenia 1 i 2 (2014 st. stacjonarne), Nowoczesne metody analizy żywności
PROGRAM ĆWICZEŃ 2014, medycyna, histologia
Botanika systematyczna program ćwiczeń 2014, biologia, Biologia I rok, Botanika systematyczna
Plan cwiczen V X 2014
10 ćwiczenie 2014
9 ćwiczenie 2014
OZP ćwiczenia 2014(1)
Ćwiczenia 2014
Ćwiczenia 2014 (puste)
OZP ćwiczenia 2014
OZP ćwiczenia 2014
Ćwiczenia i seminarium 1 IV rok 2014 15 druk
Ekonomia ćwiczenia program PS1 2014 2015 (1)
Harmonogram ćwiczeń s5 2014 TABL 03 (08 10 14 )
Pytania z wejściówek, analityka medyczna UMP 2014, chemia fizyczna, ćwiczenia
więcej podobnych podstron