Ćwiczenie 4: Izolacja plazmidu do klonowania z wykorzystaniem zestawów
do izolacji DNA (miniliza), trawienie i elektroforeza plazmidu
Materiał do kolokwium - instrukcja, metody izolacji DNA plazmidowego, klonowanie
Wstęp teoretyczny
Izolacja DNA plazmidowego
Plazmidy to pozachromosomowe jednostki dziedziczenia występujące w komórce bakteryjnej. Ich cechą charakterystyczną jest budowa kolista. Zamknięta cząsteczka dodatkowo zwinięta jest wokół własnej osi tworząc tzw. superhelisę. Podczas izolacji DNA plazmidowego wykorzystuje się różnice właściwości między niewielkim kolistym DNA plazmidu, a DNA pochodzącym z chromosomu bakteryjnego. Czynnikiem różnicującym obydwie cząsteczki jest reakcja na proces denaturacji spowodowanej podwyższonym pH (liza alkaliczna) bądź wysokimi temperaturami. DNA plazmidowe łatwiej ulega renaturacji w momencie neutralizacji środowiska bądź obniżenia temperatury, podczas gdy DNA chromosomu bakteryjnego pozostaje w formie zdenaturowanej ze względu na swoją wielkość. W łagodniejszych warunkach obydwie formy mogą renaturować, przy czym DNA plazmidowe renaturuje znacznie szybciej. W metodzie lizy alkalicznej ważny jest odpowiedni czas traktowania czynnikiem denaturującym, ponieważ zbyt długie działanie wysokiego pH może nieodwracalnie denaturować obydwa rodzaje DNA. Neutralizację NaOH wykorzystywanego do lizy alkalicznej przeprowadza się octanem potasu o pH=4,8.
Do oczyszczenia plazmidu z obecnych w preparacie fragmentów DNA genomowego może w obecności związków interkalujących, np. bromku etydyny (EtBr). Związek ten wnikając pomiędzy zasady azotowe powoduje ich rozsunięcie i związane z nim częściowe rozplecenie nici DNA. To zjawisko jest odpowiedzialne za zmniejszenie jego gęstości pławnej. W plazmidowym DNA, ze względu na jego superzwiniętą strukturę, rozplatanie nici jest utrudnione, w związku z tym mniejsza ilość cząsteczek EtBr wiąże się z DNA. Powoduje to zróżnicowanie gęstości pomiędzy fragmentami liniowymi i kolistymi DNA. Zróżnicowane pod względem gęstości pławnej cząsteczki kwasów nukleinowych można następnie rozdzielać w roztworze chlorku cezu (CsCl). W wyniku wirowania wytwarza się gradient gęstości CsCl, a DNA stabilizuje się w tym miejscu, w którym jego gęstość pławna równa się gęstości CsCl.
Rys.1. Obraz UV po wirowaniu w gradiencie CsCl
Izolacja plazmidu do klonowania z wykorzystaniem zestawów do izolacji DNA
z kolumną
Metoda ta jest wykorzystywana przede wszystkim do izolacji plazmidowego DNA z wielu małych hodowli. Może być wykorzystywana do identyfikacji klonów.
Schemat. Kolejność głównych etapów podczas izolacji plazmidowego DNA
Procedura opiera się na tzw. metodzie alkalicznej, w której roztwory zasadowe użyte są
do denaturacji DNA, a następnie wykorzystując różnice w czasie renaturacji (koliste plazmidy, zawierające sczepione ze sobą nici DNA renaturują znacznie szybciej, niż DNA chromosomu bakteryjnego) można oczyścić DNA plazmidowe od DNA chromosomowego. Roztwór GL3 zobojętnia środowisko, a równocześnie powoduje wytrącenie DNA chromosomowego i białek. Istotne jest w tej metodzie przestrzeganie czasu trwania denaturacji alkalicznej, ponieważ zbyt długie przetrzymywanie DNA w roztworach zasadowych powoduje nieodwracalną denaturację. Takie DNA nie poddaje się reakcjom trawienia lub ligacji.
Zasada działania kolumny opiera się na zdolności DNA do adsorpcji na powierzchni krzemionki w obecności związków chaotropowych (denaturujących białka,
np. chlorowodorek guanidyny). Po przemyciu czysty DNA jest wymywany buforami o niskiej sile jonowej lub wodą.
Ocenę jakościową izolacji wykonuje się poprzez pomiar absorbancji próbki roztworu zawierającego DNA. Kwasy nukleinowe, nukleotydy, nukleozydy i zasady azotowe absorbują promieniowanie UV z maksimum przypadającym na 260 nm. Wiedząc, że dla białek wartość ta wynosi 280 nm, można określać stopień czystości wyizolowanego DNA na podstawie stosunku absorbancji przy konkretnych długościach fal:
A260/A280 - 1,8 dla czystego dsDNA
Wartości mniejsze od 1,8 sugerują zanieczyszczenie białkiem, z kolei wartości wyższe mówią o zanieczyszczeniu RNA. Wynika to z faktu iż stosunek ten dla czystych białek wynosi 1, a dla czystego RNA wynosi 2.
Klonowanie genów
Do czasu wynalezienia nowoczesnych metod inżynierii genetycznej, manipulacje polegające na wyodrębnianiu i przenoszeniu genów z jednej komórki do drugiej wykonywano jedynie na bakteriach przy zastosowaniu bakteriofagów. W przypadku eukariotycznych organizmów manipulacje genetyczne stały się możliwe dopiero po wprowadzeniu metod rekombinacyjnych i klonowania DNA.
Proces klonowania genów można podzielić na kilka etapów. Pierwszym jest izolacja oraz fragmentacja DNA z komórek organizmu, którego geny mają zostać sklonowane. W celu fragmentacji DNA używane są enzymy restrykcyjne lub mechaniczne sposoby fragmentacji (rozciąganie, przeciąganie przez kapilary, rozpylanie roztworów). Analogicznie należy rozciąć nośnik, którym należy się posłużyć w celu klonowania wybranego fragmentu. Takim nośnikiem może być wektor plazmidowy (jak na ćwiczeniach), może też być nim wektor fagowy lub sztucznie skonstruowane hybrydy plazmidowo-fagowe, czy plazmidowo-chromosomowe. Trzeba pamiętać, że obecnie stosowane nośniki zawsze są przekonstruowane w celu osiągnięcia najlepszego efektu klonowania.
Kolejnym elementem procesu klonowania genów jest ligacja - łączenie fragmentów DNA z wektorem. Aby zapobiec odtworzeniu wyjściowego wektora poprzez cyrkularyzację jego formy liniowej (bez połączenia z klonowanym fragmentem DNA), można rozciąć wektor i klonowane DNA za pomocą dwóch różnych, odpowiednio dobranych enzymów restrykcyjnych. W efekcie końce zlinearyzowanego wektora nie są już wzajemnie komplementarne, co znacznie zmniejsza prawdopodobieństwa zamknięcia samego wektora do formy kolistej. Połączenie zrekombinowanych cząsteczek DNA katalizowane jest przez ligazę DNA. Jest to enzym wytwarzający wiązania fosfodiestrowe pomiędzy dwoma fragmentami DNA, w obecności źródła energii (ATP). Aktywność ligacyjna wymaga obecności grupy fosforanowej na końcu 5' i grupy -OH na końcu 3' łączonych fragmentów DNA. Ligaza DNA działa najefektywniej podczas tworzenia wiązań fosfodiestrowych między cząsteczkami posiadającymi zhybrydyzowane, komplementarne końce. Ligaza T4 posiada również zdolność łączenia tępych końców nici DNA. Jednak wydajność tego typu procesu jest niska i wymaga w związku z tym zwiększonej ilości enzymu oraz przedłużonego czasu reakcji. Zligowany z dodatkowym DNA plazmid należy następnie umieścić w komórkach gospodarza, w których będą mogły się powielać (transformacja). Ostatnim etapem, po otrzymaniu szeregu kolonii organizmów zawierających wektor, jest analiza i identyfikacja interesujących nas fragmentów DNA.
Podsumowując, główne etapy klonowania w komórkach bakteryjnych to:
1)Trawienie próbki DNA enzymem restrykcyjnym.
2) Trawienie DNA wektora enzymem restrykcyjnym.
3) Ligacja próbki DNA z wektorem (rekombinacja).
4) Transformacja bakterii (E. coli) produktem ligacji.
5) Wzrost bakterii na płytkach agarowych z antybiotykiem selekcyjnym.
6) Detekcja interesujących nas klonów.
Najczęściej używane metody transformacji bakterii to:
Metoda chemiczna: wykorzystująca CaCl2 (również mieszaniny innych soli, przede wszystkim z I grupy, i chlorek sześcioaminokobaltowy) oraz szok termiczny.
Elektroporacja: oparta na krótkim pulsowym zadziałaniu prądu elektrycznego
o wysokim napięciu.Biolistyka: bombardowanie komórek mikrocząstkami opłaszczonymi DNA.
Rys. 2. Transformacja chemiczna - schemat
Komórki bakteryjne, inkubowane w roztworze CaCl2 w lodzie można przechowywać przez wiele miesięcy w temp. -800C i posługiwać się nimi w razie potrzeby. Takie komórki, przygotowane do przyjęcia obcego DNA nazywamy komórkami kompetentnymi. Po dodaniu do nich plazmidu, komórki należy poddać szokowi termicznemu (420C), a następnie schłodzić. Czas trwania szoku termicznego musi być ściśle przestrzegany, żeby uzyskać odpowiednią wydajność transformacji.
Rys. 3. Elektroporacja
Elektroporacja jest procesem znacznie wydajniejszym od transformacji chemicznej, jednak wymaga dosyć kosztownego sprzętu, dlatego stosuje się ją przede wszystkim wtedy, gdy taka wydajność jest bezwzględnie konieczna, np. przy tworzeniu bibliotek genowych, czy genomowych. Czas trwania i napięcie elektroszoku jest zależne od wielkości i rodzaju komórek, dlatego te parametry dobierane są eksperymentalnie.
Biolistyka (gene-gun)
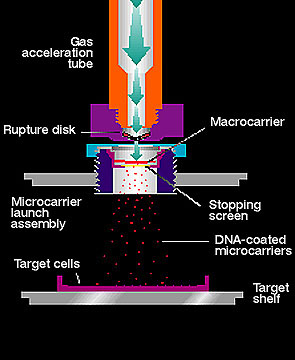
Bombardowanie komórek drobinkami złota, bądź tytanu, opłaszczonymi fragmentami DNA lub plazmidami nazywane jest strzelbą genową (ang. gene-gun). Metoda ta jest szczególnie skuteczna w stosunku do komórek zawierających ścianę komórkową, przede wszystkim komórek roślinnych.
Rysunek pokazuje schemat urządzenia i jego zasadę działania.
Aby niepotrzebnie nie przeszukiwać kolonii nie zawierających plazmidu, komórki po transformacji wysiewa się na podłoże selekcyjne, na którym przeżywają jedynie komórki zawierające plazmid. Każdy wektor, również wektory plazmidowe, zawiera tzw. marker selekcyjny, gen, którego obecność pozwala na odróżnienie komórek zawierających i nie zawierających plazmidu. W wektorach plazmidowych jako markery selekcyjne wykorzystywane są głównie geny oporności na różne antybiotyki, na ćwiczeniach będzie to gen oporności na ampicylinę. Wysianie komórek na podłoże zawierające ten antybiotyk pozwoli na wzrost jedynie komórkom zawierającym plazmid z odpowiednim markerem selekcyjnym.
Materiały i odczynniki:
Całonocna hodowla bakteryjna w 2 ml pożywki z antybiotykiem, schłodzona w lodzie
i odwirowana w temp. pokojowej, zamrożona
Kolumna do izolacji DNA (pojemność 20 μg DNA)
Roztwór L1 (do zawieszania bakterii: 50 mM Tris:HCl, pH 8.0; 10 mM EDTA; 100 ug/ml RNaza A)
Roztwór L2 (do lizy bakterii: 0.2 N NaOH; 10% SDS)
Roztwór GL3 (zobojętniający: 3 M CH3COOK; 5% CH3COOH; 8 M chlorowodowek guanidyny)
Roztwór W (do przemywania kolumny)
Roztwór A1 (do przemywania kolumny)
H2O (do wymywania DNA z kolumny)
Mikrowirówka
Termoblok
Enzym restrykcyjny EcoRI (10 U/μl)
Bufor do trawienia EcoRI 10x: (0.5 M Tris-HCl pH 7.5; 0.1 M MgCl2; 1 M NaCl;
0.2% Triton X-100; 1mg/ml albuminy)
Agaroza (naważka 0.24 g)
Bromek etydyny (20 mg/ml)
Bufor LB do obciążania DNA (0.25% błękit bromofenolowy; 0.25% cjanol ksylenu;
30% glicerol)
Marker do DNA (DNA /HindIII)
Bufor TAE do elektroforezy 50x (0.2 M Tris:octan; 0.05 M EDTA)
Aparat do elektroforezy horyzontalnej
Transiluminator
Wykonanie ćwiczenia
Osad bakterii z całonocnej hodowli należy zawiesić dokładnie w 200 μl roztworu L1, dodać 200 μl roztworu L2 i wymieszać delikatnie przez odwracanie probówki 5-6 razy. Inkubować 3 min. w temperaturze pokojowej.
Dodać 400 μl roztworu GL3 i obracać probówkę delikatnie 6-8 razy. Odwirować
przy maksymalnych obrotach przez 10 minut.Supernatant nanieść na kolumnę i wirować 1 minutę na maksymalnych obrotach.
Przefiltrowany roztwór odrzucić, kolumnę przemyć 500 μl roztworu W, wirować
1 minutę na maksymalnych obrotach, przefiltrowany roztwór odrzucić.Przemyć kolumnę 700 μl roztworu A1, wirować 2 minuty na maksymalnych obrotach, przefiltrowany roztwór odrzucić.
Do elucji DNA przenieść suchą kolumnę do nowej probówki (podpisanej - DNA plazmidowe) i dodać 40 μl H2O bezpośrednio na membranę. Inkubować 3 minuty
w temperaturze pokojowej i odwirować 1 minutę na maksymalnych obrotach. Kolumnę wyrzucić.4 μl DNA zawiesić w 200 μl H2O, zmierzyć ilość DNA spektrofotometrycznie, wykorzystując wzór:
C [μg/μl] = (A260 -A320) x rozcieńczenie x 0.05
W dzienniku laboratoryjnym musi być zapisane stężenie DNA plazmidowego oznaczone na ćwiczeniach.Trawienie plazmidu enzymem EcoRI - Sporządzić mieszaninę do trawienia:
DNA 0,5 μg
10x bufor do EcoRI 2 μl
EcoRI 0,5 μl (dodaje prowadzący ćwiczenia)
H2O do 20 μl
Trawić w temperaturze 370C, 1 godzinęW tym czasie z naważki agarozy przygotować i wylać żel (0.8%, 100 ml)
i przygotować preparat DNA nietrawionego:
DNA 0.25 μg + H2O do 10 μl + 2 μl Buforu LBPo trawieniu nanieść próby DNA nietrawionego i DNA potrawionego EcoRI (10 μl + 2 μl buforu LB). Elektroforezę prowadzić 15-30 minut.
Preparaty nietrawionego i trawionego DNA należy dokładnie podpisać nazwą grupy, sekcji i datą, a następnie oddać do zamrożenia.
Wyszukiwarka
Podobne podstrony:
tematy cwiczen - ii rok biologii, Biol UMCS, IV semestr, Biologia molekularna, Egzamin
Ćwiczenie 1 - oznaczanie stalej i stopnia dysocjacji, Biotechnologia PWR, Semestr 3, Chemia fizyczna
Ćwiczenie 2 - liczby przenoszenia i ruchliwosc jonow, Biotechnologia PWR, Semestr 3, Chemia fizyczna
Sprawko 1, Biotechnologia POLSL, Semestr IV, MBwOŚ, Laboratorium, Sprawozdana
Ćwiczenie 1 - oznaczanie stalej i stopnia dysocjacji, Biotechnologia PWR, Semestr 3, Chemia fizyczna
0 Ćwiczenie 4 II rok, Biotechnologia, III semestr, BKiIG, Instrukcje
0 Ćwiczenie 2 II rok + spraw, Biotechnologia, III semestr, BKiIG, Instrukcje
Sprawozdanie 8 - Auksyny, Biotechnologia POLSL, Semestr II, FiB laboratorium, Sprawozdania
0 Cwiczenie 6 II rok spraw, biotechnologia inż, sem3, BKiIG, laborki, sprawka
0 Cwiczenie 1 II rok spraw, biotechnologia inż, sem3, BKiIG, laborki, sprawka
cwiczene 5, II rok, II rok CM UMK, Giełdy, od Joe, histologia i embriologia, HISTOLOGIA, Materiały n
Sprawozdanie 4 - Optyka, Biotechnologia POLSL, Semestr II, FiB laboratorium, Sprawozdania
0 Cwiczenie 3 II rok spraw, biotechnologia inż, sem3, BKiIG, laborki, sprawka
Biochemia egzamin czesc II opracowanie dowydruku, Biotechnologia POLSL, Semestr V, Biochemia, Egzami
Ćwiczenie 1, II rok, Immunologia
cwiczenie 3, II rok, Wytrzymałość Materiałów
Zagadnienia z BIOCHEMII-oprac studentow z korekta KM, Biotechnologia POLSL, Semestr V, Biochemia, Eg
więcej podobnych podstron