
Budowa i wlasciwosci kwasow nukleinowych.
Kwasy nukleinowe zajmuja wyjatkowa pozycje wsrod czasteczek chemicznych tworzacych
materie ozywiona. Podwojny heliks DNA zawiera informacje o budowie bialek, a rozne
rodzaje RNA decyduja o tym, ze moze ona ulegac ekspresji. Efektem ekspresji informacji
genetycznej jest zlozona struktura bialkowa determinujaca funkcje komorek i calego
organizmu. Jednoczesnie, szczegolna struktura czasteczki DNA umozliwia precyzyjne
powielenie materialu genetycznego, bez ktorego nie byloby mozliwe rozmnazanie sie
organizmow oraz dziedziczenie cech.
Do kwasow nukleinowych zaliczamy:
Kwas deoksyrybonukleinowy - DNA,
Kwas deoksyrybonukleinowy - DNA.
DNA - kwas deoksyrybonukleinowy jest polinukleotydowa czasteczka, majaca postac
dlugiego nierozgalezionego podwojnego heliksu. Deoksyrybonukleotydy stanowiace
podjednostki DNA skladaja sie z zasady azotowej (jadnej z czterech), pentozowego
pierscienia cukru - deoksyrybozy oraz reszty kwasu ortofosforanowego.
Zasady
azotowe
wchodzace
w
sklad
nukleotydow
(nazwa
zwyczajowa
dla
deoksyrybonukleotydow) to: adenina, guanina, cytozyna lub tymina (oznaczane kolejno jako:
A,G,C i T). Pierwsze dwie sa zasadami purynowymi, natomiast dwie ostatnie to pirymidyny.
W polinukleotydowym lancuchu deoksyrybonukleotydy polaczone sa ze soba kowalencyjnie -
wiazaniami fosfodiestrowymi, wystepujacymi miedzy sasiadujacymi resztami cukrowymi.
Tworzy to uklad: cukier - reszta fosforanowa - cukier, stanowiacy glowna nic, od korej
odchylone sa zasady.
Ponizszy rysunek przedstawia budowe zasad azotowych oraz sposob polaczenia kolejnych
deoksynukleotydow
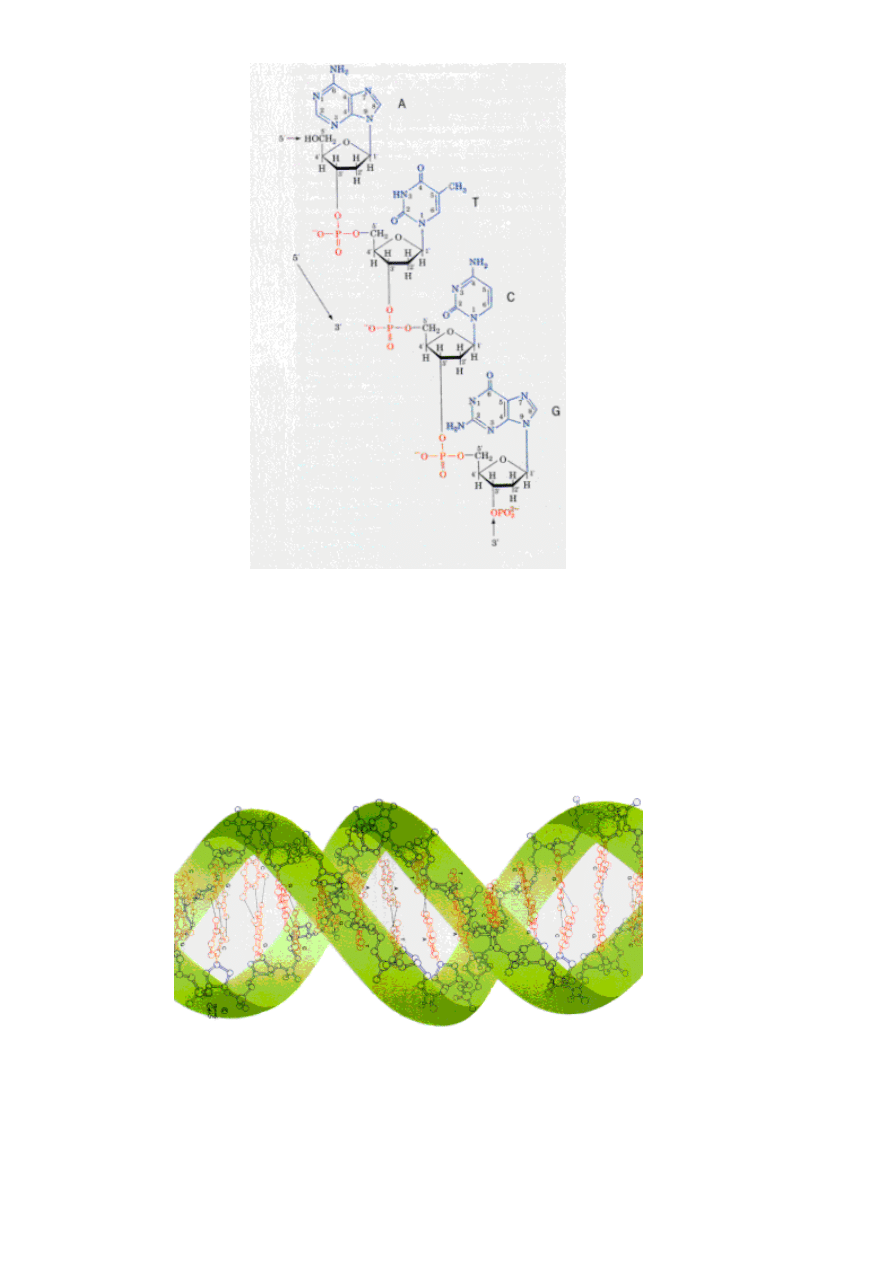
Rysunek 1
Konce polinukleotydowej nici nie sa rownocenne. Wynika to ze struktury wiazania
fosfodiestrowego w DNA. Reszta fosforanowa wiaze ze soba rozne atomy pierscienia
cukrowego, sa to atomy wegla zwane ze wzgledu na polozenie 3' i 5.' Analogicznie do nazw
atomow wegla, z ktorymi wiaza sie reszty fosforanowe nazywane sa konce nici. Wyrozniamy
wiec 3' koniec, na ktorym znajduje sie grupa -OH, dolaczona do wegla 3' oraz koniec 5' z
reszta fosforanowa, dolaczona do wegla 5'(patrz rysunek).
Kluczowe znaczenia dla funkcji biologicznych DNA ma jego struktura przestrzenna. Jak, juz
wspomniano czesteczka ta jest podwojnym heliksem, znaczy to, ze sklada sie z dwoch nici
polinukleotydowych owinietych wokol siebie.
Rysunek 2
Chrakterystyczne ulozenie obu nici wynika z wlasciwosci fizycznych ich poszczegolnych
elementow. W roztworze wodnym, stanowiacym srodowiska wewnetrzne komorki, silnie
hydrofobowe zasady azotowe ukladaja sie do wewnatrz, natomiast elementy hydrofilowe
jakimi
sa
reszty
fosforanowe
i
cukrowe
polozone
sa
zewnetrznie.
Wazne jest, ze nici tworzace heliks biegna w przeciwnych kierunkach, czyli po jednej stronie
znajduja sie konce 3' i 5'. Polaczenie polinukleotydowych nici odbywa sie przez wiazania
wodorowe miedzy zasadami azotowymi. Calosc czastki DNA mozna porownac do spiralnie
skreconej drabiny, gdzie szczeblami sa oddzialywujace ze soba zasady azotowe, zas
wspierajacymi je listwami deoksyrybozy polaczone wiazaniami fosfodiestrowymi.
Prawoskretne skrecenie calej struktury wynika z odleglosci pomiedzy poszczegolnymi
elementami, a liczba nukleotydow przypadajaca na pelny obrot heliksu wynosi ok. 10.5 (12
dla
sztucznie
syntetyzowanej
formy
lewoskretnej).
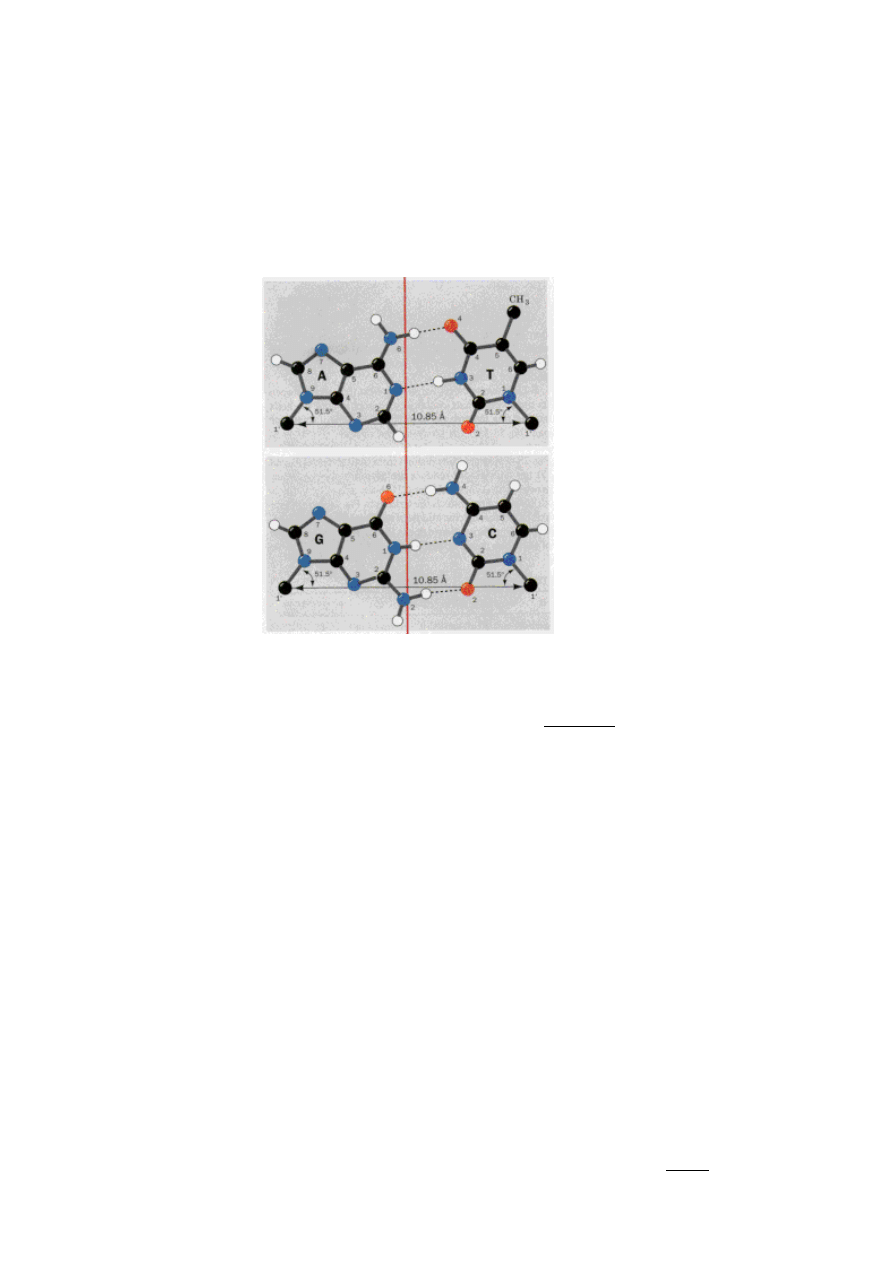
Strukture podwojnego heliksu DNA utrzymuja oddzialywania miedzy zasadami azotowymi.
Sa to oddzialywania specyficzne, adenina laczy sie z tymina, zas guanozyna z cytozyna (pary
A-T i G-C). Zasady tworzace para sa do siebie komplementarne. Ponadto dlugosci wiazan
wodorowych pomiedzy poszczegolnymi zasadami roznia sie tylko nieznacznie, przez co
srednica heliksu jest niemal jednakowa na calej dlugosci.
Rysunek 3
Komplementarnosc zasad azotwych jest istotna dla kopiowania informacji genetycznej.
Sekwencja jednej nici, niesie w sobie kolejnosc ulozenia nukleotydow w drugiej nici.
Rozplecenie i dobudowanie nici komplementarnych do lancuchow wyjsciowych wystarcza do
otrzymania dwoch idenycznych czasteczek DNA (Porownaj replikacja )
Kwas rybonukleinowy - RNA.
W komorce wystepuja rozne rodzaje RNA, a ich wielkosc i struktura przestrzenna jest na tyle
odmienna, ze kazdy z nich zasluguje na oddzielne omownienie. Niemniej jednak czesc
elementow budowy RNA jest wspolna dla roznych rodzajow i te zostana omowione na
wstepie.
Budowa chemiczna RNA jest zblizona do DNA. Monomerami polinukleotydowej czesteczki
sa nukleotydy, tyle, ze sa to rybonukleotydy. Znaczy to, ze w ich sklad wchodzi reszta
fosforanowa, zasada azotowa i ryboza, a nie jak w przypadku DNA 3'-deoksyryboza.
Rybonukleotydy sa polaczone ze soba wiazanimi fosodiesrowymi analogicznie do DNA.
Odmienny jest natomiast sklad zasad azotowych, wchodzacych w sklad RNA, wystepuje tu
adenina, guanina i cytozyna, lecz tymina zostala zastapiona uracylem.
Uracyl, zastepujac tymine, moze tworzyc pare komplementarna z adenina.
RNA mimo zdolnosci do tworzenia komplementarnych par zasad nie wystepuje w postaci
jednolitego podwojnego heliksu. Jednak zdolnosc do komplementowania pozwala na
tworzenie roznorodnych struktur. Jedna z nich jest tzw. struktura "szpilki do wlosow" (ang.
hairpin), inna, wystepujaca w tRNA, podobna do liscia koniczyny (Porownaj tRNA).

Kwasy rybonukleinowe ze wzgledu na pelnona funkcje biologiczna mozna podzielic na
szereg rodzajow, najwazniejszymi z nich sa:
mRNA (przekzywanie informacji, okreslajacej kolejnosc laczenia aminokwasow w
biosyntezie bialka),
tRNA (czasteczki przenoszace aminokwasy w procesie biosyntezy bialek i innych
szlakach anabolicznych),
rRNA (czasteczki biorace udzial w budowie rybosomu),
maloczasteczkowe RNA (czasteczki pelniace szereg funkcji w komorce, zwiazanych
z ekspresja materialu genetycznego).
Informacyjne RNA - mRNA.
Czasteczki mRNA naleza do najbardziej zroznicowanych pod wzgladem wielkosci i
stabilnosci. Ich funkcja jest przekazywanie informacji o strukturze bialek - stanowia matryce,
na ktorej polimeryzuja aminokwasy.
Czasteczka mRNA jest pojedyncza nicia polinukleotydowa, wystepujaca w roznych
postaciach pod wzgledem struktury przestrzennej (rzadko w postaci liniowej). Czynnikami
umozliwiajacymi zroznicowanie strukturalne mRNA jest wystepowanie wiazan wodorowych
oraz chemiczny charakter poszczegolnych elementow.
Na koncu 5' mRNA znajduja sie elementy, ktore decyduja o przylaczaniu sie rybosomu. W
komorkach prokariotycznych jest to specjalna sekwencja nukleotydowa (sekwencja Shine-
Dalgarno). U organizmow eukariotycznych natomiast, jest to charakterystyczna struktura
zwana "czpeczka" (ang. cap).
Kolejnym elementem mRNA jest tzw. ramka odczytu, czyli wlasciwa sekwencja kodujaca
okreslone bialko, ograniczona sygnalami start i stop dla translacji.
Koniec 3' mRNA u organizmow eukariotycznych ma dolaczony polimer zbudowany z 200-
250 nukleotydow adenylowych (tzw. ogon poliA).
Opisujac budowe mRNA nalezy wspomniec, ze w przypadku organizmow eukariotycznych
nie jest ono bezposrednim produktem transkrypcji na matrycy DNA. Forma dojrzalego
mRNA jest poprzedzona wystepowaniem jadrowego heterogennego RNA (hnRNA), ktore
dopiero po odpowiedniej obrobce (m.in. wycieciu intronow) staje sie gotowa matryca do
syntezy bialek.
Transferowe
RNA - tRNA.
Czasteczki tRNA stanowia lacznik w procesie przekazywania informacji zwartej w mRNA na
jezyk aminokwasow. Ich zadaniem jest dostarczenie odpowiednich aminokwasow do stynezy
bialka. Musza zatem miec miejsce odpowiedzialne za przylaczenie sie aminokwasu oraz
czesc rozszyfrowujaca informacje niesiona przez mRNA (patrz translacja).
Czasteczki tRNA maja dlugosc od 74 do 95 nukleotydow, Wewnetrzna komplementarnosc
zasad pozwala na tworzenie charakterystycznej struktury, podobnej do liscia koniczyny. Lecz
nie jest to idealny "lisc koniczyny", gdyz obok czterech duzych ramion wystepuje dodatkowo
mniejsze, zwane ramieniem dodatkowym (to male ramie jest czesto podstawa do klasyfikacji
tRNA).
Jedno z czterech (a wlasciwie pieciu) ramion w tRNA, zwane ramieniem akceptorowym,
stanowi miejsce przylaczania sie aminokwasow. Laczenie odpowiednich aminokwasow ze
swoistymi tRNA ma charakter enzymatyczny i katalizowane jest przez odpowiednie enzymy

zwane aminoacylo-tRNA syntetazami - reakcje te wymagaja energii z ATP. Produkt
otrzymany po dolaczeniu aminokwasu do tRNA nazywany jest aminoacylo-tRNA.
W kazdej komorce istnieje przynajmniej 20 rodzajow czasteczek tRNA, poniewaz kazdemu
aminokwasowi musi odpowiadac co najmniej jedno tRNA. Zwykle liczba tRNA,
odpowiadajaca danemu aminokwasowi jest wieksza od jednego (patrz kod genetyczny).
Na przeciw ramienia akceptorowego lezy ramie antykodonowe . Na jego koncu znajduje sie
trojka nukleotydow, ktora rozpoznaje komplementarna trojke na nici mRNA podczas
translacji (patrz translacja). Ten nukleotydowy triplet na czasteczce tRNA nazwany zostal
antykodonem.
Wiadomo, ze w sklad jednoniciowego lancucha tRNA wchodzi wiele zmodyfikowanych
zasad azotowych. Modyfikacje te polegaja na dolaczeniu roznych grup funkcyjnych i
dokonywane sa w procesie dojrzewania (obrobki posttranskrypcyjnej).
Rybosomalne RNA - rRNA.
rRNA jest to czasteczka, ktora wraz ze specyficznymi bialkami buduje rybosom. W sklad
duzej i malej podjednostki rybosomu wchodza rozne rodzaje rRNA, ktore w wiekszosci
przypadkow powstaja na terenie jadra w wyniku przeksztalcenia prekursorowej czasteczki
(wyjatkiem jest 5s rRNA, ktory powstaje z innej czasteczki prekursorowej). rRNA tworzy
specyficzne strukury przestrzenne, w ktorych czesc czasteczki wystepuje w postaci
sparowanej , tworzac uklad przypominajacy szpilke do wlosow (ang. hair-pin), a pozostale
odcinki wystepuja w postaci jednoniciowej. W przyblizeniu czasteczka rRNA rybosomowego
wyglada jak klebek, w ktory powbijano szpilki.
Na uwage zasluguje fakt, ze oprocz rRNA wchodzacego w sklad rybosomu na terenie
komorki wystepuje wiele innych rodzajow rRNA, pelniacych rozne funkcje W sumie rRNA
stanowi ok. 90% calego RNA komorkowego.
Maloczasteczkowe RNA
Czasteczki maloczasteczkowego RNA moga pelnic rozmaite funkcje w komorce, glowne z
nich to:
wycinanie intronow,
potranskrypcyjna obrobka pre-mRNA,
udzial w terminacji transkrypcji niektorych genow.
Kwasy nukleinowe jako material dziedziczny
Calosc kwasu deoksyrybonukleinowego (DNA), zawierajacego informacje genetyczna
organizmu nazywamy genomem.
Ze wzgledu na istotne roznice, zarowno pod wzgledem budowy, jak i wielkosci, postac w
jakiej wystepuje DNA zostanie omowiona oddzielnie dla:
Struktura genomu organizmow prokariotycznych
BAKTERIE
DNA bakterii wystepuje w postaci dlugiej, kolistej helisy, nieoslonietej bialkami czasteczki.
Jednakze nie jest to struktura zupelnie naga, nawinieta jest bowiem na rdzen zbudowany z
RNA i bialek (bialka te sa podobne do histonow, wystepujacych w chromosomach

organizmow wyzszych). W odroznieniu do komorek eukariotycznych, prokariota nie maja
oddzielnego przedzialu komorkowego, ktory oddzielalby material genetyczny od elementow
cytoplazmatycznych.
Strukture w jakiej wystepuje genom bakteryjny okresla sie czesto mianem genoforu. Jej
dlugosc
miesci
sie
w
granicach
od
700tys.
do
9.5
mln
par
zasad.
Genofor bakterii roznie sie od chromosomu Eukaryota jeszcze jedna wlasciwoscia. Geny
kodujace bialka stanowia tylko maly procent DNA chromosomalnego u eukariota. Reszta
przypada na powtarzajace sie sekwencje nukleotydow, ktorych funkcja nadal nie jest znana. U
bakterii natomiast prawie caly DNA genoforu to geny kodujace.
Poza glowna nicia DNA u wiekszosc Prokariota wystepuja male, kolisto zamkniete struktury
podwojnej helisy zwane plazidami.
WIRUSY
Wirusy sa czastkami zlozonymi z bialek i kwasow nukleinowych. Genom wirusow moze
moze byc budowany przez:
czasteczki DNA w postaci podwojnej helisy
czasteczki DNA w postaci jednoniciowej
czasteczki RNA
Nieznane sa przypadki, aby oba kwasy jednoczesnie wchodzily w sklad genomu wirusow.
Kwasy te sa zamkniete w bialkowa otczke zwana kapsydem. Kapsyd w wiekszosci
przypadkow sklada sie z kilku rodzajow bialek. Genom wirusow musi zawierac co najmniej 3
geny:
kodujacy bialko kapsydu,
kodujacy polimeraze (enzym odpowiedzialny za powielenie wirusowych kwasow
nukleinowych),
kodujacy bialko odpowiedzialne za przylaczanie sie czastki wirusa do komorki
gospodarza.
Ale trojgenowy genom wystepuje tylko u najprostrzych czastek wirusowych. Bardziej
skomplikowane maja duzo wiecej genow, np. bakteriofag T4 ok. 150, a wirus ospy ok. 300.
Na uwage zasluguje fakt, ze niektore wirusy, atakujace komorki bakteryjne, tzw. bakteriofagi
(lub w skrocie po prostu fagi), w swych genomach maja specjalne sekwencje nukleotydowe,
ktore pozwalaja im na wlaczanie sie do genoforu bakteryjnego. Sekwencje te wykazuja
homologie (podobienstwo w kolejnosci nukleotydow) z niektorymi fragmentami DNA
bakterii, co pozwala im na latwa integracje.
Struktura genomu organizmow eukariotycznych
Struktura w jakiej wystepuje genom organizmow eukariotycznych jest znacznie bardziej
skomplikowana w porownaniu z prokariontami. Glowna tego przyczyna sa roznice w jego
wielkosci. Wieksze rozmiary DNA eukariontow powoduja koniecznosc mocniejszego
upakowania czasteczki. Nawiniecie jej na bialkowy rdzen oraz skrecenie, ulatwia dokladne
rodzielenie ogromnej nici DNA. Strukura, w ktorej DNA zwiazany jest z bialkami, a
nastepnie kilkukrotnie poskrecany nosi nazwe chromosomu.
Bialka, na ktore nawiniete jest DNA to histony. Histony wraz z DNA tworza podstawowa
strukture chromosomu zwana nukleosomem . Nukleosom to czastka o ksztalcie dysku, ktorej
bialkowa czesc buduje 8 czastek histonowych (po dwie z typu: H2A, H2B, H3, H4). Na taki
proteinowy rdzen nawinieta jest nic DNA o dlugosci ok 170 nukleotydow. Dodatkowo, obok
wpomnianego oktameru wystepuje histon H1, ktory odgrywa znaczaca role w dalszym
upakowaniu DNA

struktura nukleosomu
Rozwiniety chromosom eukariotyczny przypomina sznur korali, jednakze nic DNA nie
przebiega w srodku nukleosomow lecz jest na niego nawinieta. W skondensowanym
chromosomie (metafazalnym) wspomniany sznur korali jest skrecony, tworzac tzw. solenoid.
Rysunek 1
Solenoid moze ulegac dalszemu skrecaniu az do osiagniecia postaci postaci maksymalnie
skondensowanej przedstawionej na rysunku struktura chromosomu metafazalnego
Lecz chromosom w takiej postaci nie wystepuje przez caly cykl komorkowy. W interfazie
DNA, wystepujace na terenie jadra komorkowego ma postac mniej lub bardziej zwinietej nici
zwiazanej z bialkami matrix jadrowej (histonowymi i niehistonowymi). Takia postac DNA
jadrowego nazwano chromatyna W zaleznosci od stopnia skondensowania, chromatyne
dzielimy na:
euchromatyne - forma luzna,
heterochromatyne - forma skondensowana .
Na uwage zasluguje fakt, ze mocne upakowanie DNA uniemozliwia jego transkrypcje. Aby
moglo dojsc do ekspresji informacji genetycznej DNA musi wystepowac u luznej postaci
(euchromatyny).
Niewielka czesc informacji dziedzicznej eukariontow znajduje sie poza jadrem komorkowym,
w postaci niewielkich czasteczek DNA zawartych w organellach komorkowych. Organellami
z wlasnym genomem sa:
mitochondria,
chloroplasty, w przypadku roslin fotosyntetyzujacych.
Wlasciwosc ta decyduje o pewnej autonomicznosci tych organelli. Jednak nie sa one w pelni
niezalezne od informacji zawartej w aparacie jadrowym. Czesc bialek mitochondrialnych i
chloroplastowych eksprymowana jest dzieki DNA jadrowemi, a nastepnie bialka te sa
transportowane do odpowiednich organelli.
Replikacja DNA
Replikacja DNA jest procesem zapewniajacym przekazywanie informacji genetycznej z
komorek rodzicielskich do komorek potomnych w sposob prawie doskonaly. Doskonalosc ta
jest uzyskiwana nie tylko dzieki precyzyjnemu mechanizmowi samej replikacji, lecz rowniez
za sprawa systemow ochronnych, zdolnych do wykrywania i naprawy bledow. Dzieki tym
systemom bledy powstale w procesie replikacji pojawiaja sie najwyzej raz na miliard
nukleotydow.
Bez wzgledu na to, czy komorka ma tylko jeden chromosom, czy tez ma ich wiele, DNA
ulega replikacji zawsze tylko jeden raz na kazdy cykl podzialowy .
Podstawowa cecha replikacji jest jej semikonserwatywnosc. Znaczy to, ze synteza nowych
nici moze zachodzic tylko z udzialem nici rodzicielskich, sluzacych jako matryce. W procesie
tym wykorzystuje sie zdolnosc zasad azotowych, wchodzacych w sklad nukleotydow, do
tworzenia komplementarnych par. Dzieki tej wlasnie wlasnosci wystarczy rozplatac
podwojny heliks DNA, nastepnie do uwolnionych w ten sposob nici dobudowac
komplementarne nukleotydy, aby otrzymac dwie identyczne czastaczki DNA (kazda z
otrzymanych w ten sposob czastek bedzie miala jedna nic rodzicielska i jedna
dosyntetyzowana na nowo).
Mowiac o replikacji nalezy wprowadzic pojecie replikonu. Replikon jest to jednostka
replikacji, za ktora przyjeto uwazac odcinek DNA, zawierajacy miejsce startu oraz
przylegajace sekwencje uczestniczace w kontroli tego prosesu.
W procesie replikacji mozna wyroznic trzy zasadnicze etapy:
Inicjacja replikacji
Inicjacja, czyli poczatek replikacji zachodzi w scisle okreslonych miejscach na nici DNA.
Miejsca te nazywaja sie "origin" (w skrocie ori). Zawieraja one specyficzne sekwencje,
sluzace im do:
wiazania bialek inicjatorowych,
rozdzielenia nici i rozpoczecia procesu replikacji,
przylacznia bialek wspomagajacych proces replikacji.
Miejsce inicjacji jest bardzo wazne, poniewaz tylko tu moze odbywac sie kontrola repikacji.
Raz rozpoczety proces musi przebiegac az do zakonczenia syntezy calego replikonu. U
bakterii replikon obejmuje caly genofor, znaczy to, ze pojedyncze miejsce inicjacji kontroluje
replikacje calego genomu. Organizmy eukariotyczne natomiast, maja wiele replikonow w
kazdym chromosomie. Dzieki tej roznicy szybkosc replikacji genomu eukariota wielokrotnie
przewyzsza szybkosc replikacji genomu prokariota.
Rozdzielajace sie w miejscu syntezy lancuchy matrycowego DNA tworza tzw. "widelki
replikacyjne". O rozdzieleniu heliksu DNA decyduja bialka enzymatyczne zwane
helikazami. Wykorzystuja one energie hydrolizy ATP do zmiany ksztaltu swojej czasteczki
(podobnie jak bialka miesniowe), co umozliwia im wykonanie pracy mechanicznej. Helikazy
przesuwaja sie wzdluz dwuniciowego DNA, rozdzielajac nici i poszerzajac widelki
replikacyjne.

Najwazniejszymi enzymami odpowiedzialnymi za replikacje DNA sa polimerazy DNA. Nie
maja one jednak zdolnosci do samodzielnego rozpoczecia syntezy lancuchow DNA, moga
jedynie je wydluzac. Z tego powodu synteza DNA zaczyna sie od wbudowywania
starterowych odcinkow RNA, ktore zakonczone sa wolna grupa -OH, na koncu 3'. Za
wbudowywanie tych odcinkow odpowiedzialne sa wyspecjalizowane enzymu zwane
primazami.
Zatem kolejne etapy inicjacji replikacji to:
przylaczenie sie helikaz do odpowiednich sekwencji inicjatorowych,
rozplatanie podojnego heliksu i powstanie widelek replikacyjnych,
synteza starterowych odcinkow RNA przez primazy,
Elongacja replikacji
Porces elongacji, podobnie jak proces inicjacji, wymaga udzialu wielu bialek
enzymatycznych. Oprocz polimeraz DNA, glownych enzymow replikacji, potrzebnych jest
wiele
enzymow
odpowiedzialnych
za
katalizowanie
reakcji
analogicznych
do
przeprowadzanych podczas inicjacji replikacji (rozplatywanie i stabilizacja nici,
wbudowywanie starterow). Wszystkie te bialka tworza kompleks zawany replisomem.
Polimerazy syntetyzujace potomne nici DNA nie tylko nie sa zdolne do rozpoczecia replikacji
(patrz inicjacja replikacji), lecz rowniez przylaczanie przez nie nukleotydow moze sie
odbywac wylacznie do grup -OH (konca 3') starterow RNA. Zatem replikacja DNA przebiega
tylko w jednym kierunku - od 5' do 3' konca. Odwrotna orientacja nici w obrebie podwojnego
heliksu pozwala na ciagla synteze tylko jednej nici (tzw. nici wiodacej). Druga natomiast
musi byc w postaci krotkich fragmentow , dolaczonych do odcinkow starterowych (jest to
tzw. nic opozniona). Fragmenty te nazwano, na czesc odkrywcy, fragmentami Okazaki.
Wytworzenie wiazania estrowego miedzy reszta fosforanowa a deoksyryboza, katalizowane
przez polimerazy, wymaga dostarczenia energii. Jak w wiekszosci syntez komorkowych, tak i
tu energia pochodzi z wysokoenergetycznych wiazan miedzy resztami kwasu fosforowego.
Czasteczka DNA jest zbudowana z monofosfonukleotydow. Natomiast substratami do synezy
nowej nici w procesie replikacji sa trifosfonukleotydy (ATP, CTP, GTP, TTP), wiec
dolaczenie
jednego
nukleotydu
wiaze
sie
z
rozerwaniem
dwoch
wiazan
wysokoenergetycznych.
W fazie elongacji procesu replikacji bierze udzial kilka polimeraz, roznizcych sie
funkcjonalnie:
polimeraza I - jej rola jest usuwanie odcinkow starterowych z fragmentow Okazaki
oraz wycinanie uszkodzonych odcinkow DNA w procesie naprawy,
polimeraza II - niestety funkcja metaboliczna tego enzymu nie jest dokladnie
poznana, wiadomo natomiast, ze wykazuje aktywnosc egzonukleolityczna,
polimeraza III - jest wlasciwa replikaza DNA, jej glowna funkcja jest wstawianie
nowych nukleotydow na koncu 3' nowopowstajacej nici DNA.
Funkcje polimeraz omowiono na podstawie organizmow prokariotycznych, lecz ze wzgledu
na podobienstwo tych funkcji wzgledem eukariontow nie zachodzi potrzeba oddzielnego ich
omowienia. Na uwage zasluguje jednak fakt, ze organizmy eukariotyczne maja dodatkowa
polimeraze, odpowiedzialna za replikacje DNA jadrowego.
Kolejnymi, nie wspomnianymi dotad, enzymami koniecznymi do przeprowadzenia procesu
replikacji przez komorke sa ligazy. Ich zadaniem jest laczenie fragmentow Okazaki (juz po
wycieciu starterow przez polimeraze) w jedna dluga nic. Ligazy bakteryjne wymagaja NAD

jako koenzymu i zrodla energii, natomiast ligazy eukariotyczne wykorzystuja do tego celu
ATP.
Podsumowujac - na proces elongacji skladaja sie:
synteza fragmentow Okazaki, z wykorzystaniem odcinkow starterowych,
naprawa ewentualnych bledow, powstalych podczas trwania procesu,
wyciecie fragmentow RNA, sluzacych za startery,
laczenie (ligacja) odcinkow Okazaki w jedna dluga nic.
Terminacja replikacji
Terminacja jest koncowym etapem replikacji, w ktorym dochodzi do polaczenia DNA w
kompletne chromosomy i rozdzielenia ich pomiedzy komorki potomne. Mechanizmy
terminacji u prokaroiota i eukariota znacznie roznia sie sie od siebie.
U organizmow prokariotycznych, gdzie na kolistym genoforze znajduje sie tylko jeden
replikon, za koncowy etap replikacji odpowiedaja 4 sekwencje nukleotydowe , wiazace sie z
bialkiem terminacyjnym. Bialko to bedac inhibitorem replikacji, hamuje caly proces.
U eukariontow sytuacja przedstawia sie inaczej. Wystepowanie kilku repikonow w
replikujacym sie chromosomie powoduje, ze do zakonczenia replikacji wystarczy fizyczne
zetkniecie sie dwoch podazajacych ku sobie w przeciwnych kierunkach widelek
replikacyjnych.
Nie
potrzeba
tu
specjalnych
sekwencji
terminalnych.
Odmiennie przedstawia sie sytuacja z zakonczeniem syntezy chromosomow. W
przeciwienstwie do kolistych genoforow bakteryjnych, chromosomy eukariotyczne
zbudowane sa z liniowych czasteczek DNA. Gdyby replikacja u organizmow eukariotycznych
konczyla sie analogicznie do prokariota to ostatni kilkunukleotydowy fragment pozostawalby
niedoreplikowany. Na koncu kazdego chromosomu istnieja jednak charakterystyczne
sekwencje, ktore pozwalaja na utrzymanie niezmiennej dlugosci genomow. Sekwencje te
zwane sa telomerami. Telomery odpowiadaja rowniez za ochrone DNA przed laczeniem sie
z
innymi
chromosomami.
Replikacja telomerow zachodzi w sposob odmienny od reszty chromosomu. Odpowiada za
nie specjalny enzym zwany telomeraza. Jest to enzym, ktory w swoim centrum aktywnym
zawiera matryce do syntezy telomerow - czasteczke RNA. Starterami w tym procesie sa
koncowe sekwencje telomeru pozostalego z poprzedniego cyklu replikacyjnego.
Zatem na terminacje replikacji skladaja sie nastepujace procesy:
zakonczenie syntezy nowych nukleotydow w odpowienich miejscach,
polaczenie powstalego DNA w kompletne chromosomy,
rozdzielenie chromosomow pomiedzy komorki potomne.
Ekspresja informacji genetycznej
Informacja o budowie organizmu jest zapisana w DNA, te informacje nazywamy genotypem.
Aby genotyp ujawnil sie w postaci obserwowalnych cech fenotypowych, informacja
dziedziczna musi przebyc droge od DNA do bialek - czyli ulec ekspresji. O tym, ze DNA
koduja bialka, a nie inne zwiazki zadecydowalo prawdopodobnie ich szerokie spektrum
funkcjonalne. Bialka stanowiac ponad polowe suchej masy komorek, wchodza w sklad prawie
wszystkich struktur komorkowych. Dzieki aktywnosci enzymatycznej decyduja o przebiegu
reakcji chemicznych, dodatkowo zdolnosc bialek do zmian konformacyjnych powoduje, ze
moga one spelniac wiele innych funkcji.

Podczas ekspresji informacja genetyczna jest dwukrotnie przeksztalcana:
po raz pierwszy w procesie transkrypcji, ktory polega na przepisaniu informacji z
DNA na RNA z wykorzystaniem reguly komplementarnosci,
po raz drugi podczas translacji, czyli przetlumaczeniu sekwencji nukleotydow na
sekwencje aminokwasow.
Przplyw informacji w komorce mozna zatem zapisac w nastepujacy sposob:
Zapis ten byl prawidlowy az do momentu wykrycia retrowirusow. Retrowirusy maja bardzo
charakterysyczna ceche - ich materialem genetycznym jest RNA. Co wiecej informacja
zawarta w RNA tych wirusow moze byc przepisywana na DNA za pomoca specjalnego
enzymu zwanego odwrotna transkryptaza. Wlasnie wykrycie odwrotnej transkryptazy
zlamalo obowiazujacy dogmat o przeplywie informacji genetycznej, zatem przedstawiony
wyzej schemat nalezy zmodyfikowac do nastepujacej postaci:
Transkrypcja
Transkrypcja jest pierwszym etapem ekspresji genow, polega na "przepisaniu" kolejno zasad
z DNA na RNA. Proces ten jest katalizowany przez polimeraze RNA - enzym, ktory
przyczepia sie do DNA na poczatku genu i syntetyzuje RNA komplementarne do jednej z
nici. Tak powstaly RNA (po dodatkowej obrobce) w pozniejszym etapie ekspresji materialu
genetycznego stanowi matryce, na ktorej syntetyzowane sa bialka. Transkrypcja, obok
translacji, jest zatem kluczowym procesem warunkujacym zycie organizmow. Dzieki niej
mozliwe jest odczytanie informacji genetycznej, zachowujac jednoczesnie nienaruszalnosc
centralnego banku informacji - podwojnej helisy DNA.
U organizmow prokariotycznych i eukariotycznych transkrypcja rozni dosc znacznie. Roznica
ta przejawia sie glownie w pierwszym etapie - inicjacji. Ze wzgledu na to proces ten zostanie
omowiony oddzielnie dla obu tych nadkrolestw.
Transkrypcja u organizmow prokariotycznych
Proces transkrypcji u Prokaryota rozpoczyna sie od zwiazania sie polimerazy RNA
(glownego enzymu katalizujacego) do odpowiedniej sekwencji na DNA, zwanej
promotorem.
Promotory sa podobne we wszystkich genach organizmow prokariotycznych. W ich budowie
istotne sa dwie, zwykle szescionukleotydowe sekwencje. Jedna z nich - polozona blizej konca
5', jest pierwsza rozpoznawana przez polimeraze.
Polimeraza RNA jest enzymem skladajacym sie z czesci rdzeniowej, zbudowanej z trzech
roznych elementow bialkowych, oraz podjednostki odpowiedzialnej za rozpoznawanie i
wiazanie sie do sekwencji promotorowych. Podjednostka ta nazywana zostala czynnikiem
sigma. W komorkach prokariotycznych istnieje wiele takich czynnikow. Wykazuja one
specyficznosc dla okreslonych promotorow i dzieki tej wlasnosci moga wplywac na regulacje
ekspresji genow.
Aby moglo dojsc do rozpoczecia syntezy RNA, nie wystarcza zwiazanie polimerazy RNA z
elementami promotorowymi.Konieczne jest rowniez przejscowe rozdzielenie podwojnego
lancucha DNA (proces ten nazwano topnieniem).
Wszystkie wyzej opisane procesy, czyli:
rozpoznanie promotorow przez czynnik sigma,

przylaczenie polimerazy RNA do elementow promotorowych,
miejscowe rozdzielenie helisy DNA.
skladaja sie na tzw. inicjacje traskrypcji.
Kolejnym etapem trasnkrypcji jest elongacja. W czasie jego trwania polimeraza RNA
przesuwa sie po nici DNA, syntetyzujac RNA. Substratmi w tym procesie sa rybonukleotydy
(ATP, GTP, CTP i UTP). Energia zmagazynowana w ich wysokoenergetycznych wiazaniach
jest wykorzystywana do wytwarzania wiazan fofodiestrowych w RNA. W trakcjie elongacji
powstaje przejsciowa struktura hybrydowa DNA-RNA.
Omawiajac proces elongacji warto dokladniej przeanalizowac jedena czesto umykajacy
uwadze prawidlowosc dotyczaca calego procesu transkrypcji.
Czasteczka DNA sklada sie z dwoch pojedynczych nici. Jedna z nich jest wlasciwa
nicia kodujaca bialka (tzw. nic sensowna), natomiast droga, komplementarna do niej,
to nic niekodujaca - nonsensowna. Taka struktura DNA pozwala nie tylko na latwe
kopiowanie DNA (patrz replikacja), jak i na przepisywanie informacji na RNA.
Podczas transkrypcji powstajaca nic RNA jest komplementarna tylko do jednej nici
DNA - nonsensownej (inaczej mowiac nic nonsensowna jest matryca). Dzieki temu
nowo powstale RNA jest, co do kolejnosci nukleotydow, takie same jak nic kodujaca
(nie zapominajac o tym, ze zamiast tyminy, w RNA wystepuje uracyl).
Transkrypcja konczy sie w scisle okreslonym miejscu DNA zwanym terminatorem. Tu
zachodzi oddysocjowanie polimerazy RNA od DNA. Istnieja dwa mechanizmy terminacji
transkrypcji.
Transkrypcja u organizmow eukarotycznych
W odroznieniu od organizmow prokariotycznych, u organizmow eukariotycznych istnieja trzy
rodzaje polimerazy, odpowiedzialne za "odczytywanie" roznych klas genow.
Nazwa
Produkty transkrypcji
Polimeraza I
rozne rodzaje rybosomalnego RNA (rRNA)
Polimeraza II
matrycowe RNA (mRNA)
maloczasteczkowe RNA
Polimeraza III
niektore rodzaje maloczasteczkowego RNA
transferowe RNA (tRNA)
Zaden z tych enzymow nie potrafi samodzielnie przylaczac sie do DNA. Polaczenie takie jest
mozliwe jedynie dzieki specjalnym bialkom tzw. czynnikom transkrypcyjnym (w skrocie
TF; ang. Transcripton factor). Czynniki te lacza sie z DNA w specjalnych miejscach zwanych
promotorami. Dla wielu genow elementem niezbednym do przylaczenia sie pierwszego
czynnika transktypcyjnego jest sekwencja TATA (ang. TATA-box). Incjacja (lub regulacja)
genow nie zachodzi tylko przy udziale sekwencji promotorowych, czynniki transkrypcyjne
moga wiazac sie rowniez do tzw. sekwencji enhancerowych. Enhancery nie musza byc
polozone obok sekwencji kodujacej jak to jest w przypadku promotorow, moga byc oddalone
od niej nawet o 1000 par zasad lub znajdowac sie w intronie. Ich orientacja rowniez nie ma
znaczenia, enhancer funkcjonuje poprawnie nawet po odwroceniu go o 180 stopni. Wzajemne
oddzialywanie czynnikow transkrypcyjnych, przylaczonych do promotorow i enhancerow,
bedacych zwykle aktywatorami, decyduje o rozpoczeciu pracy przez polimeraze RNA.
Dalszy etap transkrypcji, czyli elongacja, przebiega podobnie jak u organizmow
prokariotycznych. .

Rozny jest natomiast ostatni etap traskrypcji - terminacja. U Eukaryota nie stwierdzono
czynnikow uwalniajacych, inne sa rowniez sekwencje odpowiedzialne za zakonczenie
transkrypcji. Jednakze brak danych na temat terminacji nie pozwala na dokladna
charakterystyke miejsc konca transkrypcji.
Produktem transkrypcji u organizmow eukariotycznych, katalizowanej przez polimeraze RNA
II, nie jest gotowa matryca RNA (mRNA), jest nim czasteczka prekursorowa zwana pre-
mRNA. Ze wzgledu na nieciaglosc genow eukariotycznych i wystepowanie oddzielnego
przedzialu jadrowego, zsyntetyzowany transkrypt musi ulec dodatkowym przemianom (patrz
posttranskrypcyjne etapy ekspresji genu eukariotycznego).
Posttranskrypcje etapy ekspresji genow eukariotycznych
Koniecznosc posttranskrypcyjnej obrobki materialu zawierajacego informacje genetyczna
spowodowane jest dwoma cechami organizmow eukariotycznych:
Wiekszosc genow Eukaryota ma nieciagly chrakter, znaczy to, ze skladaja sie one z
naprzemiennie wystepujacych obszarow kodujacych - eksonow oraz niekodujacych
intronow. Procesowi transkrypcji ulegaja zarowno introny, jak i eksony, ale w sklad
funkcjonalnego mRNA nie moga wchodzic elementy niekodujace. Musi zatem istniec
mechanizm umozliwiajacy ich wycinanie.
Ponadto, w przeciwienstwie do Prokaryota procesy transkrypcji i translacji sa
rozdzielone w czasie i przestrzeni. Matrycowe RNA powstaje na terenie
kompartmentu jadrowego, natomiast translacja zachodzi w cytoplazmie. W zwiazku z
tym musza istniec struktury umozliwiajace transport mRNA do cytoplazmy.
Produktem transkrypcji genu jest zawierajaca introny i niezdolna do eksportu z jadra
czasteczka prekursorowego mRNA (pre-mRNA). Taki pierwotny transkrypt musi ulec
trzem procesom, aby stac sie matryca do translacji.
Na koncu 5' dosyntetyzowana musi byc struktura odpowiedzialna za przylaczenie sie
rybosomu, tzw. czapeczka (od ang. cap proces ten nazwano capping). Czapeczka jest
zbudowana z trzech reszt fosforanowych oraz zmetylowanej guaniny.
Koniec 3' formowany jest przez specjalny kompleks bialkowy. W sklad tego
kompleksu wchodzi poliA polimeraza, ktorej zadaniem jest dosyntetyzowanie "ogona"
zlozonego z samych nukleotydow adenylowych, tzw. poliA ogon. Taka
poliadenylacja jest niezbedna do przetransportowania mRNA do cytoplazmy,
jednoczesnie chroniac je przed degradacja. Poliadenylacja jest uwatunkowana
wystepowaniem specyficznych sekwencji na 3' koncu pre-mRNA.
Najbardziej skomplikowanym procesem podczas "dojrzewania" mRNA jest wycinanie
intronow, czyli tzw. splicing. W wyciananiu intronow bierze udzial specjalny
kompleks bialkowy zwany splicesomem. Proces ten polega na odpowiednim
wypetleniu intronow z udzialem endonukelaz i polaczeniu eksonow w jedna nic.
Laczonie eksonow katalizowane jest ligazami. Istnieje kilka mechanizmow usuwania
intronow, m.in. samowycinanie (ang. self-splicing). W przypadku samowycinania role
katalizatorow moga pelnic czasteczki mRNA, zdolne do przyjmowania odpowiednich
konformacji
Kod genetyczny
Ekspresja materialu dziedzicznego jest uwarunkowana istnieniem sposobu wedlug, ktorego
mozna przetlumaczyc informacje genetyczna zawarta w sekwencji nukleotydow na sekwencje
aminokwasow. W polowie lat piedziesiatych zwrocono uwage na pewna zaleznosc
matematyczna dotyczaca kodowania informacji genetycznej. Skoro informacja na temat
bialek zapisana jest w postaci DNA, a czasteczka ta sklada sie z czterech roznych
nukleotydow, to do zapisania w niej informacji dotyczacej 20 elementow (tyle wlasnie
aminokwasow buduje bialka), potrzeba jednostek kodujacych zlozonych z trzech
nukleotydow. Pogrupowanie czterech nukleotydow z roznymi zasadami w dowolne trojki,
pozwala na otrzymanie 64 kombinacji. Ewentualne ulozenie ich w dwojki, pozwoliloby
jedynie na uzyskanie 16 kombinacji, natomiast z ukladu czworkowego wynikloby 256, co jest
wielkoscia zbyt duza. Zatem przyroda wybrala mozliwie najprostrza wersja zapisu
chemicznego:
Proste permutacje czterech nukleotydow zgrupowanych w trojki, daja mozliwosc
zakodowania wszystkich 20 aminokwasow.
Trojke, ktorej odpowiada jeden aminokwas nazwano kodonem.
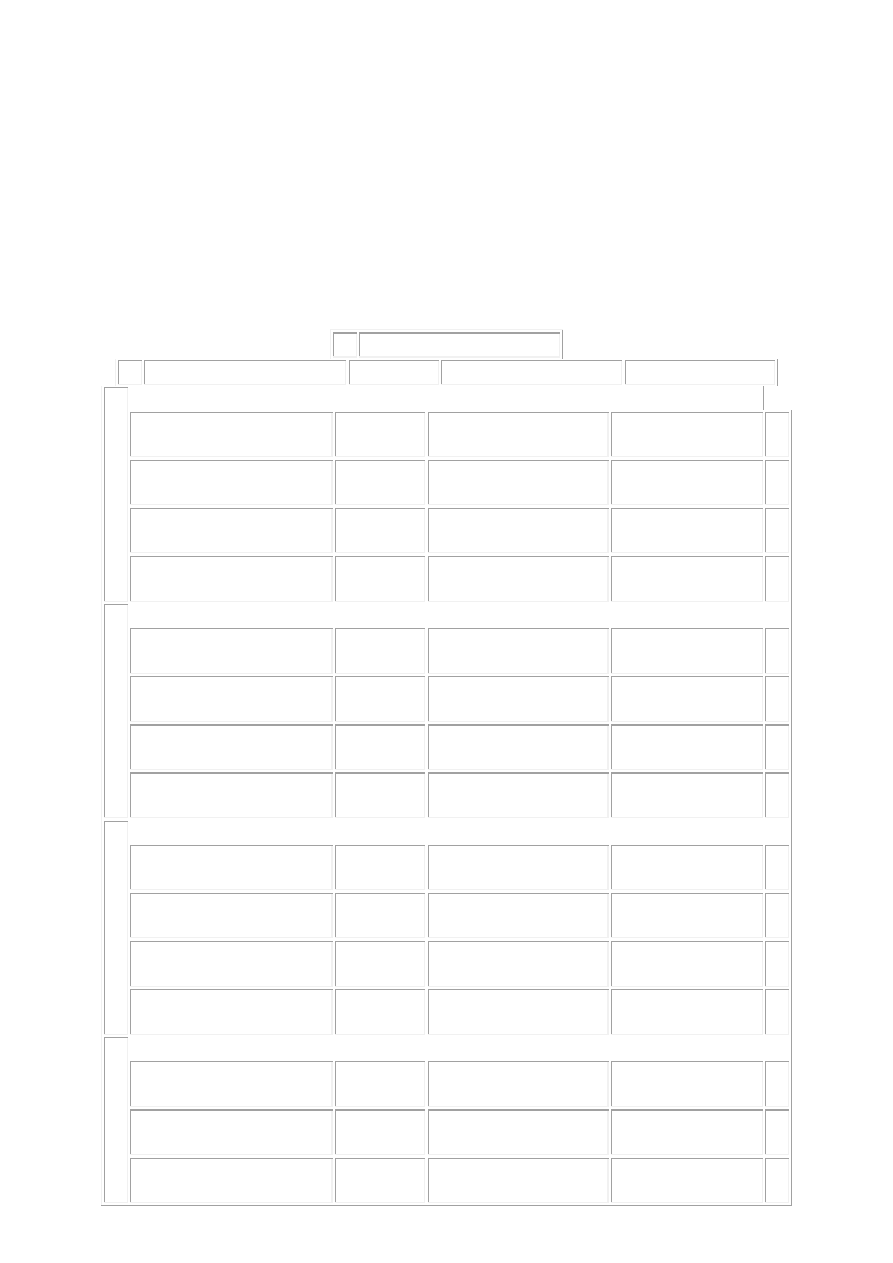
Tabela kodu genetycznego.
U
C
A
G
U
UUU
Fenyloalanina
UCU
Seryna
UAU
Tyrozyna
UGU
Cysteina
U
UUC
Fenyloalanina
UCC
Seryna
UAC
Tyrozyna
UGC
Cysteina
C
UUA
Leucyna
UCA
Seryna
UAA
Stop translacji
UGA
Stop translacji
A
UUG
Leucyna
UCG
Seryna
UAG
Stop translacji
UGG
Tryptofan
G
C
CUU
Leucyna
CCU
Prolina
CAU
Histydyna
CGU
Arginina
U
CUC
Leucyna
CCC
Prolina
CAC
Histydyna
CGC
Arginina
C
CUA
Leucyna
CCA
Prolina
CAA
Glutamina
CGA
Arginina
A
CUG
Leucyna
CCG
Prolina
CAG
Glutamina
CGG
Arginina
G
A
AUU
Izoleucyna
ACU
Treonina
AAU
Asparagina
AGU
Seryna
C
AUC
Izoleucyna
ACC
Treonina
AAC
Asparagina
AGC
Seryna
A
AUA
Izoleucyna
ACA
Treonina
AAA
Lizyna
AGA
Arginina
G
AUG Metionina
(Inicjaca translacji)
ACG
Treonina
AAG
Lizyna
AGG
Arginina
G
G
GUU
Walina
GCU
Alanina
GAU
Kw. asparaginowy
GGU
Glicyna
U
GUG
Walina
GCC
Alanina
GAC
Kw. asparaginowy
GGC
Glicyna
C
GUA
Walina
GCA
Alanina
GAA
Kw. glutaminowy
GGA
Glutamina
A

GUG
Walina
GCG
Alanina
GAG
Kw. glutaminowy
GGG
Glicyna
G
Jak widac w tabeli kod trojkowy zapewnia 64 kombinacje. Jednak nie wszystkie z tych trojek
koduja aminokwasy, istnieja bowiem 3 kodony, stanowiace sygnal zakonczenia translacji,
ktorym nie odpowiada zaden z nich. Pozostale 61 kodonow ma swoj odpowiednik
aminokwasowy. Latwo wiec zauwazyc, ze na jednen aminokwas przypadaja srednio trzy
trojki kodujace. Przez te wlasciwosc kod genetyczny nie jest wzajemnie jednoznaczny,
inaczej mowiac jest zdegenerowany.
Mowiac o niejednoznacznosci nalezy zwrocic uwage na pewne zagadnienie.
Przeksztalcajac informacje zawarta w lancuch polipeptydowym do postaci lancucha
nukleotydowego - uzyskuje sie kilka rozwiazan. W te strone kod jest
niejednoznaczny.
Przy odtwarzaniu ukladu aminokwasow w polipeptydzie z kolejno ulozonych
nukleotydow otrzyma sie tylko jedno rozwiazanie. W te strone kod jest jednoznaczny.
Dodatkowa wlasnoscia kodu genetycznego jest jego uniwersalnosc. Caly swiat ozywiony
uzywa tego samego sposobu zapisu informacji o bialkach. Wszystkie aminokwasy obecne w
bialkach roznych organizmow sa zakodowane przy uzyciu tych samych tripletow (wykryto
tylko nieliczne wyjatki m.in. w mitochondrialnym kodzie genetycznym).
Zatem kod genetyczny jest :
trojkowy,
zdegenerowany,
uniwersalny (w takej samej formie u prawie wszystkich organizmow),
bezprzecinkowy,
Translacja
Translacja, czyli biosynteza bialek jest ostatnim etapem ekspresji informacji genetycznej. Po
przepisaniu informacji z nukleotydowej sekwencji DNA na RNA (mRNA), translacja
pozwala na przetlumaczenie szyfru nukleotydowego na jezyk aminokwasow w bialku.
System translacji dziala podobnie zarowno u organizmow prokariotycznych, jak i u
eukariotycznych, z ta roznica, ze u Eucariota nastepuje przestrzenne oddzielenie transkrypcji
od translacji (translacja odbywa sie na terenie cytoplazmy co wiaze sie z koniecznosca
transportu mRNA z przedzialu jadrowego).
Przed omowieniem samego procesu translacji, nalezy wspomniec o strukturach niezbednych
do jego przeprowadzenia, sa nimi:
Obok tych struktur, w procesie biosyntezy bialka biora udzial rowniez: matrycowe RNA
(mRNA) oraz wysokoenergetyczne zwiazki, ktorych zadaniem jest dostarczenie energii
(najprawdopodobniej sa nimi czasteczki GTP).
W procesie translacji poszczegolne skladniki musza znalesc sie w scisle okreslonym
polozeniu w stosunku do siebie, zatem czasteczki bioroce w nim udzial musza zostac
uporzadkowane przestrzennie. Taka porzadkujaca funkcje pelnia rybosomy. Latwosc
dysocjacji i asocjacji obu podjednostek rybosomalnych umozliwia skutecznie wiazanie sie
tych organelli z mRNA oraz aminoactlo-tRNA.

Rozpoznanie odpowiedniego tripletu nukleotydowego na mRNA odbywa sie dzieki dzieki
uniwersalnej dla oddzialywan miedzy kwasami nukleinowymi regule komplementarnosci
(antykodon z tRNA musi sie laczyc z odpowiednim kodonem polozonym na mRNA).
Po zwiazaniu sie mRNA pomiedzy dwie podjednostki, w miejsca A i P rybosomu dolaczane
sa odpowiednie aminoacylo-tRNA. Pierwszym, dolaczanym do kodonu inicjatorowego
(AUG), jest zwykle aminoacylo-tRNA, zwiazane z N-formylomietionina. (tzw. f-Met-tRNA).
Dalej proces translacji zachodzi w nastepujacych po sobie cyklach. Poszczegolne fazy
jednego z takich cykli przedstawiaja sie nastepujaco:
W miejscu P rybosomu znajduje sie tRNA z dolaczonym f-Met-tRNA lub tRNA z
fragmentem lancucha polipeptydowego, jesli jest to jeden z dalszych cykli.
Oczywiscie wymienione poprzednio tRNA musza miec antykodony komplementarne
do kodonow na mRNA.
Do wolnego miejsca A dolacza sie nowy aminoacylo-tRNA z antykodonem
odpowiadajacym trojce, znajdujacej sie wlasnie w tym miejscu.
Nastepnym etapem jest wytworzenie wiazania peptydowego pomiedzy aminokwasem
znajdujacym w miejscu A a fragmentem lancucha polipeptydowego. Dzieki temu
lancuch wydluza sie o jeden aminokwas i przenoszony jest w miejsce A rybosomu.
Tworzenie lancucha polipeptydowego ma charakter enzymatyczny, katalizatorem jest
w niej fragment RNA z duzej podjednostki.
W kolejnej fazie zdeacylowany tRNA z jest odrzucany z miejsca P, a na jego miejsce
przesuwa sie tRNA z dolaczonym fragmentem polipeptydu. Towarzyszy temu
przemieszczenie sie mRNA (translokacja), przy uzyciu energii z
wysokoenergetycznych wiazan GTP. W ten sposob, ze naprzeciw wolnego juz miejsca
A pojawia sie kolejny kodon, odpowiadajacy nowemu aminokwasowi.
Proces translacji konczy sie, gdy w miejscu A pojawi sie jeden z kodonow terminacyjnych
(UGA - opal, UAA - ochre lub UAG - amber). Zadnemu z trzech kodonow stop nie
odpowiada aminokwas. W rezultacie dochodzi do dolaczenia czynnikow terminacyjnych
(RF), ktore powoduja uwalnianie polipeptydowego produktu oraz dysocjacje calego
kompleksu rybosomowego.
Powstaly polipeptyd musi ulec pewnym modyfikacjom chemicznym m.in. proteolitycznemu
obcieciu N-formylometioniny, a nastepnie przyjac odpowiednia konformacje, ktora pozwoli
na pelnienie odpowiedniej funkcji.
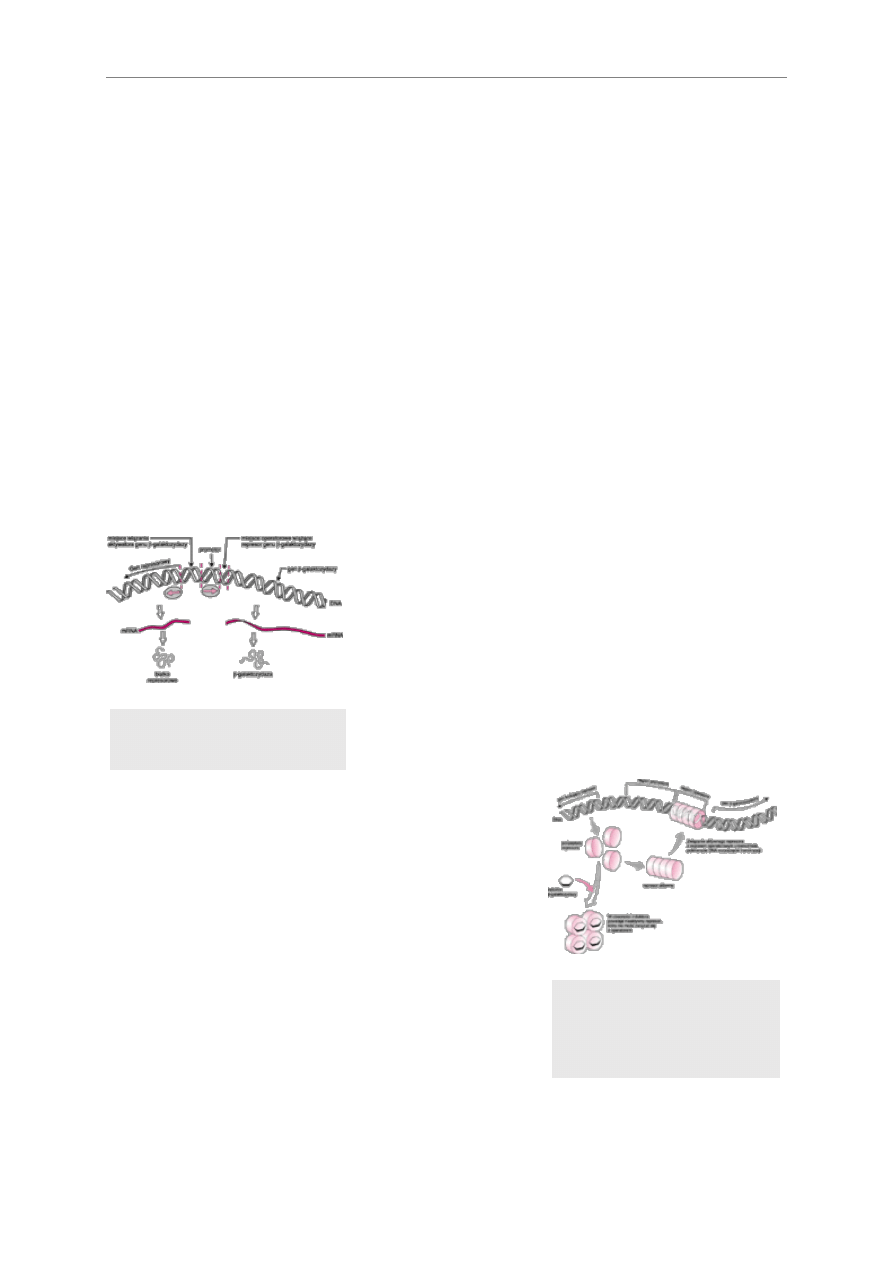
Włączanie i wyłączanie genów
oznawanie tr
anskrypcji i translacji genów to zaledwie część badań nad ich ekspresją. Innym
problemem jest sposób, w jaki ekspresja ta jest regulowana, to znaczy jak na tempo i siłę ekspresji
genu wpływają warunki zewnętrzne. Postęp w rozumieniu mechanizmów transkrypcji i translacji rzucił
światło na regulację genów. Badania genetyczne bakterii dostarczyły narzędzi i koncepcji potrzebnych
do wyjaśnienia, jak regulowana jest ekspresja genów. W pewnych warunkach wiele genów
bakteryjnych w ogóle nie ulega ekspresji, podczas gdy poziom ekspresji innych różni się nawet
kilkaset razy. Zmiana warunków może spowodować aktywację genów uprzednio milczących oraz
wyciszać geny aktywne. Ta cecha pozwala komórkom bakteryjnym na dużą elastyczność w
dostosowaniu się do zmian środowiska zewnętrznego – na przykład do zmiennego zaopatrzenia w
składniki odżywcze lub tlen.
P
Ekspresja genu zazwyczaj regulowana jest na poziomie transkrypcji, czyli syntezy mRNA.
Najczęściej regulowanym etapem jest inicjacja transkrypcji. O tym, czy konkretny gen będzie
przepisywany na mRNA, czy też nie, decydują białka wiążące się przejściowo ze specjalnymi
sekwencjami DNA bezpośrednio poprzedzającymi gen. Oddziaływanie białek regulatorowych z DNA
może zarówno blokować transkrypcję, jak i ją umożliwiać. Odpowiednio do funkcji, białka te nazywane
są więc represorami albo aktywatorami. Aby docenić precyzję tych mechanizmów regulacji i sposób, w
jaki pozwalają one komórce bakterii E. coli reagować na bodźce środowiska, najlepiej przyjrzeć się
konkretnemu przy
kładowi.
Enzym E. coli
β-galaktozydaza rozkłada laktozę (cukier
występujący w mleku) na dwa prostsze cukry: glukozę i
galaktozę. Jeśli E. coli rośnie w obecności glukozy,
preferowanego przez nią pokarmu, nie syntetyzuje β-
galaktozydazy; jeśli jednak jedynym dostępnym cukrem jest
laktoza
– wytwarza ten enzym. Do regulacji transkrypcji służy w
genomie E. coli
kilka sekwencji DNA poprzedzających 5' koniec
odcinka kodującego β-galaktozydazę. Polimeraza RNA, która
prowadzi transkrypcję, wiąże się z jedną z nich – promotorem.
Druga sekwencja
– operator – znajduje się pomiędzy
promotorem a
sekwencją kodującą ten enzym. Operator
oddziałuje z innym białkiem – represorem. Związanie się białka
represorowego z operatorem uniemożliwia polimerazie RNA
inicjację transkrypcji.
Odcinki DNA regulujące
transkrypcję genu β-
galaktozydazy E. coli
Gdy bakterii dostarczy się laktozę, cukier ten wiąże się z
białkiem represorowym, co powoduje zmianę jego kształtu i
uniemożliwia mu wiązanie się z DNA, co z kolei pozwala
polimerazie RNA na transkrypcję, powstaje mRNA, a następnie β-
galaktozydaza i laktoza może być wykorzystana jako źródło
energii do wzrostu. Warto zwrócić uwagę, że tak jak w przypadku
anemii sierpowatej, o aktywności biologicznej białka decyduje
jego specyficzna struktura.
Różne formy represora,
obecność lub brak induktora,
odpowiednio hamuje lub
indukuje transkrypcję genu
β-galaktozydazy
Trzeba uświadomić sobie, że represor genu β-galaktozydazy rozpoznaje sekwencję operatora i
łączy się z nią. Z powodu jej długości jest bardzo mało prawdopodobne, by taka sama sekwencja
wystąpiła jeszcze w innym miejscu genomu. Dzięki temu represor nie wiąże się z innymi sekwencjami
DNA i może regulować tylko ekspresja genu β-galaktozydazy.
Oprócz negatywnej kontroli wynikającej z oddziaływania białka represorowego z operatorem, gen
ten podlega również kontroli pozytywnej.
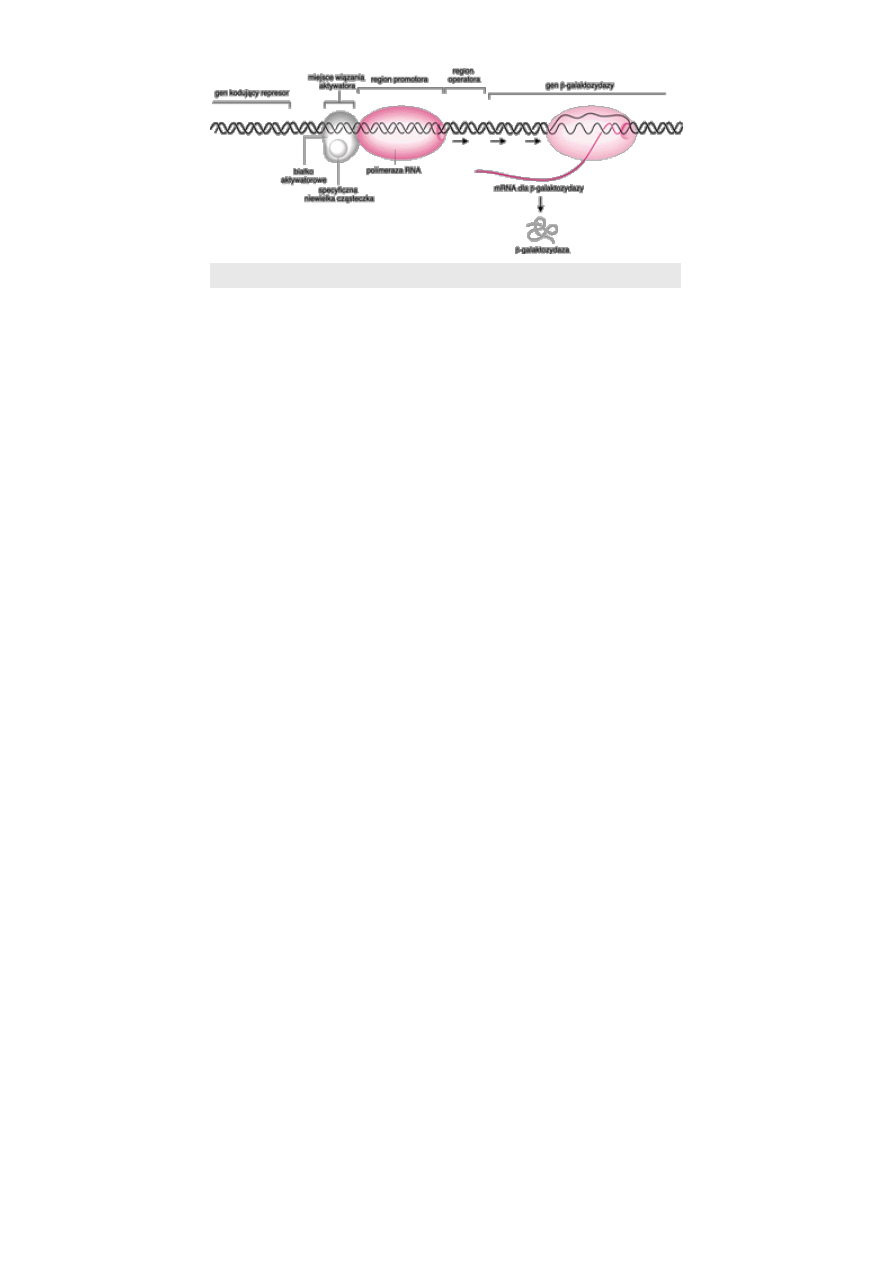
Transkrypcja genu β-galaktozydazy wymaga aktywatora
Transkrypcja genu β-galaktozydazy może rozpocząć się tylko w obecności specyficznego aktywatora.
Jest nim białko aktywne jedynie po połączeniu ze specyficzną małą cząsteczką. Pojawia się ona w
komórce, kiedy w pożywce nie ma glukozy. W tym sensie cząsteczka ta jest jakby sygnałem głodu. W
warunkach „głodu glukozowego” cząsteczka ta tworzy kompleks z białkiem aktywatorowym, które
łączy się z krótkim odcinkiem DNA niedaleko promotora i operatora. Przyłączenie kompleksu
wzmacnia zdolność polimerazy RNA do transkrypcji genu β-galaktozydazy. Oznacza to, że jego
ekspresja zależy od dwóch czynników środowiskowych: braku glukozy oraz obecności laktozy.
Takiej złożonej regulacji podlega transkrypcja większości genów bakteryjnych, ale białka represorowe i aktywatorowe nie są
jedynymi elementami układu regulującego. Czasami sam białkowy produkt genu jest regulatorem własnej transkrypcji, co tworzy
układ sprzężenia zwrotnego. Syntezę mRNA może więc regulować tempo inicjacji, a także kontrola szybkości wydłużania jego
łańcucha oraz zmiana prawdopodobieństwa, że będzie kontynuowana aż do końca genu, lub też że skończy się w jakimś
kontrolnym punkcie w jego obrębie.
Regulacja ekspr
esji genu zachodzi również podczas translacji mRNA na polipeptyd. Także i tu najczęściej kontroli podlega
inicjacja, czytanie pierwszego kodonu, choć również następne kroki syntezy łańcucha polipeptydowego mogą jej podlegać.
Dodatkowe zjawiska regulujące mogą też zachodzić podczas przekształcania się ukończonego polipeptydu w funkcjonalne
białko, ponieważ wiele peptydów, aby pełnić swoją funkcję, wymaga modyfikacji, czyli przyłączenia określonych grup
chemicznych. Na przykład w hemoglobinie zawierająca żelazo cząsteczka odpowiedzialna za transport tlenu dodawana jest do
białka już po syntezie i złożeniu obu różnych łańcuchów peptydowych. Każdą taką modyfikację przeprowadza jeden bądź wiele
enzymów, których obfitość lub poziom aktywności także mogą podlegać regulacji. Co więcej, wiele białek, żeby właściwie
funkcjonować, musi znaleźć się w odpowiednim miejscu w komórce. Na przykład w komórkach eukariotycznych białka
niezbędne do tworzenia rybosomów muszą być przeniesione z cytoplazmy, gdzie powstają, do jąderka, gdzie składane są
rybosomy. Inne białka muszą być dostarczone do błony komórkowej lub, jak hormony, uwolnione na zewnątrz komórki. W
konsekwencji kontrolowanie transportu białek do właściwego miejsca również wpływa na poziom ich aktywności. Liczne
różnorodne i złożone mechanizmy biorą udział w regulacji ilości funkcjonalnego produktu genu. Często proces ten jest swoisty
dla konkretnego genu, stanu fizjologicznego komórki lub warunków środowiska zewnętrznego.
Wyszukiwarka
Podobne podstrony:
biologia nmr id 87949 Nieznany
biologia pp o id 88225 Nieznany (2)
czynniki biologiczne 10 id 6672 Nieznany
biologia wyklady id 88204 Nieznany (2)
BIOLOGIA KOMORKI 2 id 87836 Nieznany
biologia ABCDE id 87704 Nieznany
Biologia Wyklad 9 id 88128 Nieznany (2)
Biologia nr 3(1) id 87951 Nieznany
Biologia Cwiczenia 4 id 87712 Nieznany (2)
Biologia Cwiczenia 8 id 87713 Nieznany (2)
biologia 2008 id 87683 Nieznany (2)
Biologia molekularna Wyklady UM Nieznany
Biologia Cwiczenia 9 id 87714 Nieznany (2)
biologia spektroskopia2 id 8805 Nieznany
biologia mol id 87896 Nieznany
Biologia Wyklad 6 id 88126 Nieznany (2)
5 UMYSL I MOLEKULY id 39823 Nieznany (2)
biologia synteza id 88080 Nieznany
więcej podobnych podstron