
2009-01-08
1
Bioinformatyka
– wykład 10, 16.XII.2008
Białkowa
bioinformatyka strukturalna
–
c.d.
krzysztof_pawlowski@sggw.pl

2009-01-08
2
Popularne systemy klasyfikacji
struktur białkowych
•
SCOP -
visual
inspection
& automated
methods, A. Murzin
http://scop.mrc-lmb.cam.ac.uk/scop/
•
CATH -
semi-automatic,
http://www.cathdb.info
•
FSSP –
automated
(DALI)
http://ekhidna.biocenter.helsinki.fi/dali
2009-01-08
3
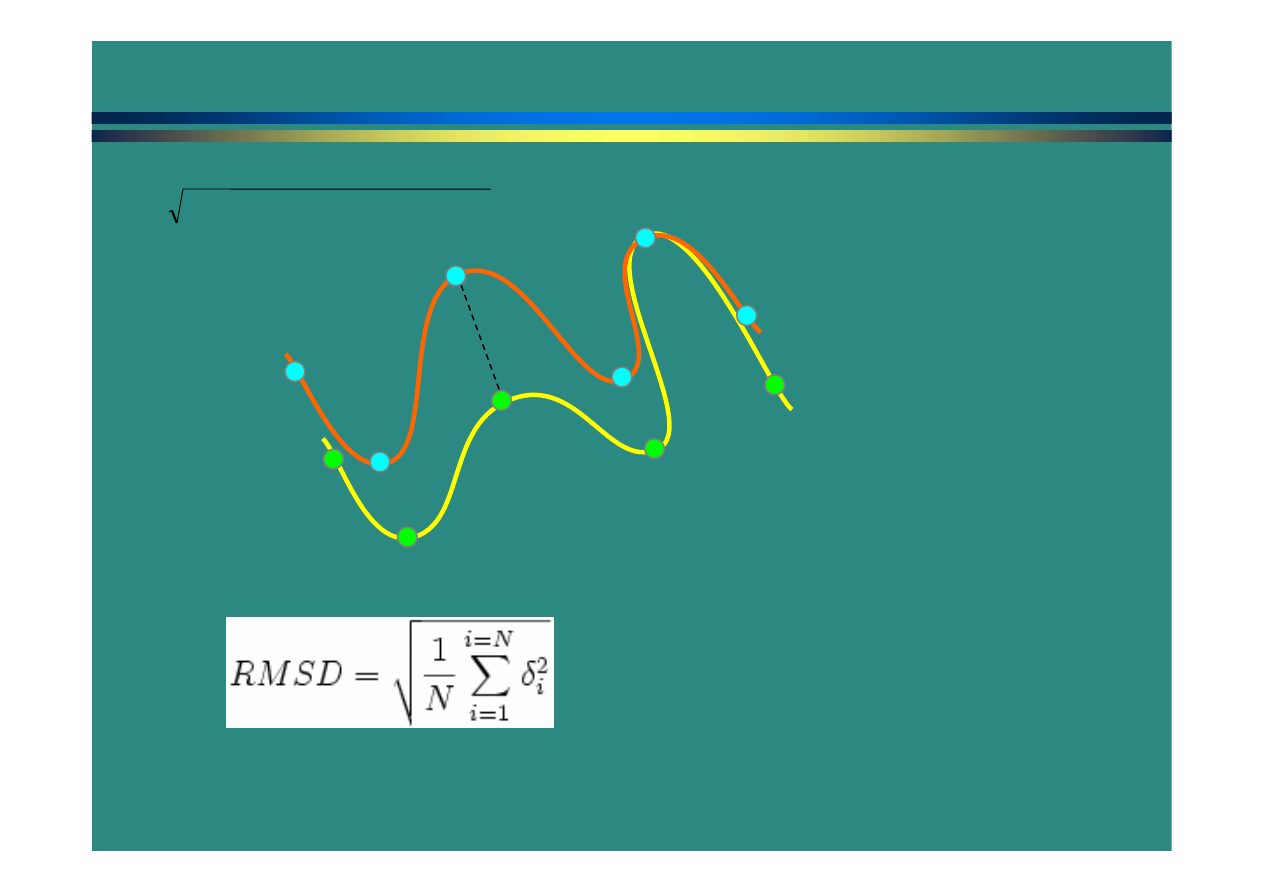
Dopasowanie strukturalne (alignment)
d
iJ
=
odległość:
(x
i
- x
J
)
2
+ (y
i
- y
J
)
2
+ (z
i
- z
J
)
2
b
c
d
e
f
α
β
γ
δ
ε
ζ
Root mean square deviation
-
zwykle C
α
2009-01-08
4
podobnie jak identyczność
sekwencji,
RMSD ma sens tylko dla określonego
dopasowania i regionu sekwencji

2009-01-08
5
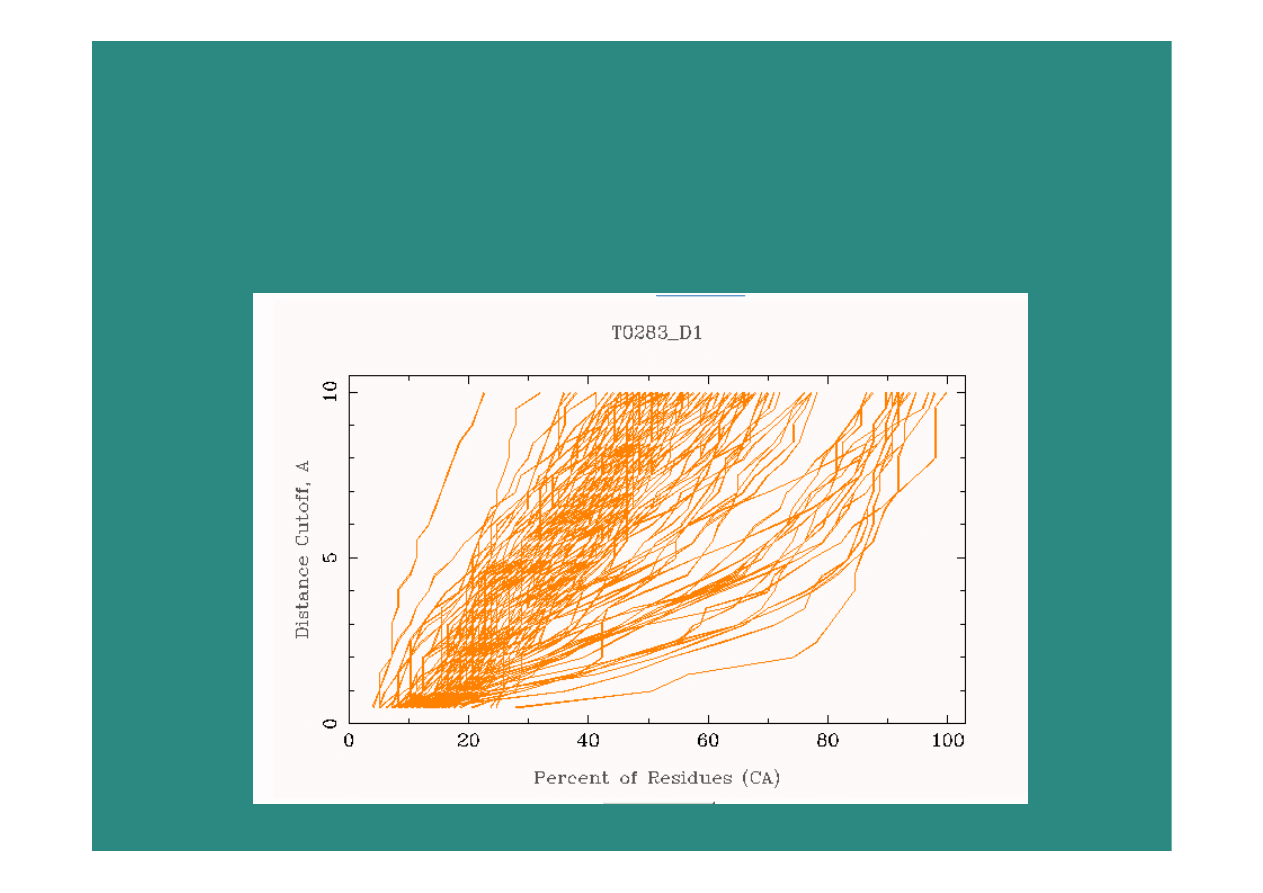
porównanie struktur -
programy
DALI
-
Distance Alignment Tool; dopasowanie macierzy
odległości
VAST
-
Vector alignment Search Tool; dopasowanie
wektorowe; wektory
opisujące
struktury
drugorzędowe
FATCAT
-
Flexible Alignment allowing Twists
LGA
–
lokalna i globalna optymalizacja RMSD
(longest
continuous
segments
& global
distance
test)
Wybór metody porównania struktur zależy od problemu

2009-01-08
6
Plan wykładu
•
doświadczalne metody badania struktur
białkowych: krystalografia, NMR, inne
•
oddziaływania decydujące o strukturze
białka
•
”pole siłowe”
-
opis energetyczny struktur
białka

2009-01-08
7
Plan wykładu
•
doświadczalne metody badania struktur
białkowych: krystalografia, NMR, inne
•
oddziaływania decydujące o strukturze
białka
•
”pole siłowe”
-
opis energetyczny struktur
białka
2009-01-08
8
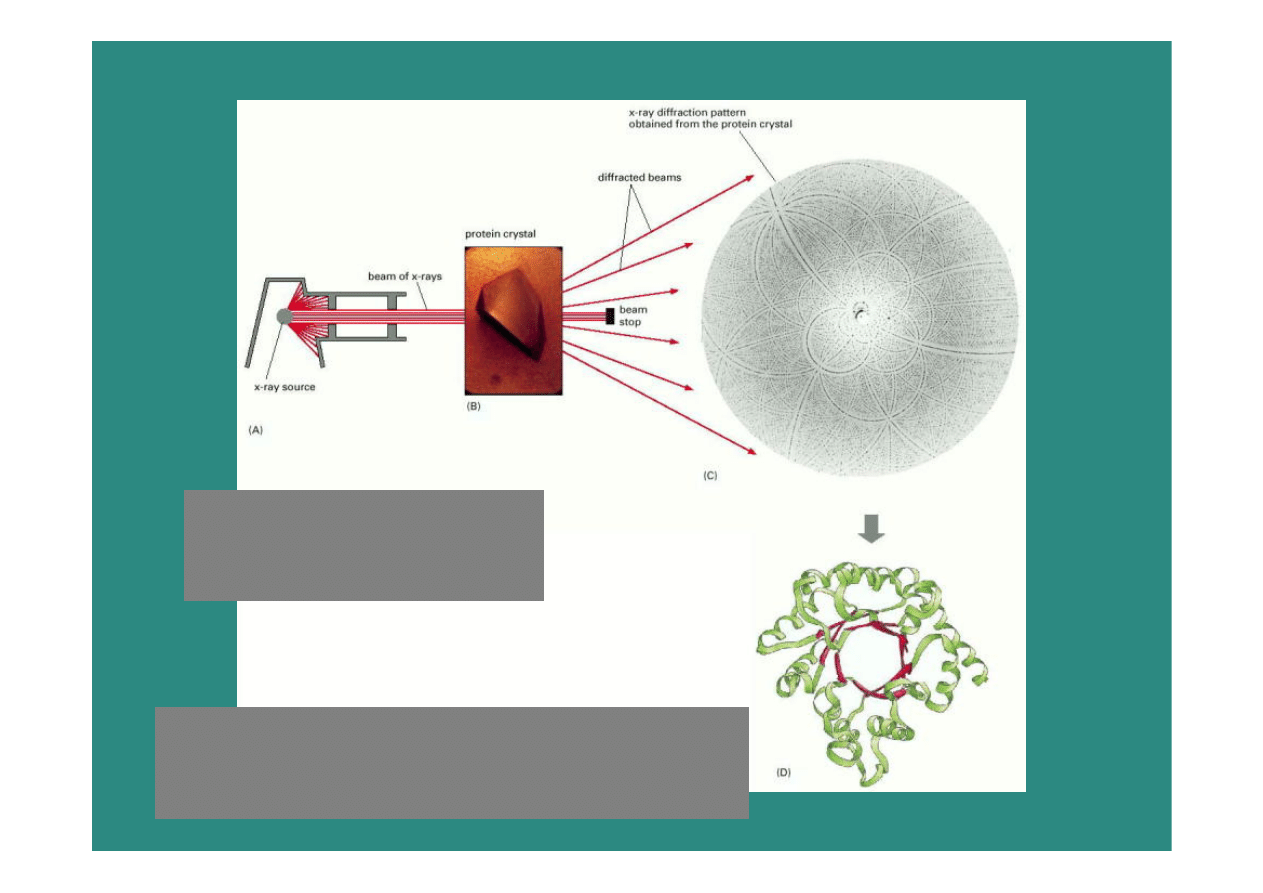
Krystalografia
promieni X
oddziaływania
promieni X z elektronami
2009-01-08
9
krystalografia
Informacje –
gęstość
elektronowa
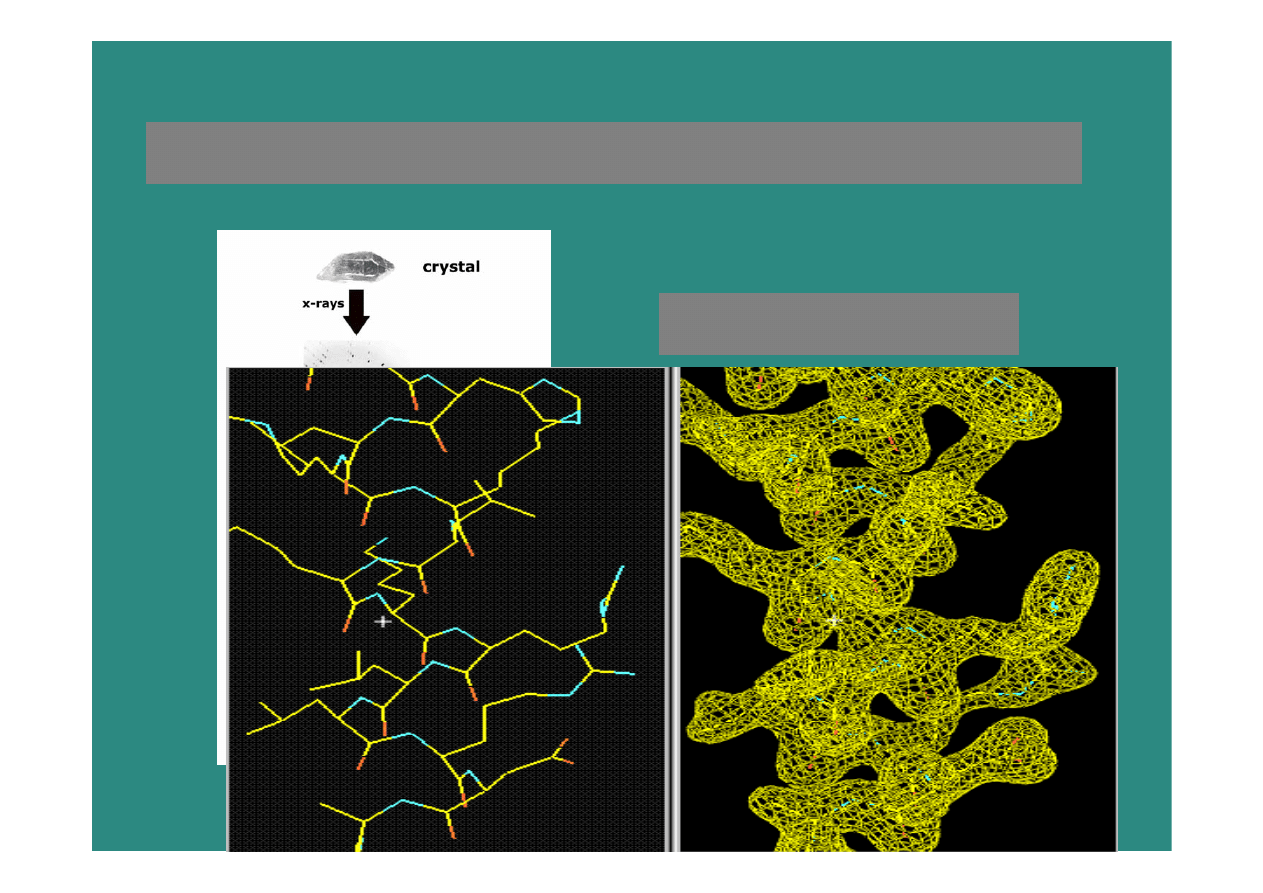
2009-01-08
10
krystalografia
Informacje –
gęstość
elektronowa

2009-01-08
11
NMR –
magnetyczny
rezonans jądrowy
oddziaływania
momentów magnetycznych jąder atomowych
z zewnętrznym polem magnetycznym
wykorzystuje się
protony, C13, N15

2009-01-08
12
Ze sprzężeń
spin-spin ->
informacje: kąty torsyjne
i więzy na odległości
Otrzymuje się
ensembl
struktur spełniających więzy
NMR –
magnetyczny
rezonans jądrowy

2009-01-08
13
a
krystalografia promieni X
najwięcej rozwiązanych struktur
krystalizacja!
a
NMR
białka w roztworze, ale ograniczenia wielkości białek, kosztowne
znakowanie izotopowe
a
mikroskopia elektronowa
duże struktury, np. kompleksy, zwykle z użyciem struktur krystalogr.
elementów
a
krystalografia neutronowa
protony są widoczne
wszystkie doświadczalne metody wyznaczania struktur
obejmują
elementy modelowania struktur

2009-01-08
14
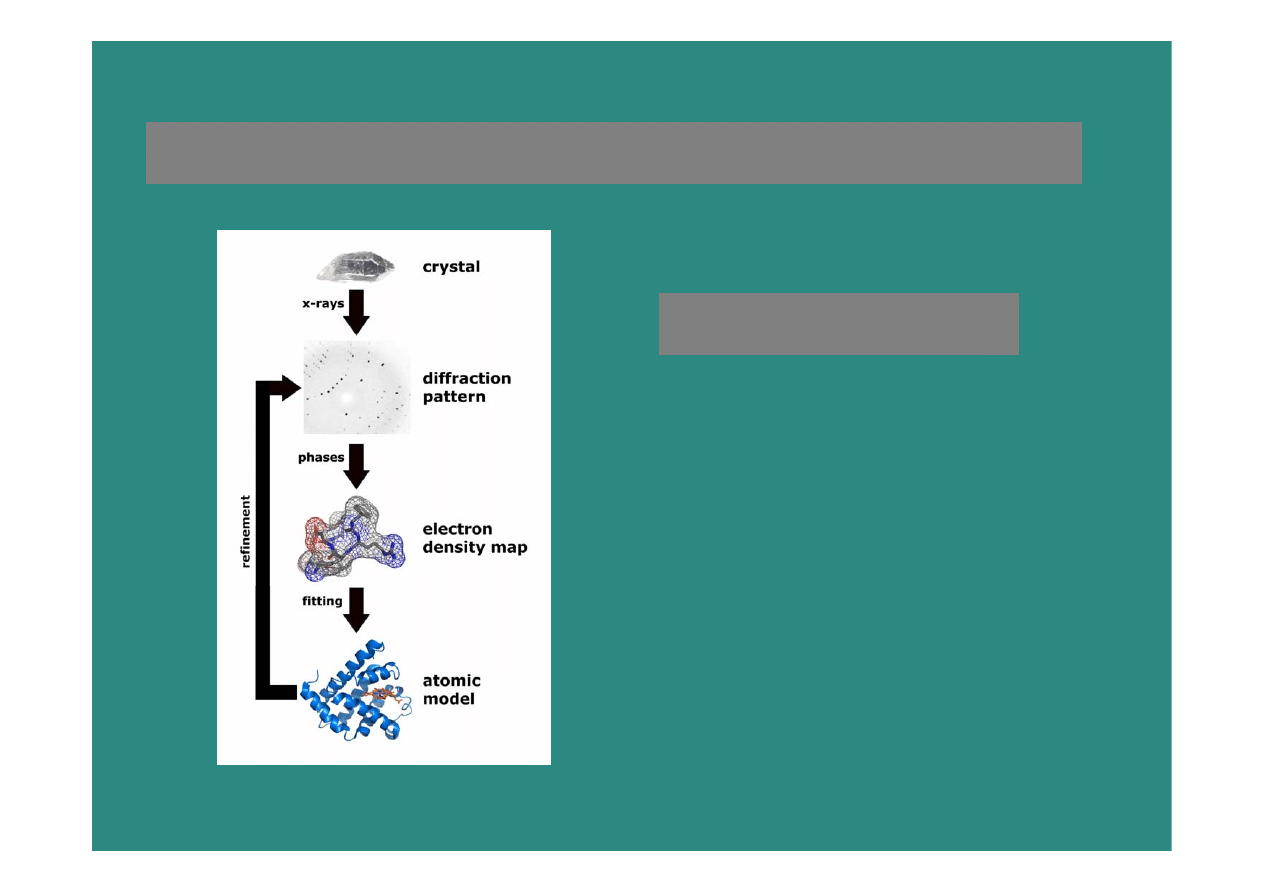
Typical steps in experimental
structure determination (X-ray)
•
Cloning and expression
•
Purification
•
Crystallization
•
X-ray data collection
•
X-ray data processing, model building and refinement
We get “the real thing”, however:
In the past the whole process could take a few years
Problems in any step can halt the structure solving process
Recently: substantial progress in speeding up and automation of structure
solving
The structure -
sequence gap: millions
of
sequences in NCBI,
54000+ protein structures in the PDB
(as of
mid
Dec 2008)

2009-01-08
15
Plan wykładu
•
doświadczalne metody badania struktur
białkowych: krystalografia, NMR, inne
•
oddziaływania decydujące o strukturze
białka
•
”pole siłowe”
-
opis energetyczny struktur
białka

2009-01-08
16
Oddziaływania decydujące
o strukturze białka
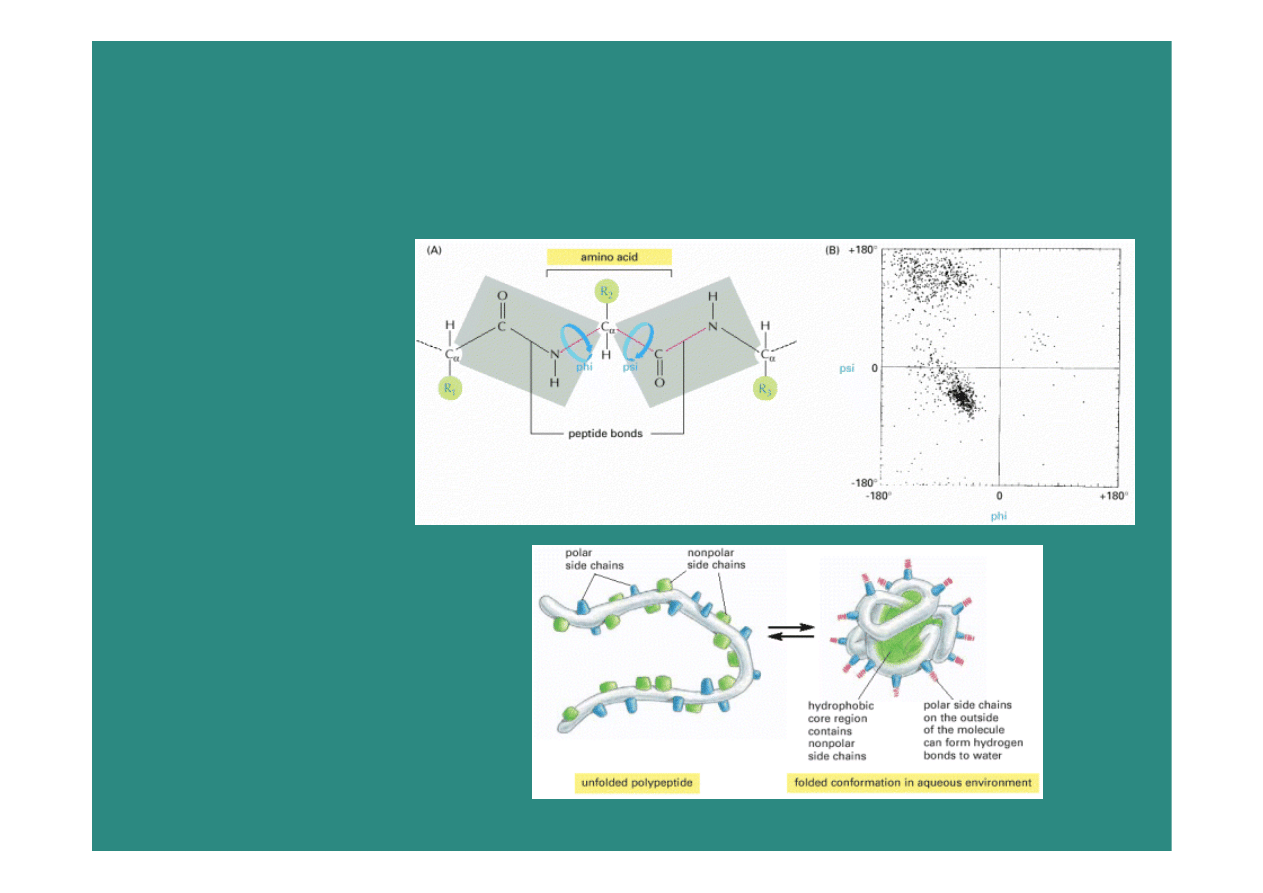
2009-01-08
17
Oddziaływania decydujące
o strukturze białka
lokalne
długozasięgowe

2009-01-08
18
F = U –
TS
Prawdopodobieństwo
stanu ~ exp(-F/k
B
T)
Struktura natywna
–
minimum energii swobodnej

2009-01-08
19
Wiązania kowalencyjne

2009-01-08
20
Electrostatics in uniform media:
potential
ϕ
1
= q
1
/
εr
Interaction of two charges
:
U =
ϕ
1
q
2
=
ϕ
2
q
1
= q
1
q
2
/
εr
ε
= 1 vacuum
ε ≈ 3 protein
ε ≈ 80 water
Protein/water interface
In non-uniform media:
ε
= ?
At atomic distances:
ε
= ?
Oddziaływania elektrostatyczme

2009-01-08
21
Oddziaływania van
der Waalsa
•
Oddziaływanie elektrostatyczne dipol-dipol
(uśrednione)
•
Oddziaływanie indukcyjne (dipol-dipol indukowany)
•
Oddziaływanie dyspersyjne (dipol indukowany -
dipol
indukowany)
~ 1/r
6

2009-01-08
22
Siły
odpychania
walencyjnego
•
zakaz Pauliego
(na jednym orbitalu
molekularnym
nie mogą
się
znajdować
więcej niż
dwa
elektrony).
•
E ~ exp(-r), często przybliżana E ~ 1/r
12
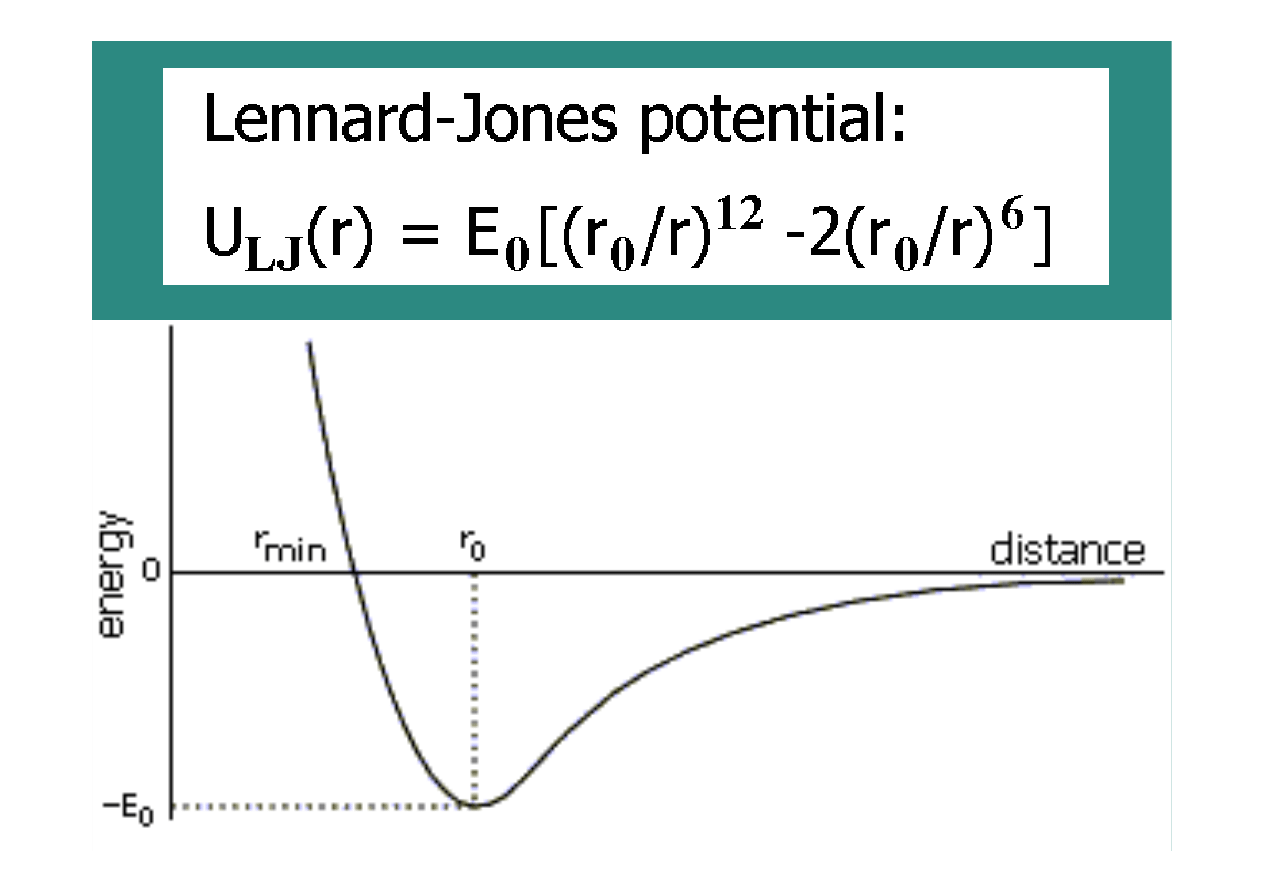
2009-01-08
23
oddziaływania van
der Waalsa
oraz siły
odpychania
walencyjnego
2009-01-08
24
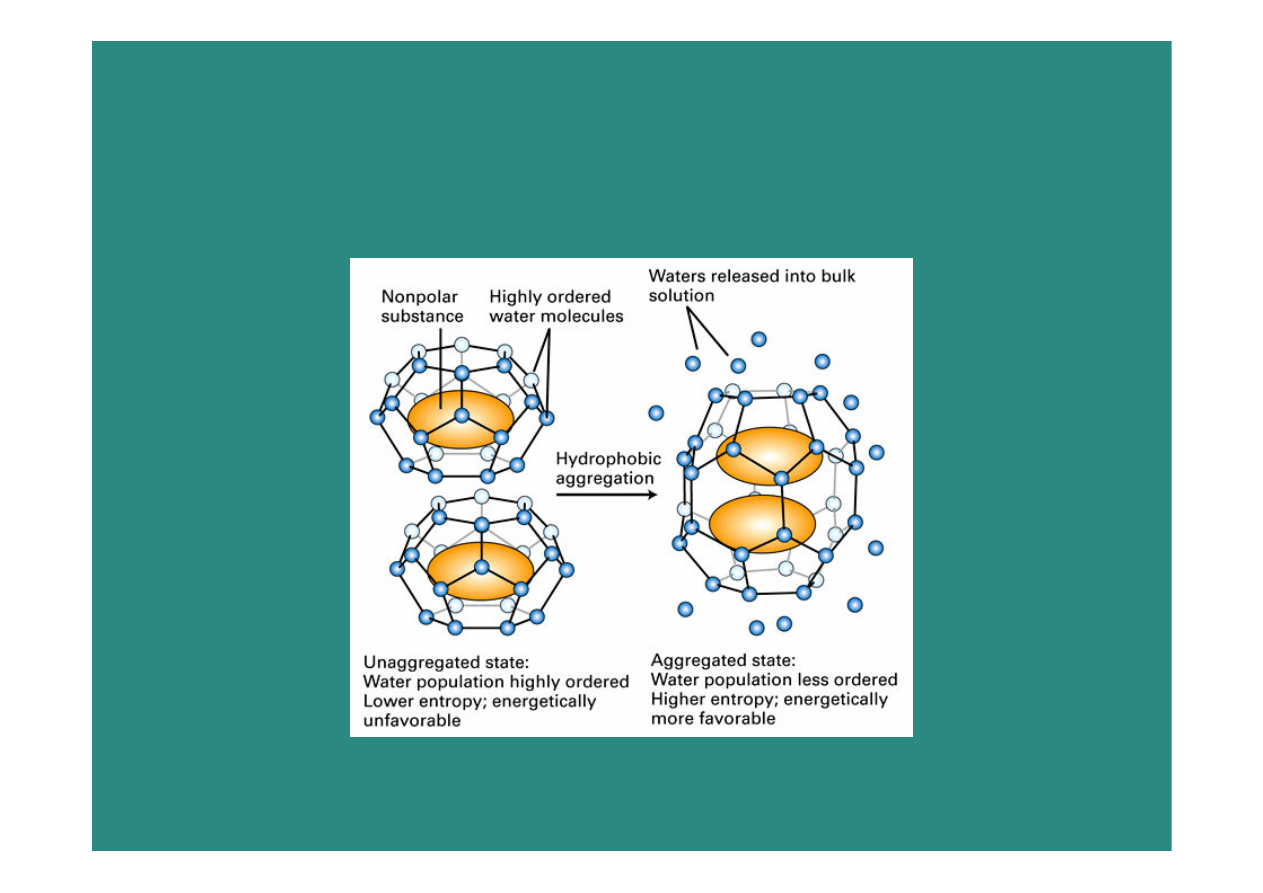
Efekt hydrofobowy

2009-01-08
25
Efekt hydrofobowy
Entropia!
węglowodór
fosforan

2009-01-08
26
Plan wykładu
•
doświadczalne metody badania struktur
białkowych: krystalografia, NMR, inne
•
oddziaływania decydujące o strukturze
białka
•
”pole siłowe”
-
opis energetyczny struktur
białka

2009-01-08
27
Opis energetyczny struktury białka
Przyzwoity opis
układu molekularnego –
równanie falowe Schrödingera
...ale dla większości zastosowań
wystarczy
klasyczny model białka

2009-01-08
28

2009-01-08
29
Wesołych Świąt!
...następny wykład 6.I.2009
Document Outline
- Bioinformatyka – wykład 10, 16.XII.2008 Białkowa bioinformatyka strukturalna – c.d.
- Popularne systemy klasyfikacji struktur białkowych
- Slide Number 3
- Slide Number 4
- Slide Number 5
- Plan wykładu
- Plan wykładu
- Slide Number 8
- Slide Number 9
- Slide Number 10
- Slide Number 11
- Slide Number 12
- Slide Number 13
- Typical steps in experimental structure determination (X-ray)
- Plan wykładu
- Slide Number 16
- Slide Number 17
- Slide Number 18
- Slide Number 19
- Slide Number 20
- Oddziaływania van der Waalsa
- Siły odpychania walencyjnego
- Slide Number 23
- Efekt hydrofobowy
- Efekt hydrofobowy
- Plan wykładu
- Slide Number 27
- Slide Number 28
- Wesołych Świąt!
Wyszukiwarka
Podobne podstrony:
bioinformatyka w13 2008 9 web
bioinformatyka w2 2008 web
bioinformatyka w9 2008 web
bioinformatyka w6 2008 web
bioinformatyka w11 2008 web
bioinformatyka w4 2008 web
bioinformatyka w12 2008 9 web
bioinformatyka w3 2008 web
bioinformatyka w7 2008 web
bioinformatyka w1 2008 web
bioinformatyka w8 2008 web
bioinformatyka w5 2008 web
bioinformatyka w13 2008 9 web
bioinformatyka w2 2008 web
bioinformatyka w9 2008 web
więcej podobnych podstron