
co najczęściej uszkadza DNA?
* endogenne mutageny:
• H
2
O – hydroliza → usuwanie zasad, deaminacja C do U
• O
2
– oksydacja → modyfikacje i rozpad zasad lub deoksyrybozy,
pękanie DNA (nick)
• czynniki alkilujące → modyfikacje zasad i szkieletu fosfodiestrowego
* światło słoneczne (UV)
* mutageny, karcinogeny np. BBQ, palenie i żucie tytoniu,
aflatoksyny tworzone przez pleśnie
Uwagi do tematu: mutacje/naprawa DNA
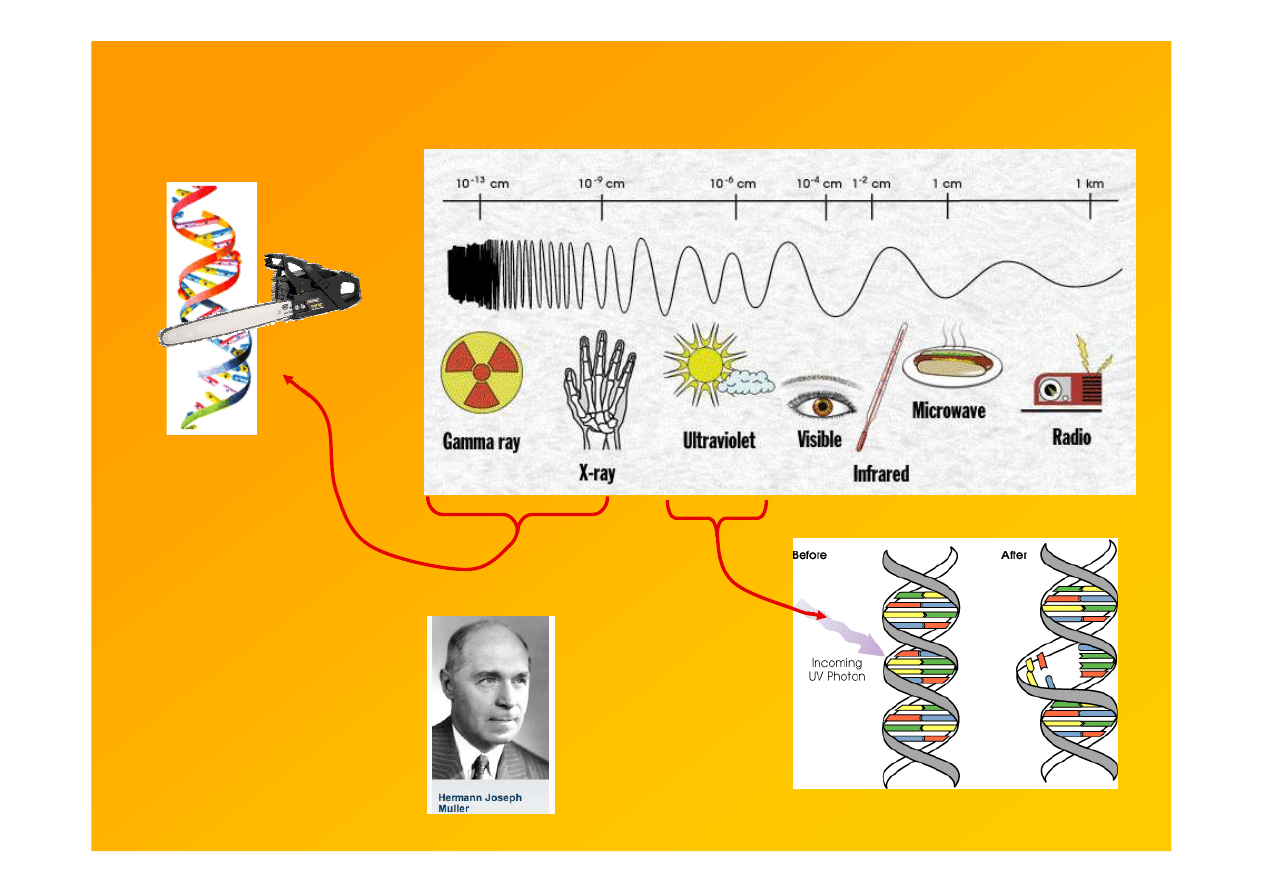
2
Mutagens
: electromagnetic radiation
UV light
- causes intrastrand
pyrimidine dimers
Gamma-rays, X-rays
- ionizing radiation
- break DNA strands
http://www.stanford.edu/class/bio203/lecture_files/stearns3.ppt#312,9,Slajd 9
1927 odkrył,
że pr.X jest
mutagenne,
Nobel 1946
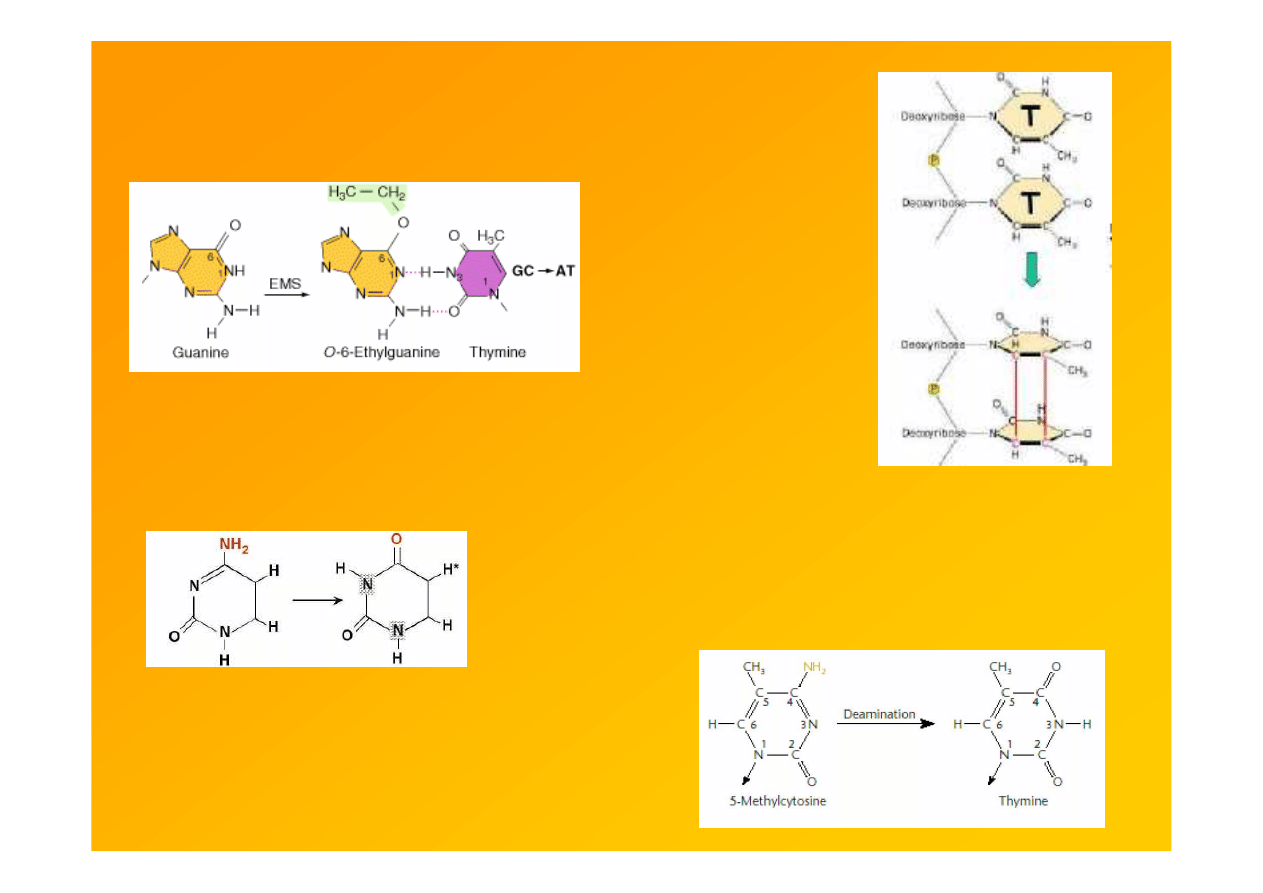
mutageny wykazują specyficzność
np. EMS: G-C
→
A-T
UV: dimery Py
cytozyna uracyl
deaminacja cytozyny
spontaniczna lub wynik działania kwasu
azotawego, hydroksylaminy, HSO
3
-
5mC jest gorącym m-scem deaminacji; C
→
T
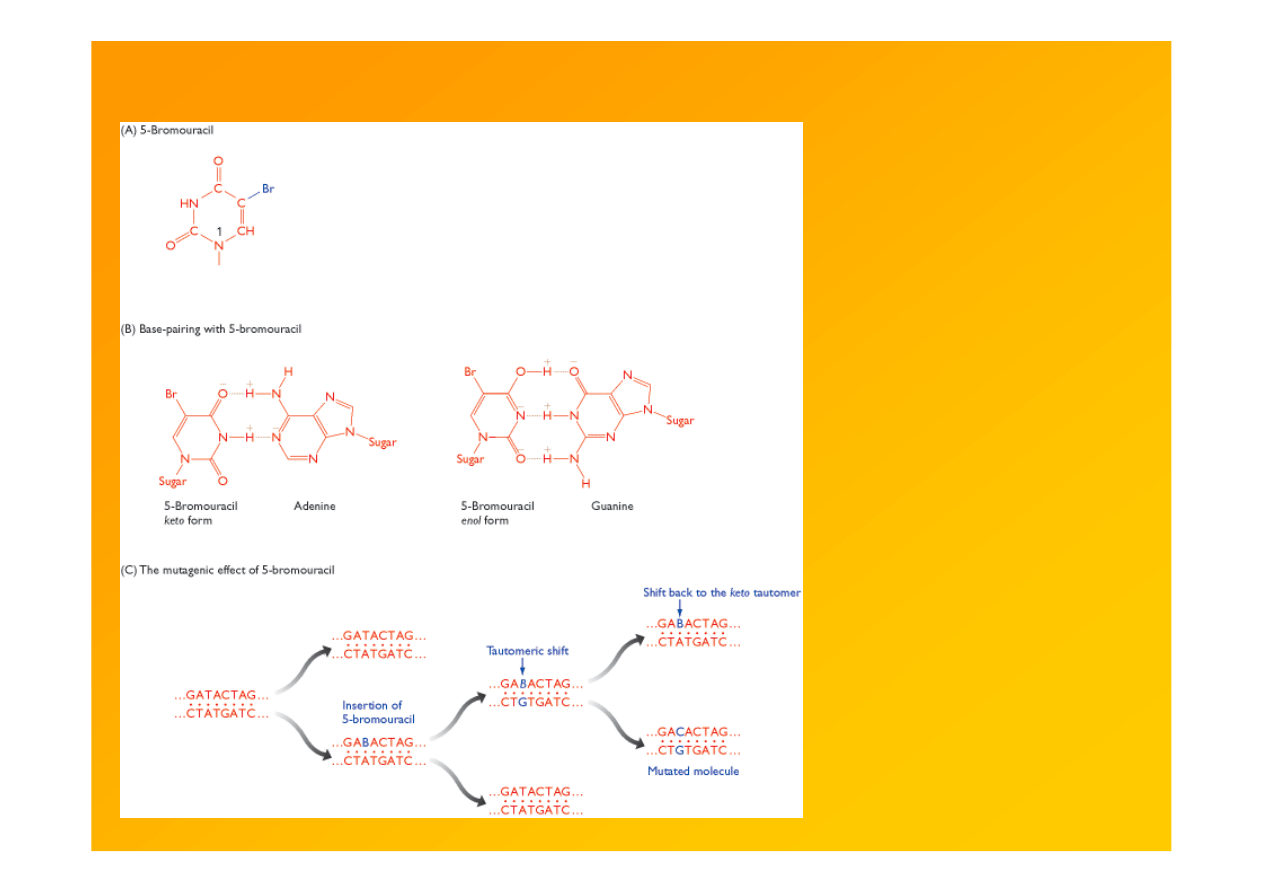
Brown TA 2002 Genomes 2nd ed. Garland Sci.
Ryc. 14.6. 5-Bromouracil and its mutagenic effect
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=genomes.figgrp.8387
bromouracyl: T-A→C-G

Patogenne mutacje zachodzą w obrębie 3 typów sekwencji DNA:
• kodujących
• wewnątrzgenowych niekodujących
• regulatorowych
mutacje nonsens – wynik podstawienia lub insercji lub delecji
skutki:
• niestabilny mRNA - najczęściej
• skrócony peptyd
• opuszczenie eksonu z mutacją nonsens - rzadko
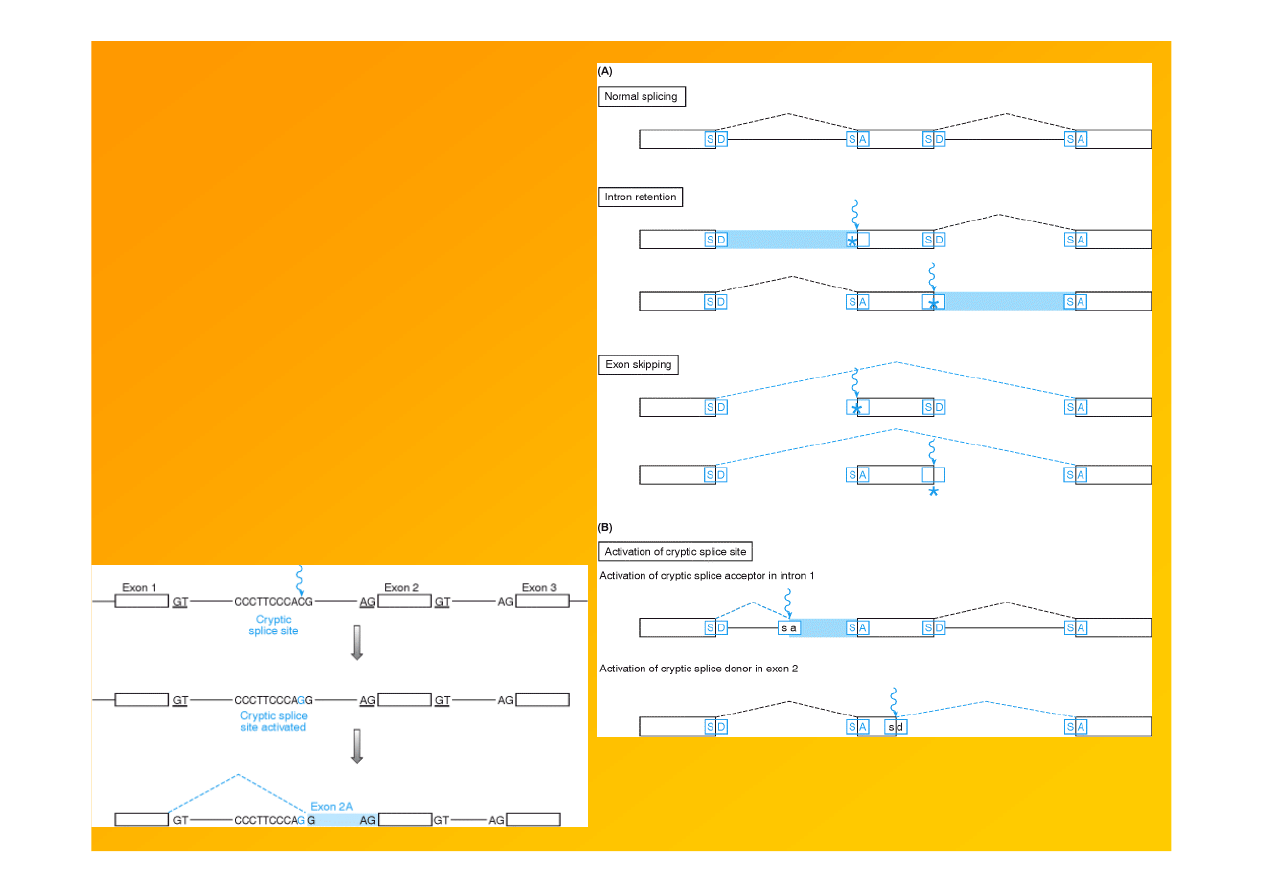
Fig.9.11. Zaburzenia splicingu -wynik
mutacji naturalnych sekwencji miejsc
donorowych lub akceptorowych
splicingowych lub aktywacji
kryptycznych m-sc splicingowych.
splice donor (SD)
splice acceptor (SA)
Strachan & Read 1999 Human Molecular Genetics 2,
Garland Sci.
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.1124
Fig. 9.13. Mutations can cause abnormal RNA
splicing by activation of cryptic splice sites.
Strachan & Read 1999 Human Molecular Genetics 2,
Garland Sci.
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.1125
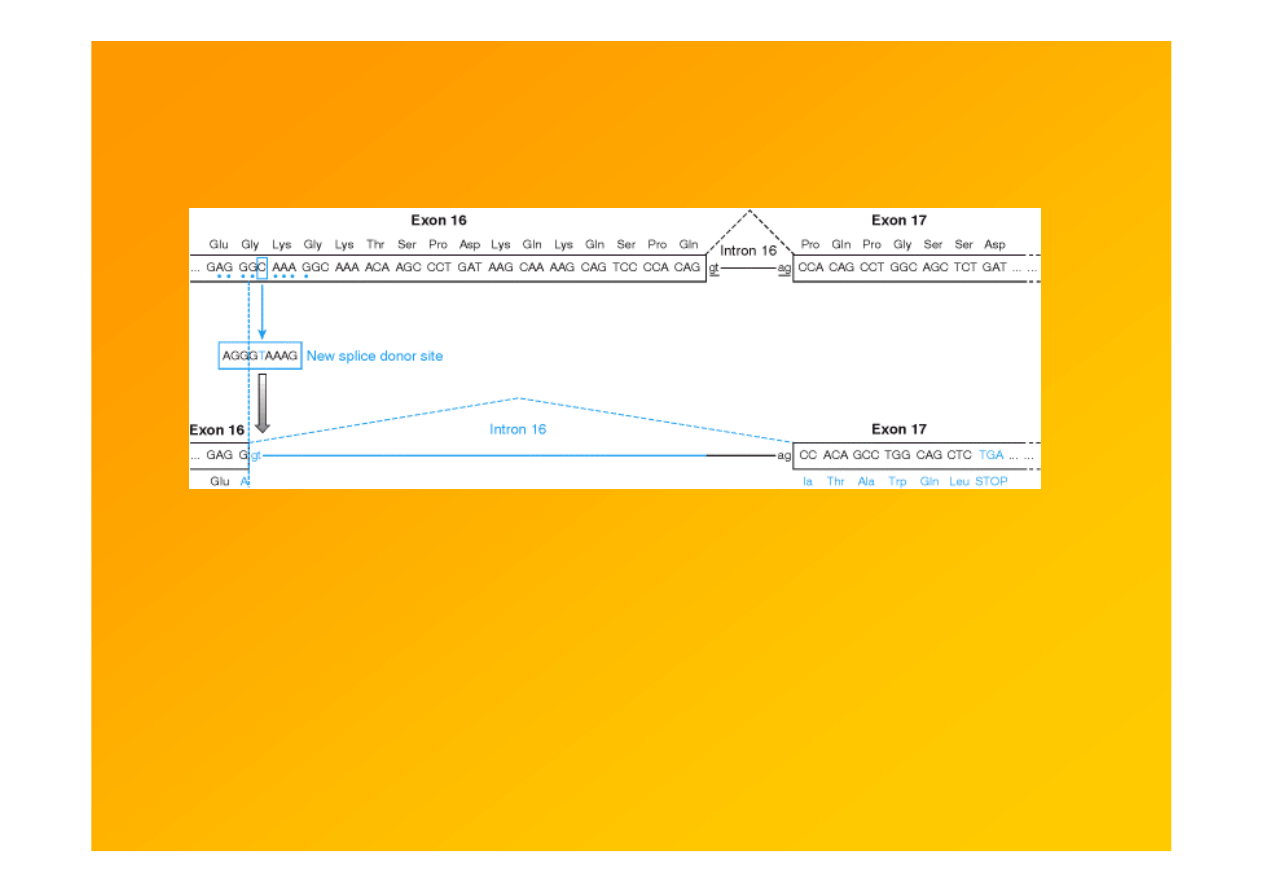
Fig. 9.12.
When a silent mutation is not silent.
Mutacja w genie kalpainy 3 u pacjenta z dystrofią
obręczowo-kończynową LGMD2A. Kodon glicynowy (GGC) zostaje zastąpiony innym glicynowym (GGT), co
powoduje powstanie kryptycznego donorowego m-sca splicingowego w eksonie 16 , a w konsekwencji
utratę części sekwencji eksonu 16 i zmianę ramki odczytu ( Richard i Beckmann, 1995).
Strachan & Read 1999 Human Molecular Genetics 2, Garland Sci.
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.1072

geny zawierające
niestabilne powtórzenia trójnukleotydowe
:
• z umiarkowaną ekspansją powtórzeń (CAG)
n
w sekwencji kodującej
typowo stabilny niepatologiczny allel ma n=10–30, a niestabilny
patologiczny 40–200;
transkrypcja i translacja genu nie zmienia się, ale produkt nabywa
funkcję: jego długi trakt poliglutaminowy powoduje agregację białka
w niektórych komórkach i ich śmierć
• z dużą ekspansją niekodującego powtórzenia
powtórzenia różnych trójek (np. CGG, CCG, CTG, GAA) w promotorze,
UTRach lub intronach
allel niepatologiczny ma n= 5-50, a niestabilny patologiczny setki-
tysiące
powoduje to zahamowanie ekspresji genu, utratę funkcji
przykłady chorób spowodowanych
transpozycją elem. ruchomych
:
• u 2 ze 140 niespokrewnionych pacjentów hemofilia A była spowodowana
insercją
de novo
elem. LINE-1 do eksonu w genie czynnika VIII
(Kazazian et al., 1988)
• w neurofibromatozie typu I znane są przypadki inaktywacji poprzez
insercję elem. Alu (Wallace et al., 1991)
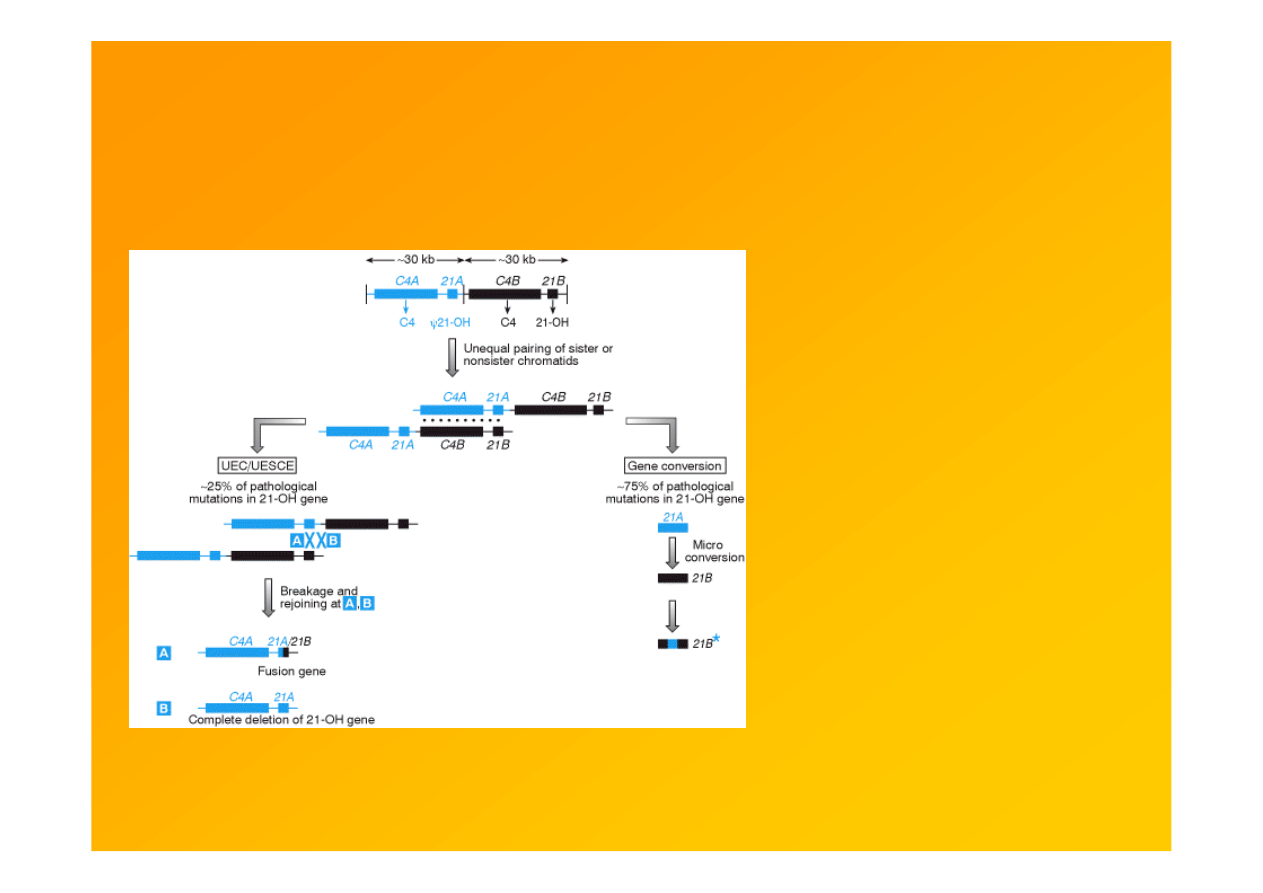
Tandemowe powtórzenia i rodziny zgrupowanych genów mają skłonność
do patogennych nierównych c-o oraz konwersji genów
Fig. 9.17. Niemal wszystkie
mutacje w genie 21-hydroksylazy
steroidowej CYP21 są wynikiem
wymiany sekwencji z blisko
spokrewnionym pseudogenem.
Zduplikowane geny dopełniacza C4 i 21-
hydroksylazy steroidowej tworzą 30-kb
tandemowe powtórzenia o 97%
identyczności sekwencji. Geny C4A, C4B
i CYP21B ulegają ekspresji, CYP21A
(21A) jest pseudogenem. Ok. 25%
patologicznych mutacji locus 21B
stanowią 30-kb delecje spowodowane
nierównym c-o (unequal crossover, UEC)
lub nierówną wymianą siostrzanych
chromatyd (unequal sister chromatid
exchange, UESCE). Pozostałe mutacje to
na mała skalę konwersje genowe CYP21B
– mały fragment CYP21A zawierający
szkodliwą mutację jest kopiowany i
zastępuje oryginalna sekwencję CYP21B
Strachan & Read 1999 Human Molecular
Genetics 2, Garland Sci.
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.1135
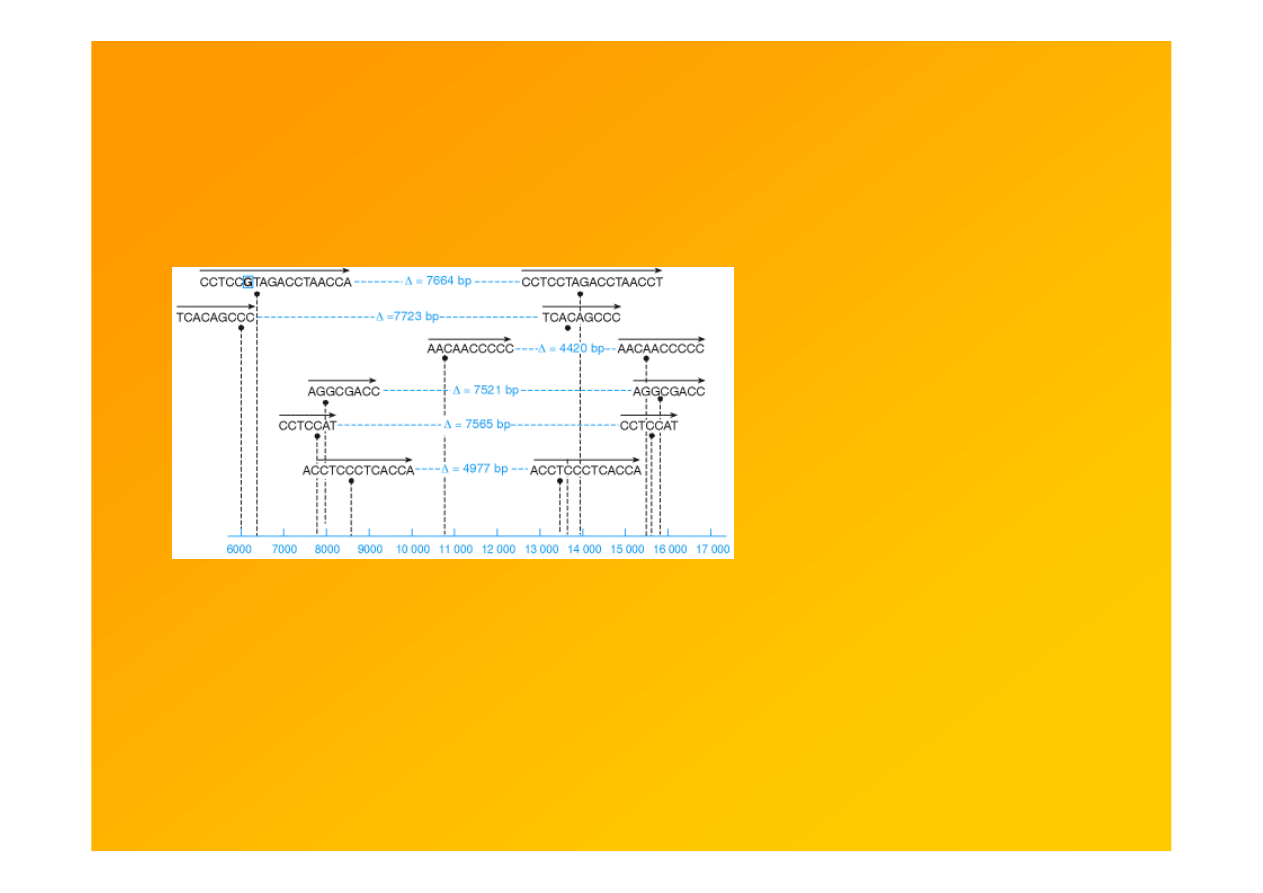
Fig. 9.19. Short direct repeats mark the endpoints of many pathogenic deletions
in the mitochondrial genome. Scale represents nucleotide position in the
mitochondrial genome. Position 1 occurs within the D loop region and numbering
increases in a clockwise direction for the illustration in Figure 7.2.
Note that as
recombination does not occur within the mitochondrial genome, one likely mechanism
to explain the deletions is slipped strand mispairing.
Strachan & Read 1999 Human Molecular Genetics 2, Garland Sci.
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.1147
powtórzenia rozproszone (krótkie proste oraz Alu)
predysponują do dużych delecji i duplikacjji
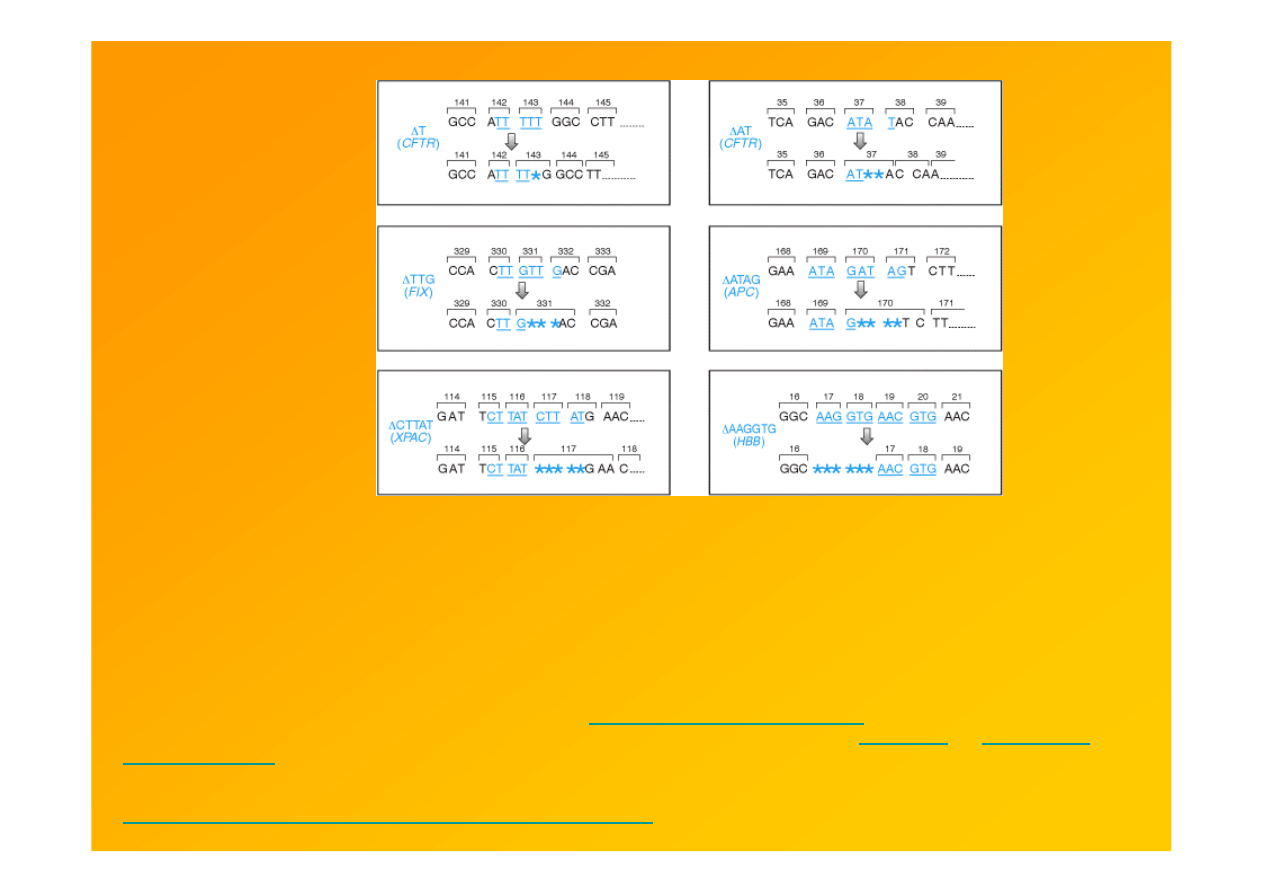
Fig. 9.14. Krótkie tandemowe powtórzenia (1-6-nt) są gorącymi m-scami delecji/insercji.
CFTR, cystic fibrosis transmembrane regulator
FIX, factor IX (hemophilia B)
APC, adenomatous polyposis coli
XPAC, xeroderma pigmentosa complementation group C
HBB, β-globin (β-thalassemia).
Original references are listed in Appendix 3 of
Cooper and Krawczak (1993)
. Though not illustrated here,
small insertions are often tandem repeats of sequences flanking them (see
Table 8.1
of
Cooper and
Krawczak, 1993
).
Strachan & Read 1999 Human Molecular Genetics 2, Garland Sci
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.1134
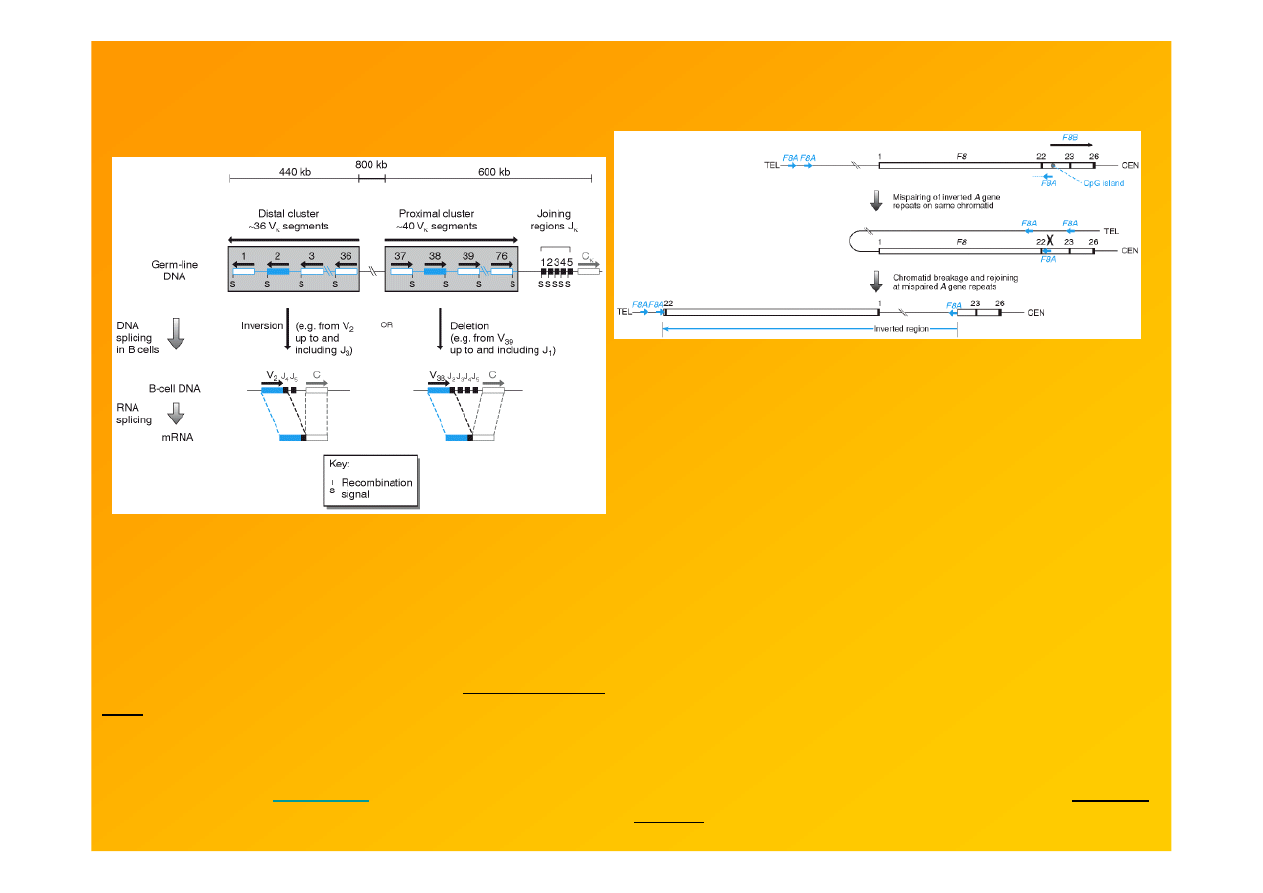
(patogenne) inwersje mogą być skutkiem
wewnątrzchromatydowej rekombinacji między odwróconymi powtórzeniami
Figure 8.28. Inversion or deletion results in V-J splicing
to produce functional Ig κ light chain genes. The human κ
light chain gene cluster contains about 76 Vκ segments
arranged in two large clusters, in opposite orientations. V
segments in the distal cluster have the opposite orientation
to the Jκ segments and the single Cκ sequence. As a result,
the DNA rearrangements used to splice distal Vκ segments
to a Jκ segment are megabase inversions ( Weichhold
et al.
,
1990). Those in the proximal cluster can undergo V-J joining
by a somatic recombination resulting in a deletion of the
intervening chromosomal segment, most likely through an
intrachromatid recombination event such as that used in
class switching (see
Figure 8.29
).
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.900
Figure 9.20. Inversions disrupting the factor VIII gene
result from intrachromatid recombination between inverted
repeats. For the sake of clarity only exons 22, 23 and the
first and last exons (1 and 26) of the factor VIII gene (
F8
;
open box) are shown. Intron 22 of this gene contains a CpG
island from which the
F8B
gene internal is transcribed in the
same direction as the factor VIII gene, and expression
involves splicing of a novel exon within intron 22 on to exons
23–26 of the factor VIII gene. The internal
F8A
gene is also
transcribed from the intron 22 CpG island but in the opposite
direction. Two other sequences closely related to
F8A
are
found about 500 kb upstream of the factor VIII gene and are
transcribed in the same direction as the factor VIII gene and
the opposite direction to that of the
F8A
gene. The high
degree of sequence identity between the three members of
the
F8A
gene family means that pairing of the
F8A
gene with
one of the other two members on the same chromatid can
occur by looping back of the chromatid. Subsequent chromatid
breakage and rejoining can result in an inversion of the region
between the
F8A
gene and the other paired family member,
resulting in disruption of the factor VIII gene (see Lakich
et
al.
, 1993).
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.1137

uszkodzenia DNA/jądro ludzkiej komórki (46 chr., 6×10
9
pz)
typ
tempo indukcji [#/godz]
tempo naprawy [#/godz]
Hydroliza
utrata zasad
Pu (A, G)
500
szybkie (BER)
Py (C, T)
25
Deaminacja
C, 5mC
100-500
szybkie (BER)
Oksydacja
8oxoG
100-500
szybkie (BER)
nicks
2 500
20 000 (ligaza DNA)
Alkilacja
3mA
25
szybkie (BER)
O
6
mG
?
? (transferaza Me)
UV (dimery Py)
50 000
50 000 (NER)
http://www.biology.ualberta.ca/courses/genet408/index.php?Page=6827, Lec02
W ludzkim genomie jest co najmniej 130 genów białek naprawy DNA – mutacje niektórych,
np. Ogg1 naprawiającego 8oxoG, nie są letalne, bowiem Reperacja, Replikacja, Rekombinacja
działają w skoordynowany sposób, ale najczęściej nieprawidłowe działanie któregokolwiek
ze składników 3R prowadzi do niestabilności genomu.
Wyszukiwarka
Podobne podstrony:
lab1 12 id 258878 Nieznany
II CSK 330 12 1 id 209820 Nieznany
Cwiczenie 12 id 99084 Nieznany
Calki, IB i IS, 2011 12 id 1073 Nieznany
zestaw 12 id 587976 Nieznany
ldm rozmaite 12 id 264070 Nieznany
IMG 12 id 210985 Nieznany
Instrukcja 12 id 215508 Nieznany
pamieci 12 id 348557 Nieznany
LITERATURA 12 id 270804 Nieznany
BiolMol 10 id 87436 Nieznany
Fizyka i astronomia 12 id 17675 Nieznany
img 12 id 211037 Nieznany
KPG 433 12 id 249386 Nieznany
PMWEU 12 id 363556 Nieznany
Fizjologia Cwiczenia 12 id 1743 Nieznany
cw 12 id 122179 Nieznany
II CSK 445 12 1 id 209821 Nieznany
więcej podobnych podstron