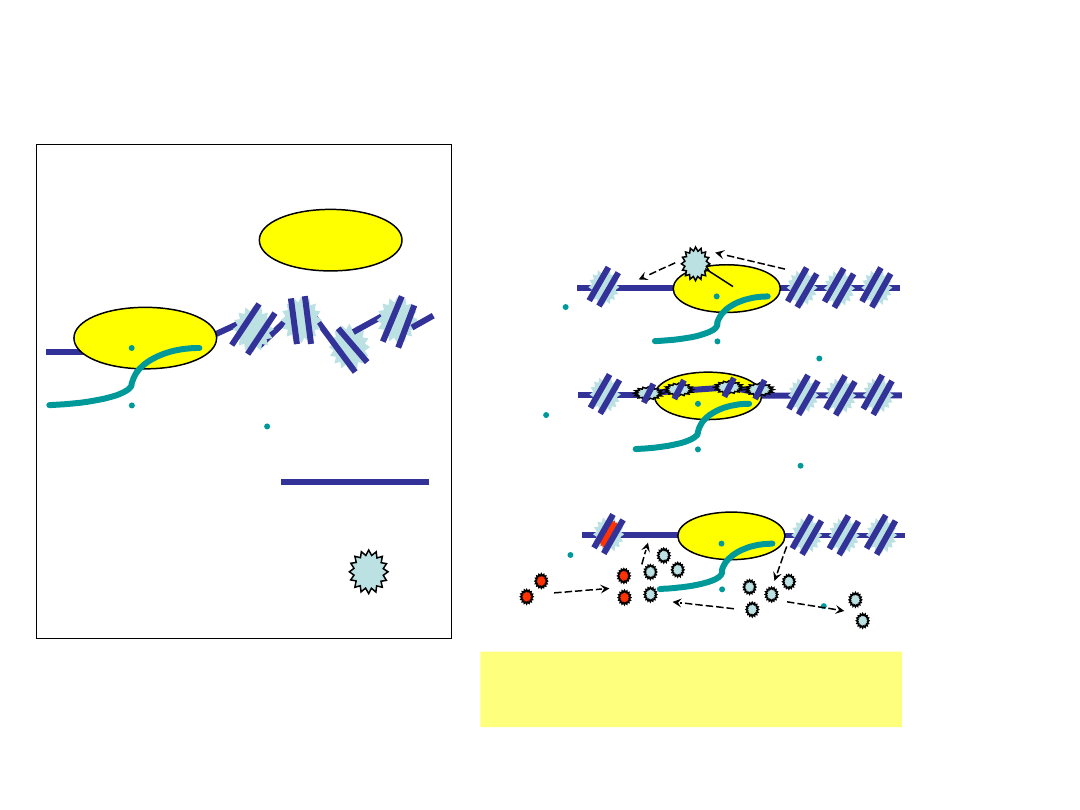
transkrypcja chromatyny
problem:
problem:
kompleks pol II RNA
500 kD +
nukleosom:
145 bp
DNA
= 100 kD
rdzeń histonowy = 100 kD
skip:
split:
strip:
Jak to może działać
Jak to może działać
:
:
Skip czy Split czy Strip
?
dysocjacja
histonów?
NO
NO
YES
Thoma 1991 Trends Genet 7: 175
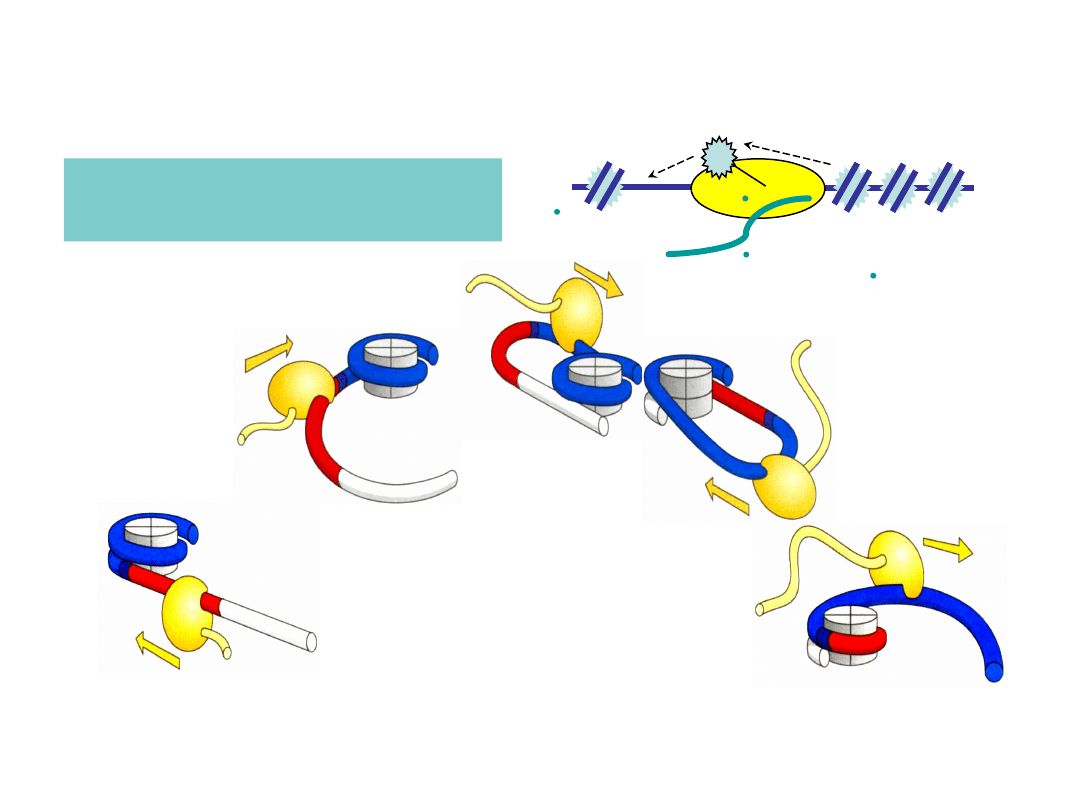
skip ?
Jak by to się mogło
Jak by to się mogło
dziać?
dziać?
Studitsky, Clark, Felsenfeld 1974 Cell 76, 371
przesuwanie się
wzdłuż pętli DNA
tak działa pol III RNA i fagowe pol RNA
(także zależne od ATP kompleksy
remodelujące)
Studitsky i in. 2004 Trends Bioch Sci 29: 127-135
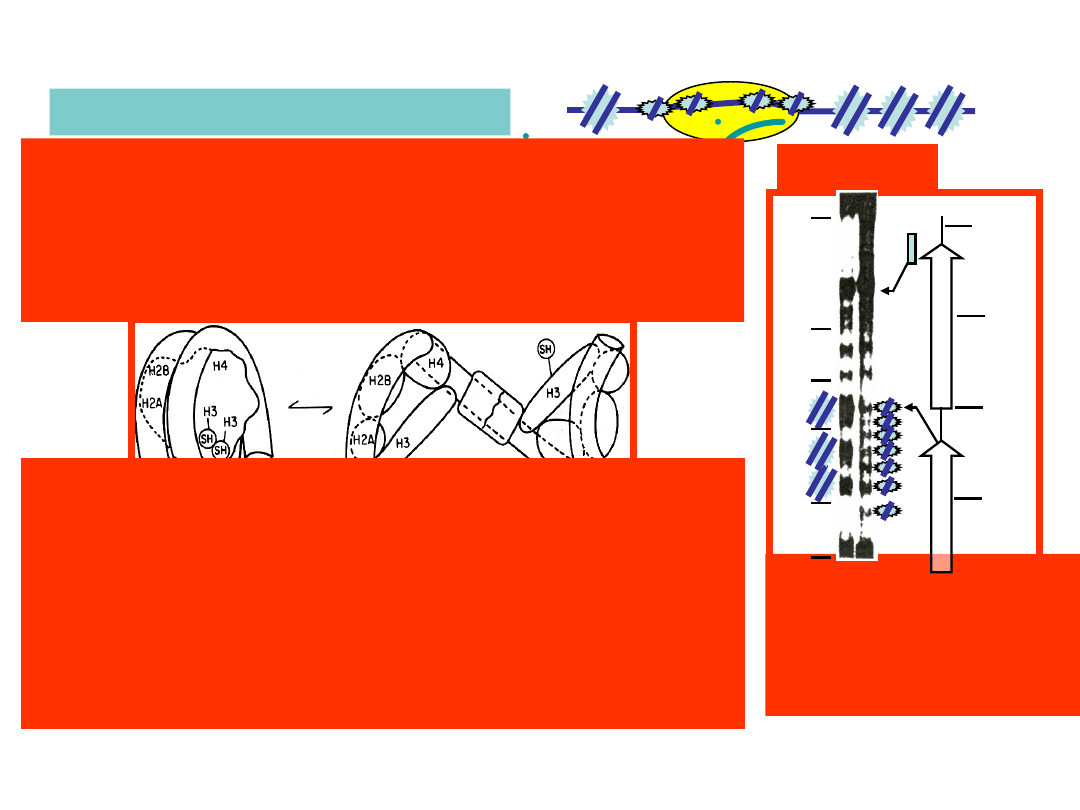
split ?
Jak by to się mogło dziać?
Jak by to się mogło dziać?
w ME transkrybowana chromatyna wydaje się być
„gładka” (o jednakowej grubości), nić jest
grubsza niż gołe DNA,
przeciwciała wykrywają obecność histonów
czy to są
artefakty?
a
Lee & Garrard 1991 EMBO J. 10,
607
w transkrybowanej
chroma-tynie po
trawieniu DNazą I
pojawiają się
powtórzenia połówek
nukleosomów
+2.0kb
+2.5kb
+3.0kb
+3.5kb
C
IN
2
H
S
P
8
2
kb
3.53
2.03
1.58
1.33
0.98
0.82
Transcription
-
+
Prior & Allfrey 1983 Cell 34, 1a0a33a
transkrypcja eksponuje dwie normalnie ukryte
reszty
cysteinowe H3 na działanie czynników
modyfikujących
i chromatografię powinowactwa Hg
mononukleosomy wydają się być niesfałdowane w
postaci
dwu tetramerów histonów + nieduże białka
niehistonowe
powolna transkrypcja przez pol II RNA powoduje wyparcie z
nukleosomu jednego dimeru H2A-H2B
Studitsky i in. 2004 Trends Bioch Sci
29: 127-
135
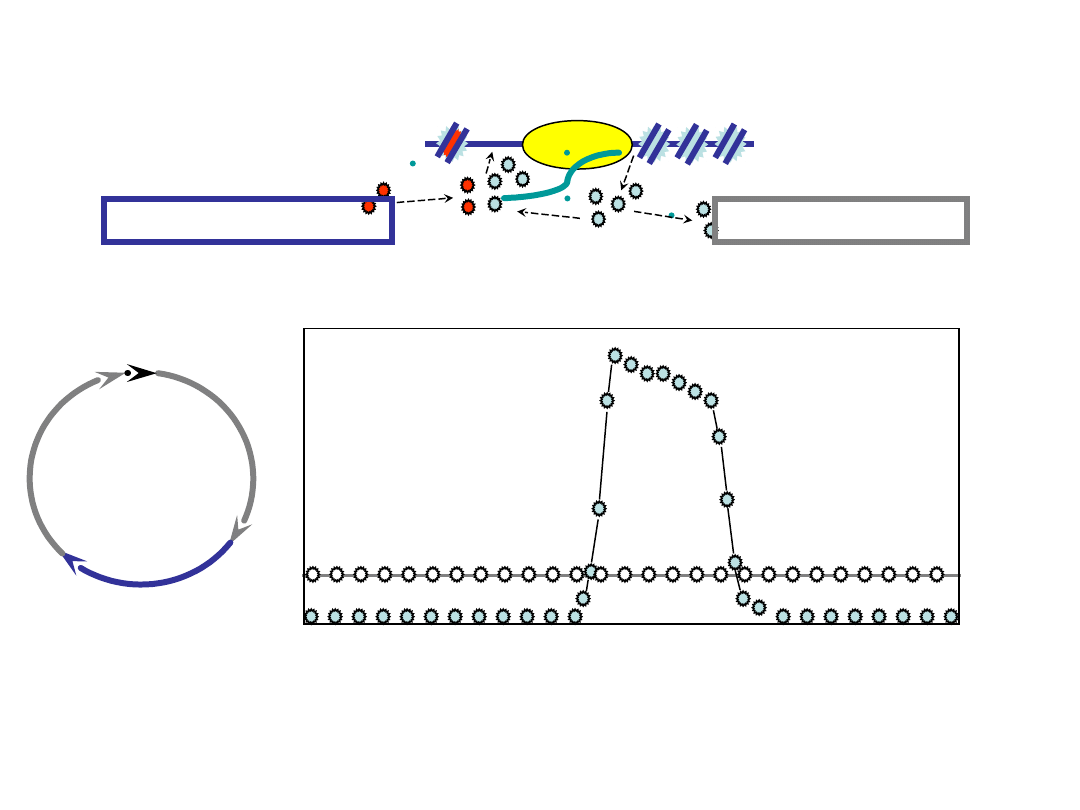
strip ?
problem:
które histony są dostępne do tworzenia nukleosomów ?
synteza histonów
obrót histonów
faza S
faza
G
faza
G
2
mitoz
a
M
faza G
1
faza S
faza G
2
M
zależne od replikacji
konstytutywne zastępcze histony
u wszystkich eukariota H3.3, u roślin wysoce
zacetylowany H3.2
aktywna transkrypcja przez pol II RNA
powoduje rozpad
nukleosomów
Kulaeva i in. 2007 Mutat Res 618: 116-129

dynamika struktury
chromatyny
• w obrębie wysp CpG jest zależna od stanu
metylacji DNA
• modyfikacje histonów - „kod histonowy”
• remodelowanie chromatyny
* zależne od ATP
* niekiedy za pośrednictwem interferencji RNA
(RNAi)
NOBEL 2006 !
przykłady zależnej od RNA heterochromatynizacji:
- inaktywacja piętnowanego chromosomu X (Xi),
-
wyciszenie centromerów i loci typu
koniugacyjnego u droż-
dży rozszczepkowych
Schizosaccharomyces pombe
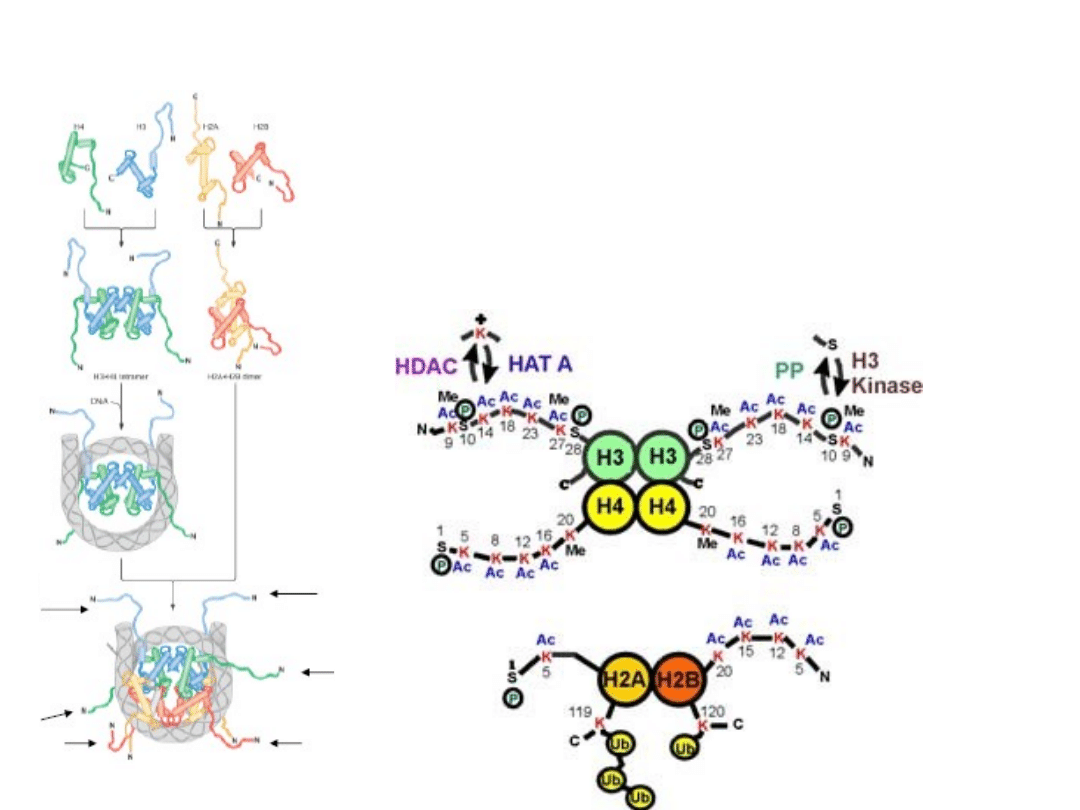
modyfikacje histonów
K – lizyna
S - seryna
acetylacja lizyn
metylacja lizyn (mono i di) oraz arginin (mono, di i tri)
fosforylacja seryn i treonin
ubikwitynacja C-końcowych lizyn
ADP-rybozylacja
koniugacja SUMO do lizyn
Allfrey i in. PNAS 51, 786-794
Bradbury, BioEssays 14, 9-16
H4 N-terminus
H3 N-terminus
Ac-S-G-R-G-
K
-G-G-
K
-G-L-G-
K
-G-G-A-
K
-R-H-R-
K
-V-L-R-D-
+
+
+
+
+
+
+ + +
+
Ac or Me
Ac
Ac
Ac
Ac
5
8
12
16
20
A-R-T-
K
-Q-T-A-R-
K
-S-T-G-G-
K
-A-P-R-
K
-Q-L-A-T-
K
-A-A-R-
K
-S-A-P-
Me
Ac or Me
Ac
Ac
Ac
Ac
+
+
+ +
+
+
+
+
+
4
9
14
18
23
27
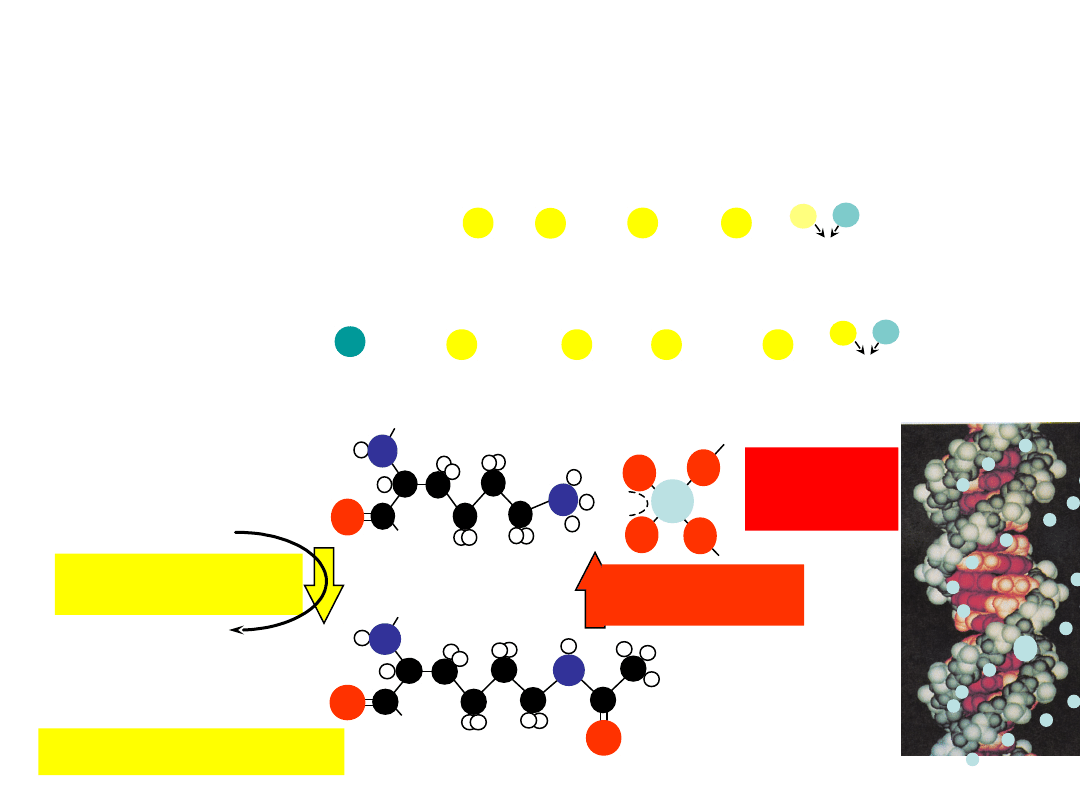
lizyna
-N-Acetyl-Lysine
HAT (Histone
Acetyl-Transferase)
HDAC (Histone
Deacetylase)
odwracalne reakcje
O
N
C
C C
C
C
C
N
+
P
O
O
O
P
O
-
wiązanie
DNA
brak wiązania DNA
O
N
C
C C
C
C
C
N
O
C
C
Acetyl-CoA
CoA
P
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
acetylacja konserwowanych lizyn
(N końce H3 i H4 oraz wzorce ich acetylacji są konserwowane)
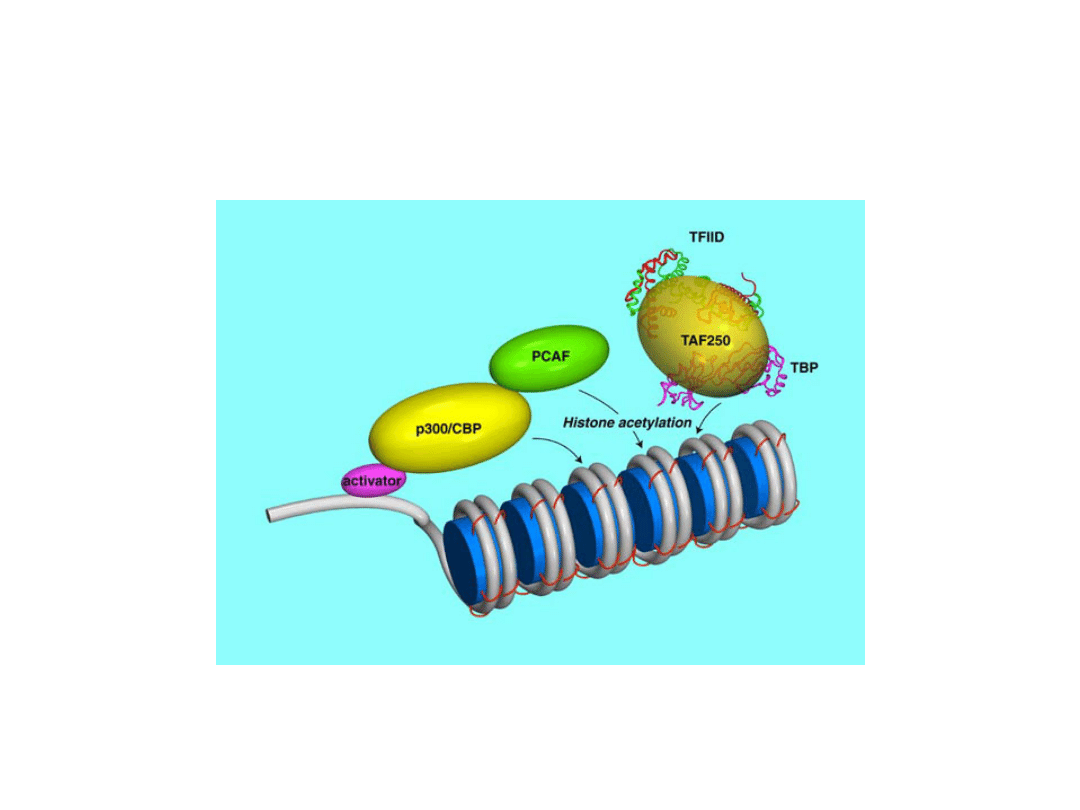
wielość kompleksów HAT
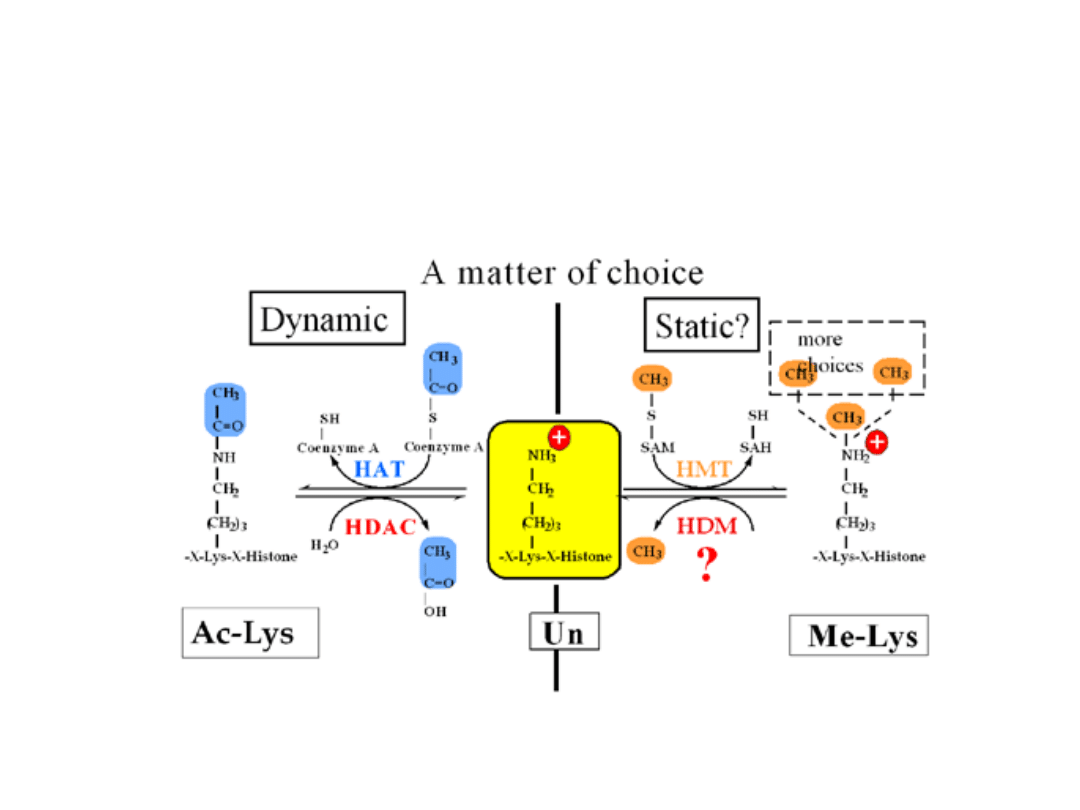
modyfikacje lizyny
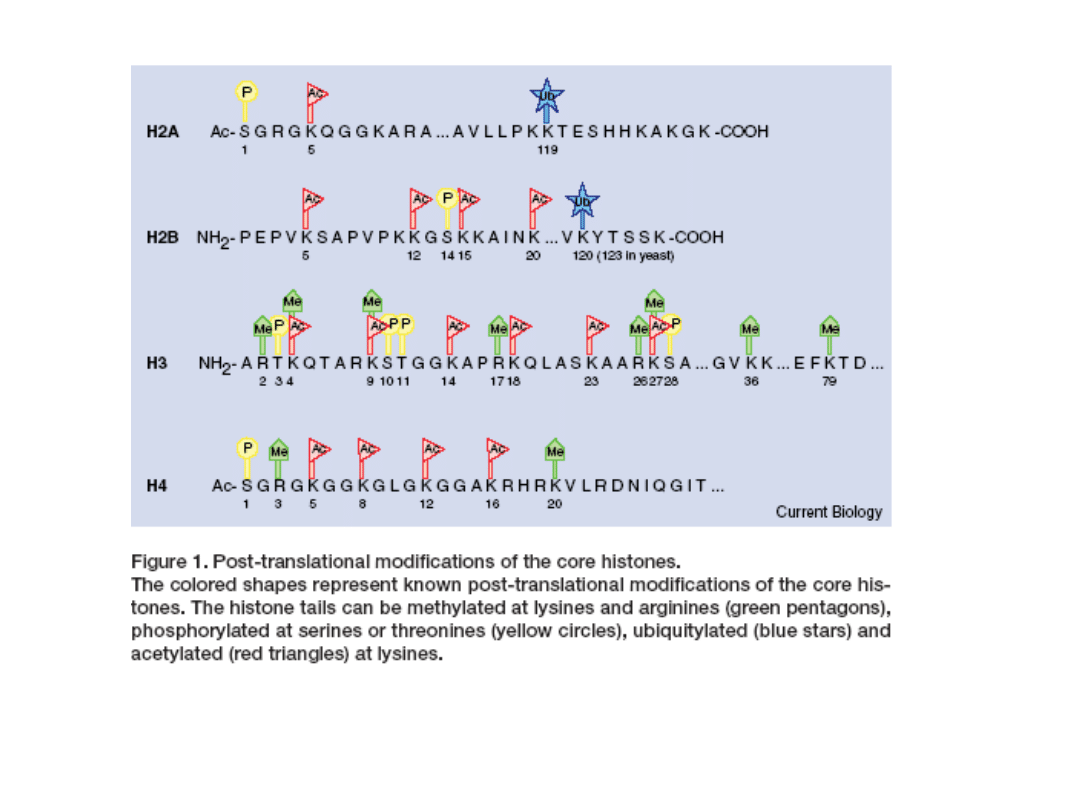
Peterson, Laniel 2004 Curr. Biol. 14:R546-51

kod histonowy daje możliwość
acetylacji H4 - w 4 pozycjach,
fosforylacji H3 – w 2 pozycjach…
>4 000 kombinacji modyfikacji pojedynczego
nukleosomu
kod histonowy
H4AcK8
H3AcK14
H3PhS10
H3MeK9 (trimetylacja)
brak acetylacji H3 lub
H4
H4AcK4
H4AcK12
H2APhS1
H2APhT119
H3PhT3
H3PhS10
H3PhS28
= represja transkrypcji
= tworzenie nowych nukleosomów w
fazie S
= transkrypcja
= skondensowana mitotyczna chromatyna
Peterson, Laniel 2004 Curr Biol 14,
R546
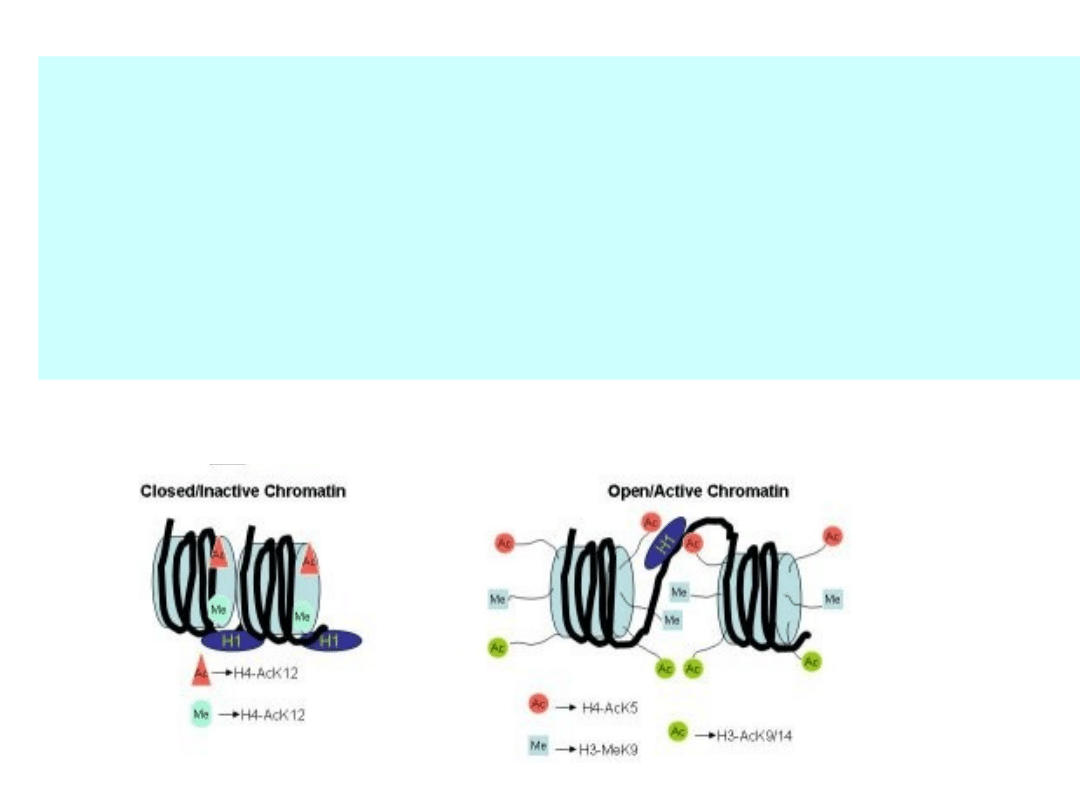
ogólne cechy chromatyny
nieaktywnej
aktywnej
* deacetylacja histonów
* H4AcK5, H3AcK9/14
* H4MeK12
* H3MeK9
* 5mCpG
* brak metylacji DNA
* obecność HP1 (izoformy α,β,γ)
* m-sca nadwrażliwe na
DNazę I
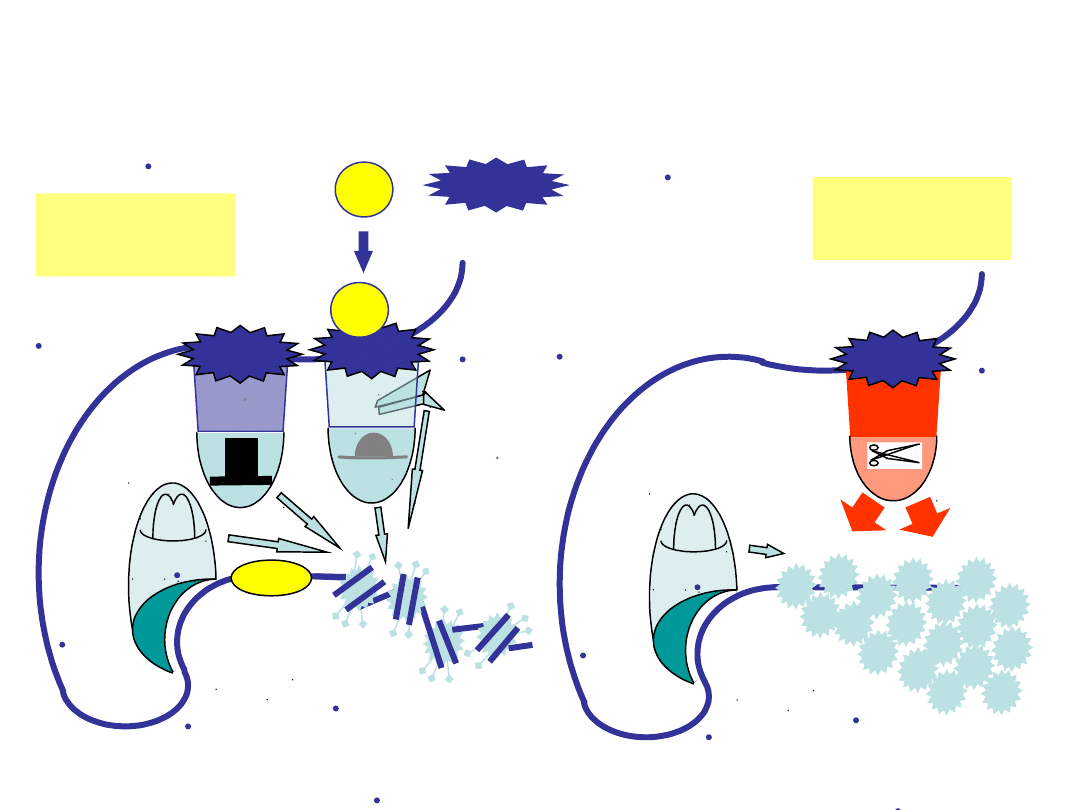
acetylacja histonów na
promotorach
represja
transkrypcji
receptor hormonu
tarczycy
przykład czynnika
transkrypcyjnego
korepresor
TAF 250
II
TB
P
HAT
TA
TA
TACCCG
N-CoR
Sin3
RPD3
HDAC
brak hormonu
Wolffe 1997 Nature 387: 16 zmienione
TAF 250
II
TB
P
HAT
TA
TA
pol.II
HAT
GCN5
ACGGTC
ADA2
ADA3
aktywacja
transkrypcji
koaktywator
HAT
TR/RXR
TH
h
o
rm
o
n
+
HAT
P/CAF
p300
CPB
HAT
TACCCG
TH
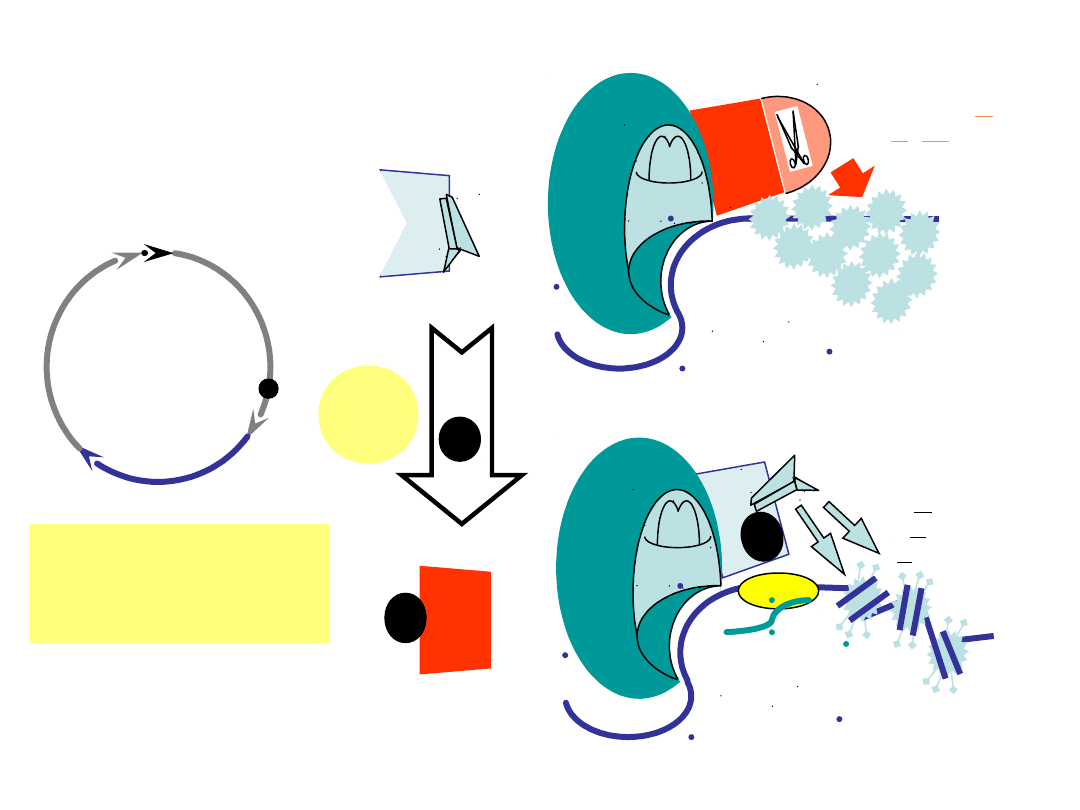
aktywacja
genów fazy
S
Ait-Si-Ali et al. Nature 396, 184 (1998) zmienione
kontrola przejścia
G1-S punkt R
(ang. Restriction)
HDAC1 (ang. Histone
Deacetylase)
Rb
TAF 250
II
TB
P
HAT
TA
TA
geny fazy S WYŁĄCZONE
korepresor Rb
CPB
HA
T
P
TAF 250
II
TB
P
HAT
TA
TA
geny fazy S WŁĄCZONE
aktywny HAT
(ang. Histone
Acetyl
Transferase)
Rb
P
słaba aktywność
H
A
T
CPB
niska aktywność HAT
koaktywator
S
G1
G2
mitoza
R
cyklina E/
/kinaza
cdk2
P
+
pol.II
korepresor
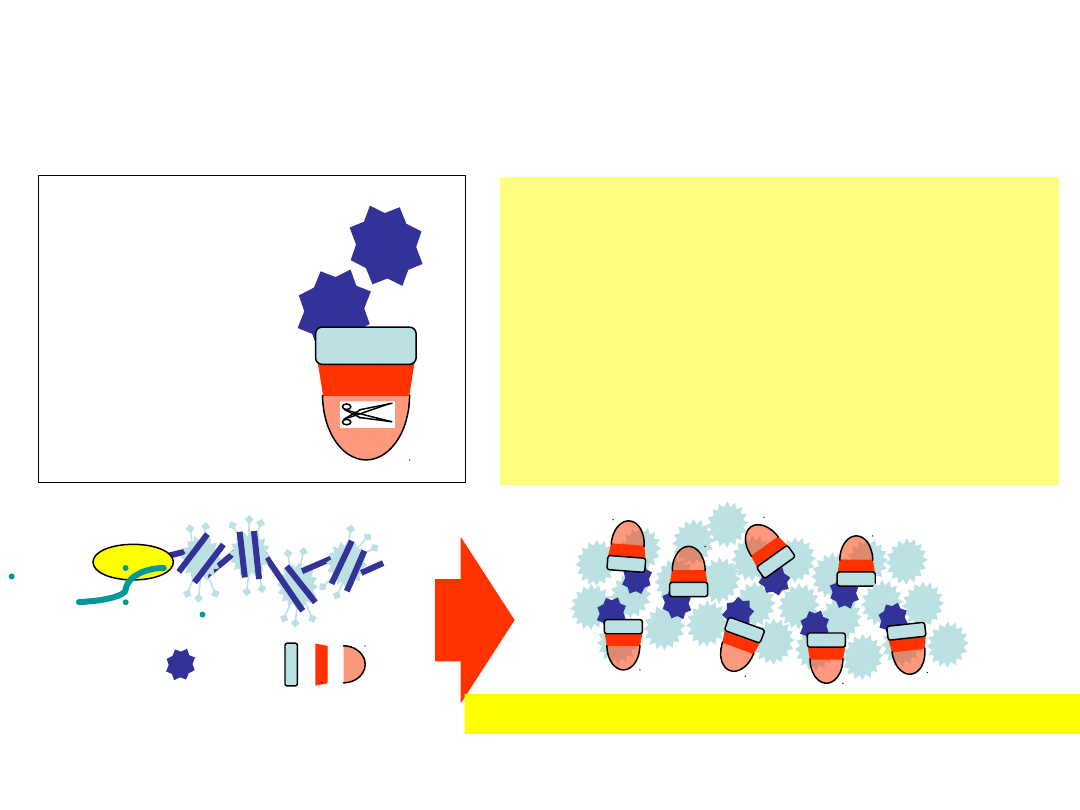
deacetylacja chromatyny
wyciszenie transkrypcyjne – inaktywacja chromosomu X
Nan et al. Mol.Cell.Biol. 16, 414 (1996); Cell 88, 1 (1997); Jones et al. Nat.Genet. 19, 187 (1998)
metylacja C w CpG występuje w
wyciszonych genach i inaktywowanych
chromosomach X
metylacja CpG jest utrzymywana po
replikacji dzięki metylazie DNA
specyficznej dla hemimetylowanego DNA
5mC wiąże represor transkrypcyjny MeCP2
(MethylC-binding Protein-2)
MeCP2 wiąże korepresor Sin3 z deacetylazą
histonów RPD3
+
+
hypoacetylowane wyciszone włókno chromatyny
represor transkrypcji
HDAC
Sin3
RPD3
MeCP2
5’
..
p
C
p
G
p
..
3’
me
5
3’
..
p
G
p
C
p
..
5’
5
me
korepresor
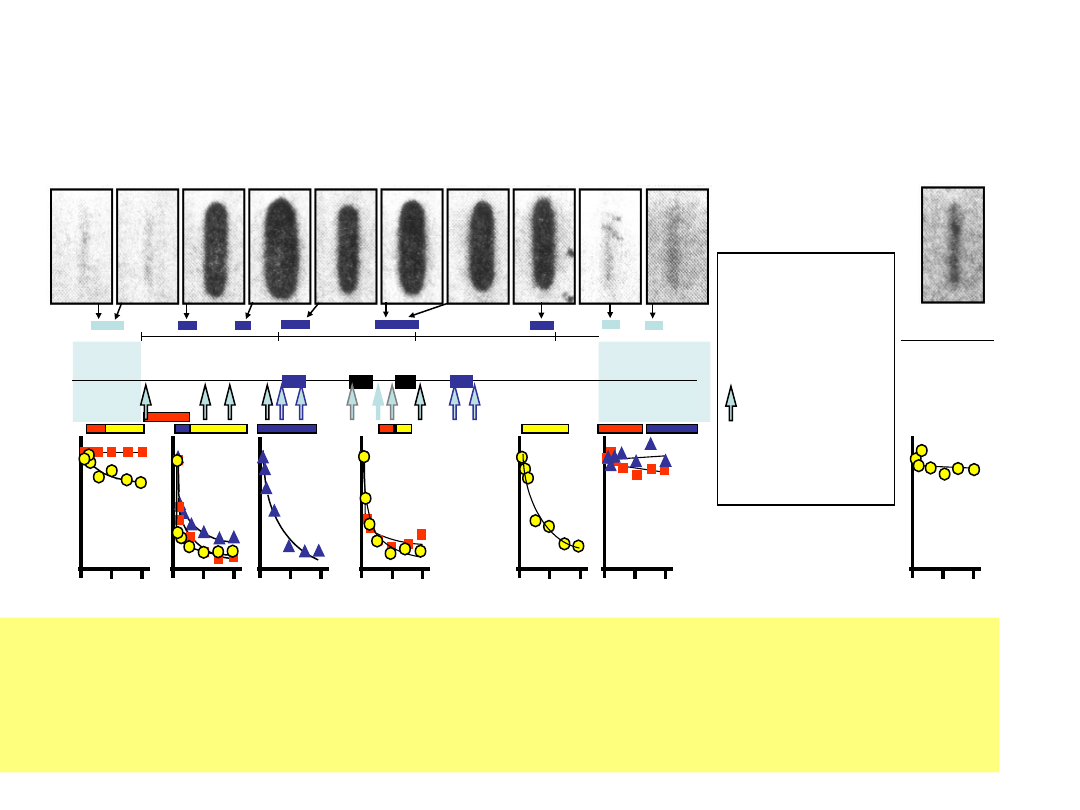
acetylacja domen
chromatynowych
Hebbes, Clayton, Thorne, Crane-Robinson 1994 EMBO J. 13, 1823
przykład: domena kurzych genów -globinowych
wysoki poziom acetylacji chromatyny w całych domenach (pętlach
DNA) indukuje ogólną wrażliwość chromatyny na DNazę I
wewnątrz tych domen w obrębie funkcjonalnych genów lub miejsc wiązania
czynników transkrypcyjnych w strukturze chromatyny występują małe miejsca
nadwrażliwe na DNazę I
domain
boundary
domain
boundary
H
A
0
10
20
30kb
m-sce nadwraż-
liwe na DNazę I
geny -globiny
kompleksy nukleoso-
mów z antyAcK
DNA loop domain
ogólna wrażliwość
na DNazę I
kontrola:
nieaktywny gen
kurzej
owalbuminy
0 1 2
DNase I (u/ml)
D
N
A
r
e
m
a
in
in
g
DNase I
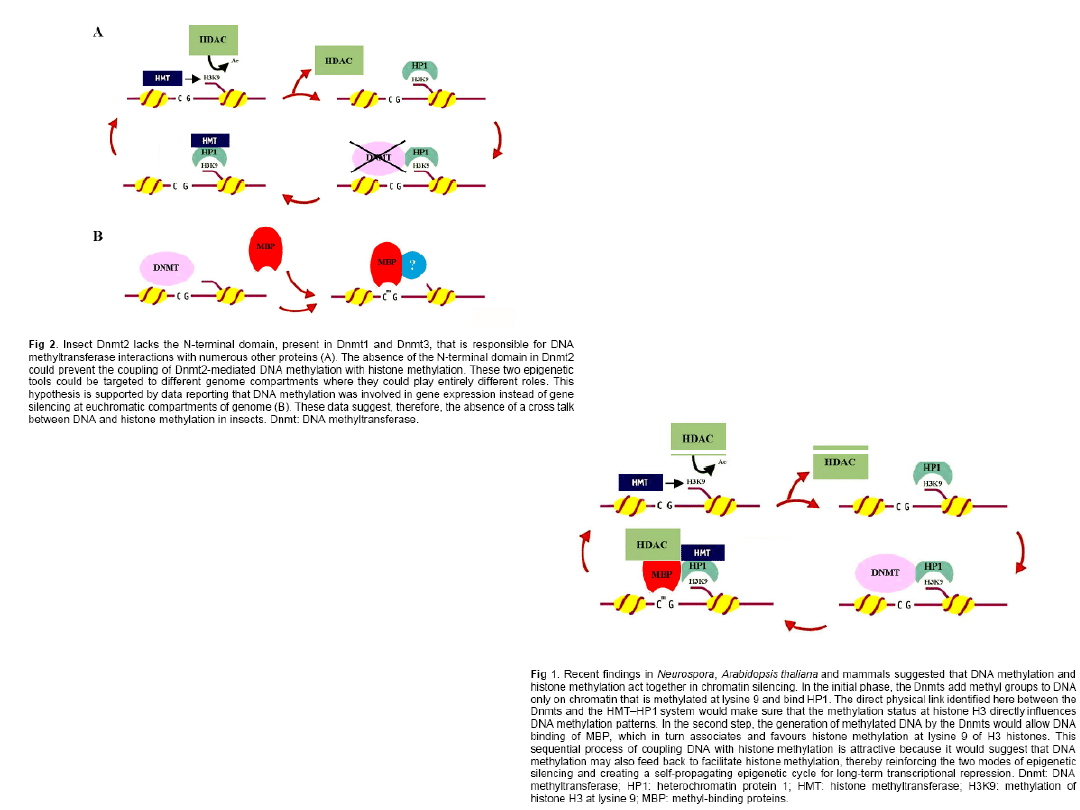
różnice gatunkowe
interakcji metylacji
DNA i histonów
owady – Dnmt2 nie ma N-końcowej
domeny odpowiedzialnej za
interakcje z in. białkami -
metylacja DNA i his - tonów nie są
powiązane
Neurospora, Arabidopsis,
człowiek – metylacja DNA i
histonów współdziałają w
wyciszaniu genów
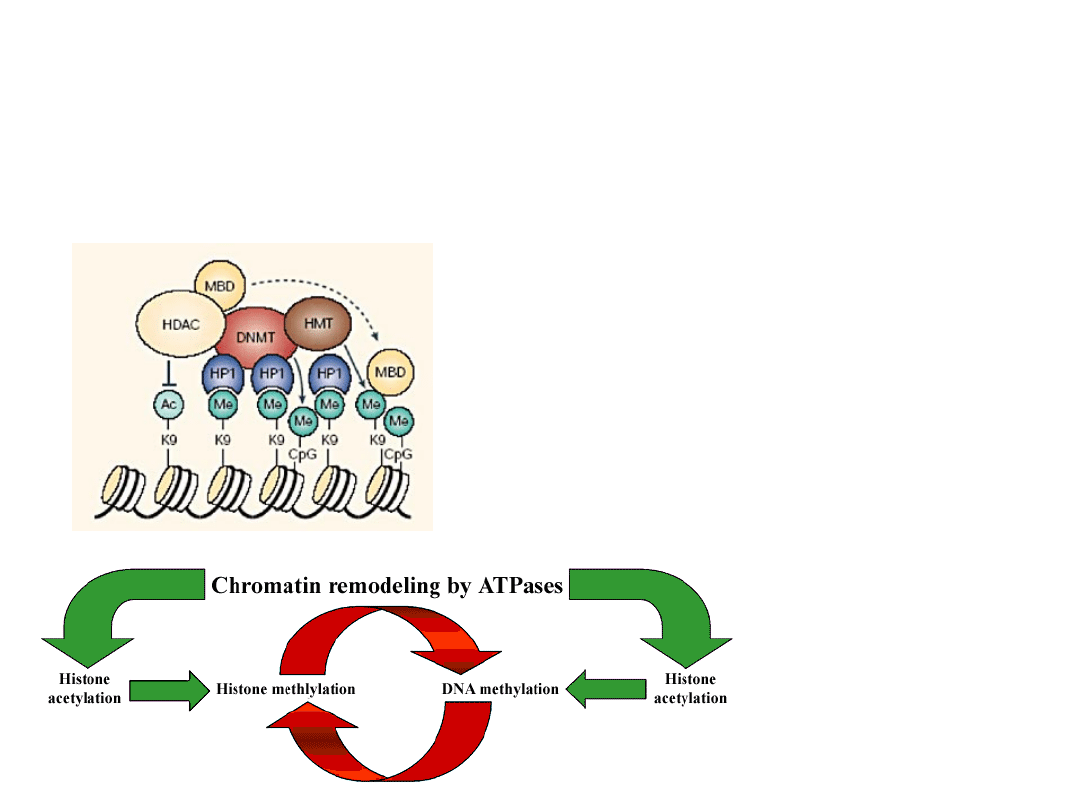
kolejność
deacetylacji
histonów,
metylacji
histonów,
metylacji DNA
jest różna
DNMT oddziałują z:
deacetylazami histonów (HDACs)
metylazami histonów (HMTases)
zależnymi od ATP kompleksami
remodeluja- cymi chromatynę
białkami strukturalnymi chromatyny (z
rodzi-
ny HP1)
kooperatywna i samowzmacniająca się
organizacja maszyneri modyfikujących
chromatynę i DNA odpowiedzialnych za
wyciszanie genów w komórkach normalnych
i nowotworowych
www.med.ufl.edu/biochem/keithr/fig4pt1.html
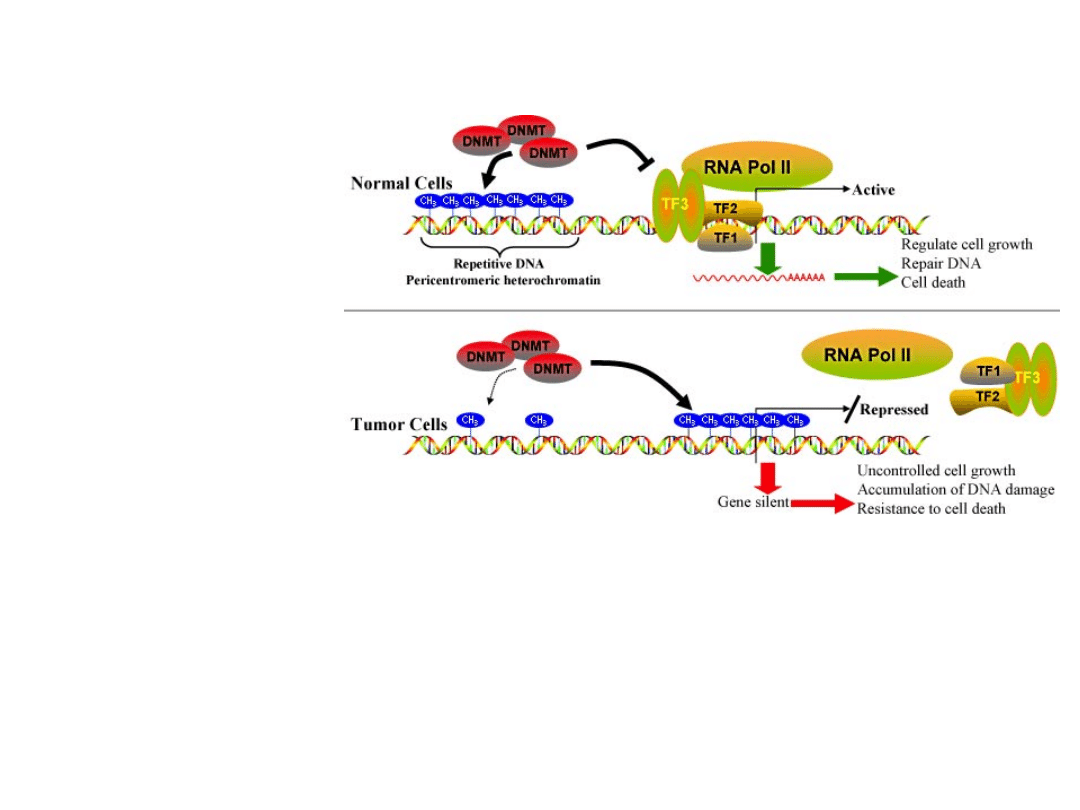
wiązanie DNMT do odpowiednich regionów
prawdopodobnie zachodzi dzięki interakcjom białko–białko
http://www.med.ufl.edu/biochem/keithr/fig3.html
komórki normalne –
metylacja DNA dotyczy
regionów
repetytywnych a
większość wysp CpG w
promotorach jest
niezmetylowana
komórki nowotworowe –
powtarzalne DNA traci
metylację, a wyspy
CpG promotorów
zyskują metylację, co
powoduje wyciszenie
bliskiego genu
Esteller, Almouzni 2005 EMBO Rep 6: 624-8
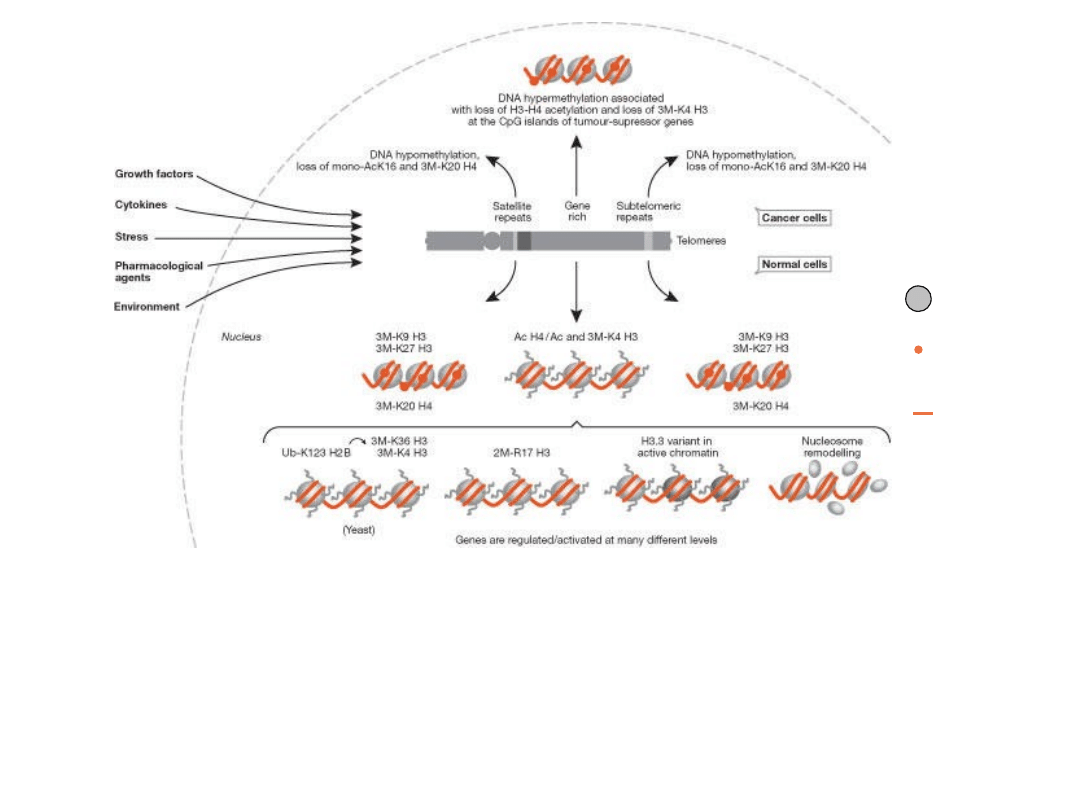
modyfikacje histonów i metylacja DNA integrują funkcje
jądrowe
zależność struktury nukleosomowej, lokalizacji
chromosomowej i aktywności transkrypcyjnej.
nukleosom
metylowan
e CpG
DNA
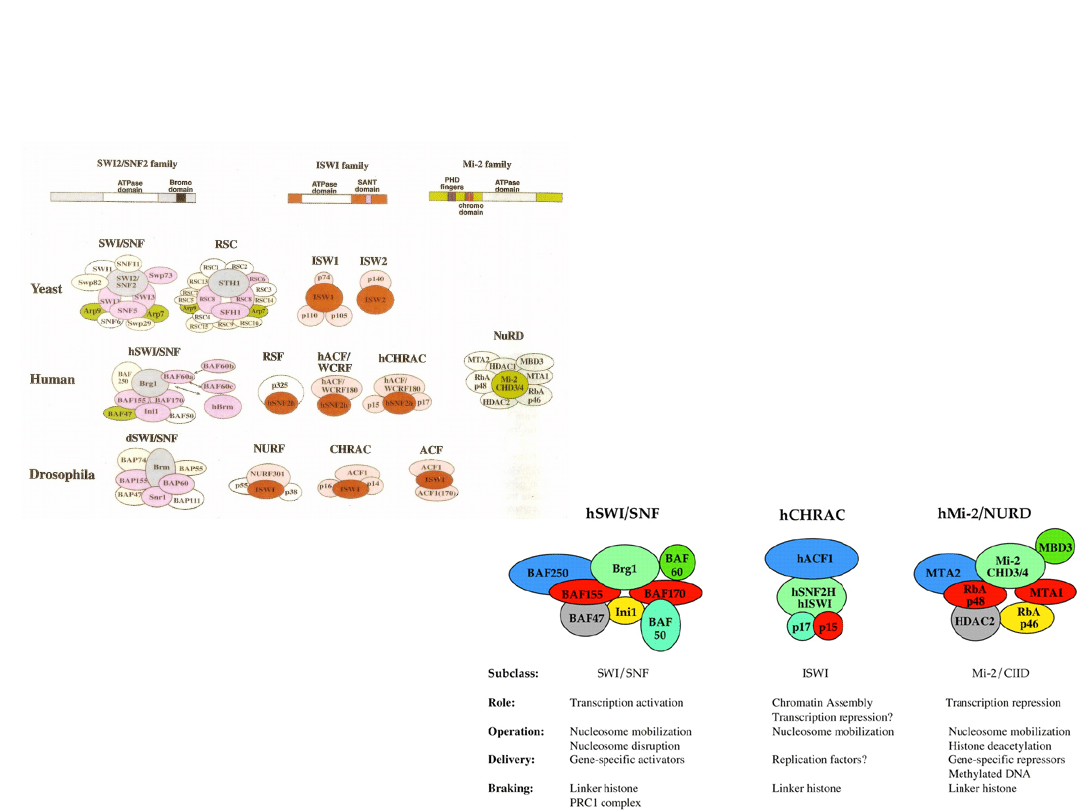
ATP- zależne kompleksy remodelujące chromatynę
3
klasy kompleksów konserwowane pomiędzy S.c., D.m., H.s.
Peterson 2002 EMBO Rep 3: 319–322
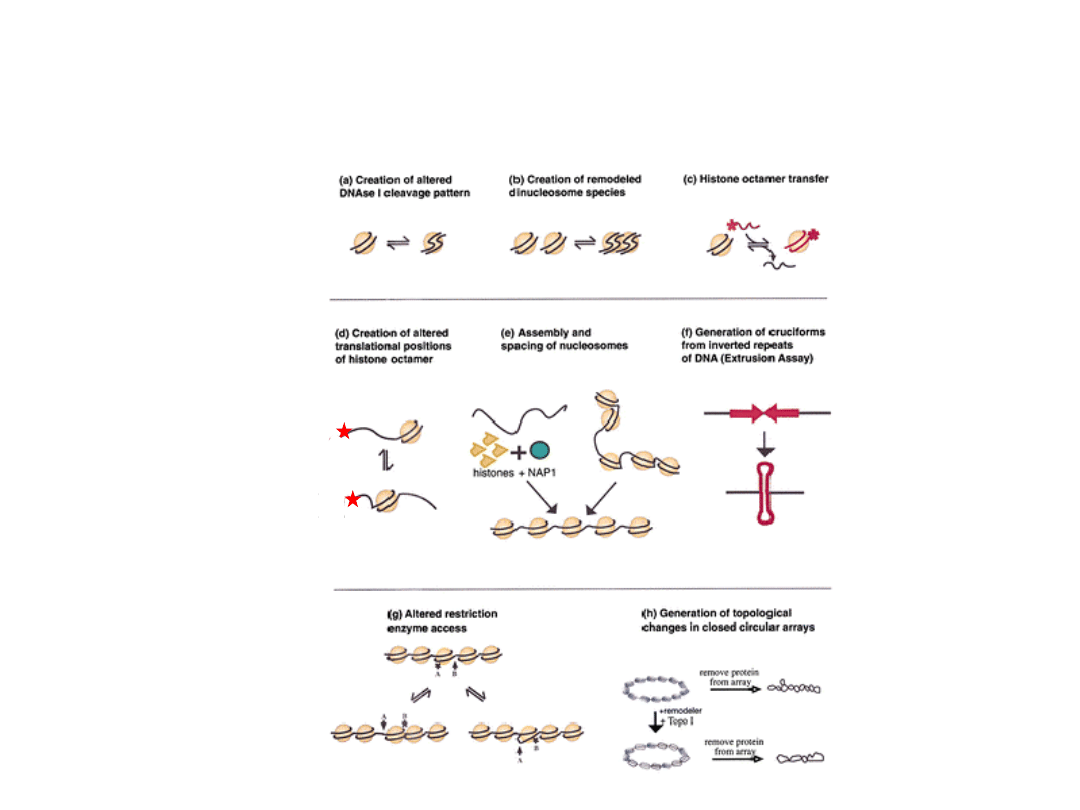
biochemiczna aktywność ATP-zależnych
kompleksów remodelujących chromatynę
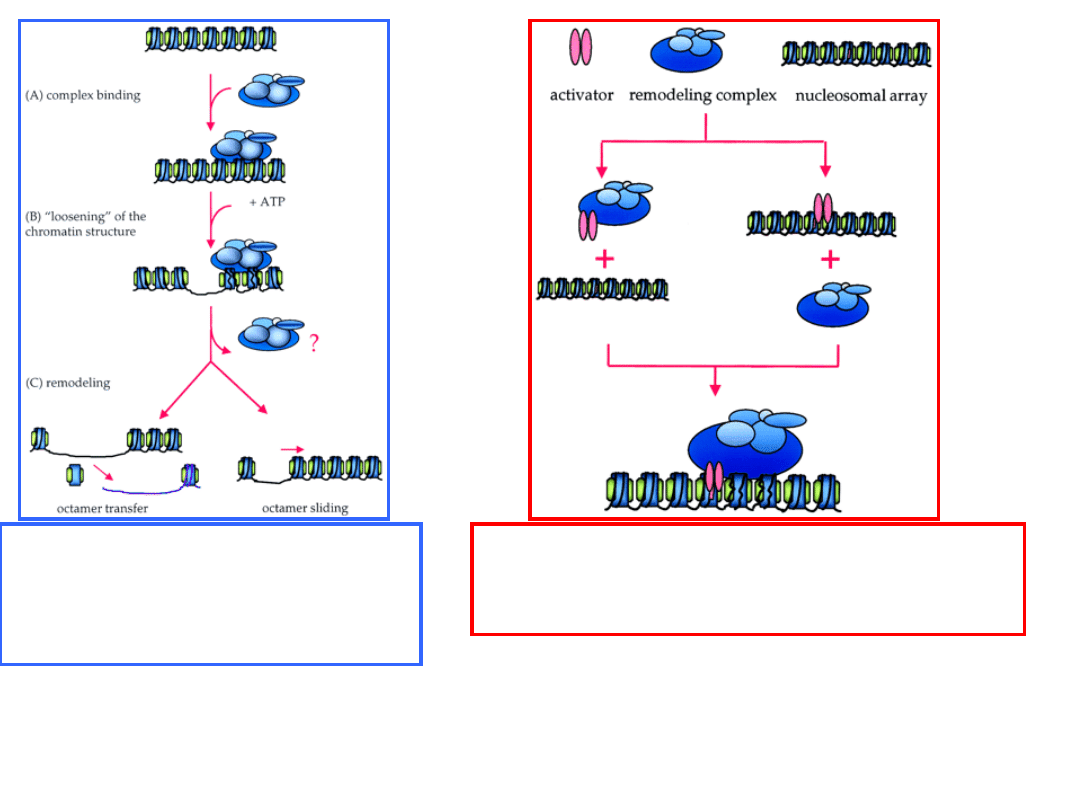
interakcje z czynnikami
transkrypcyjnymi decydują o
kierowaniu kompleksów do miejsc
docelowych
etapy działania kompleksów
prowadzące do przesunięcia,
transferu, rozpadu
nukleosomu
Vignali i in. 2000 Mol Cell Biol 20: 1899-1910
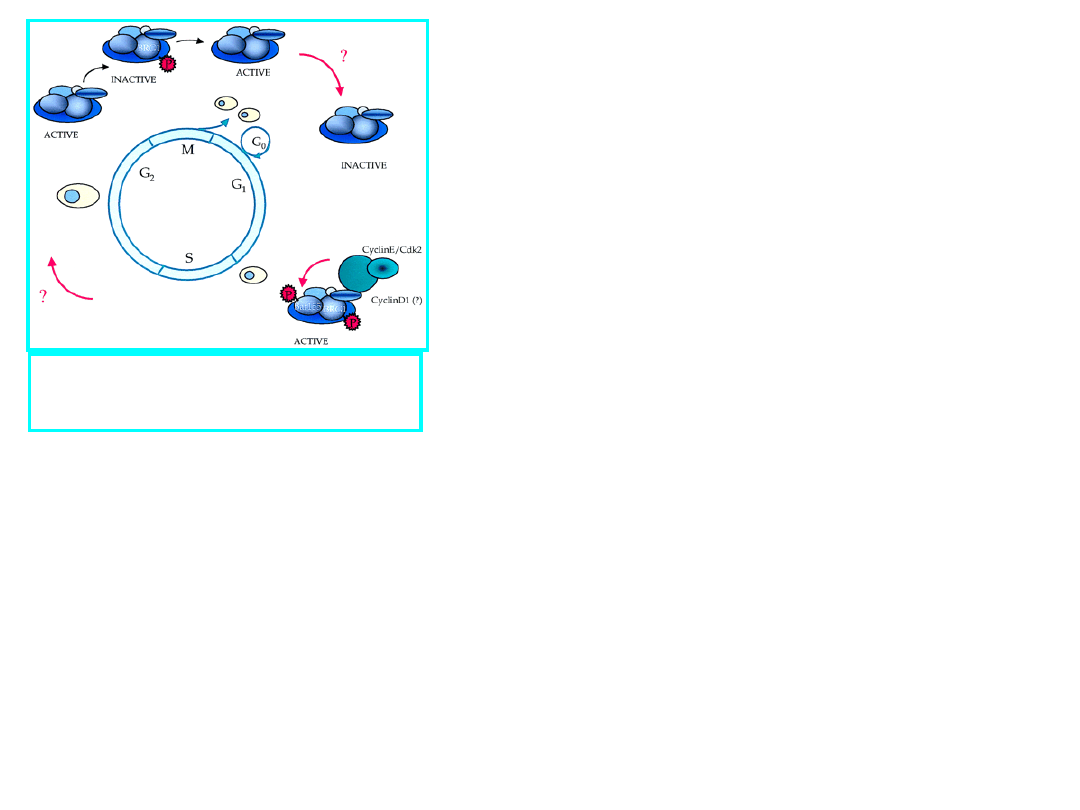
regulacja cyklu komórkowego
przez kompleks SWI/SNF
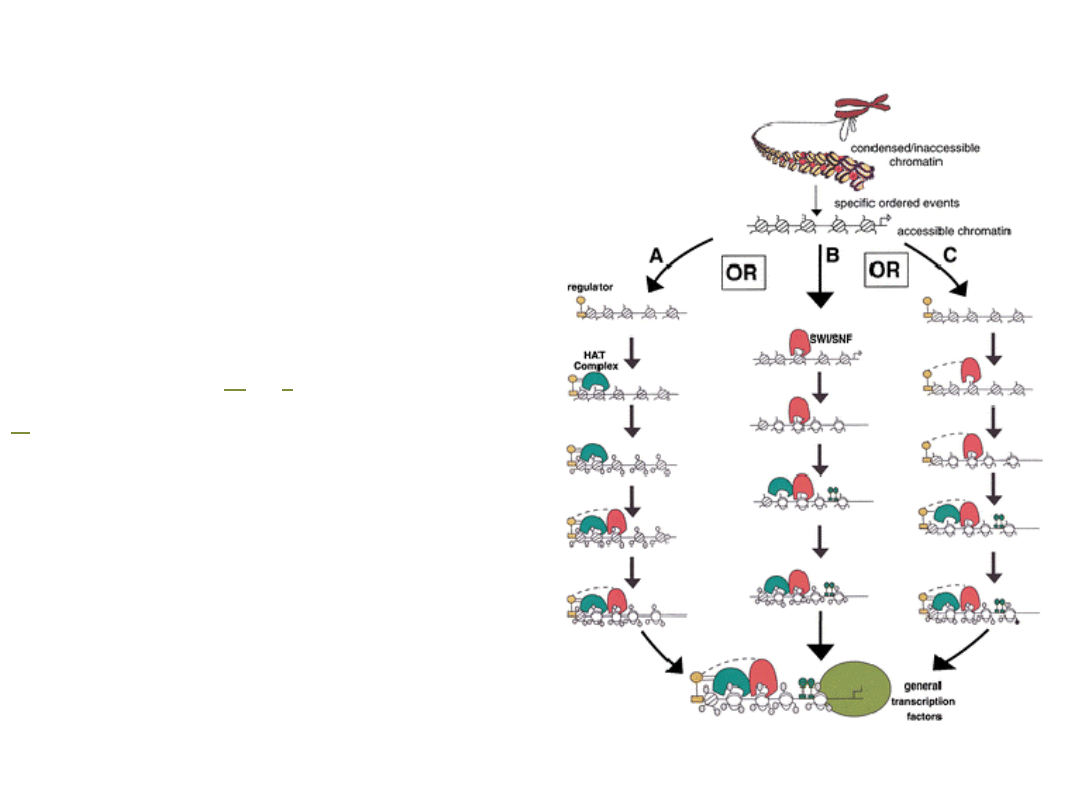
oddziaływania między:
kompleksami HAT,
kompleksami
remodelującymi
i
czynnikami transkrypcyjnymi
umożliwiają utworzenie
PIC
(ang. preinitiating
complex)
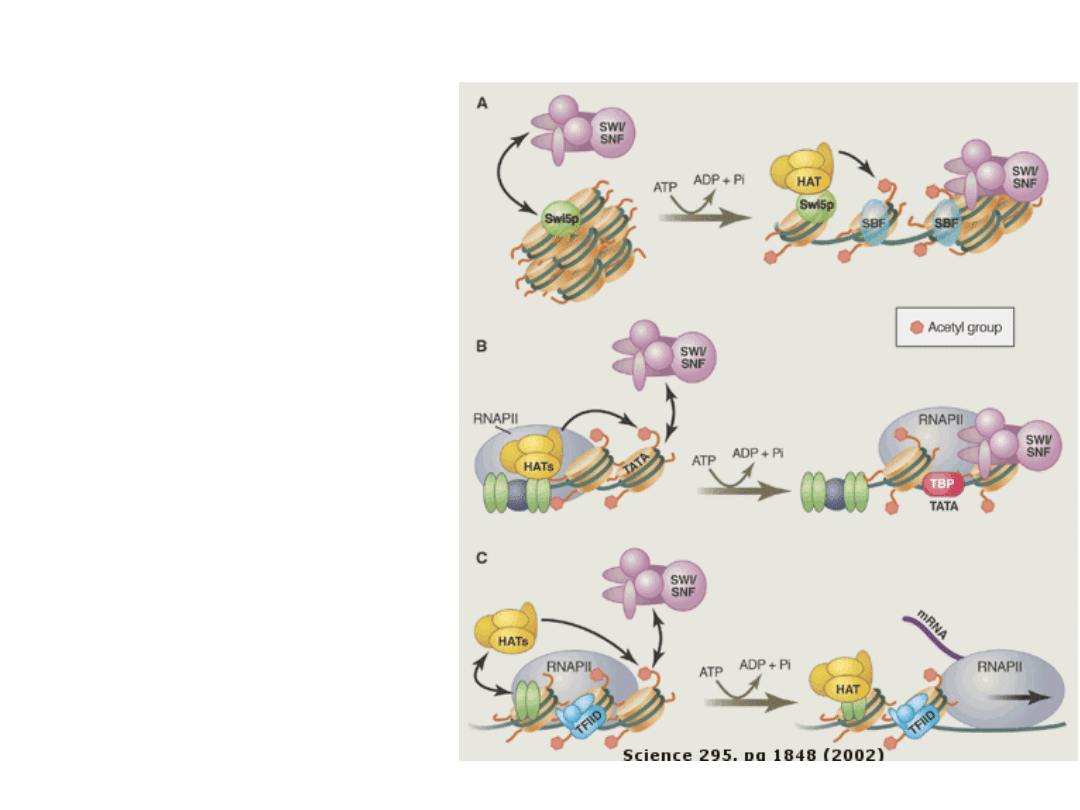
A.
promotor drożdżowego
genu HO - przed powstaniem
PIC aktywator
Swi5p
rekrutuje
SWI/SNF
i
HAT
(Gcn5p), co zwabia aktywator
SBF
B.
promotor ludzkiego IFN-β –
związane wyżej aktywatory
rekrutują
HATy
podczas
tworzenia PIC, acetylacja
histonów ułatwia
przyłączenie
SWI/SNF
, który
niszczy strukturę nukleosomu
związanego z promotorem
C.
promotor ludzkiego α
1
-AT-
kompleksy
HAT
(CBP, P/CAF) i
SWI/SNF
przyłączają się do
promotora po utworzeniu PIC
stymulując ekspresję
enzymy remodelujące chromatynę ułatwiają różne etapy aktywacji genów
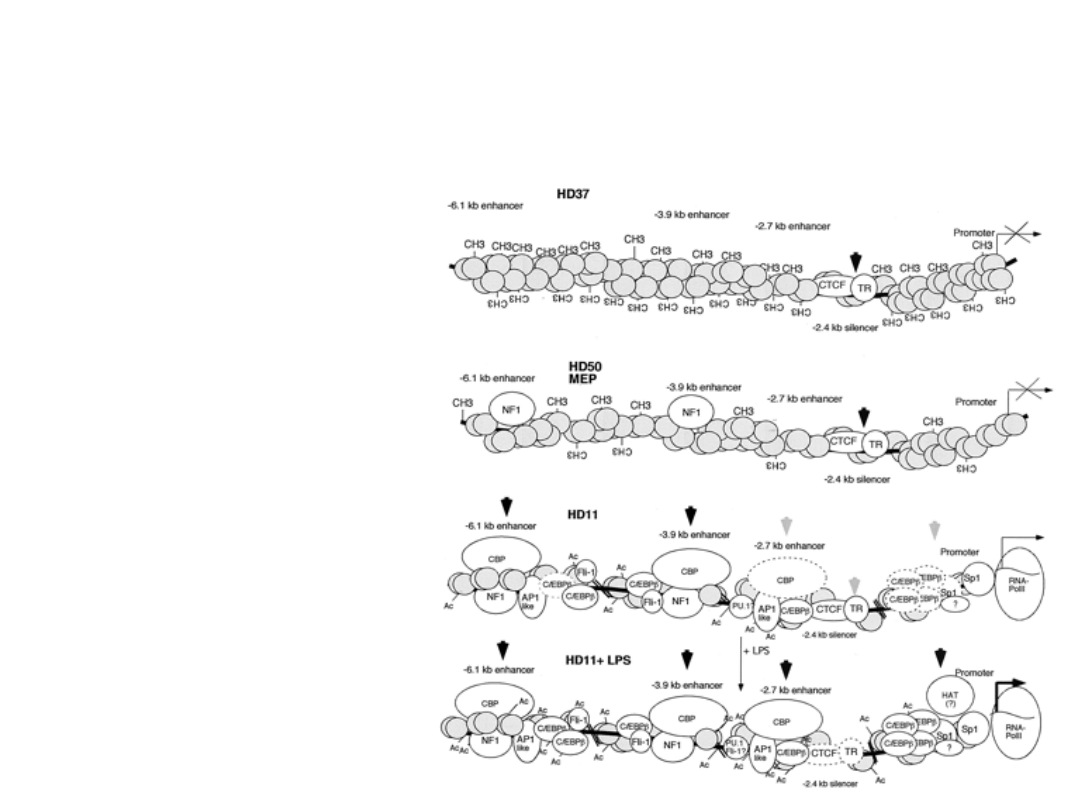
białe owale – czynniki
transkrypcyjne związane z
rdzeniami enhancerów,
promotorem i -2.4-kb
silencerem;
TR- receptor hormonu tarczycy,
związany do -2.4-kb silencera
obok m-sca CTCF
CH
3
i Ac - m-sca metylacji DNA
i acetylacji histonów
szare kółka – nukleosomy
owale z linia kreskowaną –
kompleksy czynników
transkrypcyjnych obecne w
niektórych komórkach lub
stabilizowane po stymulacji
LPS
strzałki czarne i szare –
odpowiednio silne i słabe m-
sca nadwrażliwe na DNazę I
Lefevre i in. 2003
Mol. Cell. Biol. 23: 4386-4400
zmiany struktury chromatyny, rekrutacji czynników
transkrypcyjnych i kofaktorów w rejonie regulatorowym
locus
lizozymu
w linii erytroblastów HD37 (Lys
¯ ¯
), komórkach
prekursorowych HD50 MEP (
Lys
¯
), niestymulowanych (
Lys
+
) i
stymulowanych LPS komórkach HD11 (
Lys
++
)
Lys
¯
¯
Lys
¯
Lys
+
Lys
++
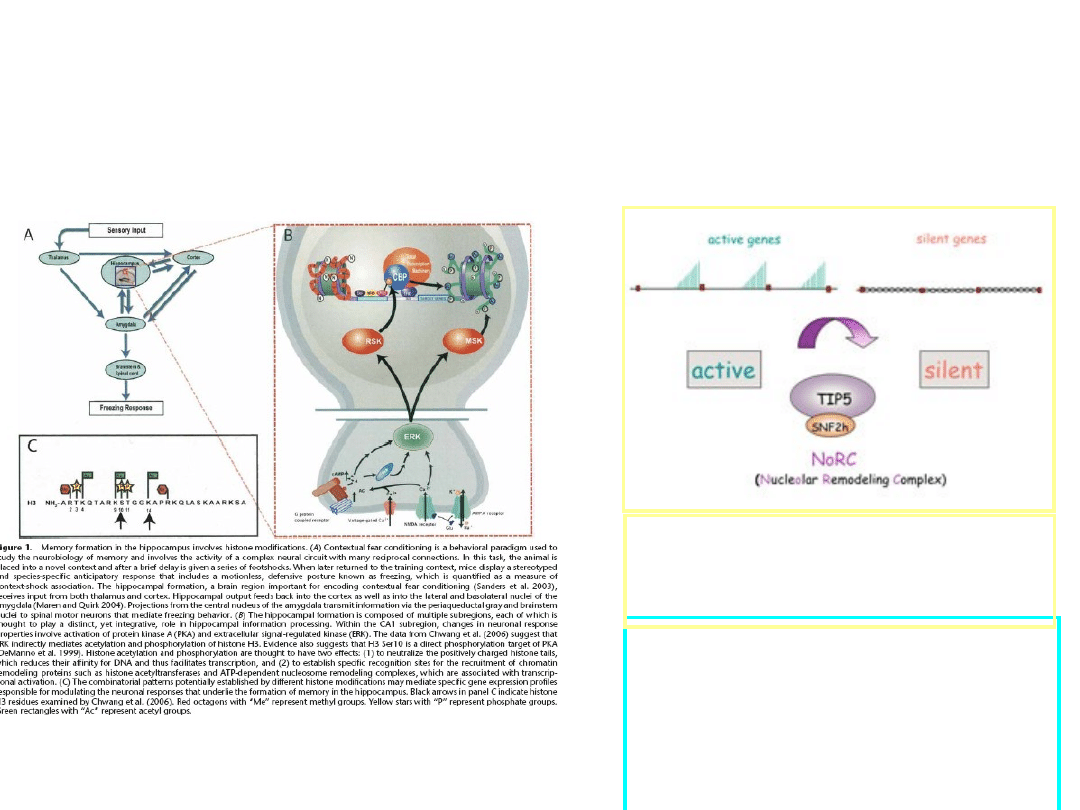
Wood i in. 2006 Combinatorial chromatin
modifications and memory storage: A code for
memory? Learn Mem 13: 241-244
naprawa DNA – NER, NHEJ,
rekombinacja homologiczna, naprawa
genów transkrypcyjnie aktywnych
Shim i in. 2005 Mol Cell Biol 25: 3934-
44
Kruhlak i in. 2006 Cell Biol 172: 823-
834
Chai i in. 2005 Genes Dev 19: 1656-
1661
inne procesy zachodzące przy udziale enzymów
modyfikacji histonów i/lub remodelujących chromatynę
Mayer i in. 2006 Intergenic
transcripts regulate the
epigenetic state of rRNA genes
Mol Cell 22: 351-361
kompleks
remodelujący
NoRC wycisza
geny rRNA
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
Wyszukiwarka
Podobne podstrony:
Struktura chromatyny
04 Struktury spoleczne. Dynamika struktur, studia, wprowadzenie do socjologii
APP 11 Dynamiczne Struktury Danych
Dynamiczne struktury danych
07 STRUKTURA CHROMATYNY A
Dynamika struktura+v+2007bs
dynamiczna struktura danych
dynamika i struktura samobojstw
Podstawy programowania II 5 Dynamiczne struktury danych(1)
Dynamika i struktura przychodów (3)
Dynamika i struktura kosztów (1)
DYNAMICZNE STRUKTURY DANYCH
Dynamika i struktura przeobrazen zatrudnienia
Barczyński Dynamika struktura2006
KWASY NUKLEINOWE I STRUKTURA CHROMATYNY
41 Jakie białka dominują w strukturze chromatyny i jaka jest ich funkcja
19 Odprzęganie we wy, struktura układu, dynamika zerowa
Analiza struktury i dynamiki
więcej podobnych podstron