
Mechanizmy rekombinacji
DNA
Rearanżacje genów
Rekombinacja homologiczna (ogólna) polega na
wymianie fragmentów nici pomiędzy dwoma cząsteczkami
DNA w dowolnym miejscu genomu, w miejscach największej
homologii sekwencji.
Rekombinacja zlokalizowana polega na wymianie
fragmentów DNA (informacji genetycznej) z udziałem
specyficznych białek rozpoznających określone sekwencje
w miejscu rekombinacji, np. rearanżacje genów u bakterii z
udziałem plazmidów lub bakteriofagów.
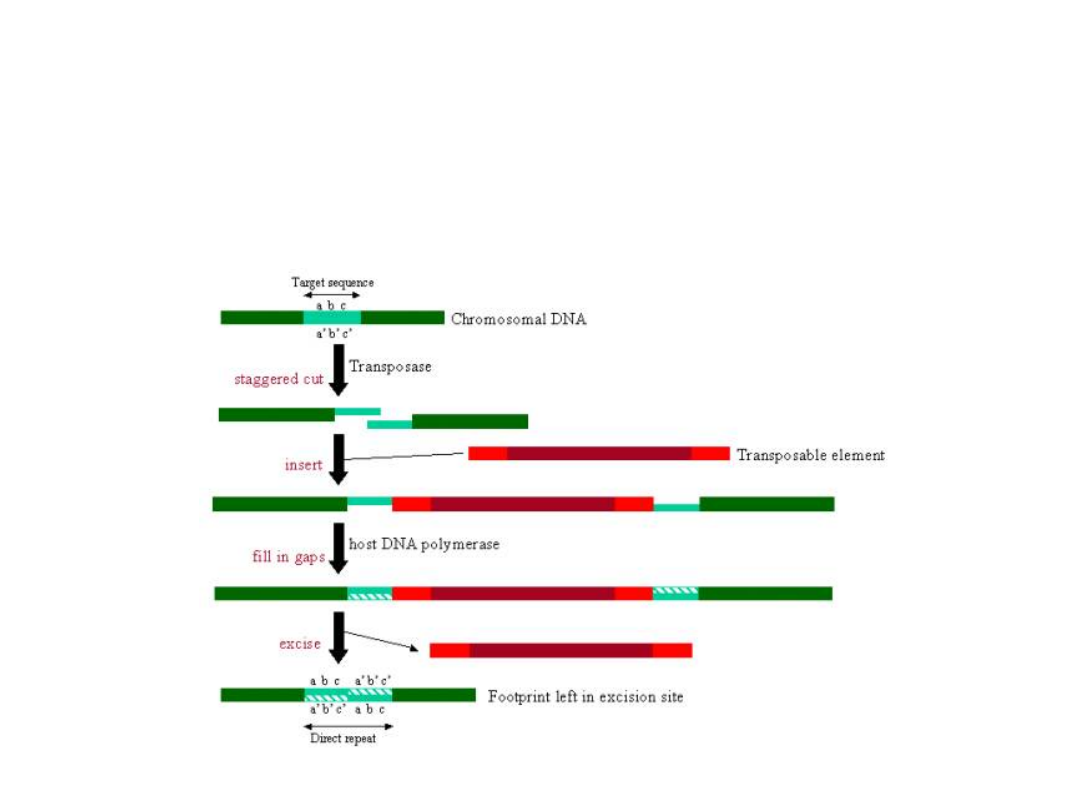
Transpozycja to przeniesienie specyficznych fragmentów
DNA z jednego chromosomu na drugi lub z jednego miejsca w
inne w obrębie tego samego chromosomu.
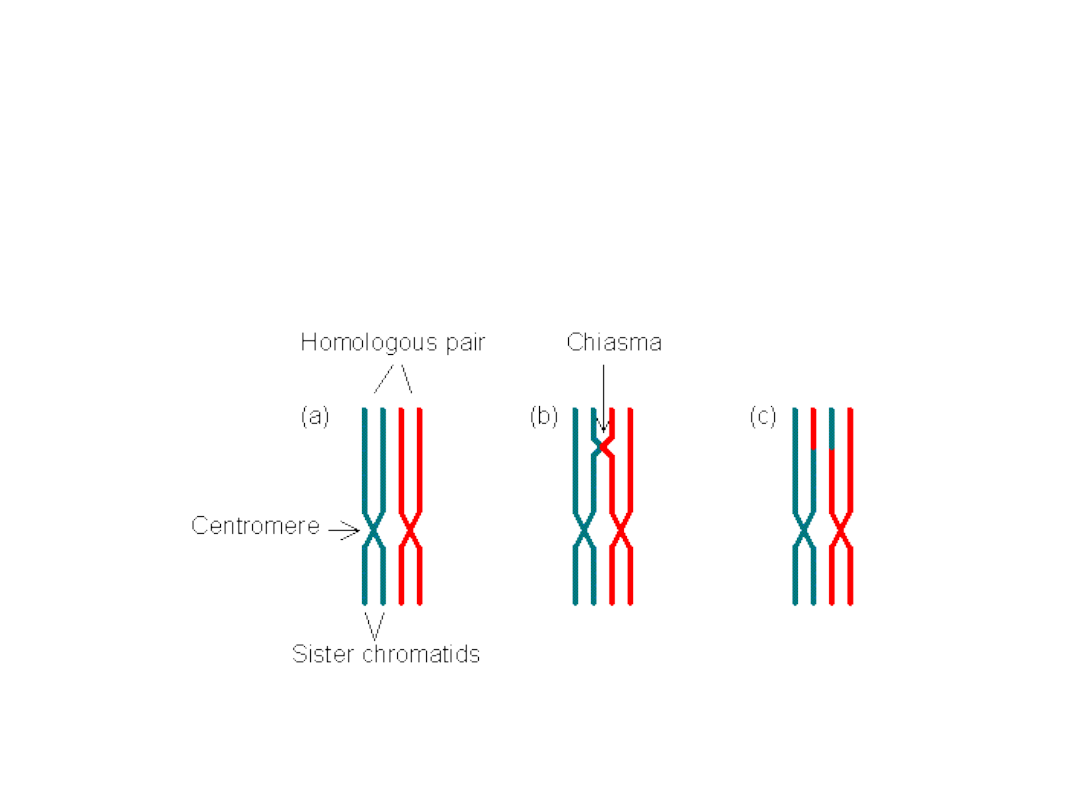
Homologiczna
rekombinacja
zachodzi
pomiędzy
dwuniciowymi cząsteczkami DNA posiadającymi analogiczne
lub bardzo podobne sekwencje nukleotydów.
W komórkach eukariotycznych do rekombinacji homologicznej
dochodzi głównie podczas mejozy w profazie pierwszego podziału
redukcyjnego. W wyniku tzw. crossing over dochodzi do wymiany
fragmentów nici DNA pomiędzy
niesiostarzanymi
chromatydami.
Taka rekombinacja homologiczna prowadzi do powstania nowych układów alleli
(nowych kombinacji genów).
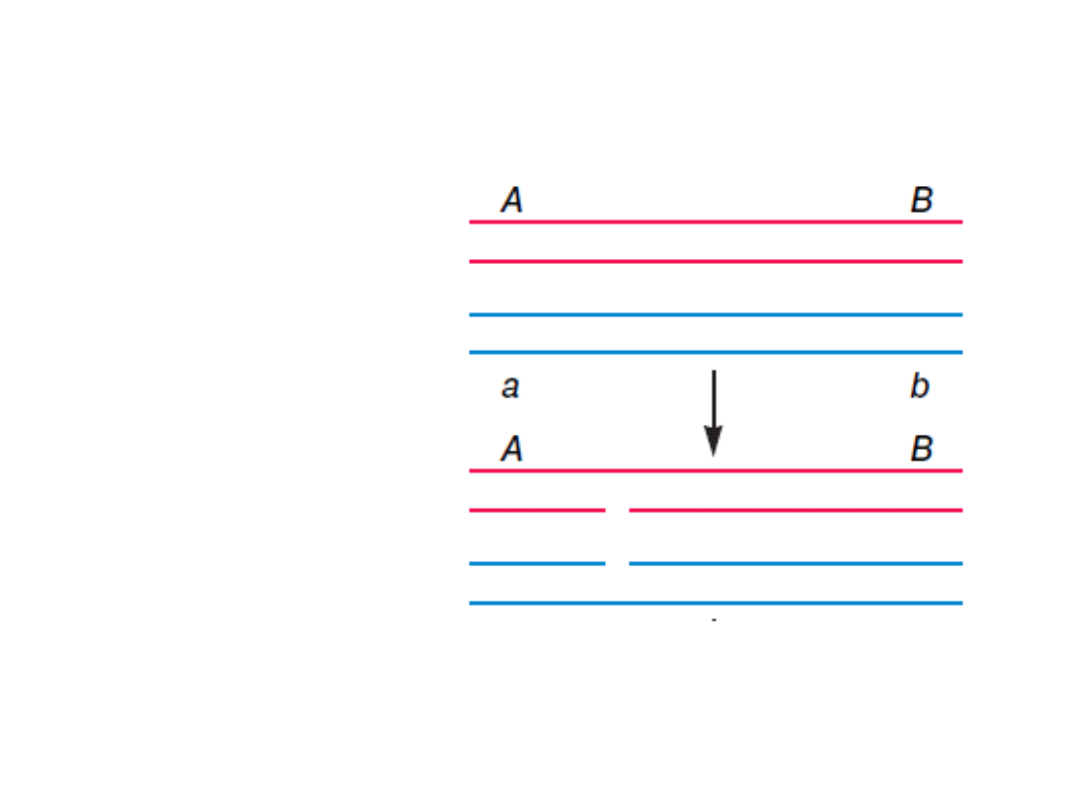
Model homologicznej rekombinacji
(Robin Holliday, 1964)
Dwie cząsteczki DNA
układają się tak względem
siebie, że ich
homologiczne fragmenty
leżą na przeciw siebie.
Homologiczną
rekominację
zapoczątkowuje
rozcięcie
jednego
z
łańcuchów w każdej z
dwóch cząsteczek DNA.
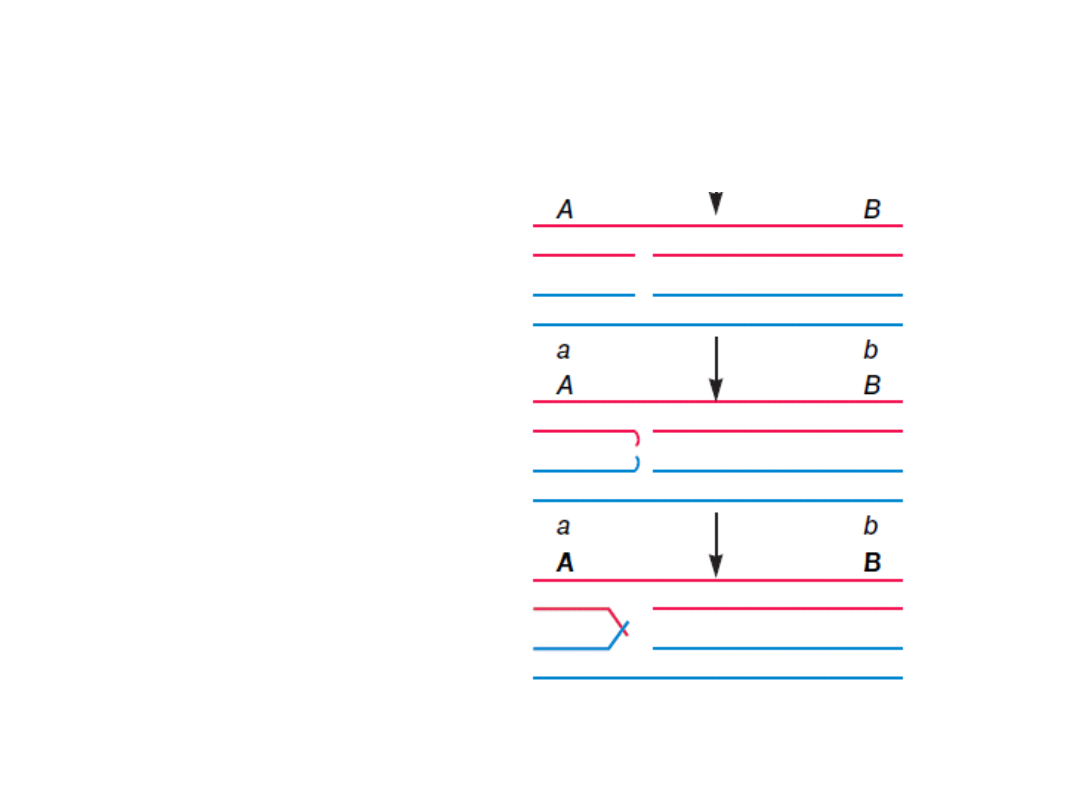
Model homologicznej rekombinacji
(Robin Holliday, 1964)
Wolne końce każdej z
przeciętych nici dokonują
inwazji
na
drugą
cząsteczkę
DNA,
odszukując
rejony
komplementarne.
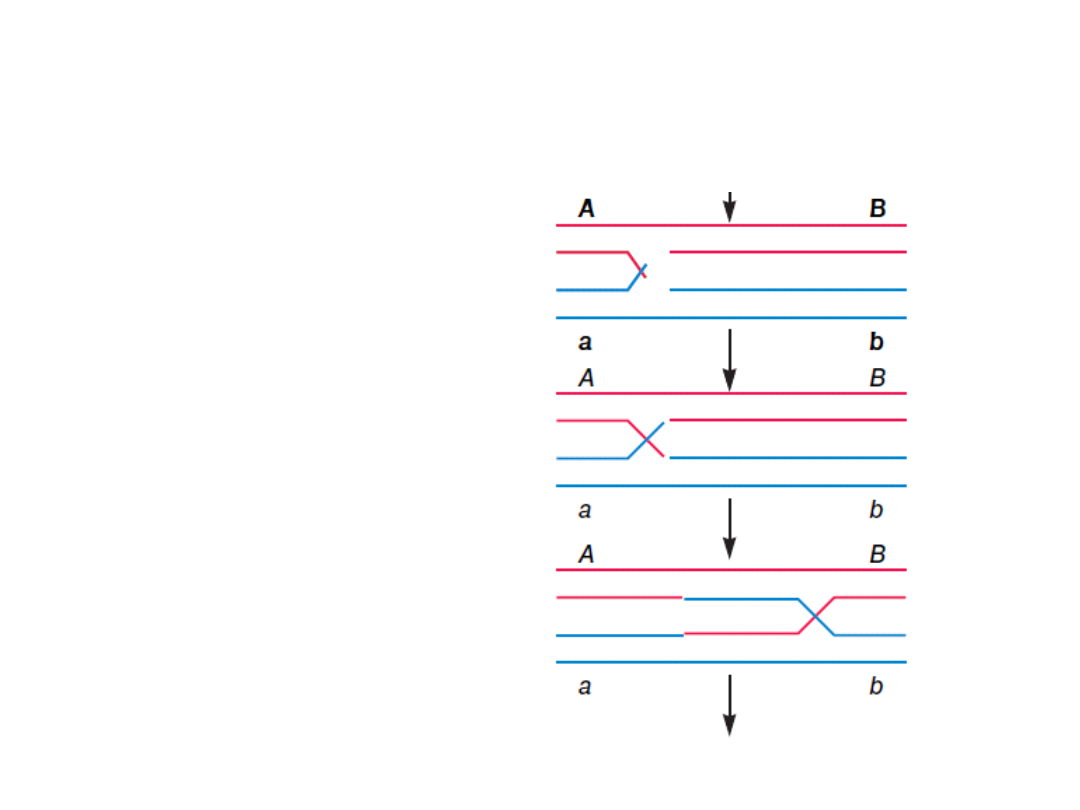
Model homologicznej rekombinacji
(Robin Holliday, 1964)
Rozcięte łańcuchy DNA po
do-konaniu inwazji sąsiedniej
nici,
podlegają
ligacji.
Struktura ta jest dynamiczna
i ulega przesunięciom na
znaczne
odległości,
co
określa
się
mianem
przemieszczania
ramion
lub
wędrówki
rozgałęzienia.
Taka struktura, na którą
składają
się
dwie
nici
krzyżujące się i dwie nici
nieskrzyżowane nosi nazwę
połączenia Hollidaya lub
krzyżowej
wymiany
łańcuchów.
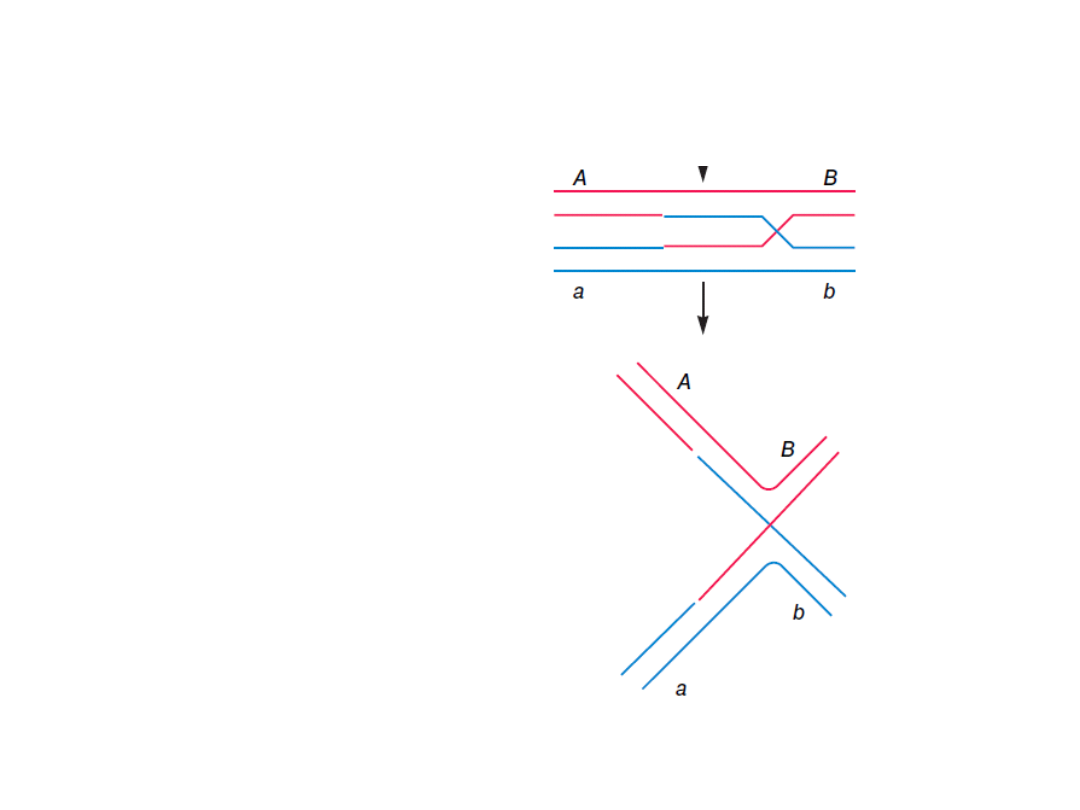
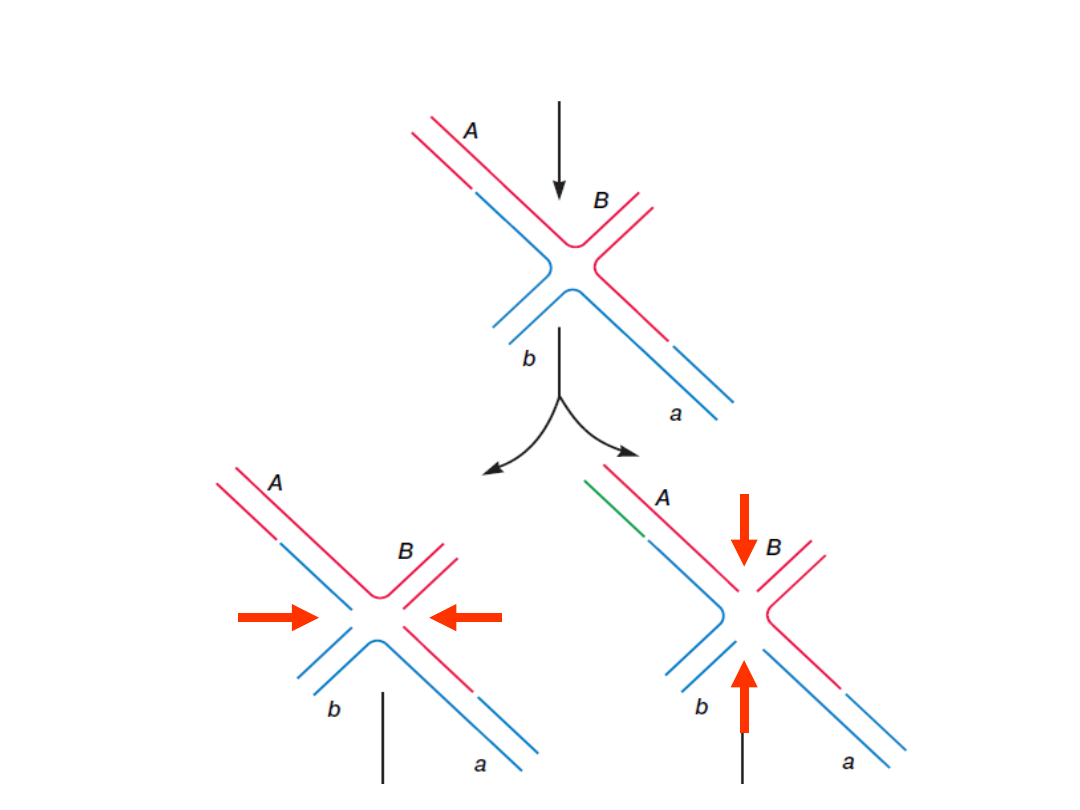
Model homologicznej rekombinacji
Dwie równoważne
struktury
Połączenie Hollidaya
Model homologicznej rekombinacji
Połączenie Hollidaya
może być przecinane i
łączone na dwa
sposoby.
Przecięcie nici inwazyjnych
Przecięcie nici
nieinwazyjnych
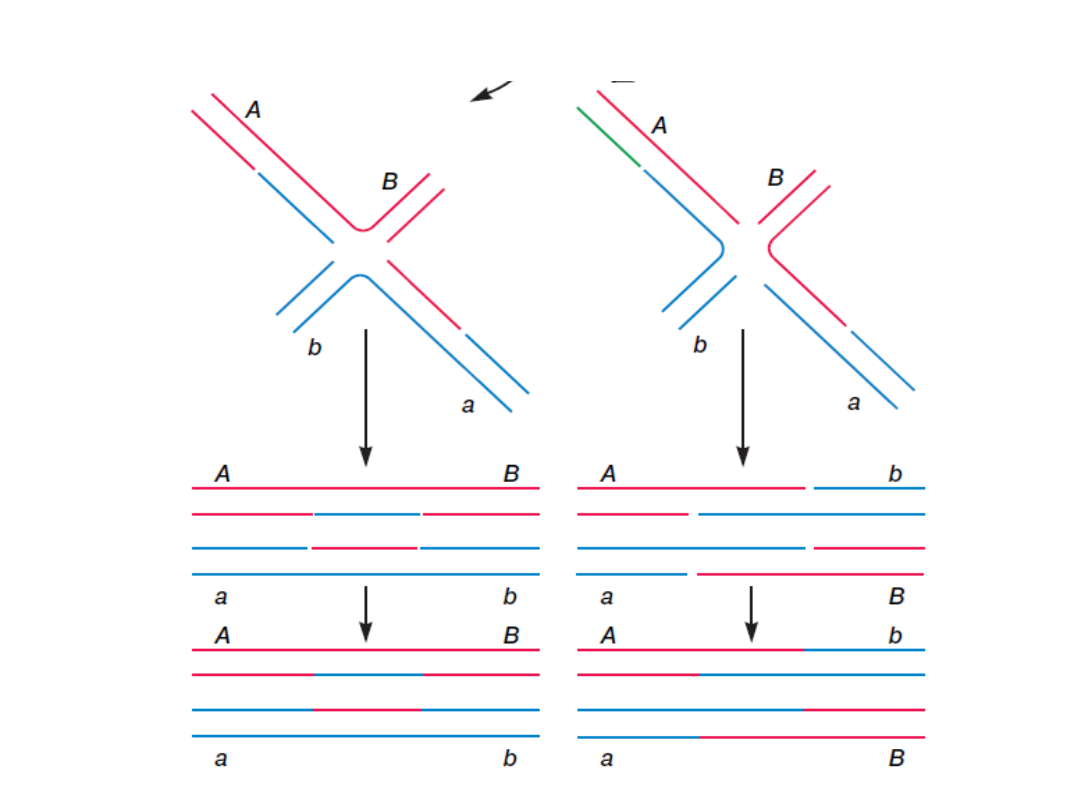
Model homologicznej rekombinacji
Sliced
Patched
Powstają takie sa-
me cząsteczki jak
rodzicielskie
z
wyjątkiem wymie-
nionych rejonów.
Powstaje
zrekom-
binowana
cząsteczka
DNA.
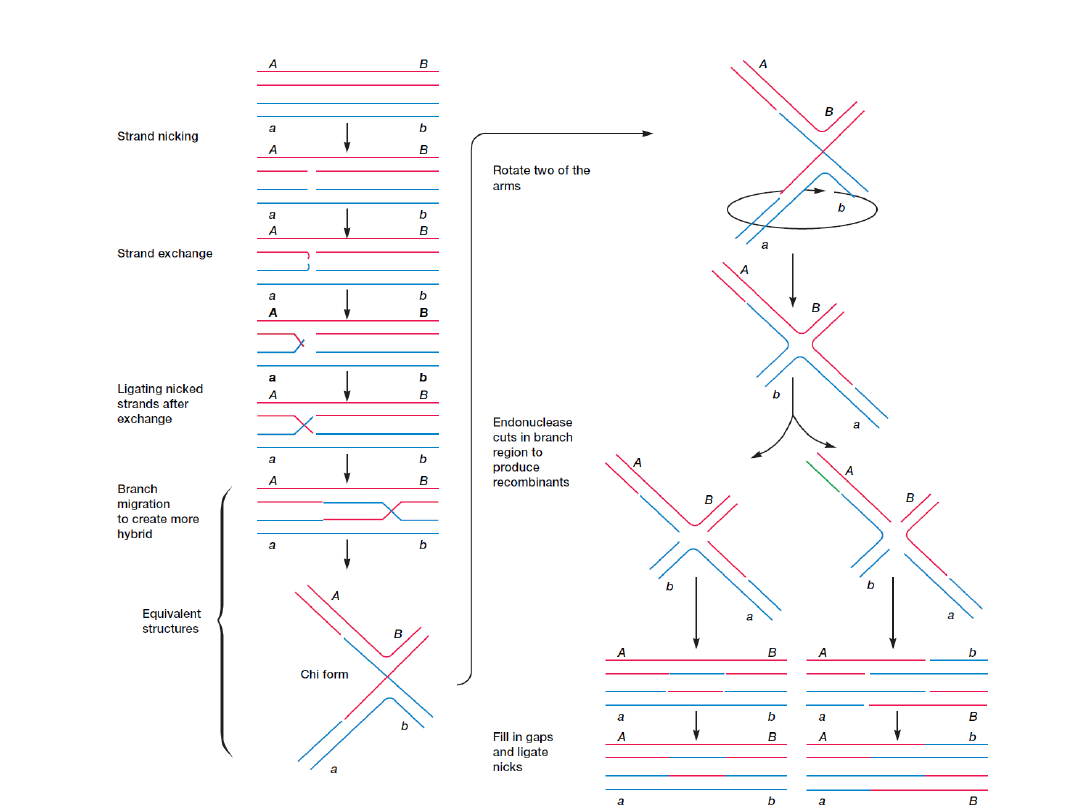
Model homologicznej rekombinacji wg
Hollidaya
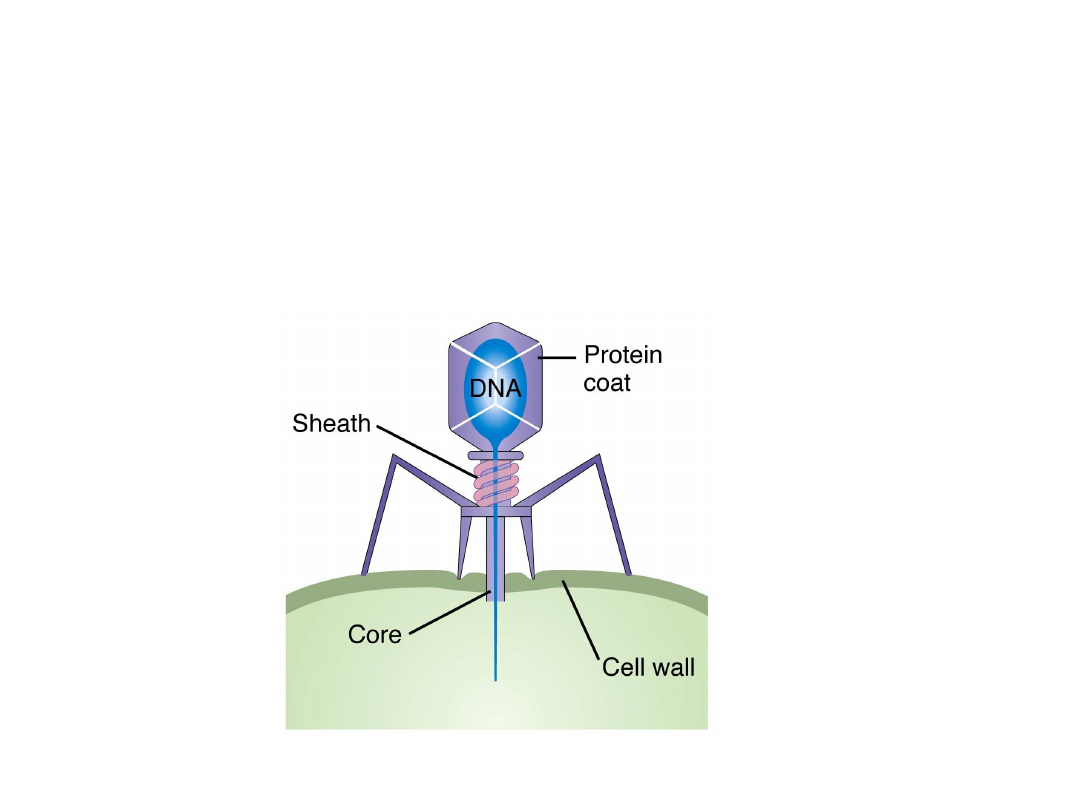
Przykładem rekombinacji zlokalizowanej jest przenoszenie
genów między komórkami bakteryjnymi przy udziale
bakteriofagów.
Rekombinacja zlokalizowana
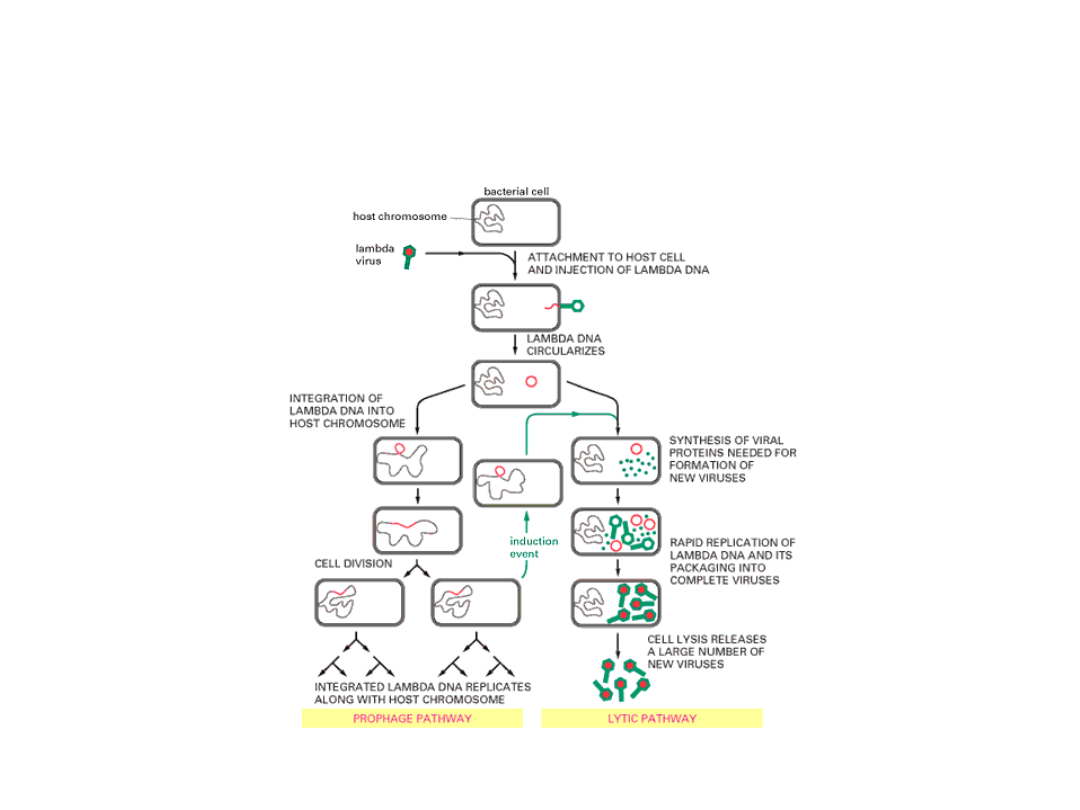
Cykle życiowe bakteriofaga
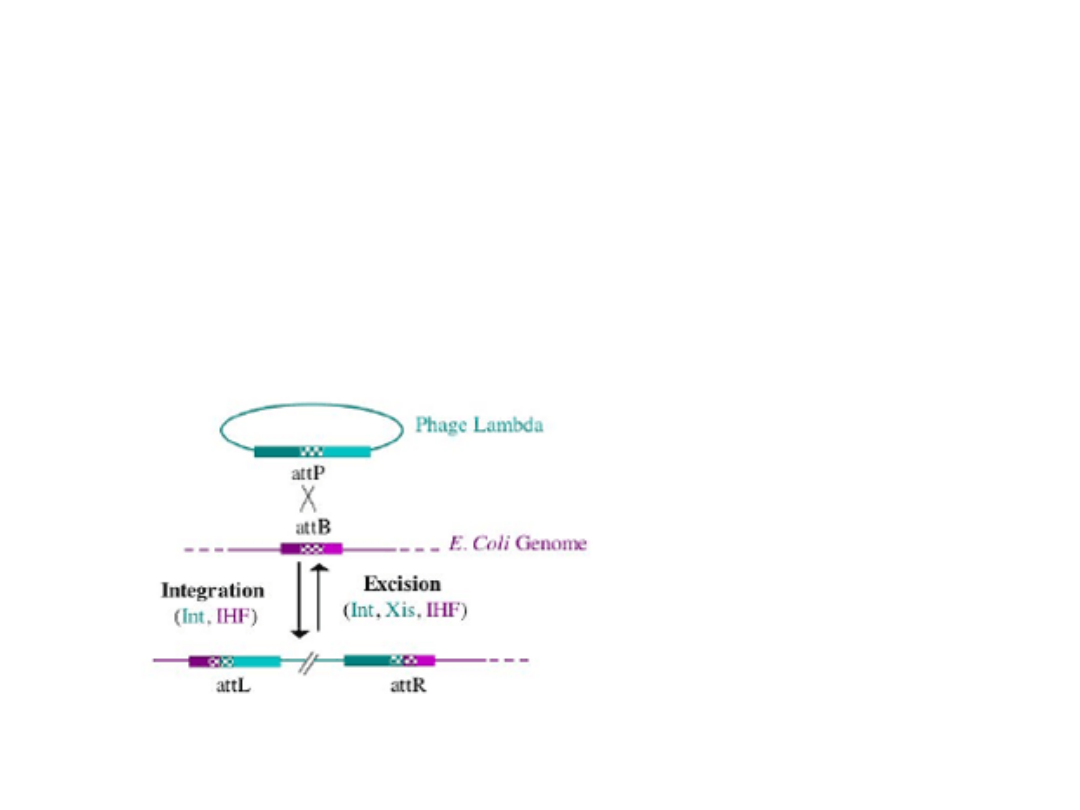
Genom faga integruje z chromosomem bakteryjnym w
wyniku rekombinacji zlokalizowanej. Za ten proces
odpowiedzialny
jest
specjalny
enzym
–
integraza,
wytwarzana przez faga. Integraza rozpoznaje i rozcina
specyficzne sekwencje DNA zarówno w genomie bakteryjnym
(attB) jak i fagowym (attB), a następnie łączy ze sobą te dwie
cząsteczki DNA.
Obok integrazy w tym
pro-cesie bierze udział
czynnik integracyjny
gospodarza IHF
(integration host factor).
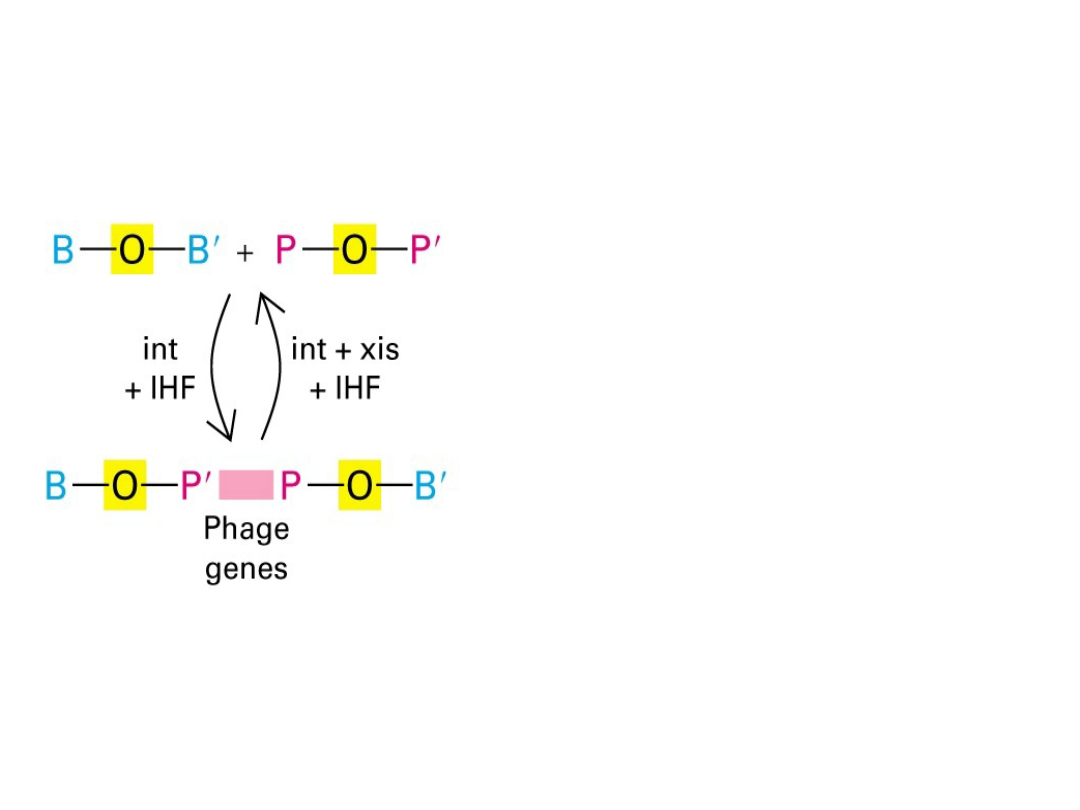
Integraza
rozpoznaje
bakteryjną
sekwencję attB (B-O-B’) oraz fagową
attP (P-O-P’). B i B’ to sekwencje
specyficzne dla E. coli, P i P’ - dla faga
lambda; O - to identyczne sekwencje
obecne zarówno w DNA bakterii jak i
faga.
W
wycinaniu
faga
z
genomu
bakteryjnego udział bierze, obok
integrazy
i
białka
IHF,
enzym
ekscyzynaza.
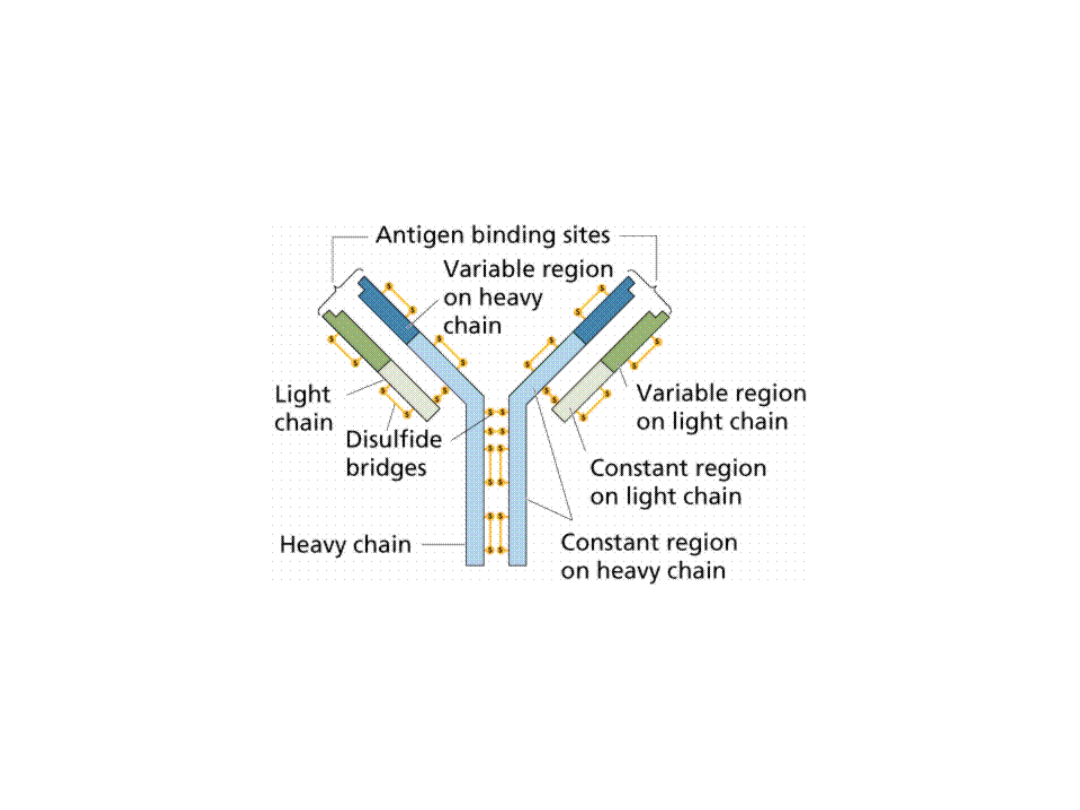
Rearanżacje genów dla
immunoglobulin/przeciwciał

Powstanie różnorodności przeciwciał wiąże się
z:
1. procesem somatycznej rekombinacji
obejmującym rearanżacje i delecje genów dla
immunoglobulin,
2. procesami cięcia i składania (splicing)
mRNA.
Budowa genów dla łańcuchów lekkich (L)
Trzy
geny
kodują
każdy
łańcuch
lekki
immunoglobulin.
Część zmienna (V) kodowana jest przez dwa
geny:
- gen V, który zawiera informację
genetyczną dotyczącą kolejności pierwszych 95
aa części zmiennej łańcucha L. Na ludzkim
chromosomie 2 obecnych jest ponad 200 takich
genów dla łańcucha L
kappa.
- gen J (segment łączący) kodujący
kolejnych 13 końcowych aminokwasów (pozycje
96 – 108) części zmiennej łańcucha L. Istnieje 5
takich genów J
1
– J
5
.
Część stała (C) kodowana jest przez gen C
kodujący sekwencję aminokwasową części stałej
(pozycje aa 109 – 214). W przypadku genu C
lambda istnieje kilka jego kopii, odpowiadającym
podtypom tego łańcucha L.
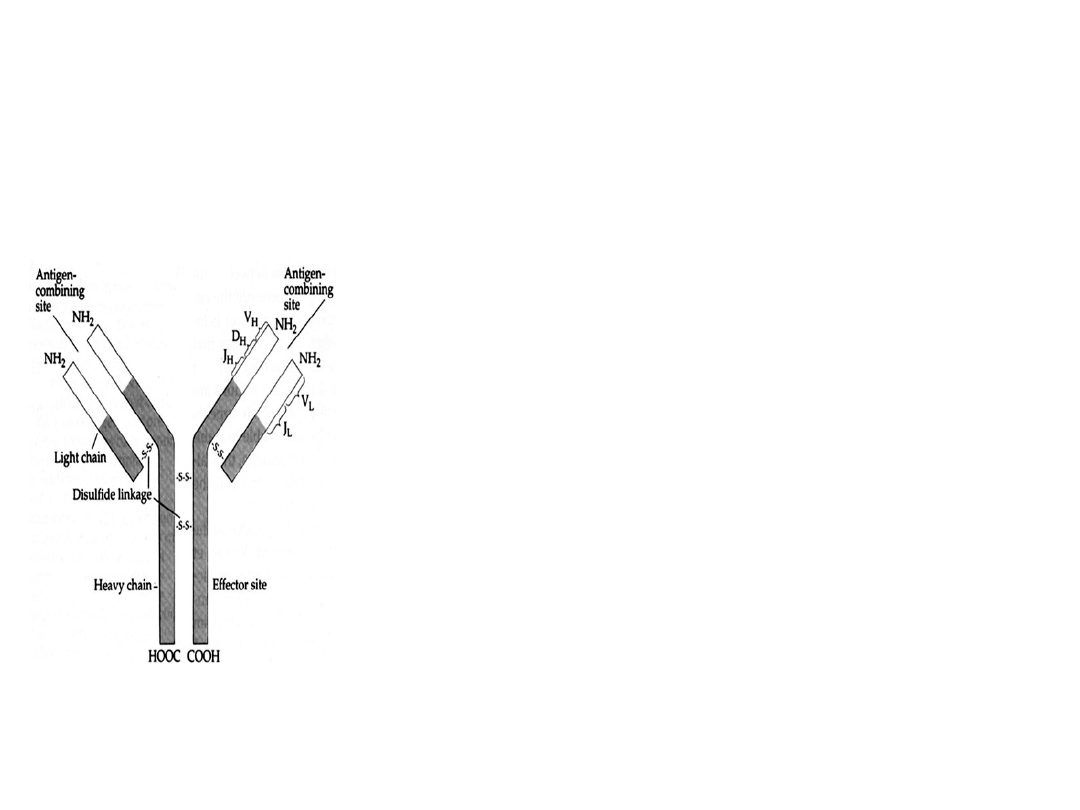
Budowa genów dla łańcuchów lekkich (L) (c.d.)
Każdy z genów V może łączyć się z dowolnym genem J, co
prowadzi do powstawania genu (egzonu) V/J. Ten nowo powstały
gen (egzon) reprezentuje jeden z możliwych genów (egzonów)
kodujących część zmienną łańcucha L.

Podczas procesu transkrypcji gen V/J, wspólnie z genem C, ulega
transkrypcji do pre-mRNA, który w wyniku procesów cięcia i
składania przekształca się w dojrzały mRNA gotowy do translacji.
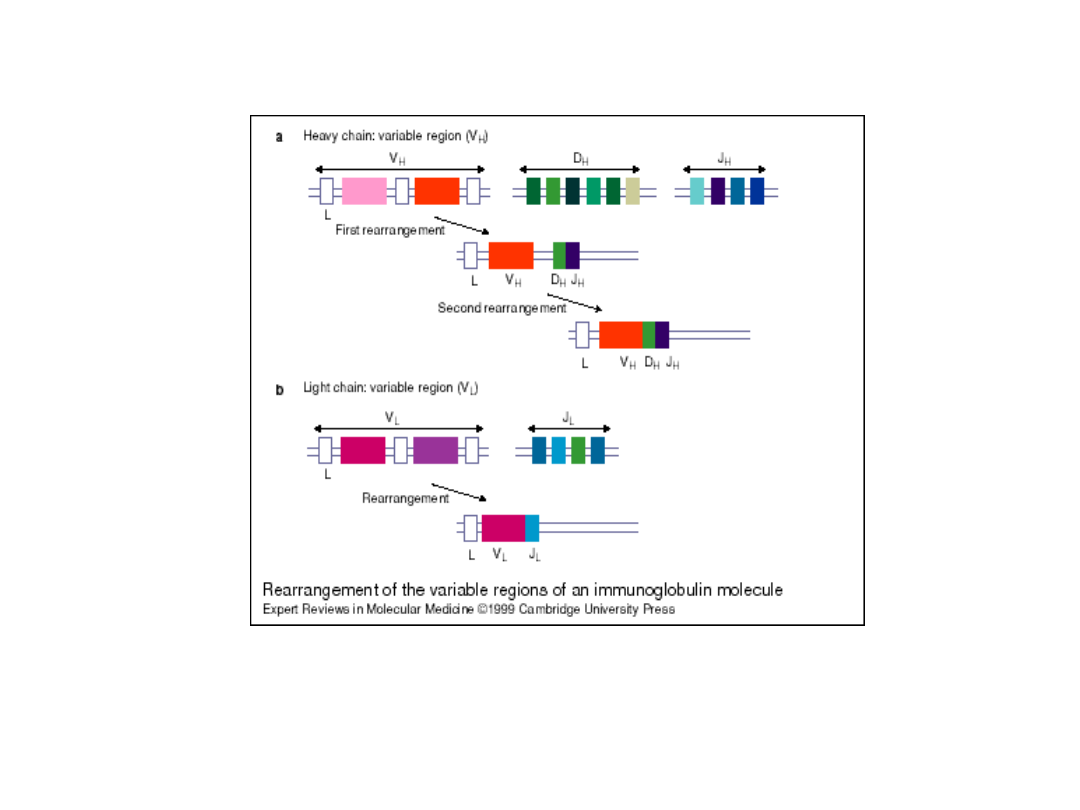
Budowa genów dla łańcuchów ciężkich (H)
Budowa genów dla łańuchów H
podobna jest do budowy genu dla
łańcucha L, ale bardziej złożona
ponieważ:
- gen (egzon) kodujący część
zmienną utworzony jest z trzech genów,
- gen (egzon) kodujący część
stałą koduje trzy/cztery domeny C,
- gen dla części stałej musi
kodować odrębne części stałe dla
każdej z 5 klas immunoglobulin.
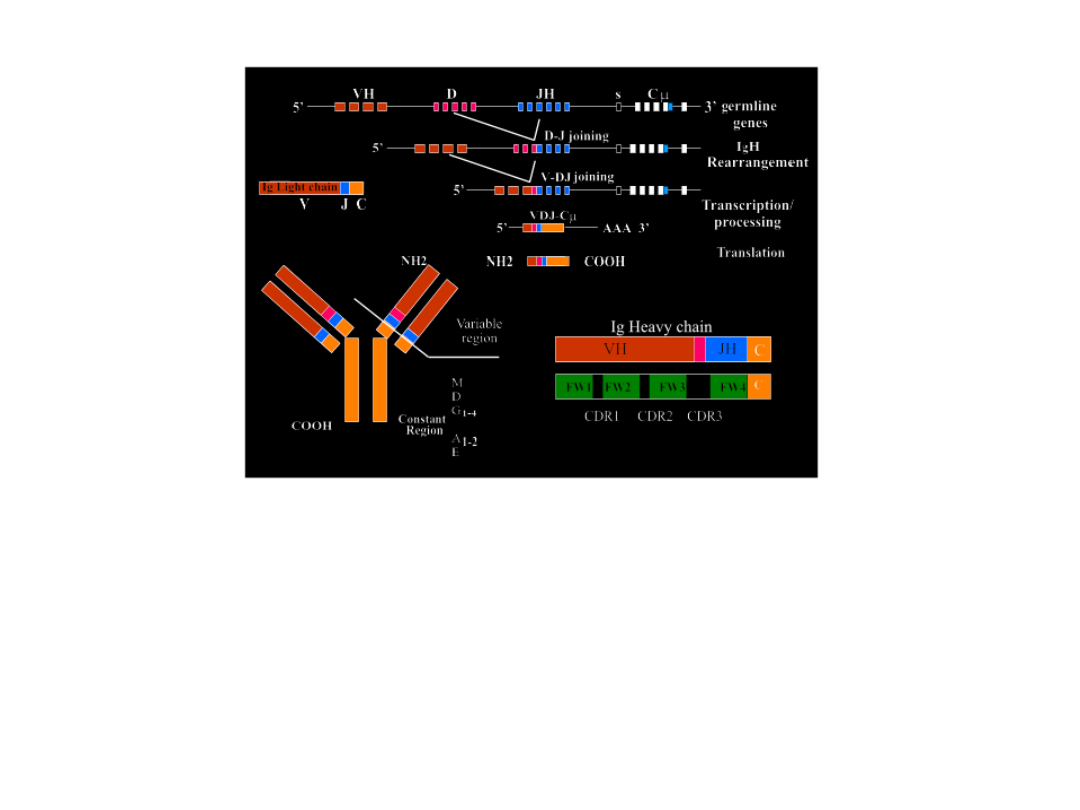
Część zmienną łańcucha H kodują:
gen V
H
kodujący
pierwszych 100 aminokwasów,
gen D
H
kodujący 2 – 13
kolejnych aa i
gen J
H
kodujący 4 – 6 ostatnich aa.
Istnienie ponad 200 genów V
H
, około 20 genów D
H
i 6 genów
J
H
, co warunkuje ogromną różnorodność regionów zmiennych
łańcuchów H.
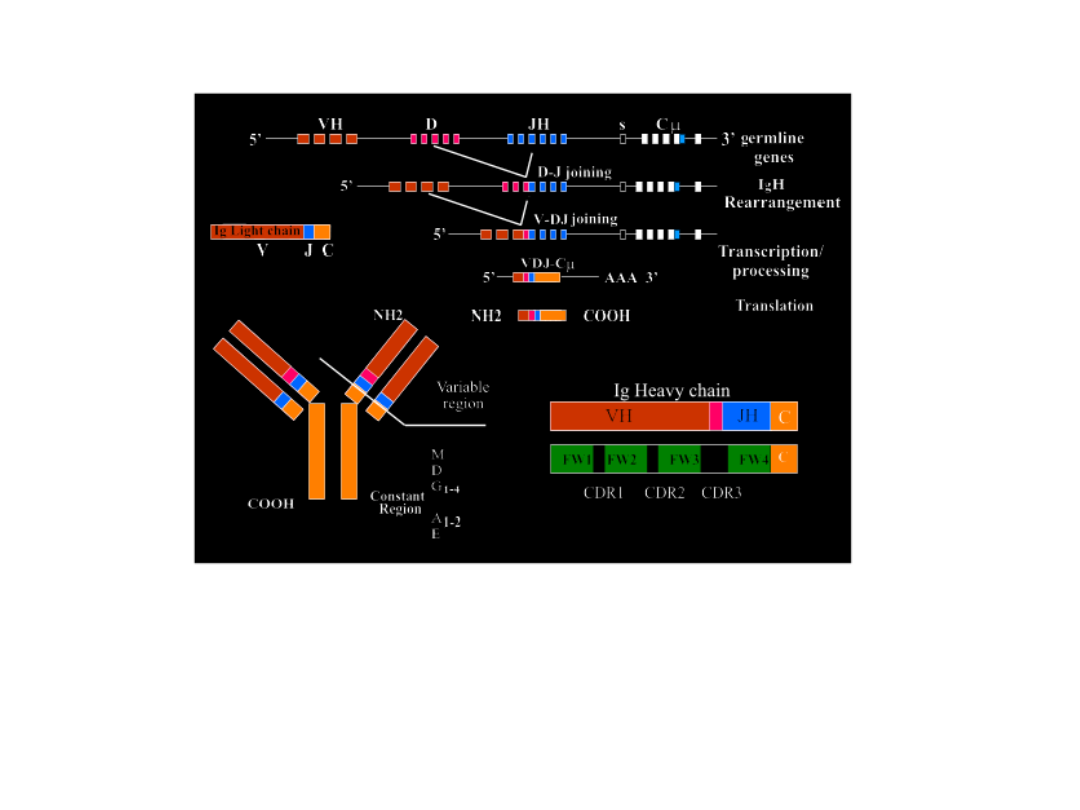
Budowa genów dla łańcuchów ciężkich (H) c.d.
Powstanie egzonu V/D/J:
- rearanżacje (rekombinacje) prowadzą najpierw do połączenia
genu D
H
z genem J
H
, a następnie powstały segment przyłączony zostaje
de genu V
H
.
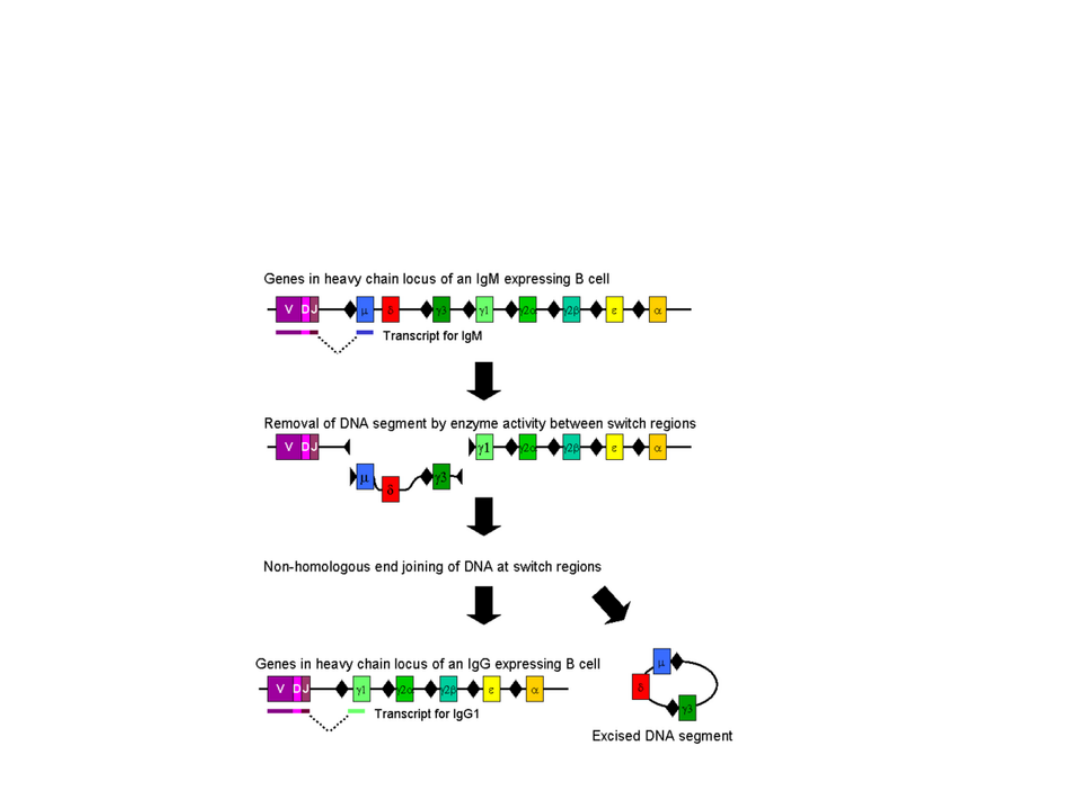
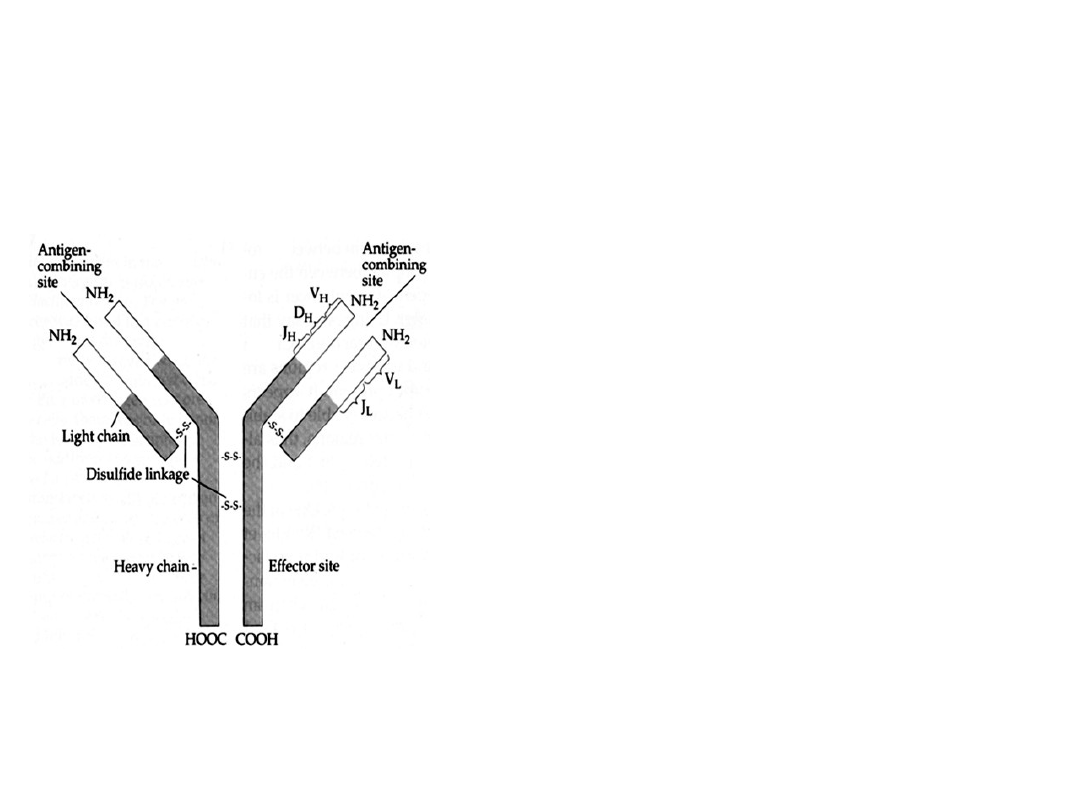
Przełączanie klas immunoglobulin
Za przełączanie klas immunoglobulin odpowiedzialny jest
proces rekombinacji, który prowadzi do przemieszczenia
genu VDJ w pobliże właściwego genu C.

Transpozycje
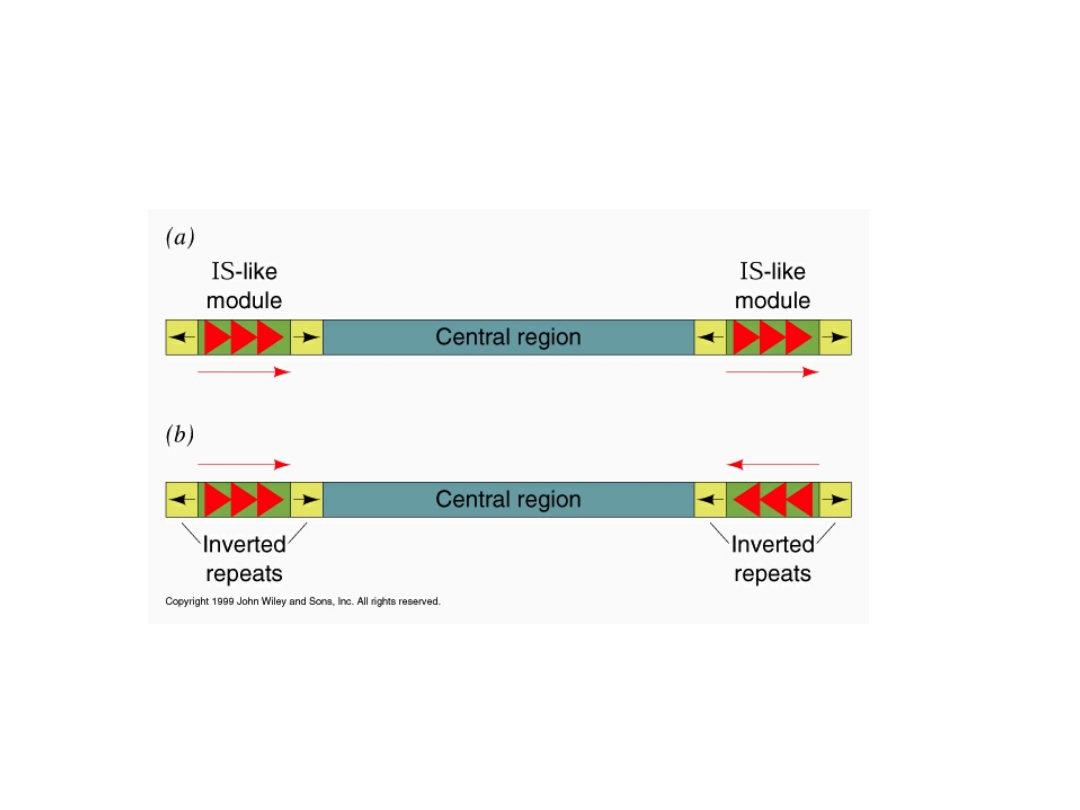
Transpozycje
Transpozomy
to
ruchome
elementy
genetyczne
przemieszczające się z jednego miejsca na drugie w
chromosomie. Występują one zarówno o prokariota jak i
eukariota.
W przemieszczaniu transpozomów biorą udział kodowane przez
nie enzymy – transpozazy. Transpozycje nie wymagają
homologii sekwencyjnych pomiędzy cząsteczkami DNA.
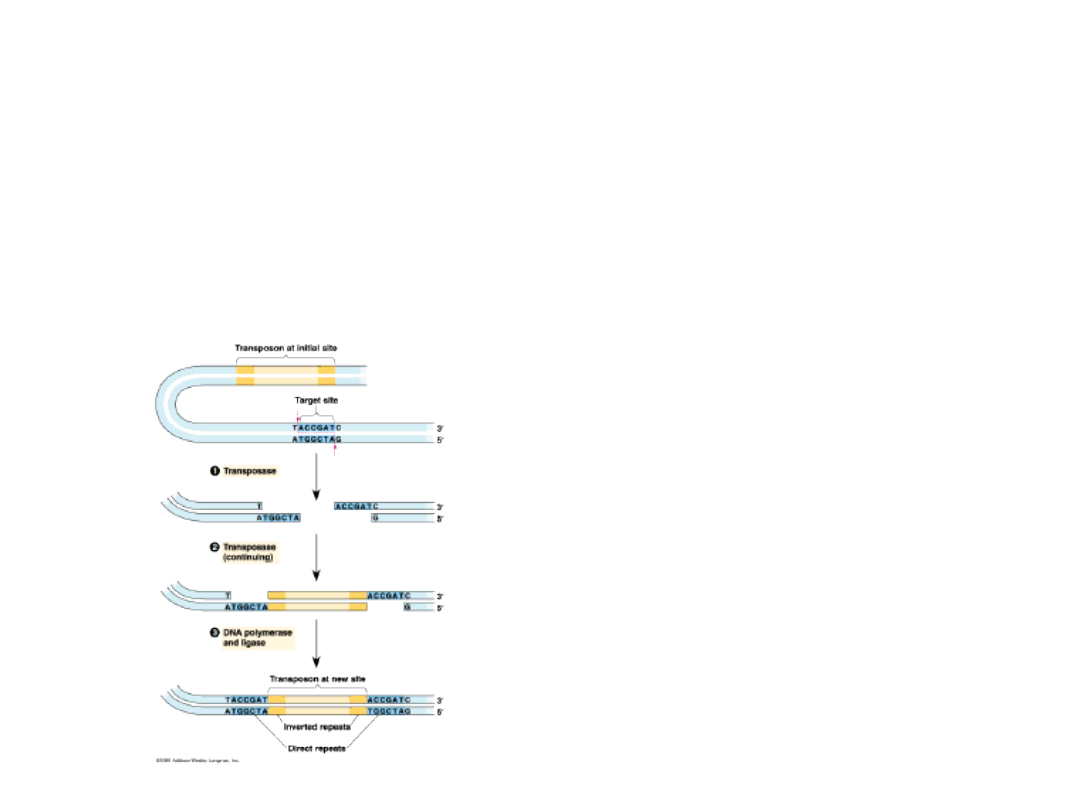
Transpozomy prokariotyczne
Sekwencje insercyjne (IS), należące do najprostszych
transpozomów, występują zarówno w chromosomach
bakteryjnych jak i plazmidach. Zwykle mają one wielkość
poniżej 2 kpz i obok krótkich odwróconych powtórzeń
końcowych, zawierają gen dla transpozazy i gen regulatorowy.
Sekwencje docelowe w DNA
gospodarza, rozpoznawane przez
transpozazy, charakteryzuje je nie
kolejność nukleotydów, ale ich
długość.
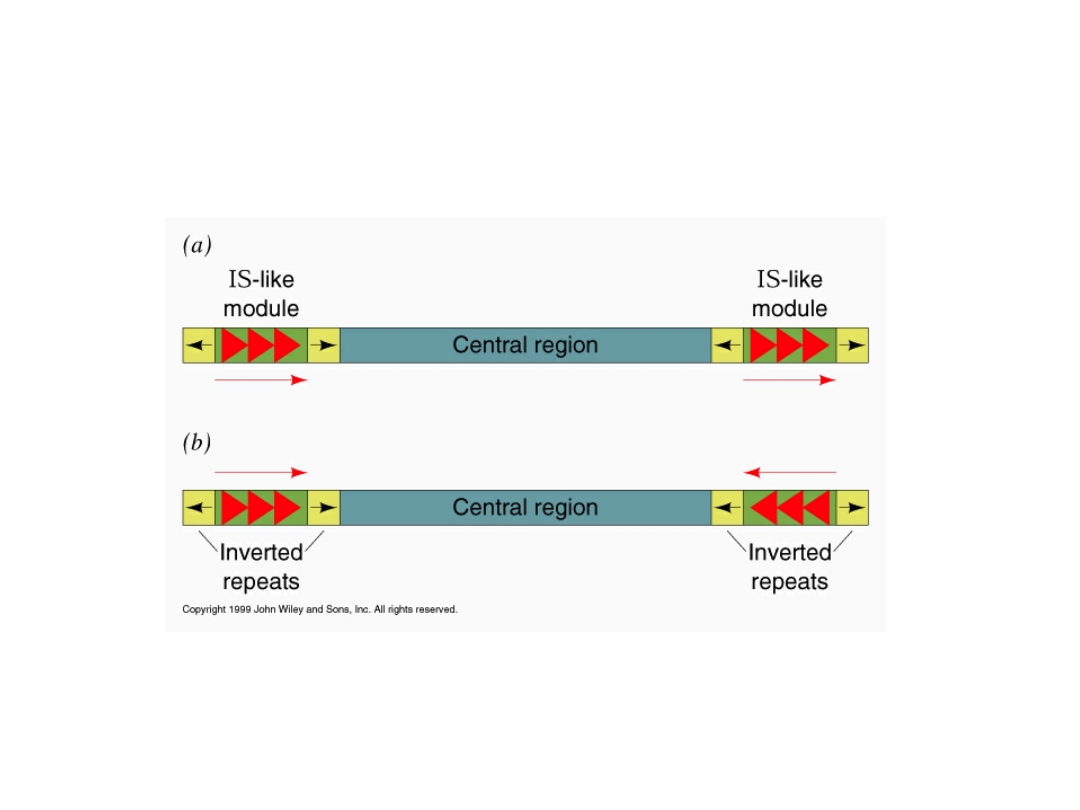
Transpozomy u prokariota
Transpozomy złożone zawierają inne geny, nie biorące
bezpośrednio udziału w procesie transpozycji, np. geny
oporności na antybiotyki.
Transpozon Tn3 – część centralną stanowią 3 geny (tnpA kodujący
transpozazę, tnpR kodujący białko represorowe i resolwazę, amp
kodujący -laktamazę).
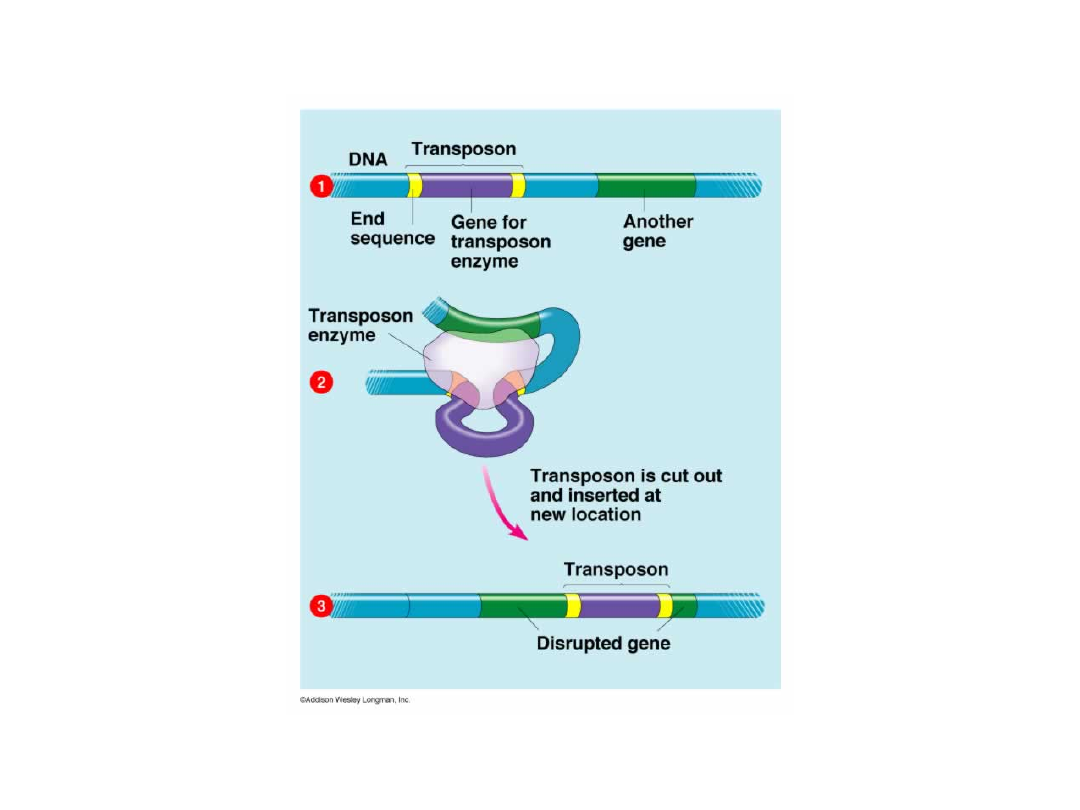
Mechanizmy przemieszczania się transpozomów
Transpozycja niereplikacyjna typu „wytnij i wklej”.

Transpozomy, w przypadku bakterii, odgrywają następującą
rolę:
- mogą brać udział w regulacji ekspresji genów ze
względu na obecność sekwencji promotorowych;
- umożliwiając homologiczną rekombinację są
zaangażowane w rearanżacje genomów bakteryjnych;
- ze względu na obecność genów oporności na
antybiotyki odpowiedzialne są za szerzenie się oporności
antybiotykowych;
- mogą uczestniczyć w tworzeniu nowych białek.
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
Wyszukiwarka
Podobne podstrony:
W10 2009 Rearanżacja genów immunoglobulin
11 BIOCHEMIA horyzontalny transfer genów
Genetyka regulacja funkcji genow
Ekspresja genów
Gradient ekspresji genów w regulacji morfogenezy u ssaków, Medycyna ŚUM, Rok 1, Biologia medyczna, T
wprowadzenie genów
Mapowanie genów na przykładzie Drosophila melanogaster(1)
3M3 Whiteris G składnik aktywny stymulujący ekspresję trzech kluczowych genów
EKSPRESJA GENÓW KLONOWANYCH W WEKTORACH PLAZMIDOWYCH W ZREKOMBINOWANYCH SZCZEPACH E COLI(1)
W1 Współdziałanie genów
19 Podstawy genetycznej?terminacji płci oraz dziedziczenia genów sprzężonych z płci u ssaków
Lekcja 8 Podstawy mapowania genów
Ekspresja genów część I Rodzaje kwasów rybonukleinowych i kod genetyczny1
18 Babik, Ewolucja genomow i powstawanie nowych genow (2009)
2 Regulacja funkcji genów Zmienność i mutacje
10 WYK X Regulacja funkcji genów u bakterii1id 10654 ppt
Nowa mapa naszych genów ujawnia ogromne różnice między1
więcej podobnych podstron