
REGULACJA EKSPRESJI
GENÓW

Regulacja ekspresji
genów
• Istnieje ścisłe powiązanie między aparatem
genetycznym komórki a jej stanem
czynnościowym
• Produkty kodowane przez różne geny są
wytwarzane w ilościach określonych
właściwościami strukturalnymi i
funkcjonalnymi danej komórki
• Regulacja tego systemu odbywa się przez
włączanie i wyłączanie odpowiednich genów

Regulacja ekspresji
genów
• Regulacja ekspresji genów dotyczy
zarówno organizmów
prokariotycznych jak i
eukariotycznych
• W pojedynczej komórce eukariotycznej
tylko około 15% genów ulega
ekspresji; przy czym w różnych typach
komórek aktywowane są różne geny

Regulacja ekspresji genów
u Prokaryota
U Prokaryota ekspresja genów
regulowana jest na dwóch poziomach:
• TRANSKRYPCJI – przez regulację liczby
tworzonych cząsteczek mRNA
• TRANSLACJI – przez regulację liczby
kopii polipeptydów powstałych na
matrycy konkretnej cząsteczki mRNA

Regulacja przez kontrolę
szybkości inicjacji
transkrypcji
• REGULACJA WEWNĘTRZNA – dotyczy
poziomu transkrypcji jednego genu
względem innego w zależności od
promotora; jest to inaczej stopień
oddziaływania między polimerazą RNA a
promotorem
• REGULACJA ZEWNĘTRZNA – poziom
transkrypcji regulowany jest przez czynniki
zewnętrzne, niezależne od struktury genu

Promotory u Prokaryota
• Geny zawierające promotory
słabe, wykazują niski poziom
ekspresji – są to głównie geny
kodujące produkty potrzebne w
niedużych ilościach
• Promotory silne indukują z
większą częstością procesy inicjacji
trankrypcji – nawet co 2 sekundy
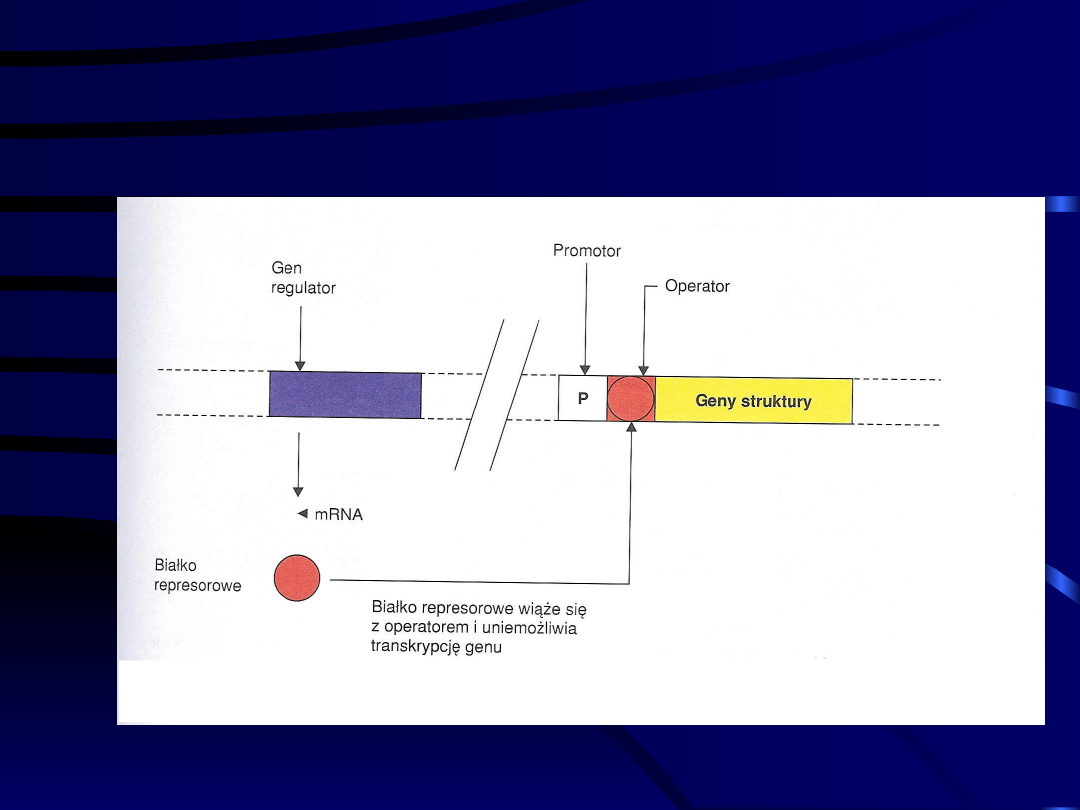
Struktura operonu
bakteryjnego

Efektory allosteryczne
• Cząsteczki represorów są białkami
allosteryczymi
• Efektory allosteryczne przyłączają się do
centrum allosterycznego (inne niż centrum
aktywne)
• Efektorami allosterycznymi mogą być
aminokwasy, czy też hormony steroidowe
• Wiązania między białkami allosterycznymi i
efektorami są słabe, a oddysocjowanie
efektora zależy od jego stężenia
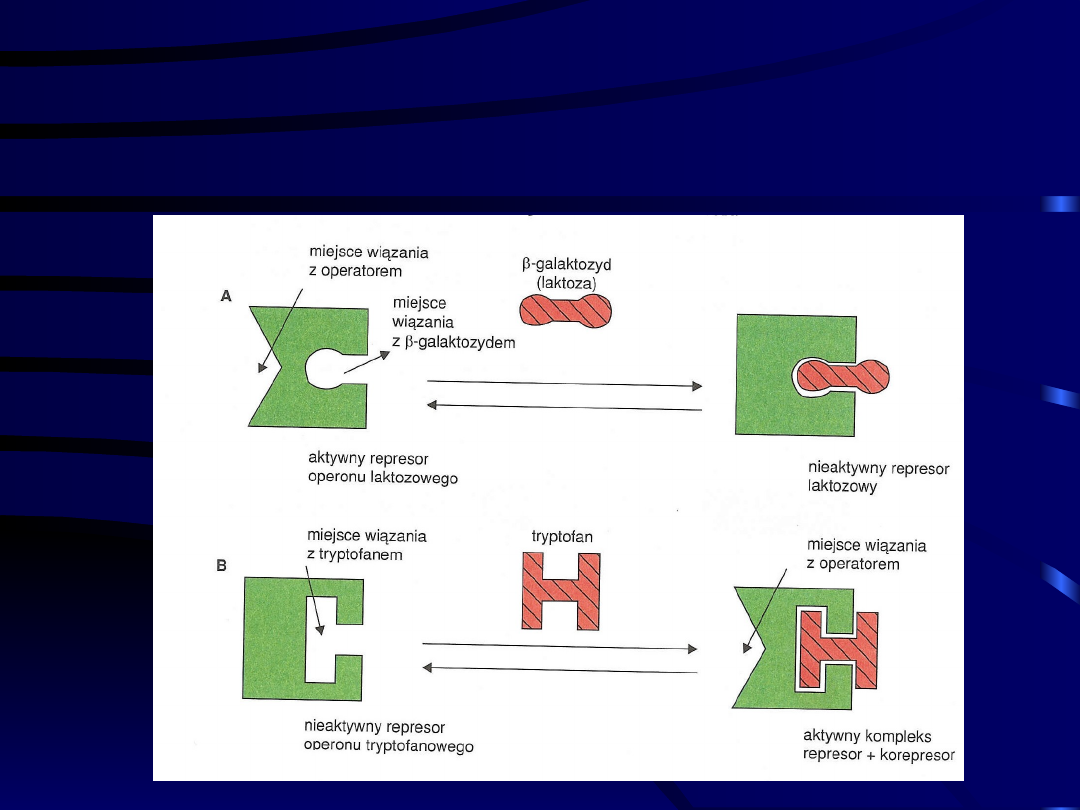
Aktywność allosterycznych represorów
pod wpływem: A - induktora i B -
korepresora

Rodzaje operonów
bakteryjnych
• INDUKOWANE (KATABOLICZNE)- produkcja
enzymów jeśli substrat obecny w środowisku
• ULEGAJĄCE REPRESJI (ANABOLICZNE)-
produkcja enzymów jeśli substancja
syntetyzowana nie istnieje w komórce
• PODLEGAJĄCE REGULACJI POZYTYWNEJ –
transkrypcja w obecności induktora
• PODLEGAJĄCE REGULACJI NEGATYWNEJ –
blokowanie transkrypcji przez wolny represor
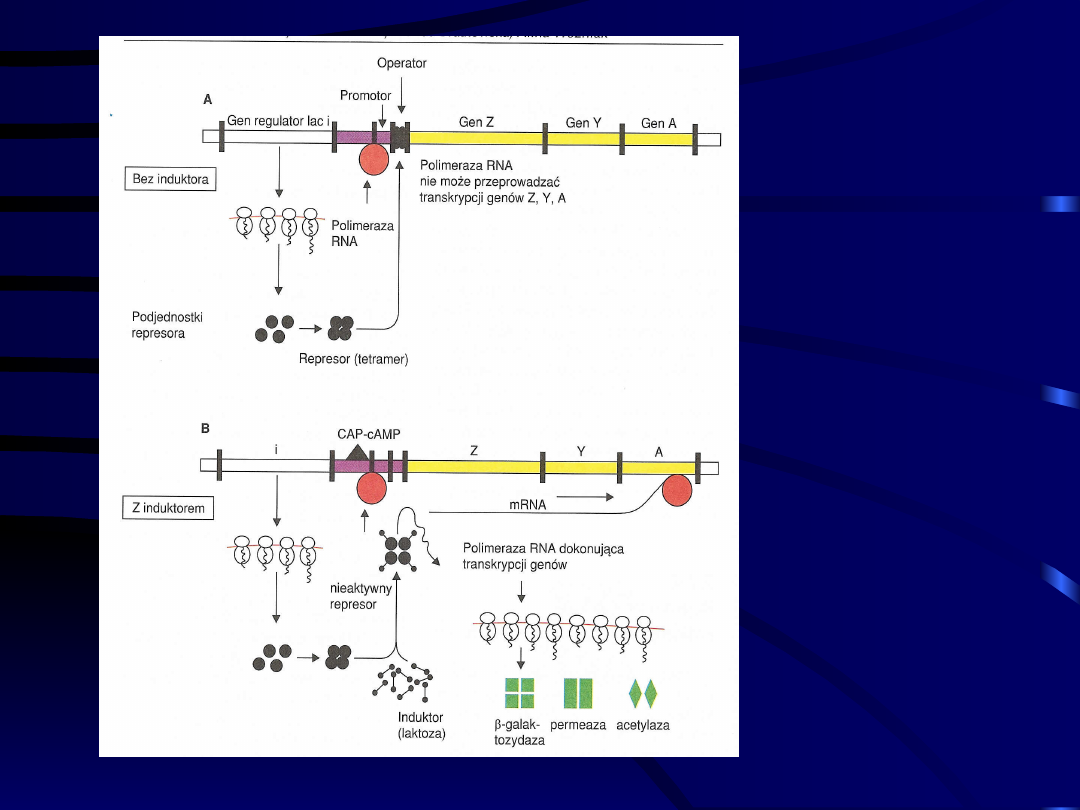
Operon
lakozow
y E.
Coli.
Przykład
regulacji
negatywnej
(A) i
pozytywnej
(B)
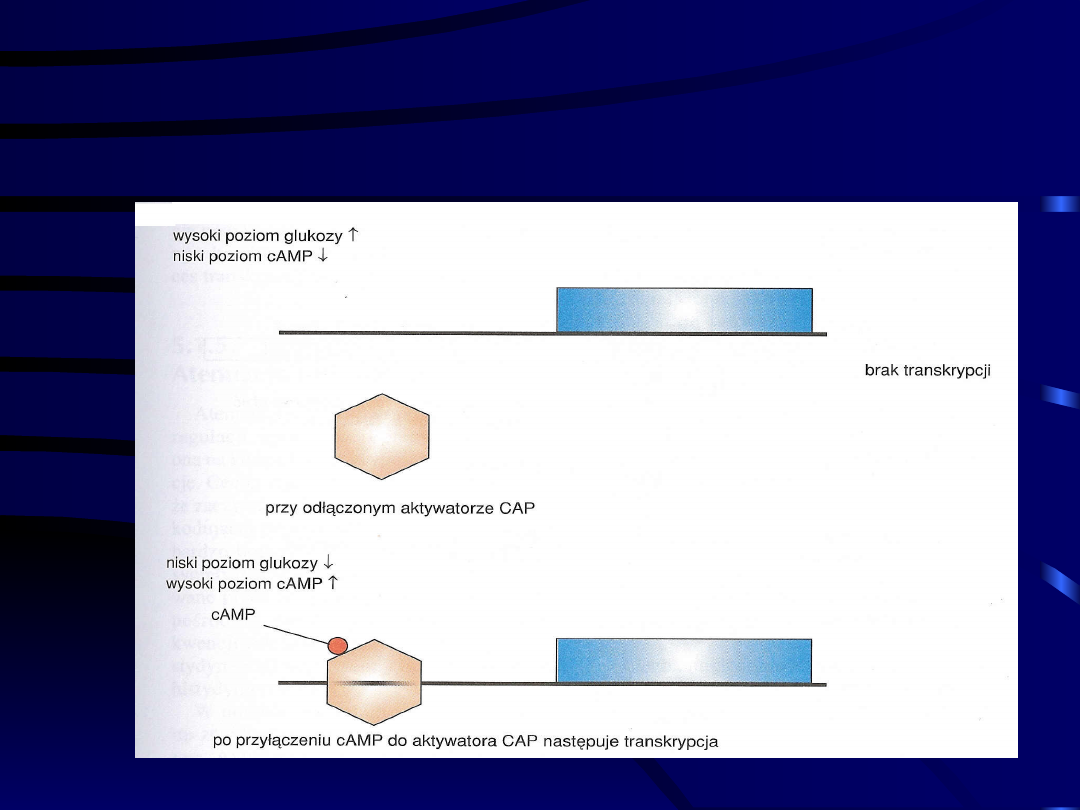
Represja kataboliczna
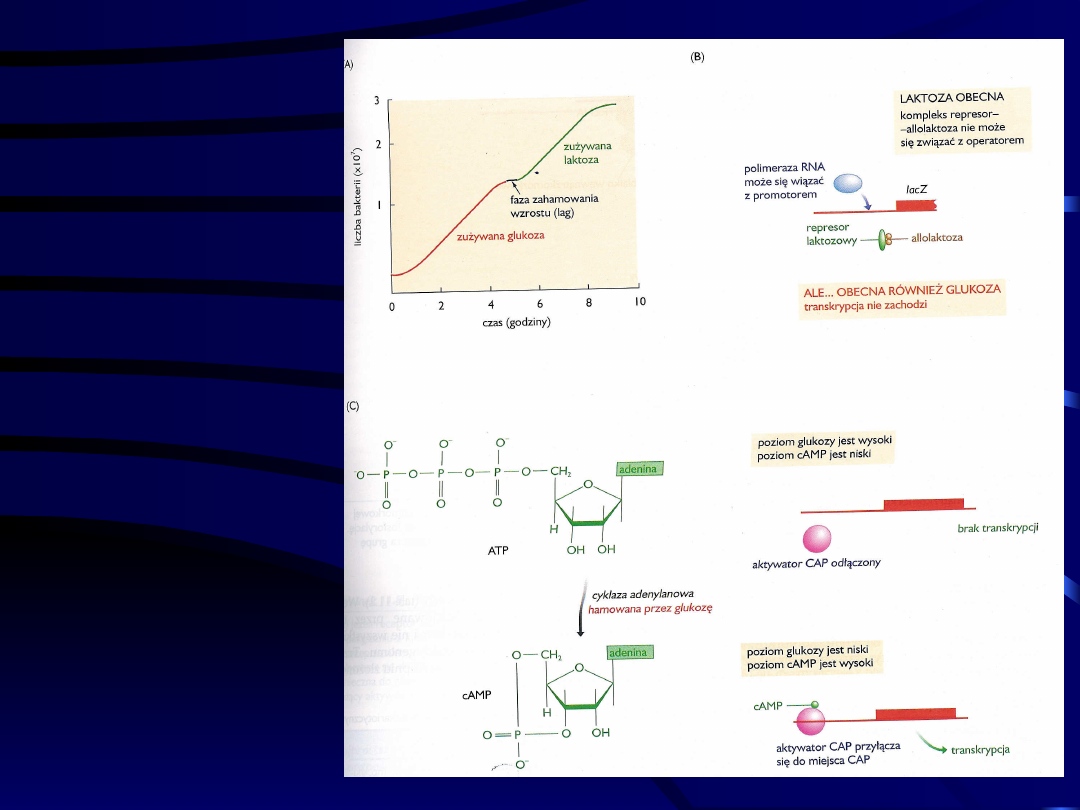
(A) Krzywa
wzrostu E.
Coli w
pożywce
zawierajacej
laktozę i
glukozę. (B)
Mechanizm
znoszenia
działania
repesora
laktozowego
przez
glukozę. (C)
Regulacja
ilości cAMP
w komórce
przez
glukozę.
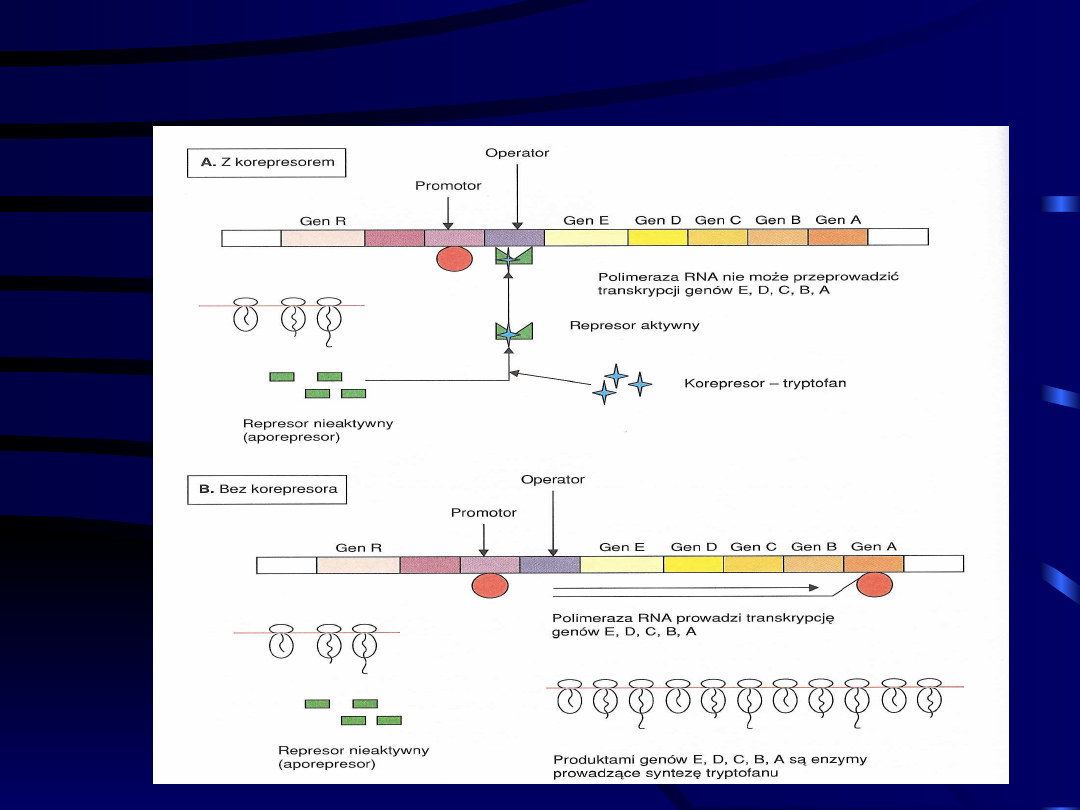
Operon tryptofanowy
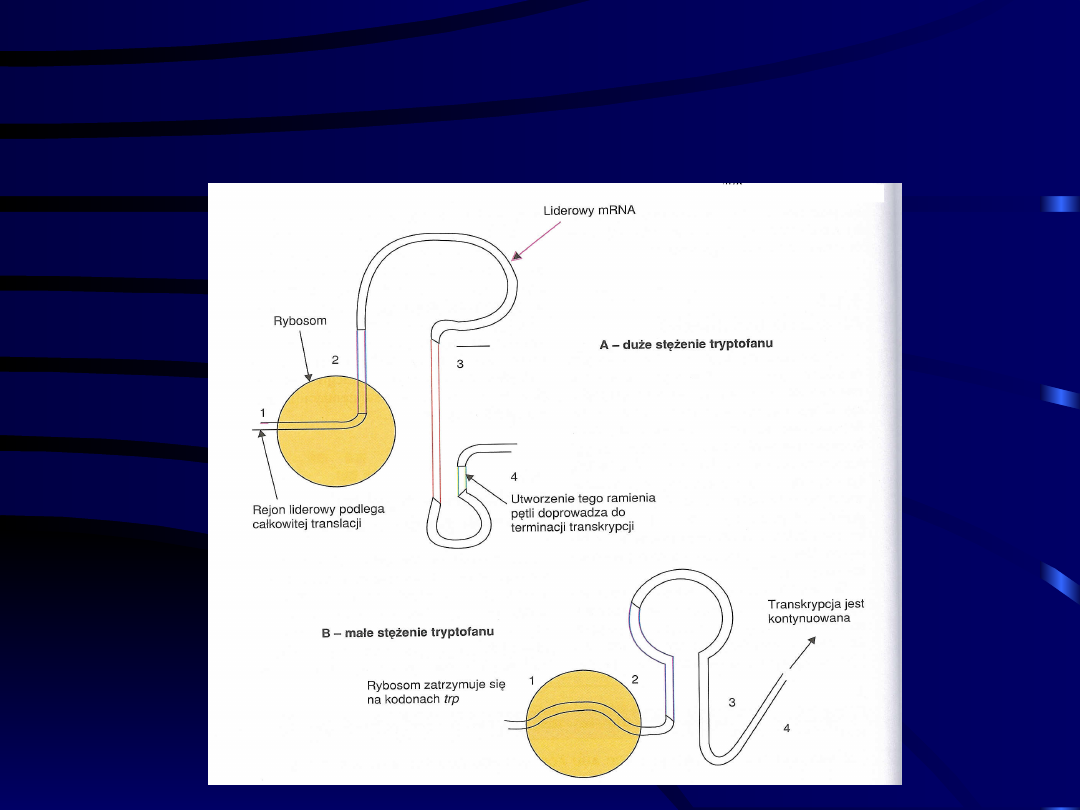
Model atenuacji w operonie
tryptofanowym
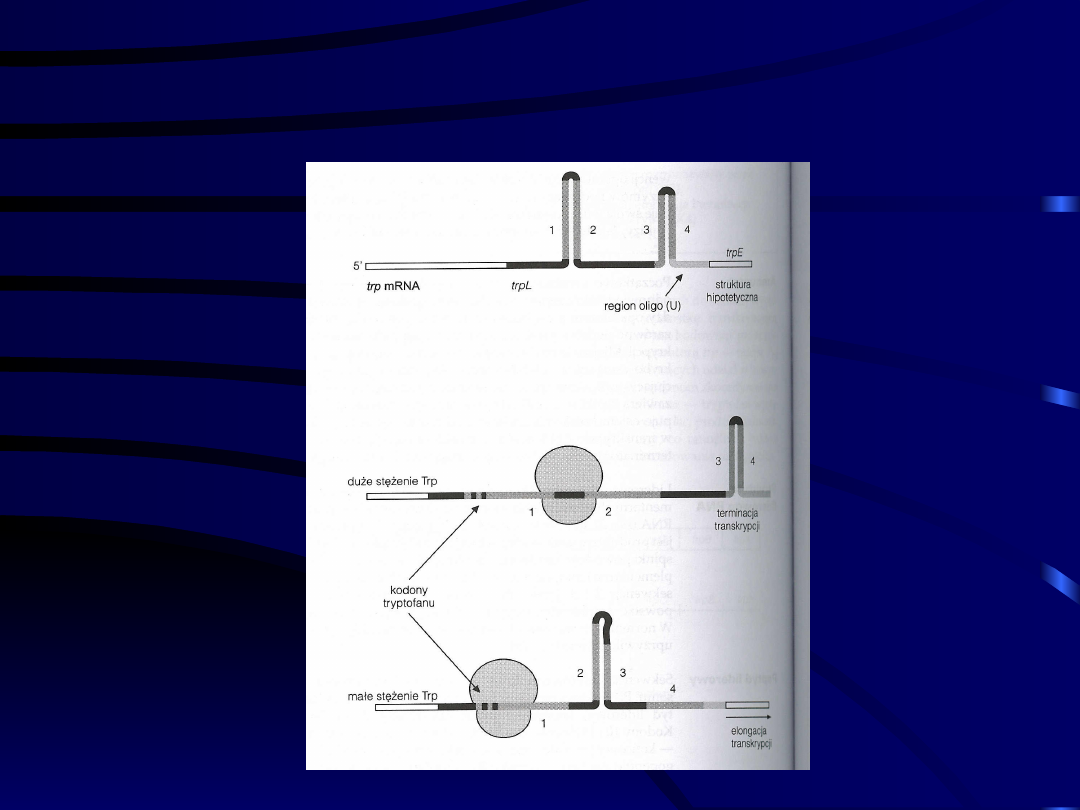
Tranksrypcyjna atenuacja
operonu trp
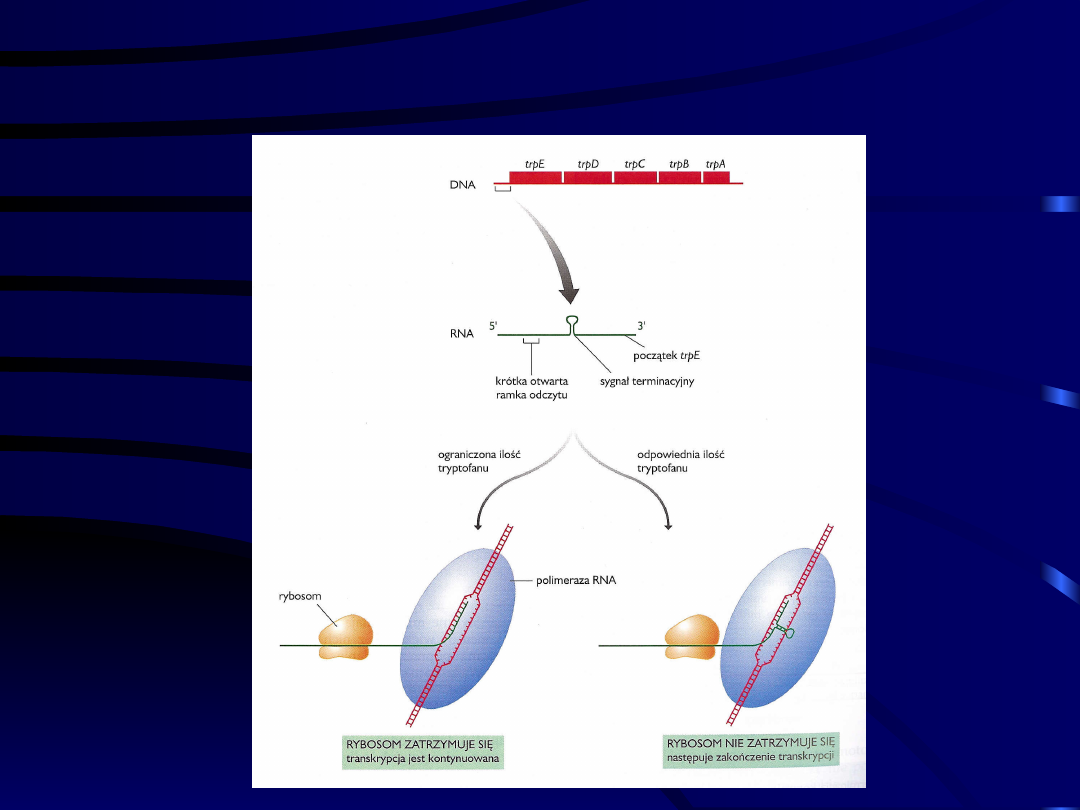
Atenuacja w operonie
trp
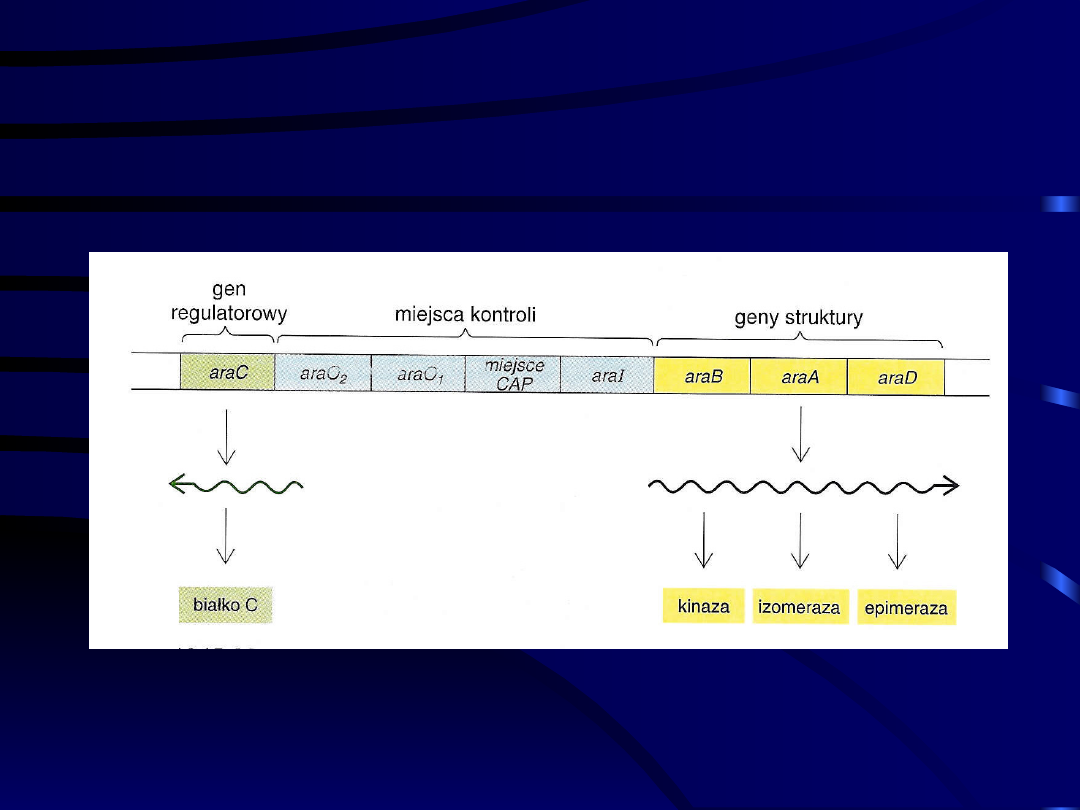
Mapa operonu
arabinozowego
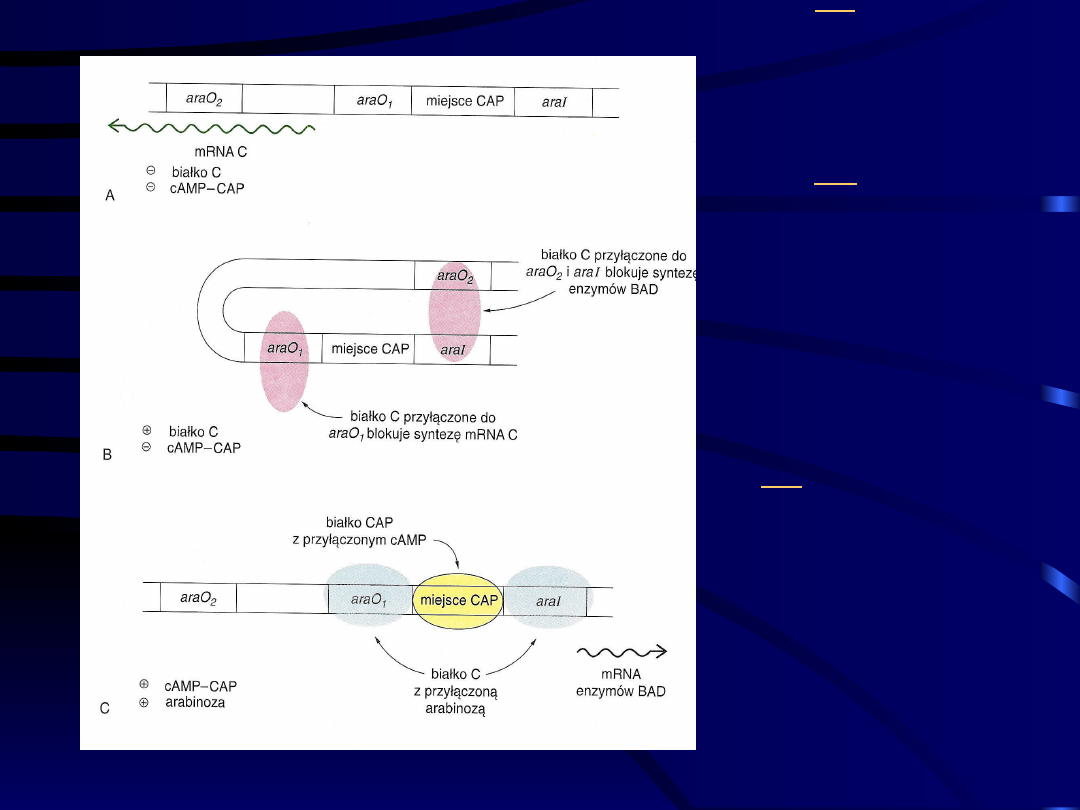
A. Małe
stężenie białka
C – synteza
białka C
B. Małe
stężenie
kompleksu
cAMP-CAP i
duża ilość
białka C – brak
syntezy mRNA
C. Duża ilość
arabinozy i
kompleksu
cAMP-CAP –
synteza mRNA
enzymów
katalizujących
arabinozę
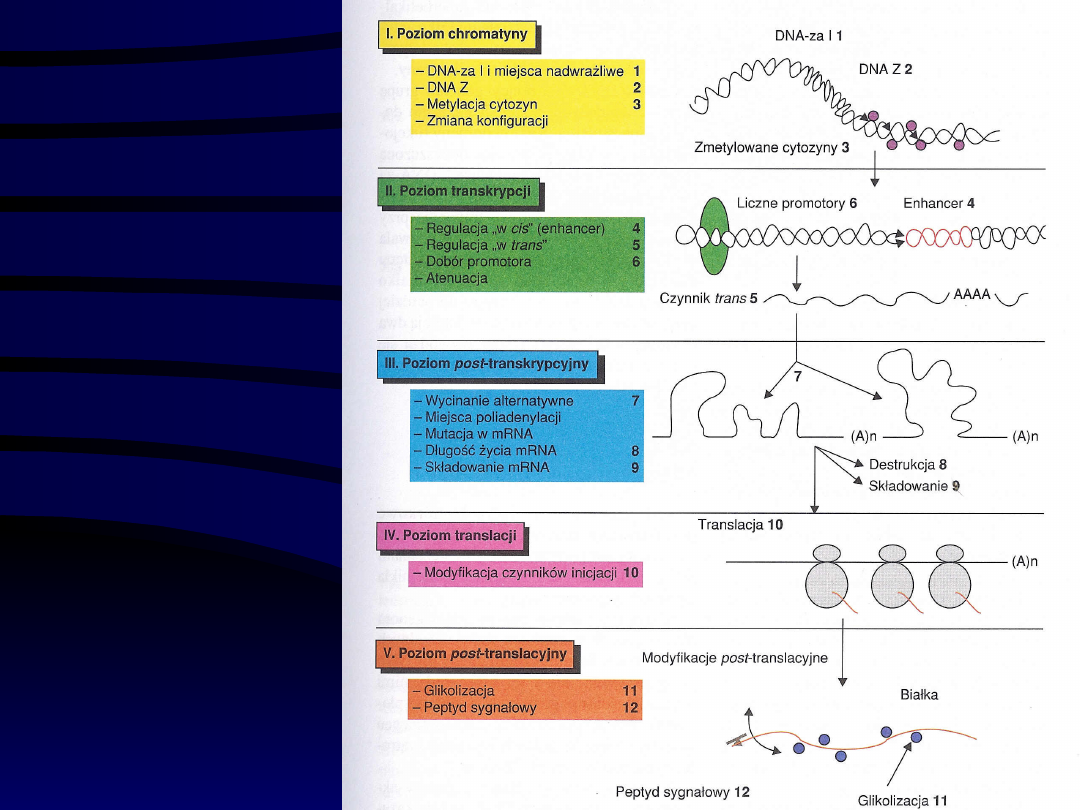
Mechaniz
my
regulacji
ekspresji
genów u
Eukaryota
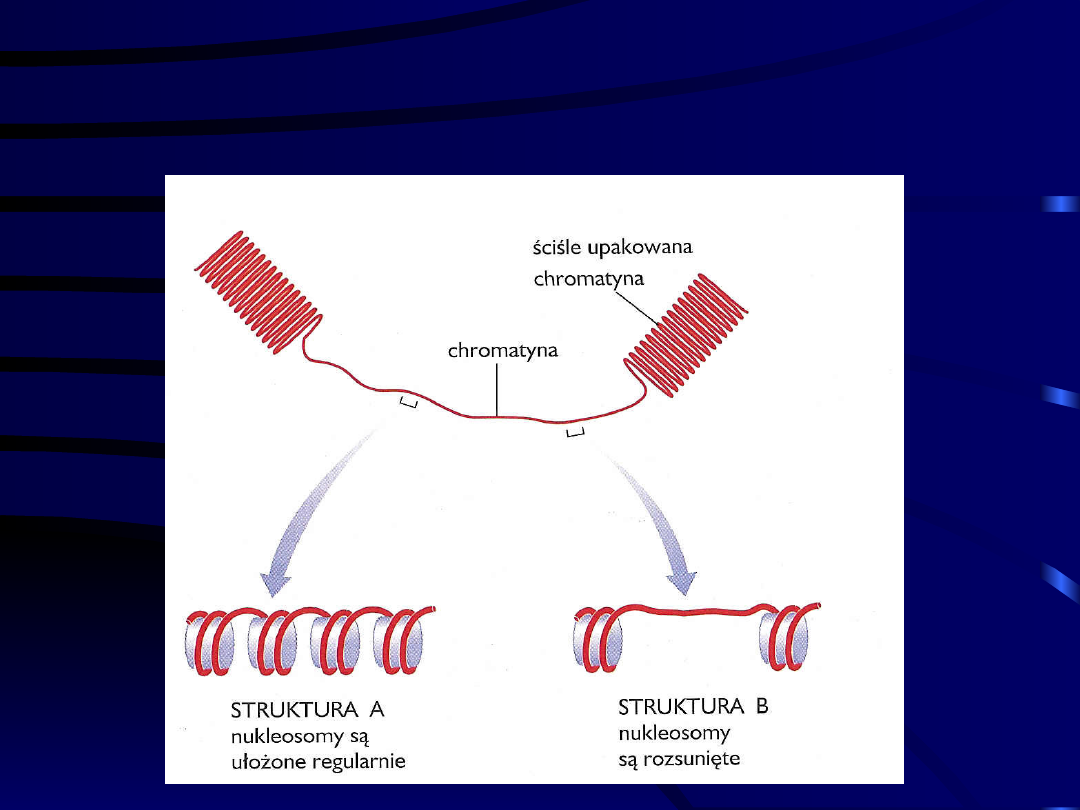
Wpływ struktury chromatyny
na ekspresję genów
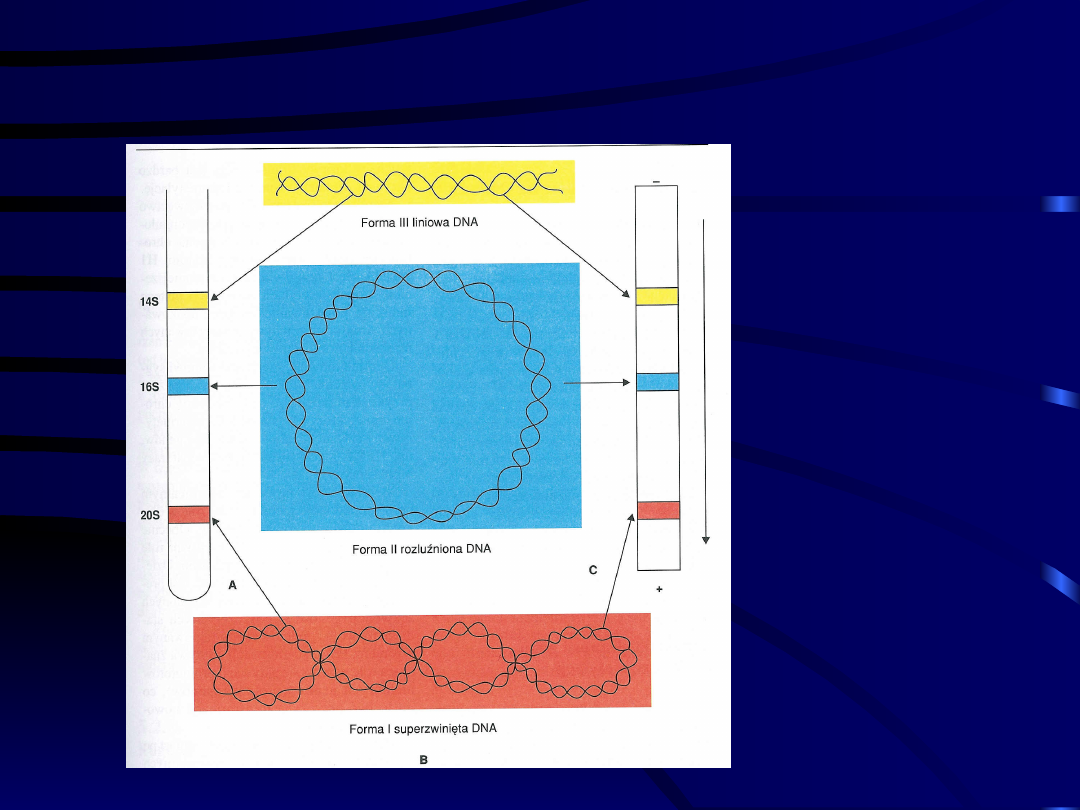
Formy przestrzenne
DNA
A –
ultrawirowanie
B – mikroskopia
elektronowa
C - elektroforeza
Kolor czerwony
– forma I
Kolor niebieski –
forma II
Kolor żółty –
forma III

Miejsca wrażliwe na DNA-
zę
• Miejsca wrażliwe na DNA-zę
odpowiadają miejscom aktywnym
• Znajdują się w specjalnej konformacji
chromatyny, która czyni je bardziej
dostępnymi
• Specyficzna struktura przestrzenna jest
najwyższym poziomem regulacji
• Miejsca nadwrażliwe na DNA-zę – nie ma
tu nukloesomów i białka wiążące mają tu
łatwy dostęp do DNA
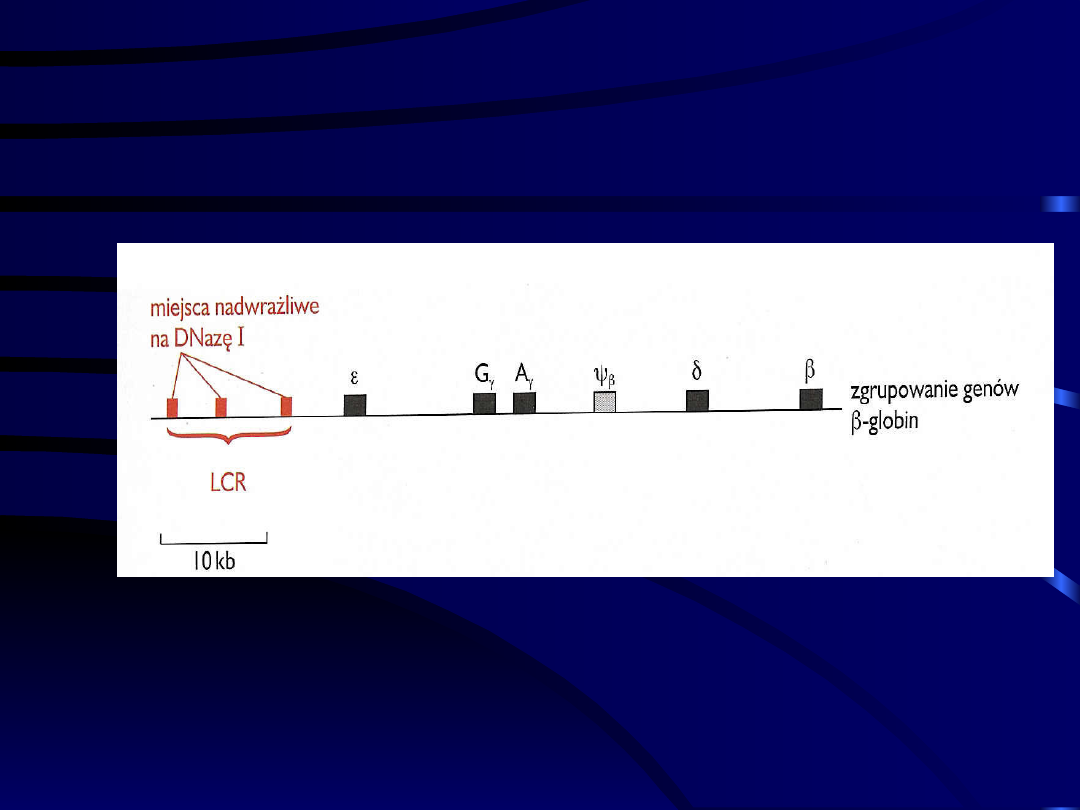
Miejsca nadwrażliwe na DNazę
wskazują położenie LCR (locus
control region) w ludzkim genomie

Metylacja DNA
• Metylacja DNA polega na enzymatycznym
przyłączaniu reszt metylowych (-CH
3
) do
nukleotydów
• Procesowi temu podlegają cytozyny wchodzące w
skład dinukleotydu CpG
• Metylacja cytozyn związana jest z obniżeniem
aktywności transkrypcyjnej – stanowi sygnał
wyłączenia genu
• Jest procesem odwracalnym
• Spełnia istotną rolę w długoterminowej inaktywacji
genów (chromosom X, protoonkogeny)
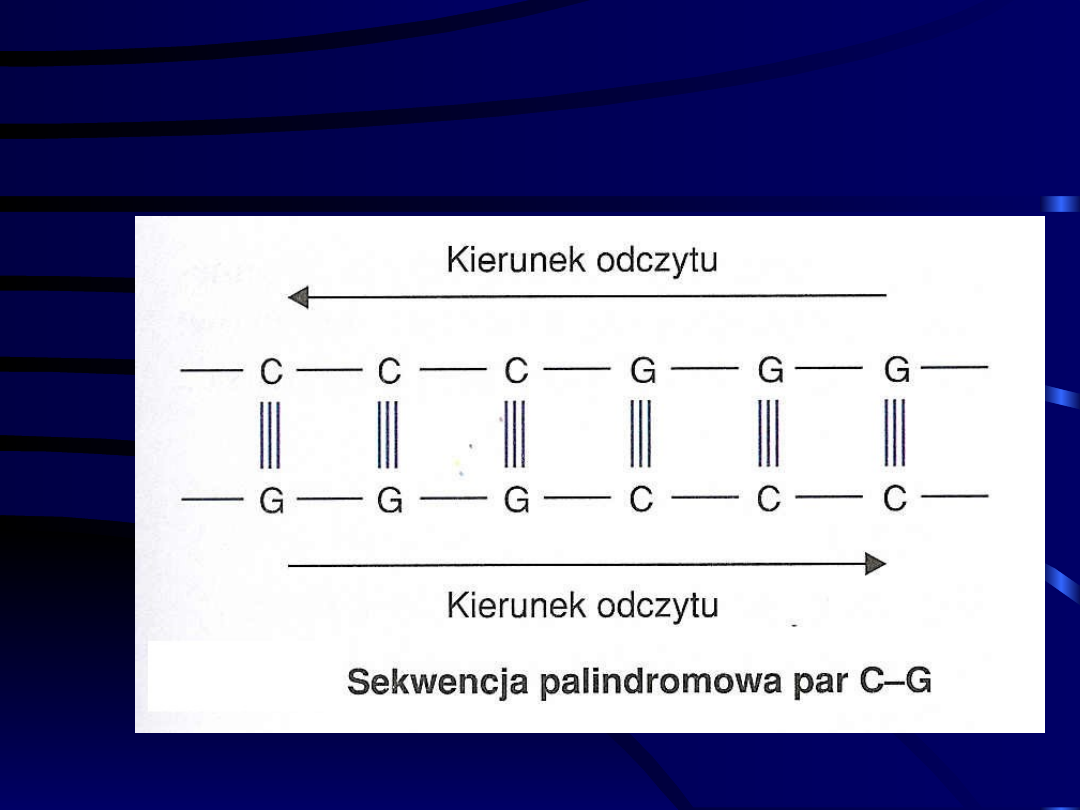
Sekwencja CpG

Czynniki transkrypcyjne
• Są to czynniki kontroli ekspresji
genów
• Zdolne są przyłączyć się do DNA do
specyficznych sekwencji
• Mogą regulować poziom transkrypcji
dodatnio lub ujemnie
• Kodowane są przez geny
regulatorowe, które stanowią 5-10 %
genomu człowieka
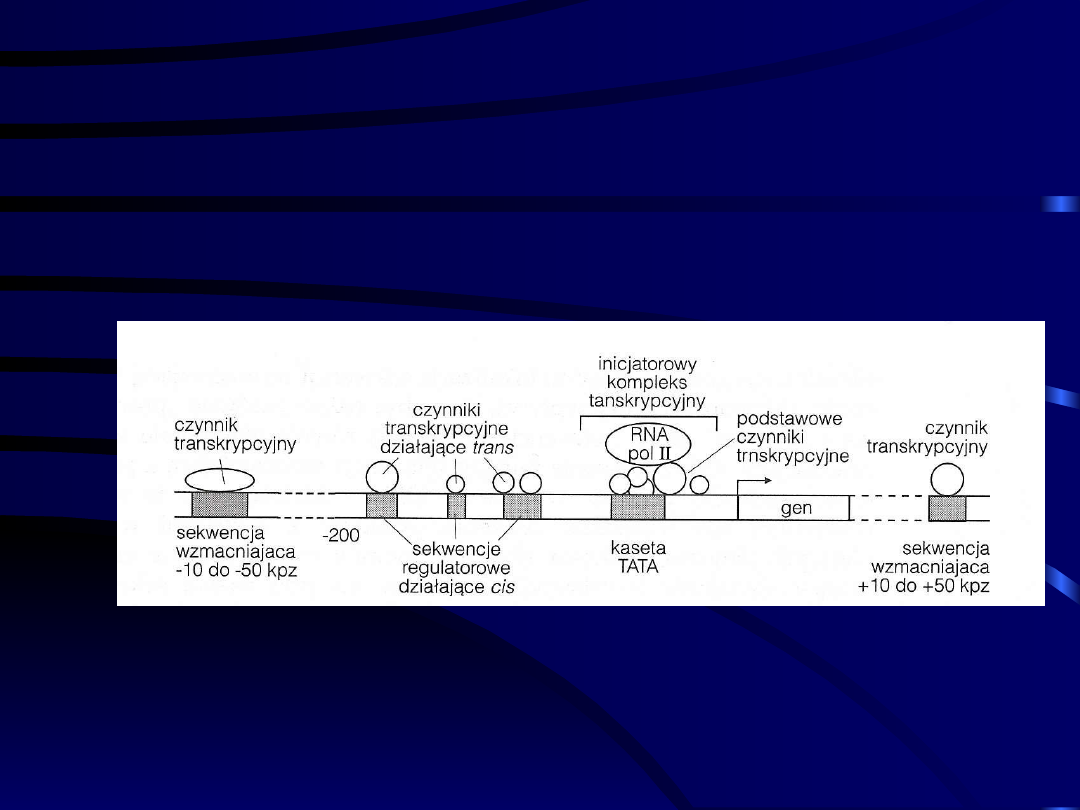
Miejsca przyłączania czynników
transkrypcyjnych w komórkach
eukariotycznych

Czynniki transkrypcyjne
• Mają budowę modularną i składają
się z domen białkowych
pełniących określone funkcje:
- DOMENY WIĄŻĄCE DNA
- DOMENY ODPOWIEDZIALNE ZA
DIMERYZACJĘ
- DOMENY TRANSAKTYWUJĄCE
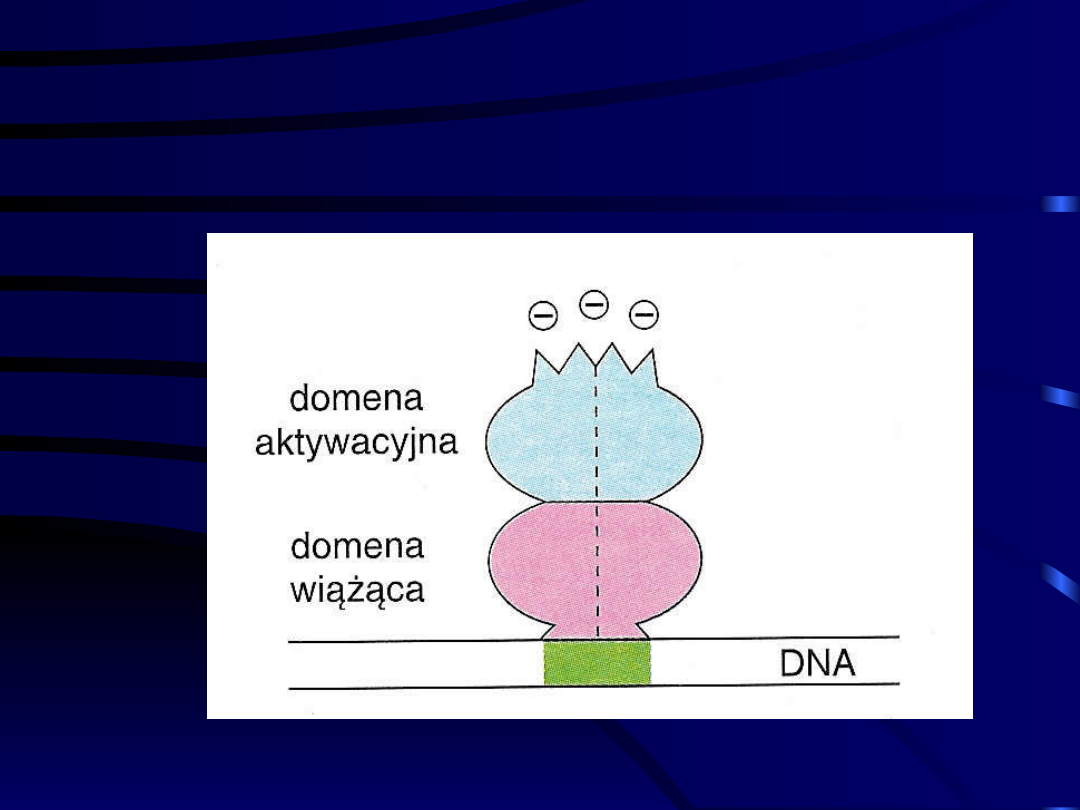
Schematyczna budowa
aktywatorów
transkrypcji

Czynniki transkrypcyjne
• Scharakteryzowano trzy typy domen wiążących
na podstawie występujących w nich motywów:
- helisa-zwrot-helisa
- palce cynkowe
- helisa-pętla-helisa
• W domenach odpowiedzialnych za dimeryzację
wyróżnia się dwa typy motywów:
- suwak leucynowy
- helisa-pętla-helisa
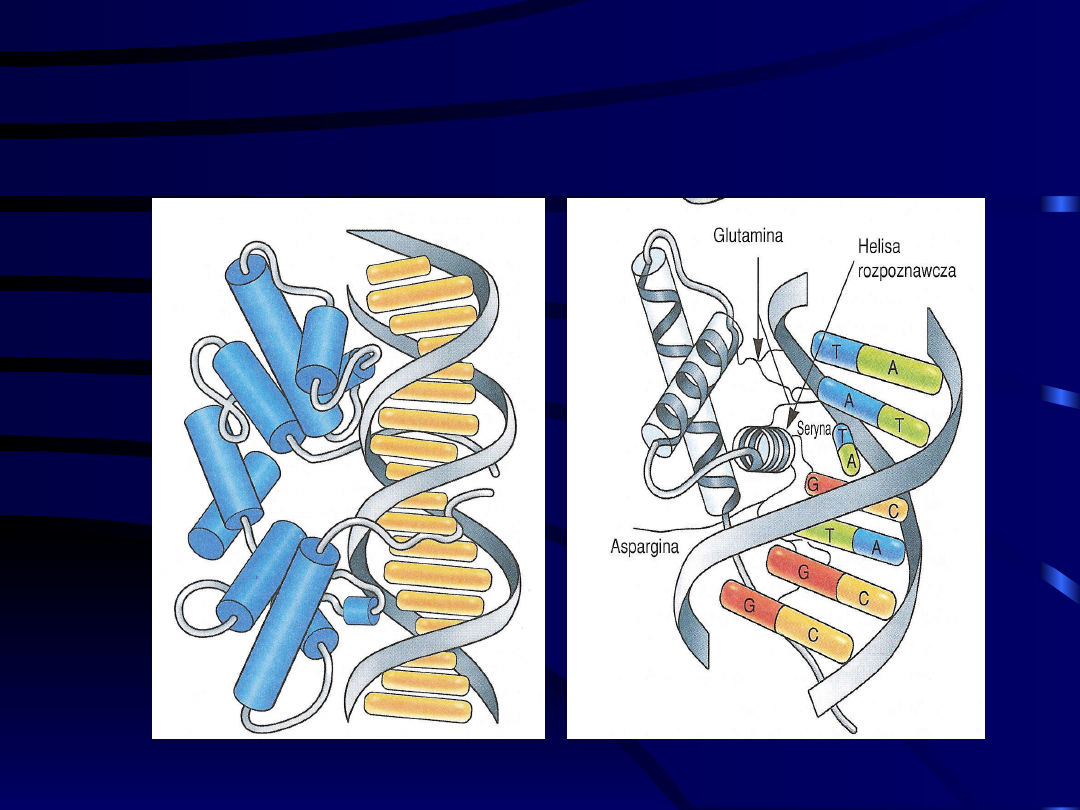
Struktura helisa–zwrot-
helisa
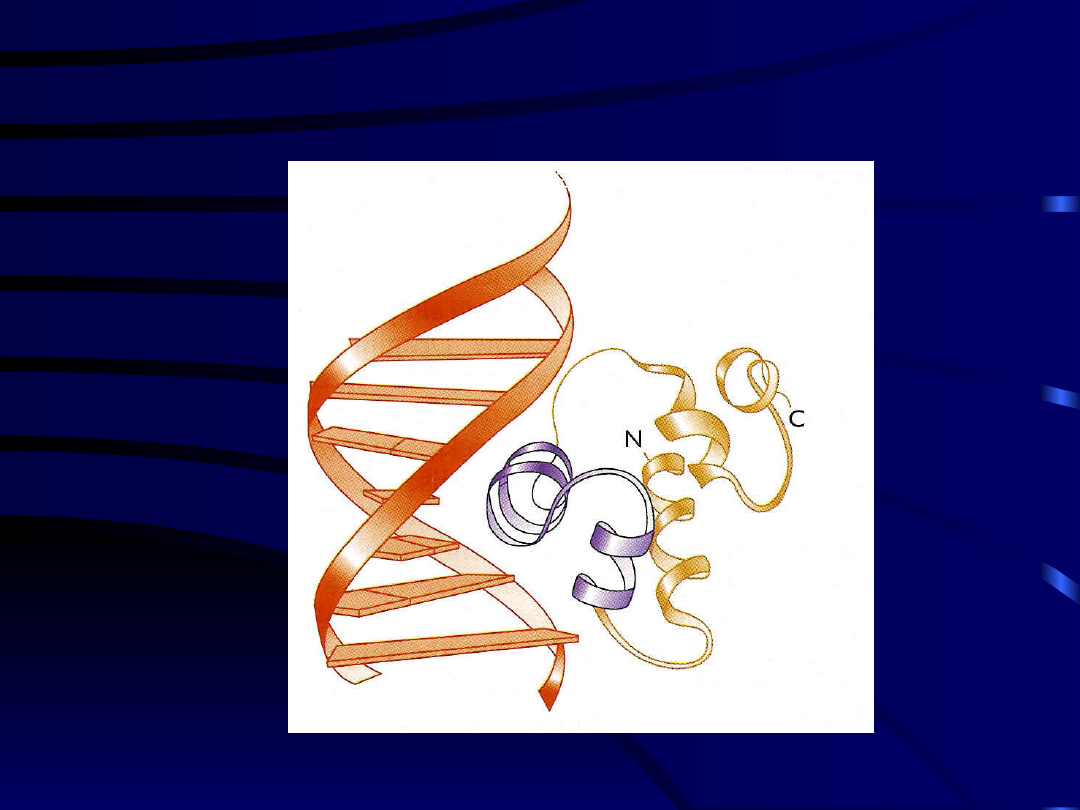
Domena typu helisa-skręt-helisa
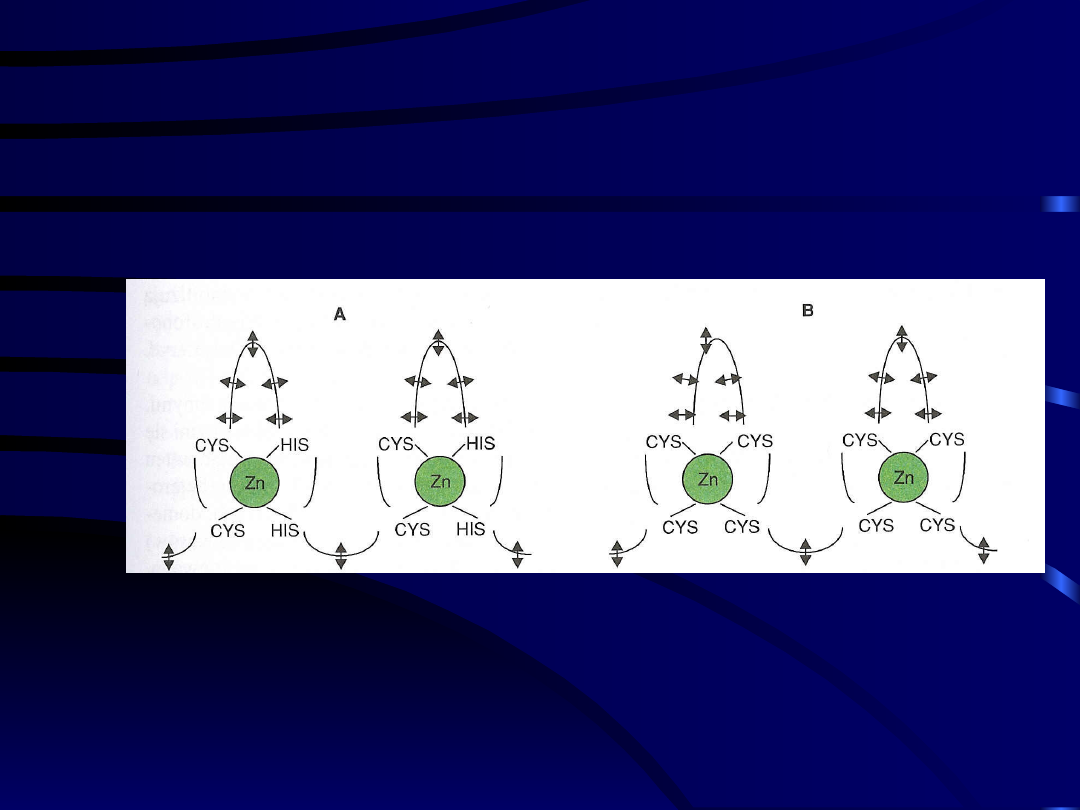
Motyw „palca
cynkowego”
A i B – struktury „palców cynkowych”
różniące się rodzajem aminokwasu
wiążącego cynk
- strzałki oznaczają miejsca
oddziaływania z DNA
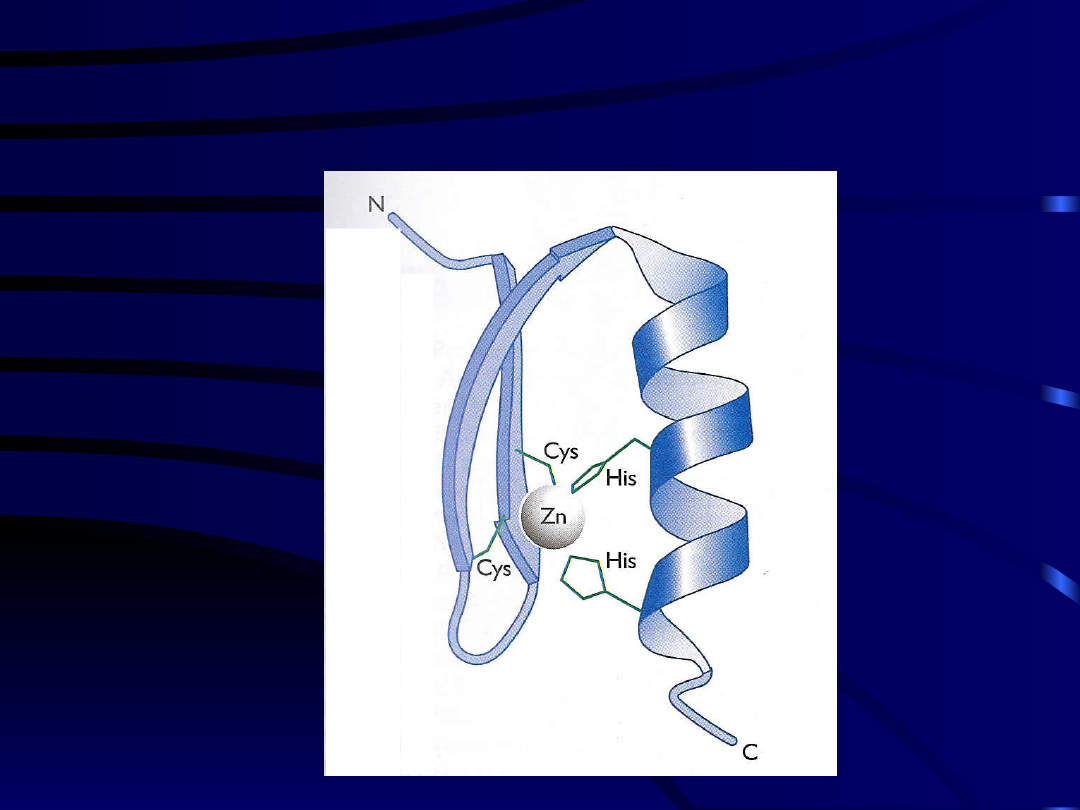
Palec cynkowy Cys
2
His
2
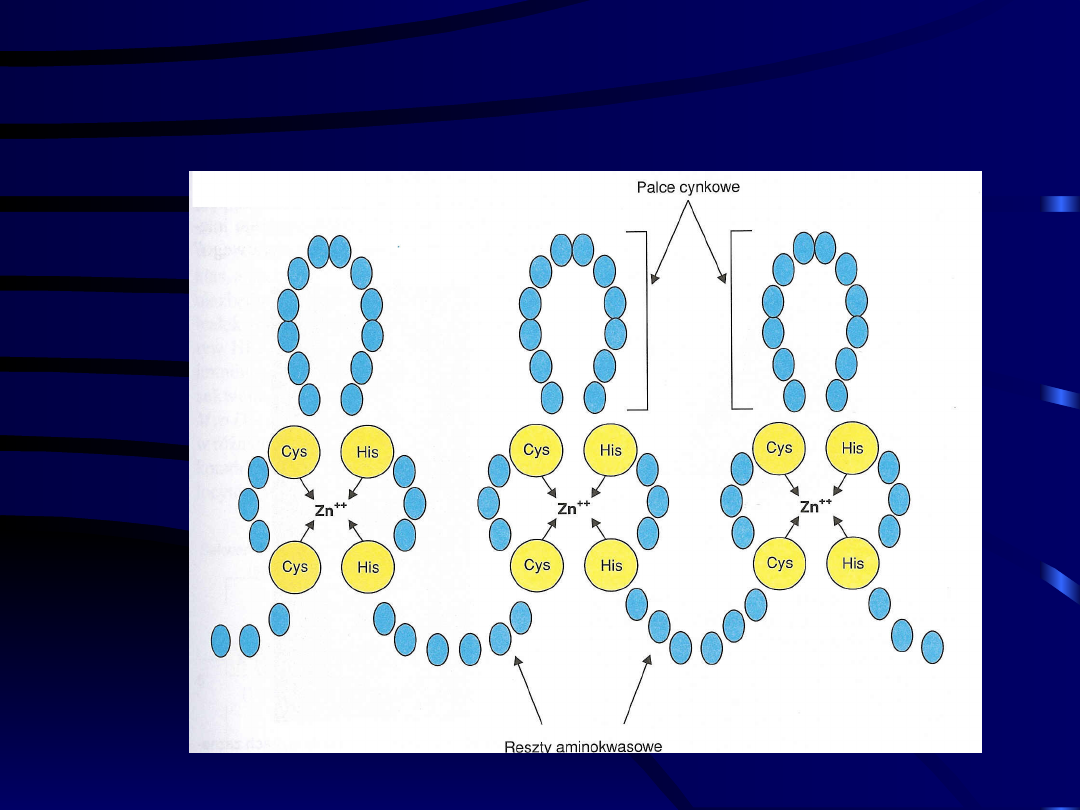
Czynnik transkrypcji
Sp1
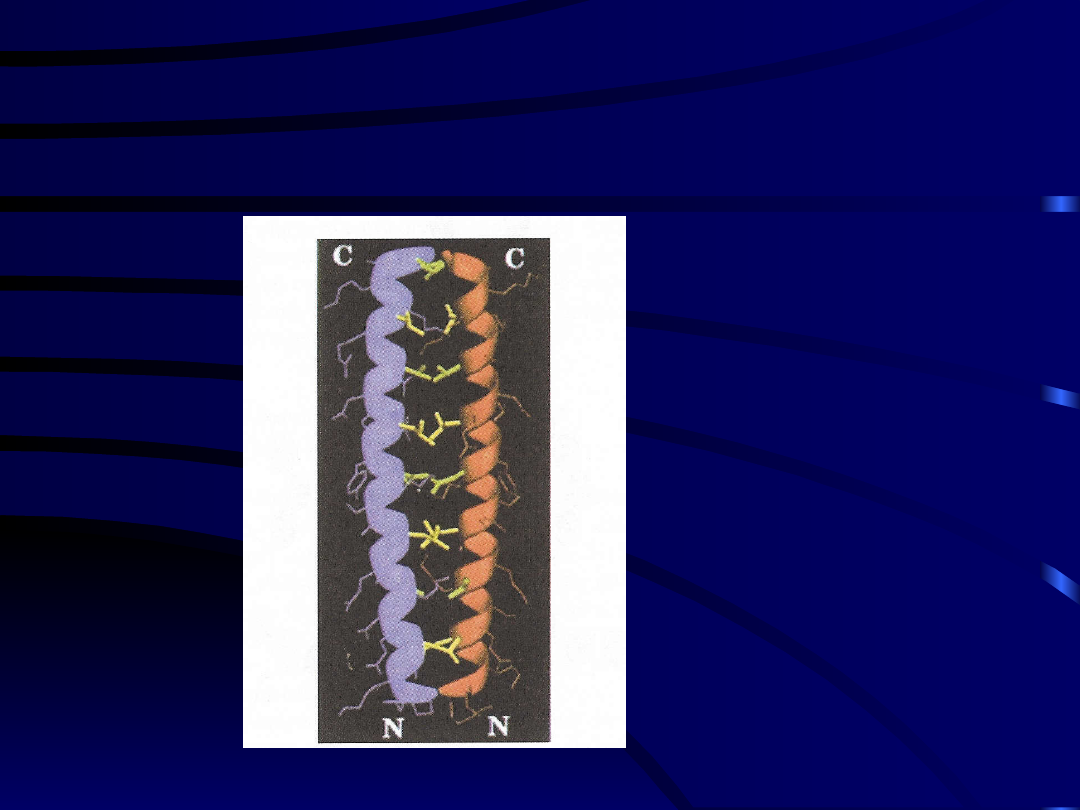
Motyw „suwaka
leucynowego”
kolor żółty i
zielony =
miejsca
kontaktu na
leucynach
Wstęgi = Helisy
Rozgałęzione
linie =
Łańcuchy
boczne
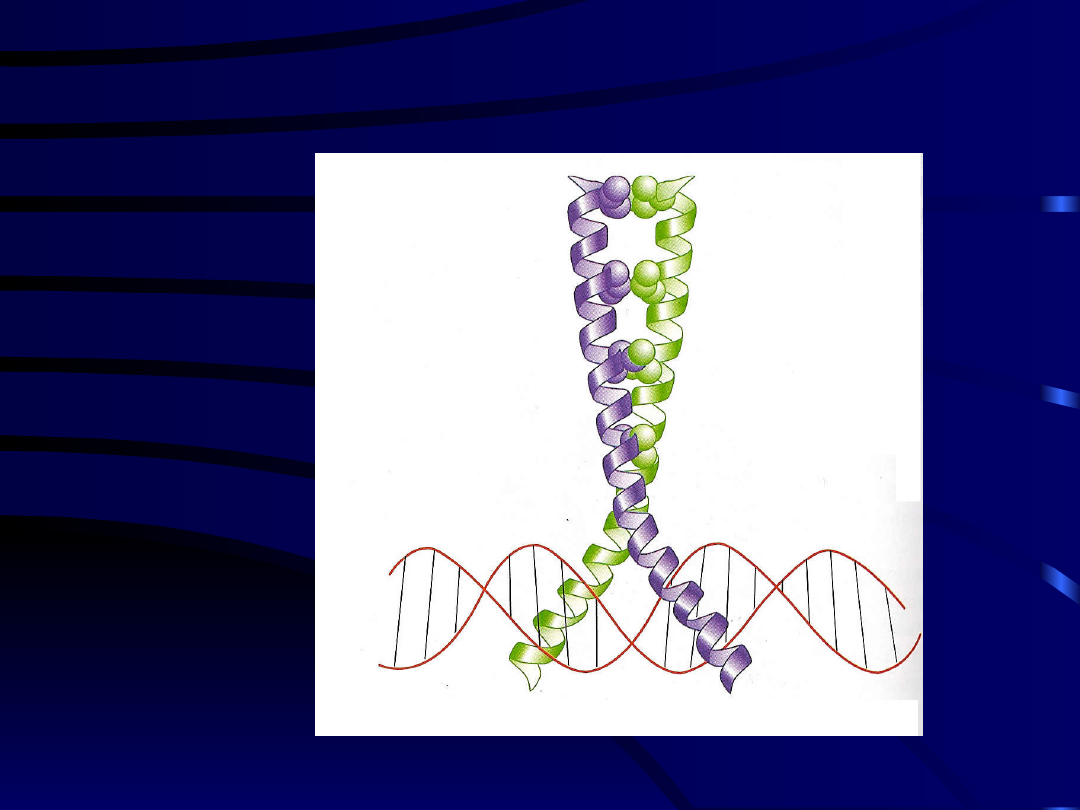
Suwak leucynowy

Domena typu helisa-wstęga-
helisa
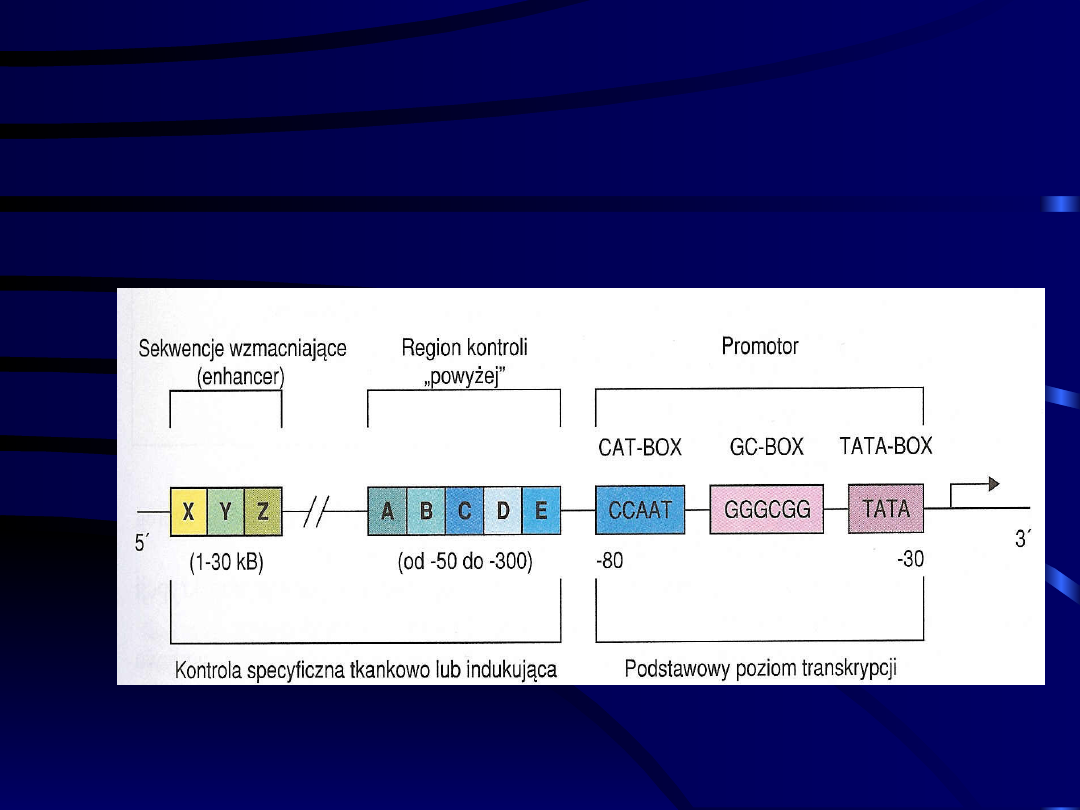
Różne miejsca wiązania
czynników transkrypcyjnych z
DNA

Sekwencje wzmacniające
(enhancer)
• Wzmagają znacznie poziom transkrypcji
genu, z którym są związane
• Mogą występować jako sekwencje
wyciszające (silencer) i odpowiadać za
ograniczenie transkrypcji
• Ich inwersja nie oznacza utraty wpływu
na transkrypcję lecz jej obniżenie
• Utrzymują swe właściwości aktywatora
nawet jeśli są przemieszczone o kilka lub
kilkaset tysięcy par zasad na nici DNA
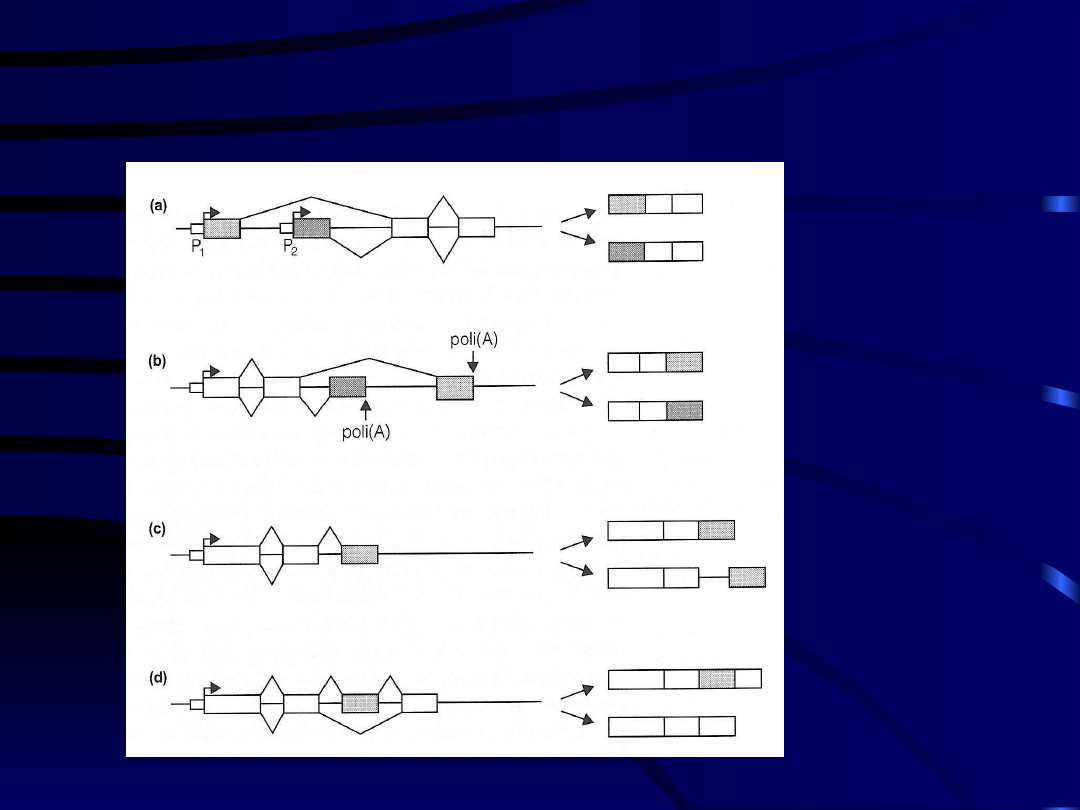
Splicing alternatywny
(a)Alternatyw
ny wybór
promotoró
w
(b)Alternatyw
ny wybór
miejsc
rozcięcia i
poliadenyl
acji
(c) Zachowani
e intronu
(d)Ominięcie
eksonu
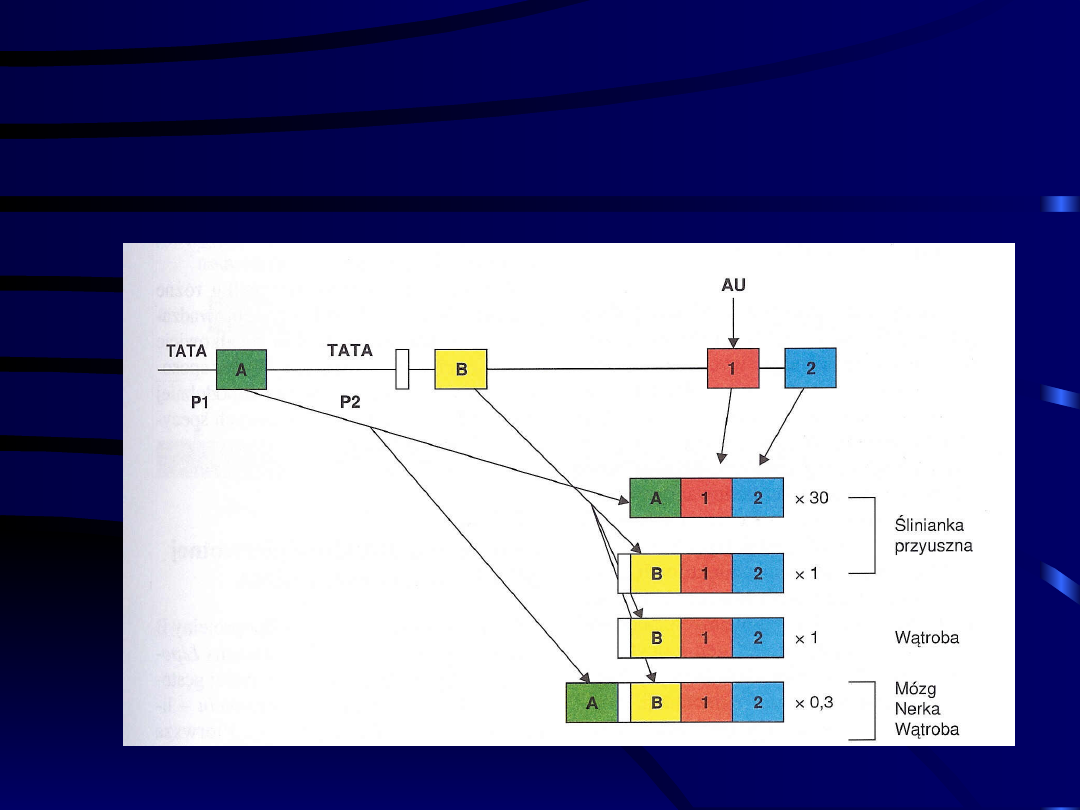
Rożnicowanie transkryptów przez
promotory alternatywne – model
genu alfa-amylazy (promotory P1 i
P2)
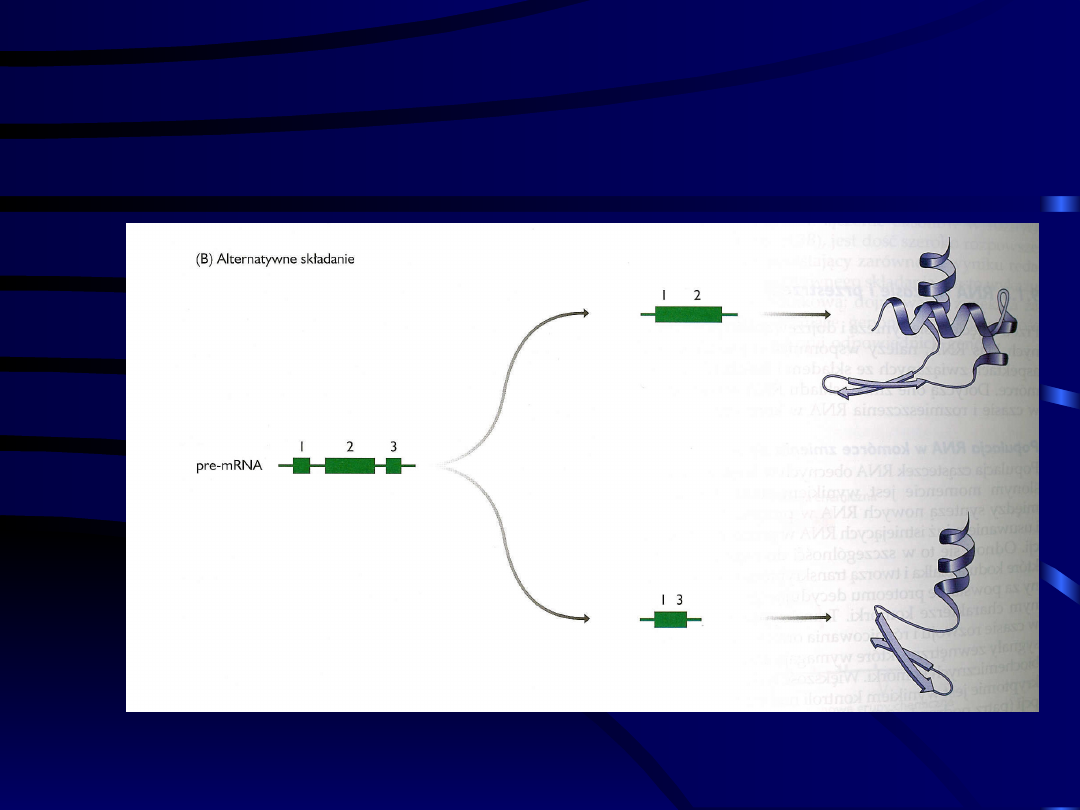
Alternatywne składanie
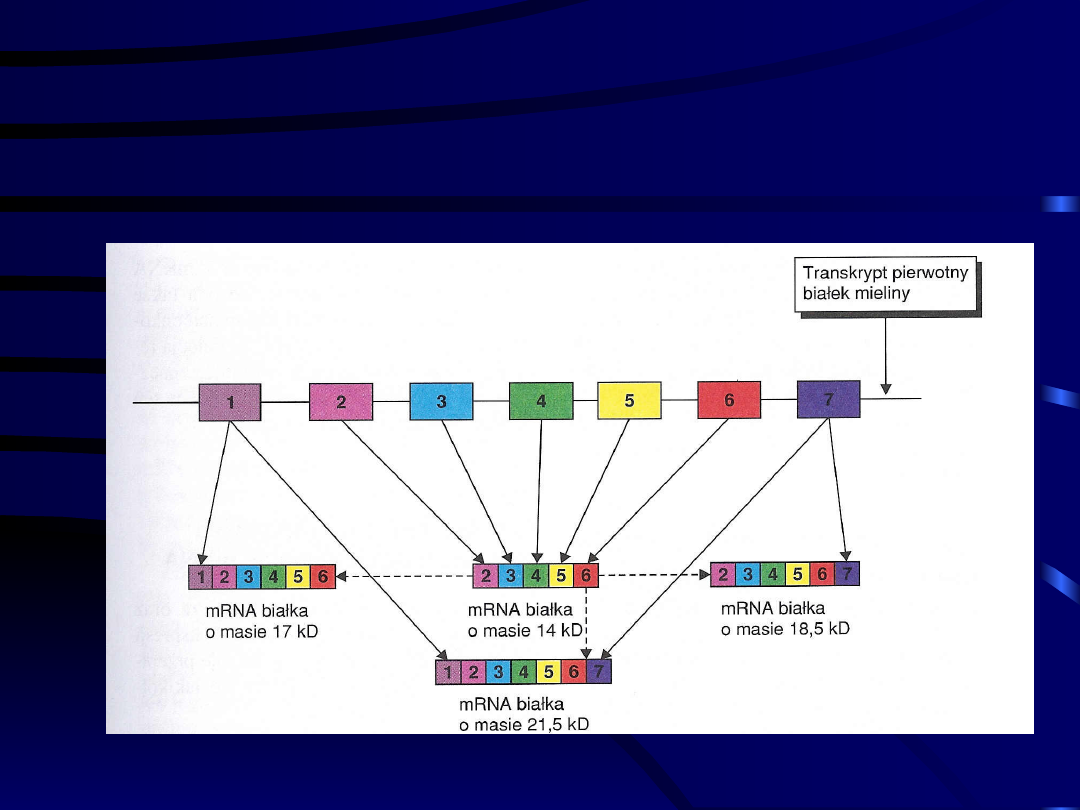
Cztery białka mieliny otrzymane
przez alternatywne wycinanie
eksonów
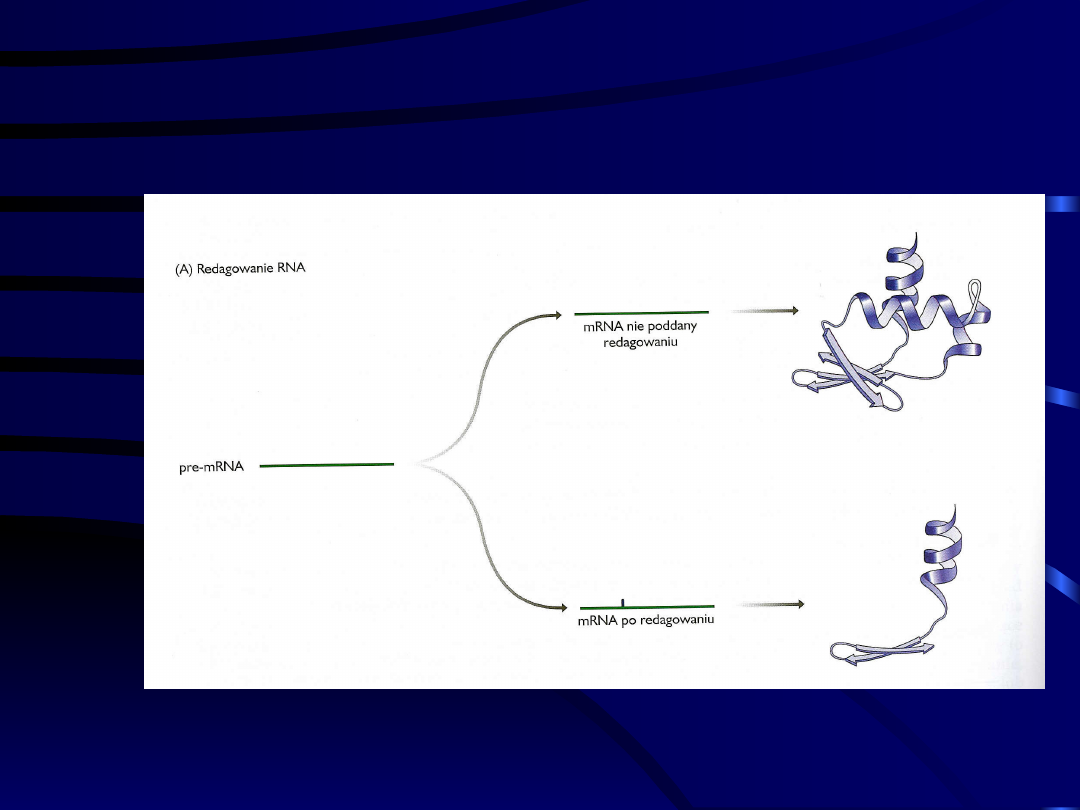
Redagowanie RNA
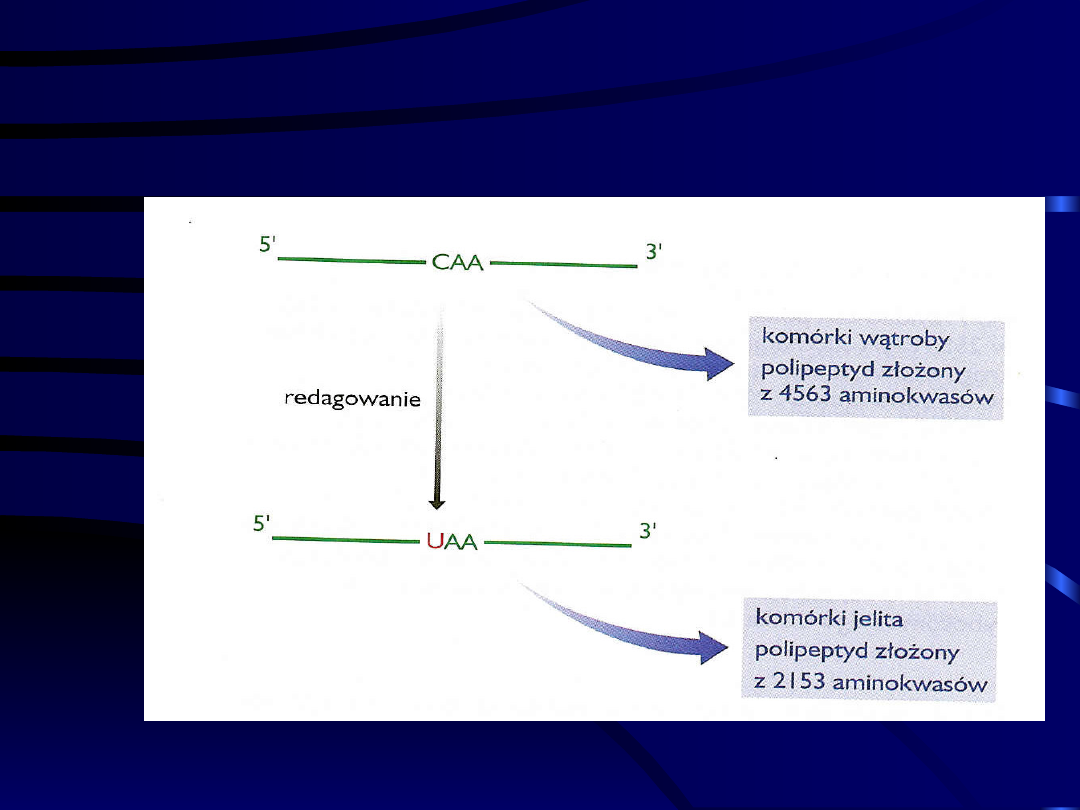
Redagowanie mRNA ludzkiej
apolipoproteiny B

Przykłady redagowania RNA u
ssaków
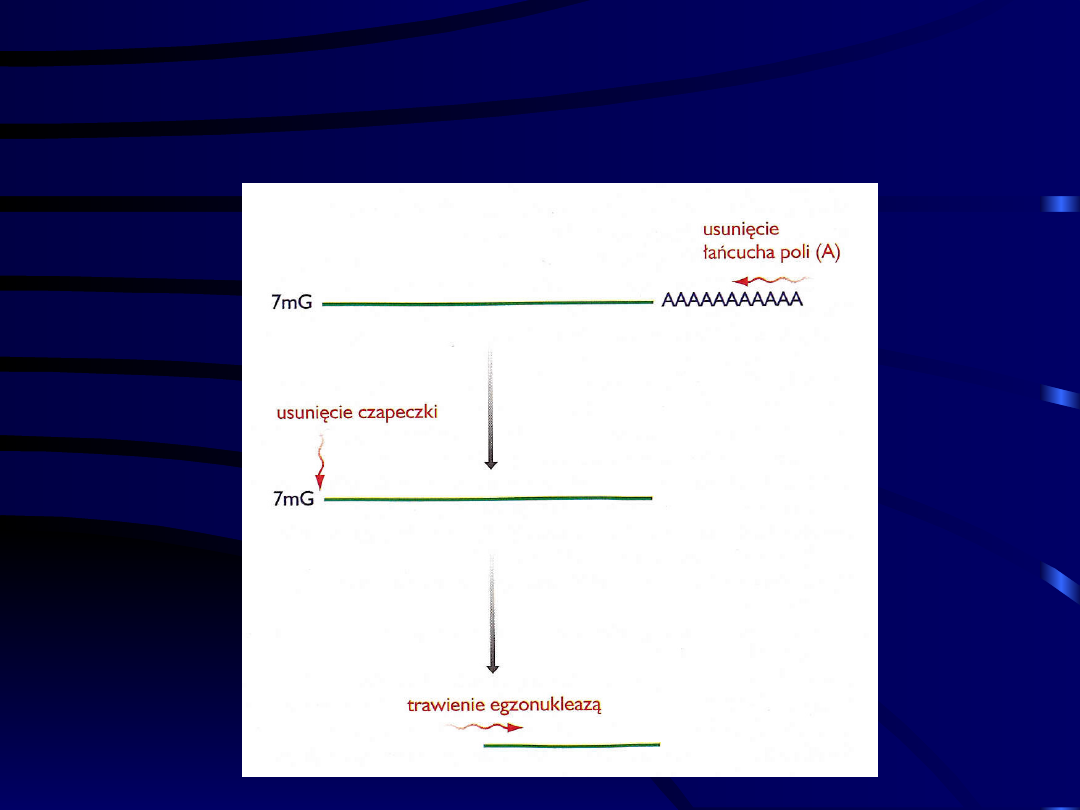
Szlak degradacji mRNA zależny
od deadenylacji
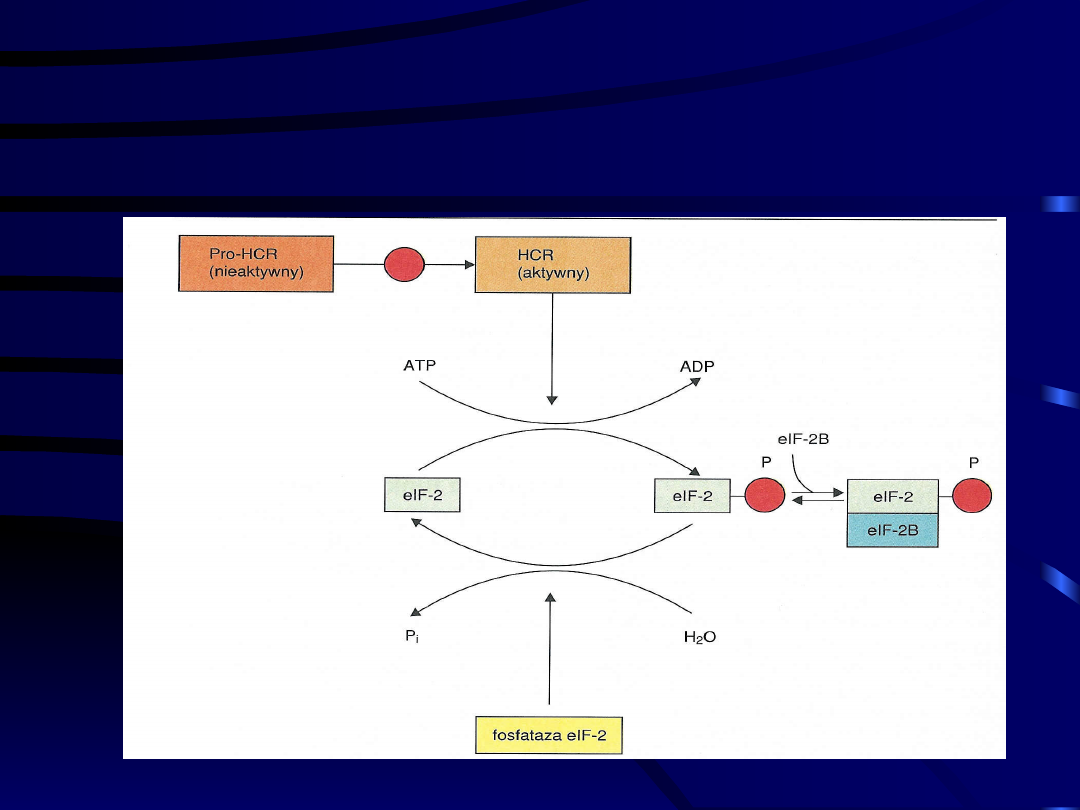
Regulacja na poziomie translacji
- model syntezy białek w
retikulocytach kontrolowanej
przez hem
HCR – Heme Controlled Represor

Białka szoku cieplnego
(HSP)
Białka szoku cieplnego syntetyzowane są
w wyniku działania czynników stresowych tj:
• podwyższona temperatura
• infekcja wirusowa
• czynniki uszkadzające DNA
• analogi aminokwasów
• zmiany pH środowiska
• obecność etanolu

Struktura białka Hsp70
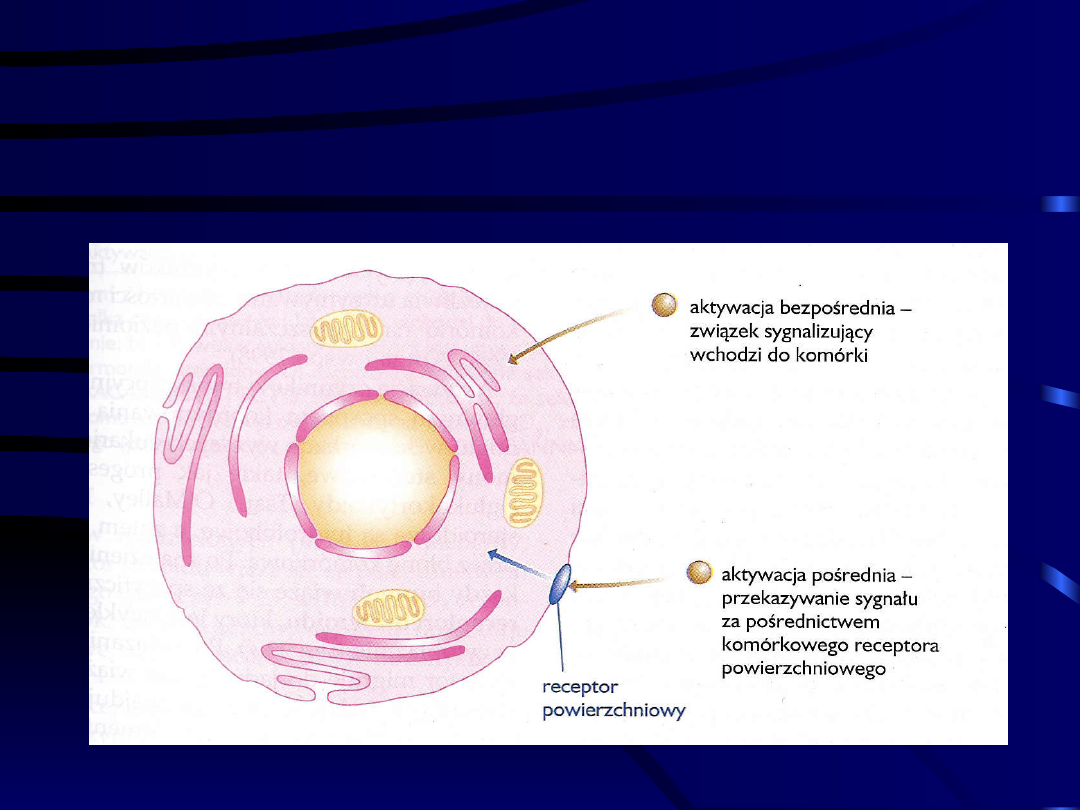
Możliwe sposoby wywierania wpływu na
zdarzenia zachodzące wewnątrz komórki
przez zewnątrzkomórkowy związek
sygnalizujący
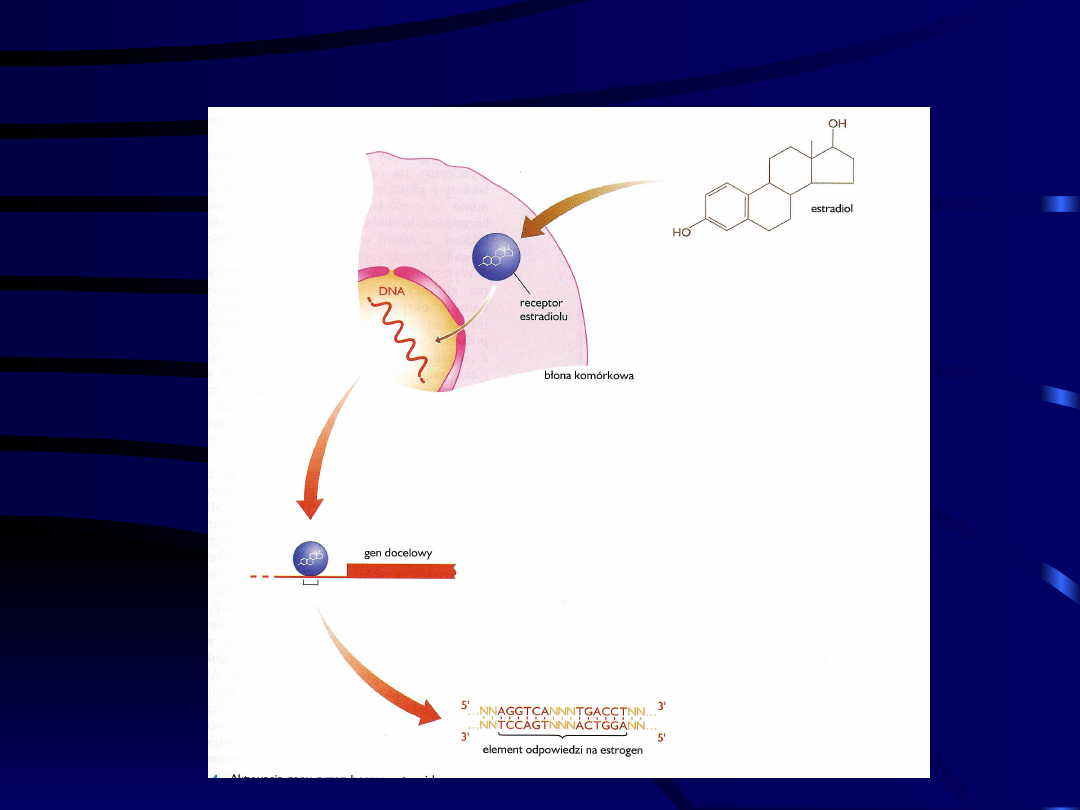
Aktywacja genu przez hormon
steroidowy

Receptory hormonów
steroidowych
Wszystkie receptory hormonów steroidowych
posiadają identyczną strukturę na którą składają się:
• Koniec HN
2
-terminalny (tzw. domena A-B genu)
specyficzny dla receptora, niekonserwatywny
• Domena C – bardzo konserwatywna, składa się z
około 65 aminokwasów, domena ta oddziałuje z DNA
• Region niekonserwatywny o zmiennej długości
• Domena E o zmiennej długości – do niej wiąże się
hormon
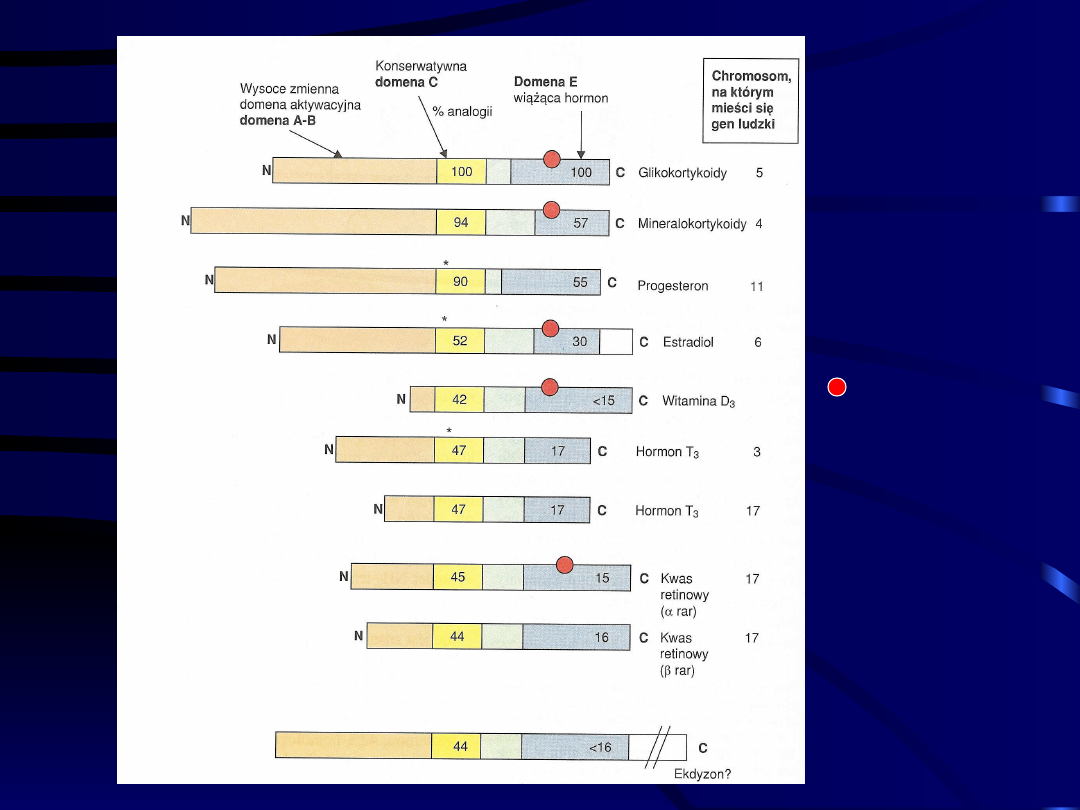
Schemat
budowy
jądrowych
receptorów
hormonów
steroidowy
ch
-
receptory,
dla
których
zbadano
już
wiązanie
hormonu
* -
receptory,
dla których
zbadano już
wiązanie z
DNA
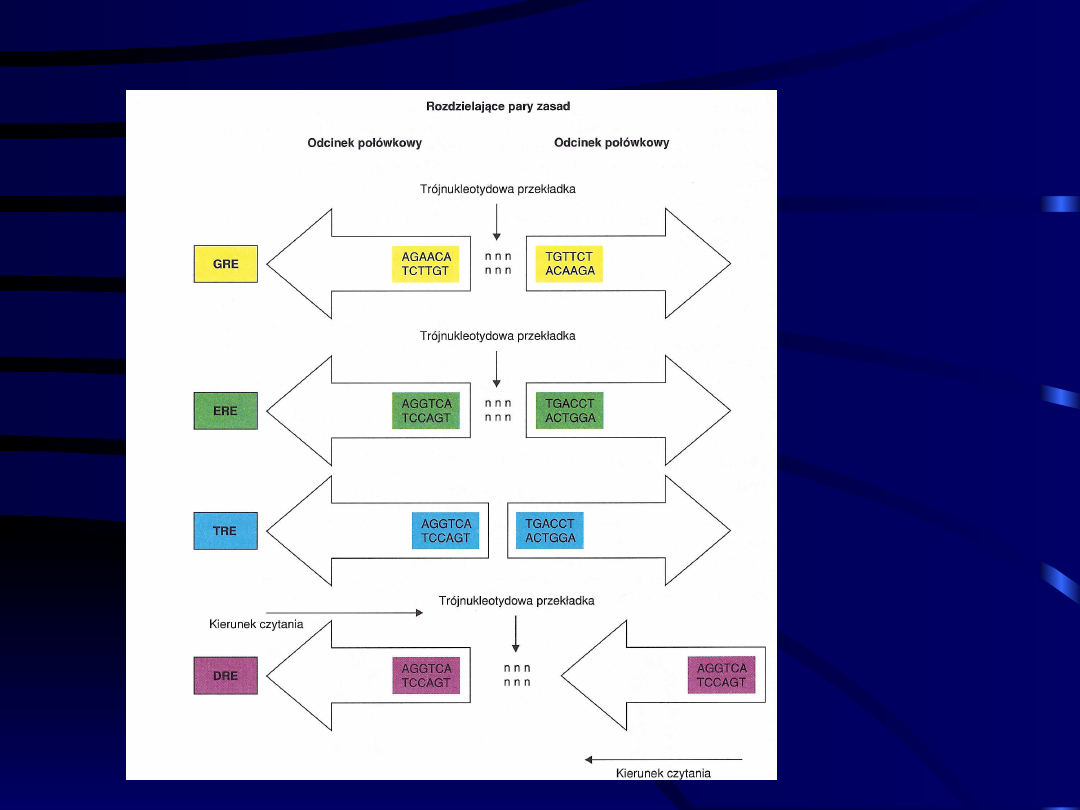
Budowa elementu odpowiedzi
hormonalnej
GRE –
Glucocorticoid
Hormone
Response
Element
ERE – Estradiol
Hormone
Response
Element
TRE – Thyroid
Hormone
Response
Element
DRE – sekwencja
wiążąca
receptory
„sieroce”
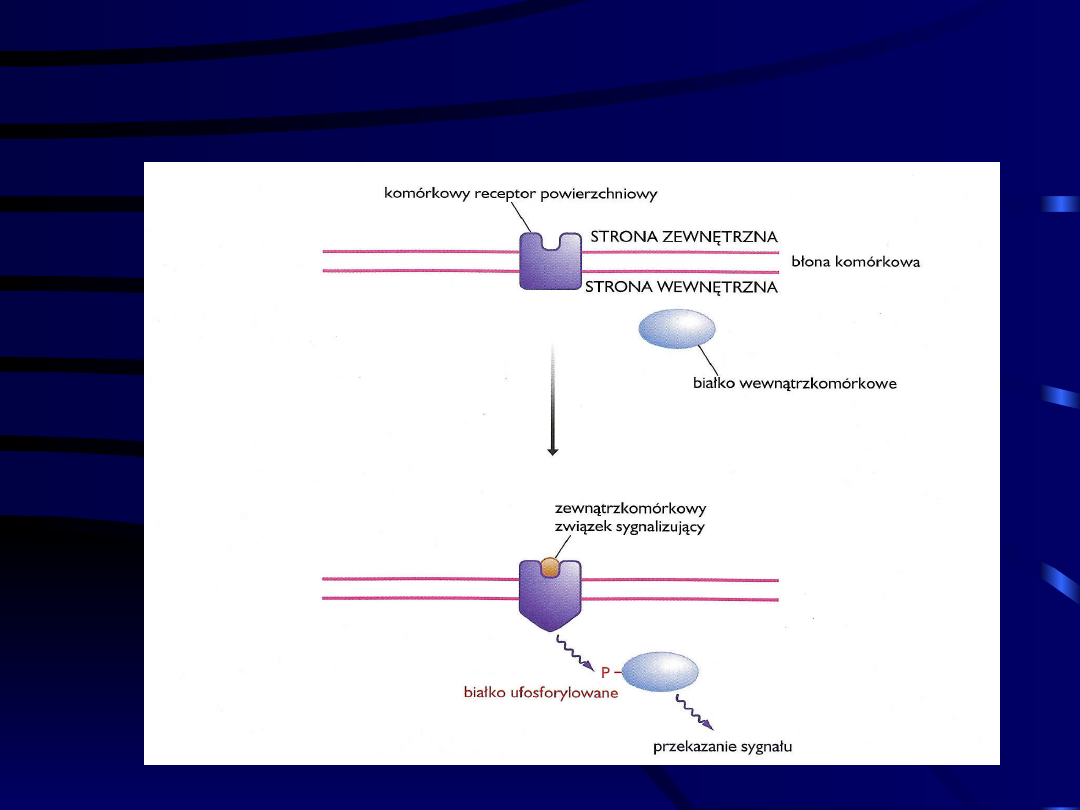
Rola komórkowego receptora
powierzchniowego w przekazywaniu
sygnałów
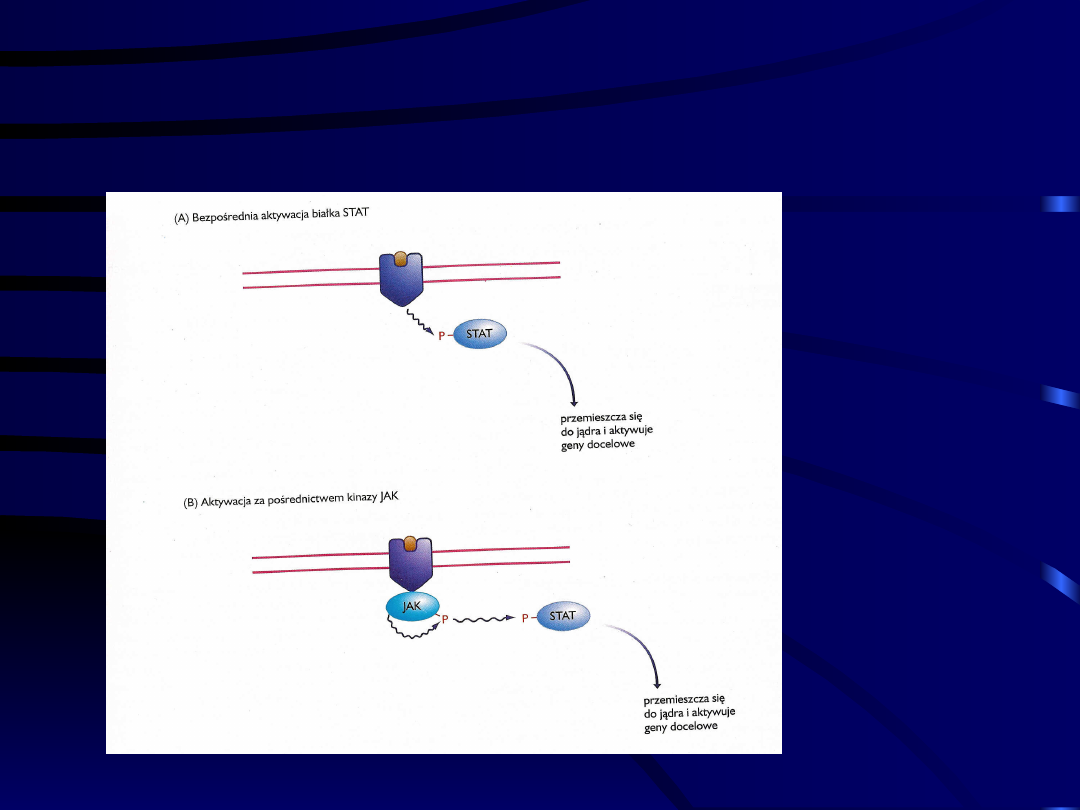
Przekazywanie sygnału z
udziałem białek STAT
A –
receptor
należy do
rodziny
kinaz
tyrozynowy
ch
B –
receptor w
połączeniu
z kinazą
tyrozynową

ZMIENNOŚĆ I MUTACJE

ZMIENNOŚĆ
Zmienność-występowanie dziedzicznych
lub niedziedzicznych różnic:
▪ zmienność wewnątrzosobnicza -
pomiędzy komórkami danego organizmu
▪ zmienność osobnicza - pomiędzy
osobnikami należącymi do tej samej
populacji ,
▪ zmienność grupowa - pomiędzy
populacjami .

ZMIENNOŚĆ
1. Mutacyjna
-
mutacje genowe
(tranzycje, transwersje,
delecje, insercje, inwersje)
-
mutacje chromosomowe
:
- strukturalne (inwersje, translokacje,
duplikacje,
delecje, izochromosomy, chromosomy
koliste)
- liczbowe-aneuploidie (monosomie,
nullisomie,
trisomie)
-
euploidie
(genomowe):
- autopoliploidie (triploidie, tetraploidie,
itd.)
- allopoloploidie (amfiploidie)

2. Rekombinacyjna
(rekombinacja
homologiczna,
rekombinacja zlokalizowana, rekombinacja
transpozycyjna)
3. Fluktuacyjna
(ciągła)
4. Alternatywna
(skokowa)
ZMIENNOŚĆ

• Zmienność fluktuacyjna (ciągła) – daje się
określić w jednostkach miary (wzrost, masa
ciała, IQ, liczba krwinek, pigmentacja włosów i
skóry)
• Zmienność alternatywna (skokowa, nieciągła) –
np. układ grupowy Rh
ZMIENNOŚĆ FENOTYPOWA

REKOMBINACJE
– procesy wymiany
fragmentów DNA między chromosomami
homologicznymi lub dwuniciowymi
helisami DNA. Rekombinacje nie prowadzą do
wytworzenia nowych alleli genów, ale do
ciągłego ich przetasowywania i powstawania
różnych kombinacji genotypów.
ZMIENNOŚĆ DZIEDZICZNA
(REKOMBINACJE I MUTACJE)

•
Zjawisko prowadzące do rekombinacji genetycznej
sprzężonych genów polegające na wymianie
odpowiadających sobie położeniem odcinków
między homologicznymi grupami sprzężeń
(chromosomami)
•
Zachodzi w wyniku symetrycznych pęknięć i
ponownego połączenia się odcinków chromatyd
chromosomów homologicznych po ich wzajemnej
wymianie w pachytenie i/lub diplotenie mejozy
•
Występuje również somatyczny , czyli mitotyczny
crossing over , który polega na wymianie
siostrzanych chromatyd w obrębie jednego
chromosomu. Występuje rzadko i z różną
częstością w chromosomach .
REKOMBINACJA HOMOLOGICZNA
(crossing over)
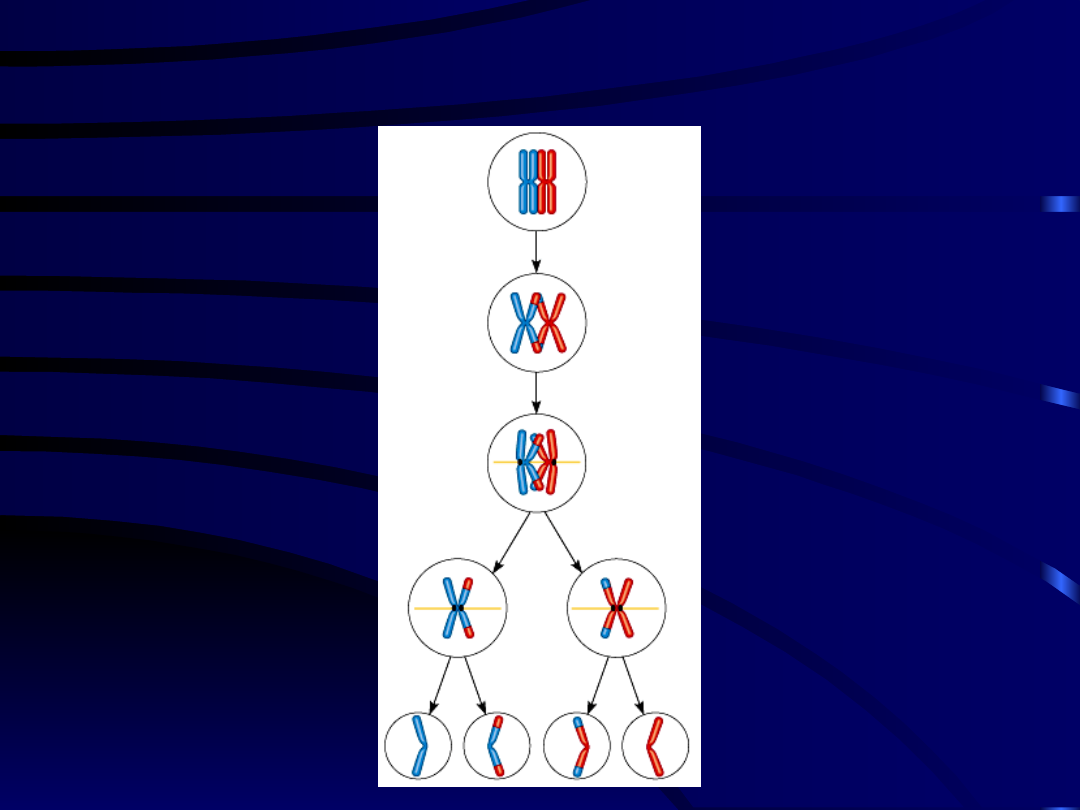
REKOMBINACJA HOMOLOGICZNA
(crossing over)

• Dotyczy wymiany niehomologicznych, ale
specyficznych fragmentów
• Przykładem tego typu rekombinacji jest
tworzenie przeciwciał i receptorów limfocytów
T
• W komórkach bakteryjnych rekombinacja
zlokalizowana zachodzi podczas
wbudowywania plazmidów do genomu bakterii.
REKOMBINACJA ZLOKALIZOWANA

• Zachodzi podczas wbudowywania transpozonów w
nowe miejsce genomu
• Transpozony są to ruchome elementy genomu
zawierające geny kodujące transpozazę (enzym o
aktywności nukleazy)
• Najprostszym przykładem transpozonów są
sekwencje insercyjne (IS)
• W rekombinacji transpozycyjnej wymagane jest
istnienie specyficznej sekwencji DNA w miejscu
akceptorowym dla IS. Sekwencja ta ulega duplikacji,
a IS wbudowywana jest między podwojony fragment.
• Oprócz genów transpozazy transpozon może
zawierać inne geny – jest to tzw. transpozon złożony
REKOMBINACJA TRANSPOZYCYJNA

• Termin „
mutacja
” wprowadził H. De Vries w
1909r
• Mutacja
jest to zmiana dziedziczna powstająca
na skutek zmiany genu w jego nowy allel
(mutacja genowa), zmiany struktury
chromosomu (mutacja chromosomowa),
zmiany liczby chromosomów (mutacja
liczbowa), bądź zwielokrotnienie haploidalnego
zestawu chromosomów (mutacja genomowa)
MUTACJE

• każda zmiana sekwencji nukleotydów w
obrębie genu , inna od sekwencji genu
wyjściowego (powstaje nowy allel genu)
• dotyczy genów kodujących białka
strukturalne i enzymatyczne
MUTACJE GENOWE

• Tranzycja
– zamiana jednej zasady purynowej
na drugą purynową , lub pirymidynowej na
inną pirymidynową.
• Traswersja
– zamiana zasady purynowej na
pirymidynową lub odwrotnie.
• Delecja
– wypadnięcie pojedynczej lub większej
liczby par nukleotydów z danego genu.
• Insercja
– wstawienie pojedynczej lub większej
liczby par nukleotydów do danego genu .
MUTACJE GENOWE

• Mutacje nonsensowne
i zmiany
odczytu prowadzą zwykle do
całkowitej utraty aktywności enzymu
(przerwanie syntezy).
• Mutacje zmiany sensu
prowadzą do
syntezy enzymów o zmienionych
właściwościach chemicznych i
fizycznych .
• Mutacje nieme
– nowy kodon jest
synonimiczny z kodonem przed
mutacją.
MUTACJE GENOWE

1.
Aberracje chromosomowe strukturalne
• Inwersja
– (odwrócenie o 180
o
) na skutek
pęknięć jednego chromosomu i połączenia
wolnych jego końców w odwrotnym
kierunku .
→ paracentryczna obejmuje odcinek
chromosomu bez
centromeru
→ perycentryczna obejmuje fragment z
centromerem .
ABERRACJE CHROMOSOMOWE

• Translokacja
– przemieszczenie się fragmentu chromosomu
w inne miejsce tego samego lub innego chromosomu.
→ T. intrachromosomalna (wewnętrzna) między homologicznymi
chromosomami
→ T. interchromosomalna (zewnętrzna) między chromosomami
niehomologicznymi
→ T. wymienna (wzajemna) wzajemna wymiana odcinków między
chromosomami niehomologicznymi, całkowita liczba
chromosomów pozostaje nie zmieniona, a dwa spośród nich
mają
nieprawidłowe kształty .
Traspozycja
– translokacja polegająca na przeniesieniu
odcinka z
jednego chromosomu do drugiego .
ABERRACJE CHROMOSOMOWE
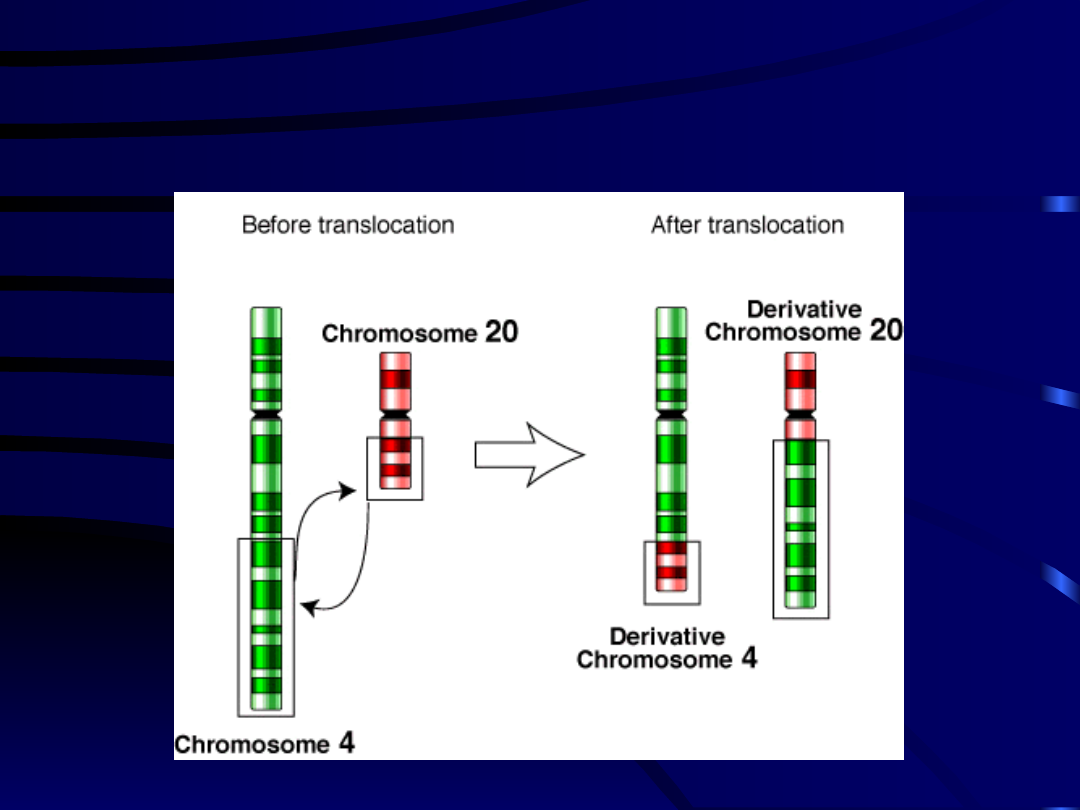
TRANSLOKACJA WYMIENNA
(WZAJEMNA)

• TRANSLOKACJA ROBERTSONOWSKA:
Łączą się całe lub prawie całe ramiona
długie dwóch różnych chromosomów
(połączenia centryczne). Miejscem połączenia
jest rejon centromeru. Dochodzi do utraty
funkcjonalnie nieistotnej części materiału
genetycznego ( ramiona krótkie ) .
Translokacja robertsonowska zrównoważona
-
nie zmienia się ilość materiału genetycznego.
Brak objawów fenotypowych.
Translokacja robertsonowska
niezrównoważona
-ilość materiału genetycznego
powiększa się. Fenotypowe ujawnienie choroby
ABERRACJE CHROMOSOMOWE
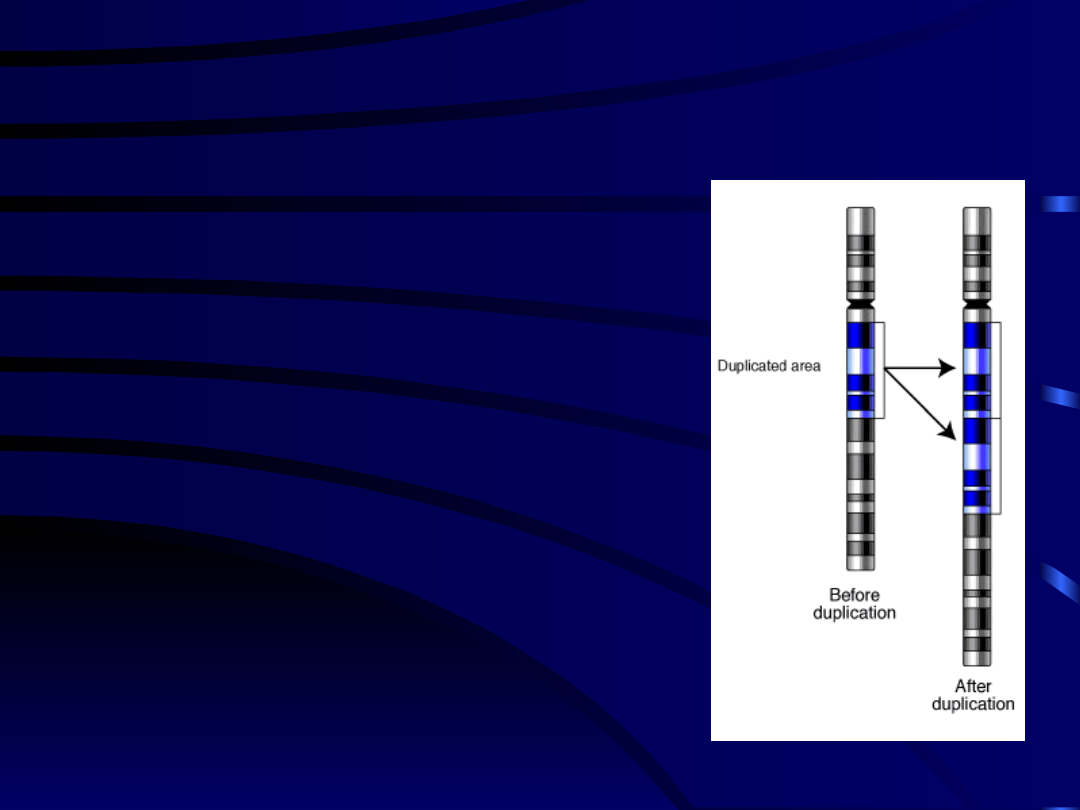
• Duplikacja
– podwojenie
tych samych odcinków
chromosomów (podwojenie
kopii genów). Podwojone
fragmenty mogą występować
jako bezpośrednie
powtórzenia (proste
powtórzenia tandemowe) lub
jako odwrócone względem
siebie powtórzenia
fragmentów chromosomów.
ABERRACJE CHROMOSOMOWE
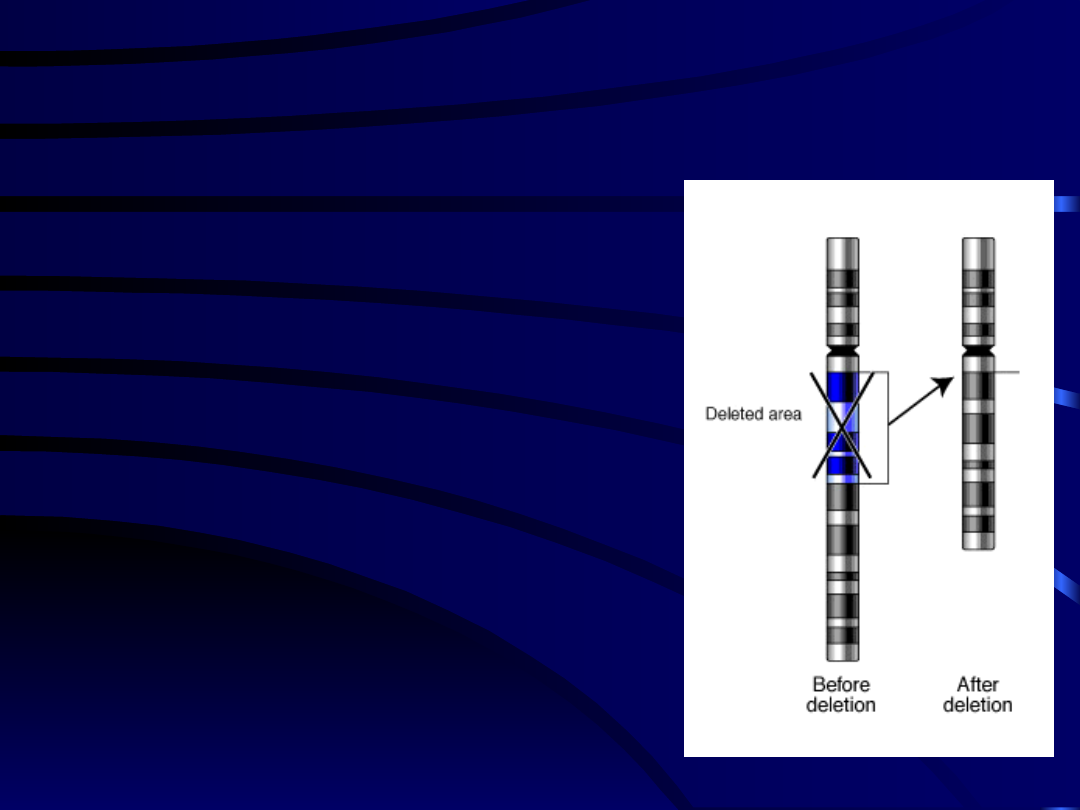
• Delecja (deficjencja)
–
utrata odcinka
chromosomu.
→ d. terminalna obejmuje
część
dystalną chromosomu
→ d. interstycjalna obejmuje
fragment środkowy
ABERRACJE CHROMOSOMOWE
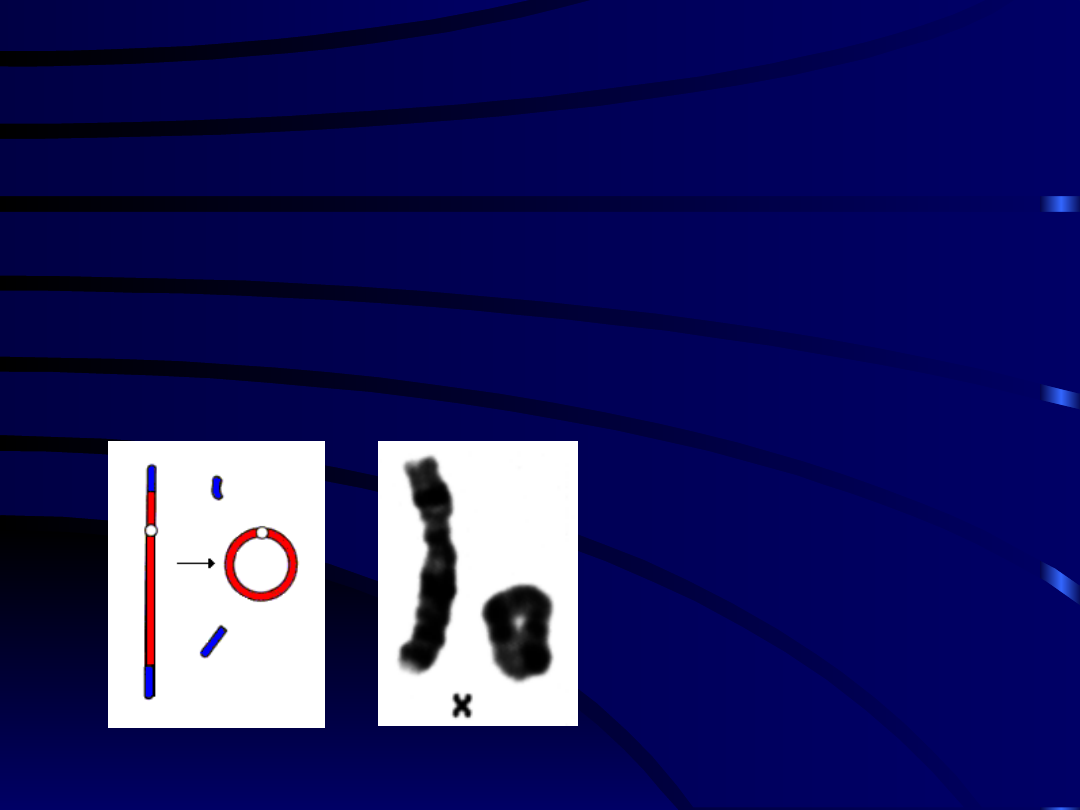
• Chromosom kolisty
– powstaje w wyniku
pęknięcia, a następnie połączenia końców
chromosomu.
U człowieka chromosomy koliste powstają
najczęściej z chromosomów 4 , 13 , 18 pary
oraz chromosomu X .
ABERRACJE CHROMOSOMOWE

• Izochromosom
– powstaje w
wyniku nieprawidłowego ,
poprzecznego podziału
centromeru chromosomu
metafazowego. Składa się on
tylko z połączonych ramion
długich lub krótkich. Powstanie
izochromosomów powoduje
ubytek genów zawartych w
utraconych ramionach i
podwojenie ich liczby w
ramionach , które utworzyły
chromosom. Powstają zarówno z
autosomów jak i chromosomu X .
ABERRACJE CHROMOSOMOWE

2. Aberracje liczbowe
• Aneuploidie
– powstają w wyniku zwiększenia
lub zmniejszenia diploidalnej liczby chromosomów
o pojedyncze chromosomy .
Zapis
2n + a
lub
2n – a
, gdzie
n
to haploidalna
liczba chromosomów ,
a
to liczba chromosomów
podlegających zmianom ilościowym .
Powstawanie uniparentalnej disomii (UPD)
- polega na obecności u diploidalnego potomka
pary
chromosomów pochodzących tylko od jednego
rodzica .
ABERRACJE CHROMOSOMOWE
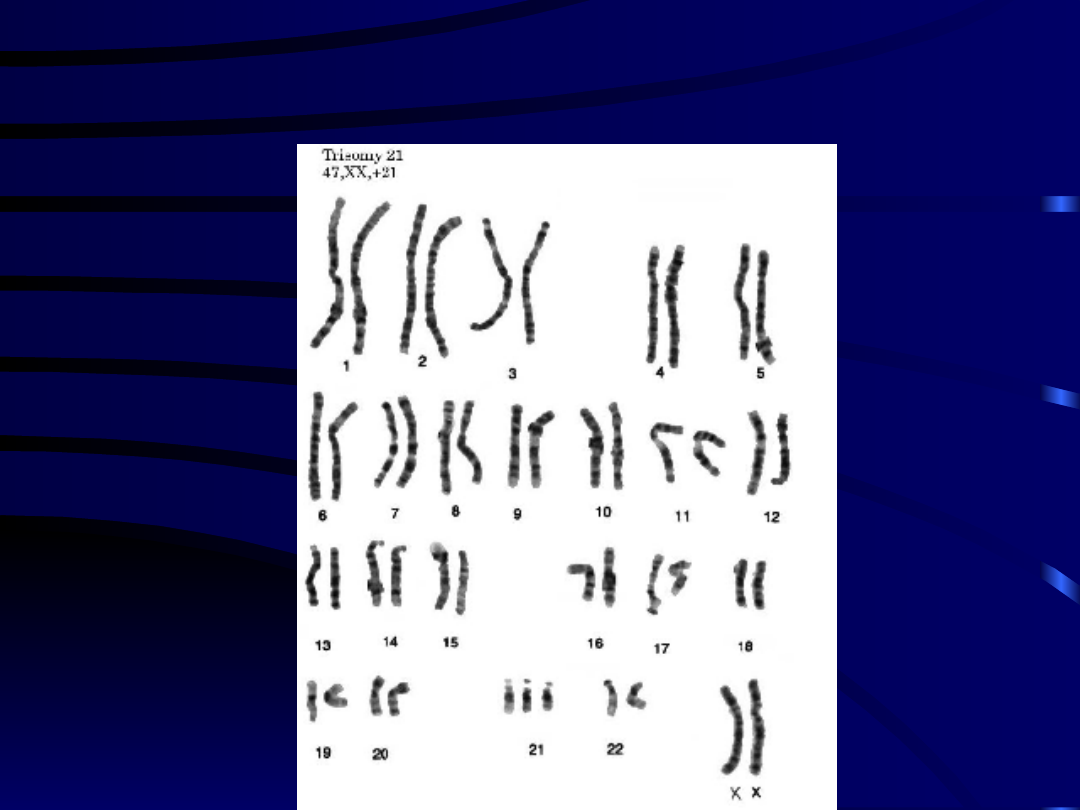
TRISOMIA 21 PARY CHROMOSOMÓW
47,XX,+21 (ZESPÓŁ DOWNA)

• Euploidie
– zwielokrotnienie całego podstawowego zespołu
chromosomów. Zapis 3n, 4n, 5n, itd.
→
autopoliploidie
- garnitur chromosomów jest
zwielokrotniony
o ten sam zestaw chromosomów.
Autopoliploidie są u człowieka letalne i prowadzą do
poronień.
np. AABB(2n) + AABB(2n) = AAAABBBB(4n)
→
allopoliploidie (amfiploidie)
zwielokrotnienie
niehomologicznych
zespołów chromosomów . Powstają najczęściej na skutek
podwojenia liczby chromosomów u mieszańców
międzygatunkowych. Ten typ aberracji nie występuje u
człowieka .
np. AABB(2n) + CCDD(2n) = AABBCCDD(4n)
ABERRACJE CHROMOSOMOWE

Mutageny
to czynniki indukujące powstawanie
mutacji znacznie ponad poziom mutacji
spontanicznych.
Mutageny mogą powodować:
• Transformację nowotworową (kancerogeneza)
• Wady rozwojowe (teratogeneza)
• Śmierć komórek (całego organizmu)
Czynniki mutagenne podzielono na:
• Fizyczne (np. UVA, UVB, promieniowanie X)
• Chemiczne (np. niektóre leki, środki konserwujące i
inne)
• Biologiczne (wirusy brodawczaka ludzkiego i inne)
CZYNNIKI MUTAGENNE

• uszkodzenia DNA, przed ich naprawą, są
rozpoznawane przez enzymy reparacyjne:
- polimerazy DNA: jądrowe (α, β, δ, ε)i
polimeraza mitochondrialna (γ)
- ligazy DNA
- glikozylazy DNA
- endonukleazy apurynowe/apirymidynowe (5’-
AP i 3’-AP)
- białka pomocnicze
- fotoliazy DNA
- metyotransferaza o
6
-metyloguanina-DNA
(MGMT)
MECHANIZMY NAPRAWY DNA

• Naprawa DNA może być kompletna i niekompletna
• W przypadku braku możliwości naprawy DNA
komórka powinna ulec programowanej śmierci
komórki-apoptozie
• Procesy naprawy zachodzą w okresie
przedreplikacyjnym, podczas replikacji lub w
okresie poreplikacyjnym
• Szybkość naprawy chromatyny aktywnej
transkrypcyjnie jest większa niż nieaktywnej
chromatyny
MECHANIZMY NAPRAWY DNA

-
usuwanie błędnie sparowanej zasady
- naprawa przez wycinanie zasad
azotowych
- naprawa przez wycinanie
nukleotydów
- naprawa rekombinacyjna
- odpowiedź SOS
MECHANIZMY NAPRAWY DNA
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
- Slide 44
- Slide 45
- Slide 46
- Slide 47
- Slide 48
- Slide 49
- Slide 50
- Slide 51
- Slide 52
- Slide 53
- Slide 54
- Slide 55
- Slide 56
- Slide 57
- Slide 58
- Slide 59
- Slide 60
- Slide 61
- Slide 62
- Slide 63
- Slide 64
- Slide 65
- Slide 66
- Slide 67
- Slide 68
- Slide 69
- Slide 70
- Slide 71
- Slide 72
- Slide 73
- Slide 74
- Slide 75
- Slide 76
- Slide 77
- Slide 78
- Slide 79
- Slide 80
- Slide 81
- Slide 82
- Slide 83
- Slide 84
- Slide 85
- Slide 86
- Slide 87
- Slide 88
Wyszukiwarka
Podobne podstrony:
2 Regulacja funkcji genów Zmienność i mutacje
Genetyka regulacja funkcji genow
10 WYK X Regulacja funkcji genów u bakterii1id 10654 ppt
10 WYK X Regulacja funkcji genów u?kterii1
Regulacja funkcji genów pod wpływem temperatury
Regulacja funkcji genów pod wplywem temperatury
Regulacja funkcji genów pod wplywem temperatury 1
Genetyka regulacja funkcji genow
4 6 Różniczki funkcji dwóch zmiennych
5 Rachunek różniczkowy funkcji jednej zmiennej
4 pochodna funkcji jednej zmiennej
ZMIENNOSC I MUTACJE, fizjoterapia, biologia medyczna
C 04,5 Rachunek różniczkowy funkcji wielu zmiennych
10 Pochodna funkcji jednej zmiennej
Funkcja jednej zmiennej ciagi
funkcje wielu zmiennych UWM id Nieznany
10 Funkcje wielu zmiennych
1 funkcje dwoch zmiennych
więcej podobnych podstron