
IDENTYFIKOWANIE
IDENTYFIKOWANIE
POLIMORFIZMÓW
POLIMORFIZMÓW
GENETYCZNYCH
GENETYCZNYCH
MAGDALENA SĘDEK

PODSTAWOWE METODY
PODSTAWOWE METODY
UŻYWANE W BIOLOGII
UŻYWANE W BIOLOGII
MOLEKULARNEJ:
MOLEKULARNEJ:
• ELEKTROFOREZA
• PCR
• SEKWENCJONOWANIE

ENZYMY RESTRYKCYJNE
ENZYMY RESTRYKCYJNE
(ENDONUKLEAZY)
(ENDONUKLEAZY)
• W biologii molekularnej wykorzystuje
się enzymy restrykcyjne typu II
• Tną one DNA w ściśle określonych
miejscach
• Rozpoznają miejsca kilku nukleotydowe
• Mogą dawać lepkie albo tępe końce
• Izolowane są z różnych gatunków
drobnoustrojów od których biorą swoje
nazwy np. EcoRI z Escherichia coli
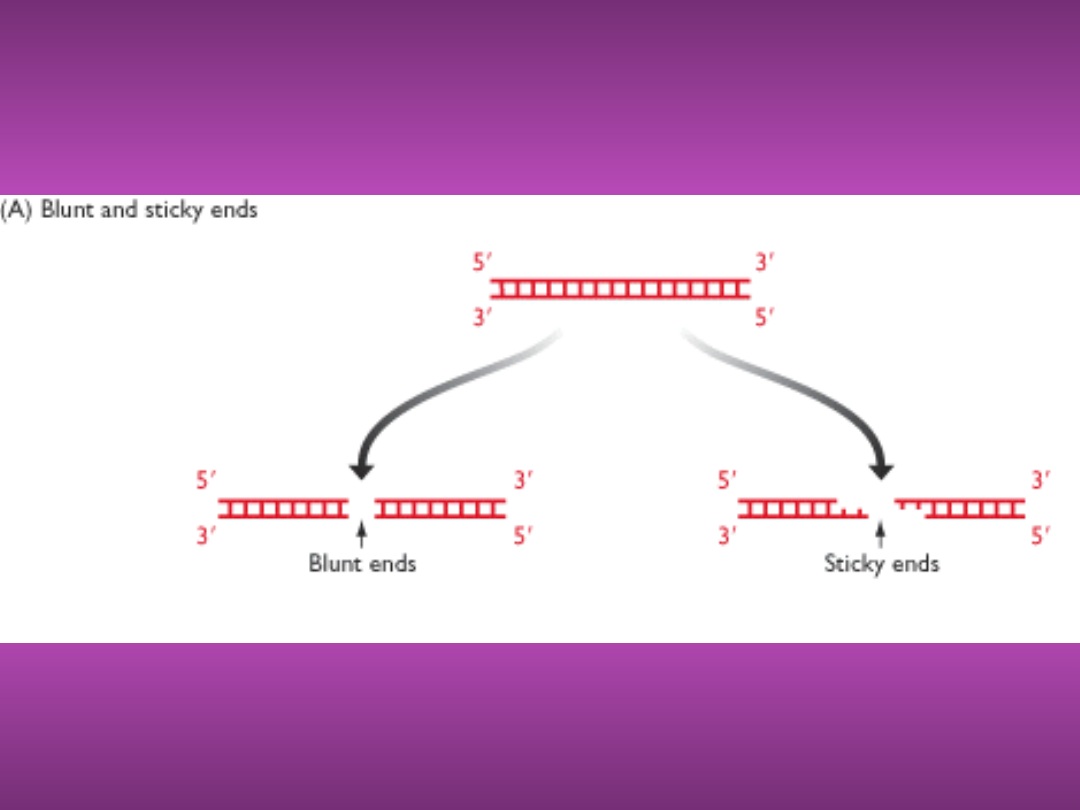
LEPKIE I TĘPE KOŃCE
LEPKIE I TĘPE KOŃCE
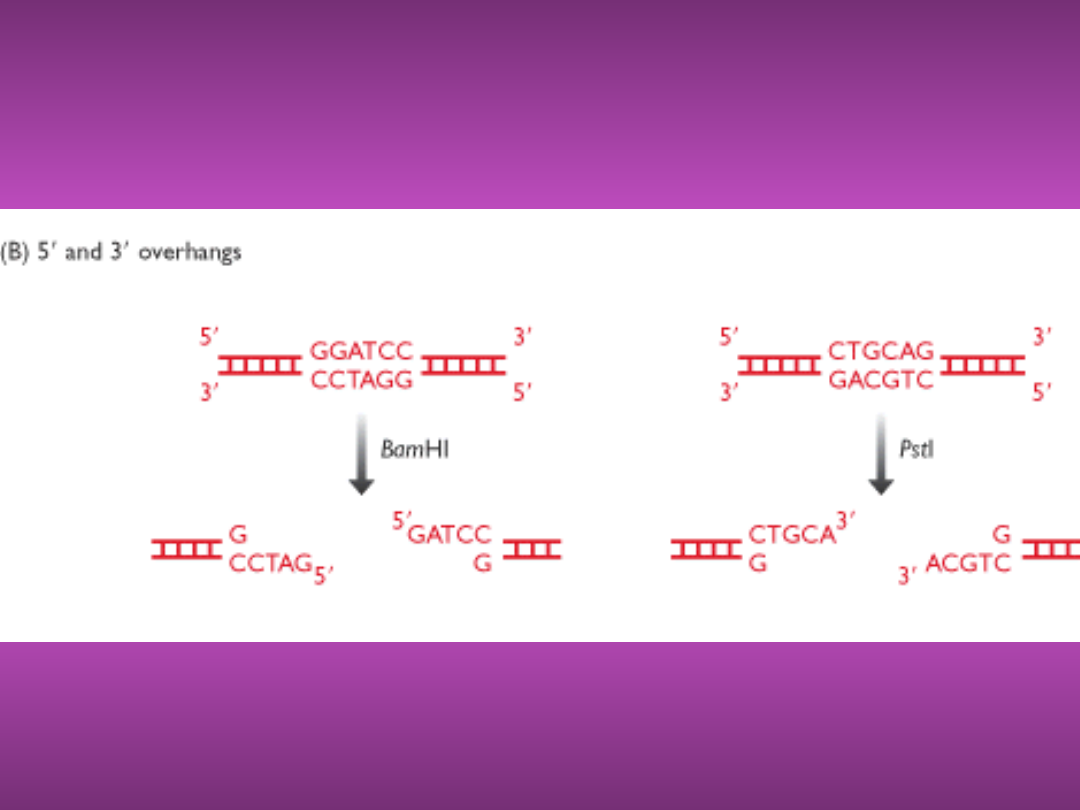
LEPKIE KOŃCE -
LEPKIE KOŃCE -
PRZYKŁAD
PRZYKŁAD
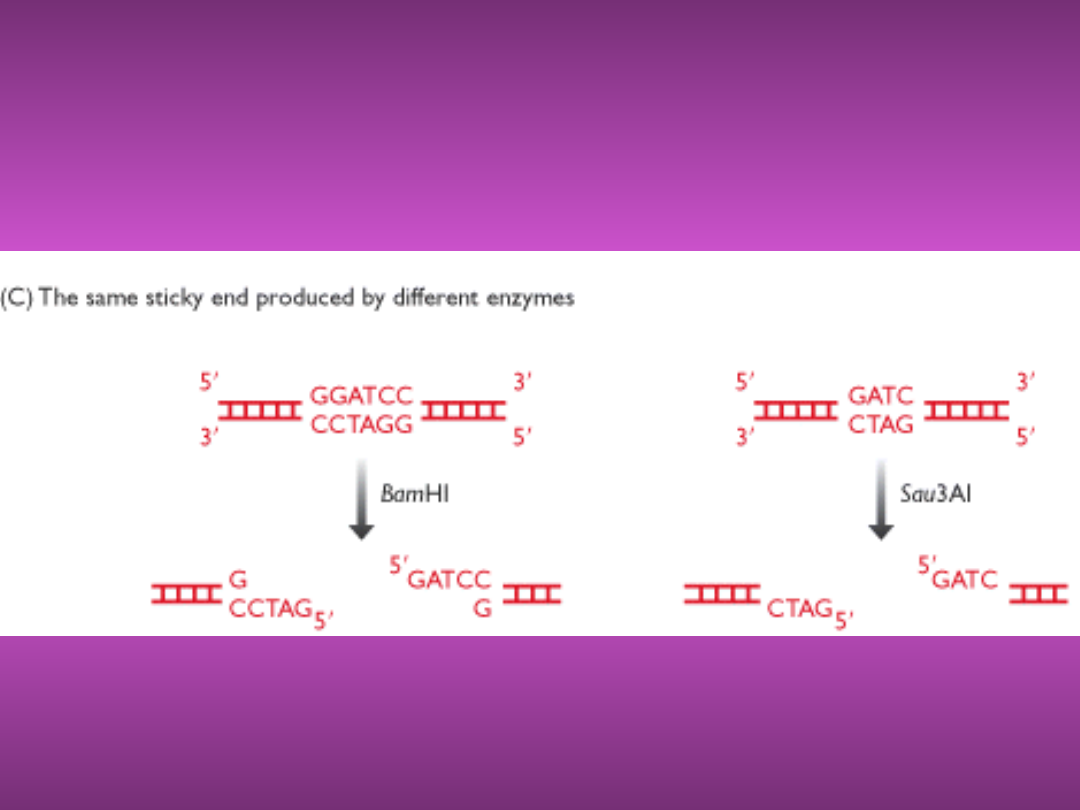
LEPKIE KOŃCE -
LEPKIE KOŃCE -
PRZYKŁAD
PRZYKŁAD
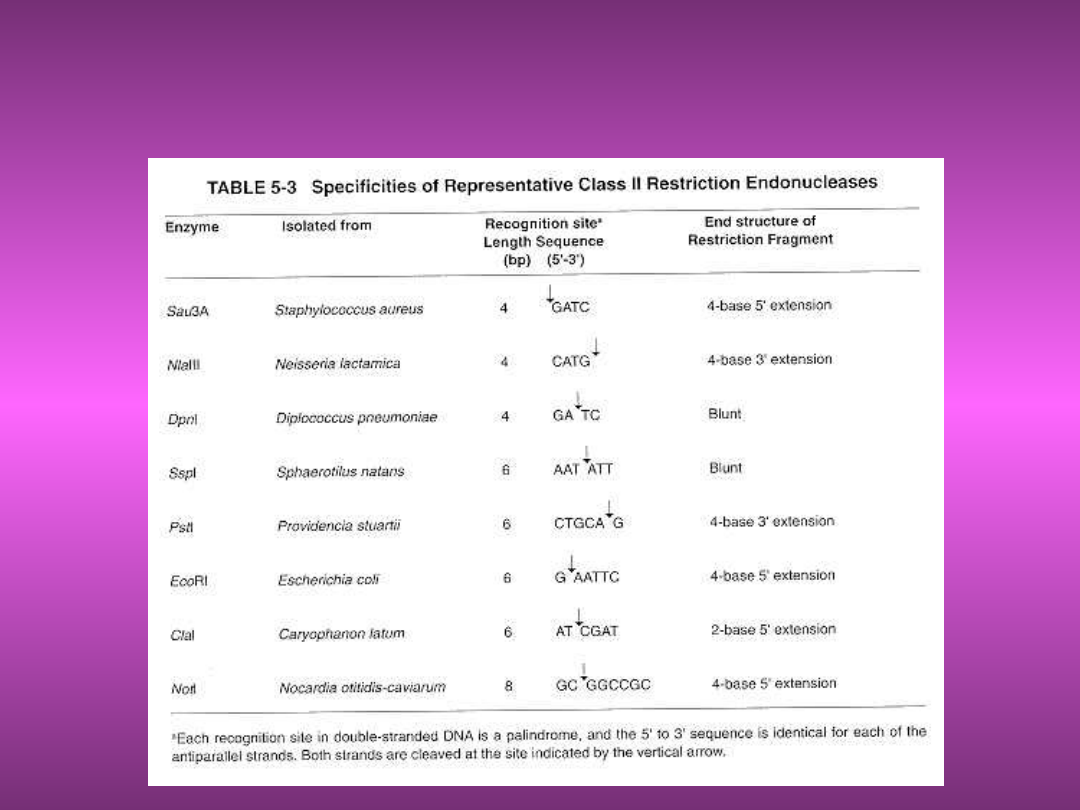
PRZYKŁADY ENZYMÓW
PRZYKŁADY ENZYMÓW
RESTRYKCYJNYCH
RESTRYKCYJNYCH

ELEKTROFOREZA W
ELEKTROFOREZA W
ŻELU
ŻELU
• Metoda polegająca na rozdzieleniu
kwasów nukleinowych (DNA, RNA) lub
białek w żelu w zależności od ich
wielkości, ładunku, konformacji itp.
• Żel zawiera pory przez które cząsteczki
migrują pod wpływem pola
elektrycznego

ELEKTROFOREZA
ELEKTROFOREZA
• Rozdział zachodzi w żelu:
• Agarozowym – zawiera duże pory,
używany do rozdziału dużych
cząsteczek, głównie DNA ale też RNA,
metoda szybka
• Poliakrylamidowym – w zależności od
stężenia możemy uzyskać bardzo duże
lub bardzo małe pory, głównie używany
do rozdziału białek ale też DNA, metoda
wolniejsza ale bardziej dokładna

ELEKTROFOREZA DNA
ELEKTROFOREZA DNA
• DNA posiada ładunek ujemny i migruje od
anody (-) do katody (+)
• Duże cząsteczki (dłuższe) migrują wolniej
a mniejsze (krótsze) szybciej
• DNA jest bezbarwny, aby go zobaczyć
musimy dodać do żelu bromku etydyny,
który interkaluje do DNA
• Bromek etydyny fluoryzuje pod wpływem
promieniowania UV

AGAROZA – POLIMER
AGAROZA – POLIMER
WĘGLOWODANOWY IZOLOWANY ZE
WĘGLOWODANOWY IZOLOWANY ZE
ŚCIANY KOMÓRKOWEJ KRASNOROSTÓW
ŚCIANY KOMÓRKOWEJ KRASNOROSTÓW

1.
1.
Odważamy odpowiednią
Odważamy odpowiednią
ilość agarozy
ilość agarozy

2. Dodajemy odpowiednią
2. Dodajemy odpowiednią
ilość buforu
ilość buforu

3. Całość podgrzewamy aby
3. Całość podgrzewamy aby
rozpuścić agarozę
rozpuścić agarozę

4. Przygotowujemy aparat
4. Przygotowujemy aparat
do wylania żelu
do wylania żelu

5. Wylewamy żel
5. Wylewamy żel

6. Czekamy około pół
6. Czekamy około pół
godziny aż żel stężeje
godziny aż żel stężeje

6. Żel umieszczamy w
6. Żel umieszczamy w
aparacie i zalewamy
aparacie i zalewamy
buforem
buforem
7. Przygotowujemy próbkę
7. Przygotowujemy próbkę
do nałożenia na żel
do nałożenia na żel

8. Nakładamy próbkę na
8. Nakładamy próbkę na
żel
żel

9. Podłączamy aparat do
9. Podłączamy aparat do
źródła prądu
źródła prądu

10. Czekamy około godziny
10. Czekamy około godziny
aż żel się rozwinie
aż żel się rozwinie

11. Umieszczamy żel pod
11. Umieszczamy żel pod
źródłem UV
źródłem UV
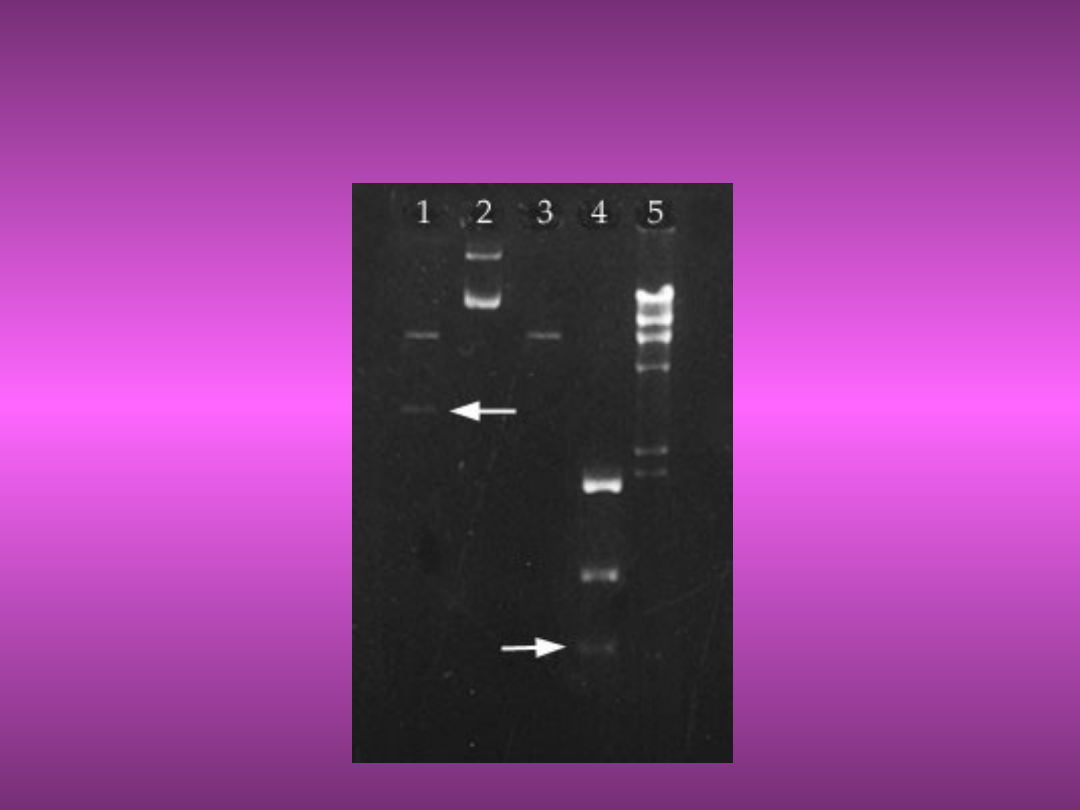
12. Robimy zdjęcie i
12. Robimy zdjęcie i
analizujemy żel
analizujemy żel

PCR
PCR
(Polymerase Chain
(Polymerase Chain
Reaction) Reakcja
Reaction) Reakcja
Łańcuchowa Polimerazy
Łańcuchowa Polimerazy
•Reakcja polegająca na
amplifikacji (powieleniu)
danego fragmentu DNA

CO POTRZEBUJEMY ABY
CO POTRZEBUJEMY ABY
ZASZŁA REAKCJA PCR?
ZASZŁA REAKCJA PCR?
• Matryca – cząsteczka DNA której
fragment chcemy amplifikować
• dNTPy - Deoksyrybonukleotydy
trójfosforanów (Literki A,C,T,G)
• Startery – krótkie (do 20 bp)
oligonukleotydy
• Termostabilna Polimeraza – zdolna do
pracy w wysokej temperaturze (>70°C)
• Bufor, jony magnezu


POKAZ PCR
POKAZ PCR

ZALETY
ZALETY
• Szybkość
• Czułość
• Specyficzność
• Łatwość w zastosowaniu
• Bardzo szerokie zastosowanie
• Umożliwia pracę na bardzo małych
ilościach DNA - wystarczy jedna
komórka

OGRANICZENIA
OGRANICZENIA
• Musimy mieć informacje o sekwencji
którą klonujemy aby zaprojektować
startery
• ALE: możliwość użycia losowych
starterów
• Możliwość klonowania niewielkich
fragmentów DNA (do kilku tysięcy par
zasad)
• ALE: użycie kombinacji kilku polimeraz
umożliwia klonowanie fragmentów nawet
do 42 kb

OGRANICZENIA METODY
OGRANICZENIA METODY
PCR
PCR
• Niedokładność - polimeraza nie
posiada aktywności egzonukleazy, im
dłuższy fragment tym większe
prawdopodobieństwo błędu
• ALE: nowsze polimerazy posiadają
aktywność egzonukleazy i dużo
rzadziej się mylą

INNE PCR
INNE PCR
•Reverse Transcriptase PCR
– PCR z użyciem odwrotnej
transkryptazy
•Real Time PCR – PCR w
czasie rzeczywistym

SEKWENCJONOWANIE
SEKWENCJONOWANIE
• Technika ustalania kolejności
nukleotydów w sekwencji DNA
• Dzięki elektroforezie w żelu
poliakryloamidowym następuje
rozdział cząsteczek DNA różniących
się jednym nukleotydem
• Dwie metody wprowadzone w
latach 70tych

METODY SEKWENCJONOWANIA
METODY SEKWENCJONOWANIA
METODA CHEMICZNEJ DEGRADACJI DNA
(Maxama i Gilberta)
• Sekwencję DNA określa się działając na
wyznakowany radioaktywnie dwuniciowy DNA
związkami chemicznymi, które rozszczepiają
cząsteczki w specyficznych miejscach
poszczególnych nukleotydów
METODA TERMINACJI ŁAŃCUCHA
(Sangera,dideoksy)
• Sekwencję cząsteczki jednoniciowego DNA określa
się dzięki syntezie komplementarnych łańcuchów a
reakcje są zatrzymywane losowo w pozycjach
określonych nukleotydów
• Automatyczne sekwencjonowanie z
wykorzystaniem dideoksyrybonukleotydów
wyznakowanych fluorescencyjnie

CO POTRZEBUJEMY DO REAKCJI
CO POTRZEBUJEMY DO REAKCJI
SEKWENCJONOWANIA METODĄ
SEKWENCJONOWANIA METODĄ
TERMINACJI ŁAŃCUCHA?
TERMINACJI ŁAŃCUCHA?
• Jednoniciowa matryca – przed reakcją nić
DNA denaturujemy
• Starter
• Polimeraza DNA
• Trifosforany deoksyrybonukleotydów
dNTPy (Literki A, T, C, G)
• Trifosforany dideoksyrybonukleotydów
ddNTPy – pozbawione grupy 3’-
hydroksylowej, niezbędnej do utworzenia
reakcji z następnym nukleotydem

POKAZ
POKAZ
SEKWENCJONOWANIA
SEKWENCJONOWANIA
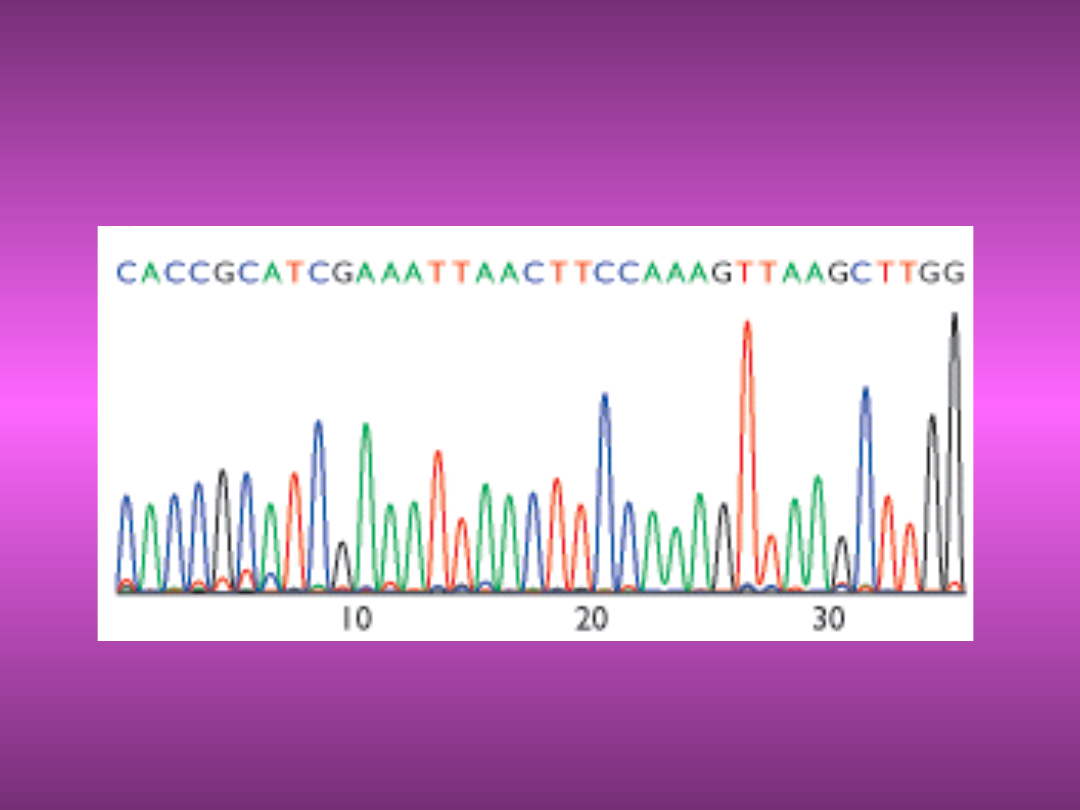
CO OTRZYMUJEMY NA
CO OTRZYMUJEMY NA
KOŃCU?
KOŃCU?
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
Wyszukiwarka
Podobne podstrony:
Polimorfizm genetyczny1.2, BIO, Kurs genetyki molekulranej 26-30.09.2011
Polimorfizm genetyczny oznacza występowanie w populacji 2
Polimorfizm genetyczny transporteów dla leków
Prelekcja 10 - cz 1 - Mutacje genowe (punktowe) i polimorfizmy DNA, ROK 1, Genetyka
Prelekcja 10 - cz 1 - Mutacje genowe (punktowe) i polimorfizmy DNA, Genetyka
ZMIANY NOWOTWOROWE IMPLIKACJE W GENETYCZNEJ IDENTYFIKACJI OSÓB
Identyfikacja genetyczna ofiary postrzału po czterech latach od zgonu
Seminarium3 Inne zaburzenia genetyczne
Genetyka regulacja funkcji genow
Analiza genetyczna w medycynie sądowej
03 PODSTAWY GENETYKI
Prezentacja Genetyka Schizofrenii
Genetyka mendlowska wyklad
04) Kod genetyczny i białka (wykład 4)
więcej podobnych podstron