OPRACOWANIE PYTAŃ Z BIOCHEMII
2.Przepływ informacji genetycznej w komórce.(dział 5 str.96 Stryer- fajny niedługi dział warty przeczytania, ja wybrałam najistotniejsze informacje z tego zakresu)
Wstęp
- bezpośrednią matrycą do syntezy białek jest RNA
- kodony są kolejno odczytywane przez cząstki tRNA, które w procesie biosyntezy białka pełnia rolę adaptera
- istnienie u Eukarytoa intronów i eksonów ma duże znaj\czenie w ewolucji.
Wszystkie komórkowe RNA są syntetyzowane przez polimerazy RNA
-polimeraza RNA zależna od DNA(enzym) potrzebuje do przeprowadzenia syntezy RNA następujących składników:
+ matryca- optymalną matrycą jest dwuniciowy DNA.(RNA nie jest efektywną matrycą podobnie jak hybrydy DNA- RNA
+ aktywowanych prekursorów. Konieczne są wszystkie cztery trifosforany rybonukleozydów: ATP, GTP, UTP i CTP.
+ dwuwartościowego jonu metalu. Efektywne są Mg2+ lub Mn2+. In vivo wymagania te spełnia Mg2+.
POLIMERAZA katalizuje inicjację syntezy i stopniowe wydłużanie łańcucha RNA.
(RNA)n reszt + trisosforan rybonukleozydu = (RNA)n+1 reszt + PPi
Mechanizm
A. Podobieństwa do syntezy DNA
- synteza przebiega w kierunku 5'-3'
- nukleofilowy atak grupy 3'-OH z końca rosnącego łańcucha na a-ortofosforan dołączonego trisosforanu nukleozydu
- hydroliza pirofosforanu napędza reakcję w kierunku wydłużania łańcucha
B. Różnice:
- polimeraza RNA nie wymaga primera
- synteza RNA na matrycy DNA jest konserwatywna, podczas gdy synteza DNA jest semikonserwatywna
- polimeraza nie ma aktywności nukleazowej
Tytuły nagłówków:
- Polimeraza RNA czerpie instrukcje z matrycy DNA.
- Transkrypcja rozpoczyna się w pobliżu miejsc promotowrowych i kończy w miejscach terminacji.
- Transportujący RNA pełni w procesie biosyntezy białka funkcji cząsteczki adapterowej
- Aminokwasy są kodowane przez grupy trzech zasad. Odczyt kodu zaczyna się w określonym punkcie.
- Rozszyfrowanie kodu genetycznego: syntetyczny RNA może służyć jako mRNA.
- Trimukleotydy pobudzają wiązaniespecyficznych transportujących RNA przez rybosomy.
- Do rozszyfrowania kodu genetycznego użyto również kopolimerów o zdefiniowanej sekwencji.
- Głowne cechy kodu genetycznego
- Informacyjny RNA zawiera sygnały start i stop dla syntezy białka.
- Kod genetyczny jest prawie uniwersalny.
- Sekwencja zasaf w genie i sekwencja aminokwasów w jego polipeptydowym produkcie są współliniowe.
- Większość genów eukariontów jest mozaiką intronów i eksonów.
- Eksony kodują domeny białkowe.
- Podczas ewolucji RNA pojawił się prawdopodobnie przed DNA i białkami.
3. Struktura kwasów nukleinowych.
(Harper;1998; 476-487; Struktura i funkcje kwasów nukleinowych)
Genomy część 7
-DNA zawiera informacje genetyczną
-DNA składa się z 4 deoksyrybonukleotydów
a. przeciwlegle nici
b. pasmo matrycowe i pasmo kodujące
c. podwójna helisa
Chemiczną naturę jednostek deoksynukJeo-tydowych DNA — deoksyadenylan, deoksygua-nylan, deoksycytydylan i tymidylan. Te monomeryczne jednostki DNA są utrzymane w postaci polimeru przez mostki 3', 5'-fosfodiestrowe, tworząc w ten sposób pojedyncze pasmo.Treść informacyjna DNA (kod genetyczny) iest zawarta w sekwencji, w której te monomery - deoksyrybonukleotydy purynowe i pirymi-dynowe — są zorganizowane. Cząsteczka polimeru DNA, jak to pokazano, jest polarna; na jednym końcu ma grupę 5'-hydroksylową lub fosforanową, a na drugim grupę 3'-fosforanową lub hydroksylową. Znaczenie tej popularności zostanie wyjaśnione w dalszej części rozdziahi. Ponieważ informacja genetyczna jest zawarta w kolejno ułożonych jednostkach monomerycznych w cząsteczkach polimeru, musi istnieć mechanizm reprodukowania, czyli replikowania tej swoistej informacji mającej bardzo duży stopień wierności.
-Denaturacja (topnienie) DNA jest używana do analizy jego struktury
-cząsteczka DNA ma rowki(wiekszy i mniejszy)
-DNA istniej w postaci zrelaksowanej i silnie skręconej
-DNA służy jako matryca do replikacji i transkrypcji
-RNA różni się od DNA pod względem właściwości chemicznych
a. cukier ryboza
b .zamiast tyminy uracyl
c. występuje jako pasmo pojedyncze
d .ilość guaniny nie musi równać się ilości cytozyny, ani ilość adeniny nie musi równać się ilości uracylu
e .RNA może być hydrolizowany w środowisku alaklicznym.
4. Synteza RNA przez polimerazy RNA zależne od DNA
(Harper,1998, Synteza, przekształcenie i metabolizm RNA 513-531)
RNA jest syntetyzowany na matrycy DNA przez polimerazę RNA
Pasmo, które ulega transkrypcji nazywa się pasmem DNA. Drugie pasmo Dna często określa się jako pasmo kodujące
Informacja na pasmie matrycowym jest odczytywna w kierunku 3'-5'
DNA- zależna polimeraza RNA jest enzymem odpowiedzialnym za polimeryzację rybonukleotydów o sekwencji komplementarnej do psama matrycowego genu
Jednostka transkrypcji jest to region rozciągający się między promotorem a terminatorem.
5.Rodzaje RNA i ich funkcje(HARPER)
Rybosomalny RNA (rRNA) jest syntetyzowany jako duża cząsteczka prekursorowa
W komórkach ssaków 2 większe cząsteczki rRNA i 1 mniejsza cząsteczka rRNA są przepisane jako część pojedynczej dużej cząsteczki prekursorowej. Cząsteczka prekursorowa jest następnie na terenie jąderka przekształcana, w wyniku czego powstają składowe RNA dla podjednostek rybosomów znajdujących się w cytoplazmie. Geny rRNA są umiejscowione w jąderkach komórek ssaków. W każdej komórce znajdują się setki kopii tych genów. Geny rRNA są przepisywane jako jednostki, z których każda koduje (w kierunku 5'-*3') 18S, 5,_8S i 28S rybosomalny RNA. Pierwotny transkrypt jest cząsteczką o masie 45S silnie zmetylowana na terenie jąderka. Prekursorów a cząsteczka 45S, dająca ewentualnie segment 28S, zawiera 65 grup metylowych w rybozach i 5 grup metylowych w zasadach. Metylowane są tylko te części prekursora, które ewentualnie staną się stabilnymi cząsteczkami rRNA. Cząsteczka prekursorowa 45S jest przekształcana nukleolitycznie, ale sygnały do tego przekształcania są zupełnie inne niż te, które znajdują się w hnRNA. Stąd przekształcanie nukleolityczne zachodzi na drodze mechanizmu swoistego i odmiennego od tego, który jest odpowiedzialny za przekształcenie hnRNA do mRNA.
Jak przedstawiono na ryc. 39-11 prawie połowa oryginalnego pierwotnego transkryptu jest degradowana. W czasie przekształcania rRNA zachodzi dalsza metylacja i ewentualnie na terenie jad erek łańcuchy 28S łączą się z białkami rybosomalnymi i tworzą większą podjednostkę rybosomalną o masie 60S. Cząsteczka 5,8S rRNA powstaje także z prekursorowego .Rhl A 45S na terenie jąderka i staje się integralną częścią podjednostki rybosomalnej. Mniejsze rybosomalne podjednostki (40S) tworzą się przez połączenie odpowiednich białek ryboso-malnych z cząsteczką 18S rRNA.
Obecnie odkryto, że w procesie usuwania sekwencji intronowych z pre-mRNA tworzy sie niezwykła cząsteczka RNA i przypominająca. lasso. Okazuje się, że koniec 5' sekwencji wtrąconej, łączy się przez wiązanie fosfodiestrowe 2r—-5' z resztą nukleotydu adenylowego 28-37 nukleotydów w górę (na lewo) od końca 3' sekwencji wtrąconej (intronu). Proces ten i omawiana struktura jest schematycznie przedstawiona na ryc. 39-9.
Wydaje się, że rozwiązano tajemnicę wzajemnych relacji hnRNA i odpowiadającego mu dojrzałego mRNA w komórkach eukariotycz-nych. Cząsteczki hnRNA są pierwotnymi trans-kryptami oraz produktami swoich wczesnych przekształceniu "dodaniu czapeczek i sekwencji poli(A) oraz zabraniu części odpowiadającej intronom, cząsteczki te są transportowane do cytoplazmy jako dojrzałe cząsteczki mRNA.
INFORMACYJNY RNA (mRNA) JEST MODYFIKOWANY NA KOŃCACH 5' i 3'
Jak wspomniano wyżej, cząsteczki mRNA ssaków mają strukturę zwaną czapeczką na końcu 5' i większość z nich ma sekwencję (ogon) poli(A) w końcu 3'. Struktura czapeczki jest dodana do końca 5' nowo przepisywanego prekursora mRNA w jądrze przed transportem cząsteczki mRNA do cytoplazmy. Sekwencja poli(A)(gdy występuje) jest dodana albo w jądrze, albo w cytoplazmie. Wtórne metylacje cząsteczek mRNA w obrębie grup 2'-hydroksylowych i atomu N6 reszt adenylanowych występują po pojawieniu się cząsteczki mRNA w cytoplazmie. Czapeczka 5' transkryptu RNA jest niezbędna do wytworzenia kompleksu rybo nukleoprotei nowego, do reakcji składania, i może być włączona w transport mRNA oraz rozpoczęcie translacji, a także osłania koniec 5' mRNA przed atakiem egzonukleaz działających w kierunku 5'—*3r.
Rola sekwencji poli(A) jest nieznana, ale wydaje się, że polega ona na osłonie końca 3' RNA przed atakiem cgzonukleazy działającej w kierunku 3'-+ 5'. W żadnym przypadku występowanie lub brak sekwencji poli(A) nie determinuje, czy prekursorowa cząsteczka obecna w jądrze pojawi się w cytoplazmie, ponieważ nie wszystkie cząsteczki hnRNA zawierające sekwencję poli{A) wchodzą w skład cytoplazmatycznego mRNA, ani nie wszystkie cytoplazmatyczne cząsteczki mRNA zawierają sekwencję poli(A) (histony są najlepiej zauważalnym przykładem).
Wielkość cząsteczek cytoplazmatycznego mRNA, nawet po usunięciu sekwencji poli(A), jest znacznie większa, często 2—3 razy, od wielkości wymaganej do kodowania swoistych białek, dla których stanowią one matrycę. Dodatkowe nukleotydy występują w regionach nie ulegających translacji (niekodujących) zarówno po stronie 5', jak i 3' regionu kodującego; najdłuższe sekwencje nie ulegające translacji znajdują się zwykle przy końcu 3'. Właściwa rola tych sekwencji nie jest znana, ale są one włączone w przekształcanie RNA, transport, degradację i translację.
Transportujący RNA (tRNA) jest znacznie modyfikowany
Czas teczki _tRNA, jak to opisano w rozdz. 37 i 40,służą jako cząsteczki adaptacyjne do translacji mRNA na sekwencje aminokwasów Cźąsteczki tRNA zawierają liczne modyfikacje zasad podstawowych A, U, G i C. Niektóre" z nich są to proste metylowane pochodne, a niektóre mają przekształcone wiązania glikozydowe. Cząsteczki tRNA u prokariontów i u eukariontów są przepisywane w postaci dużych cząsteczek prekursorowych, zawierających często więcej niż 1 tRNA i wtedy podlegają przekształceniom nukleolitycznym i zmniejszeniu wielkości; proces jest katalizowany przez swoistą klasę rybonukleaz. W dodatku geny niektórych cząsteczek tRNA mają, bardzo blisko części odpowiadającej pętli antykodonowej pojedynczy intron o długości 10-—40 nukleotydów. Te introny w genach tRNA są przepisywane; dlatego też przekształcanie prekursorowych transkryptów wielu cząsteczek tRNA musi obejmować usunięcie intronów i odpowiednie rlp^-nie rpginn^ antykodonowego
tak, aby powstała aktywna cząsteczka adaptacyjna do syntezy białek. Enzymy nukleolity-czne, przekształcające prekursory tRNA, rozpoznają najpewniej 3-wy miarową strukturę, a nie tylko sekwencję liniową RNA. Dzięki temu ten układ enzymatyczny przekształca tylko te cząsteczki, które są zdolne do sfal-dowania w produkty mające właściwości czynnościowe.
Dalsze modyfikacje cząsteczek tRNA obejmują alkilację nukieotydów i dołączenie charak-terystycznej końcówki C-C-A przy końcu 3' cząsteczki. Ta końcówka C-C-A jest miejscem przyłączenia swoistego aminokwasu, który ma wejść w reakcję polimeryzacji podczas syntezy białka. Metylacja prekursorów tRNA u ssaków zachodzi prawdopodobnie w jądrze, podczas gdy o"9cTęcie i dołączenie C-C-A są czynnościami cytoplazmatycznymi, ponieważ końcówki te mają szybszy metabolizm niż same cząsteczki tRNA. W cytoplazmie komórek ssaków są enzymy niezbędne do dołączenia aminokwasów do reszt C-C-A.
snRNA- małocząsteczkowe trwałe RNA- drobne jądrowe sarna są mocno wciągnięte w obróbkę mRNA i regulację genów. Spośród wielu sarna U1, U2, U4, U5 i U6 są wciągnięte w usuwanie intronów i przekształcanie hnRNA w mRNA. U7 snRNA jest wciągnięty w utworzenie prawidłowego końca 3'histonowego mRNA. U4 i U6 snRNA mogą być także potrzebne dla obróbki poli(A).
Pyt. 6
Struktury i wiązania występujące w białkach.
7.Białka metabolizmu podstawowego.
(Harper, Białka: Mioglobina i hemoglobina; str 75)
(harper dzial Substancja pozakomórkowa- kolagen str 831)
mioglobina
hemoglobina
kolagen
Mioglobina jest silnie upakowaną, w przybliżeniu kulistą cząsteczką o wymiarach 4,5 x 3,5 x 2,5 nm. Około 75% łańcucha występuje w formie ośmiu prawoskrętnych helis α o długości 7 - 20 aminokwasów.[2] Poszczególne helisy oznacza się literami od A do H. Aby odnieść się do konkretnego aminokwasu określamy w której helisie się on znajduje (np. F) oraz liczbę wskazującą pozycję od końca N (aminowego) w tej helisie. I tak np. "His F8" odpowiada helisie F i ósmemu aminokwasowi na niej, który okazuje się być histydyną.Grupę prostetyczną mioglobiny stanowi hem. Znajduje się on w zagłębieniu pomiędzy helisami E i F, a w jego centrum jest związane koordynacyjnie żelazo w formie jonu Fe2+. Utlenienie jonu żelaza Fe2+ do Fe3+ powoduje utratę aktywności biologicznej białka.Zewnętrzna część cząsteczki mioglobiny jest polarna, natomiast jej wnętrze jest niepolarne. Dzieje się tak, gdyż reszty aminokwasowe zawierające grupy polarne kierowane są na zewnątrz cząsteczki, a reszty niepolarne kierowane są do środka. Jedynym wyjątkiem od tej reguły są dwie polarne reszty histydyny (F8 i E7), które skierowane są do wnętrza cząsteczki. His F8 określana jest mianem histydyny proksymalnej, a His E7 - dystalnej.
Rodzaje mioglobiny:
oksymioglobina - mioglobina utlenowana, magazynująca tlen;
karboksymioglobina - mioglobina związana z tlenkiem węgla(II). W warunkach fizjologicznych stanowi ok. 1% mioglobiny. Wędzonemu mięsu nadaje charakterystyczny, jasno czerwony kolor.
apomioglobina (apoMb) - mioglobina pozbawiona hemu;
metmioglobina - mioglobina z utlenionym atomem żelaza.
Hemoglobiny "prawidłowe"
HbA (HbA1) (2α2β) - prawidłowa hemoglobina dorosłych
HbA2 (2α2δ) - prawidłowa hemoglobina dorosłych; stanowi około 1,5% - 3% hemoglobiny
HbF (2α2γ) - hemoglobina płodowa; ma większe powinowactwo do tlenu niż HbA, dzięki czemu jest w stanie pobrać tlen z krwi matki przez łożysko i uwolnić go w tkankach płodu. W życiu pozamacicznym jest zastępowana, gdyż słabiej uwalnia tlen w tkankach przy wyższym ciśnieniu parcjalnym tlenu. U dorosłych do 2%
hemoglobiny embrionalne - mają podobne właściwości jak HbF:
Hemoglobina Gower 1 (ξ2ε2)
Hemoglobina Gower 2 (α2ε2)
Hemoglobina Portland (ξ2γ2)
HbA1c - HbA z przyłączoną nieenzymatycznie, trwale cząsteczką glukozy do N-końcowych aminokwasów łańcuchów globiny. Duże stężenie świadczy o proporcjonalnie podwyższonej glikemii, co może pozwolić na określenie średniego poziomu glukozy w surowicy przez okres 2-3 miesięcy, a to ma znaczenie np. w ocenie skuteczności leczenia cukrzycy. Norma stanowi 4-6% ogólnej ilości hemoglobiny. Produkty przejściowe pomiędzy HbA1 a HbA1c stanowią formy HbA1a oraz HbA1b będące zasadami Schiffa, w których glukoza jest przyłączona odwracalnie. Ogólna liczba wszystkich form glikowanej hemoglobiny powinna mieścić się w zakresie 6-8% ogólnej ilości hemoglobiny.
Hemoglobiny nieprawidłowe
HbS -mutacja punktowa - podmiana hydrofilowego kwasu glutaminowego w pozycji A2 (6β) na hydrofobową walinę co powoduje powstanie lepkich miejsc i tworzenia agregatów nieutlenowanej HbS, które zniekształcają erytrocyty prowadząc do niedokrwistości sierpowatokrwinkowej
Hemoglobina typu Bristol - zmiana waliny na kwas asparaginowy w pozycji 67 łańcucha β. Zmiana nie powoduje zaburzenia funkcji
Hemoglobina typu Sydney - zmiana waliny na alaninę w pozycji 67 łańcucha β. Zmiana nie powoduje zaburzenia funkcji
Hemoglobina typu Hikari - zmiana lizyny na asparaginę w pozycji 61 łańcucha β. Zmiana nie powoduje zaburzenia funkcji
Hemoglobina typu Milwaukee - zmiana waliny na kwas glutaminowy w pozycji 67 łańcucha β. Zmiana nie powoduje zaburzenia funkcji
Hemoglobina typu Lepore - (2α2Lepore) - hemoglobina w jednym z typów β-talasemii, wynik delecji genów kodujących łańcuchy β i δ. Ich resztki tworzą gen kodujący łańcuch Lepore.
Kolagen ma nietypowy skład aminokwasów. Zawiera duże ilości glicyny i proliny oraz dwa aminokwasy nie pochodzące bezpośrednio z translacji w rybosomach - hydroksyprolinę i hydroksylizynę, z czego tę pierwszą w dość dużych ilościach. Aminokwasy te są formowane z proliny i lizyny już w gotowym produkcie translacji w procesie enzymatycznym, która wymaga obecności witaminy C. Szczegóły tego procesu wciąż nie są dobrze poznane. To właśnie ten proces wymaga konieczności występowania stałego stężenia witaminy C w organizmie, gdyż zablokowanie syntezy kolagenu skutkuje chorobą zwaną szkorbutem, polegającą na uszkodzeniach skóry, błon śluzowych i wypadaniu zębów.
Inną rzadką cechą kolagenu jest regularność rozmieszczenia aminokwasów, w każdym z jego α-łańcuchów. Łańcuchy te składają się z regularnych triad aminokwasów: Gly-X-Y, gdzie Gly - to glicyna a X i Y to inne aminokwasy. Na ogół X to prolina, zaś Y to hydroksyprolina. Niewiele innych białek wykazuje taką regularność. Regularność ta powoduje, że łańcuchy α mają tendencję do przyjmowania ściśle określonej konformacji, na skutek oddziaływań między sobą. Trzy cząsteczki kolagenu skręcają się spontanicznie w podjednostki zwane tropokolagenem. Tropokolagen ma strukturę potrójnej, ściśle upakowanej helisy, o skoku tylko 0,3 nm (nanometra) w porównaniu ze skokiem 0,36 nm, typowym dla innych białek. Wiązania kowalencyjne i wodorowe tworzone przez hydroksylizynę i hydroksyprolinę odgrywają kluczową rolę w stabilizowaniu helisy kolagenu, a także mają silny wpływ na ostateczny kształt włókien zbudowanych z kolagenu.
8.Degeneracja kodu genetycznego.
Trzy spośród kodonów nie kodują swoistego aminokwasu, nazwano je kodonami nonsensownymi. Przynajmniej 2 z tych nonsensownych kodonów są używane w komórce jako sygnały terminacji. Pozostałych 61 kodonów koduje 20 aminokwasów. Zatem kod genetyczny jest zdegenerowany tj. liczne kodony muszą odpowiadać temu samemu aminokwasowi. Niektóre aminokwasy sa kodowane przez kilka kodonów.
Jednoznaczny, ale zdegenerowany kod może być objaśniony w kategoriach molekularnych. Rozpoznanie swoistych kodonów w mRNA przez adaptacyjne cząsteczki tRNA jest zależne od ich regionu antykodonowego i reguł swoistego parowania zasad. Każda cząsteczka tRNA zawiera swoistą sekwencję komlementarną do kodonu, która nazywa się antykodonem. Dla danego kodonu w mRNA tylko pojedynczy rodzaj cząsteczki tRNA ma odpowiedni antykodon. Ponieważ każda cząsteczka tRNA może być obarczona tylko 1 swoistym aminokwasem, dlatego każdy kodon określa tylko 1 aminokwas. Jednak niektóre cząsteczki tRNA mogą używać antykodonu do rozpoznawania więcej niż 1 kodonu.
Z nielicznymi wyjątkami dany swoisty kodon będzie wprowadzał tylko 1 swoisty aminokwas, chociaż dany swoisty aminokwas może być rozpoznany przez więcej niż 1 kodon.
Pyt. 9
Funkcje biologiczne białek.
<Stryer rozdz. 2 ` Struktura i funkca białek'>
Białka odgrywają decydującą rolę właściwie we wszystkich procesach biologicznych. Znaczenie i szeroki zakres pełnionych przez nich funkcji ilustrują następujące przykłady
Kataliza enzymatyczna - prawie wszystkie reakcje enzymatyczne zachodzące w układach żywych są katalizowane przez swoiste makrocząsteczki, enzymy. Niektóre z nich, jak uwodnienie dwutlenku węgla, to reakcje całkiem proste, inne - bardzo skomplikowane, np. replikacja chromosomu. Enzymy są bardzo silnymi katalizatorami. Na ogół zwiększają szybkość reakcji chemicznej przynajmniej milion razy. Jeśli brak enzymów, przemiany chemiczne in vivo bardzo rzadko zachodzą z zauważalną szybkością. Dotychczas scharakteryzowano kilka tys. enzymów i wiele z nich uzyskano w formie krystalicznej. Prawie wszystkie znane enzymy są białkami. Białka zatem są odpowiedzialne za kierunek przemian w układach biologicznych.
Transport i magazynowanie - transport wielu małych cząsteczek i jonów zachodzi z udziałem swoistych białek. Przykładem jest hemoglobina, przenosząca tlen w krwinkach czerwonych oraz mioglobina, pokrewne białko, odpowiedzialne za transport tlenu w mięśniach. Żelazo jest przenoszone w osoczu krwi przez transferynę, a przechowywan w wątrobie, w kompleksie z innym białkiem - ferrytyną.
Ruch uporządkowany - białka są głównym składnikiem mięśni. Przesunięcie się dwu rodzajów włókien białkowych względem siebie prowadzi do skurczu mięśnia. W skali mikroskopowej ruchy uporządkowane, takie jak przemieszczanie chromosomów podczas mitozy lub poruszanie się plemników za pomocą wici, są także rezultatem działań białkowych układów kurczliwych
Funkcje mechaniczno - strukturalne - dużą elastyczność mięśni oraz tkanki kostnej zapewnia obecność kolagenu, białka fibrylarnego.
Ochrona immunologiczna - białka o dużej swoistości, które rozpoznają substancje obce dla ustroju i łącza się z nimi, to przeciwciała. Obcymi dla ustroju mogą być wirusy, bakterie lub komórki innych organizmów. Białka odgrywają zatem istotną rolę w rozróżnianiu między tym, co własne i obce dla danego organizmu.
Wytwarzanie i przekazywanie impulsów nerwowych - reakcja komórek nerwowych na specyficzne bodźce przebiega z udziałem białek receptorowych. Przykładem jest rodopsyna, białko fotoreceptorowe, występujące w komórkach pręcikowych siatkówki. Cząsteczki receptorów, podatne na pobudzenie przez małe swoiste cząsteczki, np. acetylocholinę, są odpowiedzialne za przenoszenie impulsów w synapsach, czyli połączeniach między komórkami nerwowymi.
Kontrola wzrostu i różnicowanie - kontrola odpowiedniej kolejności ekspresji informacji genetycznej jest zasadniczym warunkiem uporządkowanego wzrostu i różnicowania komórek. Tylko mała część informacji zawartej w genomie ujawnia się w określonym przedziale życia komórki. U bakterii istotnym elementem kontroli są białka represorowe, „wyciszające” określone fragmenty DNA komórki. W organizmach wyższych wzrost i różnicowanie kontrolują białkowe czynniki wzrostu. Na przykład czynnik wzrostu nerwu kieruje tworzenie sieci nerwowej. Aktywność różnych komórek w organizmach wielokomórkowych koordynują hormony. Wiele z nich, jak insulina czy hormon tyreotropowy, są białkami. W istocie białka funkcjonują w komórkach jako czujniki kontrolujące przepływ energii materii.
10.Enzymy uczestniczące w degradacji cząsteczek DNA.
(Stryer 2007 str 245 dział: strategie katalityczne)
Enzymy restrykcyjne, inaczej restryktazy - enzymy z grupy endonukleaz przecinające nić DNA w miejscu wyznaczanym przez specyficzną sekwencję DNA. Rozpoznawana sekwencja z reguły ma charakter symetryczny o długości od 4 do 8 par zasad (pz), choć zdarzają się częste wyjątki. Restryktazy wraz z metylazami DNA stanowią system restrykcji i modyfikacji DNA, który w organizmach prokariotycznych stanowi mechanizm obronny zapobiegający włączeniu DNA bakteriofaga do genomu bakterii. Niespecyficzność enzymów restrykcyjnych w niektórych warunkach nazywa się aktywnością star.
Enzymy restrykcyjne naturalnie występują u bakterii i sinic, stanowiąc element tzw. systemu "restrykcji-modyfikacji". System ten chroni komórkę przed wnikaniem obcego DNA (np. DNA bakteriofagów).
System ten zakłada istnienie w komórce mikroorganizmów dwóch rodzajów enzymów:
restrykcyjna endonukleaza - rozpoznaje specyficzne miejsce cięcia
metylotransferaza - chroni przed cięciem
Modyfikacja taka chroni DNA przed atakiem własnych enzymów restrykcyjnych.
Wyróżnia się następujące typy enzymów restrykcyjnych:
Typ I
wielopodjednostkowe kompleksy enzymatyczne zawierające aktywności metylazy i restryktazy. Przecinają DNA z dala od rozpoznawanej sekwencji, w bliżej nieokreślonym miejscu. Z tego powodu nie mają większego zastosowania praktycznego.
Typ II
przecinają DNA w zdefiniowanym miejscu, w obszarze rozpoznawanej sekwencji lub w jej pobliżu. Składają się z pojedynczych polipeptydów. Rozpoznają sekwencje symetryczne.
Typ IIs
zbliżone do typu II, przecinają z jednej strony rozpoznawanej sekwencji, która jest asymetryczna.
Typ III
duże kompleksy, które wymagają dwóch sekwencji rozpoznawanych w pobliżu siebie. Bez znaczenia praktycznego.
Typ IV
zbliżone do typu II. Zawierają aktywność metylazy i restryktazy w tym samym polipeptydzie. Przecinają DNA w zdefiniowanym obszarze poza sekwencją rozpoznawania. Aktywności metylazy i restryktazy nie mogą działać równocześnie. Enzym "przełącza" się w zależności od substratu.
Enzymy restrykcyjne rozpoznające tę samą sekwencję DNA a przecinające DNA w odmiennych miejscach nazywamy neoschizomerami.
Enzymy restrykcyjne, różniące się sekwencją swojego polipeptydu i pochodzące z odmiennych organizmów, a rozpoznające tę samą sekwencję DNA i przecinające DNA w takim samym miejscu nazywamy izoschizomerami. Interesującą własnością izoschizomerów jest to, że pomimo tej samej specyficzności substratowej, z reguły mają zupełnie odmienną sekwencję i strukturę trzeciorzędową. Wskazuje to na ich osobne pochodzenie ewolucyjne.
11.Rola starterów w syntezie DNA.
-są niezbędne z dwóch powodów:
po pierwsze do rozpoczynanaia syntezy łańcucha komplementarnego na nici prowdzącej
po drugie do rozpoczęcia nieciągłej syntezy wszystkich kolejnych fragmentów DNA na nici opóźnionej
Odcinek RNA syntetyzowany przez prymazę jest starterem umozliwiającym rozpoczęcie syntezy DNA
Wszystkie polimerazy DNA potrzebują do rozpoczęcia syntezy łańcucha polinukleotydowego odc strterowego z wolna grupą 3'-OH.
Pyt. 12
Polimerazy DNA (funkcje)
Pyt. 13
Odwrotne transkryptazy.
Pyt. 14
Ligaza DNA - funkcje
Pyt. 16
Nukleosom.
< Stryer, rodz. 37 - „ Chromosomy i ekspresja genów w kom. eukariot.”>
Nukleosomy - pierwsze stadium kondensacji DNA. Nukleosomy są powtarzającymi się jednostkami strukturalnymi chromatyny. W 1974 roku Roger Kornberg stwierdził, że chromatyna jest zbudowana z powtarzających się jednostek strukturalnych, z których każda zawiera po dwie cząsteczki histonów H2A, H2B, H3 i H4 oraz odcinek DNA o długości 200pz. Powtarzające się jednostki strukturalne chromatyny nazwano nukleosomami. Większość DNA jest owinięta wokół pistonowego rdzenia. Pozostały DNA, nazywany łącznikiem, wiąże sąsiednie nukleosomy i nadaje włóknom chromatyny elastyczność. Tak więc włókna chromatyny są łańcuchami elastycznie połaczonych nukleosomów, przypominającymi sznur koralików.
Rdzeń nukleosomu składa się z odcinka DNA o długości 140par zasad owiniętego wokół pistonowego oktameru.
Zawartość DNA w nukleosomach pochodzących z różnych organizmów waha się w granicach od ok. 160 - 240 par zasad.
W badaniach stwierdzono, że centrum nukleosomu zajmuje tetrametr utworzony z dwóch histonów H3 i dwóch H4, otoczony dwiema parami, czyli dimerami histonów H2A-H2B.
Tworzenie się nukleosomów i struktur wyższego rzęduw chromosomach może zachodzić dzięki elastyczności DNA. DNA kontaktuje się z rdzeniem pistonowym niemal przy każdym skręcie dwuniciowej helisy DNA; kontakty te ograniczają się do jej wewnętrznej powierzchni. Histony nie otaczają DNA i nie wystają poza skrętami superhelisy. Najsilniejsze połączenie jest utworzone między tetrametrem H3-H4 a środkową pętlą super helisy. Odcinki o strukturze helisy alfa, wystające z dimeru H3, ściśle dopasowują się do małych rowków (małych bruzd) w DNA po obu stronach osi podwójnej symetrii nukleosomu.
Histony mogą oddziaływać z większością sekwencji DNA, pełniąc funkcje urządzenia do upakowania DNA.
Dimer H2A-H2B oddziałuje z każdą odsłoniętą stroną tetrameru H3-H4.
Rdzeń nukleosomu jest stabilizowany przez połączenie dimeru H2A-H2B z każdym ostatnim półobrotem superhelisy.
Rozmontowanie nukleosomu, które zachodzi podczas replikacji DNA jest prawdopodobnie inicjowane przez dysocjacje lub odciągnięcie dimeru H2A-H2B.
Histon H1 odgrywa kluczową rolę w tworzeniu następnego poziomu struktury chromosomu, służąc jako most, łączący sąsiednie nukleosomy. Histon H1 jest ulokowany na zewnątrz nukleosomu, gdzie wiąże się z łącznikowym DNA i oddziałuje z podjednostką H2A rdzenia. Jeśli cząsteczka DNA zostanie skrócona z 160 do 140 praz zasad, to H1 zostaje uwolniony z nukleosomu. Stosunek stechiometryczny histonów H1 i nukleosomów wynosi 1:1, w odróżnieniu od pozostałych histonów, dla których stosunek ten wynosi 2:1.
Istnieje kilka typów histonów H1, podczas gdy pozostałe histony są wysoce zachowawcze. Co więcej, bezpośrednio przed mitozą H1 ulega fosforylacji, a jest defosforylowany po mitozie, co sugeruje, że jego zdolnośc do upakowania DNA jest regulowana przez kowalencyjne metody.
Pyt. 17
Stopnie upakowania DNA w chromatynie.
cząsteczka DNA
DNA jest nawinięte na proteiny wiążące DNA. Taki kompleks jest nazwany chromatyną. Wśród protein wiążących wyróżniamy mediatory transkrypcji genowej lub replikacji DNA oraz proteiny tworzące strukturę chromatyny i organizujące DNA tzw. histony. Mamy 5 rodzajw histonów - H1, H2a, H2b, H3 i H4 - które tworzą konstrukcję dla podstawowej jednostki chromatyny jaką jest nukleosom.
Każdy nukleosom zawiera ściśle powiązany pakiet 8 histonów (po 2 z H2A, H2B, H3 i H4) wraz z DNA na nie nawiniętym. Nukleosomy są ze sobą powiązane za pomocą histonu H1. Całośc włókno o długości 30 nm, które jest podstawową jednostką widzianą w interfazie podziałów komórkowych w mikroskopie elektronowym.
Włókno chromatynowe jest następnie zwinięte i tworzą domeny o znaczeniu strukturalnym i funkcjonalnym.
Dalsza kondensacja następuje przez kolejne fazy wijania i pakowania domen chromatynowych oraz interakcje pomiędzy podtrzymującymi proteinami, co prowadzi do powstania chromosomu metafazowego o szerokości ok. 700 nm
< Stryer, rodz. 37 - „ Chromosomy i ekspresja genów w kom. eukariot.”>
< Drewa, rodz 6, `Genom człowieka' >
Struktura chromosomów
Za przestrzenną organizację DNA w chromosomie odpowiedzialne są głównie białka zasadowe, zwane histonami. Wyróżnia się pięć typów histonów: H1, H2A, H2B, H3 i H4. Histony te kompleksie z DNA tworzą nukleonom, czyli podstawową jednostkę upakowania chromatyny. Osiem cząsteczek histonów ( po dwa histony H2A, H2B, H3 i H4) tworzy rdzeń nukleosomu, dookoła którego owinięty jest DNA długości 146 par zasad (1,8 zwoju). Sąsiednie nukleosomy połączone są łącznikowym DNA zawierającym ok. 60 par zasad. Histon 1 ulokowany jest na zewnątrz nukleosomu, gdzie wiąże się z łącznikowym DNA i oddziałuje z podjednostką H2A rdzenia. Odgrywa tym samym istotną rolę w tworzeniu następnego poziomu struktury chromatyny, służąc za most łączący sąsiadujące nukleosomy. Szerokość podwójnej helisy DNA wynosi 2nm, natomiast szerokość upakowania nukleosomowego wynosi 11nm. Łańcuch nukleosomów tworzy następnie helisę solenoidu, Skondensowanie DNA w tej helisie jest 40-krotne, a szerokość solenoidu wynosi 30nm. Pofałdowanie solenoidu prowadzi z kolei do utworzenia struktur wyższego rzędu, w których tworzeniu bierze udział szereg białek niehistonowych.
Najwyższy stopień skondensowania chromatyny stanowią chromosomy metafazowe.
<Drewa>
Najsilniej upakowany DNA odkryto w plemnikach, gdzie histony zostały zastąpione przez protaminy, białka bogate w argininę, które łącząc się z DNA przybierają strukturę prawie całkowicie alfa-helikalną.
Nawiniecie DNA na rdzeń nukleosomowy skraca jego liniowe wymiary i przyczynia się do jego upakowania w komórce.
Prosty odcinek DNA złożony z 200pz miałlby długość 68nm, natomiast w nuklosomie jego liniowe wymiary zredukowane są do ok. 10nm. Stopień skondensowania DNA w nukleosomach wynosi zatem ok. 7.
Łańcuch nukleosomów tworzy kolejną helisę - solenoid. Średnica solenoidu - 36nm, stopień skondensowania DNA - ok. 40.
41. Replikacja: miejsce rozpoczęcia, rola startera, fragmenty Okazaki, rola helikaz
Inicjacja replikacji:
PROKARIOTA |
EUKARIOTA |
|
|
ROLA STARTERA:
Do zainicjowania replikacji konieczny jest starter RNA („primer”, ma ok. 5 nukleotydów), syntetyzowany przez polimerazę RNA zwaną prymazą. Starter jest wydłużany przez polimerazę DNA III, która syntetyzuje DNA na obu matrycowych niciach DNA, tj. na nici wiodącej i opóźnionej. Polimeraza DNA I degraduje starter (wykorzystuje swą aktywność egzonukleazową 5'-3') i zastępuje go odcinkiem DNA, a następnie ligaza DNA łączy końce DNA.
FRAGMENTY OKAZAKI:
Synteza DNA przebiega w kierunku 5'-3' na każdej nici wyjściowego DNA. Na jednej z nici- wiodącej (3'-5') nowo powstający DNA jest syntetyzowany w sposób ciągły. Na drugiej nici-opóźnionej (5'-3') synteza jest nieciągła-najpierw tworzą się krótkie sekwencje: fragmenty Okazaki (długości około 1000 nukleotydów), które następnie ulegają połączeniu.
ROLA HELIKAZ:
Helikazy to grupa enzymów, które łączą się niespecyficznie z podwójną helisą DNA, sparowanymi nićmi RNA lub hybrydami DNA-RNA, rozplątują je i rozrywają wiązania wodorowe pomiędzy zasadami azotowymi, powodując rozdzielenie nici.
Wprowadzane są one do otwartego kompleksu w miejscu inicjacji replikacji wraz z innymi białkami, których uwolnienie aktywuje helikazę po czym rozpoczyna się proces replikacji.
Brak jednej z helikaz odpowiedzialny jest za zespół Blooma - niedobór odporności.
Rozplatają podwójną helisę na koszt ATP
Wielkość: 300 kDa
Liczba cząstek na komórkę: 20
42. Polimerazy DNA- właściwości korekcyjne
Podstawowa reakcją chemiczną katalizowana przez polimerazy DNA jest reakcja syntezy DNA prowadzona zawsze tylko w kierunku 5'-3'
Aktywność 3'-5' egzonukleazy jest właściwością wielu matrycozależnych polimeraz DNA, zarówno bakteryjnych, jak i eukariotycznych. Aktywność ta umożliwia polimerazie usuwanie nukleotydów od końca 3', a więc końca łańcucha aktualnie syntetyzowanego. Uważa się, że jest to aktywność korekcyjna, której rolą jest naprawa błędów popełnionych przypadkowo podczas dopasowywania wiązań wodorowych par zasad włączanych w budowany łańcuch polinukleotydowy
Aktywność 5'-3' egzonukleazy występuje mniej powszechnie. Mają je te polimerazy, których funkcja spełniana w czasie replikacji wymaga zdolności do usuwania co najmniej części fragmentów polinukleotydowych wcześniej dołączonych do matrycy właśnie przez nią kopiowanej. Aktywność ta jest np. niezbędna u bakterii podczas łączenia fragmentów DNA syntetyzowanych w czasie replikacji nieciągłej na matrycy opóźnionej
Zależne od matrycy polimerazy DNA przeprowadzające replikację DNA muszą zwiększać dokładność procesu łączenia komplementarnych par zasad o kilka rzędów wielkości (normalnie częstość błędów tego procesu: 5-10%). Na taką poprawę wydajności składają się dwa mechanizmy:
Polimeraza DNA ma mechanizm selekcji nukleotydów, który istotnie zwiększa dokładność syntezy DNA zależnej od matrycy. Selekcja ta odbywa się zapewne na trzech różnych etapach reakcji polimeryzacji- odrzucanie niewłaściwego nukleotydu zachodzi wówczas, gdy nukleotyd po raz pierwszy wiąże się z polimerazą DNA, gdy jest przenoszony do centrum aktywnego enzymu oraz gdy jest dołączany do końca 3' syntetyzowanego łańcucha polinukleotydowego
Dokładność syntezy DNA jest jeszcze większa wówczas, gdy polimeraza DNA wykazuje aktywność egzonukleazy działającej w kierunku 3'-5', dzięki czemu może usuwać niewłaściwy nukleotyd, który przeszedł przez proces selekcji zasad i został przyłączony do końca 3' nowego łańcucha polinukleotydowego. Nazywa się to korekcją błędów . Proces ten nie opiera się na żadnym aktywnym mechanizmie sprawdzającym. W istocie każdy etap syntezy łańcucha polinukleotydowego należy rozpatrywać jako współzawodnictwo między aktywnością polimerazową i egzonukleazową enzymu, w którym zwykle zwycięża polimeraza, ponieważ jest ona aktywniejsza od egzonukleazy, przynajmniej wtedy, gdy nukleotyd włączony na końcu 3' jest komplementarny do matrycy. Aktywność polimerazowa jest jednak słabsza wówczas, gdy końcowy nukleotyd nie pasuje do matrycy, następująca w wyniku tego przerwa w polimeryzacji umożliwia przewagę aktywnośći egzonukleazowej i usunięcie niewłaściwego nukleotydu.
Pojawiające się błędy mogą być poprawione przez „korektę”, jeśli polimeraza ma aktywność egzonukleazy 3'-5'. Jeżeli ostatni wstawiony nukleotyd jest komplementarny do matrycy, wówczas przeważa aktywność polimerazy. Jeżeli jednak nie jest on dopasowany, wtedy górę bierze aktywność egzonukleazy.
W Drewie była taka fajna tabela o tych polimerazach, jaką mają aktywność, gdzie występują, za co odpowiadają, także warto tam zajrzeć
43. Telomeraza-mechanizm działania
Telomery- sekwencje DNA znajdujące się na końcach chromosomów eukariotycznych.
Telomerowy DNA (końce chromosomów) jest syntetyzowany przez specyficzny enzym- telomerazę. Jest to polimeraza DNA, zawierająca Większość telomerowego DNA ulega kopiowaniu w prawidłowym procesie replikacji DNA. W celu uzupełnienia braków stwarzanych przez proce swój własny RNA, który służy jako matryca do syntezy powtarzających się krótkich sekwencji telomerowego DNA. Podczas replikacji telomery są wydłużane w zupełnie niezależnym procesie syntezy, katalizowanym przez enzym- telomerazę.
Telomeraza:
Zbudowana z elementu białkowego, jak i łańcucha RNA
Składa się z 450 nukleotydów
w pobliżu końca 5' sekwencja: 5'CUAACCCUAAC3', której region centralny jest sekwencją odwrotnie komplementarną do powtarzalnej sekwencji telomerów ludzkich: 5'TTAGGG3'. Sekwencja ta umożliwia telomerazie wydłużanie DNA od końca 3'-OH polinukleotydy, za pomocą następującego mechanizmu: Sekwencje DNA z powtarzalnym motywem telomeru ludzkiego: 5'TTAGGG3'. Łańcuch RNA telomerazy tworzy pary zasad z końcowym fragmentem cząsteczki DNA i przedłuża łańcuch polinukleotydowy DNA o krótki odcinek. Następnie telomer azowy RNA ulega translokacji do następnego miejsca parowania zasad wzdłuż polinukleotydowego łańcucha DNA i znowu wydłuża go o kolejny krótki odcinek. Proces ten może być powtarzalny, aż do uzyskania końca chromosomu odpowiedniej długości.
Telomeraza może syntetyzować tylko nić bogatą w G. Nie wiadomo, jak przebiega synteza drugiej nici, bogatej w C, lecz wydaje się, że gdy nić G zostanie wydłużona w sposób dostateczny, do jej końca dołącza się polimeraza DNA α i inicjuje syntezę nici komplementarnej, której dalsza replikacja przebiega w sposób normalny. Paradoksalnie nić C będzie krótsza od nici G, gdyż jej inicjacja wymaga syntezy startera RNA
Telomerazie w nieustannym przedłużaniu telomerów przeszkadzają białka wiążące telomerowy DNA (TBP). Nadprodukcja białek TBP prowadzi do skracania telomerów, natomiast wprowadzenie mutacji uniemożliwiającej wiązanie się TBP z telomerowym DNA powoduje, że telomery ulegają znacznie większemu wydłużeniu niż zwykle.
Mechanizm działania telomerazy nie jest do końca wyjaśniony. Przyjmuje się, że cząsteczka RNA telomerazy tworzy wiązania wodorowe z końcem telomeru. Wykorzystując następnie swój RNA jako matrycę do syntezy DNA (enzym ten jest więc odwrotną transkryptazą) telomeraza dołącza 6 nukleotydów do końca 3' telomeru. W następnym etapie telomeraza dysocjuje od DNA, ponownie łączy się z końcem telomeru i powtarza proces wydłużania nicic DNA. Cykl ten telomeraza powtarza setki razy, zanim ostatecznie zostanie odłączona od chromosomu. Nowo wydłużona nić DNA służy następnie jako matryca w normalnej replikacji DNA, w wyniku czego powstaje dwuniciowy chromosomowy DNA.
44. Czynniki mutagenne, rodzaje mutacji
Czynnikami mutagennymi są:
Czynniki fizyczne:
Promieniowanie nadfioletowe (UV): o λ=260nm indukuje dimeryzację sąsiadujących ze sobą zasad pirymidynowych, zwłaszcza gdy są to dwie T, powodując powstanie dimeru cyklobutylowego. Tworzenie dimerów w kolejności częstości powstawania: CT>TC>CC (wszystkie od 5' do 3'). Dimery puryn są znacznie rzadsze. Inny rodzaj fotoproduktu stanowi uszkodzenie (6-4), w którym atomy węgla o numerach 4 i 6 sąsiadujących ze sobą pirymidynach tworzą wiązanie kowalencyjne
Promieniowanie jonizujące: mogą wystąpić mutacje punktowe, delecje, insercje, a także poważne uszkodzenia DNA uniemożliwiające replikację genomu (w zależności od rodzaju i natężenia promieniowania), niektóre rodzaje promieniowania działają bezpośrednio, inne pośrednio- tworząc nadtlenki
wysoka temperatura: stymuluje zależnie od wody cięcie wiązania β-N-glikozydowego (łączy zasadę z cukrem w nukleotydzie). Zdarza się to częściej w purynach, niż w pirymidynach i powoduje powstanie miejsca AP (apurynowego/apirymidynowego), czyli pozbawionego zasady. Pozostający fosforan cukru jest niestabilny, ulega szybkiej degradacji, tworząc lukę w dwuniciowym DNA
czynniki chemiczne:
kwas azotowy (III) - HNO2 - powoduje usunięcie grup aminowych z zasad azotowych, co powoduje np. zamianę cytozyny w uracyl
związki alkilujące (np. iperyt i jego pochodne) - powodują dołączanie do zasad azotowych grup alkilowych, co również zmienia ich charakteranalogi zasad azotowych (np. 5-bromouracyl odpowiada tyminie; 2-aminopuryna-analog adeniny) - nie są prawidłowo odczytywane podczas transkrypcji
barwniki akrydynowe (np. oranż akrylowy, akryflawina, proflawina) - powodują wstawianie lub wycinanie sekwencji nukleotydowych
alkaloidy - np. kolchicyna, blokująca tworzenie wrzeciona podziałowego, co powoduje, że chromosomy nie rozchodzą się podczas podziału
sole metali ciężkich
czynniki deaminujące (kwas azotawy-deaminuje A,C i G; dwusiarczan sodowy- usuwa C) powodują usunięcie grupy aminowej. Deaminacja G nie jest mutagenna- powstaje ksantyna, która blokuje replikację. Deaminacja A daje hipoksantynę, tworzy ona pary z C, zamiast z T. Deaminacja C daje U, który tworzy pary z A, zamiast z G.
czynniki alkilujące (dimetylonitrozoamina, metanosulfonian etylowy- EMS, halogenki metylu, produkty metabolizmu azotynów) powodują mutacje punktowe, dodają grupy alkilowe do nukleotydów w cząsteczkach DNA. Efekt alkilacji zależy od pozycji, w której zostaje zmodyfikowany nukleotyd, jak również od rodzaju dodanej grupy alkilowej. Metylacja często prowadzi do mutacji punktowej (nukleotydy o zmienionych zdolnościach tworzenia komplementarnych wiązań), inne alkilacje blokują replikację, tworząc połączenia między dwiema niciami DNA lub dodając duże grupy alkilowe-brak postępu kompleksu replikacyjnego
czynniki interkalujące: (bromek etydyny); zwykle powodują insercje, mogą one się wsuwać pomiędzy pary zasad, bo są płaskimi cząsteczkami i spowodować niewielkie rozwiniecie helisy- zwiększenie odległości między sąsiednimi parami zasad
czynniki metaboliczne (np. brak jonów Mg2+ lub Ca2+)
Dwa sposoby powstawania mutacji:
Spontaniczne błędy w replikacji, wymykające się spod kontroli mechanizmów korekcyjnych polimeraz DNA. Mutacje te nazywane są błędami niedopasowania nukleotydów, ponieważ polegają na wstawieniu w danej pozycji nukleotydu nie komplementarnego do nukleotydu z matrycowej nici DNA. Jeżeli brak komplementarności utrzyma się w podwójnej helisie, wtedy jedna z cząsteczek drugiego pokolenia potomnego będzie zawierać w obu niciach wersje mutacji wprowadzoną na stałe
Mutacje powstające, gdy mutagen oddziałuje z DNA rodzicielskim, wywołując zmianę struktury, wpływającą na właściwości komplementarnego wiązania zmienionego nukleotydu. Z reguły zmiana dotyczy tylko jednej nici rodzicielskiej-tylko jedna czasteczka potomna niesie mutację, natomiast w drugim pokoleniu potomnym znajdzie się ona w dwóch spośród utworzonych cząsteczek
(rysunki ze strony 341 Genomów)
Przyczyny mutacji:
Błędy w replikacji- przyczyna mutacji punktowych. Normalnie częstość błędów wynosi 5-10%, ale do zwiększenia wydajności tej reakcji przyczyniają się polimerazy DNA (patrz pytanie 42). Nie wszystkie błędy można przypisać polimerazom: niekiedy wstawiany jest „właściwy” nukleotyd, jednak nukleotydy (dokładnie zasady azotowe) mogą występować w postaci 2 tautomerów - izomerów strukturalnych
Tymina- forma ketonowa (prawidłowa) i enolowa. Jeśli wstawiona zostanie forma enolowa, to komplementarną zasadą będzie guanina, a nie adenina (T-G)
Adeniana i jej tautomer iminowy, który wiąże się z cytozyną (A-C)
Guanina i jej forma enolowa komplementarna do tyminy (G-T)
Błędy w replikacji mogą doprowadzić do mutacji typu insercji i delecji. Mutacje tego typu często nazywa się mutacjami zmiany fazy, ponieważ może nastąpić przesunięcie fazy odczytu przy translacji białka kodowanego przez gen. Insercja lub delecja 3 lub wielokrotności 3 nukleotydów jedynie dodaje lub usuwa jeden lub kilka kodonów- brak zmiany fazy odczytu. Insercje i delecje szczególnie częste, gdy matryca DNA ma krótkie powtarzające się sekwencje, które mogą wywołać poślizg replikacji, przy którym nić matrycowa i jej kopia przesuwają się względem siebie, przez co część matrycy jest powielana dwukrotnie lub opuszczana. Zjawisko to stanowi główną przyczynę dużej zmienności sekwencji mikrosatelitarnych, jak również odpowiada za choroby powtórzeń tri nukleotydowych. Niektóre choroby związane z upośledzeniem umysłowym powstają w wyniku ekspansji tri nukleotydowych w obszarze liderowym genu- powstaje wtedy miejsce kruche, czyli obszar w chromosomie, który łatwo ulega pęknieciu
RODZAJE MUTACJI:
duże rearanżacje genowe
- delecje - utrata części sekwencji DNA (alfa-talasemia, niedobór hormonu wzrostu, rodzinna hipercholesterolemia, dystrofia mięśniowa Duchenne'a)
duplikacje - powielenie sekwencji DNA (rodzinna hipercholesterolemia, dystrofia mięśniowa Duchenne'a)
insercje - wbudowanie dodatkowej sekwencji DNA (hemofilia, neurofibromatoza)
mutacje punktowe
Najczęstsze mutacje w genomie ludzkim
Typy:
insercje
delecje
tranzycje (zastąpienie zasady purynowej inną zasadą purynową lub pirymidynowej inną pirymidynową)
transwersje (zastąpienie puryny pirymidyną lub odwrotnie)
Efekt:
- mutacje missens (zmiany sensu) -zmiana nukleotydu w pierwszej lub drugiej pozycji kodonu powoduje zmianę kodowanego przez triplet aminokwasu. Zmienia się aminokwas w białku.
- mutacje neutralne (ciche) - nie powodują zmiany kodowanego aminokwasu, ponieważ zmiana nukleotydu następuje w trzeciej pozycji tripletu lub też mutacja nie dotyczy sekwencji kodujących lub z nimi związanych.
- typu stop kodon (nonsense), powodujące przesuniecie ramki odczytu (frameshift mutation)
mutacje transkrypcyjne
Występują w obszarze od 5' do początku kodonu inicjacyjnego w sekwencji DNA (np. sekwencje TATA). Jest to miejsce krytyczne dla inicjacji transkrypcji . Efektem tych mutacji jest spadek produkcji białka.
mutacje translacyjne
mutacje nonsens (stop) - w wyniku mutacji powstaje przedwcześnie kodon terminacyjny stop, czego efektem jest białko skrócone
mutacje zmiany ramki odczytu - delecja lub insercja w rejonie kodującym (nie będąca wielokrotnością trzech) genu zmienia wszystkie występujące za mutacja kodony; następuje zmiana sekwencji aminokwasowej - zmienia sie kodowane białko
mutacje RNA
zmieniają prawidłowe miejsca łączące w regionie połączenia intronu i eksonu
tworzą nowe miejsca łączenia wewnątrz intronów lub eksonów
w wyniku takiej mutacji może powstać niestabilne szybko degradujące się RNA
mutacje dynamiczne: mutacje występujące tylko u człowieka, wykazujące dużą zmienność populacyjną:
mutacja polegająca na wzroście liczby powtórzeń trójnukleotydowych
korelacja miedzy ciężkością choroby oraz wiekiem pojawienia się objawów, a ilością powtórzeń
ilość powtórzeń może wzrastać w kolejnych pokoleniach
objawy mogą nasilać sie lub występować wcześniej w kolejnych pokoleniach (antycypacja)
premutacja - wzrost ilości powtórzeń poniżej wartości granicznej - nie powoduje wystąpienia choroby - bezobjawowi nosiciele
Mutacje dynamiczne stanowią podłoże molekularne 14 jednostek chorobowych i można je podzielić na dwie grupy:
Typ I - choroby neurodegradacyjne: ch. Huntingtona, rdzeniowo-opuszkowy zanik mięśni, ataksje móżdżkowo-rdzeniowe,
Typ II - dystrofia miotoniczna, zespół kruchego chromosomu X, upośledzenie umysłowe związane z kruchym miejscem FRAXE, ataksja Friedreicha.
negatywne mutacje dominujące
zmutowany allel wytwarza produkt, który interferuje z produktem allelu niezmutowanego
geny kodujące białka strukturalne - geny kolagenu, elastyny
choroby: osteogenesis imperfecta, zespół Marfana
45. Naprawa uszkodzeń DNA- podstawowe mechanizmy
Większość komórek ma 5 różnych kategorii systemów naprawy DNA:
Systemy naprawy bezpośredniej: działają bezpośrednio na uszkodzone nukleotydy, przywracając każdemu z nich oryginalną strukturę, naprawa jest możliwa w następujących przypadkach:
Pęknięcia: naprawiane przez ligazę DNA, jeśli przerwane było wiązanie fosfodiestrowe, bez uszkodzenia grupy 5'-fosforanowej oraz 3'-hydroksylowej po obu stronach pęknięcia. Często tak się dzieje pod wpływem promieniowania jonizującego
Niektóre uszkodzenia przez alkilację są bezpośrednio odwracalne dzięki aktywności enzymów przenoszących grupę alkilową z nukleotydu na własny łańcuch polipeptydowy np. enzym Ada u E.coli usuwa grupy alkilowe przyłączone do atomów tlenu w pozycji 4 i 6, odpowiednio w T i G, może też naprawiać zmetylowane wiązania fosfodiestrowe ; MGMT (metylotransferaza O6-metyloguanino-DNA) usuwa grupy alkilowe z pozycji numer 6 guaniny
Dimery cyklobutylowe naprawiane przez zależny od światła bezpośredni system zwany fotoreaktywacją. U E.coli jest to fotoliza DNA, jeśli λ=300-500nm, to enzym ten wiąże się z dimerami cyklobutylowymi i zamienia je z powrotem na wyjściowe nukleotydy mononumeryczne. Fotoliaza jest u licznych bakterii, eukariontów, kręgowców, ale NIE MA jej u człowieka! Podobnie działa fotoliza fotoproduktów (6-4)-występuje u licznych mikroorganizmów, nie ma go u E.coli ani u człowieka
Naprawa z wycinaniem zasad: usuwanie uszkodzonej zasady azotowej, wycięcie krótkiego fragmentu wokół powstałego tam miejsca AP i ponowna synteza z udziałem polimerazy DNA. Proces inicjowany jest przez glikozydazę DNA, która przecina wiązanie β-N-glikozydowe między zasadą a cukrem. Każda glikozydaza ma zawężoną specyficzność. Większość organizmów potrafi usuwać uracyl (deaminowana cytozyna) i hipoksantyna (deaminowana adenina), produkty utleniania: 5-hydroksycytozyna i glikol tyminowy oraz zmetylowane zasady: 7-metyloguanina i 2-metylocytozyna. Glikozydaza DNA usuwa uszkodzoną zasadę przez „odwrócenie” struktury-jest ona na zewnątrz helisy-zostaje odłączona, a następnie powstałe miejsce AP zamieniane jest w jednonukleotydową lukę. Etap ten może przebiegać wieloma sposobami: najczęściej endonukleaza AP (egzonukleaza III lub endonukleaza IV) przecina wiązanie fosfodiestrowe po stronie 5'ASTR. Niektóre endonukleazy usuwają dodatkowo z miejsca AP cukier, inne tego nie potrafią i działają razem z odrębną fosfodiestrazą. Alternatywny szlak przemiany miejsca AP w lukę-wykorzystanie aktywności endonukleazowej niektórych glikozydaz DNA, które dokonują cięcia po stronie 3' AP, prawdopodobnie z równoczesnym usunięciem uszkodzonej zasady. Następnie cukier jest usuwany przez fosfodiestrazę. Jednonukleotydowa lukę wypełnia polimeraza DNA (wykorzystuje nieuszkodzoną zasadę z drugiej nici). U E.coli lukę wypełnia polimeraza DNA I, drożdże wykorzystują polimerazę δ, a ssaki polimerazę DNA β. Po wypełnieniu luki wiązanie fosfodiestrowe tworzone jest przez ligazę DNA.
Naprawa z wycinaniem nukleotydów: podobnie jak naprawa z wycinaniem zasad, nie jest jednak poprzedzona usunięciem uszkodzonej zasady i może działać na bardziej uszkodzone obszary DNA. Naprawa ta może usuwać połączenia wewnątrz nici czy zasady z przyłączonymi dużymi grupami chemicznymi. Może też usuwać dimery cyklobutylowe w procesie naprawy ciemnej-czyli u organizmów nie wyposażonych w system fotoreaktywacji naprawy tych dimerow. Podczas naprawy z wycinaniem nukleotydów fragment jednoniciowego DNA z uszkodzonym nukleotydem jest usuwany i zastąpiony nowym DNA. Procesu tego nie poprzedza jednak selektywne usuwanie zasady, a wycinany fragment łańcucha jest dłuższy. Najlepiej zbadany przykład to proces naprawy łat u E.coli- wycinany i łatany nastepnie fragment jest stosunkowo krótki: ma 12 nukleotydów. Naprawę krótkich łat inicjuje kompleks enzymów: endonukleaza UvrABC (ekscinukleaza). W pierwszej fazie w miejscu uszkodzonego DNA przyłącza się trimer mający dwa monomery białka UvrA i jeden UvrB. Elementem najbardziej zaangażowanym w lokalizację uszkodzenia jest najprawdopodobniej UvrA- białko to dysocjuje bowiem po odnalezieniu miejsca uszkodzenia. Odłączenie się UvrA pozwala na przyłączenie się UvrC, tworzy się wtedy dimer UvrBC przecina łańcuch polinukleotydowy po obu stronach uszkodzenia. UvrB nacina w piątym wiązaniu fosfodiestrowym poniżej uszkodzonego nukleotydu, a UvrC w ósmym powyżej- przez co wycinanych jest 12 nukleotydów. Wyciety fragment jest kolejno usuwany w postaci całego polinukleotydu przez helikazę DNA II. Odłączają się również UvrC, a UvrB zostaje na miejscu i zapełnia powstałą lukę. Luka jest wypełniana przez polimerazę DNA I, a ostatnie wiązanie fosfodiestrowe syntetyzowane przez ligazę DNA. E.coli ma też system naprawy długich łat- fragment wycietego DNA może mieć do 2 kb. Działa on najprawdopodobniej w obszarach zmodyfikowanych całych grup nukleotydów. U eukariota system długich łat obejmuje wymianę 24-29 nukleotydów. U człowieka w system ten jest zaangażowanych co najmniej 16 białek. Pierwsze ciecie tak, jak u E.coli. Obu cieć dokonują endonukleazy. Najprawdopodobniej DNA wokół uszkodzenia jest rozplatany przez helikazę, a za aktywność tą odpowiada TFIIH- pełni funkcje transkrypcji i naprawie. Obecnie uważa się, że między tymi procesami istnieje bezpośredni związek. Dowód: odkrycie naprawy sprzężonej z transkrypcją, nici genów do syntezy RNA są naprawiane szybciej niż inne obszary genomu.
Naprawa niedopasowanych nukleotydów: poprawia błędy replikacji, wycina fragment jednoniciowy DNA, zawierający niewłaściwy nukleotyd i wypełnia powstałą lukę. System ten musi wykrywać nie tyle sam niedopasowany nukleotyd, ile brak komplementarności między nicią matrycową, a potomną. Po wykryciu niedopasowania system wycina część łańcucha potomnego i wypełnia lukę podobnie jak naprawa z wycinaniem zasad i nukleotydów. System naprawczy musi zatem odróżniać nić matrycową od potomnej. U E.coli nić potomna jest słabiej zmetylowana. DNA E.coli podlega metyzacji dzieki metylazie adeninowej DNA (Dam)-zamienia ona A na 6-metyloadeninę w sekwencji5'GATC3' oraz metylazie cytozy nowej DNA (Dcm) zamienia C na 5-metylocytozynę w sekwencjach 5'CCAGG3' oraz 5'CCTGG3'. Metylacje te nie są mutagenne. Między replikacją DNA, a metyzacją nici potomnej występuje opóźnienie, które daje systemowi naprawczemu czas na wyszukanie niedopasowanych nukleotydów. E.coli ma trzy systemy takiej naprawy: długich/krótkich/bardzo krótkich łat. System długich łat: wymienia nawet ponad 1kb DNA, wymaga białek MutH(rozróznia obie nici DNA, wiążąc się z niezmetylowanymi sekwencjami 5'GATC3'), MutL(rola nie jest jasna, koordynacja pozostałych białek: by MutH wiązało siew dobrym miejscu) i MutS(wykrywa brak komplementarności) oraz helikazy DNA II. Po związaniu MutH przecina wiązanie fosfodiestrowe przed G w etylowanej sekwencji, a helikaza oddziela pojedynczą nić. Nie ma enzymu przecinającego nić poniżej miejsca dopasowania-oddzielony fragment jednoniciowy jest degradowany przez egzonukleazę. Luka jest następnie wypełniana przez polimerazę DNA I i ligazę DNA. Podobnie jest w systemach naprawy krótkich (wycina mniej niż 10 nukleotydów; uruchamiany po rozpoznaniu niedopasowanej pary A-G lub A-C przez MutY) i bardzo krótkich łat (naprawia niedopasowane G-T, rozpoznawane przez endonukleazę Vsr). U Eukariota homologi białek Mut. Różnica jest to, że do rozróżnienia obu nici nie zawsze służy metyzacja. (DNA muszki owocowej i drożdży nie jest silnie zmetylowany). Możliwe jest połączenie systemu naprawczego z kompleksem replikacyjnym-naprawa sprzężona jest z syntezą DNA, lub wykorzystanie białek wiążących się z jednoniciowym DNA, oznaczających nić rodzicielską.
Naprawa rekombinacyjna: wykorzystywana do naprawy dwuniciowych przerw w DNA. W proces ten zaangażowane są 4 grupy genów. Kodują one wieloskładnikowy kompleks białkowy, który kieruje ligazę DNA do miejsca pęknięcia. W skład kompleksu wchodzi białko Ku (złożone z dwóch różnych podjednostek), które wiąże się z końcami DNA po obu stronach pęknięcia wraz z kinazą białkową, zwaną DNA-PKCS, która aktywuje prawdopodobnie trzecie białko XRCC4, które oddziałuje z ligazą DNA IV ssaków, kierując to białko naprawcze w miejsce dwuniciowego pęknięcia. Taki proces to łączenie końców niehomologicznych (NHEG). Traktuje się go jako rodzaj rekombinacji, ponieważ wykorzystywany jest nie tylko do naprawy przerw, ale też do łączenia cząsteczek/fragmentów uprzednio niepołączonych, tworząc nowe kombinacje. (prawdopodobnie wykorzystywany do tworzenia genów Ig i receptorów limfocytów T)
46. Aktualne teorie tłumaczące proces onkogenezy
Najpowszechniej akceptowany jest pogląd, że mutacje w obrębie niewielkiej grupy specjalnych genów zatrzymują syntezę białek hamujących rozwój nowotworu i uaktywniają onkoproteiny. Ostatnio jednak rosnącym zainteresowaniem cieszą się trzy teorie alternatywne. Jedna z nich jest modyfikacją teorii standardowej według której w komórkach przedrakowych następuje ogromny wzrost nakładających się na siebie losowych mutacji. Dwie inne hipotezy koncentrują się na roli aneuploidii.
TEORIA STANDARDOWA
Kancerogeny np. promieniowanie UV czy dym tytoniowy bezpośrednio wpływają na zmianę sekwencji DNA w genach związanych z rakiem
W wyniku mutacji genów supresorowych w komórce nie ma prawidłowo działających białek, które powstrzymują jej wzrost. Komórka dzieli się nadal, choć nie powinna.
Równocześnie mutacje onkogenów powodują zwiększoną aktywność onkoprotein; stymulują one dodatkowo rozmnażanie komórek
Nadmiar onkoprotein i brak białek supresorowych nowotworów powoduje szybki wzrost liczby zmutowanych komórek
Po wielu cyklach i ekspansji… ze zmienionych…pokonuje wszel… jej rozwój. Rak atakuje
W najbardziej zaawansowanym stadium komórki…
ZMODYFIKOWANA TEORIA STANDARDOWA (MUTATOROWA)
Jakiś czynnik zakłóca działanie jednego lub większej liczby genów potrzebnych do syntezy lub naprawy DNA
Nie usuwane mutacje nakładają się na siebie w kolejnych podziałach komórki. Ich liczba sięga dziesiątki tysięcy. W pewnym momencie ofiarom zmian padają też geny związane z rakiem
Tak jak w teorii standardowej eliminacja białek supresorowych nowotworów i aktywacja onkoprotein wyłącza mechanizmy samozniszczenia
Jak wyżej
Jak wyżej
TEORIA WCZESNEJ NIESTABILNOŚCI
Coś wycisza (unieczynnia) jeden lub kilka genów głównych odpowiedzialnych za podział komórek
W procesie powstania pojawiają się błędy. Niektóre komórki potomne otrzymują niewłaściwą liczbę chromosomów lub chromosomy są uszkodzone (np. pozbawione jednego ramienia). Po każdym podziale komórki zaburzenia stają się poważniejsze. W ich wyniku zachodzą zmiany w składzie i ilości genów komórkowych (dodanie lub usuwanie fragmentów chromosomów zmienia układ genów w komórce)
W pewnym momencie poziom białek supresorowych nowotworów spada poniżej wartości krytycznej, a dodatkowe kopie onkogenów powodują wzrost ilości wytwarzanych onkoprotein
Jak wyżej
Jak wyżej
W najbardziej zaawansowanym stadium komórki rakowe przedostają się do krwi. W odległych miejscach organizmu tworzą się..
TEORIA PANANEUPLOIDIA
W wyniku błędu w procesie podziału powstają komórki aneuploidalne
Niewłaściwe rozmieszczenie lub uszkodzone chromosomy powodują zmianę proporcji występowania tysięcy genów. Zaczynają zawodzić enzymy współdziałające ze sobą przy powielaniu lub naprawie DNA. W rezultacie większość uszkodzonych komórek ginie
Te, które przeżyją wytwarzają komórki potomne. Są one również aneuploidalne, choć w sposób różniący się od komórek wyjściowych
W pewnym momencie kilka komórek uzyskuje odpowiedni zestaw cech- `uprawnienia specjalne”… Mnożą się, tworząc niezłośliwy nowotwór
Podczas..
KOMÓRKI MACIERZYSTE
Komórki macierzyste, inaczej komórki pnia- komórki, które jednocześnie:
Są zdolne do potencjalnie nieograniczonej liczby podziałów
Mają zdolność do różnicowania się jednego typu komórek
Rodzaje komórek macierzystych:
Embrionalne
Płciowe-w narządach płciowych
Somatyczne- w różnych rodzajach narządów
Symetryczny i niesymetryczny podział komórek pnia
Nowotwór, a komórki macierzyste:
komórki macierzyste umożliwiają regenerację uszkodzonych lub martwych komórek
Liczba komórek macierzystych (SC) pozostaje stała w organizmie- samoodnowa
Namnażanie się SC jest ściśle kontrolowane przez sygnały genetyczne.
Usunięcie sygnałów-niekontrolowane podziały.
Nowotwór powstaje w wyniku nagromadzenia się licznych mutacji w komórce.
Długo żyjące SC mogą nagromadzić odstatecznie dużą liczbę mutacji
czynniki mutagenne
kom. macierzysta
nowotworu
nie wytwarzają genów nowotworowych po wszczepieniu ich zwierzętom
WPŁYW NOWOTWORU NA ROZWÓJ RAKA
Nisza, w której znajduje się guz wywiera duży wpływ na inicjacje i przebieg procesu nowotworowego. Zmutowane SC przeniesione do innej niszy nie wytworzy guza. Zdrowe SC przeszczepione do uszkodzonych niszy dają początek nowotworów.
47. Regulacja wydzielania przeciwciał przez limfocyty B na poziomie ekspresji genetycznej ( z zakazanej książki)
Hipoteza komórek linii płciowej, zgodnie z którą wszystkie przeciwciała są kodowane przez geny w komórkach linii zarodkowej jest błędna: człowiek ma w genomie 105 genów, a potrafi wytworzyć przynajmniej 1015 rodzajów przeciwciał o rożnej swoistości
W rzeczywistości komórki linii zarodkowej nie zawierają kompletnych genów łańcuchów lekkich i ciężkich. Geny te mają postać oddzielnych kodujących części (segmentów), składanych w procesie zwanym rekombinacją somatyczną, która zachodzi podczas dojrzewania limfocytu B. Proces składania odbywa się w każdym limfocycie B. Poprzez składanie rożnych fragmentów DNA powstają całkowicie nowe geny immunoglobulin, co stanowi podstawę różnorodności przeciwciał
Rekombinacja genów łańcuchów lekkich:
Część zmienna (V) łańcucha lekkiego κ jest kodowana przez sekwencję DNA odrębną od sekwencji kodującej część stałą (C). Obie sekwencje kodujące znajdują się na tym samym chromosomie, ale są od siebie oddalone.
Podczas dojrzewania limfocytu B fragmenty DNA kodujące część V i C zbliżają się do siebie i zostają połączone, tworząc tym sposobem funkcjonalny gen łańcucha lekkiego. Proces ten określany mianem rekombinacji somatycznej, polega zwykle na usunięciu DNA znajdującego się między odcinkami kodującymi części V i C w komórce linii zarodkowej, w niektórych przypadkach może też wykorzystywać mechanizm inwersji
W komórce linii zarodkowej segment V genu koduje tylko 95 aminokwasów części zmiennej łańcucha lekkiego (od końca -N). Pozostałych kilka aminokwasów części V (reszty 96-108) jest kodowanych przez odcinek DNA nazywany segmentem J genu. Segment ten nie jest równoznaczny z polipeptydem J w pentametrze IgM! W komórce linii zarodkowej segment J genu poprzedza segment C genu, od którego oddzielony jest tylko intronem. Co więcej, istnieją liczne warianty segmentu V genu (ok. 300) i 5 wariantów segmentu J genu, z których jeden jest nieaktywny.
Podczas dojrzewania limfocytów B jeden z ok. 300 alleli Vx zostaje precyzyjnie połączony z jednym z alleli Jy genu, co prowadzi do powstania genu łańcucha lekkiego. Ten proces jest nazywany łączeniem VJ. Transkrypcja rozpoczyna się w pobliżu początku zrekombinowanego segmentu genu V i przebiega aż do końca segmentu C genu. Pozostałe allele J genu ulegają również transkrypcji, ale sekwencje te są usuwane podczas zachodzącego następnie dojrzewania RNA- w jego wyniku usuwany jest intron poprzedzający segment genu C. Powstały ostatecznie mRNA zawiera jedynie sekwencje VxJyC i koduje odpowiedni polipeptyd łańcucha lekkiego. Zależnie od tego, który z 300 alleli genu V zostanie wybrany i połączony z jednym z alleli J genu, powstaje olbrzymia różnorodność łańcuchów lekkich.
Geny łańcucha lekkiego λ powstają także dzięki rekombinacje somatycznej, zachodzącej podczas dojrzewania limfocytów B, ale jest mniej alleli V i J, niż w przypadku genów kodujących łańcuchy κ. Większość cząsteczek przeciwciał zawiera łańcuchy lekkie κ, a nie łańcuchy lekkie λ
Rekombinacja genów łańcuchów ciężkich:
Powstają w analogiczny sposób, z tym że kodujące je geny składają się z 4 segmentów tworzonych przez odcinki V, J, D i C. Wyróżnia się ok. 200-1000 alleli VH, ok. 15 aktywnych alleli DH i 4 allele JH. Gdy cześć zmienna łańcucha lekkiego jest kodowana przez allele V i J, część zmienna łańcucha ciężkiego jest kodowana przez allele V, D i J.
W systemie genów łańcucha ciężkiego wyróżnia się kilka alleli C, po jednym dla każdego typu łańcucha ciężkiego: Cμ, Cδ, Cγ ( o różnych podklasach) Cε i Cα, kodujące odpowiednio części stałe łańcuchów ciężkich μ, δ, γ, ε i α.
Podczas dojrzewania limfocytu zachodzą dwie rearanżacje genu łańcucha ciężkiego. Najpierw dany allel genu DH łączy sie z allelem JH (łączenie DJ) i następnie zrekombinowany fragment DHJH łączy się z danym allelem VH (łączenie VDJ).
W procesie składania genów łańcucha ciężkiego i lekkiego końce różnych segmentów DNA, które mają zostać połączone, mogą w trakcie przebiegu procesu rekombinacji podlegać modyfikacjom, co prowadzi do zmian w obrębie kodonów istniejących w miejscach połączeń lub nawet powoduje powstanie nowych kodonów, dzięki czemu dodatkowo zwiększa się różnorodność przeciwciał. Poza tym, dla genów kodujących przeciwciała stwierdza się większe niż zwykle tempo mutacji
Przełączenie klas:
Kiedy transkrypcji podlega gen łańcucha ciężkiego (V3D2J2CμCδCγCεCα), dochodzi do powstania łańcucha ciężkiego IgM, ponieważ za zrekombinowanym fragmentem VDJ jest położony segment utworzony przez gen Cμ i to on ulega transkrypcji. Aby doszło do przełączenia syntezy łańcucha ciężkiego na łańcuch przeciwciała innej klasy np. IgA, DNA limfocytu musi ulec kolejnej rekombinacji, prowadzącej do przesunięcia odcinka Cα w bezpośrednie sąsiedztwo zrekombinowanego fragmentu VDJ i usunięcia zbędnych alleli genu C. W wyniku ekspresji uzyskanego w ten sposób genu powstaje łańcuch ciężki Cα przeciwciała klasy IgA zamiast powstającego uprzednio łańcucha ciężkiego Cμ przeciwciała klasy IgM. Istota przełączania klas/przełączania łańcuchów ciężkich polega na tym, że zmianie ulega tylko część C syntetyzowanych łańcuchów ciężkich, część zmienna pozostaje ta sama. Swoistość przeciwciała jest określana przez miejsce wiązania antygenu, które z kolei tworzą części zmienne łańcucha ciężkiego i lekkiego. Tak więc nawet jeśli zajdzie zjawisko przełączenia klasy, prowadzące do zastąpienia wytwarzanego przez limfocyt przeciwciała klasy IgM przeciwciałem klasy IgA, swoistość tego przeciwciała pozostaje nie zmieniona.
48. Stała Michaelisa-definicja i opis
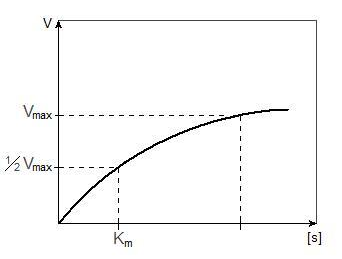
WYSOKA wartość Km- enzym ma MAŁE powinowactwo do substratu
NISKA wartość Km- enzym ma DUŻE powinowactwo do substratu
Wartość stałej Km dla większości enzymów przyjmuje wartości z zakresu 10-3 do 10-5 mol/dm3
Stężenie substratu, przy którym osiąga się szybkość reakcji równą połowie vmax, równa jest stałej dysocjacji kompleksu E-S; stężenie to od nazwiska twórcy nazywa się stałą Michaelisa (KM). Wymiar: mol/l, przy tym stężeniu enzym jest w połowie nasycony.
Przy małym stężeniu substratu reakcja I rzędu i szybkość reakcji uzależniona od stężenia substratu = [S]. Po zwiększeniu stężenia substratu osiąga się maksymalną szybkość reakcji, niezależną od stężenia substratu = k*[E]
Wytłumaczenie: gdy stężenie substratu jest małe, niektóre cząsteczki enzymu w danej chwili nie są połączone z substratem i niecałkowite wysycenie enzymu jest przyczyną tego, że nie wykazuje on maksymalnej aktywności katalitycznej. W miarę zwiększenia stężenia substratu dochodzi się do momentu, kiedy wszystkie cząsteczki enzymu będą z nim połączone i wtedy dopiero enzym pracuje z maksymalną szybkością. Wobec tego, że enzym jest już całkowicie wysycony substratem dalsze zwiększanie stężenia substratu nie wpływa na zwiększenie szybkości reakcji- reakcja przebiega ze stałą szybkością. Fakt ten tłumaczy się powstaniem kompleksu E-S w trakcie katalizy enzymatycznej kompleks ulega pewnym zmianom i powstaje produkt oraz enzym w postaci wolnej.
Co wpływa na stałą Michaelisa:
rodzaj i stężenie substratu
pH
temperatura
siła jonowa
Stała Km nie zależy od stężenia enzymu!
Wyprowadzenie:
Km= (K-1+K2)/K1= [E][S]/[ES]
49. Acetylotransferaza karnitynowa- rola w metabolizmie kwasów tłuszczowych
Transport acylo-CoA przez błonę mitochondriom
Acylotransferaza karnitynowa- jest to enzym związany z rozpadem kwasów tłuszczowych, czyli β-oksydacją. Zaktywowany kwas tłuszczowy: połączony wiązaniem tioestrowym z CoA, nazwany acylo-CoA, by przejść przez wewnętrzną błonę mitochondrialną wymaga specyficznego mechanizmu transportu-sprzężenia z polarną cząsteczką karnityny. Reakcja sprzęgania katalizowana jest przez enzym umiejscowiony na zewnętrznej powierzchni wewnętrznej błony mitochondrialnej-acylotransferazę karnitynową I i polega na usunięciu CoA oraz zastąpieniu go cząsteczką karnityny. Powstała acylokarnityna transportowana jest przy udziale translokazy przez wewnętrzną błonę mitochondriom do matriks mitochondrialnej, gdzie cząsteczki karnityny są uwalniane, czemu towarzyszy przeniesienie grupy acylowej z powrotem na CoA. Reakcję tę katalizuje acylotransferaza karnitynowa II znajdująca się na wewnętrznej błonie mitochondrialnej od strony matriks.
58.Produkty dekarboksylacji aminokwasów.
Reakcje dekarboksylacji aminokwasów polegają na rozerwaniu wiązania między grupą karboksylową -COOH i resztą cząsteczki aminokwasu, w wyniku czego wydziela się CO2 i powstaje odpowiednia amina. Reakcję katalizują dekarboksylazy aminokwasowe. Dekarboksylacja aminokwasowa jest źródłem amin biogennych - substancji o dużej aktywności fizjologicznej, np.
Histamina - po dekarboksylacji histydyny
Tyramina - po dekarboksylacji tyroksyny
Tryptamina - po dekarboksylacji tryptofanu
Serotonina = 5-hydroksytryptamina - po dekarboksylacji 5-hydroksytryptofanu
Pultrescyna - powstaje z ornityny
Kadaweryna - powstaje z lizyny
Cysteamina - składnik CoA, produkt dekarboksylacji cysteiny
W wyniku dekarboksylacji niektórych aminokwasów tworzą się ważne części składowe koenzymów, np.
2-propanolamina (składnik koenzymu B12)
cysteamina (składnik koenzymu A)
Z kwasu glutaminowego powstaje kwas gamma-aminomasłowy GABA, który należy do neurotransmiterów hamujących.
Pyt. 59
Budowa i podział aminokwasów (gluko- i ketogenne, egzo- i endogenne, niezbędne i nie niezbędne).
(Stryer rozdz. 2 `Struktura i funkcja białek'; Kłyszejko; Kędryna)
Aminokwasy są podstawowymi jednostkami strukturalnymi białek. Aminokwas jest zbudowany z grup: karboksylowej, aminowej, atomu wodoru oraz charakterystycznych grup R, które wiążą się kowalencyjnie z atomem węgla, określanym jako węgiel alfa. Ten atom węgla nazwano alfa, gdyż sąsiaduje on z grupą karboksylową (kwaśną). Grupa R nazywana jest łańcuchem bocznym aminokwasu.
W roztworze o pH obojętnym aminokwasy występują zasadniczo w formie zjonizowanej, jako jony obojnacze (jony dwubiegunowe).
Równoczesna obecność w cząsteczkach aminokwasów grup karboksylowej i aminowej sprawia, że aminokwasy są związkami amfoterycznymi, których charakter uzależniony jest od stężenia jonów wodorowych w środowisku.
Aminokwas w postaci jonu obojnaczego (amfolitu) ma aminową (NH3+) oraz zjonizowaną grupę karboksylową (COO-). Ze zmianami pH roztworu zmienia się stan jonizacji cząsteczki aminokwasu.
W roztworze kwaśnym (np. w pH 1) cofa się dysocjacja grupy karboksylowej - staje się ona niezjonizowana (COOH). W roztworze zasadowym (np. pH 11) zjonizowana jest grupa karboksylowa (COO-), a niezjonizowana grupa aminowa (NH2).
Donorowe lub akceptorowe oddziaływanie aminokwasów z jonami H+, modyfikuje więc równowagę pomiędzy jonami obojnaczymi, kationami i anionami.
Istnieje takie pH roztworu, przy którym cząsteczki aminokwasów zachowują się tak, jak gdyby nie były naładowane (nie wędrują w polu elektrycznym). W rzeczywistości występują one w formie jonu obojnaczego, którego ładunek sumaryczny jest równy 0, czyli liczba ładunków ujemnych jest równa liczbie ładunków dodatnich. Takie pH nosi nazwę punktu izoelektrycznego (pI)
Tetraedryczne ułożenie czterech różnych podstawników wokół atomu węgla alfa, nadaje aminokwasom charakter związków optycznie czynnych. ( z wyjątkiem glicyny wszystkie, aminokwasy zawierają cztery różne podstawniki wokół węgla alfa). (W białkach L-aminokwasy)
Grupy funkcyjne aminokwasów - karboksylowa i aminowa - wykazują typowe dla nich reakcje, np. tworzenia soli, estryfikację, acetylację.
Aminokwasy:
Aminokwasy z alifatycznym łańcuchem bocznym:
Glicyna - Gly - G
Alanina - Ala - A
Walina - Val - V
Leucyna - Leu - L
Izoleucyna - Ile - IAminokwasy z łańcuchem bocznym zawierającym grupy hydroksylowe (OH):
Seryna - Ser - S
Treonina - Thr - T
Tyrozyna - Tyr - YIminokwasy:
Prolina - Pro - PAminokwasy z łańcuchem bocznym zawierającym atomy siarki:
Cysteina - Cys - C
Metionina - Met - MAminokwasy zawierające pierścień aromatyczny
Histydyna - His - H
Fenyloalanina - Phe - F
Tyrozyna - Tyr - T
Tryptofan - Trp - WAminokwasy z łańcuchem bocznym zawierającym gr. Kwaśne lub ich amidy:
Kwas asparaginowy - Asp - D
Asparagina - Asn - N
Kwas glutaminowy - Glu - E
Glutamina - Gln - QAminokwasy z łańcuchem bocznym zawierającym grupy zasadowe:
Arginina - Arg - R
Lizyna - Lys - K
Histydyna - His - H
Podziały kolejne:
Niepolarne Ile, Ala, Leu, Met, Phe, Pro, Trp, Val
Polarne pozostałe
Ketogenne Lys, Leu
Ketogenne i glukogenne Ile, Phe, Trp, Tyr
Glukogenne pozostałe 14
Egzogenne Ile, Lys, Leu, Met, His, Phe, Thr, Trp, Val
Endogenne pozostałe
Niezbędne (Essential) Arg, His, Ile, Leu, Lys, Met, Phe, Thr, Trp, Val
Nie niezbędne (Nonessential) Ala, Asp, Asn, Cys, Glu, Gln, Gly, Pr, Ser, Tyr
Aminokwasy z ładunkiem “+” Arg, His, Lys
Aminokwasy z ładunkiem ujemnym „-„ Asp, Glu
Hydrofobowe Ala, Leu, Met, Phe, Trp, Tyr, Val
Glukogenne aminokwasy to takie, które mogą być substratami w szlaku glukoneogenezy, odpowiedzialnym za syntezę glukozy z niecukrowych prekursorów
Egzogenne aminokwasy to takie, których przemiany dostarczają b-ketokwas - acetooctan, który jest prekursorem ciał ketonowych. Spontaniczna dekarboksylacja acetooctanu dostarcza aceton, natomiast redukcja acetooctanu przekształca go w 3--hydroksymaślan.
Selenocysteina, 21.L-alfa-aminokwas <Harper rozdz. 3 `Aminokwasy i peptydy'>
Jest to aminokwas wykrywany w niektórych białkach. Jej nazwa wskazuje, że atom seleu zastępuje siarkę w jej strukturalnym analogu - cysteinie. Wartość pK3 selenocysteiny 5,2 , jest o 3 jednostki mniejsza niż cysteiny. Z uwagi na fakt, że selenocysteina jest wbudowywana do polipeptydów podczas translacji, jest ona powszechnie nazywana „21. - aminokwasem”. W przeciwieństwie jednak do pozostałych 20 aminokwasów kodowanych genetycznie, selenocysteina nie jest określona przez trzyliterowy kodon. (więcej o niej w rodziale 27 Harpera „Biosynteza aminokwasów, które nie muszą być dostarczane z pożywienia”)
60.Biosynteza kwasów tłuszczowych. ( Stryer,strony rozdział 24 - metabolizm kwasów tł.)
Synteza ma miejsce w cytozolu. (rozkład w matriks)
Związki pośrednie (intermediaty) biosyntezy kwasów tłuszczowych są kowalencyjnie związane z grupą hydrosulfidową białkowego nośnika grup acylowych (ACP), podczas gdy produkty pośrednie rozkładu kwasów tłuszczowych wiązą się z koenzymem A.
Enzymy uczestniczące u wyższych organizmów w biosyntezie kwasów tłuszczowych są połączone w jeden łańcuch polipeptydowy, zwany syntazą kwasów tłuszczowych. Natomiast enzymy degradujące nie występują w formie zdysocjowanych.
Rosnacy łańcuch kwasu tłuszczowego ulega elongacji przed kolejne dobudowywanie jednostek dwuwęglowych pochądzących z acetylo-CoA. Aktywowanym donorem tych jednostek podczas elongacji łanńcuchów jest malonylo-ACP. Reakcja elongacji przebiega kosztem energii powstającej przez uwolnienie CO2.
W syntezie kwasów tłuszczowych związkiem redukującym jest NADPH, natomiast podczas degradacji kwasów tłuszczowych w utlenianiu bierze udział NAD+ i FAD.
Elongacja katalizowana przez kompleks sytnazy kwasów tłuszczowych zatrzymuje się po zsyntetyzowaniu łańcucha 16-węglowego - palmitynianu. Do dalszej elongacji oraz syntezy podwójnych wiązań potrzeba udziału innych układów enzymatycznych.
Synteza kwasu tłuszczowego rozpoczyna się od karboksylacji acetylo-CoA, co prowadzi do uzyskania malonylo-CoA. Ta nieodwracalna reakcja jest decydującym etapem w całej syntezie. Katalizowana karboksylazą acetylo-CoA (enzym ten zawiera biotynę jako gr. Prostetyczną). Reakcja przebiega dwuetapowo. W pierwszym etapie kosztem ATP powstaje karboksybiotyna. Aktywowana grupa CO2 ulega następnie przeniesieniu z tego intermediatu na acetylo-CoA, dając malonylo-CoA.
biotyna-enzym + ATP + HCO3-- < -- > CO2~biotyna-enzym + ADP + Pi
CO2~biotyna-enzym + acetylo-CoA --> malonylo-CoA + biotyna-enzym
Intermediaty w syntezie kwasów tłuszczowych wiązą się z białkowym nośnikiem grup acylowych (ACP).
Faza elongacji :
Rozpoczyna się od utworzenia acetylo-ACP i malonylo-ACP z acetylo-CoA i malonylo-CoA. Reakcję katalizuje transacylaza acetylowa i transacylaza malonylowa.Substancją wyjściową do syntezy kwasów tłuszczowych o nieparzystej liczbie atomów węgla jest propionylo-ACP, powstający z propionylo-CoA
Acetylo-ACP reaguje z malonylo-ACP, dając acetoacetylo-ACP. Reakcję kondeksacji katalizuje enzym kondensujący acylomalonylo-ACP (syntaza 3-oksoacylo-ACP).
W reakcji kondensacji tych cząsteczek powstaje jednostka czterowęglowa (acetyloacylo-ACP) i uwaniany jest CO2. W reakcji nie biorą udziału 2 cząsteczki acetylo-ACP (a mogłyby), bo stała równowagi była by bardzo niekorzystna. Korzystna równowaga reakcji ustala się natomiast jeśli jednym z substratów jest malonylo-ACP, ponieważ jego dekarboksylacja zmniejsza w instotny sposób energię swobodną układu.
Następne 3 etapy w syntezie kwasu tłuszczowego polegają na redukcji grupy ketonowej (przy C-3) do grupy metylenowej. [Schemat 24-13,rozdział 24]
Najpierw acetoacetylo-ACP ulega redukcji do D-3-hydroksybutyrylo-ACP. Później D-3-hydroksybutyrylo-ACP ulega dehydratacji do krotonylo-ACP,a na koniec krotonylo-ACP jest redukowany do butyrylo-ACP. Reduktorem w tych reakcjach jest NADPH.
W drugim cyklu suntezy kwasów tł. Butyrylo-ACP kondensuje z malonylo-ACP, tworząc C6-β-ketoacylo-ACP. Redukcja, dehydratacja i ponowna redukcja przekształcają C6-β-ketoacylo-ACP w C6-acylo-ACP, który może rozpocząć trzeci cykl elongacji. Cykle elongacyjne są powtarzane aż do uzyskania C16-acylo-ACP, który już nie jest substratem dla enzymu kondensującego i najczęściej ulega hydrolizie do palmitynianu i ACP.
Stechiometria syntezy kwasów tłuszczowych :
8 acetylo-CoA + 7 ATP + 14 NADPH + 6 H+ --> palmitynian + 14 NADP+ + 8 CoA + 7 ADP + 7 Pi
W komórkach eukariotycznych kwasy tłuszczowe są syntetyzowane z udziałem różnych enzymów stanowiących kompleks wielofunkcyjny.
Syntaza kwasów tłuszczowych u ssaków jest dimerem zudowanym z dwóch jednakowych podjednostek o masie po 260kDa. Każdy łańcuch jest tak zwinięty, że zawiera 3 funkcjonalne domeny, które są połączone elastycznymi rejonami.
Domena 1 : odpowiedzialna za wejście substratów i ich kondensację; zawiera aktywność transferazy acetylowej (AT), transferazy malonylowej (MT) oraz syntazy β-ketoacylowej.
Domena 2 : odpowiedzialna za redukcję; zawiera białkowy nośnik grup acylowych (ACP), reduktazę ketoacylową (KR), dehydratazę (DH) oraz reduktazę enoilową (ER).
Domena 3 : uwalnia palmitynian; zawiera tioesterazę (TE).
Dokładny opis tego co się po kolei przyłącza w syntezie - Stryer,rozdział 24, pod nagłówkiem 'Elastyczna grupa fosfopantoteinowa w ACP przenosi substraty z jednego miejsca aktywnego do drugiego'
Do syntezy palmitynianu potrzebne jest 8 cząsteczek acetylo-CoA. Musi być on przeniesiony z mitochondriów do cytozolu (tam zachodzi proces syntezy!!).Mitochondria nie są łatwo przepuszczalne dla acetylo-CoA, ale barierę tą omija cytrynian, który przenosi grupy acetylowe przez wewnętrzną błonę mitochondrialną.
Źródłami NADPH dla syntezy kwasów tłuszczowych są : szlak pentozofosforanowy, a także przenoszenie cząsteczek acetylo-CoA z mitochondriów do cytozolu (podczas przeniesienia każdej cząsteczki wytwarza się jedna cząsteczka NADPH).
Pyt. 67
Właściwości spektralne kwasów nukleinowych. <Kłyszejko str. >
Pyt. 68
β-oksydacja kwasów tłuszczowych.
Pyt. 69
Ilościowe oznaczanie białka metoda biuretową.
samoodnowa
Komórka
KLON
Wyszukiwarka
Podobne podstrony:
ZAGADNIENIA EGZAMINACYJNE 2010 biochemia, Analityka semestr IV, Biochemia
biochemia 2009, Analityka semestr IV, Biochemia, Biochemia
42 opis, Analityka semestr IV, Biochemia, Biochemia
biochemia (1) egzam giełda, Analityka semestr IV, Biochemia, Biochemia
Wersja A-TEST, Analityka semestr IV, Biochemia, tłuszcze kwasy nukleinowe
biochemia kolo III, Analityka semestr IV, Biochemia, tłuszcze kwasy nukleinowe
biochemia egzamin wersja B, Analityka semestr IV, Biochemia, giłdy edz bioch
kolokwium III, Analityka semestr IV, Biochemia, Biochemia
poprawa enzymy pytania, Analityka semestr IV, Biochemia, Biochemia
koo cukry biochemia[1], Analityka semestr IV, Biochemia, Biochemia
DENATURACJA, Analityka semestr IV, Biochemia, Biochemia
cukry, Analityka semestr IV, Biochemia, Biochemia
Kwas fusydowy, Analityka semestr IV, Biochemia, Biochemia
biochemia, Analityka semestr IV, Biochemia, Biochemia
PracowniaBiochem, Analityka semestr IV, Biochemia, Biochemia
biochemia 2009, Analityka semestr IV, Biochemia, Biochemia
zadania chrom, Analityka semestr IV, Analiza Instumentalna
więcej podobnych podstron