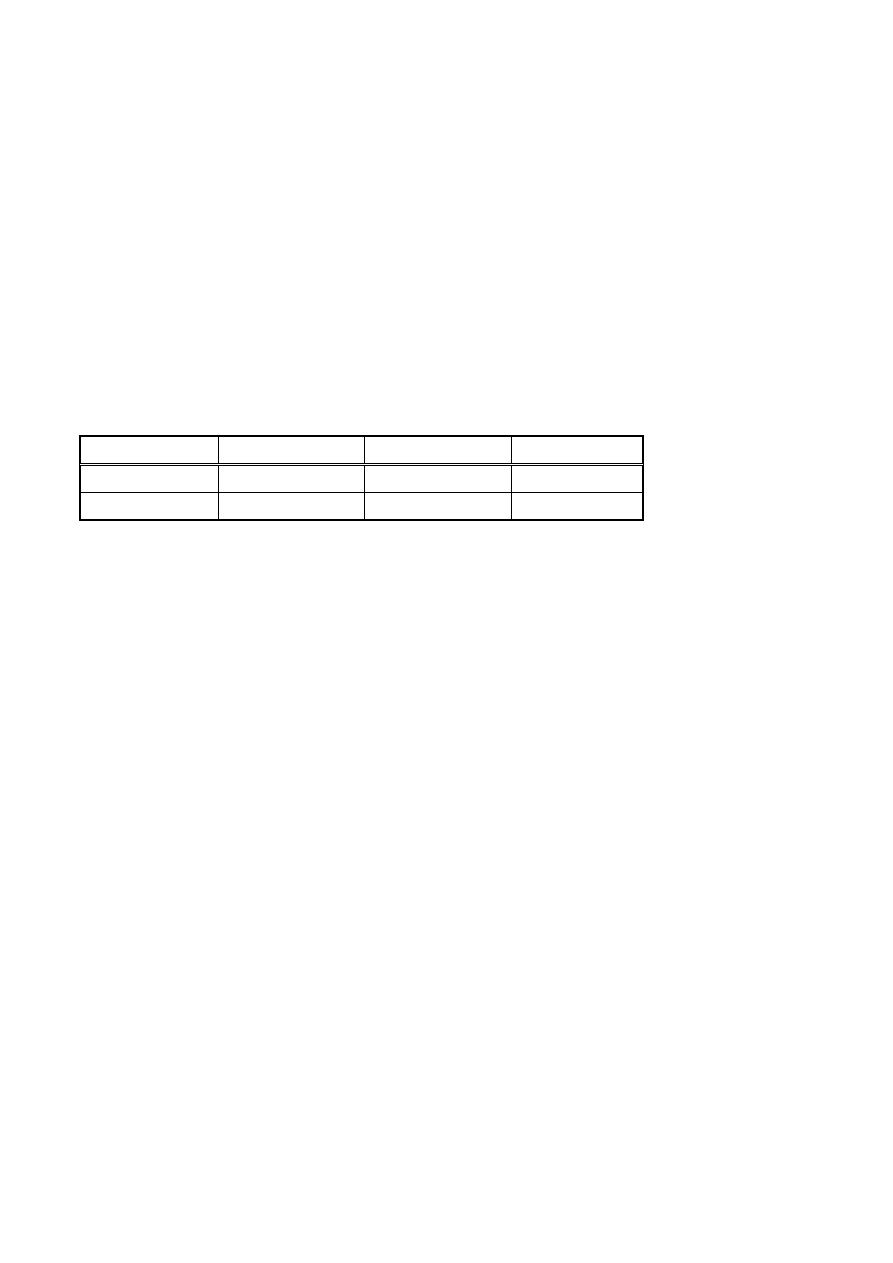
1
ćwiczenie 8
PRZENOSZENIE DNA I RNA NA MEMBRANY HYBRYDYZACYJNE.
Technika
hybrydyzacji
umożliwia identyfikację określonych sekwencji kwasów nukleinowych w badanym
materiale genetycznym. W tym celu wykorzystuje się zdolność sond hybrydyzacyjnych, czyli określonych cząsteczek
DNA lub RNA, do wiązania z homologicznymi sekwencjami obecnymi w materiale badanym. Sonda hybrydyzacyjna
przed użyciem powinna zostać wyznakowana i zdenaturowana do formy jednoniciowej. Ponieważ hybrydyzacja
pomiędzy sondą a materiałem badanym zachodzi nawet wówczas, gdy pomiędzy tymi cząsteczkami nie ma pełnej
homologii, dlatego sondami mogą być np.: oligonukleotydy przygotowane na podstawie sekwencji aminokwasowej
badanego białka, homologi danego genu uzyskane z innego gatunku, cDNA badanego genu w przypadku hybrydyzacji
z DNA genomowym lub z RNA itp. Badany DNA lub RNA, przed hybrydyzacją z sondą, powinien zostać przeniesiony
na odpowiednią membranę, tzw. filtr hybrydyzacyjny w postaci jednoniciowej. Do hybrydyzacji na ogół stosowane są
dwa typy membran: nitrocelulozowe i nylonowe. Zasadnicza różnica w ich wykorzystaniu polega na tym, że DNA
umieszczony na nitrocelulozie poddać można hybrydyzacji tylko jeden raz. DNA na membranie nylonowej po
hybrydyzacji można poddać odhybrydyzowaniu (usunięcie pierwszej sondy) i kolejnej hybrydyzacji z inną sondą.
Wśród wielu wariantów hybrydyzacji najczęściej wykonywane są techniki: southern blot, northern blot, dot blot,
hybrydyzacja kolonijna i mikromacierze.
Tab. 1 Bufory używane do przenoszenia kwasów nukleinowych na membrany hybrydyzacyjne. Uwaga! Przed
przystąpieniem do eksperymentu należy sprawdzić, jakie bufory zaleca użyć producent stosowanych membran.
Buf. depurynujący Buf.
denaturujący Buf.
neutralizujący
20 X SSC
0,25M HCl
0,5M NaOH
0,5M Tris-HCl pH7,5 0,3M cytrynian Na
1,5M
NaCl
1,5M NaCl
3,0M NaCl
Southern blot
Technika Southern blot wykorzystywana jest najczęściej do identyfikacji mapy restrykcyjnej danej sekwencji, np.
określonego genu, w genomie badanego organizmu. Metodę Southern’a wykorzystuje się również podczas analizy
RFLP (ang. restriction fragment length polymorphism), która służy do wykrywania polimorfizmu pomiędzy różnymi
organizmami.
Metoda Southern’a obejmuje następujące etapy:
1. Trawienie DNA enzymami restrykcyjnymi. W przypadku organizmów eukariotycznych, w celu identyfikacji genu
występującego w jednej kopii na genom, należy użyć około 10
μg DNA całkowitego.
2. Rozdział elektroforetyczny DNA w żelu agarozowym. Oprócz prób badanych, należy również pamiętać o
nałożeniu na żel prób kontrolnych (+ i -) oraz markera mas cząsteczkowych. Ich obecność jest niezbędna do
prawidłowej interpretacji wyniku. Czas trwania elektroforezy powinien być uzależniony od przewidywanej ilości
sygnałów hybrydyzacyjnych. W przypadku pojedynczego sygnału, wystarczy krótki rozdział, np. 5-10cm. Jeżeli
spodziewamy się uzyskać większą ilość sygnałów, należy przedłużyć elektroforezę w celu uzyskania lepszego
rozseparowania prążków.
3. Depurynacja, denaturacja i neutralizacja DNA. Etapy te wykonuje się po zakończeniu elektroforezy. Celem
depurynacji (0,25M HCl) jest fragmentacji dłuższych odcinków DNA, co ułatwia ich późniejsze przeniesienie na
membranę. Dzięki prowadzonej w warunkach alkalicznych denaturacji (0,5 M NaOH), dwuniciowe cząsteczki
DNA denaturują do formy jednoniciowej, która może podlegać hybrydyzacji z sondą. Neutralizacja wykonana po
denaturacji (0,5 M Tris pH 8,5) przywraca obojętne pH żelu, co ułatwia wiązanie DNA z membraną.
4. Przenoszenie kwasów nukleinowych na membranę nitrocelulozową lub nylonową. Transfer DNA na membrany
przeprowadza się w buforze 20 x SSC lub buforze fosforanowym. Stosuje się w tym celu jedną z trzech technik:
transfer kapilarny, próżniowy lub elektrotransfer. DNA po przeniesieniu na membranę powinien zostać na niej
utrwalony. Proces ten, polegający na wytwarzaniu wiązań kowalencyjnych pomiędzy DNA a membraną, zachodzi
podczas naświetlania UV lub ekspozycji w +80
o
C.
5. Hybrydyzacja DNA związanego na membranie z sondą DNA.
Postępowanie.
1. Przygotować 0,8% żel agarozowy z bromkiem
etydyny.
2. Nałożyć na żel badane próby, zawierające po ok.
10
μg DNA, trawionego enzymami restrykcyjnymi,
oraz marker wielkości DNA.
3. Prowadzić elektroforezę przy napięciu ok. 100V.
4. Wykonać dokumentację fotograficzną obrazu
elektroforetycznego, odciąć puste fragmenty żelu a
część przeznaczoną do transferu dokładnie zmierzyć.
5. Przeprowadzić kolejno depurynacje (25mM HCl, 7
min.) a następnie denaturację i neutralizację, płucząc
żele w odpowiednich buforach, po 15 min w temp.
pokojowej.

2
6. W czasie wykonywania etapów opisanych w p.5
przygotować:
− 2 bibuły Whatman 3MM o wymiarach
umożliwiających przepływ kapilarny buforu z kuwety
do żelu,
− 4 kawałki bibuły Whatman 3MM o wymiarach żelu,
− stos papieru lub ligniny o wymiarach żelu,
− membranę (nitrocelulozową lub nylonową) przyciętą
dokładnie do rozmiarów żelu, Uwaga, membran nie
wolno dotykać bez rękawiczek!
7. Umieścić w kuwecie stolik do transferu a następnie
przykryć go dwiema bibułami w taki sposób, aby
mogły one zasysać bufor 20xSSC z kuwety do żelu.
8. Umieścić żel na stoliku do transferu tak, aby unikać
pęcherzyków powietrza pomiędzy żelem i bibułą.
9. Przykryć żel membraną hybrydyzacyjną.
10. Na membranę nałożyć 2 bibuły Whatmana nasycone
buforem 20xSSC i dwie bibuły suche.
11. Nałożyć stos materiału ssącego i przycisnąć całość
ciężarkiem ok. 0,5 kg.
12. Prowadzić transfer przez ok. 16h.
13. Po zakończeniu transferu zdjąć stos bibuły ssącej, a
na membranie zaznaczyć pozycję kieszonek i
orientację żelu.
14. Żel wybarwić bromkiem etydyny i obejrzeć w świetle
UV, w celu sprawdzenia efektywności przeniesienia
DNA.
15. Membranę po wysuszeniu inkubować 2 godz. w
+80
o
C lub poddać naświetlaniu UV, w celu
utrwalenia znajdującego się na niej DNA.
Northern Blot
Technika Northern blot wykorzystywana jest do analizy procesu transkrypcji poszczególnych genów. Dzięki niej
można ustalić, które geny ulegają transkrypcji w badanych tkankach lub na danym etapie rozwoju organizmu. Ponadto,
technika ta umożliwia poznanie długości RNA badanych genów a także intensywności z jaką są one transkrybowane w
różnych układach biologicznych.
Metoda Northern Blott obejmuje następujące etapy:
1. Denaturację RNA i jego rozdział w denaturującym żelu agarozowym, np. w obecności aldehydu mrówkowego lub
glioksalu.
2. Przeniesienie RNA z żelu na membranę nitrocelulozową lub nylonową i jej utrwalenie. Cząsteczki RNA w
przeciwieństwie do dużych fragmentów DNA są przenoszone z żelu na membranę z dużą wydajnością, dlatego nie
ma konieczności ich uprzedniej fragmentacji.
3. Hybrydyzacja ze znakowaną sondą.
Postępowanie:
1. Przygotować 1,5 % żel agarozowy. W przypadku
żelu o objętości 50 ml, do 0,7g agarozy dodać do 42,5
ml wody i całość zagotować.
2. Po schłodzeniu agarozy do ok. +65
o
C dodać 5 ml 10x
MOPS oraz 2,5 ml 37% formaldehydu a następnie
całość wylać do uprzednio przygotowanego aparatu
elektroforetycznego. Uwaga - wszystkie te
czynności wykonywać pod dygestorium przy
włączonym nawiewie. Opary formaldehydu są
trujące!
3. Do preparatu RNA dodać 1 obj. buforu
denaturującego do RNA. Następnie całość
denaturować przez 5 min w +65
o
C.
4. Schłodzić preparat w lodzie a następnie dodać 2
μl
buforu LB i całość nałożyć na żel denaturujący.
5. Elektroforezę prowadzimy w buforze 1x MOPS pH
7,0 przy napięciu 4 V/cm.
6. Transfer wykonujemy analogicznie jak przy metodzie
Southern blot.
7. Membranę płukać krótko w 2xSSC. RNA utrwalić na
membranie inkubując ją 2 godz. w +80
0
C lub
naświetlając w świetle UV.
8. Żel po transferze wybarwić w bromku etydyny i
sprawdzić na lampie UV czy cały RNA uległ
przeniesieniu.
Dot-blot i hybrydyzacja kolonijna
Techniki Dot-blot i hybrydyzacja kolonijna wykorzystywane są na ogół do przeglądanie bibliotek genomowych i
cDNA, w celu identyfikacji klonów kodujących poszczególne geny. W przypadku techniki dot blot, na membranę
hybrydyzacyjną nakrapiany jest bezpośrednio oczyszczony DNA badanych klonów. W przypadku hybrydyzacji
kolonijnej na membranie umieszcza się kolonie bakteryjne zawierające poszczególne klony.
Przygotowanie membrany typu dot - blot.
1. Wyciąć fragment membrany nylonowej lub
nitrocelulozowej a następnie zaznaczyć na niej
ołówkiem miejsca nakrapiania DNA.
2. Na membranę nakropić DNA małymi porcjami,
susząc ją na bieżąco.
3. Po nakropieniu, DNA zdenaturować przez
umieszczenie membrany na 5 min. na bibule
nasączonej roztworem denaturującym.
4. Po denaturacji zneutralizować membranę w
roztworze neutralizującym przez 1 min.
5. Membranę wysuszyć na powietrzu.
6. Utrwalić DNA na membranie inkubując ją 2 godz. w
+80
0
C lub naświetlając w świetle UV.

3
Przygotowanie membrany do hybrydyzacji kolonijnej.
1. Wyciąć fragment membrany o formacie
odpowiadającym wielkości szalki z koloniami
przeznaczonymi do transferu.
2. Nałożyć membranę na szalkę z koloniami tak, aby
mogły one odcisnąć się na jej powierzchni,
ewentualnie przenieść indywidualnie poszczególne
kolonie na membranę oraz na szalkę dublującą w
zaznaczonych pozycjach.
3. Przenieść membranę z koloniami na bibułę
nasączoną 10% roztworem SDS w celu
przeprowadzenia lizy bakterii. Bakterie lizować przez
3 min.
4. Przenieść membranę na suchą bibułę celem
odsączenia resztek roztworu SDS a następnie
umieścić ją na 5 min. na następnej bibule, nasączonej
buf. denaturującym.
5. Po denaturacji przeprowadzić w analogiczny sposób
neutralizację i płukanie w roztworze 2x SSC.
6. Membranę wysuszyć na powietrzu.
7. Utrwalić DNA na membranie inkubując ją 2 godz. w
+80
0
C lub naświetlając w świet

4
Wyszukiwarka
Podobne podstrony:
DNA i RNA, Biochemia, Zagadnienia na kolokwia
Przenoszenie formatu z komórki na komórkę, excel
DNA i RNA cw 7
nowotwory,DNA,RNA,replikacja
NUKLEOTYDY DNA i RNA
1 Struktura i funkcja DNA i RNA Ekspresja genów
Struktura DNA i RNA 1
DNA a RNA porównanie, BIOLOGIA(1)
Biochemia Wykład VII 9 01 15 r Kwasy nukleinowe, DNA, RNA
Analizy molekularne DNA i RNA w wykrywaniu
DNA RNA
DNA RNA
Lekcja 2 DNA i RNA i reszta
mapy myśli DNA i RNA
DNA i RNA - materiały do koła, BIOLOGIA(1)
W 5 Izolacja RNA, RT PCR, hybrydyzacja 1
DNA RNA gra dydaktyczna
więcej podobnych podstron