Konspekt - Diagnostyka molekularna w medycynie
II Rok wydziału lekarskiego
1. Izolacja DNA
Ź
ródła DNA:
- krew obwodowa (limfocyty)
- komórki płynu owodniowego (AFC)
- komórki kosmówki (CVS)
- hodowle fibroblastów
- komórki epitelialne
- cebulki włosów
- plamy krwi, nasienia
- fragmenty tkanek
- szpik kostny
Metody izolacji DNA
:
1
1
.
.
I
I
z
z
o
o
l
l
a
a
c
c
j
j
a
a
D
D
N
N
A
A
z
z
z
z
a
a
s
s
t
t
o
o
s
s
o
o
w
w
a
a
n
n
i
i
e
e
m
m
f
f
e
e
n
n
o
o
l
l
u
u
i
i
c
c
h
h
l
l
o
o
r
r
o
o
f
f
o
o
r
r
m
m
u
u
c
c
e
e
l
l
e
e
m
m
o
o
d
d
b
b
i
i
a
a
ł
ł
c
c
z
z
e
e
n
n
i
i
a
a
p
p
r
r
e
e
p
p
a
a
r
r
a
a
t
t
ó
ó
w
w
2
2
.
.
I
I
z
z
o
o
l
l
a
a
c
c
j
j
a
a
D
D
N
N
A
A
p
p
r
r
z
z
e
e
b
b
i
i
e
e
g
g
a
a
j
j
ą
ą
c
c
a
a
p
p
r
r
z
z
e
e
z
z
w
w
y
y
s
s
o
o
l
l
e
e
n
n
i
i
e
e
b
b
i
i
a
a
ł
ł
e
e
k
k
z
z
l
l
i
i
z
z
a
a
t
t
ó
ó
w
w
k
k
o
o
m
m
ó
ó
r
r
e
e
k
k
3
3
.
.
I
I
z
z
o
o
l
l
a
a
c
c
j
j
a
a
D
D
N
N
A
A
p
p
o
o
p
p
r
r
z
z
e
e
z
z
z
z
w
w
i
i
ą
ą
z
z
a
a
n
n
i
i
e
e
z
z
n
n
o
o
ś
ś
n
n
i
i
k
k
i
i
e
e
m
m
,
,
o
o
d
d
m
m
y
y
c
c
i
i
e
e
z
z
a
a
n
n
i
i
e
e
c
c
z
z
y
y
s
s
z
z
c
c
z
z
e
e
ń
ń
i
i
u
u
w
w
o
o
l
l
n
n
i
i
e
e
n
n
i
i
e
e
D
D
N
N
A
A
I
I
z
z
o
o
l
l
a
a
c
c
j
j
a
a
D
D
N
N
A
A
z
z
k
k
r
r
w
w
i
i
o
o
b
b
w
w
o
o
d
d
o
o
w
w
e
e
j
j
–
–
m
m
e
e
t
t
o
o
d
d
ą
ą
w
w
y
y
s
s
a
a
l
l
a
a
n
n
i
i
a
a
b
b
i
i
a
a
ł
ł
e
e
k
k
I
I
z
z
o
o
l
l
a
a
c
c
j
j
a
a
D
D
N
N
A
A
z
z
k
k
r
r
w
w
i
i
o
o
b
b
w
w
o
o
d
d
o
o
w
w
e
e
j
j
o
o
b
b
e
e
j
j
m
m
u
u
j
j
e
e
k
k
i
i
l
l
k
k
a
a
e
e
t
t
a
a
p
p
ó
ó
w
w
:
:
1
1
.
.
L
L
i
i
z
z
a
a
k
k
o
o
m
m
ó
ó
r
r
e
e
k
k
b
b
e
e
z
z
j
j
ą
ą
d
d
r
r
z
z
a
a
s
s
t
t
y
y
c
c
h
h
2
2
.
.
L
L
i
i
z
z
a
a
l
l
e
e
u
u
k
k
o
o
c
c
y
y
t
t
ó
ó
w
w.
3
3
.
.
O
O
d
d
b
b
i
i
a
a
ł
ł
c
c
z
z
a
a
n
n
i
i
e
e
p
p
o
o
p
p
r
r
z
z
e
e
z
z
i
i
n
n
k
k
u
u
b
b
a
a
c
c
j
j
e
e
z
z
p
p
r
r
o
o
t
t
e
e
i
i
n
n
a
a
z
z
ą
ą
K
K
.
.
4
4
.
.
W
W
y
y
t
t
r
r
ą
ą
c
c
a
a
n
n
i
i
e
e
D
D
N
N
A
A
alkoholem
Główne kierunki analizy DNA
Genomowy DNA
Hybrydyzacja molekularna
Southern Blot
Northern Blot
DNA Fingerprinting
FISH (zastosowanie w cytogenetyce)
Amplifikacja DNA
Klonowanie
Amplifikacja in-vitro
PCR
Klonowanie DNA w
wektorach
Analiza sekwencji DNA

2. Hybrydyzacja (renaturacja) –
Tworzenie podwójnej helisy między komplementarnymi niciami DNA lub RNA pochodzącymi z
różnych źródeł. Oddziaływanie badanego fragmentu DNA i sondy molekularnej prowadzi do
utworzenia hybrydu (dupleksu) o dwuniciowej strukturze.
Metody hybrydyzacji molekularnej:
-Southern Blotting,
-DNA fingerprinting,
-Northern blotting,
-In situ hybridization (FISH).
S
S
o
o
n
n
d
d
ą
ą
m
m
o
o
l
l
e
e
k
k
u
u
l
l
a
a
r
r
n
n
ą
ą
m
m
o
o
g
g
ą
ą
b
b
y
y
ć
ć
:
:
-
-
s
s
y
y
n
n
t
t
e
e
t
t
y
y
c
c
z
z
n
n
e
e
o
o
l
l
i
i
g
g
o
o
m
m
e
e
r
r
y
y
-
-
s
s
y
y
n
n
t
t
e
e
t
t
y
y
c
c
z
z
n
n
e
e
b
b
ą
ą
d
d
ź
ź
n
n
a
a
t
t
u
u
r
r
a
a
l
l
n
n
e
e
g
g
e
e
n
n
y
y
l
l
u
u
b
b
i
i
c
c
h
h
f
f
r
r
a
a
g
g
m
m
e
e
n
n
t
t
y
y
-
-
c
c
D
D
N
N
A
A
-
-
f
f
r
r
a
a
g
g
m
m
e
e
n
n
t
t
y
y
c
c
h
h
r
r
o
o
m
m
o
o
s
s
o
o
m
m
ó
ó
w
w
-
-
c
c
a
a
ł
ł
e
e
g
g
e
e
n
n
o
o
m
m
y
y
b
b
a
a
k
k
t
t
e
e
r
r
y
y
j
j
n
n
e
e
S
S
o
o
n
n
d
d
a
a
m
m
o
o
l
l
e
e
k
k
u
u
l
l
a
a
r
r
n
n
a
a
m
m
u
u
s
s
i
i
b
b
y
y
ć
ć
z
z
d
d
e
e
n
n
a
a
t
t
u
u
r
r
o
o
w
w
a
a
n
n
a
a
(
(
j
j
e
e
d
d
n
n
o
o
n
n
i
i
c
c
i
i
o
o
w
w
a
a
)
)
.
.
Ł
Ł
a
a
c
c
z
z
e
e
n
n
i
i
e
e
s
s
o
o
n
n
d
d
y
y
m
m
o
o
l
l
e
e
k
k
u
u
l
l
a
a
r
r
n
n
e
e
j
j
z
z
d
d
o
o
c
c
e
e
l
l
o
o
w
w
y
y
m
m
f
f
r
r
a
a
g
g
m
m
e
e
n
n
t
t
e
e
m
m
k
k
w
w
a
a
s
s
u
u
n
n
u
u
k
k
l
l
e
e
i
i
n
n
o
o
w
w
e
e
g
g
o
o
z
z
a
a
c
c
h
h
o
o
d
d
z
z
i
i
z
z
g
g
o
o
d
d
n
n
i
i
e
e
z
z
k
k
o
o
m
m
p
p
l
l
e
e
m
m
e
e
n
n
t
t
a
a
r
r
n
n
o
o
ś
ś
c
c
i
i
ą
ą
z
z
a
a
s
s
a
a
d
d
.
.
S
S
o
o
n
n
d
d
a
a
j
j
e
e
s
s
t
t
w
w
y
y
z
z
n
n
a
a
k
k
o
o
w
w
a
a
n
n
a
a
r
r
a
a
d
d
i
i
o
o
a
a
k
k
t
t
y
y
w
w
n
n
i
i
e
e
l
l
u
u
b
b
n
n
i
i
e
e
r
r
a
a
d
d
i
i
o
o
a
a
k
k
t
t
y
y
w
w
n
n
i
i
e
e
n
n
p
p
.
.
f
f
l
l
u
u
o
o
r
r
e
e
s
s
c
c
e
e
n
n
c
c
y
y
j
j
n
n
i
i
e
e
.
.
Hybrydyzacja typu Southern (ang. Southern blot hybridization) –
Stosowana najczęściej, dotyczy hybrydyzacji DNA-DNA. Ma na celu wyszukanie sekwencji DNA
spośród mieszaniny fragmentów otrzymanych po trawieniu enzymami restrykcyjnymi.
Stosując hybrydyzację typu Southern można wykryć:
- rearanżacje genowe np. delecje albo insercje, większe od 50-100 pz
-mutacje punktowe, które tworzą nowe lub zmieniają dotychczasowe miejsce restrykcyjne dla enzymu,
stosowanego do trawienia DNA
Hybrydyzacja typu Northern Blot (ang. Northern blot hybridization) –
dotyczy
hybrydyzacji RNA pacjenta z sondą molekularną. Stosowana w celu badania ekspresji genów.
D
D
N
N
A
A
F
F
i
i
n
n
g
g
e
e
r
r
p
p
r
r
i
i
n
n
t
t
i
i
n
n
g
g
W metodzie tej wykorzystuje się obecność w genomie polimorficznych sekwencji powtórzonych VNTR
(variable number of tandem repeats) – zmienna liczba tandemowych powtórzeń. W wyniku
hybrydyzacji VNTR z sondami molekularnymi rozpoznającymi jednocześnie kilkanaście loci otrzymuje
się dla każdego osobnika charakterystyczny obraz fragmentów DNA tzw. fingerprint, odcisk
genetyczny
Zastosowanie:
-ustalanie ojcostwa
-badania medyczno-sądowe
3. Klonowanie DNA:
1. Klonowanie DNA w wektorach
2. Klonowanie DNA in-vitro PCR
Ad.1
E
E
t
t
a
a
p
p
y
y
k
k
l
l
o
o
n
n
o
o
w
w
a
a
n
n
i
i
a
a
D
D
N
N
A
A
w
w
m
m
i
i
k
k
r
r
o
o
o
o
r
r
g
g
a
a
n
n
i
i
z
z
m
m
a
a
c
c
h
h
:
:
-
-
C
C
i
i
ę
ę
c
c
i
i
e
e
e
e
n
n
z
z
y
y
m
m
e
e
m
m
r
r
e
e
s
s
t
t
r
r
y
y
k
k
c
c
y
y
j
j
n
n
y
y
m
m
D
D
N
N
A
A
-
-
Ł
Ł
ą
ą
c
c
z
z
e
e
n
n
i
i
e
e
f
f
r
r
a
a
g
g
m
m
e
e
n
n
t
t
ó
ó
w
w
D
D
N
N
A
A
z
z
w
w
e
e
k
k
t
t
o
o
r
r
e
e
m
m
-
-
W
W
p
p
r
r
o
o
w
w
a
a
d
d
z
z
e
e
n
n
i
i
e
e
w
w
e
e
k
k
t
t
o
o
r
r
ó
ó
w
w
d
d
o
o
k
k
o
o
m
m
ó
ó
r
r
e
e
k
k
-
-
P
P
o
o
w
w
i
i
e
e
l
l
a
a
n
n
i
i
e
e
D
D
N
N
A
A
w
w
k
k
o
o
m
m
ó
ó
r
r
k
k
a
a
c
c
h
h
Stosowane wektory:
Fag M13 (1kpz)
Plazmid (5kpz)
Kosmid (40kpz)
Sztuczny chromosom bakteryjny BAC (150kpz)
Sztuczny chromosom drożdżowy YAC (1000kpz)
Ad.2
A
A
m
m
p
p
l
l
i
i
f
f
i
i
k
k
a
a
c
c
j
j
a
a
D
D
N
N
A
A
m
m
e
e
t
t
o
o
d
d
ą
ą
P
P
C
C
R
R
(
(
ł
ł
a
a
ń
ń
c
c
u
u
c
c
h
h
o
o
w
w
e
e
j
j
r
r
e
e
a
a
k
k
c
c
j
j
i
i
p
p
o
o
l
l
i
i
m
m
e
e
r
r
a
a
z
z
y
y
)
)
–
–
R
R
e
e
a
a
k
k
c
c
j
j
a
a
t
t
a
a
o
o
d
d
z
z
w
w
i
i
e
e
r
r
c
c
i
i
e
e
d
d
l
l
a
a
n
n
a
a
t
t
u
u
r
r
a
a
l
l
n
n
y
y
p
p
r
r
o
o
c
c
e
e
s
s
r
r
e
e
p
p
l
l
i
i
k
k
a
a
c
c
j
j
i
i
i
i
u
u
m
m
o
o
ż
ż
l
l
i
i
w
w
i
i
a
a
w
w
w
w
a
a
r
r
u
u
n
n
k
k
a
a
c
c
h
h
i
i
n
n
v
v
i
i
t
t
r
r
o
o
s
s
z
z
y
y
b
b
k
k
i
i
e
e
p
p
o
o
w
w
i
i
e
e
l
l
e
e
n
n
i
i
e
e
w
w
y
y
b
b
r
r
a
a
n
n
y
y
c
c
h
h
o
o
d
d
c
c
i
i
n
n
k
k
ó
ó
w
w
D
D
N
N
A
A
l
l
u
u
b
b
R
R
N
N
A
A
(
(
p
p
r
r
z
z
e
e
p
p
i
i
s
s
a
a
n
n
e
e
g
g
o
o
n
n
a
a
c
c
D
D
N
N
A
A
p
p
r
r
z
z
y
y
u
u
ż
ż
y
y
c
c
i
i
u
u
r
r
e
e
a
a
k
k
c
c
j
j
i
i
o
o
d
d
w
w
r
r
o
o
t
t
n
n
e
e
j
j
t
t
r
r
a
a
n
n
s
s
k
k
r
r
y
y
p
p
c
c
j
j
i
i
)
)
.
.
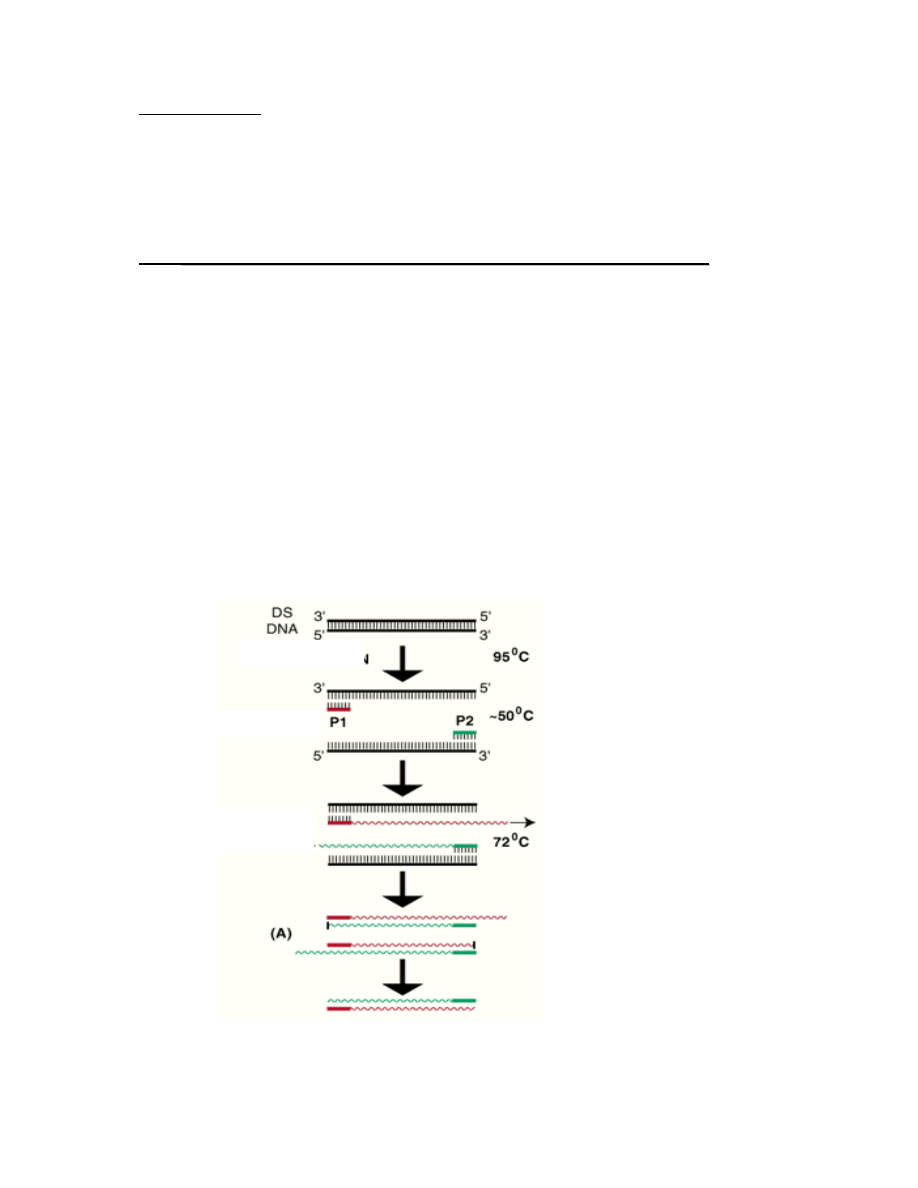
Badana sekwencja DNA jest powielana przy użyciu pary oligonukleotydowych starterów, z których
każdy jest komplementarny do jednego końca sekwencji docelowej DNA.
Startery te są wydłużane w kierunku przeciwnym, za pomocą termostabilnej polimerazy DNA, w cyklu
który obejmuje trzy podstawowe etapy:
- denaturacja dwuniciowej matrycy – 90-95°C,
- wiązanie starterów do jednoniciowej matrycy 50-65°C,
- synteza nici komplementarnej 68-72°C
Wizualizacja produktów PCR następuje poprzez rozdział elektroforetyczny. Cząsteczki DNA
umieszczone w żelu migrują w polu elektrycznym od ujemnie do dodatnio naładowanej elektrody, a ich
szybkość zależy od wielkości cząsteczki – im mniejsze tym szybciej będą się przesuwały w żelu.
Wybarwione bromkiem etydyny (mutagen interkalujący DNA) są widoczne po naświetleniu światłem
UV.
Na schemacie przedstawiono etapy reakcji PCR
Denaturacja
Wiązanie starterów
Synteza nici
komplementarne

PCR-RFLP (
analiza polimorfizmu długości fragmentów restrykcyjnych)
Polega ona na amplifikacji fragmentu DNA, w obrębie którego występuje marker RFLP (fragment DNA
zawierający miejsce restrykcyjne), a następnie jego trawieniu odpowiednim enzymem restrykcyjnym.
Zastosowanie:
- identyfikacja mutacji punktowych w obrębie miejsca rozpoznawanego przez enzym restrykcyjny
Przykład:
fenyloketonuria, anemia sierpowata, hemofilia
PCR-Multipleks
-
-
J
J
e
e
d
d
n
n
o
o
r
r
a
a
z
z
o
o
w
w
o
o
m
m
o
o
ż
ż
n
n
a
a
a
a
m
m
p
p
l
l
i
i
f
f
i
i
k
k
o
o
w
w
a
a
ć
ć
o
o
k
k
.
.
1
1
0
0
m
m
a
a
r
r
k
k
e
e
r
r
ó
ó
w
w
-
-
M
M
o
o
ż
ż
n
n
a
a
b
b
a
a
d
d
a
a
ć
ć
D
D
N
N
A
A
z
z
m
m
i
i
e
e
s
s
z
z
a
a
n
n
y
y
i
i
z
z
d
d
e
e
g
g
r
r
a
a
d
d
o
o
w
w
a
a
n
n
y
y
-
-
Z
Z
a
a
s
s
t
t
o
o
s
s
o
o
w
w
a
a
n
n
i
i
e
e
:
:
D
D
u
u
ż
ż
e
e
m
m
u
u
t
t
a
a
c
c
j
j
e
e
g
g
e
e
n
n
o
o
w
w
e
e
:
:
d
d
e
e
l
l
e
e
c
c
j
j
e
e
,
,
d
d
u
u
p
p
l
l
i
i
k
k
a
a
c
c
j
j
e
e
,
,
i
i
n
n
s
s
e
e
r
r
c
c
j
j
e
e
P
P
r
r
z
z
y
y
k
k
ł
ł
a
a
d
d
:
:
D
D
y
y
s
s
t
t
r
r
o
o
f
f
i
i
a
a
m
m
i
i
ę
ę
ś
ś
n
n
i
i
o
o
w
w
a
a
D
D
u
u
c
c
h
h
e
e
n
n
n
n
e
e
’
’
a
a
D
D
M
M
D
D
H
H
y
y
b
b
r
r
y
y
d
d
y
y
z
z
a
a
c
c
j
j
a
a
A
A
S
S
O
O
Hybrydyzacja z sondą specyficzną dla produktu PCR. Sonda rozpoznaje allel dziki (prawidłowy) lub
zmutowany.
Zastosowanie:
- wykrywanie mutacji punktowych, które nie zmieniają miejsc restrykcyjnych
Przykład:
Mukowiscydoza (cystic fibrosis)
Analiza metylacji w diagnostyce zespołu Pradera-Willego (PWS) i zespołu
Angelmana (AS)
U
U
p
p
o
o
d
d
s
s
t
t
a
a
w
w
p
p
a
a
t
t
o
o
g
g
e
e
n
n
t
t
y
y
c
c
z
z
n
n
y
y
c
c
h
h
P
P
W
W
S
S
z
z
n
n
a
a
j
j
d
d
u
u
j
j
e
e
s
s
i
i
ę
ę
r
r
e
e
g
g
u
u
l
l
a
a
c
c
j
j
a
a
e
e
k
k
s
s
p
p
r
r
e
e
s
s
j
j
i
i
g
g
e
e
n
n
ó
ó
w
w
n
n
a
a
z
z
y
y
w
w
a
a
n
n
a
a
r
r
o
o
d
d
z
z
i
i
c
c
i
i
e
e
l
l
s
s
k
k
i
i
m
m
p
p
i
i
ę
ę
t
t
n
n
e
e
m
m
g
g
e
e
n
n
o
o
m
m
o
o
w
w
y
y
m
m
.
.
P
P
W
W
S
S
s
s
p
p
o
o
w
w
o
o
d
d
o
o
w
w
a
a
n
n
y
y
j
j
e
e
s
s
t
t
b
b
r
r
a
a
k
k
i
i
e
e
m
m
a
a
k
k
t
t
y
y
w
w
n
n
o
o
ś
ś
c
c
i
i
g
g
e
e
n
n
ó
ó
w
w
w
w
r
r
e
e
g
g
i
i
o
o
n
n
i
i
e
e
c
c
h
h
r
r
o
o
m
m
o
o
s
s
o
o
m
m
u
u
1
1
5
5
p
p
o
o
c
c
h
h
o
o
d
d
z
z
ą
ą
c
c
y
y
m
m
o
o
d
d
o
o
j
j
c
c
a
a
,
,
k
k
t
t
ó
ó
r
r
e
e
u
u
l
l
e
e
g
g
a
a
j
j
ą
ą
p
p
i
i
ę
ę
t
t
n
n
o
o
w
w
a
a
n
n
i
i
u
u
(
(
s
s
ą
ą
n
n
i
i
e
e
a
a
k
k
t
t
y
y
w
w
n
n
e
e
)
)
n
n
a
a
c
c
h
h
r
r
o
o
m
m
o
o
s
s
o
o
m
m
i
i
e
e
1
1
5
5
p
p
o
o
c
c
h
h
o
o
d
d
z
z
ą
ą
c
c
y
y
m
m
o
o
d
d
m
m
a
a
t
t
k
k
i
i
.
.
Z
Z
m
m
i
i
a
a
n
n
y
y
w
w
D
D
N
N
A
A
w
w
t
t
y
y
m
m
s
s
a
a
m
m
y
y
m
m
c
c
y
y
t
t
o
o
g
g
e
e
n
n
e
e
t
t
y
y
c
c
z
z
n
n
i
i
e
e
r
r
e
e
g
g
i
i
o
o
n
n
i
i
e
e
,
,
a
a
l
l
e
e
p
p
o
o
c
c
h
h
o
o
d
d
z
z
ą
ą
c
c
e
e
o
o
d
d
m
m
a
a
t
t
k
k
i
i
o
o
d
d
p
p
o
o
w
w
i
i
e
e
d
d
z
z
i
i
a
a
l
l
n
n
e
e
s
s
ą
ą
z
z
a
a
z
z
e
e
s
s
p
p
ó
ó
ł
ł
A
A
n
n
g
g
e
e
l
l
m
m
a
a
n
n
a
a
.
.
Analiza metylacji
W celu uwidocznienia efektu metylacji, DNA modyfikowano kwaśnym siarczynem sodowym.
●
PCR prowadzono z dwoma zestawami starterów, specyficznym dla kopii metylowanej i
niemetylowanej.
Kontrola- obecność kopii matczynej i ojcowskiej
PWS - obecny tylko fragment pochodzący z kopii matczynej
AS - obecny tylko fragment pochodzący z kopii ojcowskiej
P
P
C
C
R
R
-
-
S
S
S
S
C
C
P
P
Analiza konformacji jednoniciowych DNA, polega na porównaniu konformacji badanych
jednoniciowych fragmentów kwasów nukleinowych. Zdenaturowane produkty PCR, rozdziela się w
natywnym żelu poliakrylamidowym. Jednoniciowe fragmenty DNA w warunkach elektroforezy tworzą
wewnętrzne sparowania i przyjmują określoną konformację przestrzenną zależną od sekwencji
nukleotydów. Fragmenty o takiej samej sekwencji nukleotydów, przyjmują taką samą konformację
przestrzenną i migrują w żelu z taką samą prędkością.
Zastosowanie:
- Umożliwia wykrywanie punktowych zmian w DNA.
Przykład:
zespół Marfana (Marfan syndrome)
Sekwencjonowanie DNA
M
M
e
e
t
t
o
o
d
d
a
a
p
p
o
o
l
l
e
e
g
g
a
a
n
n
a
a
e
e
n
n
z
z
y
y
m
m
a
a
t
t
y
y
c
c
z
z
n
n
e
e
j
j
r
r
e
e
p
p
l
l
i
i
k
k
a
a
c
c
j
j
i
i
j
j
e
e
d
d
n
n
o
o
n
n
i
i
c
c
i
i
o
o
w
w
e
e
j
j
(
(
d
d
e
e
n
n
a
a
t
t
u
u
r
r
o
o
w
w
a
a
n
n
e
e
j
j
)
)
m
m
a
a
t
t
r
r
y
y
c
c
y
y
D
D
N
N
A
A
,
,
r
r
o
o
z
z
p
p
o
o
c
c
z
z
y
y
n
n
a
a
j
j
ą
ą
c
c
e
e
j
j
s
s
i
i
ę
ę
o
o
d
d
j
j
e
e
d
d
n
n
e
e
g
g
o
o
s
s
t
t
a
a
r
r
t
t
e
e
r
r
a
a
,
,
a
a
k
k
o
o
ń
ń
c
c
z
z
ą
ą
c
c
e
e
j
j
w
w
b
b
u
u
d
d
o
o
w
w
a
a
n
n
i
i
e
e
m
m
d
d
i
i
d
d
e
e
o
o
k
k
s
s
y
y
n
n
u
u
k
k
l
l
e
e
o
o
t
t
y
y
d
d
u
u
(
(
z
z
m
m
o
o
d
d
y
y
f
f
i
i
k
k
o
o
w
w
a
a
n
n
e
e
g
g
o
o
n
n
u
u
k
k
l
l
e
e
o
o
t
t
y
y
d
d
u
u
)
)
d
d
o
o
n
n
o
o
w
w
o
o
p
p
o
o
w
w
s
s
t
t
a
a
ł
ł
e
e
g
g
o
o
ł
ł
a
a
ń
ń
c
c
u
u
c
c
h
h
a
a
D
D
N
N
A
A
.
.
Poszukiwanie mutacji
Mutacje znane
Mutacje nieznane
Metody przesiewowe
PCR-SSCP
PCR-HD
Sekwencjonowanie DNA
Mutacje punktowe
Rearanżacje genowe
PCR-RFLP
PCR-ASO
PCR-Multipleks
Southern Blot

Nowoczesne metody w diganostyce molekularnej
1. Mikromacierze (ang.microarrays)
Chipy DNA są zbiorem sond molekularnych związanych ze stałym podłożem w ściśle określonym
porządku, które wykrywają przez hybrydyzację komplementarne do siebie cząsteczki DNA lub RNA
Zastosowanie:
-badanie ekspresji genów, detekcja mutacji, polimorfizmów, genotypowanie (oznaczanie SNP)...
2. Real-Time PCR
Metoda ilościowa oparta na amplifikacji DNA in vitro- opiera się na pomiarze liczby kopii DNA, przez
pomiar fluorescencji
Zastosowanie: Ilościowe określanie ekspresji genów, wydajność terapii lekowych/monitorowanie
leków, Ilościowe określanie DNA wirusowego, uszkodzenia DNA (niestabilność mikrosatelitarna),
studia nad DNA mitochondrialnym, detekcja metylacji, detekcja inaktywacji chromosomu X,
genotypowanie, wykrywanie trisomii…
3. MLPA (ang. Multiplex Ligation-Dependent Probe Amplification)
“Analiza ilościowa typu Multiplex wielu genów jednocześnie”, Detekcja zmian liczby kopii 45
genomowych sekwencji DNA w jednej, prostej do przeprowadzenia reakcji PCR.
-Minimum 20 ng ludzkiego DNA lub 10-120 ng RNA
-Analiza częściowo zdegradowanego DNA
-DNA ze skrawków parafinowychn
-Tkanek w formalinien
-Płodowy DNA uzyskany z plazmy krwi matczynej.
-Rozróżnienie sekwencji które różnią się pojedynczym nukleotydem, detekcja znanych mutacji oraz
SNP
Wyszukiwarka
Podobne podstrony:
diagnostyka molekularna w medycynie 2 rokWL, biologia molekularna, interferencja RNA
diagnostyka molekularna w medycynie 2 rokWL, VI rok, Genetyka, Genetyka, Egzamin
diagnostyka molekularna w medycynie 2 rokWL
Diagnostyka molekularna w medycynie
Diagnostyka molekularna w medycynie
sem[1].01- Badania diagnostyczne w chorobach, Medycyna, Interna, Pulmonologia
Choroby dróg moczowych, ZDROWIE-Medycyna naturalna, 01-Do uporządkowania, ZIOŁA
Trochę o ziołach leczniczych, ZDROWIE-Medycyna naturalna, 01-Do uporządkowania, zdrowie(2)
Mudry - energetyczne układy dłoni, ZDROWIE-Medycyna naturalna, 01-Do uporządkowania, Zdrowie i Medyc
Choroby układu nerwowego, ZDROWIE-Medycyna naturalna, 01-Do uporządkowania, ZIOŁA
Kąpiele lecznicze, ZDROWIE-Medycyna naturalna, 01-Do uporządkowania, ZIOŁA
Organizacja Laboratorium Usługowego1B, studia-biologia, Studia magisterskie, Mgr sem III, Diagnostyk
Metody diagnostyki nowotworow, Medycyna, Onkologia
Diagnostyka molekularna niedobo Nieznany
diagnostyka molekularna 2
Żyworódka, ZDROWIE-Medycyna naturalna, 01-Do uporządkowania, ZIOŁA
Diagnoza lekarska, MEDYCYNA O, INTERNA, interna(2)
więcej podobnych podstron