
1
Cz.2_ GENETYKA_Laboratorium_2013/14
1. IZOLACJA DNA PLAZMIDOWEGO BAKTERII
Opracowano szereg metod oczyszczania plazmidowego DNA bakterii, z których ka
ż
da obejmuje
trzy etapy: - hodowl
ę
bakterii (namna
ż
anie plazmidów)
- liz
ę
bakterii, (rozpuszczenie)
- oczyszczanie plazmidowego DNA.
Plazmidy izoluje si
ę
najcz
ęś
ciej z hodowli płynnych, w których podło
ż
e uzupełnione jest
odpowiednim antybiotykiem, którego gen oporno
ś
ci znajduje si
ę
na plazmidzie. We wszystkich
metodach izolacji plazmidowego DNA wykorzystuje si
ę
dwie istotne ró
ż
nice pomi
ę
dzy DNA
genomowym a DNA plazmidowym bakterii: - DNA genomowy jest wielokrotnie wi
ę
kszy od DNA
plazmidu, - podczas procedury izolacji DNA plazmidowego DNA genomowy zostaje trwale
zniszczony, podczas gdy DNA plazmidowy pozostaje w postaci form CCC (ang. covalently closed
circle).
Odwirowane komórki bakteryjne poddawane s
ą
lizie. Bakterie ulegaj
ą
lizie pod wpływem
detergentów (SDS, Sarkozyl, Triton X-100), rozpuszczalników organicznych, roztworów
alkalicznych (liza alkaliczna) lub wysokiej temperatury (liza termiczna). Stosowany mo
ż
e by
ć
równie
ż
lizozym - enzym trawi
ą
cy
ś
cian
ę
komórkow
ą
bakterii (nie działa w pH<8.0). W metodzie lizy
alkalicznej bufor o wysokim pH zawieraj
ą
cy NaOH i SDS powoduje całkowit
ą
liz
ę
komórki jak
równie
ż
denaturacj
ę
genomowego DNA, natomiast plazmidowy DNA w formie CCC zostaje
zdenaturowany jedynie na niewielkich odcinkach. W przypadku otrzymywania plazmidowego DNA
metod
ą
termiczn
ą
, czynnikiem denaturuj
ą
cym jest wysoka temperatura, w której DNA genomowy i
plazmidowy zachowuj
ą
si
ę
podobnie jak w wysokim pH.
Nast
ę
pny etap oczyszczania DNA polega na oddzieleniu DNA od zwi
ą
zanych z nim białek.
Zwykle stosuje si
ę
do tego celu nasycony buforem roztwór fenolu lub jego mieszanin
ę
z
chloroformem i alkoholem izoamylowym. Białka mo
ż
na równie
ż
usun
ąć
przez trawienie ich
proteinazami (zwykle proteinaz
ą
K). Usuni
ę
cie kwasu RNA przeprowadza si
ę
enzymatycznie,
inkubuj
ą
c próbki z RNaz
ą
(woln
ą
od DNazy), przez s
ą
czenie molekularne lub wirowanie w
gradiencie g
ę
sto
ś
ci. W metodzie lizy alkalicznej, kiedy liniowe cz
ą
steczki DNA ulegaj
ą
denaturacji,
natomiast superzwini
ę
te formy CCC plazmidu s
ą
po denaturacji nadal splecione, neutralizacja pH
roztworami o wysokim st
ęż
eniu soli (np. octan amonu) prowadzi do renaturacji jedynie DNA
plazmidowego, podczas gdy DNA genomowy wraz z RNA i białkami wytr
ą
ca si
ę
w postaci
serowatego osadu. Z odbiałczonych próbek DNA wytr
ą
cany jest alkoholem etylowym lub
izopropanolem w obecno
ś
ci octanu potasowego, sodowego lub amonowego.
Wszystkie metody otrzymywania plazmidowego DNA, z wyj
ą
tkiem lizy alkalicznej, wymagaj
ą
oddzielenia tego DNA od DNA genomowego w gradiencie chlorku cezu w obecno
ś
ci bromku
etydyny. Wykorzystuje si
ę
tu fakt,
ż
e bromek etydyny intensywniej interkaluje do zdenaturowanego
DNA genomowego ni
ż
do DNA plazmidowego, powoduj
ą
c cz
ęś
ciowe rozwini
ę
cie helisy DNA i

2
wydłu
ż
enie cz
ą
steczki, co powoduje zmniejszenie jej g
ę
sto
ś
ci pławnej. Ró
ż
nica w g
ę
sto
ś
ci
pomi
ę
dzy form
ą
CCC a liniow
ą
i kolist
ą
OC (ang. open circle) wynosi około 0.04 g/ml i jest
wystarczaj
ą
ca do oddzielenia superzwini
ę
tego DNA podczas wirowania w gradiencie g
ę
sto
ś
ci
chlorku cezu w obecno
ś
ci bromku etydyny. Pasmo superzwini
ę
tego DNA układa si
ę
poni
ż
ej pasma
utworzonego przez liniowe fragmenty chromosomowego DNA oraz formy liniowej i OC plazmidu.
Cz
ą
steczki RNA maj
ą
najwi
ę
ksz
ą
g
ę
sto
ść
i zbieraj
ą
si
ę
na dnie probówki wirowniczej, za
ś
białka
pozostaj
ą
w górnej warstwie roztworu.
Oczyszczanie plazmidowego DNA przeprowadzi
ć
mo
ż
na równie
ż
metod
ą
wirowania lizatów
komórkowych w gradiencie alkalicznej sacharozy (w
ś
rodowisku alkalicznym formy liniowe i OC
ulegaj
ą
rozdzieleniu na pojedyncze nici i sedymentuj
ą
3-4 razy wolniej ni
ż
niezdenaturowana
superzwini
ę
ta forma CCC), stosuj
ą
c wymieniacze jonowe (np. kolumienki Qiagen) lub filtracj
ę
na
ż
elach.
PYTANIA
1. Jakie s
ą
etapy izolacji plazmidowego DNA bakterii
2. Jakie s
ą
ró
ż
nice miedzy DNA genomowym a plazmidowym bakterii?
3. Jak usuwa si
ę
białka, genomowe DNA i RNa z próby
4. Narysuj wynik wirowania gradiencie chlorku cezu w obecno
ś
ci bromku etydyny w trakcie lizy
alkalicznej
ENZYMY RESTRYKCYJNE. MAPY RESTRYKCYJNE
Enzymy restrykcyjne (ang. restriction enzymes) s
ą
enzymami izolowanymi z bakterii, zdolnymi do
rozpoznawania specyficznych sekwencji w DNA i przecinania dwuniciowej cz
ą
steczki DNA. Enzymy
restrykcyjne typu II wymagaj
ą
jako substratu dwuniciowej cz
ą
steczki DNA, zawieraj
ą
cej co najmniej
jedn
ą
sekwencj
ę
rozpoznawan
ą
przez dany enzym, oraz jonów magnezu. DNA zostaje przeci
ę
te w
obr
ę
bie lub w okolicy palindromowej sekwencji rozpoznawanej. Sekwencja palindromowa w
genetyce oznacza tak
ą
sekwencj
ę
DNA, dla której sekwencja komplementarna jest identyczna (przy
zało
ż
eniu,
ż
e obie sekwencje czytamy z uwzgl
ę
dnieniem polarno
ś
ci nici; zgodnie z przyj
ę
tym
obyczajem - od ko
ń
ca 5' do 3'):
5' A A T T 3'
3' T T A A 5'
5' A G G C C T 3'
3' T C C G G A 5'
Niektóre enzymy produkuj
ą
"lepkie ko
ń
ce" 3'. Wszystkie natomiast pozostawiaj
ą
grup
ę
fosforanow
ą
na ko
ń
cu 5', a grup
ę
hydroksylow
ą
na ko
ń
cu 3'. "Lepkie ko
ń
ce" maj
ą
du
ż
e znaczenie przy
klonowaniu genów: sekwencje "lepkich ko
ń
ców" powstałych w wyniku działania tego samego
enzymu s
ą
komplementarne. W obecno
ś
ci enzymu ligazy mo
ż
na takie cz
ą
steczki ponownie
poł
ą
czy
ć
ze sob
ą
kowalencyjnie. Nazewnictwo enzymów restrykcyjnych opiera si
ę
na literowych
skrótach, w których pierwsza litera pochodzi od rodzaju bakterii, a druga i trzecia od gatunku.
Nast
ę
pna litera oznacza szczep lub typ, a kolejne enzymy z danego typu lub szczepu otrzymuj
ą
liczby rzymskie.
Enzymy pochodz
ą
ce z ró
ż
nych szczepów, ale rozpoznaj
ą
ce te same sekwencje DNA, nazywaj
ą
si
ę
izoschizomerami.
3
Zdarza si
ę
,
ż
e dwa enzymy wytwarzaj
ą
takie same "lepkie ko
ń
ce", mino
ż
e rozpoznaj
ą
ró
ż
ne
sekwencje DNA. Enzym BamHI produkuje ko
ń
ce GATCC (rozpoznaje sekwencj
ę
GGATCC), za
ś
enzym Bgl II, wytwarzaj
ą
cy takie same "lepkie ko
ń
ce", rozpoznaje sekwencj
ę
AGATCT. Umo
ż
liwia
to klonowanie DNA strawionego BamHI w wektorze strawionym BglII, ale nie ka
ż
dy sklonowany
odcinek DNA b
ę
dzie mógł by
ć
wycinany enzymem Bgl II.
Do pojedynczej reakcji trawienia u
ż
ywa si
ę
zwykle 0,2 - 1 ug DNA w roztworze o obj
ę
to
ś
ci 20 ul lub
mniejszej. Bufory do trawie
ń
przygotowuje si
ę
w postaci 10-krotnie st
ęż
onych roztworów ró
ż
ni
ą
cych
si
ę
mi
ę
dzy sob
ą
zawarto
ś
ci
ą
soli. Je
ż
eli DNA ma by
ć
strawiony dwoma enzymami wymagaj
ą
cymi
ró
ż
nych buforów, najpierw przeprowadza si
ę
trawienie w buforze o ni
ż
szej soli, a nast
ę
pnie
podwy
ż
sza si
ę
st
ęż
enie soli w mieszaninie przez dodanie odpowiedniej obj
ę
to
ś
ci st
ęż
onego NaCl.
Inkubacj
ę
preparatów DNA z enzymami restrykcyjnymi prowadzi si
ę
w temperaturze 37
o
C w czasie
1 godziny lub dłu
ż
ej. Przerwanie reakcji (zwykle niekonieczne) mo
ż
na przeprowadzi
ć
przez
termiczn
ą
inaktywacj
ę
enzymu (15 minut, 65
o
C) lub dodaj
ą
c EDTA pH 8.0 do ko
ń
cowego st
ęż
enia
10 mM (chelatacja jonów magnezu).
Ź
ródłem informacji o enzymach restrykcyjnych (ich
termostabilno
ś
ci i wymaganiach co do st
ęż
enia soli) s
ą
katalogi firm produkuj
ą
cych te enzymy (np.
New England Biolabs, Promega). Jednostka enzymu restrykcyjnego to taka jego ilo
ść
, która trawi
kompletnie 1 ug DNA faga l (ok. 50 kb) w czasie 1 godziny w 37
o
C.
Podstawow
ą
cech
ą
produktów trawienia DNA enzymami restrykcyjnymi jest to,
ż
e otrzymywane
fragmenty DNA daj
ą
si
ę
rozdzieli
ć
na
ż
elach w taki sposób,
ż
e w ka
ż
dym pr
ąż
ku na
ż
elu znajduj
ą
si
ę
cz
ą
steczki DNA o identycznej sekwencji nukleotydowej. Fakt ten pozwala na precyzyjn
ą
obróbk
ę
DNA, m.in. konstruowanie map restrykcyjnych badanego DNA (mapa restrykcyjna
wskazuje miejsca ci
ę
te przez enzymy restrykcyjne). Analizuj
ą
c na
ż
elach produkty trawienia danego
DNA ró
ż
nymi enzymami restrykcyjnymi, stosowanymi pojedynczo i w kombinacjach, mo
ż
na ustali
ć
wzajemne poło
ż
enie i odległo
ś
ci pomi
ę
dzy sekwencjami rozpoznawanymi przez te enzymy. Oprócz
tego, DNA poci
ę
ty enzymami restrykcyjnymi mo
ż
na poddawa
ć
ligacji z wektorem i tworzy
ć
zrekombinowane (zawieraj
ą
ce sklonowany DNA) plazmidy. Aby wyznaczy
ć
wielko
ść
nieznanych
fragmentów DNA, stosuje si
ę
standardy wielko
ś
ci (cz
ą
steczki DNA o znanej wielko
ś
ci, poddawane
elektroforezie w tym samym
ż
elu co cz
ą
steczki badane).
PYTANIA
1. Narysuj wymy
ś
lon
ą
sekwencje DNA palindromowe i nie palindromowe
2. Czy ró
ż
ne enzymy restrykcyjne mog
ą
produkowa
ć
takie same lepkie ko
ń
ce
3. Co oznacza jedna jednostka enzymu restrykcyjnego?
4. Czy w jednej reakcji mo
ż
na u
ż
ywa
ć
wi
ę
cej ni
ż
jednego enzymu
4
Ć
wiczenia praktyczne
1. Otrzymywanie DNA plazmidowego na mał
ą
skal
ę
(minilizaty) za pomoc
ą
zastawu do
izolacji Plazmid Mini
Przed przyst
ą
pieniem do
ć
wicze
ń
przeczytaj ze zrozumieniem cał
ą
instrukcje do zestawu (plik
PDF).
Odczynniki i aparatura:
- pipety automatyczne i jednorazowe ko
ń
cówki
- probówki szklane (10 ml) zawieraj
ą
ce
całonocna kultur
ę
bakteryjn
ą
(LB Amp+)
- woda
- probówki Eppendorfa,
- mikrowirówka,
- pisaki do probówek/ r
ę
czniki
papierow/stojaki/alkohol do sterylizacji miejsca
pracy, r
ę
kawiczki
Wykonanie: post
ę
puj zgodnie z protokołem izolacji z uwzgl
ę
dnieniem dwóch pierwszych kroków
(nie opisanych w protokole)
1. rozla
ć
hodowle bakteryjn
ą
do probówek Eppendorfa i zwirowa
ć
bakterie w mikrowirówce przez 1
minut
ę
(2 x 1.5 ml),
2. ods
ą
czy
ć
pipet
ą
po
ż
ywk
ę
do sucha,
Podpisz swoje próby (tak
ż
eby je rozpozna
ć
do trawienia za tydzie
ń
)
2. Trawienie plazmidu pRS 416 enzymami restrykcyjnymi i sporz
ą
dzanie mapy restrykcyjnej
(planowanie na lab.II, przeprowadzanie reakcji na lab.III)
Odczynniki i aparatura:
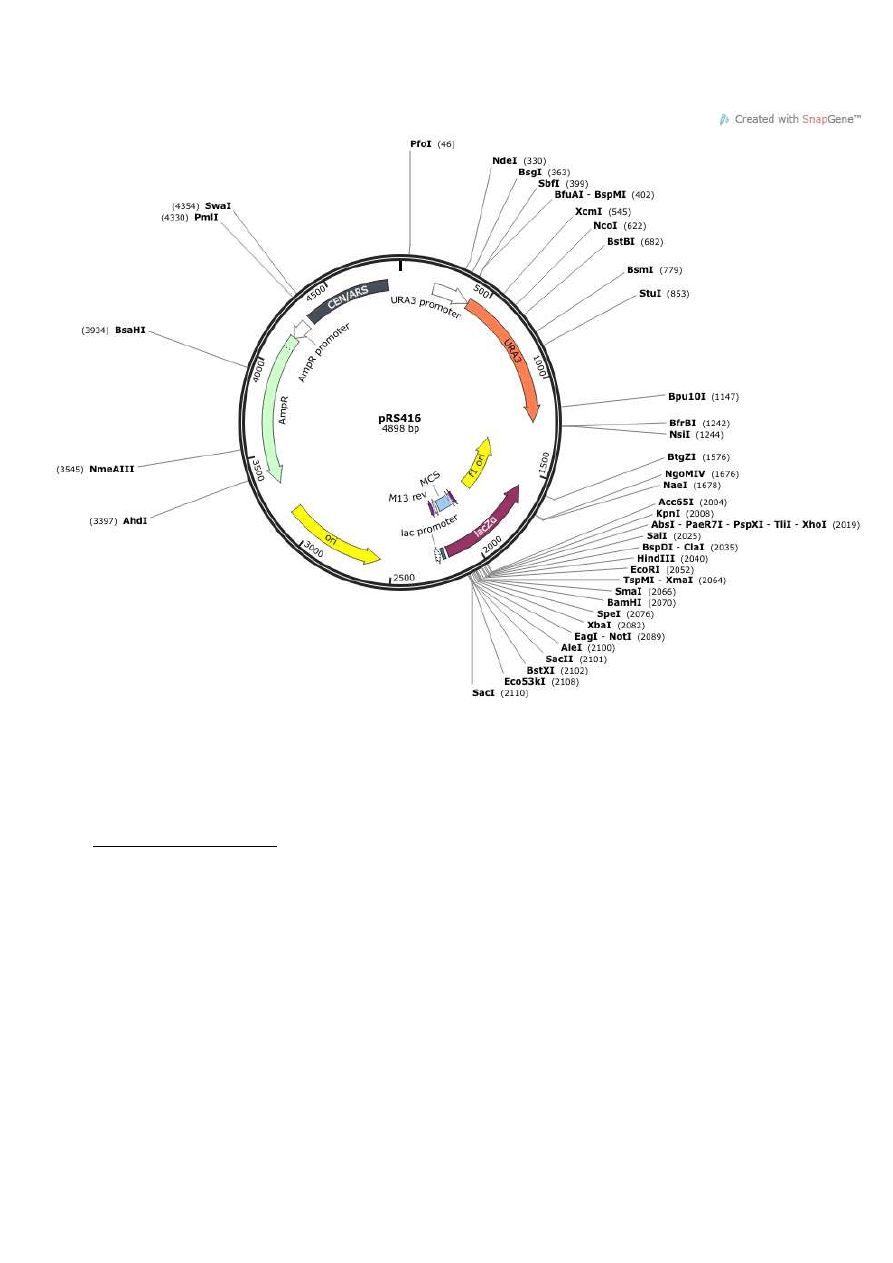
Mapa plazmidu
- enzymy - dostepna z ZGE (ApaI; BamHI; Bsp68I (NruI); Bsu15I (ClaI); Eco147I (StuI);
Eco321 (EcoRV); EcoRI; HindIII; NcoI; NdeI; NotI; NsbI (MstI); PaeI (SphI); PvuI; PvuII;
SacI; Sali; ScaI; SmaI; SmiI (SwaI); XbaI; XhoI)
- bufory,
- katalogi enzymów restrykcyjnych (podane temperatury inkubaji, bufory i dezaktywacje)
1. Zaplanuj ciecie restrykcyjne wybranymi dwoma enzymami restrykcyjnymi pojedynczo i
oboma na raz.
2. Na podstawie katalogów dobierz bufory, temperatur
ę
inkubacji i dezaktywacji.
Dostosuj do wybranych enzymów podany poni
ż
ej typowy protokół:
Zmieszaj w eppendorfie w podanej kolejno
ś
ci:
1µl 10x Buffer
6.5µl H2O
2µl DNA
0.5µl Enzyme
Inkubuj przez 1h w 37oC
3. Na postawie mapy oszacuj wielko
ść
powstałych po ci
ę
ciu fragmentów plazmidu.
4. Narysuj jak b
ę
dzie wygl
ą
dał
ż
el po elektroforezie
5
Ź
ródło:http://www.snapgene.com/resources/plasmid_files/yeast_plasmids/pRS416/
3.
Reakcja trawienia enzymami restrykcyjnymi (lab.III)
Odczynniki i aparatura:
- plazmid pRS416 (4898 bp) o st
ęż
eniu ok. 3 ug/ul, (po izolacji)
- probówki Eppendorfa,
- pipety, ko
ń
cówki do pipet
- bufory i enzymy restrykcyjne
Wykonaj reakcj
ę
zgodnie z protokołem opracowanym na Lab.II (dostosuj go do własnych
enzymów i buforów).
Reakcj
ę
przygotowuje si
ę
na lodzie!!!!!

6
4. Eektroforeza agarozowa - sprawdzenie ci
ę
cia restrykcyjnego (Lab.IV)
Odczynniki i aparatura:
- plazmid nie poci
ę
ty
- plazmid poci
ę
ty
- standard wielko
ś
ci DNA,
- obci
ąż
nik do prób (niebieski)
- probówki Eppendorfa,
- aparat do elektroforezy (laboratoria 3)
Gotowy zel agarozowy zawieraj
ą
cy barwnik
DNARed
- do strawionych próbek oraz do markera wielko
ś
ci DNA doda
ć
1/10 obj
ę
to
ś
ci barwnika do
elektroforezy,
- nanie
ść
próbki do studzienek na
ż
elu przy pomocy pipety automatycznej (Uwaga! Minimalna ilo
ść
DNA, któr
ą
wida
ć
na
ż
elu w postaci pr
ąż
ka, wynosi około 10 ng. Nie nale
ż
y przekracza
ć
200 ng
DNA w pr
ąż
ku),
- prowadzi
ć
elektroforez
ę
w buforze TBE przy napi
ę
ciu nie przekraczaj
ą
cym 5 V/cm,
- sfotografowa
ć
ż
el na transiluminatorze w
ś
wietle UV,
- oszacowa
ć
na podstawie krzywej wielko
ś
ci trawionych fragmentów DNA i porówna
ć
z planowan
ą
przez siebie.
Przygotowanie dr Dominika Włoch-Salamon na podstawie: http://arete.ibb.waw.pl/docs/Skrypt-dla-fakultetu-99.html
RAPORT - nale
ż
y odda
ć
pod koniec zaj
ęć
(
ż
eby prowadz
ą
cy wiedział jakie enzymy
przygotowa
ć
na kolejne zaj
ę
cia)
Zaplanuj ciecie restrykcyjne wybranymi dwoma enzymami restrykcyjnymi pojedynczo i oboma na
raz. Na podstawie katalogów dobierz bufory, temperatur
ę
inkubacji i dezaktywacji.
1. Wypisz dostosowany do wybranych enzymów podany poni
ż
ej typowy protokół:
Zmieszaj w eppendorfie w podanej kolejno
ś
ci:
1µl 10x Buffer
6.5µl H2O
2µl DNA
0.5µl Enzyme
Inkubuj przez 1h w 37oC
2. Podaj wielko
ś
ci fragmentów DNA powstałych po ci
ę
ciach zaplanowanych przez Ciebie
3. Narysuj jak b
ę
dzie wygl
ą
dał
ż
el po elektroforezie
Wyszukiwarka
Podobne podstrony:
Izolacja DNA plazmidy Genetyka Nieznany
Izolacja DNA plazmidowego z E.coli metodą Maxi prep, Biotechnologia kosmetologiczna, Biologia molek
IZOLACJA DNA ROŚLINNEGO METODĄ MIKRO C, Biotechnologia notatki, Genetyka - biologia molekularna
ćw 3 Protokół izolacji DNA genomowego
VI-Izolacja DNA, Genetyka
Izolacja DNA z komórek prokariotycznych i eukariotycznych
cwiczenie 8 izolacja DNA
Izolacja DNA z komorek zwierzecych
cw 4 Izolacja chloroplastów
Elektrotechnika ćw.2 Izolacyjność, Badanie parametrów stanowiska izolowanego
Etapy izolacji DNA
Izolacja DNA
DNA, AWF, Genetyka, Genetyka
Protokó- izolacji DNA na -wiczenia (1), analiza DNA
Izolacja DNA
Spr Izolacja DNA
Prelekcja 10 - cz 1 - Mutacje genowe (punktowe) i polimorfizmy DNA, ROK 1, Genetyka
Genetyka test cw, niezbędnik rolnika 2 lepszy, genetyka
więcej podobnych podstron