
Regulacja ekspresji genów u
prokariota

Co
pozwala
komórkom
na
pełnienie
przez
nie
specyficznych funkcji?
Co powoduje, że komórki różnią się między sobą?
Funkcje, jakie pełnią komórki i właściwości jakie
posiadają, są zdeterminowane
białkami
jakie je tworzą.
Od czego zależy jakie białka oraz jakie ich ilości
występują w różnych komórkach?
Odpowiedzialne są za to:
- rodzaje i ilości poszczególnych mRNA powstające
w wyniku transkrypcji określonej puli genów,
- częstość z jaką różne mRNA podlegają translacji,
- „czas życia” powstałych w czasie translacji
białek.

Innymi słowy, za właściwości i funkcje
różnych komórek odpowiedzialna jest
zróżnicowana „aktywność”/ekspresja
genów.

W komórkach prokariotycznych (bakteriach) ekspresja
genów
kontrolowana
jest
głównie
na
poziomie
transkrypcji, a więc syntezy mRNA.
Dotyczy ona:
- inicjacji transkrypcji,
- terminacji transkrypcji.

W komórkach bakteryjnych występuje wiele białek, których poziom
ekspresji zależy od środowiska zewnętrznego. Przykładem są enzymy
biorące udział w metabolizmie cukrów i aminokwasów, których
obecność/ilość w komórce zależna jest od dostępności substratu.
U bakterii, enzymy związane z określonym szlakiem metabolicznym
kodowane są przez zespoły genów pozostające pod kontrolą jednego
promotora. Taka jednostka strukturalno-czynnościowa nosi nazwę
operonu.
Większość operonów obejmuje: (i) geny struktury oraz (ii) rejon
regulatorowy (
promotor
,
operator
,
gen regulatorowy
).
Geny struktury ulegają transkrypcji w formie pojedynczego
mRNA.
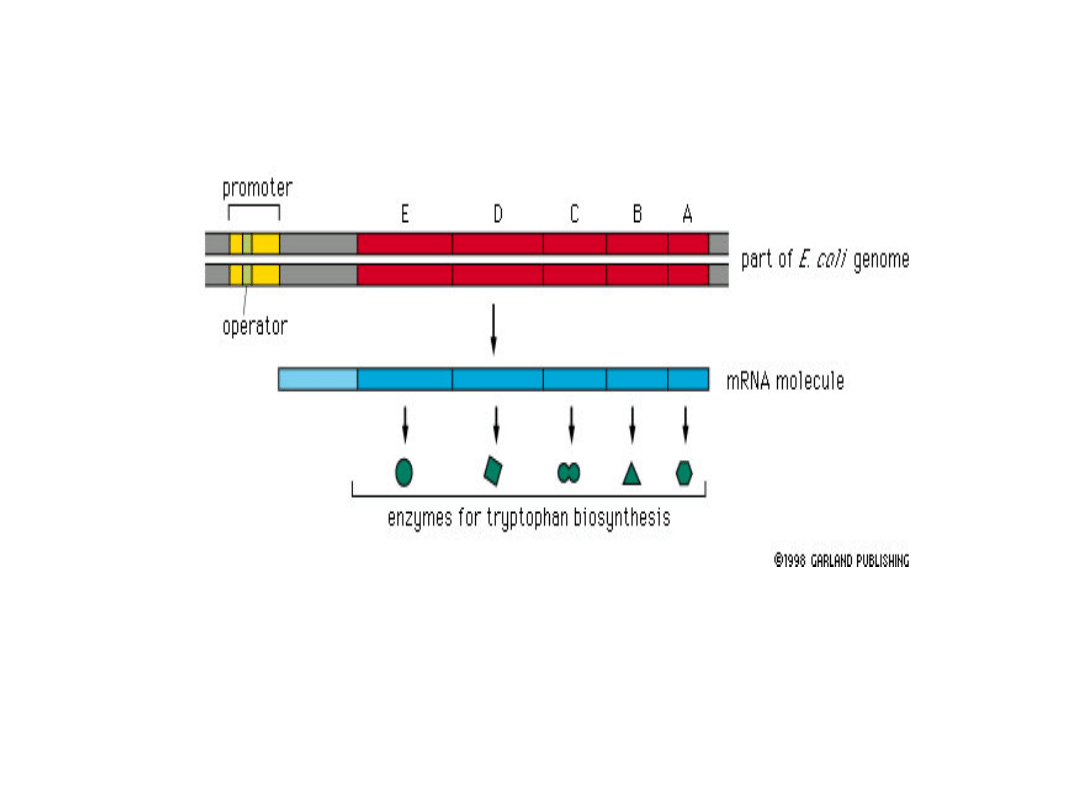
. Natomiast translacja poszczególnych łańcuchów polipeptydowych
przebiega niezależnie. Każdy gen struktury ma swój własny kodon
inicjujący i kodon stop, oraz miejsce wiązania rybosomu.

Regulacja ekspresji genów, których produkty (enzymy) biorą udział w
metabolizmie cukrów i aminokwasów oparta jest na następujących
ogólnych zasadach:
1. W przypadku metabolizmu cukrowców wzrost ilości substratu w
środowisku najczęściej aktywuje/indukuje transkrypcję danego
enzymu/enzymów, natomiast w metabolizmie aminokwasów
indukcję transkrypcji powoduje z kolei brak określonego
metabolitu (aminokwasu).
2. Niekiedy, obecność czynnika odżywczego może jednak hamować
transkrypcję
mRNA,
prowadząc
do
represji
syntezy
określonych białek enzymatycznych.
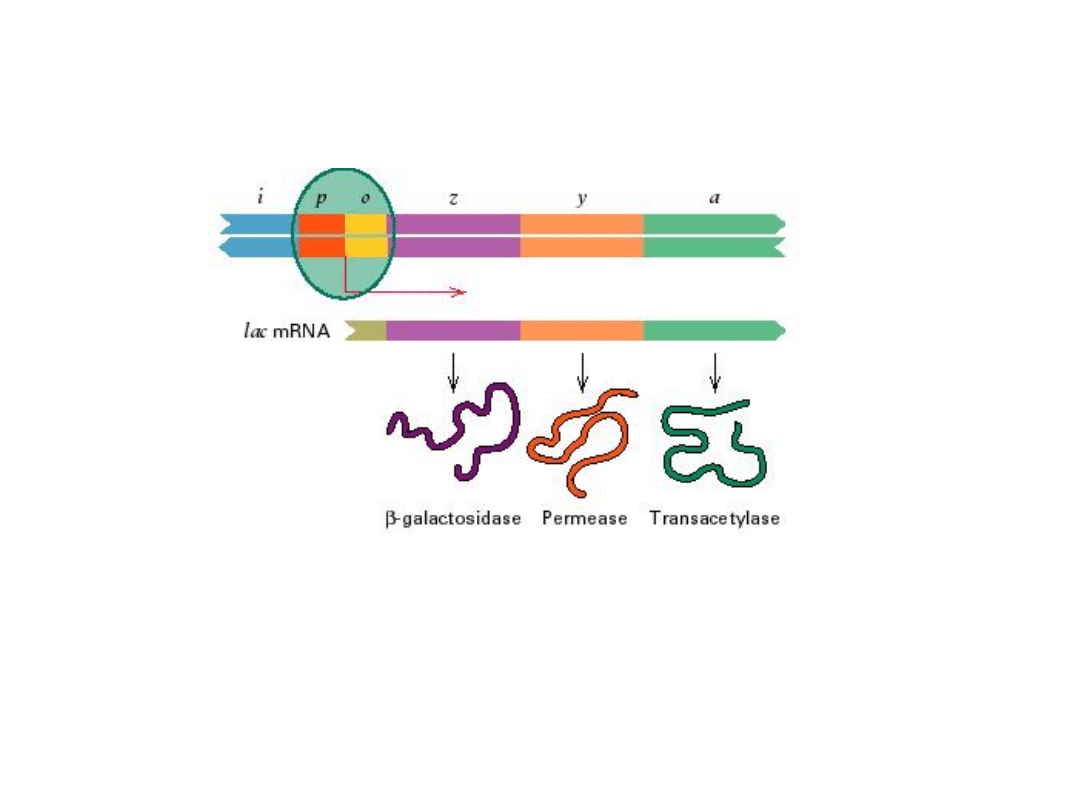
Operon laktozowy (operon lac) E. coli
W skład operonu lac wchodzą trzy geny struktury:
- -galaktozydaza,
- permeaza galaktozydowa,
- acetylotransferaza tiogalaktozydowa
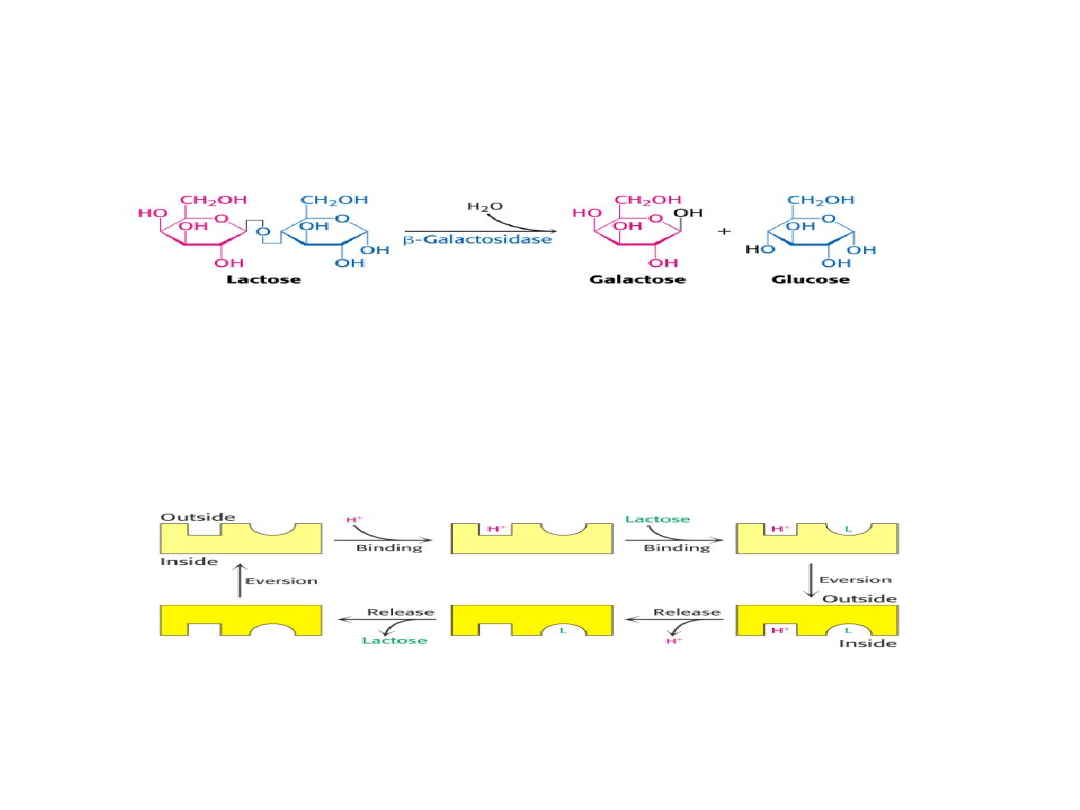
-Galaktozydaza odpowiada za rozkład laktozy do
monosacharydów.
Permeaza galaktozydowa bierze udział w transporcie
laktozy.
Acetylotransferaza tiogalaktozydowa - neutralizacja związków
dostających się do komórki podczas transportu laktozy.
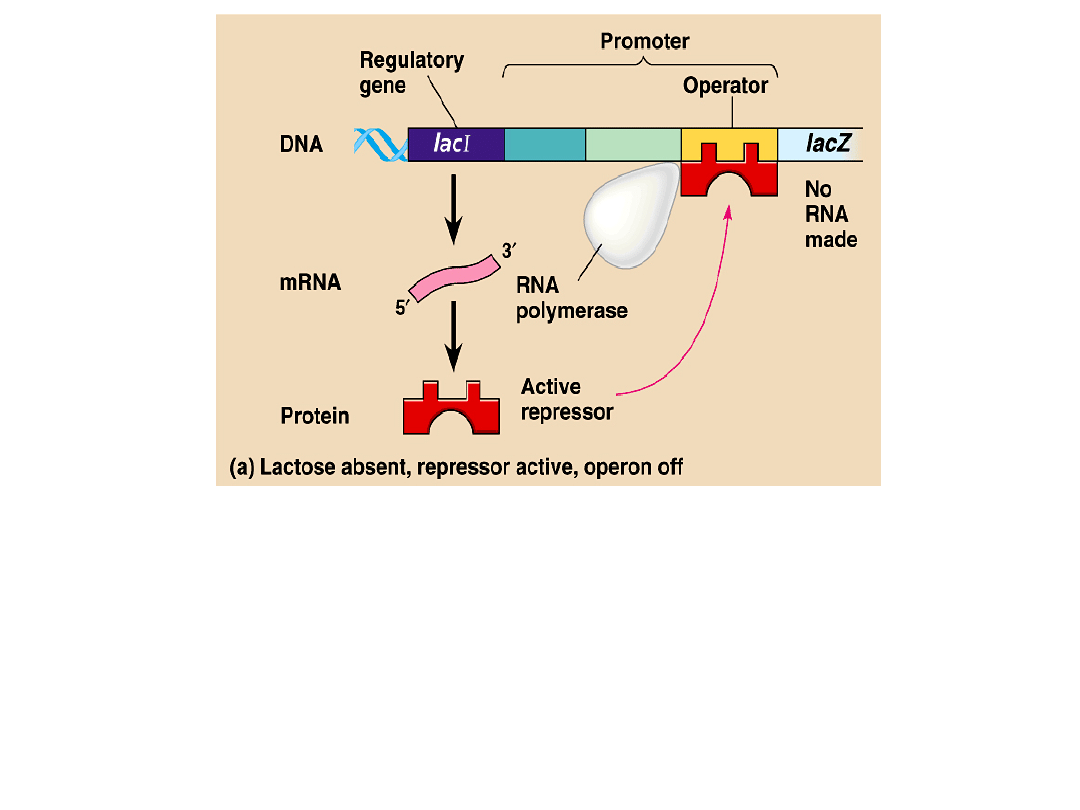
Produktem genu regulatorowego lacI jest białko represor laktozowy
(represor lac). Jest to homotetramer zbudowany z podjednostek o m.
cz. 37 kDa, który wiąże się do odcinka DNA obejmującego operator
oraz kilka nukleotydów wspólnych dla operatora i promotora. W ten
sposób obecność
represora
uniemożliwia rozpoczęcie transkrypcji przez
polimerazę RNA. Operon laktozowy jest przykładem zespołu genów
podlegających
negatywnej kontroli.
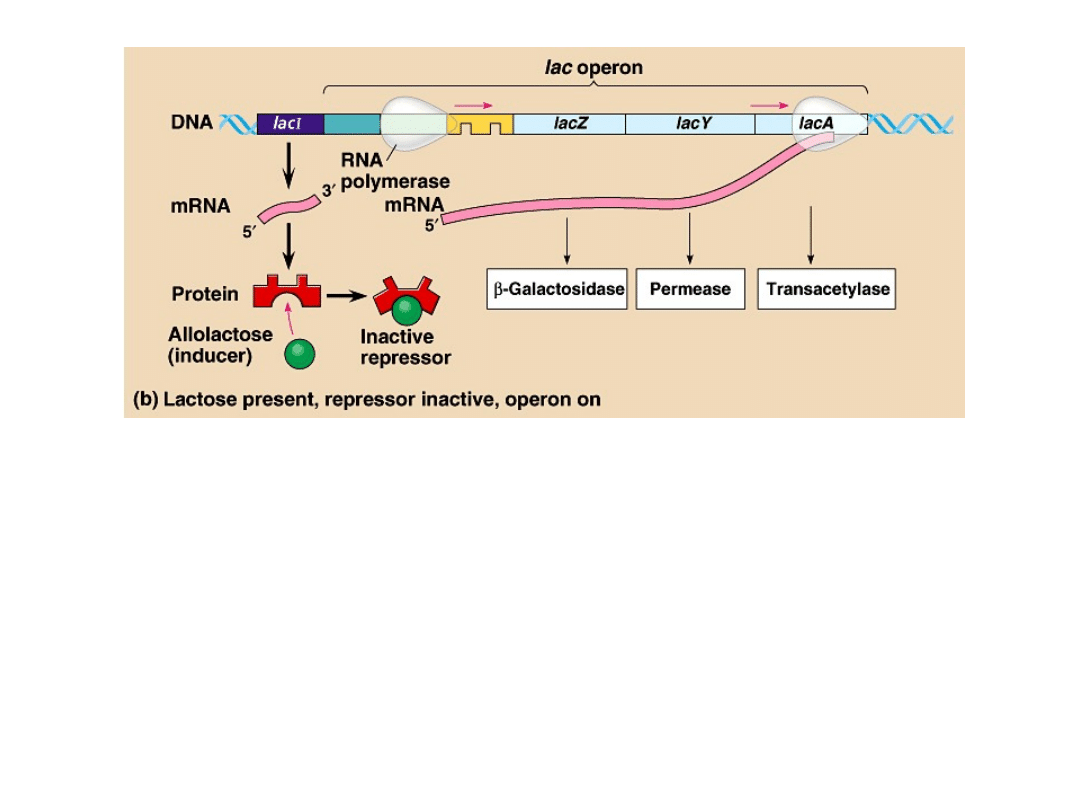
W obecności laktozy dochodzi do transkrypcji genów struktury
(zwolnienia represji) poprzez inaktywację represora w wyniku związania
induktora
. Rolę induktora pełni allolaktoza, powstająca z laktozy
przez transglikozylację katalizowaną przez -galaktozydazę.
Generalnie
induktorami
nazywamy
drobnocząsteczkowe
związki, które po związaniu z represorem hamują jego wiązanie
do operatora.
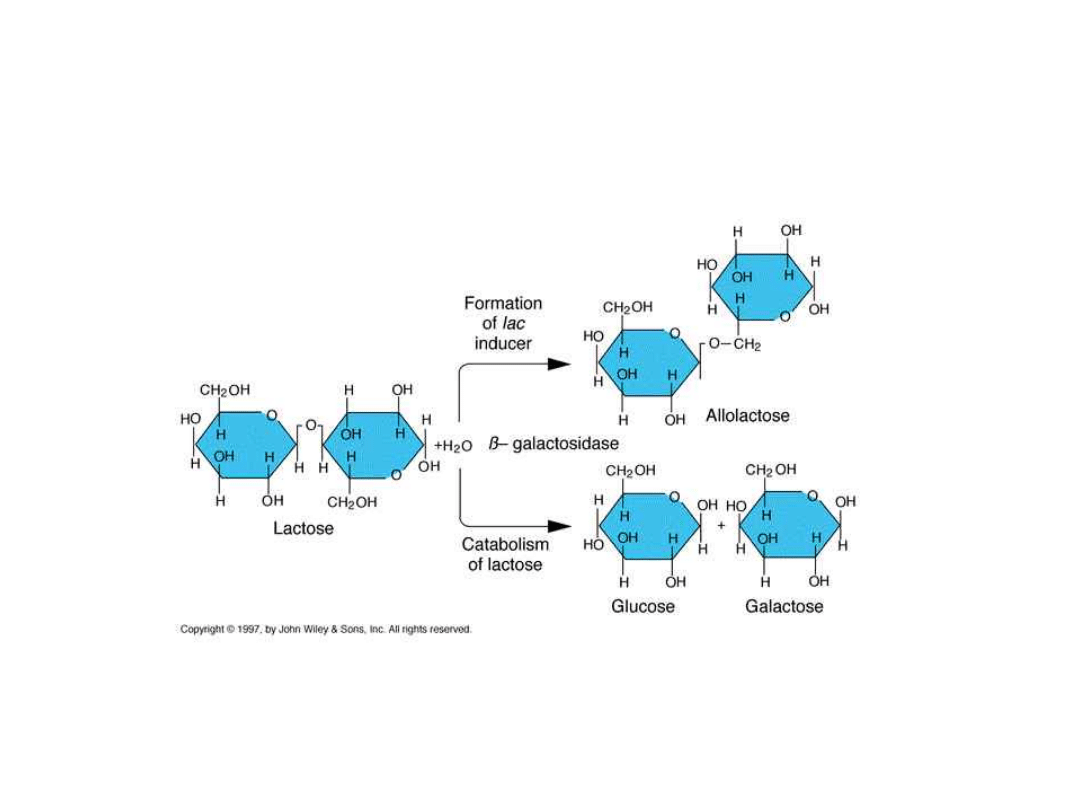
Losy laktozy po wniknięciu do wnętrza E. coli
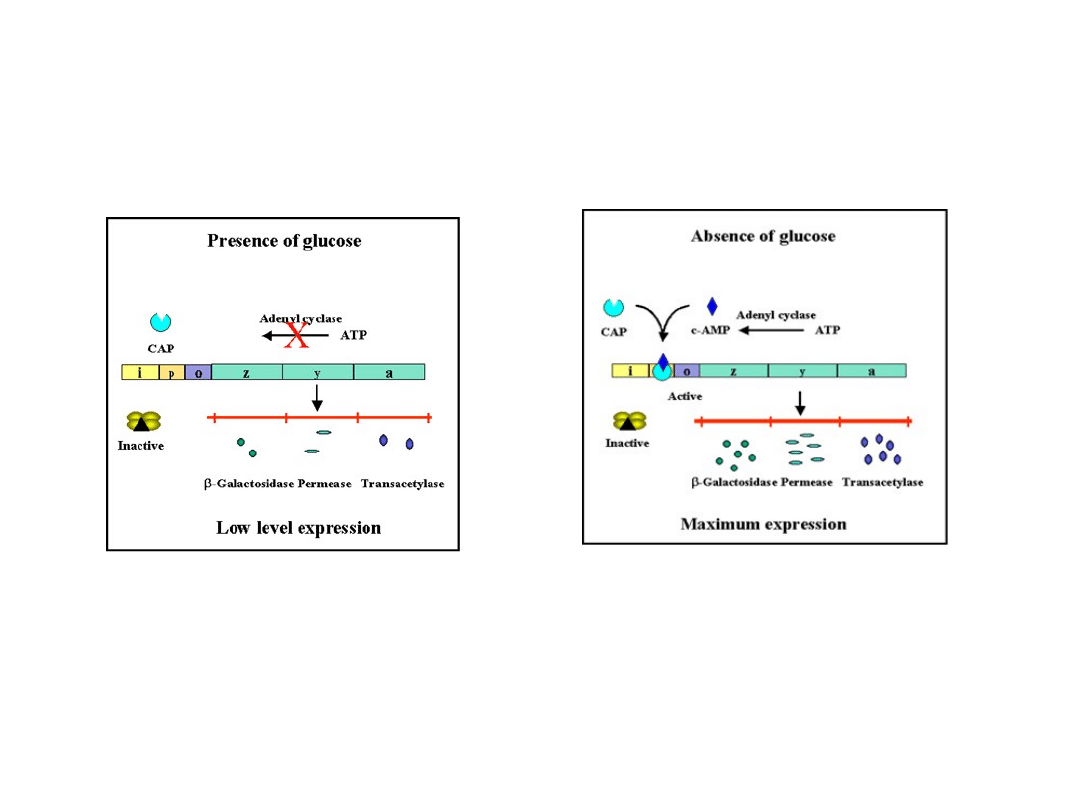
W obecności glukozy lub produktów jej rozpadu transkrypcja mRNA
dla wielu enzymów biorących udział w metabolizmie cukrów odbywa
się na niskim poziomie (
represja kataboliczna
). Dotyczy to również
operonu laktozowego.
Efekt ten może być zniesiony przez
wysokie stężenia cAMP
i
obecność
białka CAP
.
Rola białka CAP (ang. catabolite activator protein), czyli
białko aktywujące transkrypcję/białko aktywujące geny
kataboliczne i cAMP w regulacji aktywności operonu lac
Obecność glukozy Brak glukozy
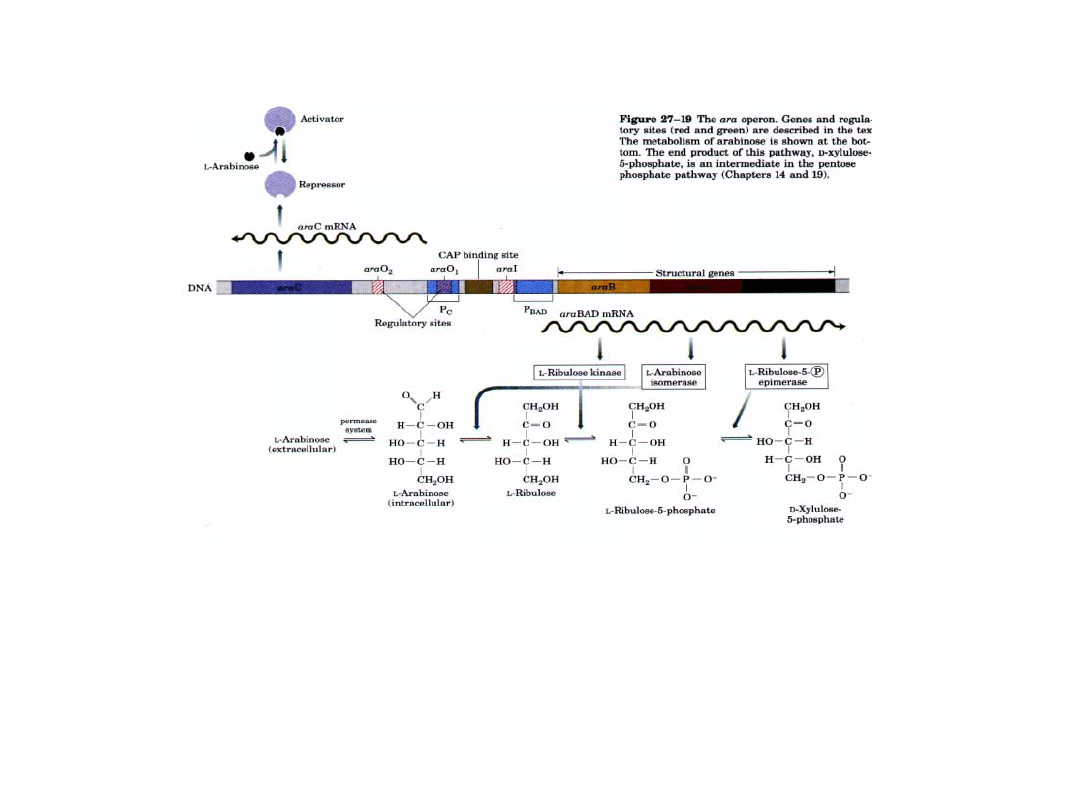
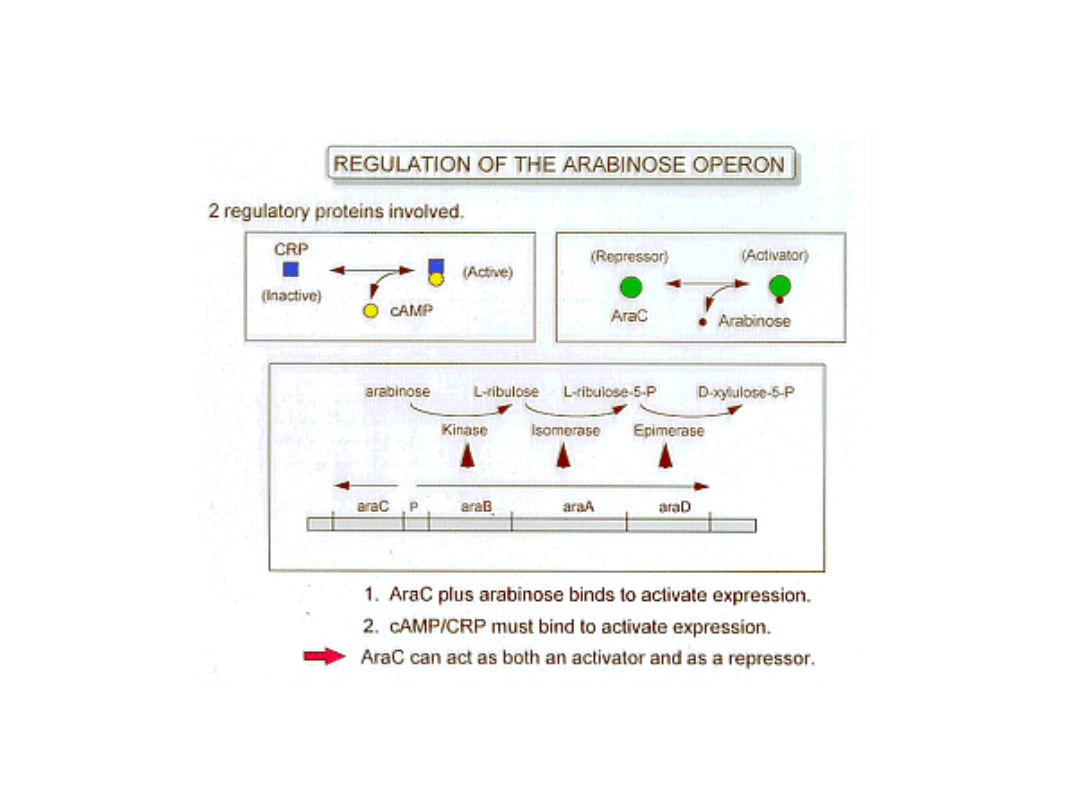
W skład operonu ara wchodzą trzy geny struktury: ara B (kodujący
kinazę L-rybulozy
), araA (kodujący
izomerazę L-arabinozy
) i araD
(kodujący
epimerazę rybulozo-5-P
).
L-Arabinoza L-rybuloza L-rybulozo-5-P D-ksylulozo-5-P
Operon arabinozowy (operon ara) E. coli
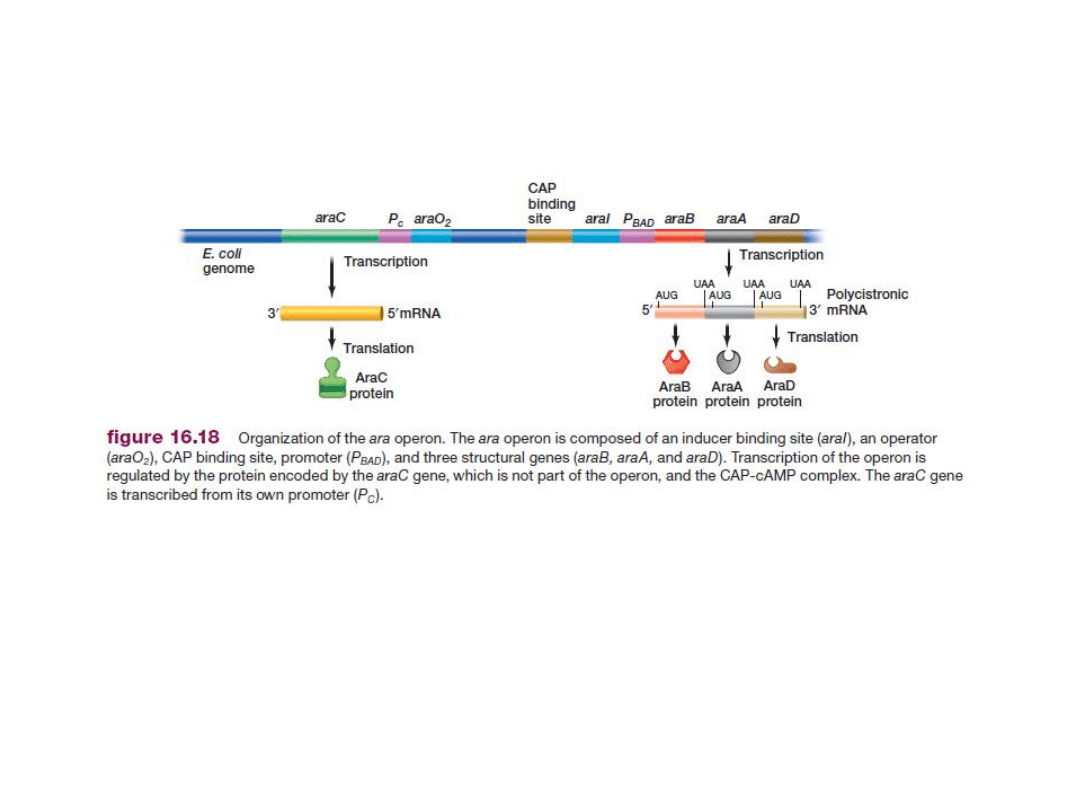
Produktem genu regulatorowego araC jest białko represorowe
AraC. Białko to wiąże się do operatora araO
2
i miejsca
wiązania induktora
araI
(araI
1
i araI
2
). Przed miejscem
wiązania induktora araI leży miejsce wiązania białka CAP.
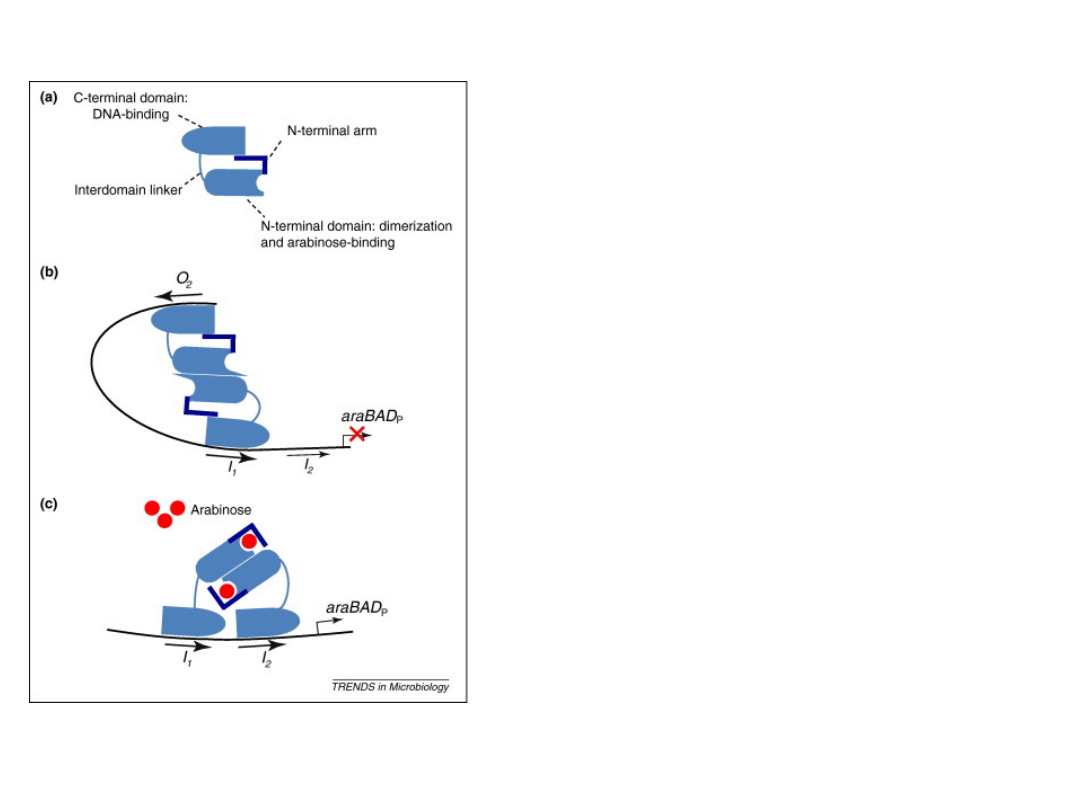
Operon arabinozowy (operon ara)
W nieobecności lub przy niskich
stężeniach arabinozy w środowisku dimery
białka AraC wiążą się do operatora araO
2
i
miejsca wiązania induktora araI (araI1)
hamując transkrypcję. W tym przypadku
białko
AraC
jest
represorem
transkrypcji
.
Przy wysokich stężeniach arabinozy, wiąże
się ona z białkiem AraC, a powstały
kompleks AraC-arabinoza odłącza się od
operatora O
2
i wiąże się do miejsca
wiązania
induktora
araI2,
co
w
konsekwencji
powoduje
rozpoczęcie
transkrypcji genów struktury.
W tym przypadku białko AraC działa jako
aktywator transkrypcji.
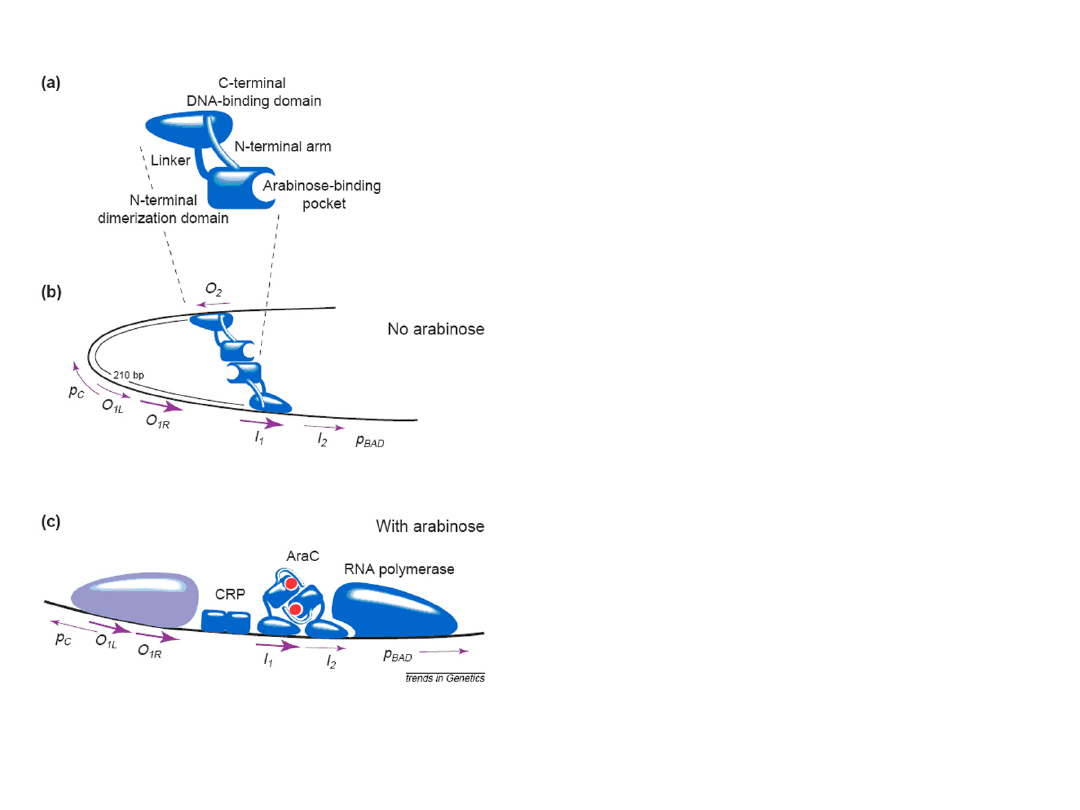
W nieobecności glukozy
W regulacji ekspresji operonu ara
ważną rolę odgrywa białko CAP. W
obecności arabinozy, dla pełnej
aktywności
transkrypcyjnej
tego
operonu konieczne jest związanie z
miejscem CAP, kompleksów białko
CAP-cAMP.
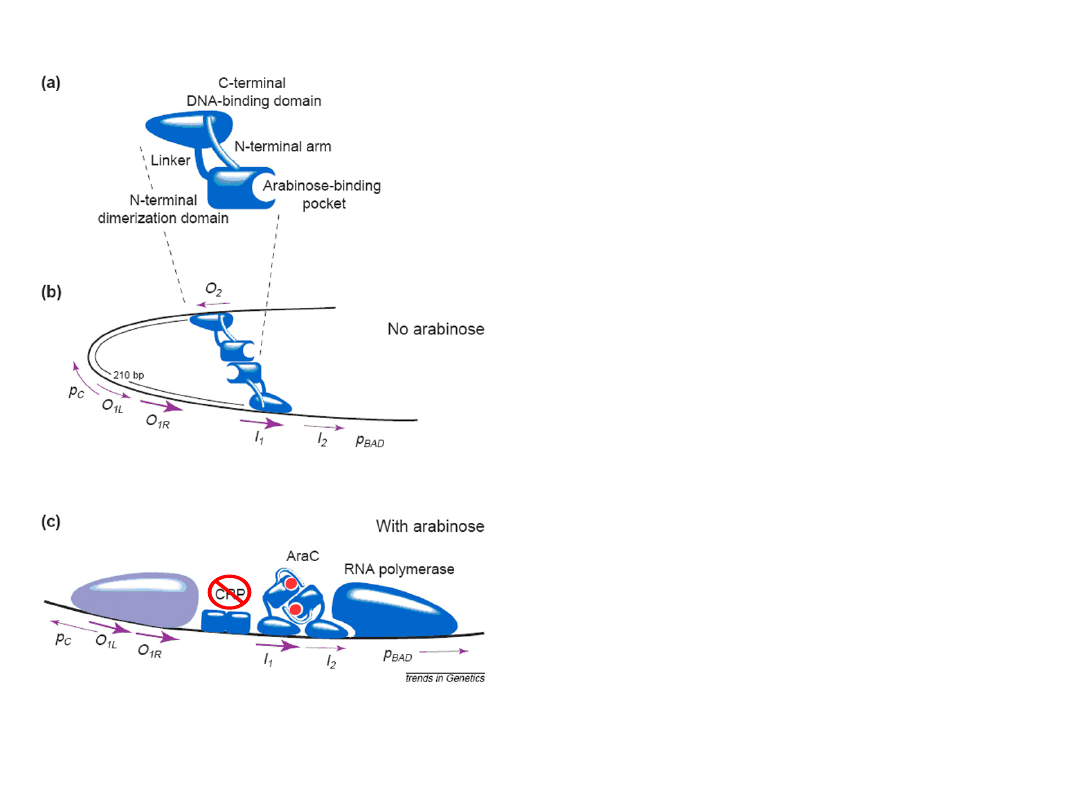
W obecności glukozy
Kiedy w środowisku, obok
arabinozy, występują
równocześnie duże ilości glukozy,
transkrypcja operonu ara
pozostaje na poziomie
podstawowym pozostaje
nieczynny transkrypcyjnie
(znajduje się on w stanie represji
katabolicznej).
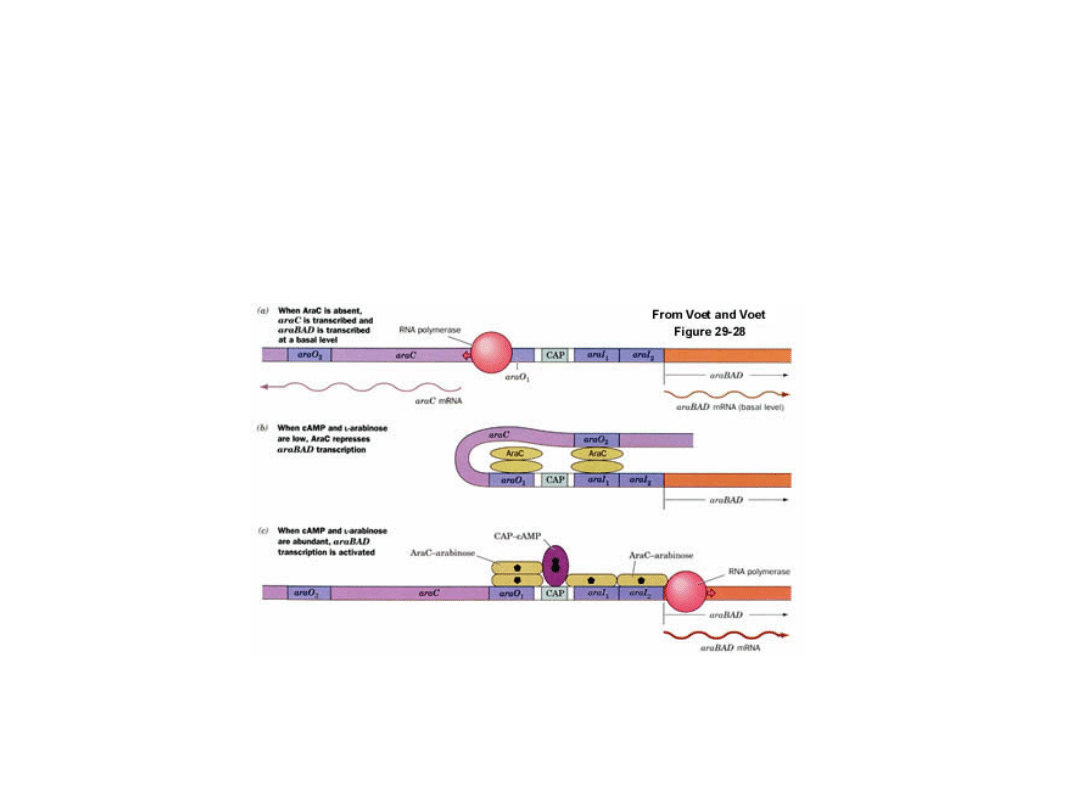
W regulacji ekspresji operonu ara ważną rolę odgrywa białko
CAP. W obecności arabinozy, dla pełnej aktywności
transkrypcyjnej tego operonu konieczne jest związanie z
miejscem CAP, kompleksów białko CAP-cAMP.
Warunek
: nieobecność w pożywce glukozy
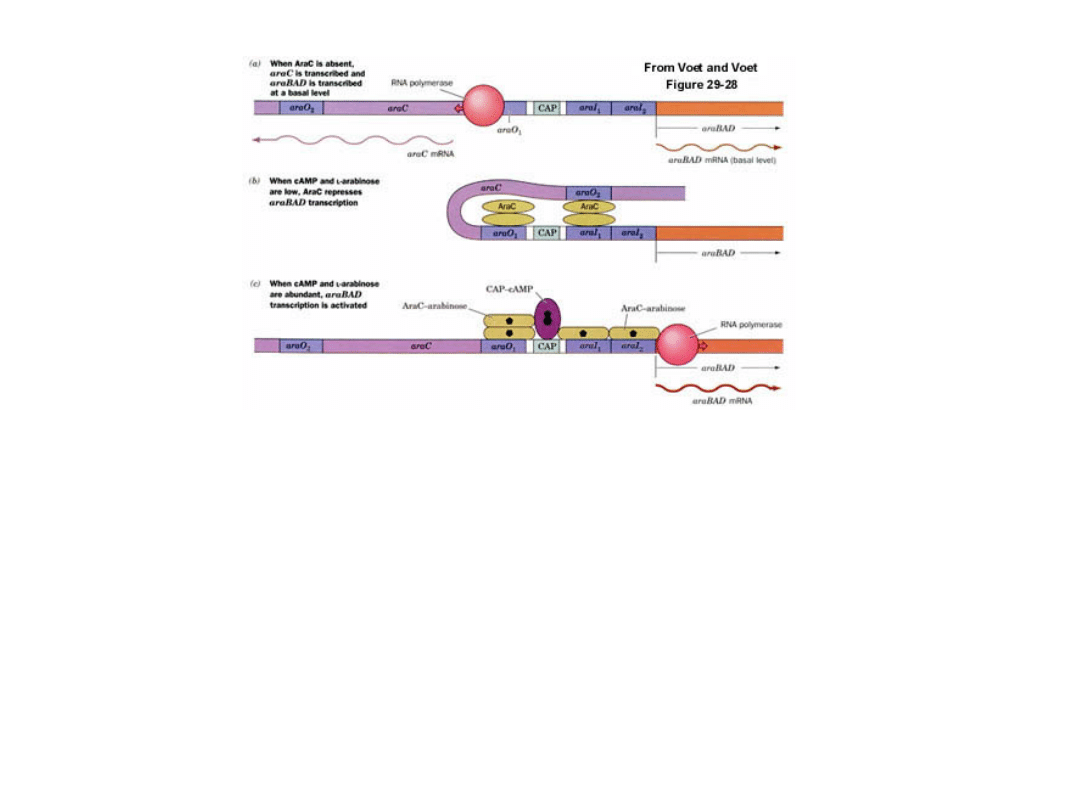
Kiedy w środowisku, obok arabinozy, występują równocześnie duże ilości
glukozy, transkrypcja operonu ara pozostaje na poziomie podstawowym
pozostaje nieczynny transkrypcyjnie (znajduje się on w stanie represji
katabolicznej).
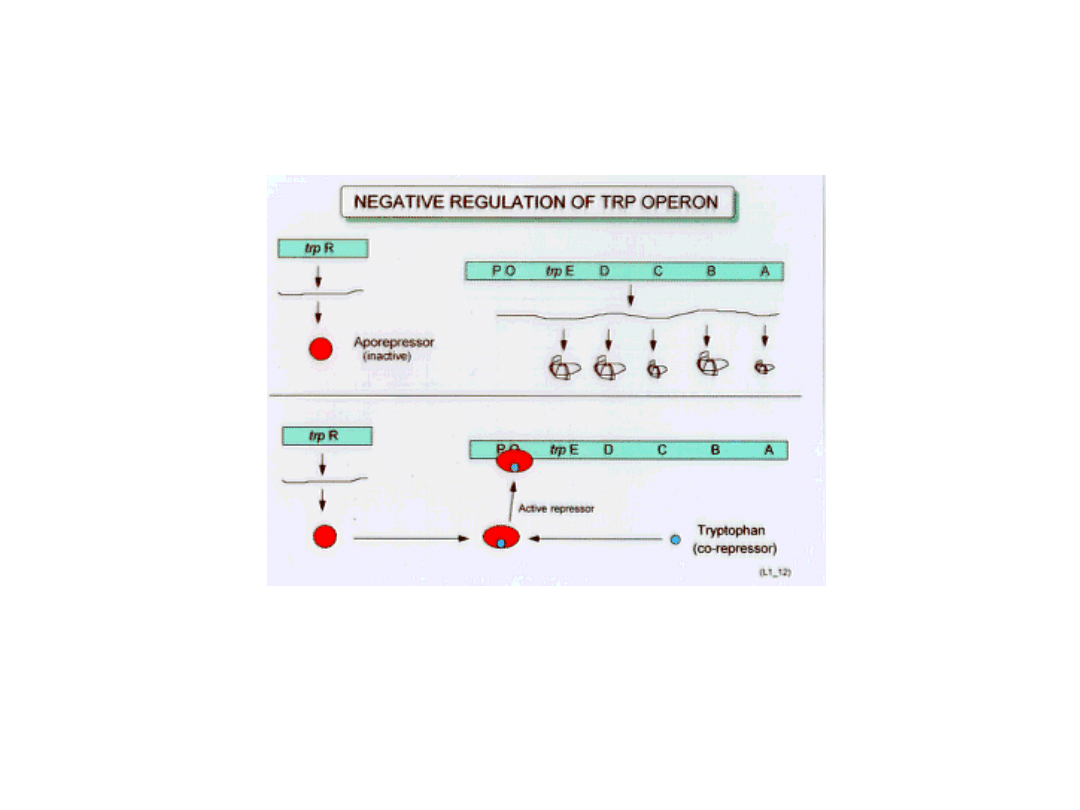
Operon tryptofanowy (operon trp)
E. coli
W nieobecności tryptofanu w pożywce, białko represorowe Trp nie
wiąże się do operatora, co umożliwia transkrypcję genów struktury.
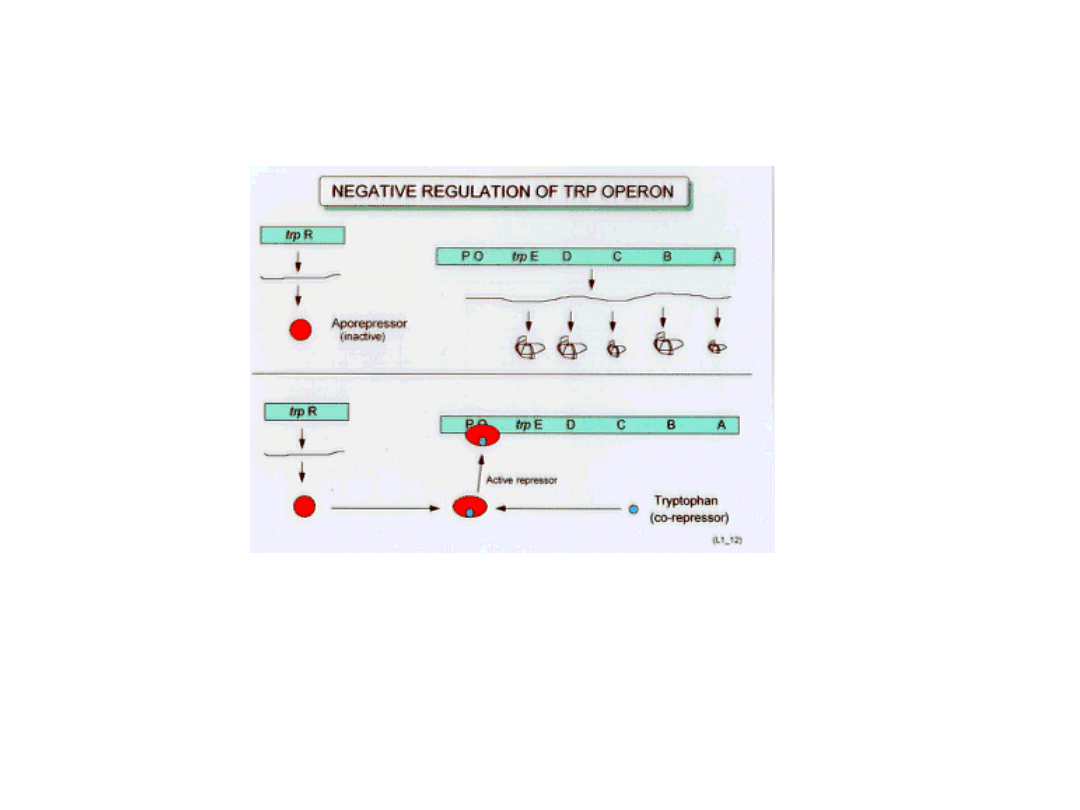
Obecność tryptofanu w środowisku hamuje transkrypcję mRNA
dla enzymów kodowanych przez operon trp.
Tryptofan pełni rolę
korepresora
, ponieważ po związaniu się z białkiem
represorowym Trp zwiększa jego powinowactwo do operatora i tym samym
umożliwia jego związanie z tym odcinkiem DNA.
Generalnie, drobnocząsteczkowe związki, które po związaniu z
represorem zwiększają jego powinowactwo do operatora, noszą nazwę
korepresorów
.
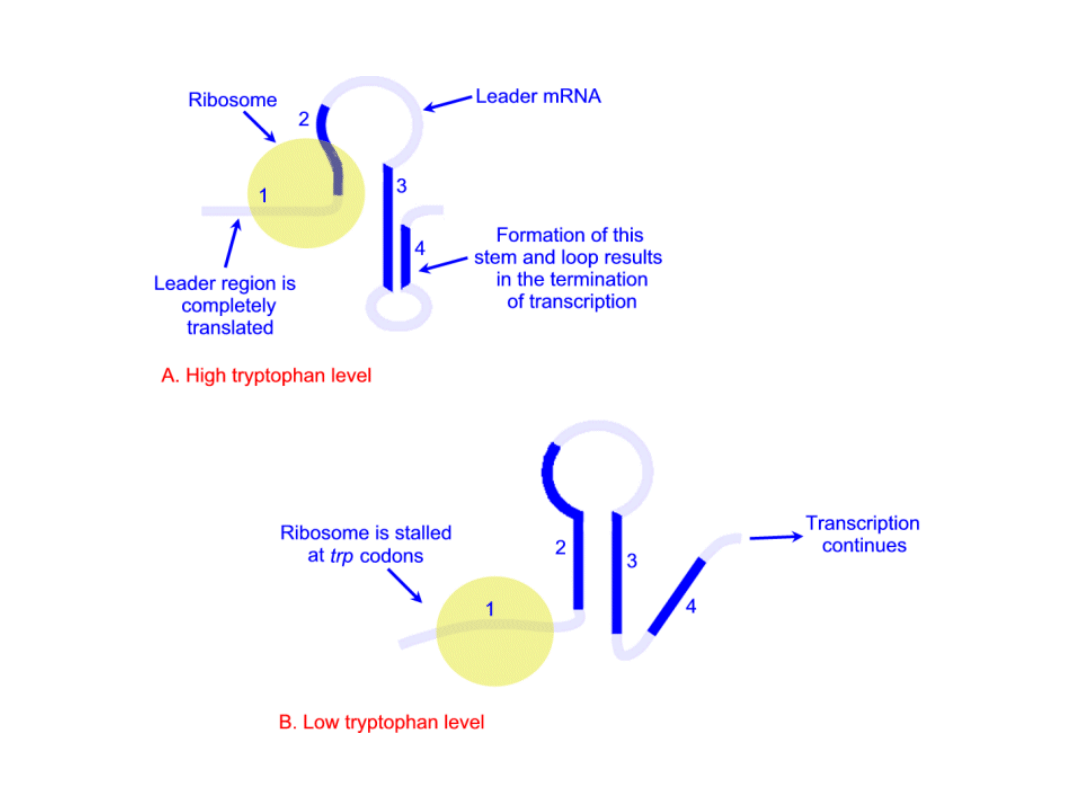
Regulacja transkrypcji na drodze atenuacji, mechanizmu
wykorzystującego ścisłą zależność pomiędzy transkrypcją i translacją
u prokariota.
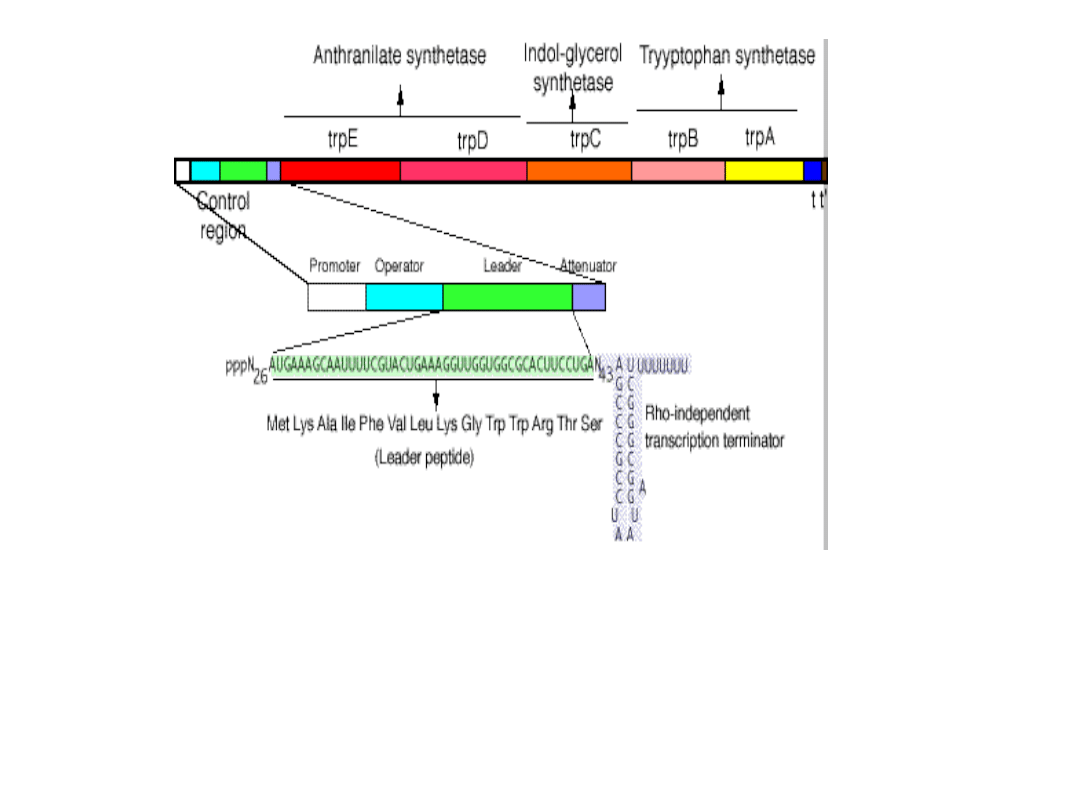
Większość
operonów
kodujących
enzymy
biosyntezy
aminokwasów posiada odcinek około 160 nukleotydów
podlegających transkrypcji i translacji, leżących za odcinkiem
promotorowo-operatorowym, który nosi nazwę sekwencji
liderowej/odcinka liderowego.
Sekwencja liderowa zakończona jest miejscem kontrolowanej
terminacji transkrypcji tzw.
atenuatorem
, tworzącym
strukturę „spinki do włosów”. W miejscu atenuatorowym
dochodzi do odłączania cząsteczek polimerazy RNA
jeszcze przed przed transkrypcją genów struktury.
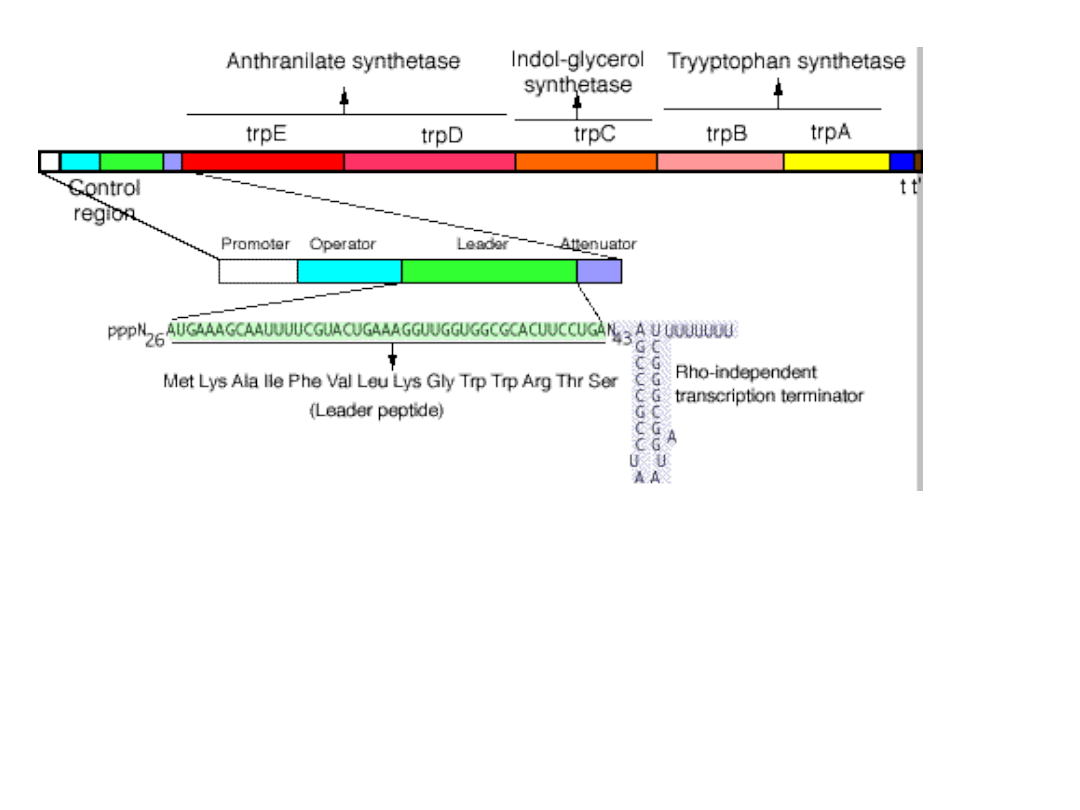
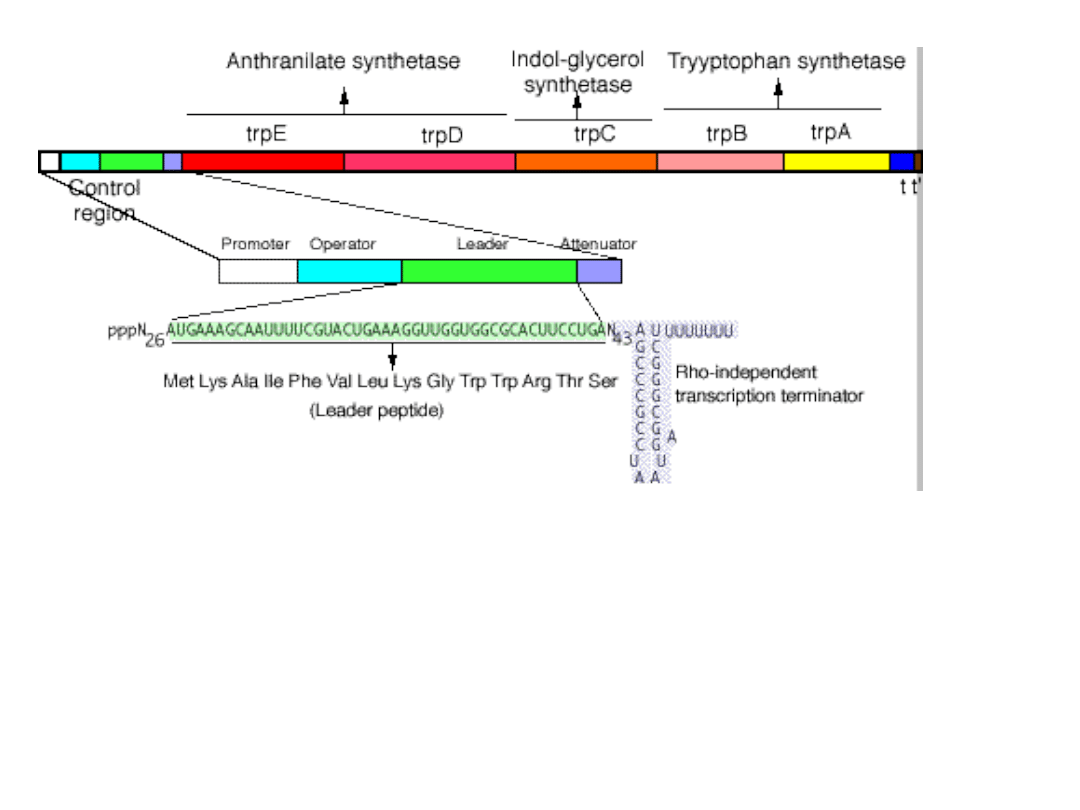
Liczba cząsteczek polimerazy RNA odłączająca się od operonu
trp w miejscu atenuatorowym zależy od stężenia tryptofanu w
środowisku. Im niższe jest stężenie tego aminokwasu, tym
większa jest liczba cząsteczek polimerazy kontynuujących
transkrypcję.
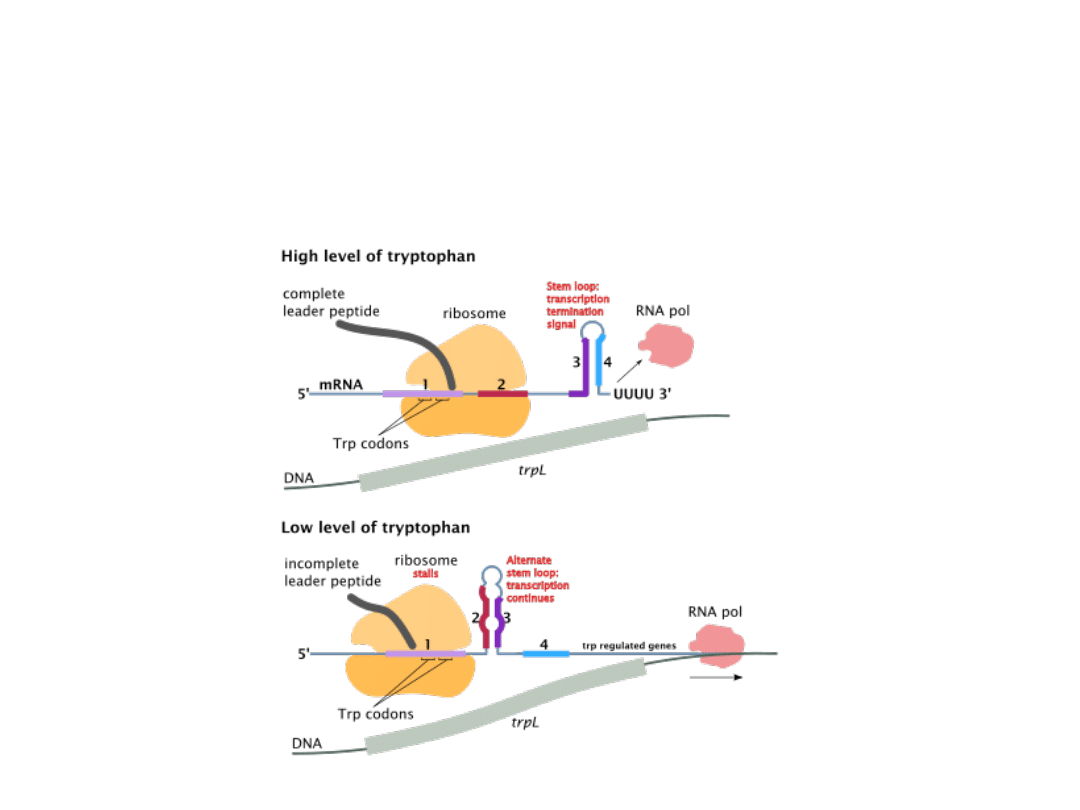
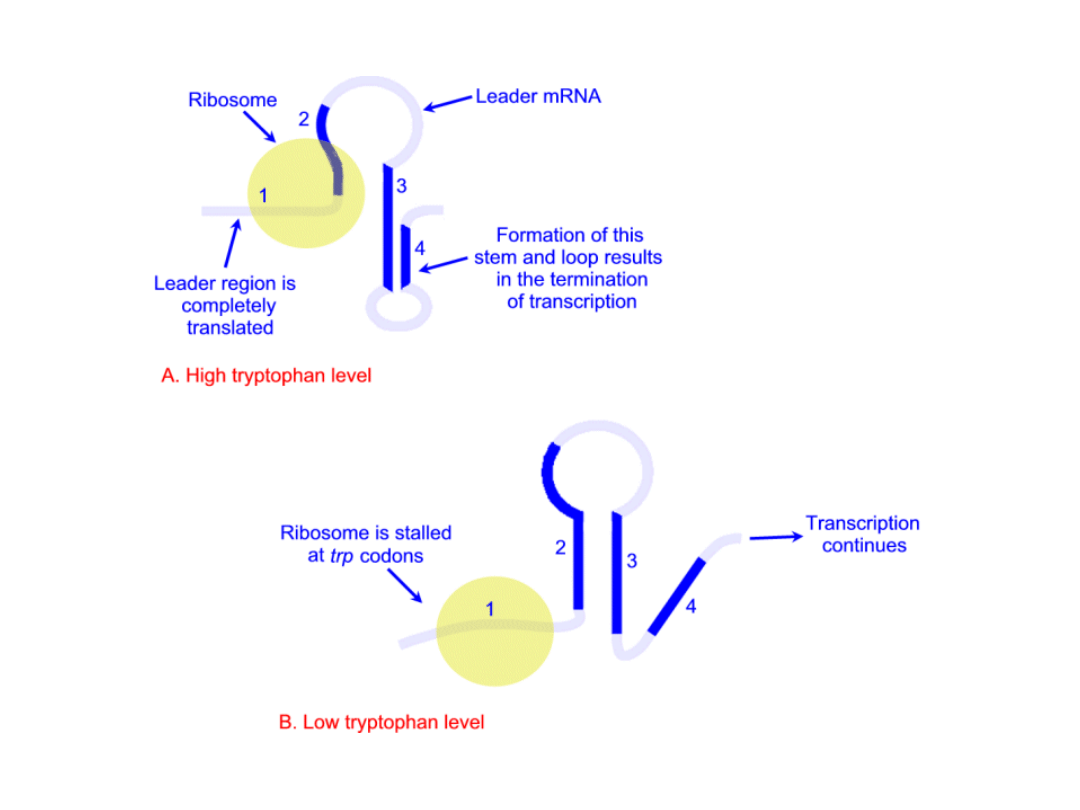
Wysokie stężenie tryptofanu
Przy wysokim stężeniu tryptofanu i tym samym wystarczających ilościach Trp-
tRNAtrp translacja odcinka liderowego przebiega w sposób niezakłócony. Pozwala
to na wytworzenie w mRNA terminacyjnej struktury „spinki do włosów”
będącej sygnałem dla zakończenia transkrypcji.
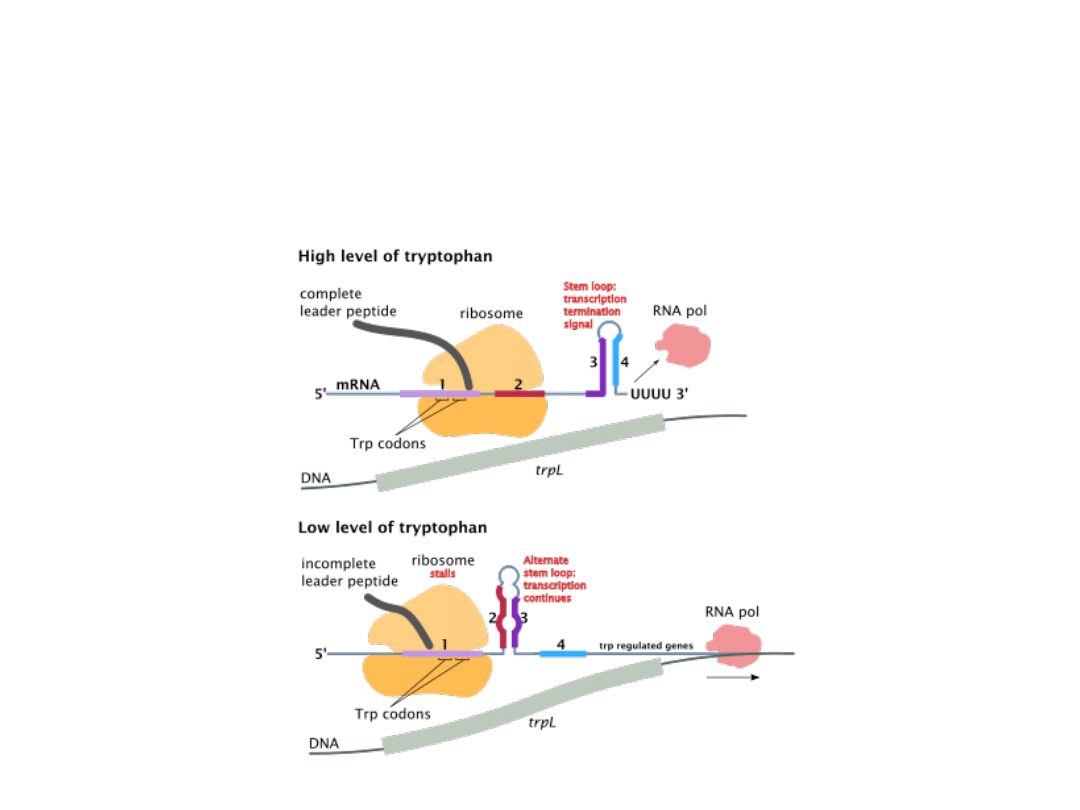
Niskie stężenie tryptofanu
Przy niskich stężeniach tryptofanu i braku wystarczających ilości Trp-tRNAtrp
dochodzi do zatrzymania translacji sekwencji liderowej w miejscu gdzie
występują obok siebie dwa kodony tryptofanowe. Zatrzymanie rybosomu
powoduje zmianę w strukturze drugorzędowej mRNA polegającą na
wytworzeniu struktury antyterminacyjnej, co pozwala na kontynuację
transkrypcji.
Regulacja ekspresji genów u Eukariota
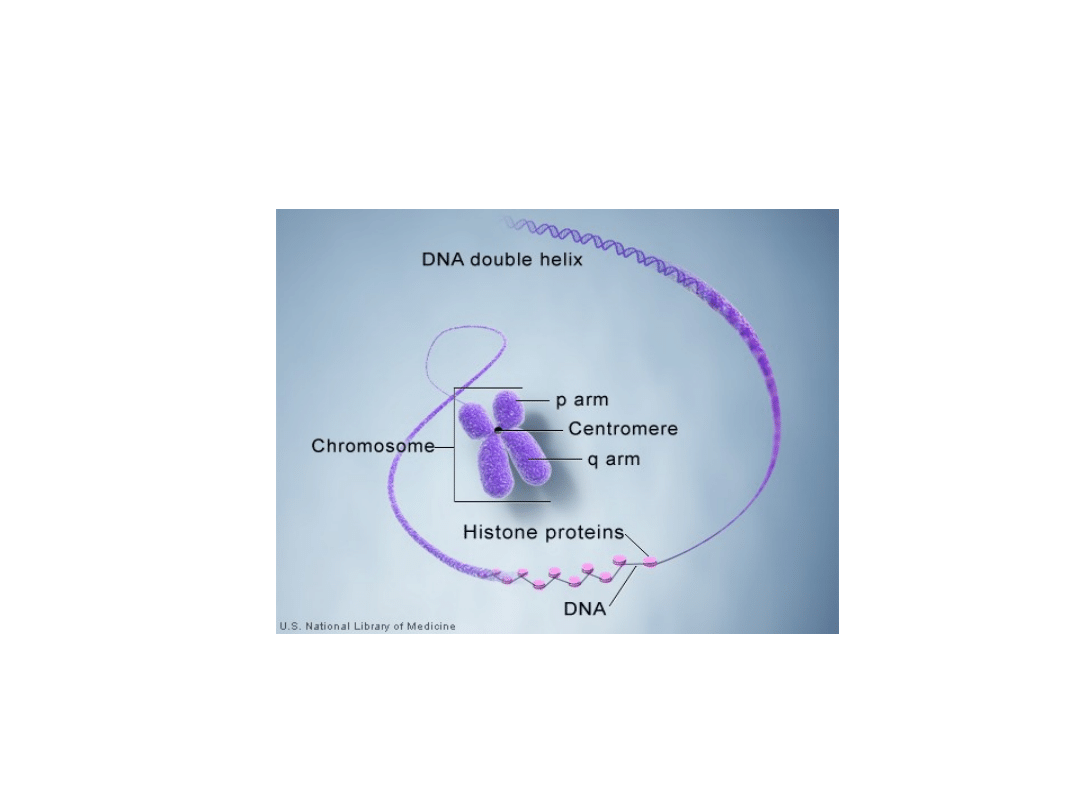
W komórkach eukariotycznych DNA zlokalizowany w jądrze
rozmieszczony jest w specjalnych strukturach zwanych
chromosomami.
Każdy chromosom utworzony jest z cząsteczki DNA, z którą
zasocjowane są białka biorące udział w jego zwijaniu w bardziej
upakowane struktury wyższego rzędu.
Nukleoproteinowy kompleks utworzony z DNA i białek nosi
nazwę chromatyny. Obok białek biorących udział w
„upakowaniu” DNA (głównie histony), w skład chromatyny
wchodzą białka zaangażowane w procesy replikacji i naprawy
DNA, transkrypcji RNA i regulację ekspresji genów.
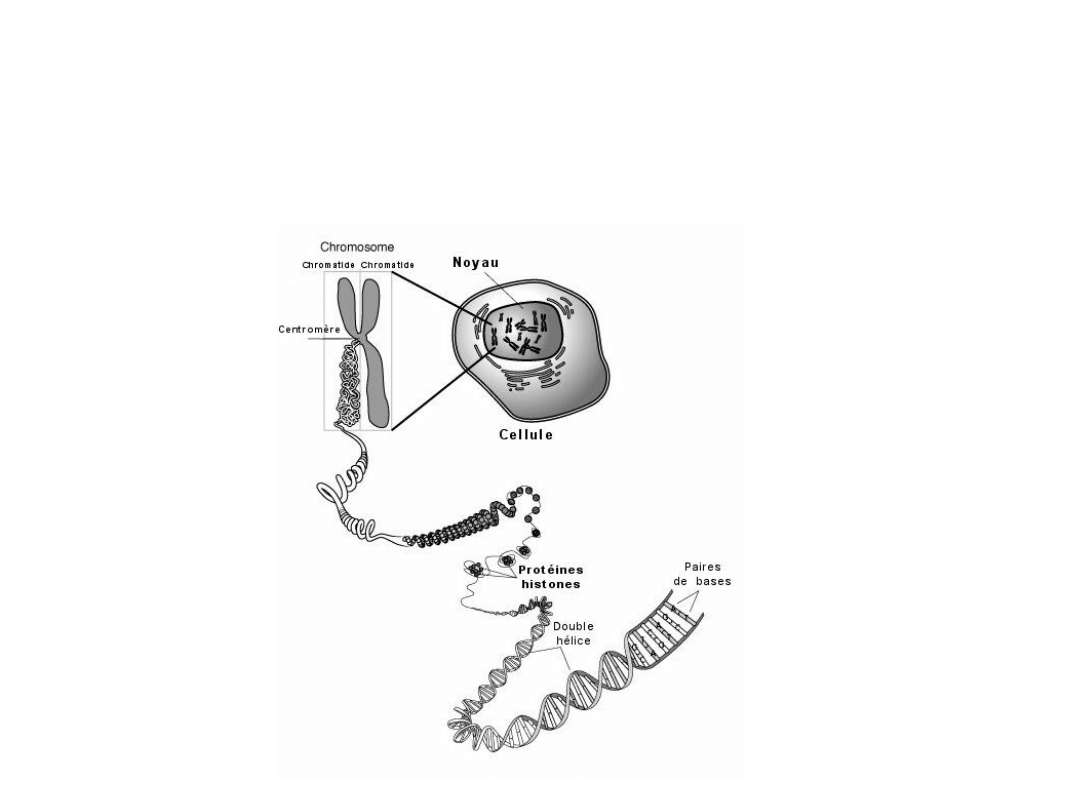
Organizacja chromatyny
Chromatyna zbudowana jest z powtarzających się
jednostek noszących nazwę nukleosomów, utworzonych z
kompleksów białkowych, wokół których prawie dwukrotnie
owinięta jest cząsteczka DNA.

Każdy z nukleosomów obejmuje kompleks białkowy, tzw.
rdzeń nukleosomu, w skład którego wchodzi 8 białek
histonowych:
- dwie cząsteczki histonu H2A,
- dwie cząsteczki histonu H2B,
- dwie cząsteczki histonu H3,
- dwie cząsteczki histonu H4,
- odcinek podwójnej helisy DNA o długości 165 - 240
nukleotydów.
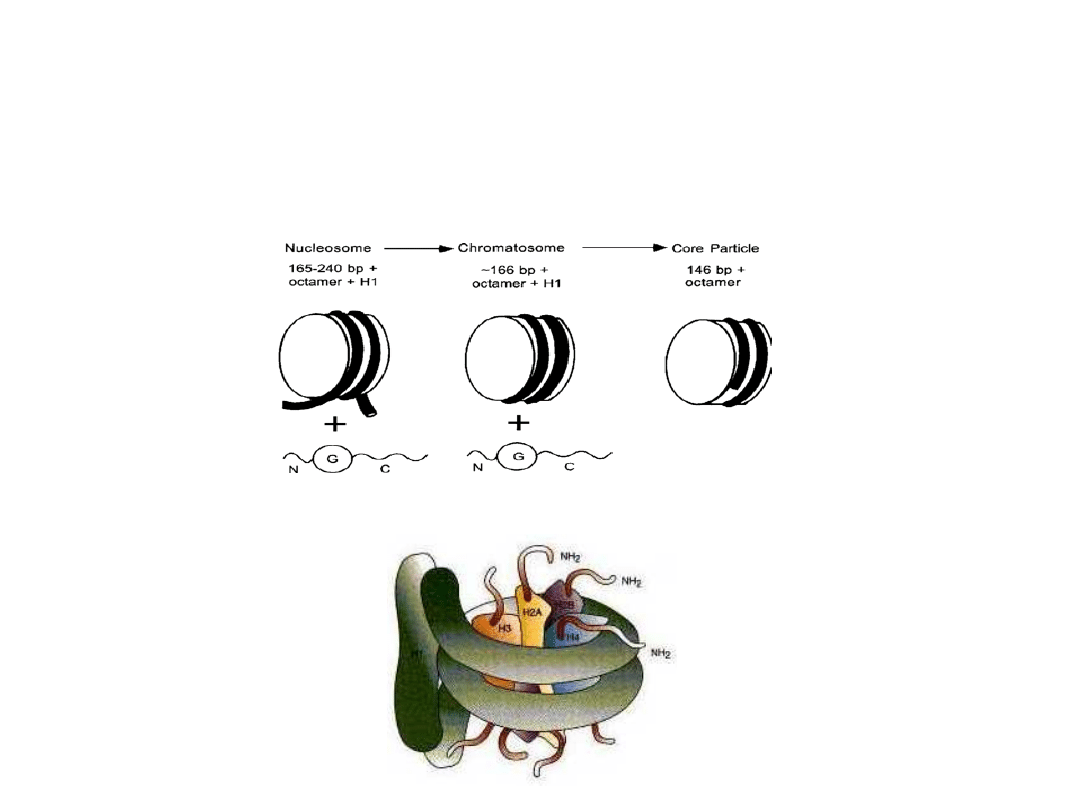
Oktamer histonów tworzy strukturę podobną do dysku.
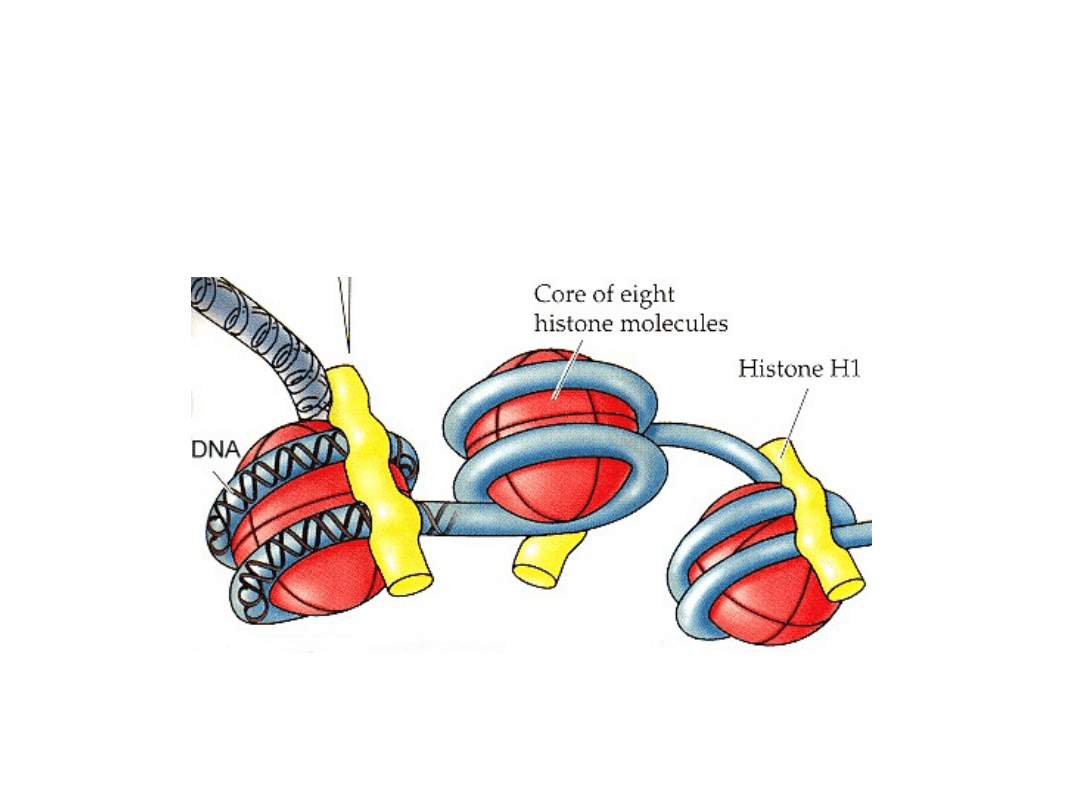
Fragment DNA łączący dwa nukleosomy nosi nazwę DNA
łącznikowego, z którym częściowo oddziałuje histon H1.
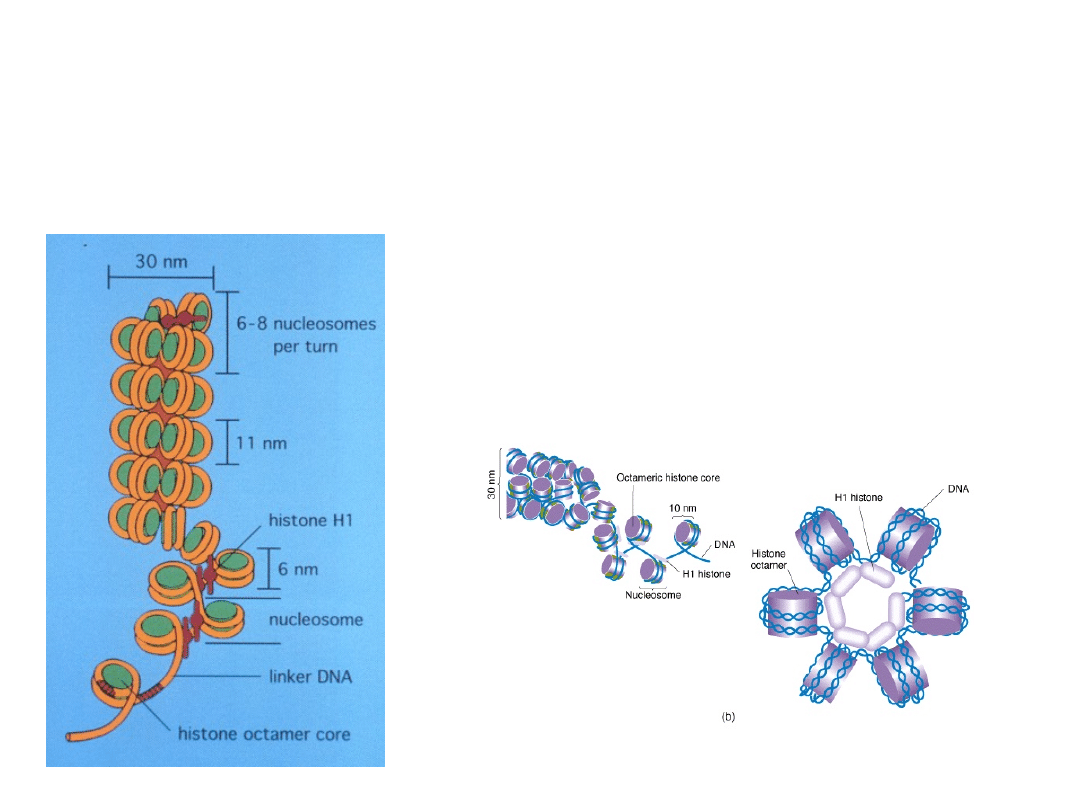
Chromatyna podlega kolejnemu poziomowi upakowania
tworząc nić chromatynową o średnicy 30 nm. Ta forma
przestrzennej organizacji DNA nosi nazwę solenoidu.
W powstaniu struktury solenoidu
udział biorą cząsteczki histonu H1,
które powodują, że nukleosomy
zostają
gęsto
upakowane
w
regularnie powtarzające się struktury
wyższego rzędu.
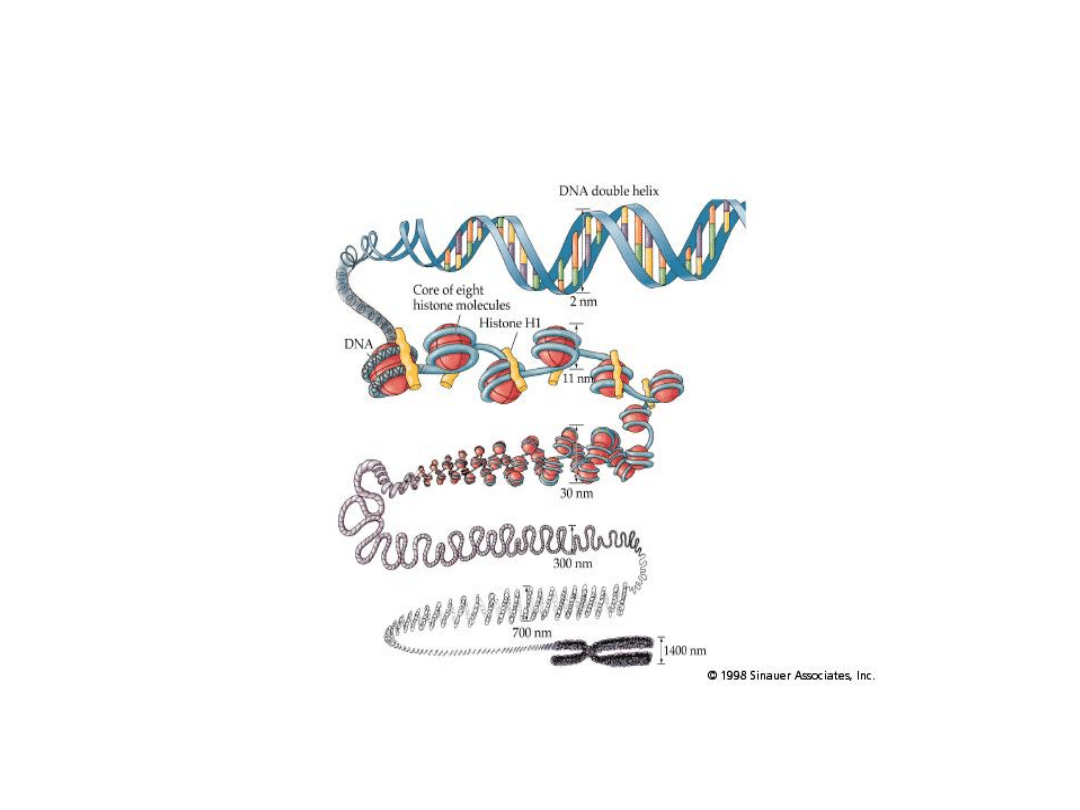
Nić chromatynowa o średnicy 30 nm ulega dalszemu
upakowaniu tworząc pętle (domeny) wiążące się z macierzą
jądrową. Biegną one na boki od osi głównej cząsteczki DNA.
Kolejne etapy kondensacji (supersolenoid, superhelisa)
prowadzą do powstania chromosomu mitotycznego.

W chromosomie mitotycznym, z powodu ścisłego
upakowania cząsteczek DNA nie dochodzi do
transkrypcji RNA, ponieważ polimerazy RNA i inne
białka nie mają dostępu do nici DNA.
Najbardziej upakowana forma chromatyny nosi
nazwę heterochromatyny. Stanowi ona około 10%
całej chromatyny i w komórkach ssaków znajdowana
jest głównie wokół centromeru i przy końcach
chromosomów. Jest ona transkrypcyjnie całkowicie
nieaktywna.
Przykładem chromosomu, który „permanentnie”
charakteryzuje
się
wysoką
zawartością
heterochromatyny jest jeden z pary chromosomów X
w komórkach osobników żeńskich u ssaków.

Ta
część
chromatyny
w
chromosomach
interfazowych, która pozostaje w mniej lub bardziej
rozwiniętym stanie nosi nazwę euchromatyny. W
typowej komórce eukariotycznej około 10% tej
chromatyny podlega aktualnie transkrypcji lub jest
gotowa
do
jej
rozpoczęcia,
tzw.
aktywna
chromatyna.
Aktywna chromatyna ma stopień organizacji
odpowiadający nukleosomom, podobnie jak DNA
podlegający replikacji.

Powtarzające się sekwencje DNA
Sekwencje DNA występujące w genomach
organizmów
prokariotycznych
jak
i
eukariotycznych dzielą się na:
- sekwencje unikatowe odpowiadające
genom kodującym białka, rybosomalne RNA, tRNA
i drobnocząsteczkowe RNA, wraz z sekwencjami
regulatorowymi,
- sekwencje powtórzone występujące w
liczbie 2 – 10
7
/komórkę,
o bliżej nieokreślonej
funkcji.
Sekwencje powtórzone występują w specjalnie
dużej liczbie w genomach człowieka i małp
naczelnych.

Sekwencje o długościach 100 – 500 pz określa się nazwą
krótkie rozproszone powtórzenia, w skrócie SINES (ang.
short interspersed repeats).
Przykładem sekwencji SINES są bardzo krótkie, kilku- do
kilkunasto-nukleotydowe sekwencje, występujące jedna za
drugą, tworzące tzw. satelitarny DNA, którego obecność
stwierdza się w centromerach i zakończeniach chromosomów.
Sekwencje liczące do kilku tysięcy nukleotydów noszą nazwę
długich rozproszonych powtórzeń, w skrócie LINES (ang.
long interspersed repeats).
W genomie człowieka typowym przykładem sekwencji typu
LINES są występujące w liczbie około 10
6
kopii sekwencje Alu o
długości 300 nukleotydów.

Głównym poziomem regulacji ekspresji genów w
komórkach eukariotycznych jest transkrypcja
Regulacja transkrypcyjna dotyczy:
1. wielu genów równocześnie,
2. indywidualnych genów.
Pierwszy typ regulacji związany jest ze stopniem
upakowania
chromatyny
(heterochromatyna
v.
euchromatyna). W stabilizacji nieaktywnej chromatyny
ważną rolę odgrywa metylacja DNA przy udziale
swoistej metylotransferazy. Dotyczy ona cytozyny przy
węglu C-5, wchodzącej w skład sekwencji CG.
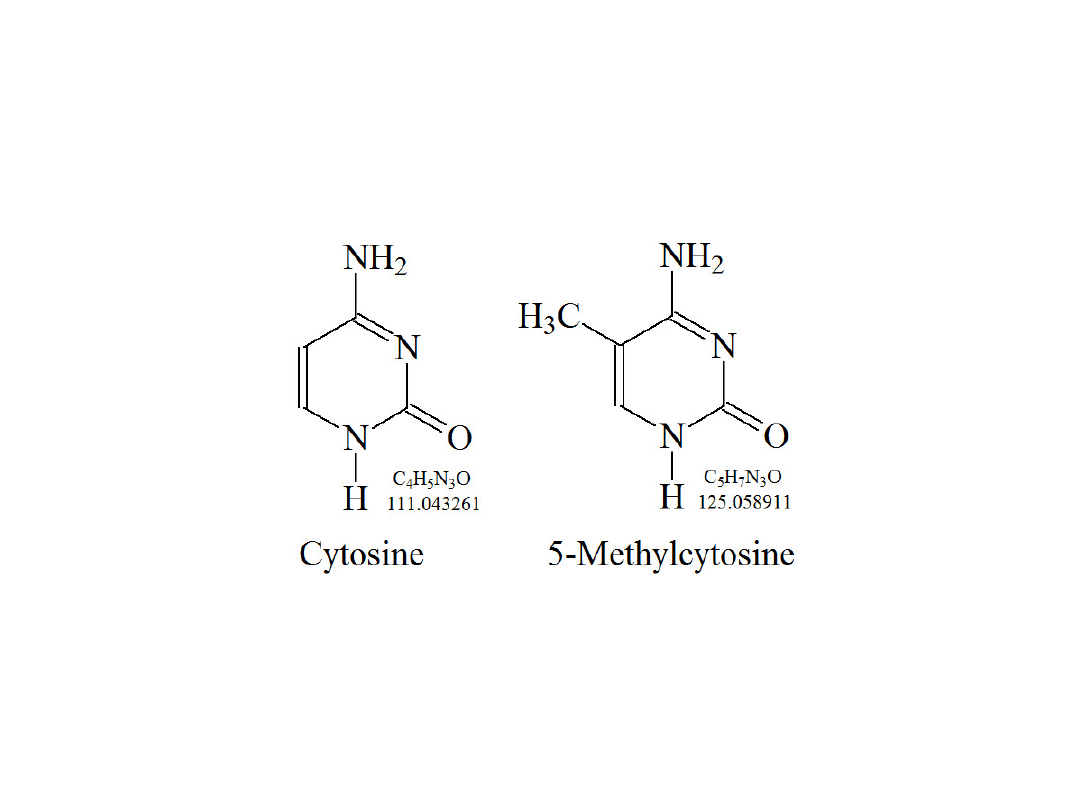
W regulacji ekspresji pojedynczych genów udział biorą dwie
klasy sekwencji regulatorowych:
- sekwencje typu trans kodujące białkowe czynniki
transkrypcyjne.
- sekwencje typu cis, do których należą: (i) sekwencje
wzmacniające i sekwencje wyciszające, (ii) elementy odpowiedzi
(ang. response elements).
Sekwencje wzmacniające lub wyciszające mają długość od
kilkudziesięciu do kilkuset nukleotydów. Mogą one działać z odległości
kilku do kilkudziesięciu tysięcy par zasad (50 kpz), niezależnie od ich
położenia (powyżej albo poniżej miejsca transkrypcji) i orientacji
względem promotora.
Elementy odpowiedzi w odróżnieniu od sekwencji
wzmacniających/wyciszających leżą w odległości kilkuset nukleotydów
powyżej promotora. Są odpowiedzialne za skoordynowaną regulację
ekspresji kilku genów. Przykład to geny regulowane przez hormony
sterydowe.
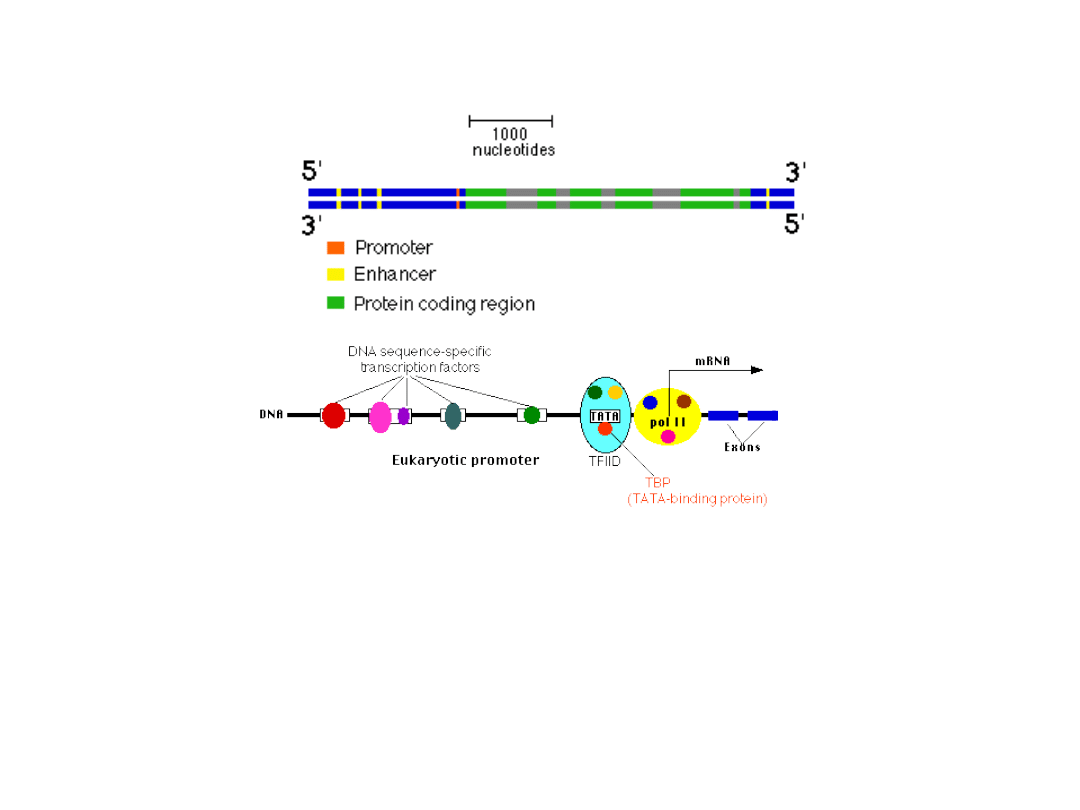
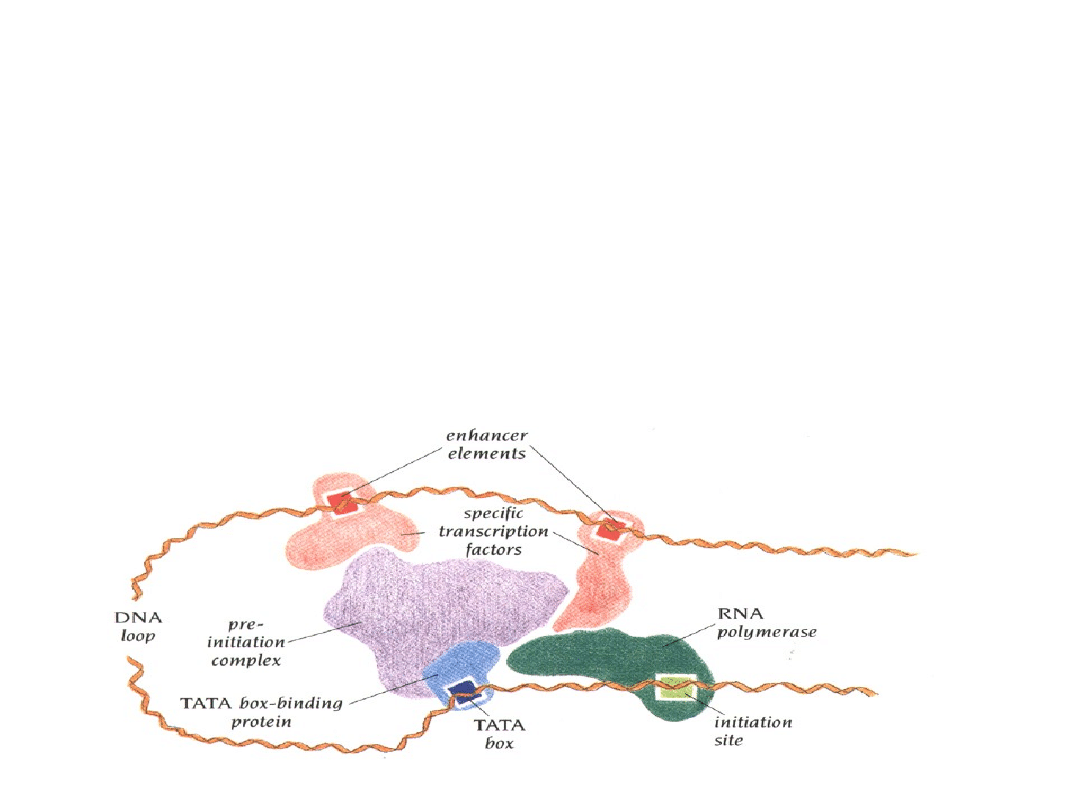
Sekwencje wzmacniające/wyciszające
Sekwencje
wzmacniające
i
wyciszające
wiążą
białka
regulatorowe - czynniki transkrypcyjne, które poprzez
zmianę struktury chromatyny, odpowiednio,
zwiększają
lub
zmniejszają
poziom transkrypcji określonych genów, czyniąc je
bardziej dostępnymi dla innych białek biorących udział w tym
procesie. To, pozwala działać sekwencjom wzmacniającym ze
znacznych odległości.
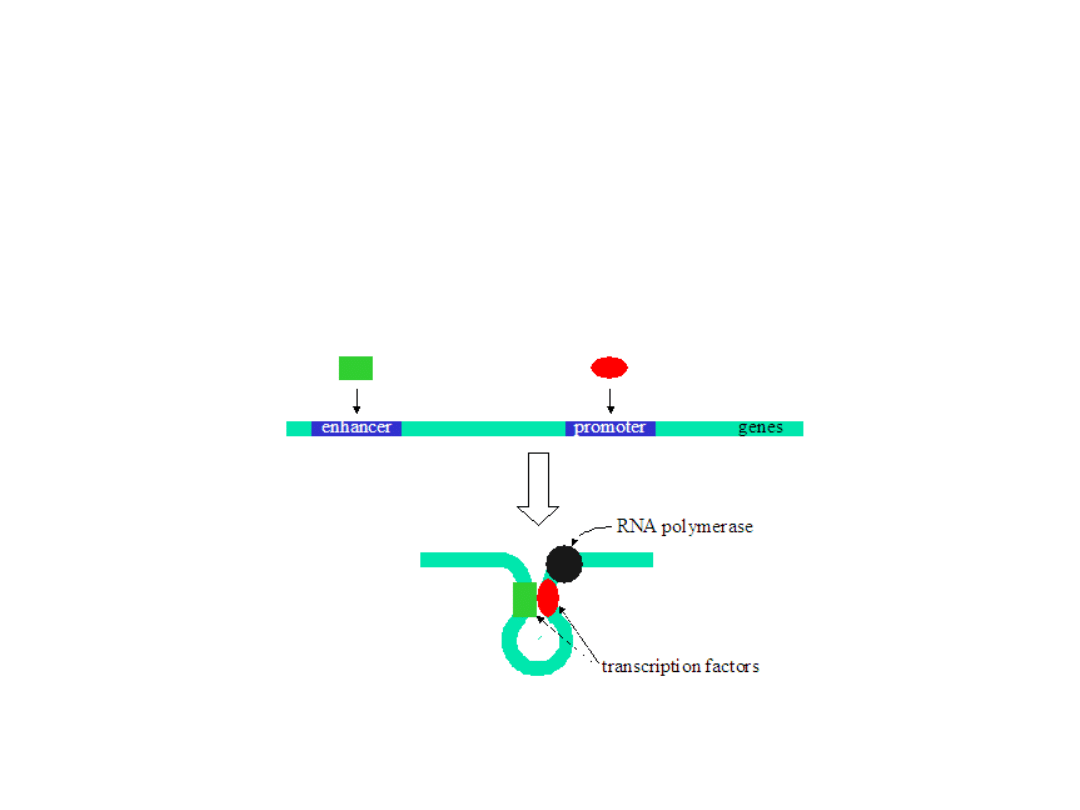
Działanie „na odległość” takich sekwencji regulatorowych
tłumaczy
się
tworzeniem
pętli
DNA
pomiędzy
wzmacniaczem i promotorem, co z kolei pozwala na wejście w
bezpośredni kontakt białka regulatorowego z polimerazą RNA
albo jednym z ogólnych czynników transkrypcyjnych.

Czynniki transkrypcyjne
Czynniki transkrypcyjne to białka oddziałujące ze swoistymi sekwencjami
regulatorowymi, odgrywające bezpośrednią rolę w regulacji ekspresji genów.
Są to białka wielodomenowe, w strukturze których wyróżniamy:
-
domenę wiążącą DNA
,
- domenę dimeryzacyjną, często stanowiącą część domeny
wiążącej DNA,
- domenę aktywującą transkrypcję odpowiedzialną za
oddziaływania z innymi białkami kompleksu transkrypcyjnego,
- domenę wiążącą ligand.
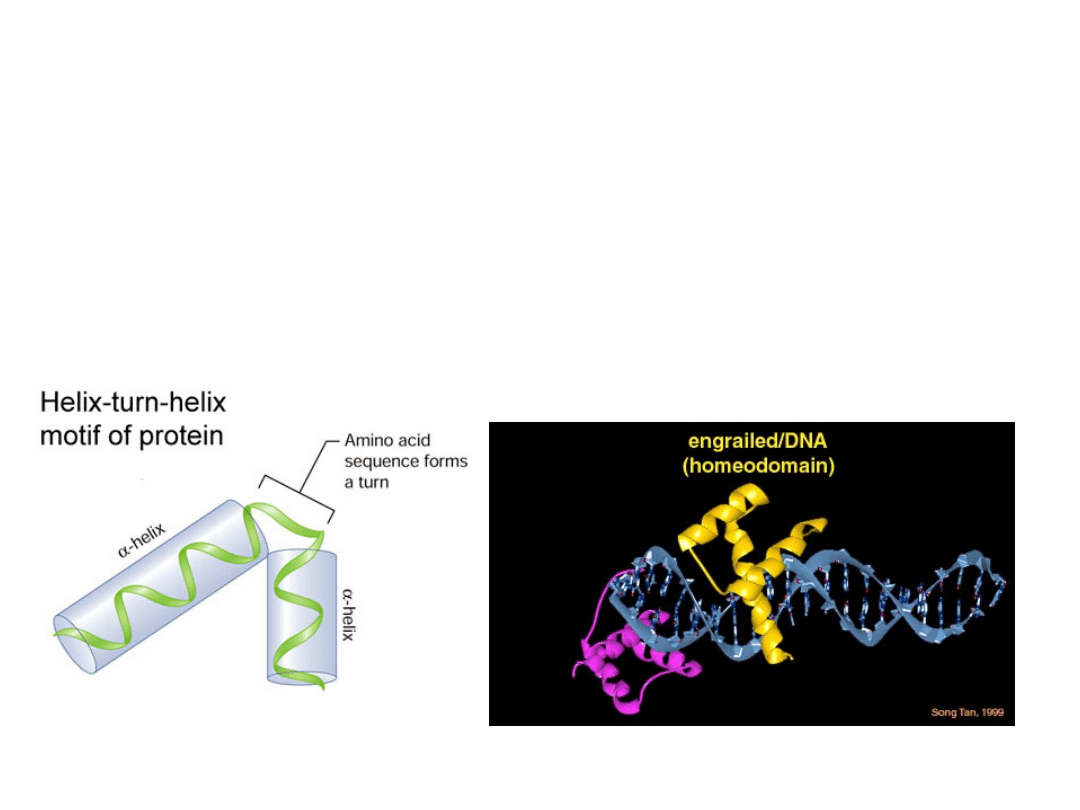
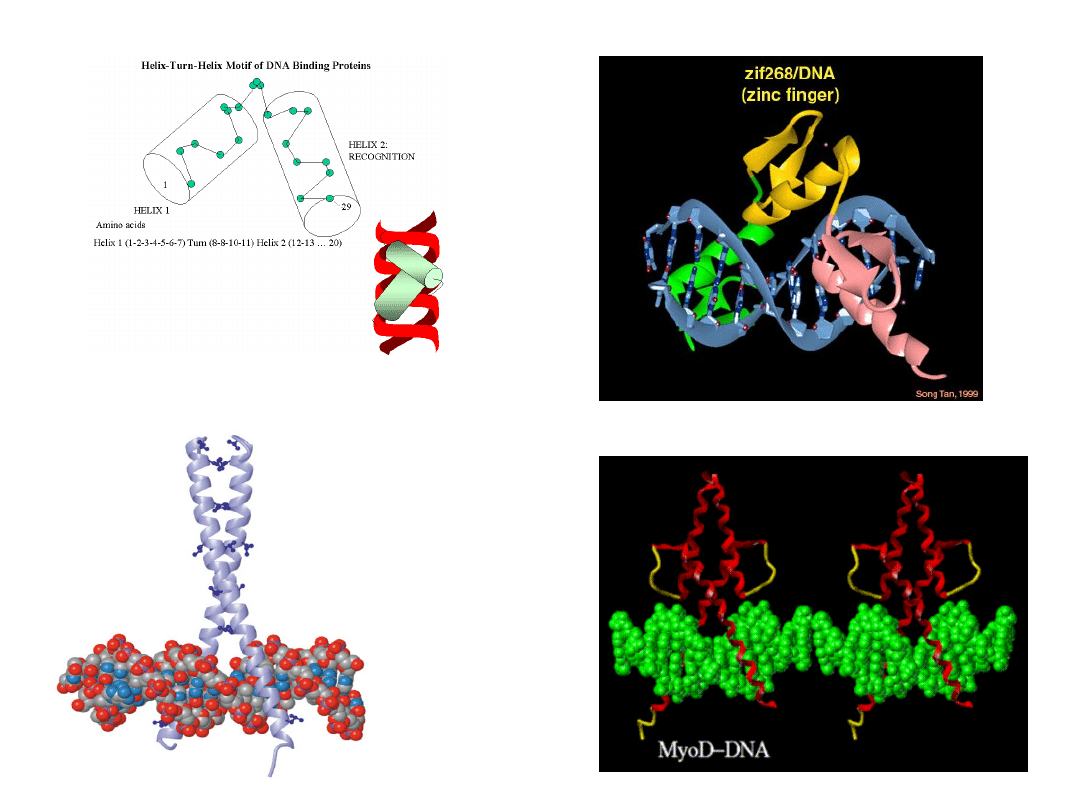
Motywy strukturalne występujące w domenach
wiążących DNA
Motyw helisa-skręt/zwrot-helisa (HTH) utworzony jest z
dwóch α-helis oraz zwrotu . Ten typ struktury występuje w
domenach
wiążących
DNA
czynników
transkrypcyjnych
zawierających tzw. homeodomeny obejmujące 60 reszt
aminokwasowych, tworzących trzy
α-helisy.
Występuje w czynnikach transkrypcyjnych biorących udział w
różnicowaniu komórek hematopoetycznych.
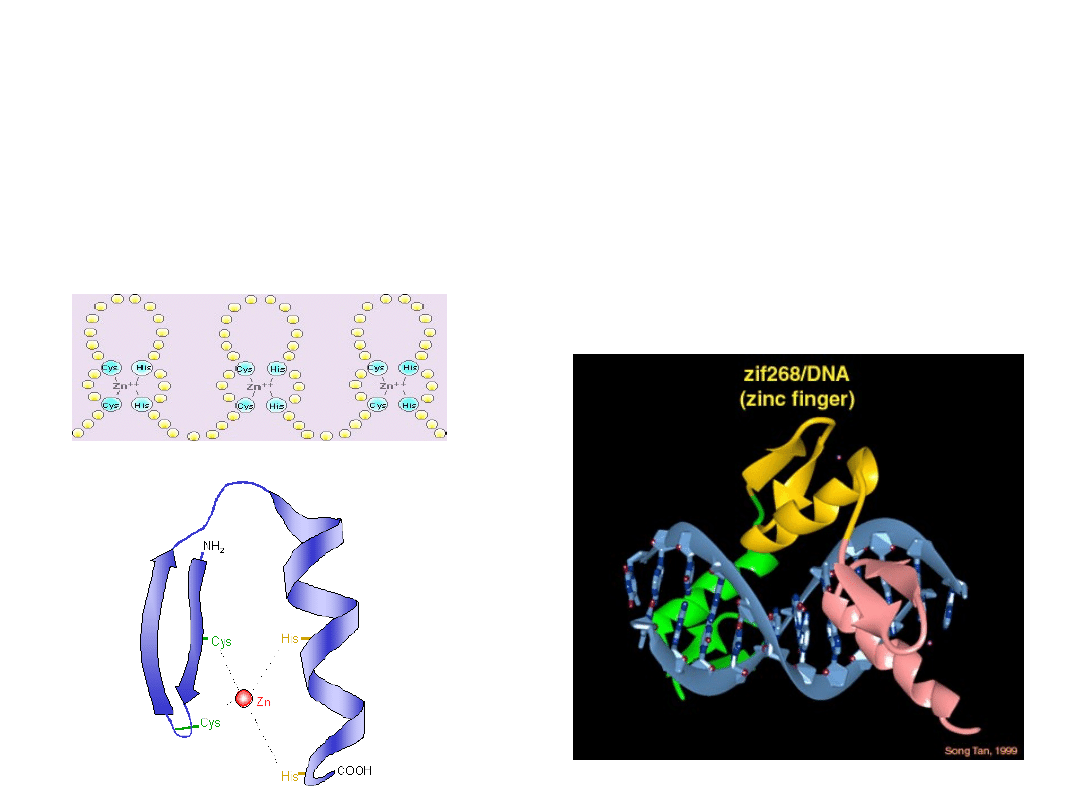
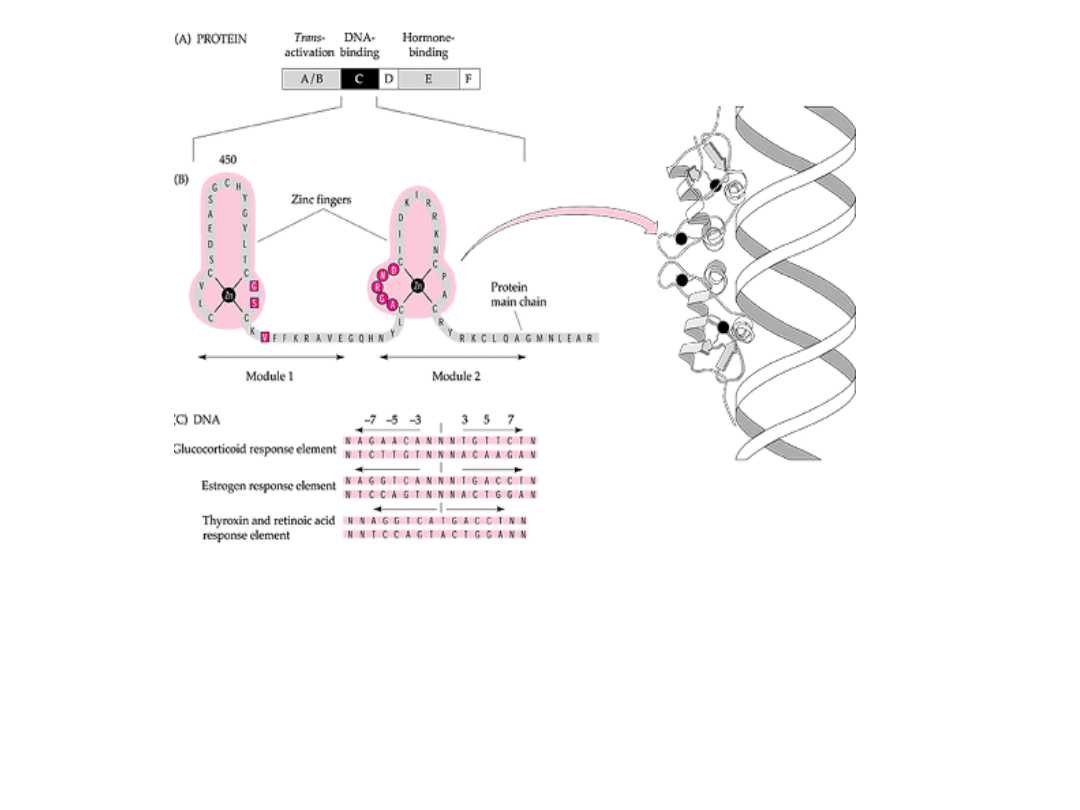
Motyw palca cynkowego tworzą albo pary histydyn i
cystein (C
2
H
2
) lub dwie pary cystein (C
4
) połączonych ze
sobą jonem cynku. To powoduje, że położony między tymi
parami aminokwasów fragment łańcucha polipeptydowego
ulega wypętleniu. Motyw palca cynkowego posiada np.
czynnik GATA-1 biorący udział w regulacji ekspresji globiny w
komórkach erytroidalnych, czy jądrowe receptory hormonów.
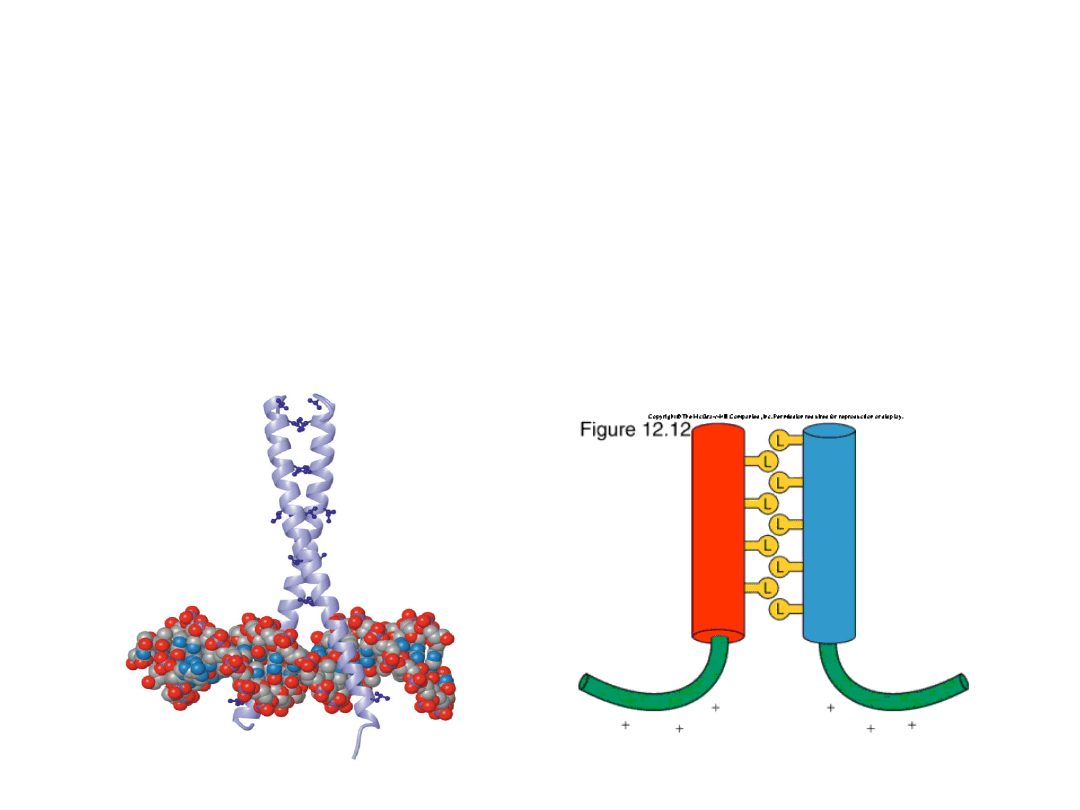
Motyw suwaka leucynowego reprezentuje rozciągniętą -
helisę, w której dokładnie co 7. pozycji występuje reszta leucyny.
Motyw
ten
odpowiada
za
dimeryzację
czynników
transkrypcyjnych
tworzących
homo-
lub
heterodimery.
Przedłużeniem „suwaka leucynowgo” jest zasadowa domena
wiążąca DNA, również o strukturze -helisy. Razem tworzą one
„zasadowy suwak leucynowy” (bZIP).
Te czynniki odgrywają bardzo ważną rolę w procesach wzrostu i
różnicowania komórek (protoonkogeny).
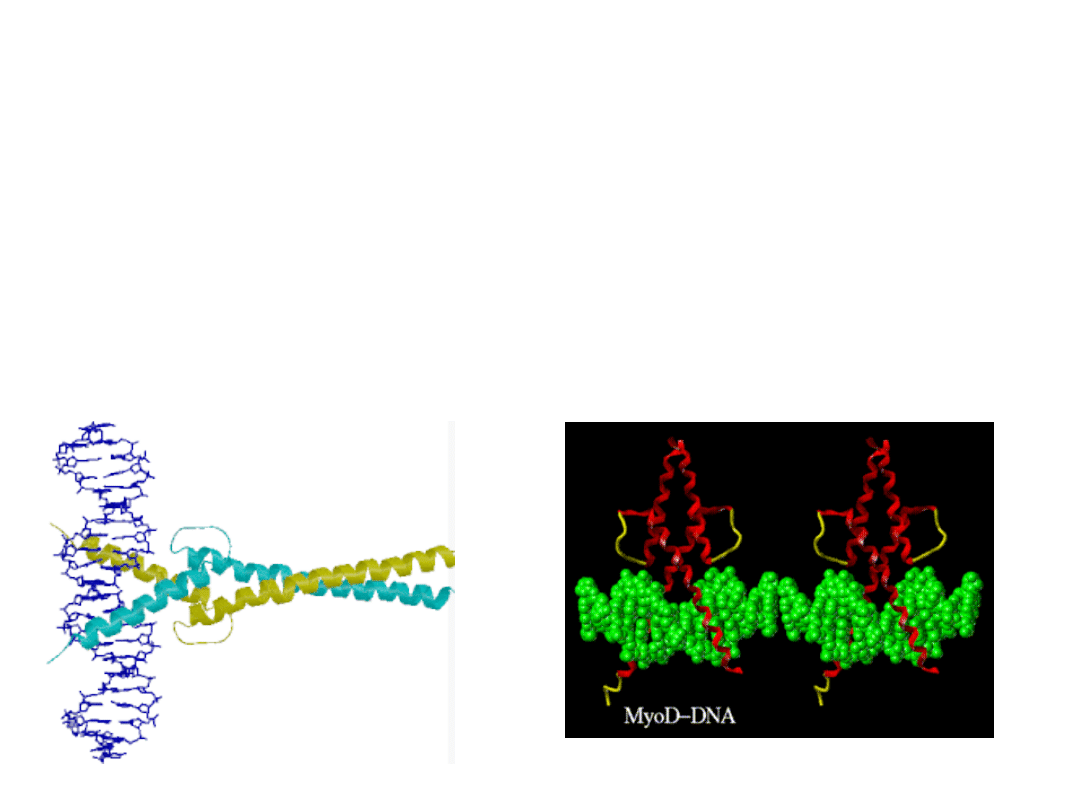
Motyw helisa-pętla-helisa (HLH) występuje np. w
takich czynnikach transkrypcyjnych jak MyoD czy
miogenina. Strukturą przypomina zasadowy suwak
leucynowy i w połączeniu z dodatkową pętlą tworzy
zasadowy motyw HLH.
Tak więc, różnica pomiędzy zasadowym suwakiem
leucynowym a zasadowym motywem HLH polega na
występowaniu dodatkowej pętli oddzielającej domenę
dimeryzacyjną od zasadowej domeny wiążącej DNA w
przypadku tej drugiej struktury.
Kombinatoryczna regulacja ekspresji genów
w komórkach eukariotycznych
Sekwencji regulatorowych, które odpowiedzialne są za poziom
ekspresji określonego genu/genów może być wiele, co
powoduje, że są one rozrzucone wzdłuż bardzo długich
odcinków DNA (nawet kilkadziesiąt tysięcy nukleotydów).
Należy pamiętać, że do każdej z tych sekwencji wiążę się
białkowy czynnik transkrypcyjny bezpośrednio odpowiedzialny
za transkrypcję.

Kombinatoryczna regulacja ekspresji genów
w komórkach eukariotycznych
Czynniki transkrypcyjne nie działają indywidualnie, ale w
zespole z innymi i dopiero współdziałanie tych wszystkich
białek regulatorowych decyduje ostatecznie o poziomie
transkrypcji danego genu.
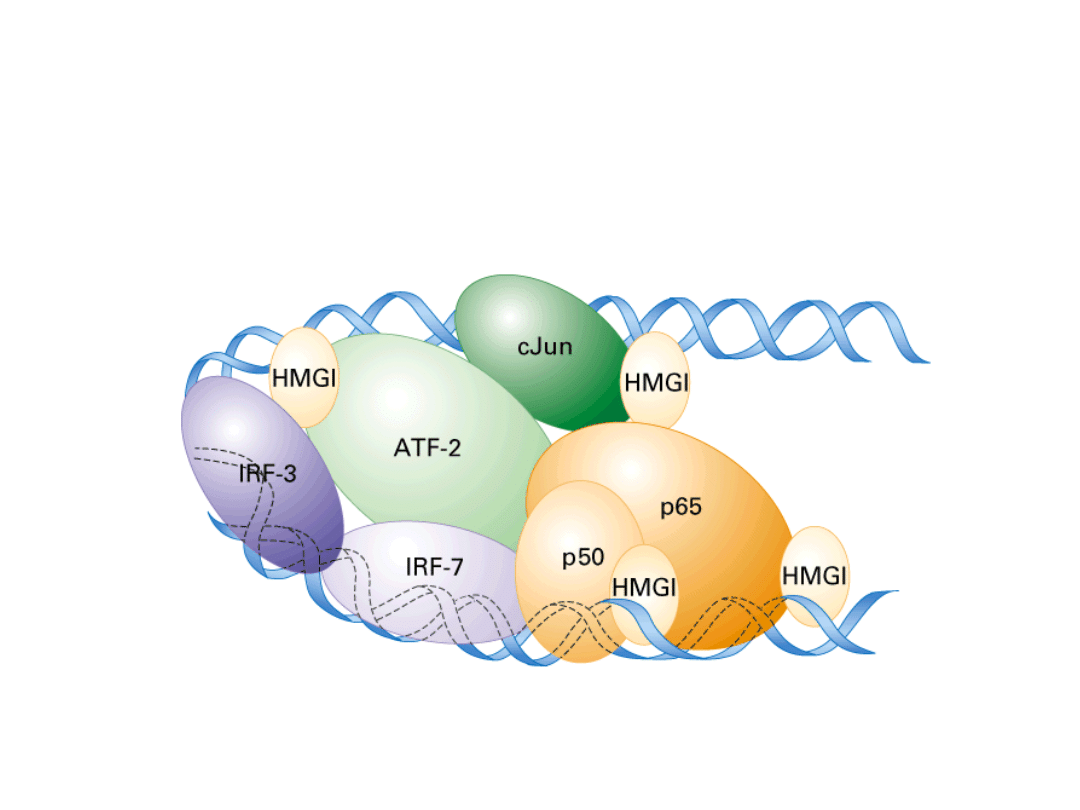
Czynniki transkrypcyjne nie tylko oddziałują z DNA, ale równie ważne
są występujące pomiędzy nimi oddziaływania typu białko – białko.
Przyłączenie jednych białek powoduje przyłączanie innych, co
prowadzi
do
powstawania
dużych
wieloskładnikowych
kompleksów regulacyjnych.
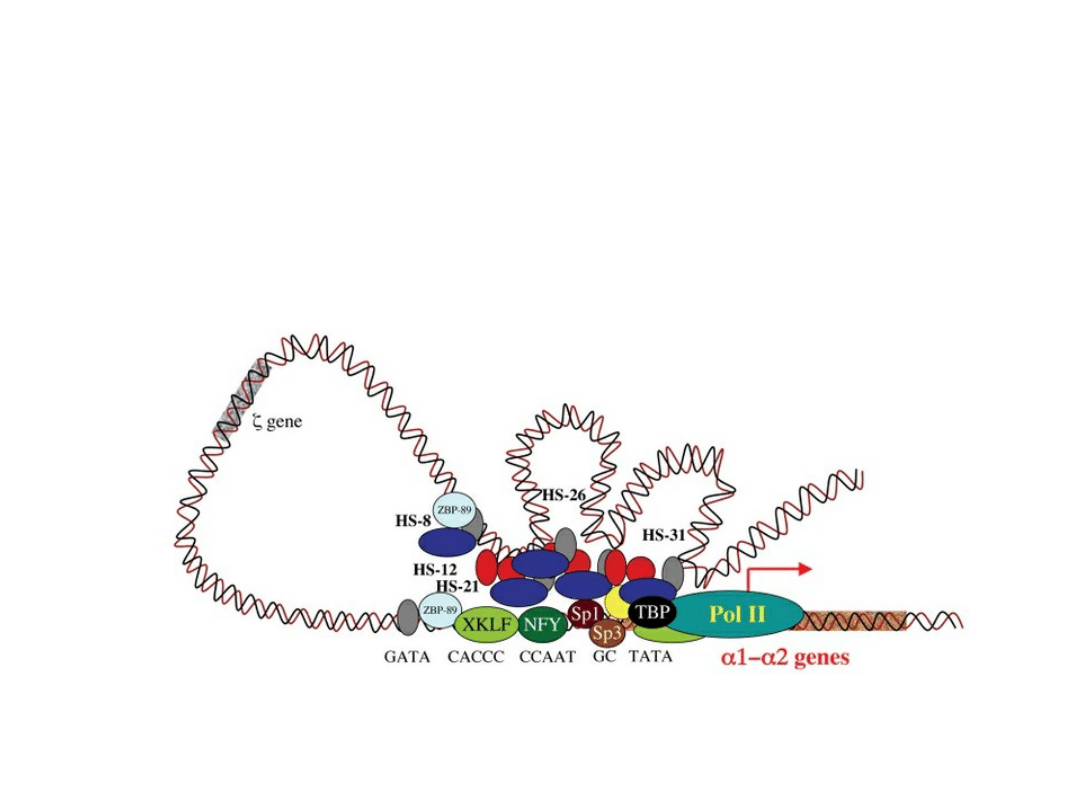
Termin
kontrola kombinatoryczna
odnosi się do
sposobu w jaki grupa białek, działając wspólnie,
determinuje ekspresję określonego genu.
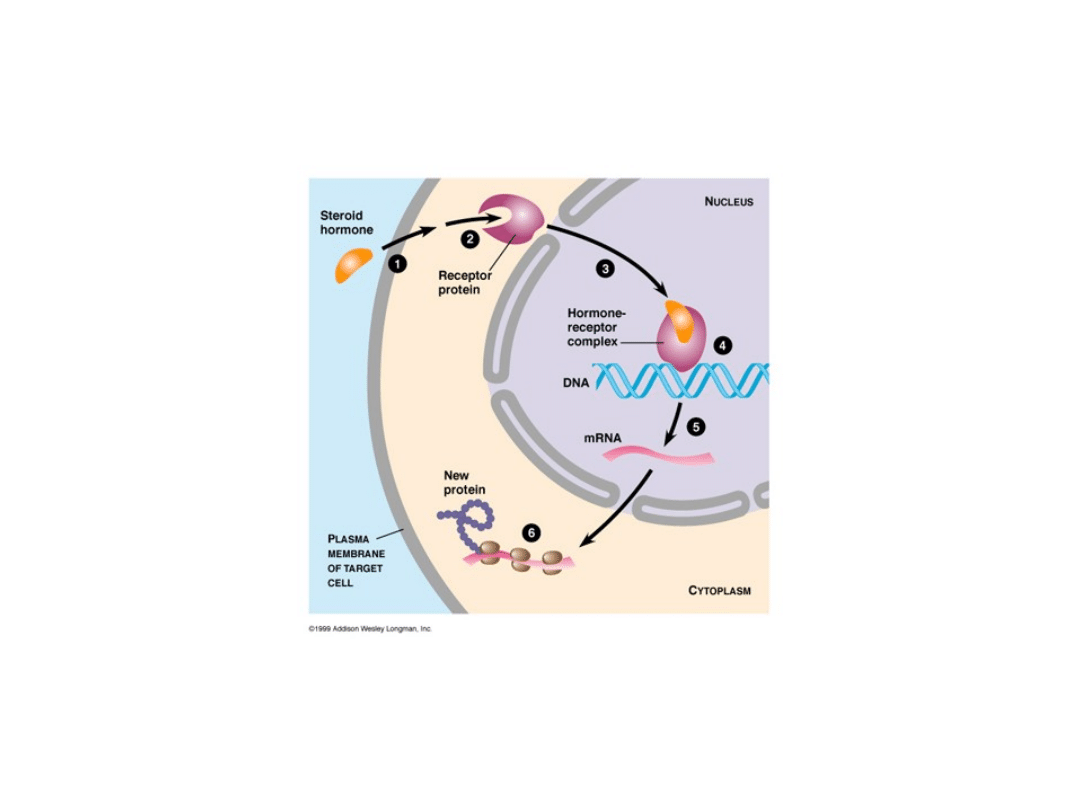
Czynniki transkrypcyjne regulowane przez hormony
sterydowe
Hormony sterydowe, hormony tyroidowe i retinoidy są ligandami dla
cytozolowych
czynników
transkrypcyjnych
noszących
nazwę
jądrowych receptorów hormonów (50).
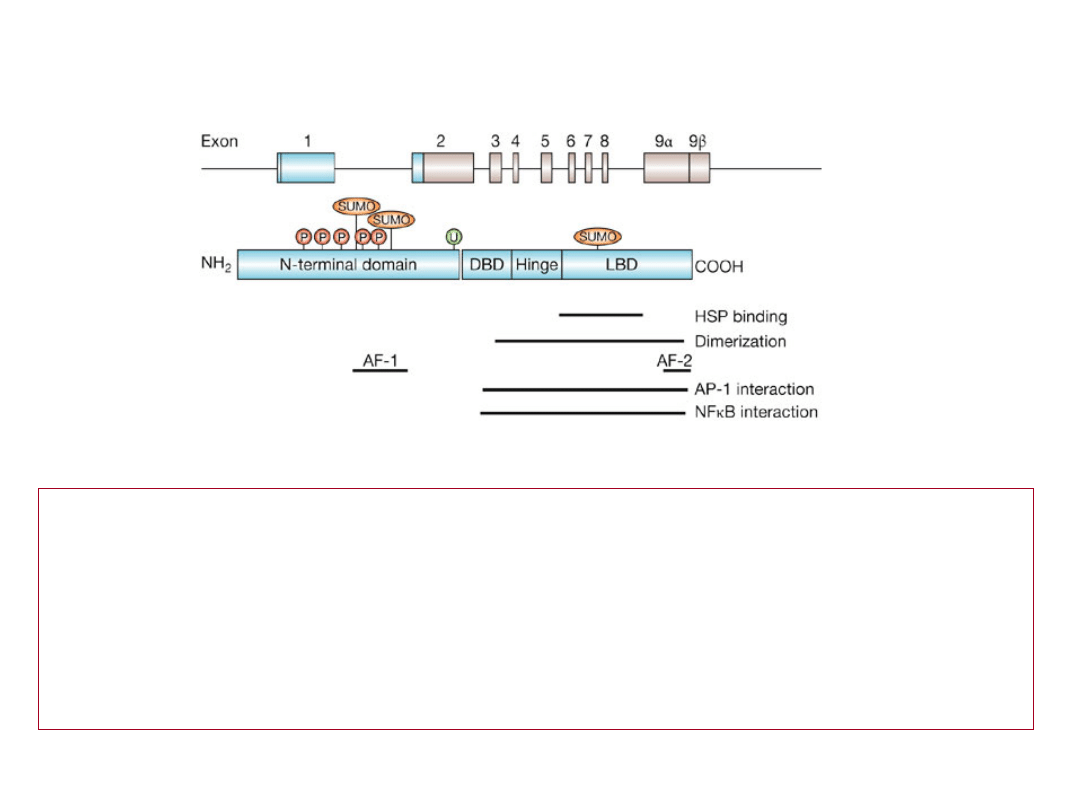
Budowa receptorów jądrowych
1. Domeny transaktywacyjne (AF)
2. Domena wiążąca DNA (
DBD
)
3. Domena wiążąca hormon (domena wiążąca ligand) (
LBD
)
4. Sygnały importu jądrowego
Jądrowe receptory hormonów
charakteryzują się obecnością dwóch
wysoce konserwatywnych domen: domeny wiążącej DNA i
domeny wiążącej ligand (hormon). Białka te wiążą się do odcinków
DNA noszących nazwę elementów odpowiedzi na sterydy (SRE,
ang. steroid response elements).
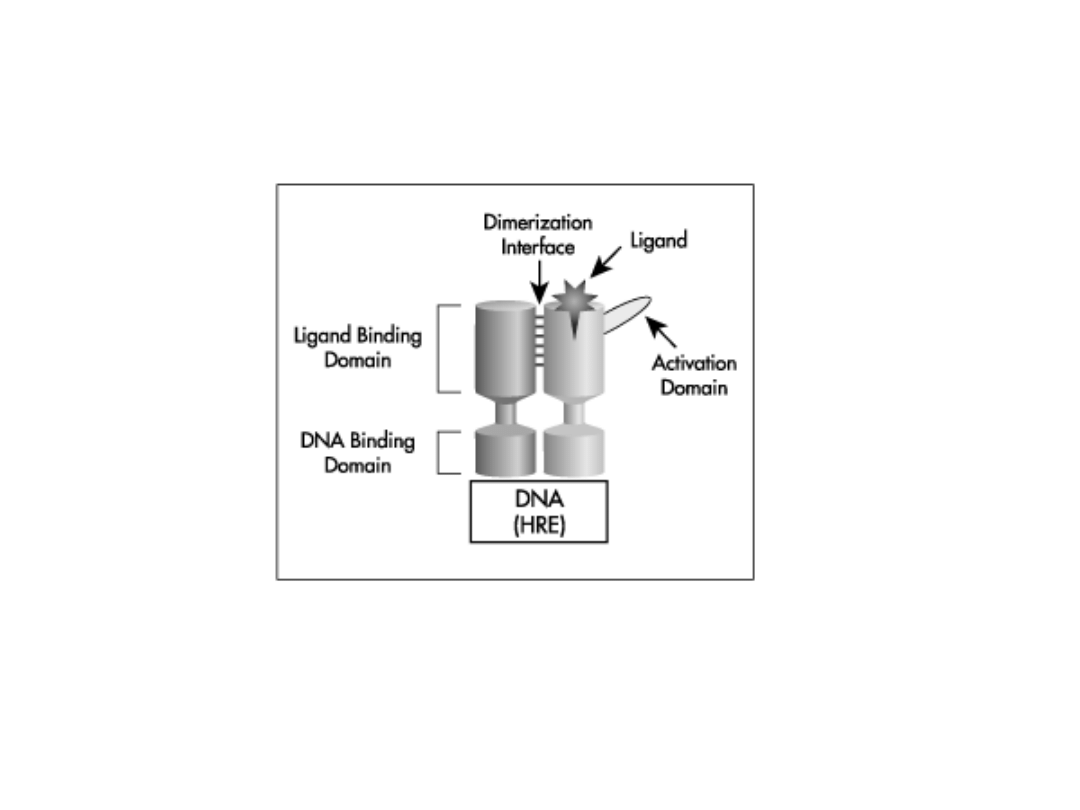
Domena wiążąca ligand jest również odpowiedzialna za
dimeryzację receptorów.
Przyłączenie hormonu do domeny wiążącej ligand powoduje, że cała
cząsteczka jądrowego receptora hormonów podlega znacznym
zmianom konformacyjnym.
Zmiany konformacyjne jakim podlegają
jądrowe
receptory hormonów
powodują przyłączanie się do
nich cząsteczek
białek regulatorowych
pełniących
funkcje
ko-aktywatorów
i
zwiększenie
transkrypcji.
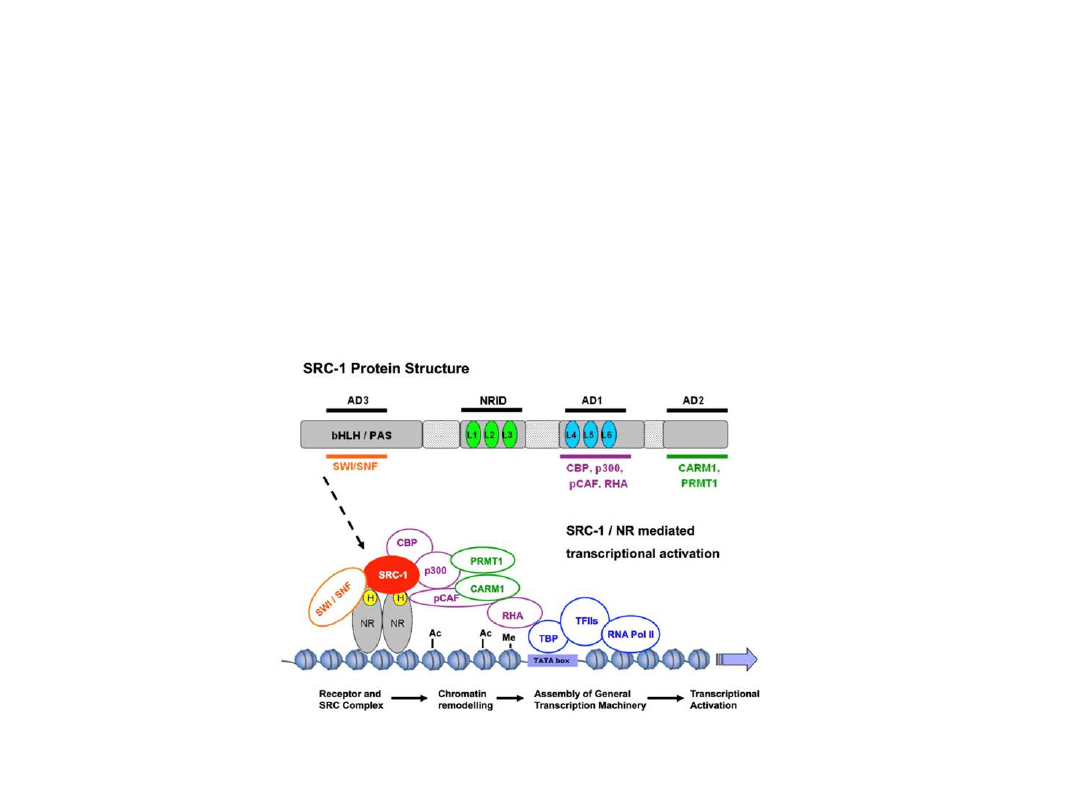
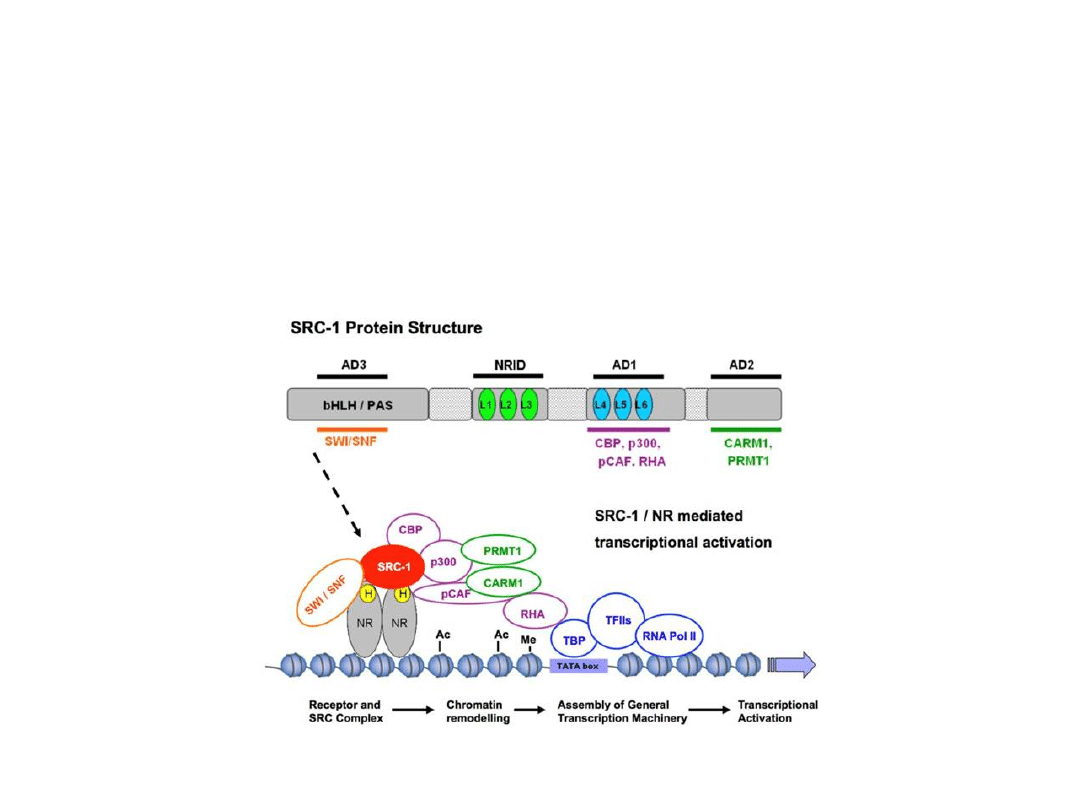
Przykłady
koaktywatorów
: białko SRC-1 (koaktywator receptora
sterydowego, ang. steroid receptor coactivator-1), białko GRIP-1
(białko oddziałujące z receptorem glukokortykoidów, ang.
glucocorticoid receptor interacting protein-1), białko NcoA-1
(koaktywator jądrowego receptora hormonu, ang. nuclear
hormone receptor coactivator-1).
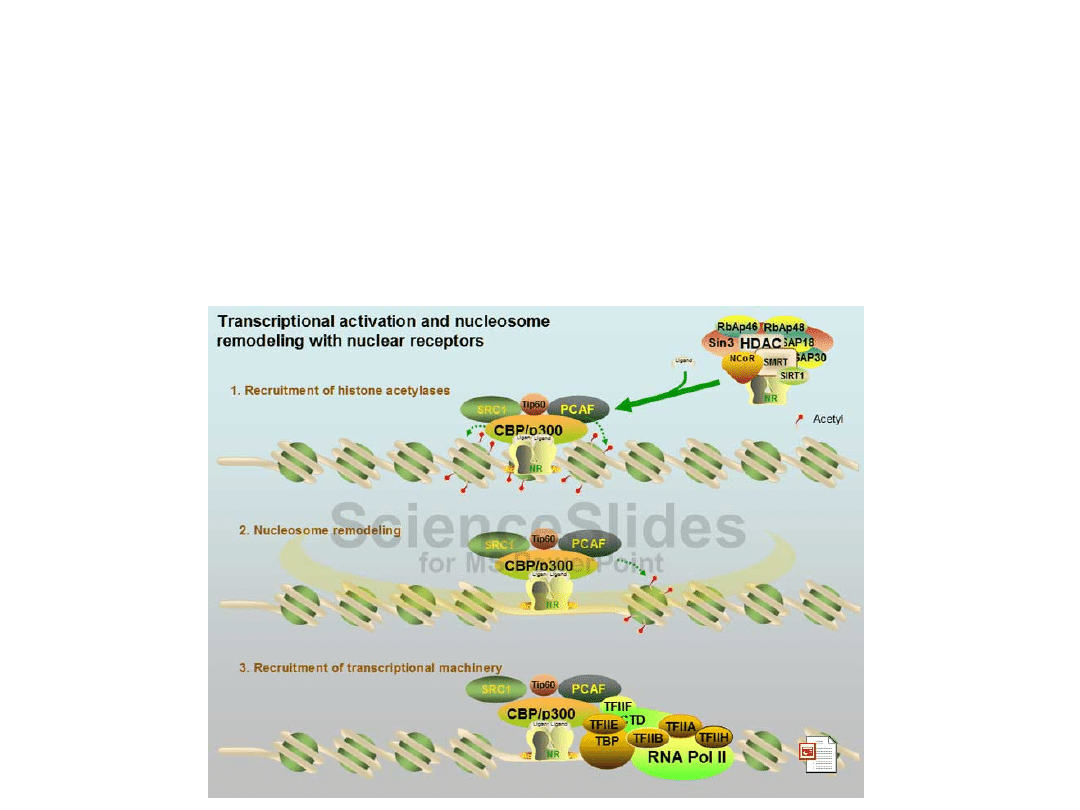
Koaktywatory uczestniczą w kowalencyjnych modyfikacjach N-końców
białek histonowych, przyczyniając się do ich acetylacji. Natomiast, w
reakcjach przenoszenia grup acetylowych z acetylo-CoA na reszty
lizynowe bezpośredni udział biorą acetylotransferazy histonów (HAT,
ang. histone acetylotransferases).
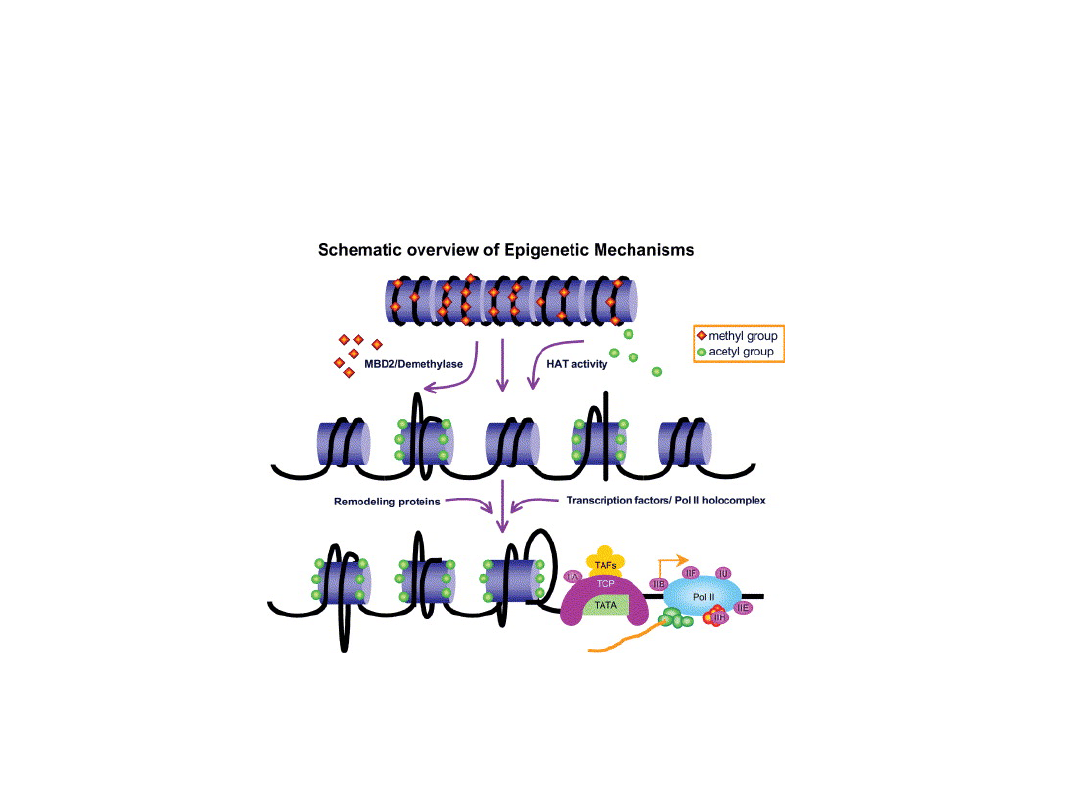
Acetylacja histonów powoduje rozluźnienie struktury chromatyny w
wyniku neutralizacji ładunków ujemnych lizyn.
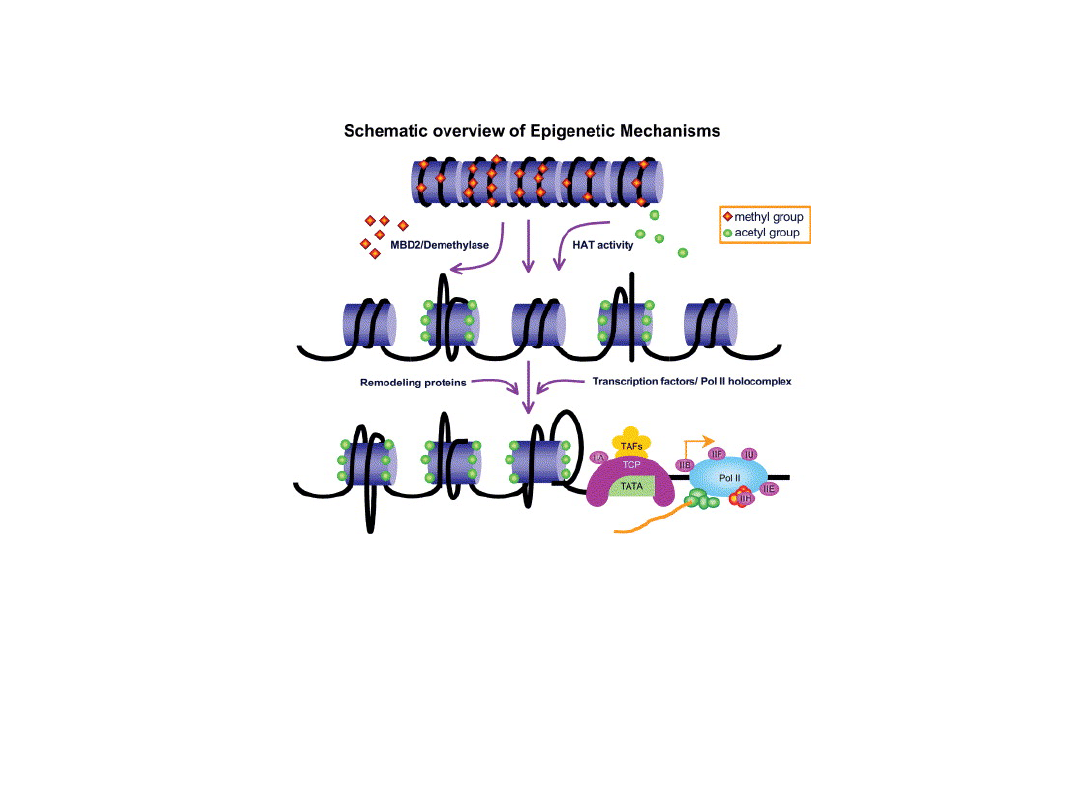
Acetyowane reszty histonów (acetylolizyny) oddziałują z wieloma
białkami biorącymi udział w regulacji transkrypcji.
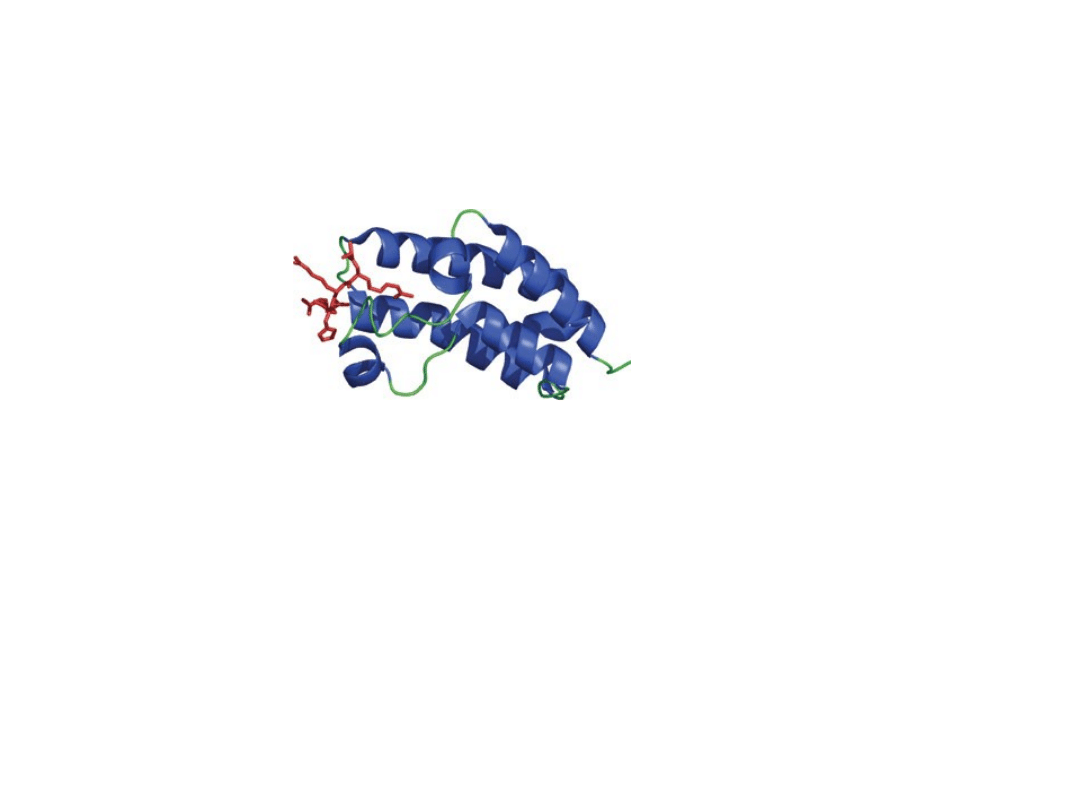
A dokładniej, acetylowane reszty lizyny oddziałują z domenami
wiążącymi acetylolizynę, zwanymi bromodomenami, które są
obecne w wielu białkach regulujących transkrypcję.
Białka zawierające bromodomenę wchodzą w skład dwóch
kompleksów białkowych uczestniczących w transkrypcji.
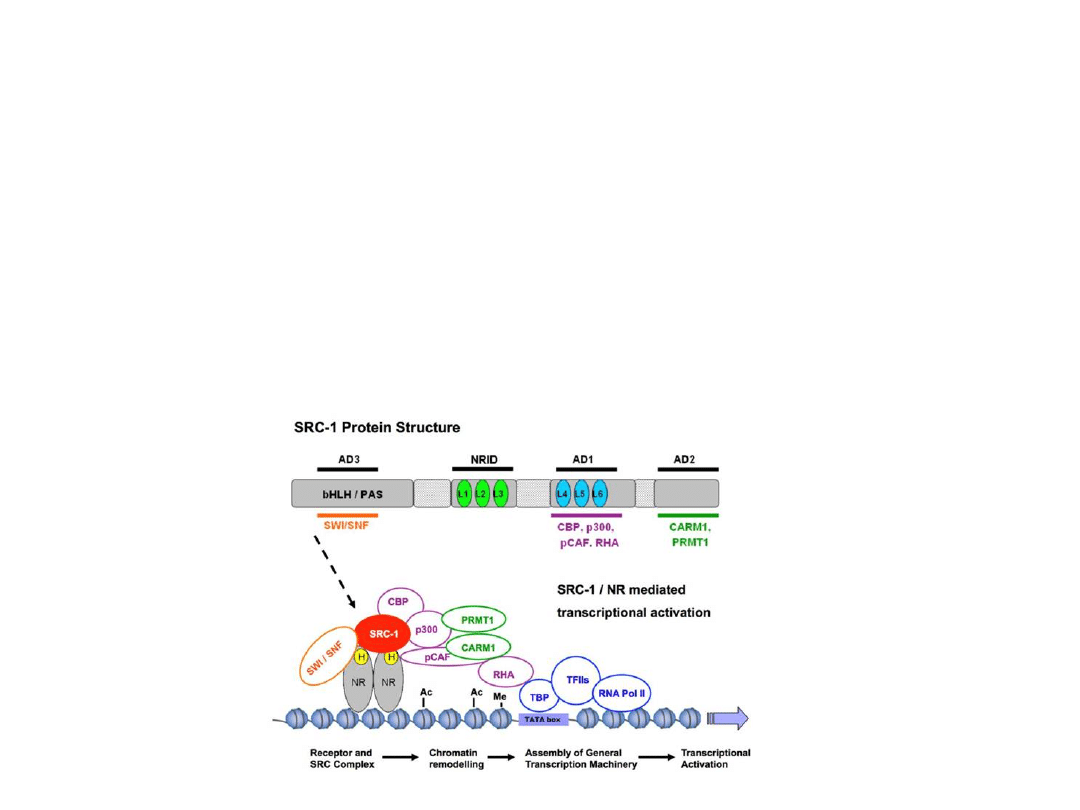
Kompleksy białkowe zawierające białka TAF (
T
ATA-box binding
protein
a
ssociated
f
actors), które wiążą się z białkiem TBP. Przykładem
takiego białka jest białko TAFII 250.
Kompleksy białkowe zwane fabrykami remodelowania
chromatyny. Kompleksy remodelujące, wykorzystujące energię z
hydrolizy ATP, przesuwają nukleosomy wzdłuż cząsteczki DNA i
powodują różne zmiany w konformacji chromatyny.
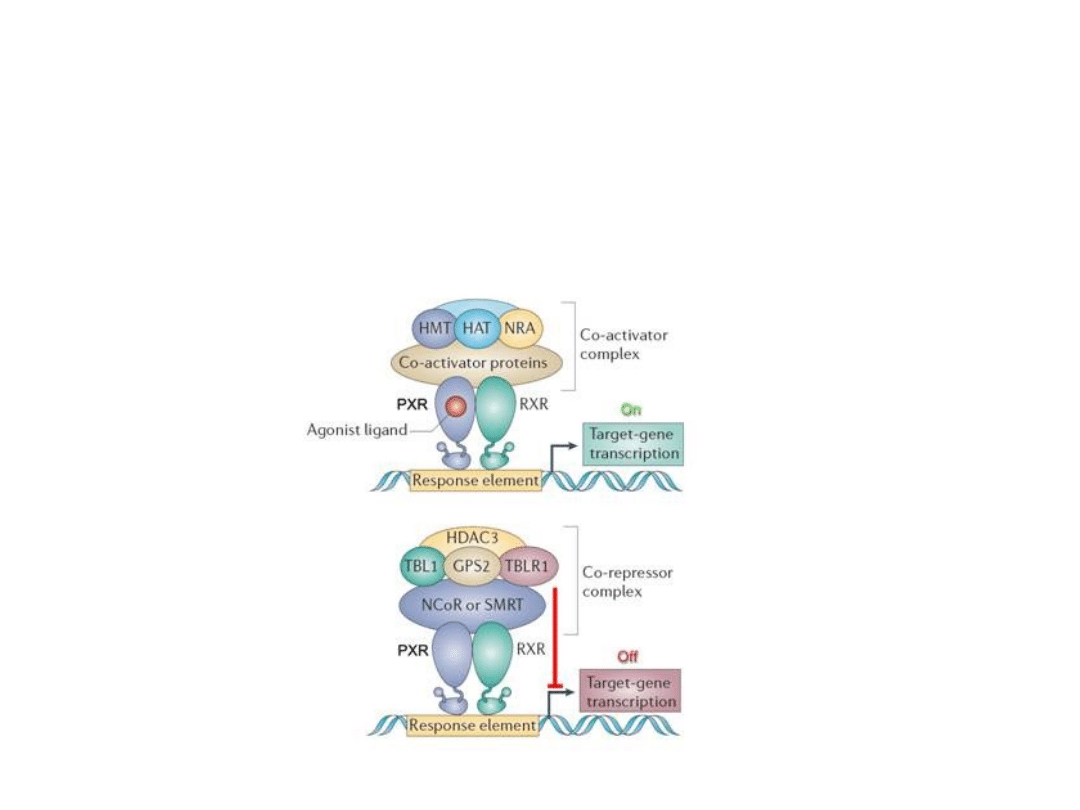
W odróżnieniu od receptorów hormonów sterydowych, receptory
hormonów tyroidowych i receptora kwasu retinojowego nie
związane z ligandem, wiążą białka korepresorowe, które hamują
transkrypcję. Dopiero związanie liganda powoduje odłączenie korepresora
i umożliwia przyłączenie koaktywatora.
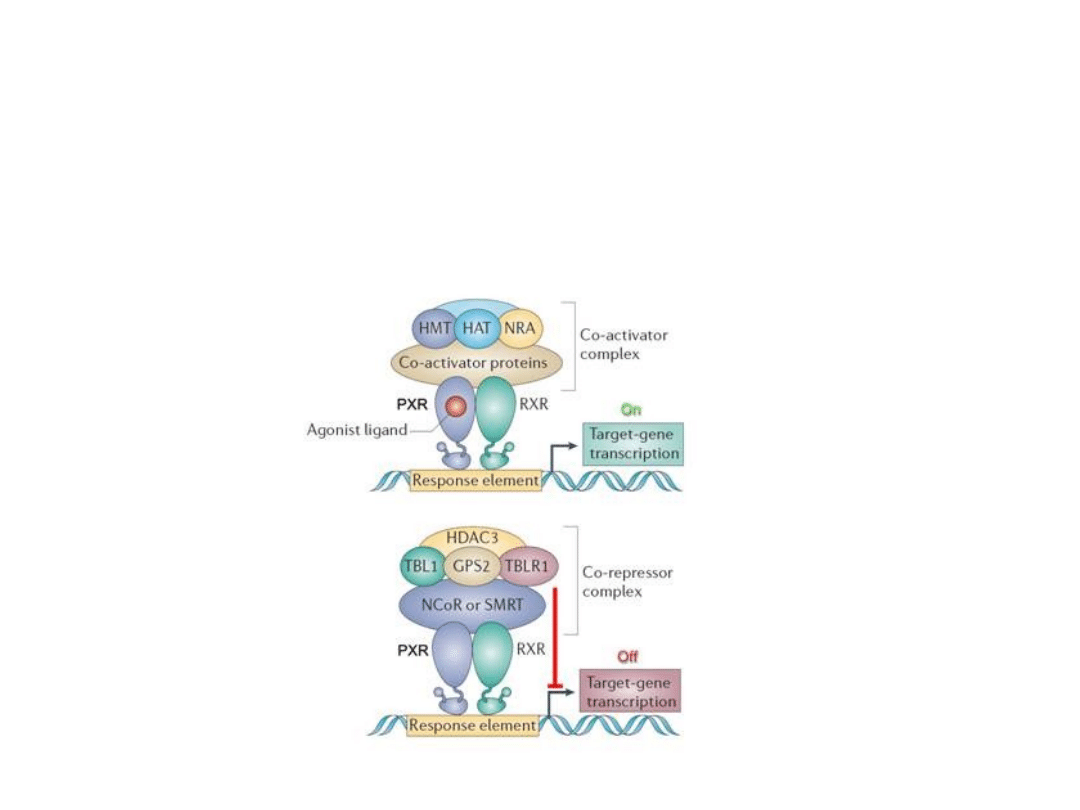
Przykładem białek korepresorowych są: białko SMRT
(cząsteczka wyciszająca receptory kwasu retinojowego i
hormonu tyroidowego, ang. silencing mediator for retinoid and
thyroid hormone receptor), białko N-Cor (korepresor jądrowego
receptora hormonalnego, ang. nuclear hormone receptor
corepressor).

Rola acetylacji histonów w aktywacji
transkrypcji
1. Redukcja powinowactwa histonów do DNA –
rozluźnienie struktury,
2. Rozpoczęcie zmian w strukturze chromatyny poprzez
ułatwienie
związania
białkowych
kompleksów
remodulujących chromatynę,
3. Rekrutacja białek tworzących kompleksy biorące udział w
transkrypcji.
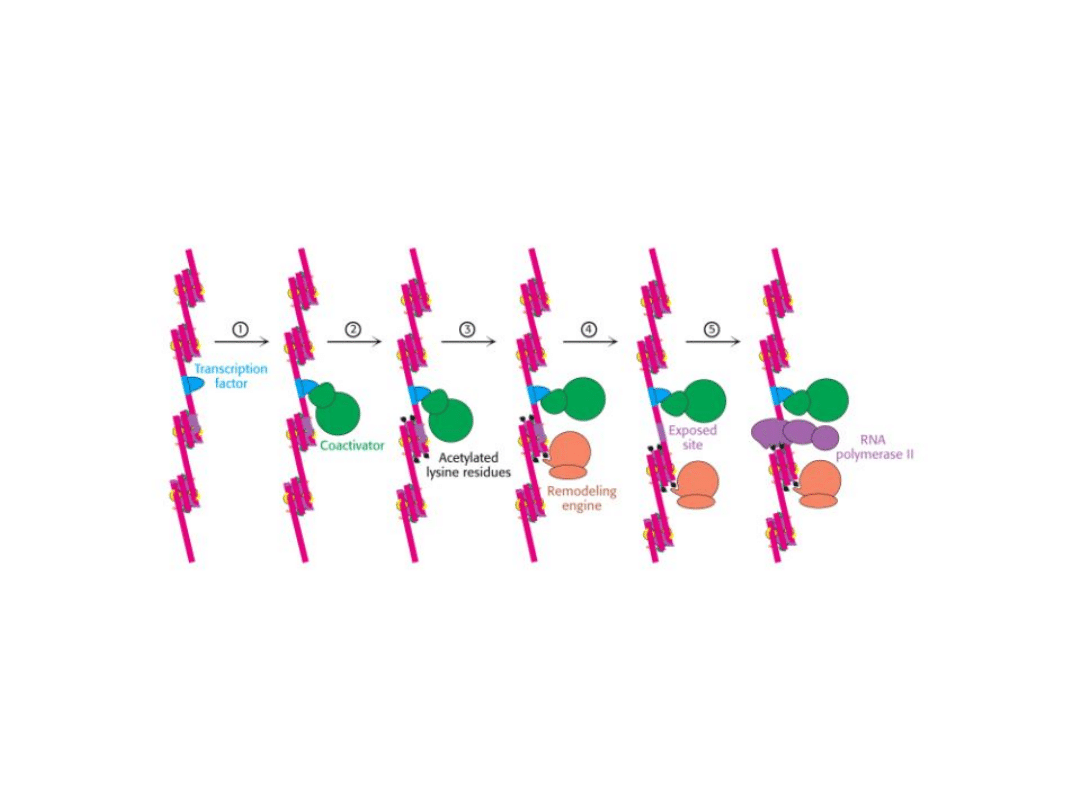
Remodelowanie struktury chromatyny

Deacetylacja
histonów,
przy
udziale
specyficznych
deacetylaz histonowych (HDAC)
prowadzi
do
zahamowania
ekspresji
aktywnych genów.

Czynniki transkrypcyjne regulowane poprzez fosforylacje
Fosforylacje czynników transkrypcyjnych mogą prowadzić do:
1. Ich translokacji z cytoplazmy do jądra komórkowego
2. Zmianie powinowactwa do sekwencji DNA
3. Ich aktywacji i funkcjonowania jako aktywatorów transkrypcji
4. Nabycia przez nie zdolności do tworzenia kompleksów
wielobiałkowych
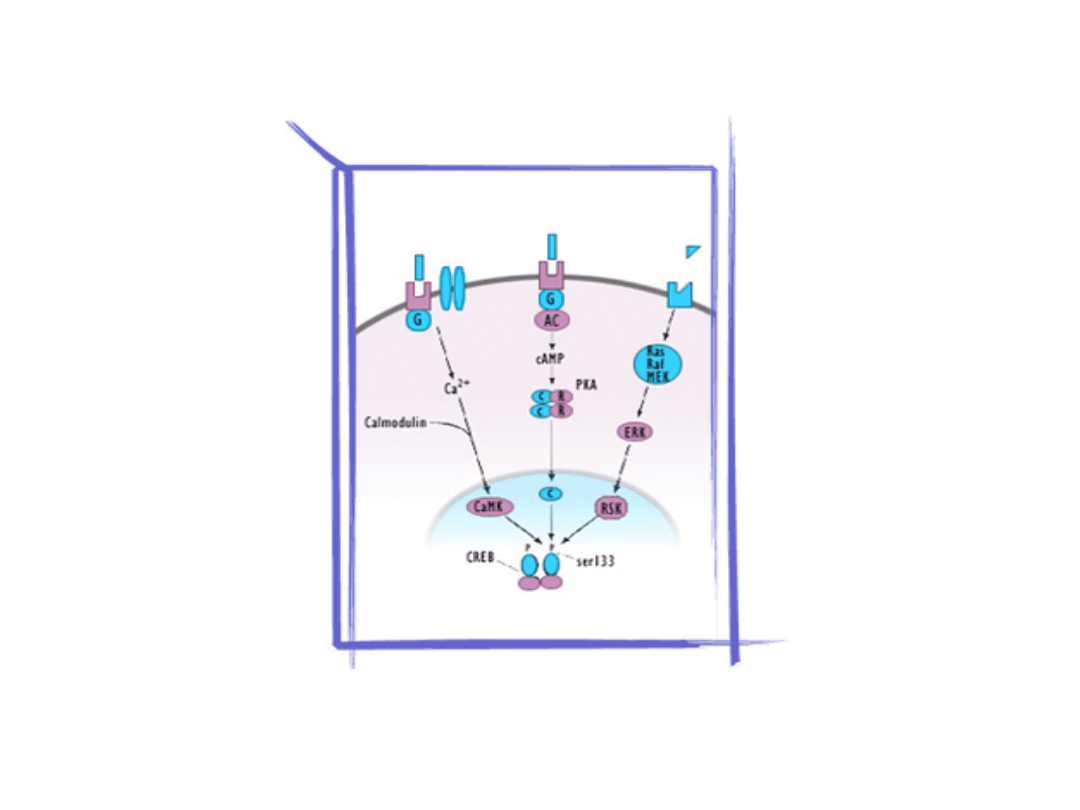
Czynniki transkrypcyjne regulowane poprzez
fosforylacje
Przykładem takiego czynnika transkrypcyjnego jest białko CREB
(cyclic AMP-response element binding protein) aktywowane przez
kinazę białek A.
Ligand
Receptor
Białko G
Cyklaza adenylanowa
cAMP
Kinaza białek A (PKA)
Podjednostka
katalityczna
Kinazy A
Białko CREB
Fosforylowane białko CREB
(białko wiążące się z elementem
odpowiedzi na cAMP) wiąże się z elementem odpowiedzi na
cAMP w genach docelowych.
Fosforylacja białka CREB powoduje również, że jest ono wiązane
przez białko CBP (CREB-binding protein), który z kolei posiada
aktywność acetylotransferazy histonowej.
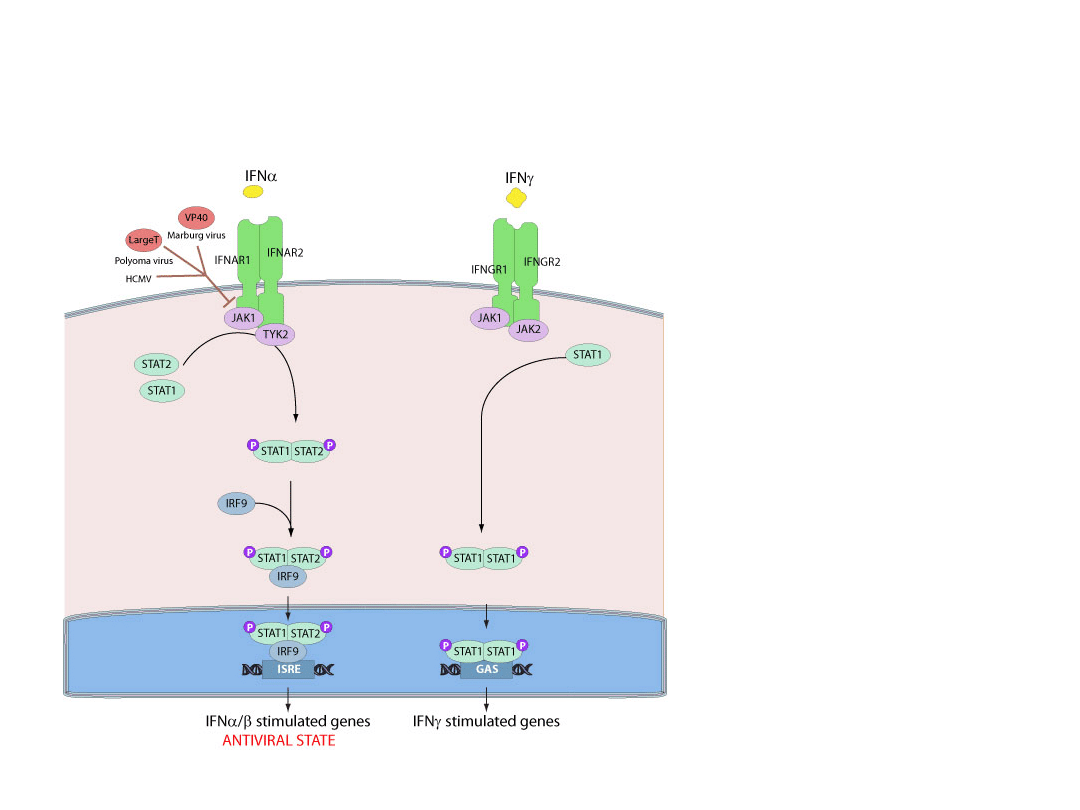
Czynniki transkrypcyjne regulowane poprzez fosforylacje
Interferon
Receptor dla interferonu
Kinazy Jak1 i Jak2
Czynniki transkrypcyjne
STAT1-α i STAT1-β
(signal transducers and activators
of transcription)

Regulacja ekspresji genów na poziomie
translacji
Przykładem takiej regulacji są geny zaangażowane w metabolizm
żelaza, kodujące takie białka jak receptor
transferynowy i
ferrytynę.
Ekspresja obu białek zależy od
poziomu żelaza, przy czym zmiany
w jego ilości nie wpływają na
transkrypcję ich genów.
Regulacja ekspresji ferrytyny
mRNA dla ferrytyny zawiera w końcu 5’ cząsteczki sekwencję o
nazwie element odpowiedzi na żelazo (IRE, ang. iron-response
element), który tworzy strukturę spinki do włosów.

Sekwencja IRE
wiąże specyficzne białko o nazwie IRE-BP,
tzn.
białko wiążące się do IRE
(ang. IRE-binding protein).
Przyłączenie tego białka hamuje translację ferrytyny.
Białko IRE-BP, przy wzroście poziomu żelaza w organizmie,
wiąże centra żelazo-siarkowe (4Fe-4S), co powoduje jego
odłączenie od mRNA dla ferrytyny, umożliwiając tym
samym translację.
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
- Slide 44
- Slide 45
- Slide 46
- Slide 47
- Slide 48
- Slide 49
- Slide 50
- Slide 51
- Slide 52
- Slide 53
- Slide 54
- Slide 55
- Slide 56
- Slide 57
- Slide 58
- Slide 59
- Slide 60
- Slide 61
- Slide 62
- Slide 63
- Slide 64
- Slide 65
- Slide 66
- Slide 67
- Slide 68
- Slide 69
- Slide 70
- Slide 71
- Slide 72
- Slide 73
- Slide 74
- Slide 75
- Slide 76
- Slide 77
- Slide 78
- Slide 79
- Slide 80
- Slide 81
Wyszukiwarka
Podobne podstrony:
Regulacja ekspresji genow poprawione
Regulacja ekspresji genów u eucaryota
3. Przyklady regulacji ekspresji genow u Eukaryota-ok, Biologia II, Biologia molekularna
Regulacja ekspresji genów, fizjoterapia, biologia medyczna
12 Regulacja ekspresji genow
1 Regulacja ekspresji genów u prokariota
Regulacja ekspresji genów - materiały teoretyczne
Epigenetyczne mechanizmy regulacji ekspresji genow?
W3 Regulacja ekspresji genów
wd 4 glukoza, lipidy metabolizm, regulatory ekspresji genów
Regulacja ekspresji genów egzamin
07) Regulacja ekspresji genów (wyklad 7)
Regulacja ekspresji genów
Regulacja ekspresji genow
Gradient ekspresji genów w regulacji morfogenezy u ssaków, Medycyna ŚUM, Rok 1, Biologia medyczna, T
4 Regulacja ekspresji
więcej podobnych podstron