
Replikacja DNA

Istota procesu:
Jest to proces umożliwiający przekazanie
informacji genetycznej z komórek rodzicielskich
kolejnym pokoleniom.
Proces ten polega na syntezie dwóch,
kompletnych, dwuniciowych helis z jednej
wyjściowej, przy czym obydwie helisy mają
sekwencję nukleotydową identyczną z helisą
wyjściową.
DNA działa jako matryca dla swojej własnej
replikacji. Każdy łańcuch DNA dyktuje
sekwencję nukleotydów łańcucha
komplementarnego.
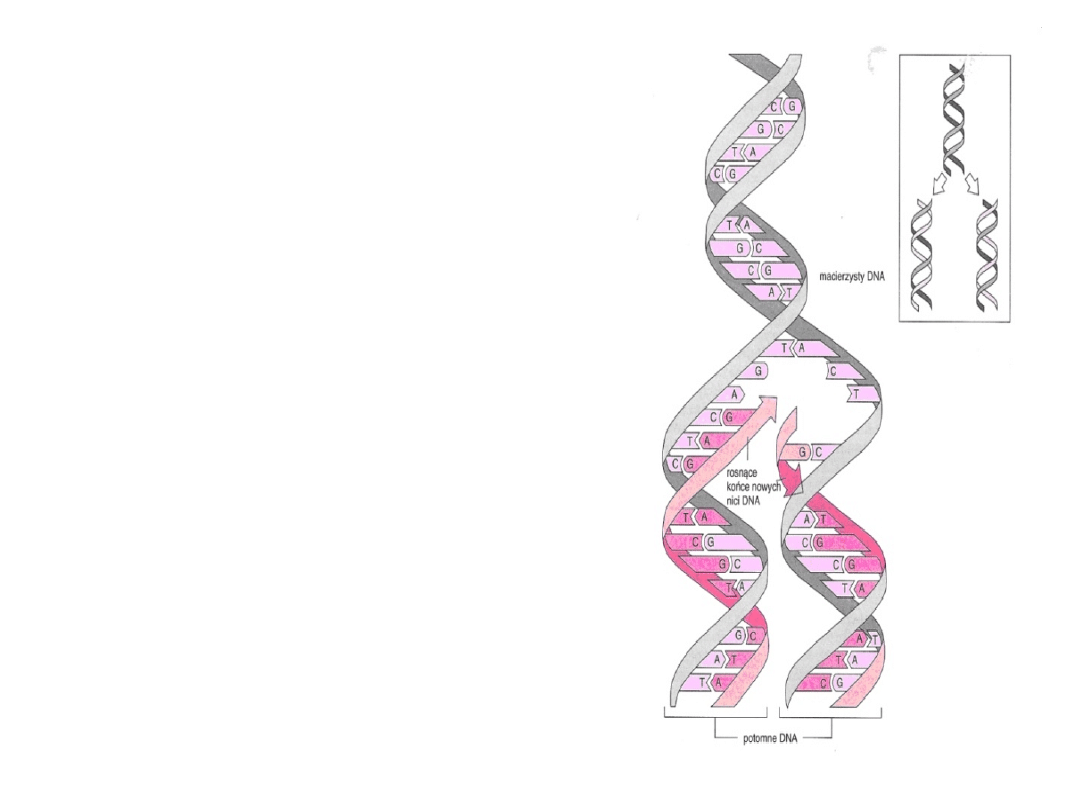
Replikacja ma charakter
semikonserwatywny. W
czasie podziału każda z
dwóch nici pełni funkcje
matrycy. Nukleotydy są
dołączane zgodnie z regułą
komplementarności zasad.
Powstaje hybryda złożona z
jednej nici starej i jednej
nowej. Każda komórka
zawiera nić starą
(macierzystą) i nową
(potomną), powstałą w
trakcie replikacji DNA
zachodzącej w fazie S
podziału komórkowego.

Substraty replikacji:
• matryca DNA
• trifosforany
deoksyrybonukleoty
dów (dNTP)
• ATP/CTP/GTP-
energia dla helikaz

Enzymy replikacji:
• Helikaza
- rozwijają nici DNA
• Topoizomeraza-
stabilizują odcinki
jednoniciowe
• DNA prymaza
- syntetyzuje startery RNA
• Białka wiążące ssDNA-
zapobiegają zwijaniu
w podwójną nić
• DNA ligaza-
łączy nowosyntetyzowane odcinki
DNA
• DNA polimerazy
• Egzonukleaza-
usuwa startery RNA z nici

Polimerazy replikacji:
• W przypadku E. coli poznano 3 polimerazy.
• Polimeraza I - zawiera jedną podjednostkę,
wykazuje aktywność egzonukleazy zarówno w
kierunku 3’-> 5’ jak i 5’->3’. Jej główną funkcją jest
synteza nici opóźnionej oraz wypełnianie po
starterach RNA
• Polimeraza II- również zawiera jedną
podjednostkę, wykazuje aktywność egzonukleazy w
kierunku 3’->5’, lecz nie w odwrotnym. Ma za
zadanie sprawdzać i naprawiać powstające nici.
• Polimeraza III- składa się na nia ok.. 10
podjednostek, aktywność egzonukleazy ujawnia
jedynie w kierunku 3’->5’, syntetyzuje nić wiodącą.

Polimerazy Eucariota:
• U Eucaryota odkryto 5 polimeraz.
• Polimeraza α-
4 podjednostki, nie ma aktywności
egzonukleazy, synteza nici opóźnionej, wypełnianie po
starterach RNA
• Polimeraza β-
1 podjednostka, również nie wykazuje
aktywności egzonukleazy, pełni funkcje naprawcze
• Polimeraza γ-
2 podjednostki, aktywność
egzonukleazy 3’->5’, synteza mitochondrialnego DNA
• Polimeraza δ-
2 lub 3 podjednostki, aktyowność
egzonukleazy 3’->5’, synteza nici wiodącej
• Polimeraza ε-
1 podjednostka, aktywność
egzonukleazy 3’->5’, pełni funkcje sprawdzające i
naprawcze.
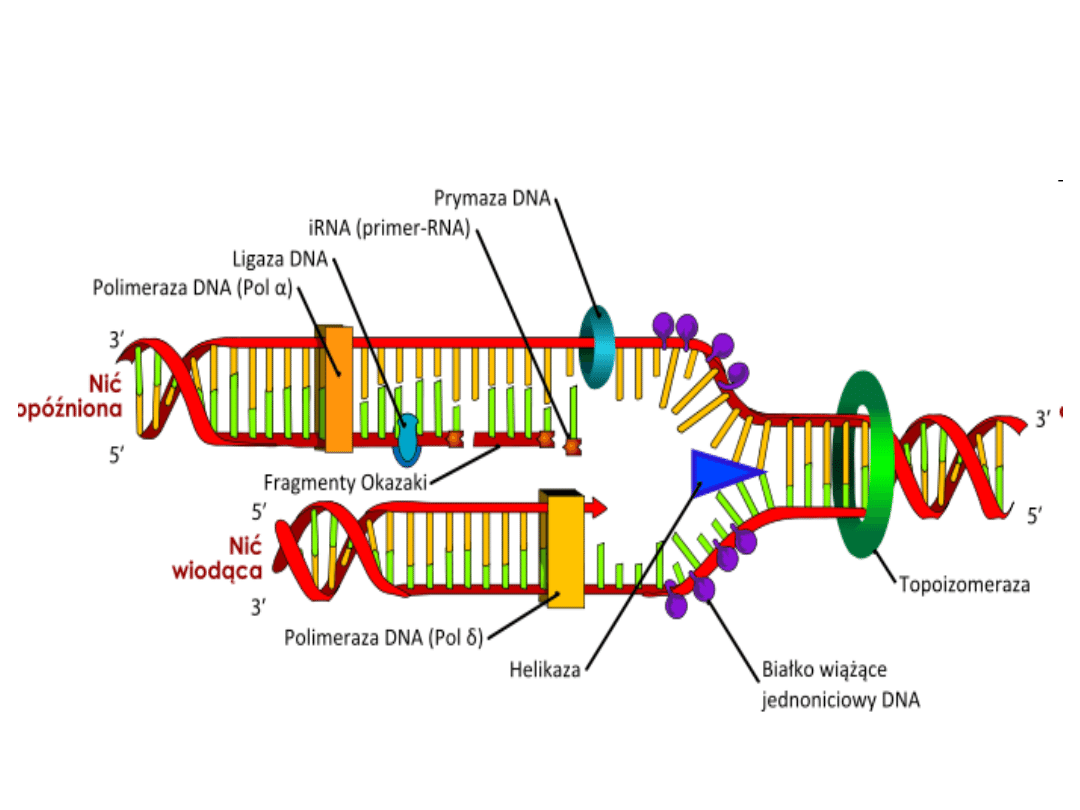
Miejsca działania
enzymów:

Inicjacja replikacji:
• Inicjacja, czyli początek replikacji zachodzi
w ściśle określonych miejscach na nici DNA.
Miejsca te nazywają się "origin" (w skrócie
ori). Zawierają one specyficzne sekwencje,
służące im do:
• wiązania białek inicjatorowych,
• rozdzielenia nici i rozpoczęcia procesu
replikacji,
• przyłączania białek wspomagających proces
replikacji.

Miejsce inicjacji jest bardzo ważne,
ponieważ tylko tu może odbywać się
kontrola replikacji. Raz rozpoczęty proces
musi przebiegać aż do zakończenia
syntezy całego replikonu.
U bakterii replikon obejmuje cały genofor,
znaczy to, że pojedyncze miejsce inicjacji
kontroluje replikację całego genomu.
Organizmy eukariotyczne natomiast, maja
wiele replikonów w każdym chromosomie.
Dzięki tej różnicy szybkość replikacji
genomu eukariota wielokrotnie przewyższa
szybkość replikacji genomu prokariota.

• Rozdzielające się w miejscu syntezy
łańcuchy matrycowego DNA tworzą
tzw. "widełki replikacyjne". O
rozdzieleniu heliksu DNA decydują
białka enzymatyczne zwane
helikazami. Wykorzystują one
energię hydrolizy ATP do zmiany
kształtu swojej cząsteczki (podobnie
jak białka mięśniowe), co umożliwia
im wykonanie pracy mechanicznej.
• Helikazy przesuwają się wzdłuż
dwuniciowego DNA, rozdzielając nici i
poszerzając widełki replikacyjne.

• Najważniejszymi enzymami
odpowiedzialnymi za replikację DNA są
polimerazy DNA. Nie mają one jednak
zdolności do samodzielnego rozpoczęcia
syntezy łańcuchów DNA, mogą jedynie je
wydłużać.
• Synteza DNA zaczyna sie od wbudowywania
starterowych odcinków RNA, które
zakończone są wolną grupą -OH, na końcu 3'.
• Za wbudowywanie tych odcinków
odpowiedzialne są wyspecjalizowane enzymy
zwane prymazami.

Elongacja replikacji:
• Proces elongacji, podobnie jak proces
inicjacji, wymaga udziału wielu białek
enzymatycznych. Oprócz polimeraz DNA,
głównych enzymów replikacji, potrzebnych
jest wiele enzymów odpowiedzialnych za
katalizowanie reakcji analogicznych do
przeprowadzanych podczas inicjacji
replikacji (rozplątywanie i stabilizacja nici,
wbudowywanie starterów). Wszystkie te
białka tworzą kompleks zwany replisomem.
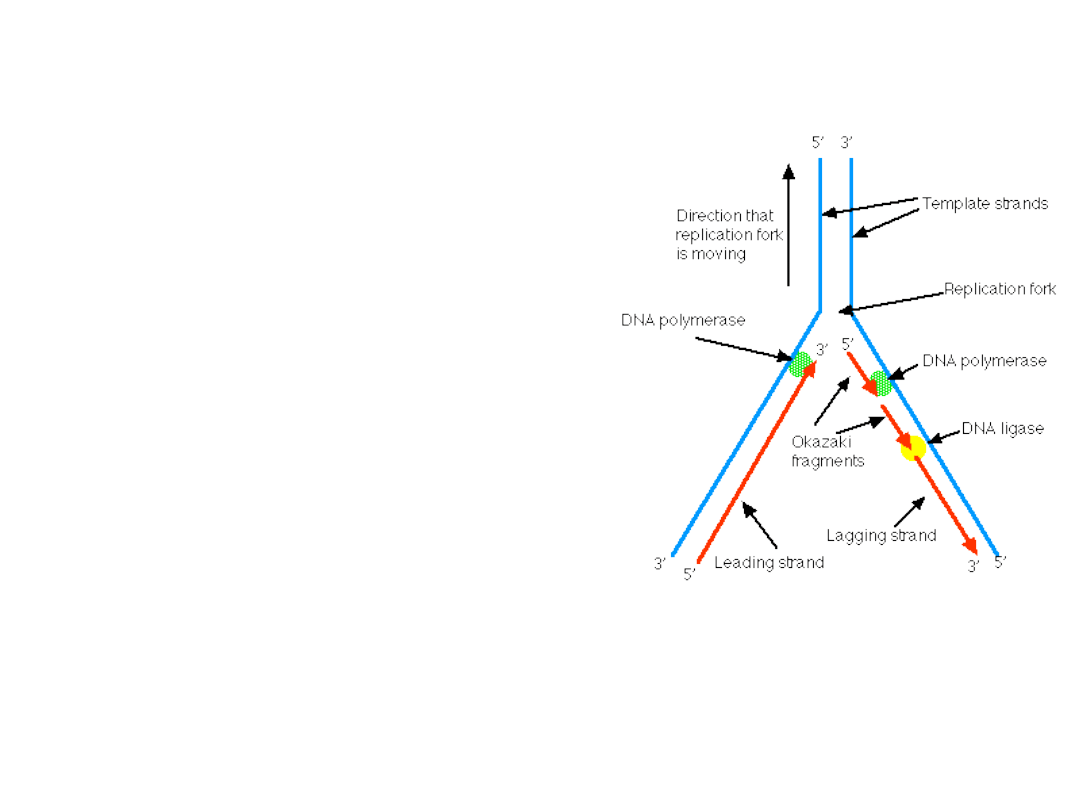
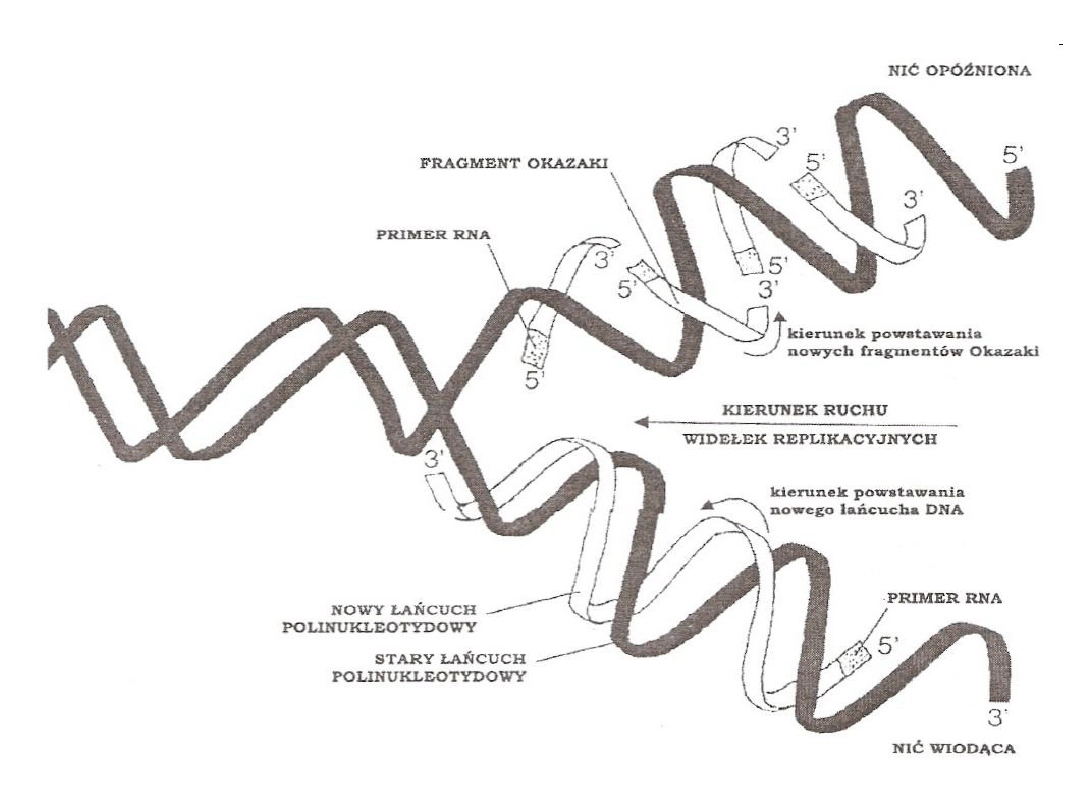
• Polimerazy syntetyzujące
potomne nici DNA nie tylko nie
są zdolne do rozpoczęcia
replikacji, lecz również
przyłączanie przez nie
nukleotydów może się
odbywać wyłącznie do grup
-OH (końca 3') starterów RNA.
• Replikacja DNA przebiega tylko
w jednym kierunku - od 5' do 3'
końca. Odwrotna orientacja nici
w obrębie podwójnego heliksu
pozwala na ciągłą syntezę tylko
jednej nici (tzw. nici wiodącej).
• Druga musi być w postaci
krótkich fragmentów,
dołączonych do odcinków
starterowych (jest to tzw. nić
opóźniona).
• Fragmenty te nazwano, na
cześć odkrywcy, fragmentami
Okazaki.

• Wytworzenie wiązania estrowego między
resztą fosforanową a deoksyrybozą,
katalizowane przez polimerazy, wymaga
dostarczenia energii. Jak w większości syntez
komórkowych, tak i tu energia pochodzi z
wysokoenergetycznych wiązań między
resztami kwasu fosforowego. Cząsteczka DNA
jest zbudowana z monofosfonukleotydów.
• Substratami do syntezy nowej nici w procesie
replikacji są trifosfonukleotydy (ATP, CTP,
GTP, TTP), więc dołączenie jednego
nukleotydu wiąże się z rozerwaniem dwóch
wiązań wysokoenergetycznych.
Ligazy
Ich zadaniem jest łączenie fragmentów
Okazaki (juz po wycięciu starterów przez
polimerazę) w jedna długa nic. Ligazy
bakteryjne wymagają NAD+ jako
koenzymu i źródła energii, natomiast
ligazy eukariotyczne wykorzystują do tego
celu ATP.

Terminacja replikacji:
• Terminacja jest końcowym etapem replikacji,
w którym dochodzi do połączenia DNA w
kompletne chromosomy i rozdzielenia ich
pomiędzy komórki potomne. Mechanizmy
terminacji u prokaroiota i eukariota znacznie
różnią się od siebie.
• U organizmów prokariotycznych, gdzie na
kolistym genoforze znajduje się tylko jeden
replikon, za końcowy etap replikacji
odpowiadają 4 sekwencje nukleotydowe ,
wiążące się z białkiem terminacyjnym. Białko
to będąc inhibitorem replikacji, hamuje cały
proces.

Terminacja u eucariota:
• Występowanie kilku replikonów w replikującym się
chromosomie powoduje, że do zakończenia
replikacji wystarczy fizyczne zetknięcie się dwóch
podążających ku sobie w przeciwnych kierunkach
widełek replikacyjnych.
• Nie potrzeba tu specjalnych sekwencji
terminalnych.
• Odmiennie przedstawia się sytuacja z
zakończeniem syntezy chromosomów. W
przeciwieństwie do kolistych genoforów
bakteryjnych, chromosomy eukariotyczne
zbudowane są z liniowych cząsteczek DNA.
• Gdyby replikacja u organizmów
eukariotycznych kończyła sie analogicznie do
prokariota to ostatni kilkunukleotydowy
fragment pozostawałby „niedoreplikowany”.
• Na końcu każdego chromosomu istnieją
jednak charakterystyczne sekwencje, które
pozwalają na utrzymanie niezmiennej
długości genomów. Sekwencje te zwane są
telomerami. Telomery odpowiadają również za
ochronę DNA przed łączeniem się z innymi
chromosomami.

Telomery:
Są one kompleksami nukleoproteinowymi.
Telomerowe DNA składa się z krótkich
tandemowych powtórzeń, bogatych w GT.
Telomery u człowieka mają długość ok. 25 kb
(dla porównania u myszy 40-80kb) i
zbudowane są z powtarzających się
fragmentów heksanulkeotydowych 5'-
TTAGGG-3'.
DNA budujące telomery jest na przeważającej
długości dwuniciowe, a ich koniec 3' ma
strukturę jednoniciową i jest bogaty w reszty
guaninowe, które tworzą strukturę
przestrzenną zwaną kwartetem G.
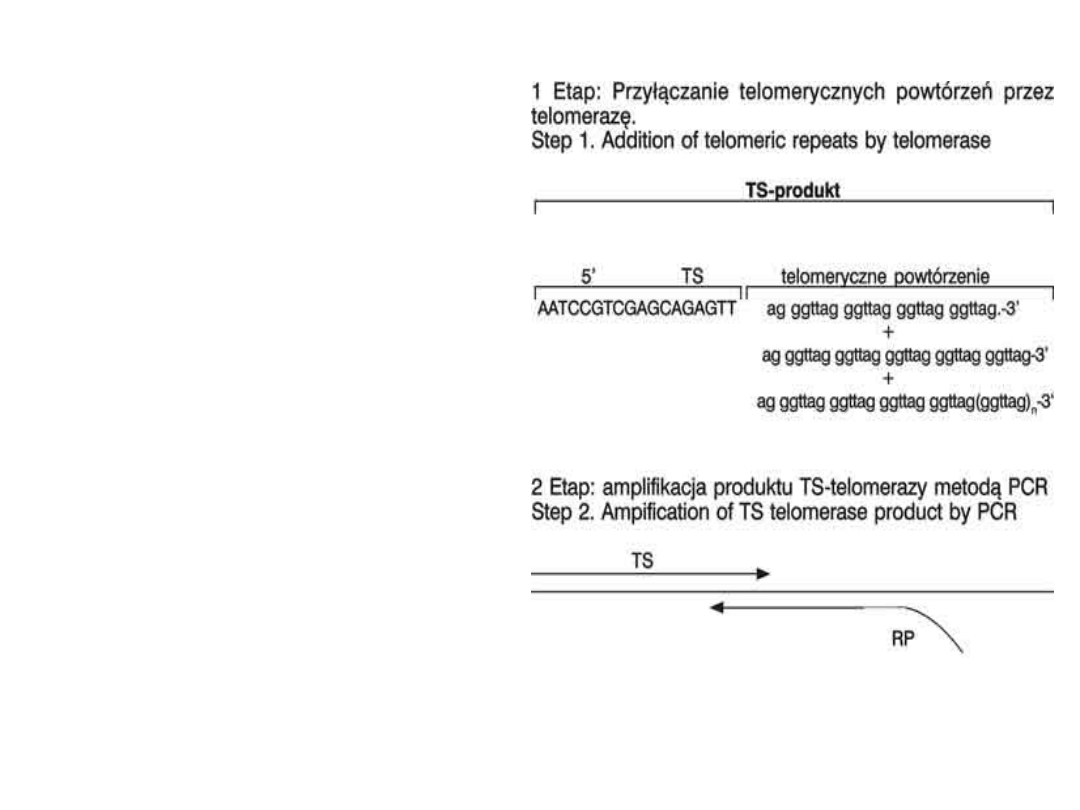
Replikacja telomerów
zachodzi w sposób
odmienny od reszty
chromosomu. Odpowiada
za nie specjalny enzym
zwany telomeraza. Jest
to enzym, który w swoim
centrum aktywnym
zawiera matrycę do
syntezy telomerów -
cząsteczkę RNA.
Starterami w tym
procesie są końcowe
sekwencje telomeru
pozostałego z
poprzedniego cyklu
replikacyjnego.

Naprawa uszkodzonego
DNA
• Naprawa niesparowanych zasad –
typu mismatch
• Naprawa przez wycięcie zasady
• Naprawa przez wycięcie nukleotydu
• Naprawa uszkodzenia dwuniciowego
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
Wyszukiwarka
Podobne podstrony:
Replikacja DNA, Pomoce do szkoły, Biologia
Replikacja DNA i choroby związane
Materialy do seminarium inz mat 09 10 czesc III
4 konta ksiegowe cwiczenia, Semestr V, Finanse i Rachunkowosc, Wyklady i materialy do seminarium
09 Replikacja DNA u?kterii
Zagadnienia do seminarium z Układu Pokarmowego
biologia, Replikacja DNA, Zjawisko transkrypcji to zjawisko powstawania rodzajów RNA ( t, m, r, )
metody wprow DNA do kom
Konstrukcja wektora plazmidowaego DNA do klonowania genów i do sekrecji w bakteriach mlekowych
Materialy do seminarium tech chem 13 14 id 284873
Materialy do seminarium inz mat Nieznany
Replikacja DNA
mat do seminarki
Od DNA do białka
REPLIKACJA DNA, biochemia
Zagadnienia do seminarium z aminokwasów i białek, Biotechnologia POLSL, Semestr V, Chemia Związków N
więcej podobnych podstron