
PODSTAWY BIOCHEMII DLA OCHRONY ŚRODOWISKA
Nukleotydy i kwasy nukleinowe
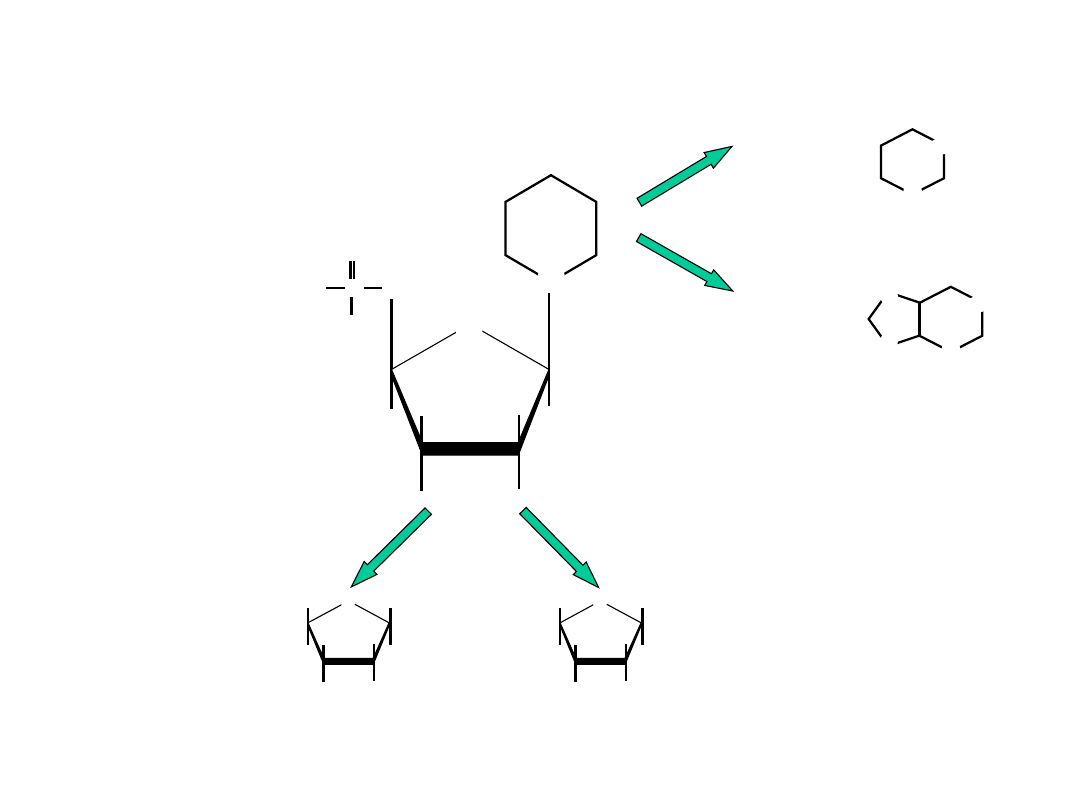
OH
OH
H
O
H
H
H
OH
HOCH
2
H
OH
H
O
H
H
H
OH
HOCH
2
ryboza
2-deoksyryboza
N
1
2
3
4
5
6
N
PIRYMIDYNA
cytozyna
tymina
uracyl
N
N
1
2
3
4
5
6
N
N
7
8
9
PURYNA
adenina
guanina
O
N
zasada
cukier
1’
2’
3’
4’
5’
CH
2
P
O
O
-
O
-
reszta kwasu
ortofosforowego
Nukleotydy
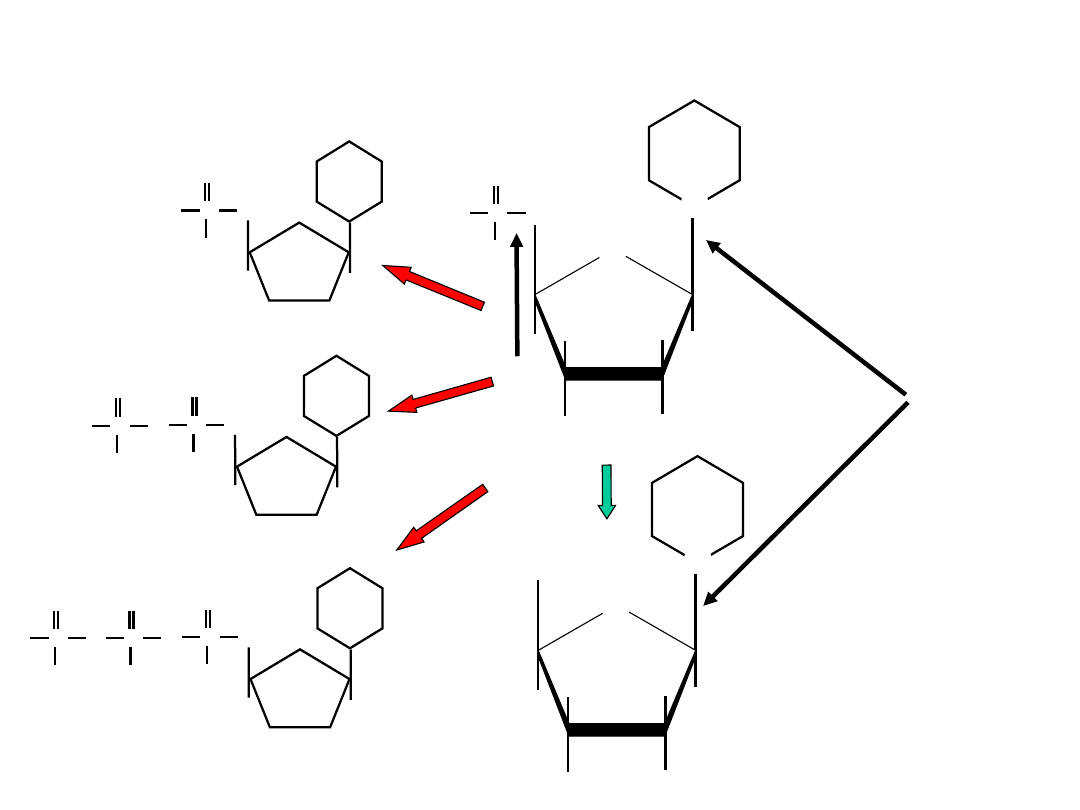
O
N
1’
2’
3’
4’
5’
CH
2
P
O
O
-
O
-
nukleotyd
O
N
1’
2’
3’
4’
5’
CH
2
OH
nukleozyd
CH
2
P
O
O
-
O
-
mono
foforan nukleozydu
CH
2
P
O
O
O
-
P
O
O
-
O
-
di
foforan nukleozydu
CH
2
P
O
O
O
-
P
O
O
-
O
P
O
O
-
O
-
tri
foforan nukleozydu
Nukleotydy
wiązanie
N-glikozydowe
wiązanie
estrowe
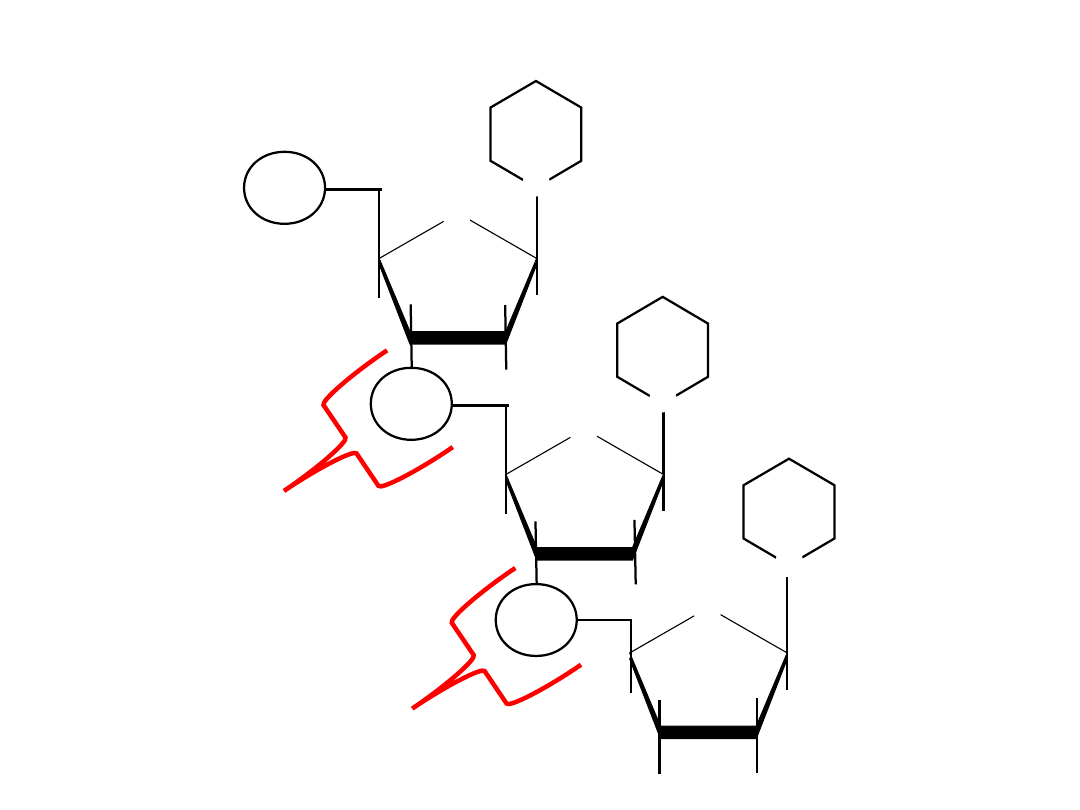
koniec 5’ łańcucha
koniec 3’ łańcucha
O
cukier
1’
2’
3’
4’
5’
fosforan
N
zasada
N
O
zasada
cukier
1’
2’
3’
4’
5’
O
cukier
1’
2’
3’
4’
5’
fosforan
fosforan
N
zasada
Fragment łańcucha kwasu nukleinowego
wiązanie
fosfodiestrowe
wiązanie
fosfodiestrowe
OH

Kwasy nukleinowe
DN
A
kwas rybonukleinowy
bierze udział
w rozkodowywaniu
informacji zawartej w DNA
koduje informację o budowie
i działaniu całego organizmu
kwas deoksyrybonukleinowy
RNA
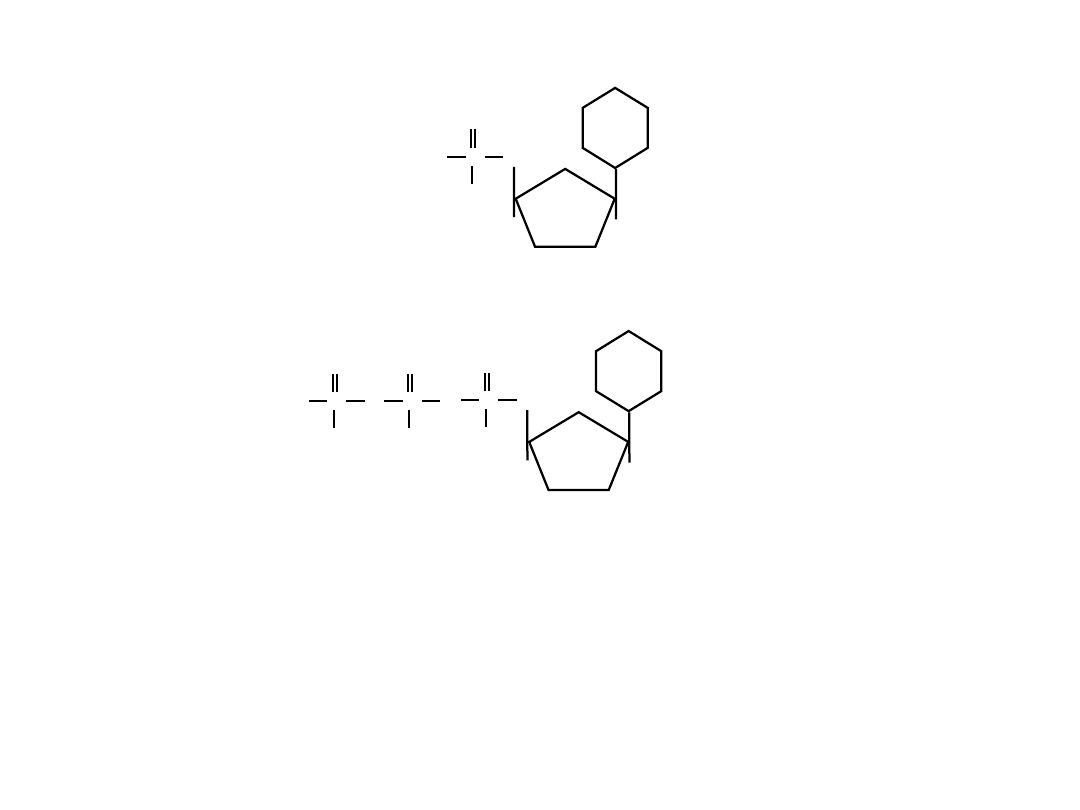
CH
2
P
O
O
-
O
-
mono
foforan nukleozydu
CH
2
P
O
O
O
-
P
O
O
-
O
P
O
O
-
O
-
tri
foforan nukleozydu
DNA dATP, dGTP, dCTP,
dTTP
RNA ATP, GTP, CTP,
UTP
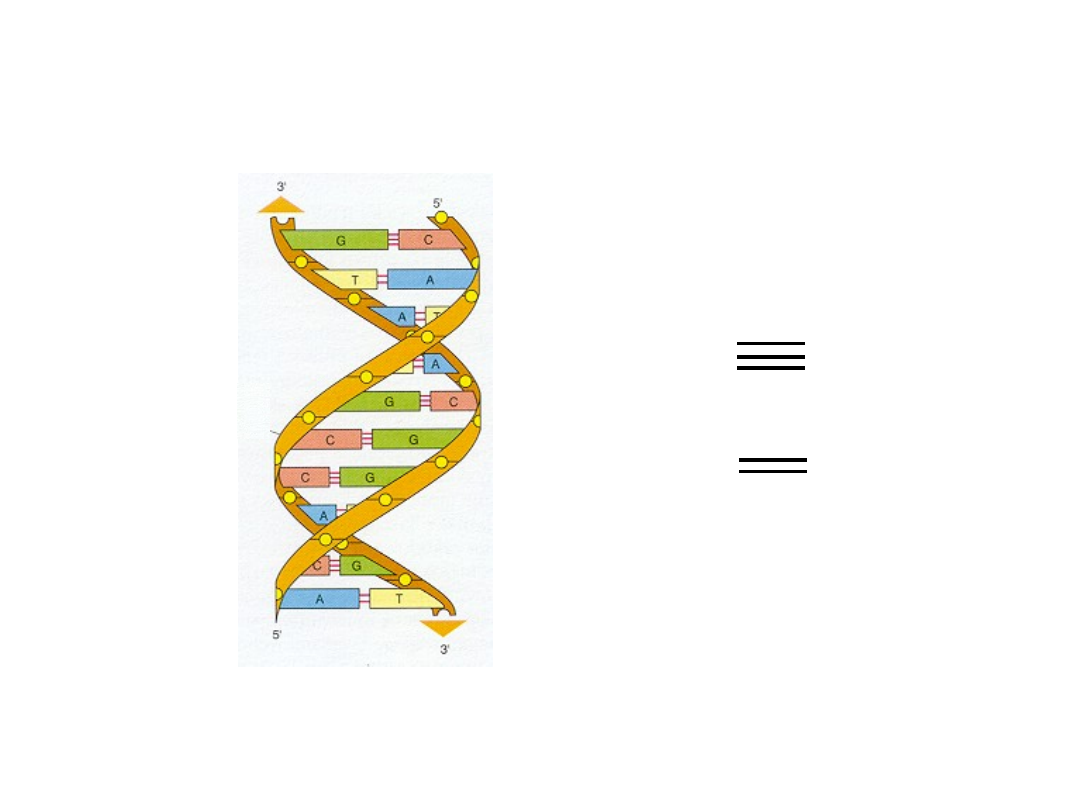
Podwójna helisa DNA
G C
A T
komplementarne parowanie zasad
5’ GGTACCAATTCAAAGCTTATG 3’
3’ CCATGGTTAAGTTTCGAATAC 5’
Enzymy restrykcyjne
Kpn I
GGTAC
C
C
CATGG
5’
GGTAC
3’
3’
C
5’
5’
C
AATTCAAAGCTTATG 3’
3’
CATGG
TTAAGTTTCGAATAC 5’
Hind III
A
AGCTT
TTCGA
A
5’ GGTACCAATTCA
A
3’
3’ CCATGGTTAAGT
TTCGA
5’
5’
AGCTT
ATG 3’
3’
A
TAC 5’
Alu I
AG
CT
TC
GA
5’
CT
TATG 3’
3’
GA
ATAC 5’
5’ GGTACCAATTCAA
AG
3’
3’ CCATGGTTAAGTT
TC
5’
5’ GGTACCAATTCA
A
|||||||||||||
||||
|||
3’ CCATGGTTAAGT
TTCGA
AGCTT
ATG 3’
|
A
TAC 5’
5’ GGTACCAATTCA
A
||||||||||||||||||||
3’ CCATGGTTAAGT
TTCGA
AGCTT
ATG 3’
|
A
TAC 5’
5’ GGTACCAATTCA
A
||||||||||||||||||||
3’ CCATGGTTAAGT
TTCGA
AGCTT
ATG 3’
|
A
TAC 5’
Hind III
A
AGCTT
TTCGA
A
Hind III
5’ GGTACCAATTCA
A
3’
|||||||||||||
3’ CCATGGTTAAGT
TTCGA
5’
5’
AGCTT
ATG 3’
|||
3’
A
TAC 5’
5’ GGTACCAATTCA
A
3’
|||||||||||||
3’ CCATGGTTAAGT
TTCGA
5’
5’
AGCTT
ATG 3’
|||
3’
A
TAC 5’
ligaza
5’ GGTACCCGGGCAA
||||||||||||||||||||
3’ CCATGGGCCCGTTTCGA
AGCTTATG 3’
|
ATAC 5’
5’ GGTACCAATTCAA
||||||||||||||||||||
3’ CCATGGTTAAGTTGGCC
CCGGTATG 3’
|
ATAC 5’
Xma I
A
CCGGT
TGGCC
A
Age I
ligaza
C
CCGGG
GGGCC
C
5’ GGTACCAATTCAA 3’
|||||||||||||
3’ CCATGGTTAAGTTGGCC 5’
5’CCGGTATG 3’
||||
3’ATAC 5’
5’ GGTAC
CCGG
T
ATG
|||||||||||||
3’ CCATG
GGCC
A
TAC
3’
5’
C
CCGGG
GGGCC
C
A
CCGGT
TGGCC
A
Xma I
Age I
5’ GGTAC
3’
|||||
3’ CCATGGGCC 5’
5’CCGGGCAAAGCTTATG 3’
||||||||||||
3’CGTTTCGAATAC 5’
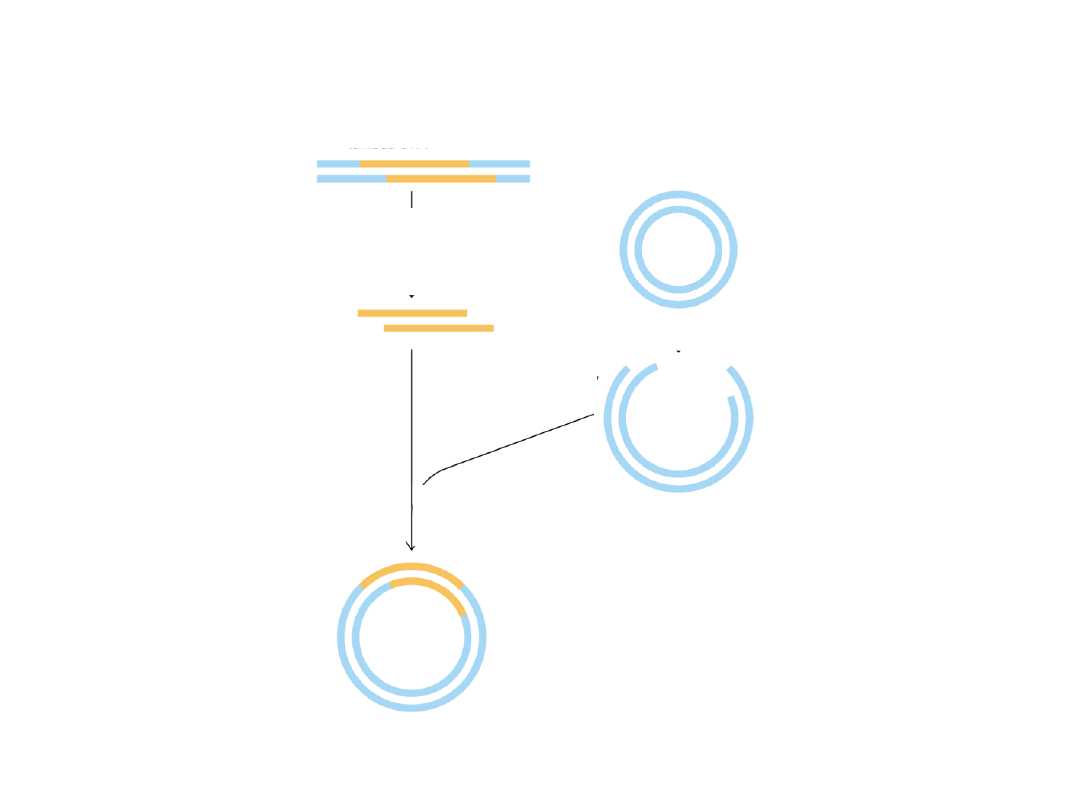
wektor
trawienie enzymem restrykcyjnym
Klonowanie DNA
DNA zawierający wstawkę,
którą chcemy przeklonować
trawienie
enzymem
restrykcyjnym
wstawka
ligacja wektora
ze wstawką
plazmid ze wstawką gotowy
do wprowadzenia do bakterii
bufor
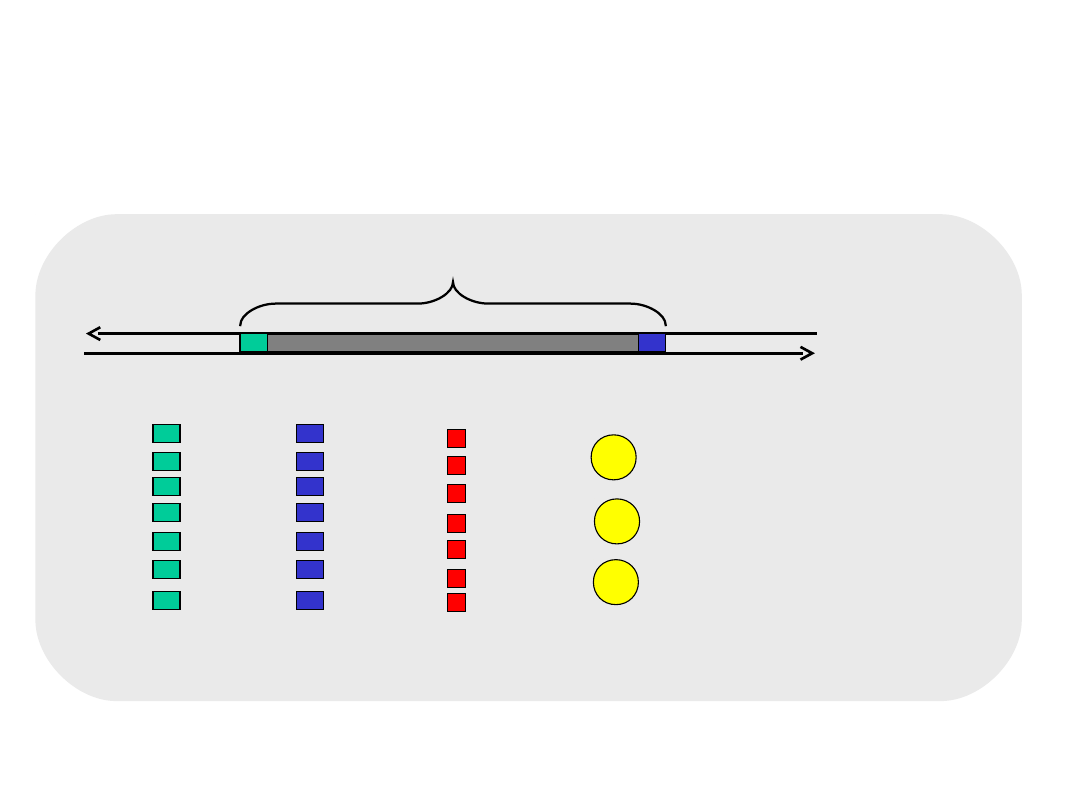
Reakcja PCR
mieszanina reakcyjna
dNTP
starter1
starter2
polimeraza Taq
dwuniciowa
matryca DNA
sekwencja docelowa

Cykl syntezy DNA w reakcji PCR
etap 1 - denaturacja matrycy
etap 2 - przyłączenie starterów
etap 3 - wydłużanie starterów
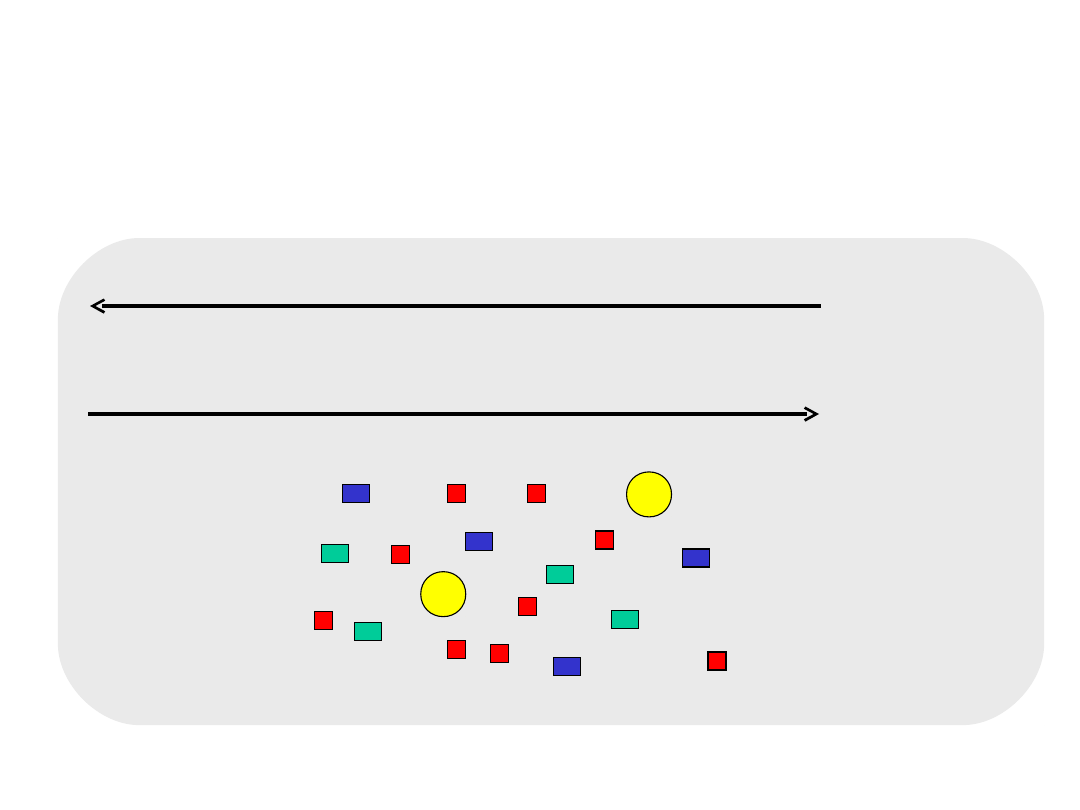
CYKL 1
denaturacja 94C
jednoniciowa
matryca DNA
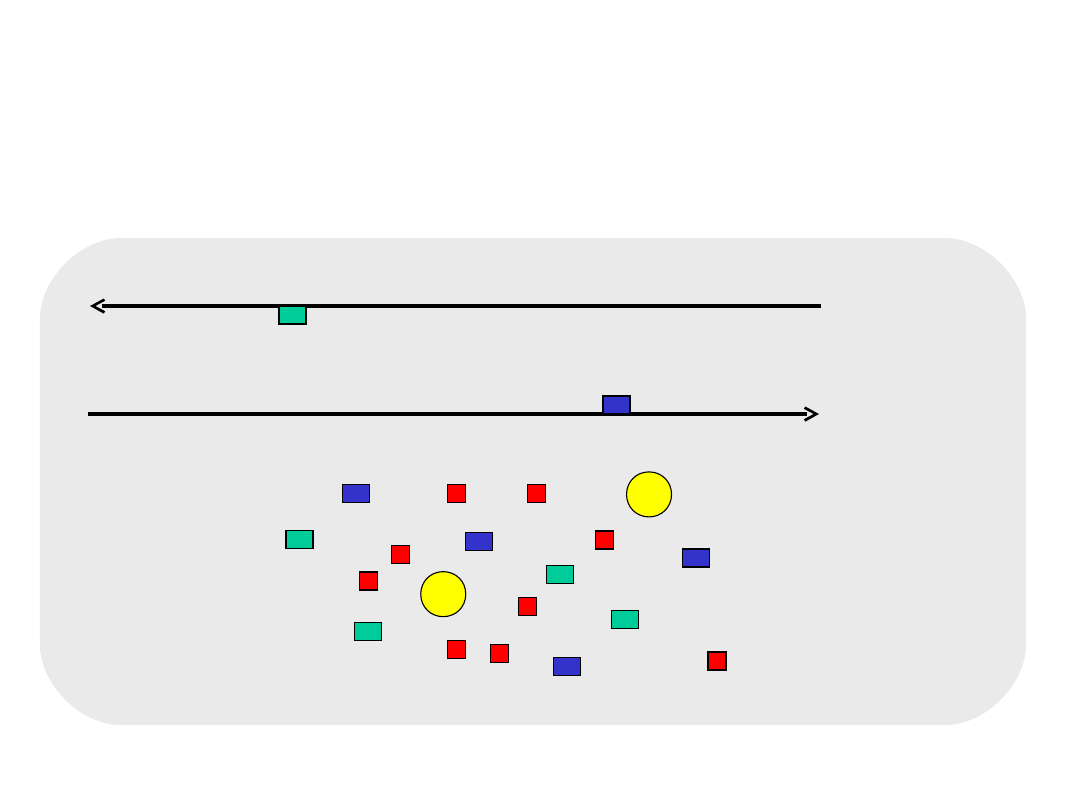
CYKL 1
przyłączenie startera do matrycy 40-65C
starter 1
starter 2
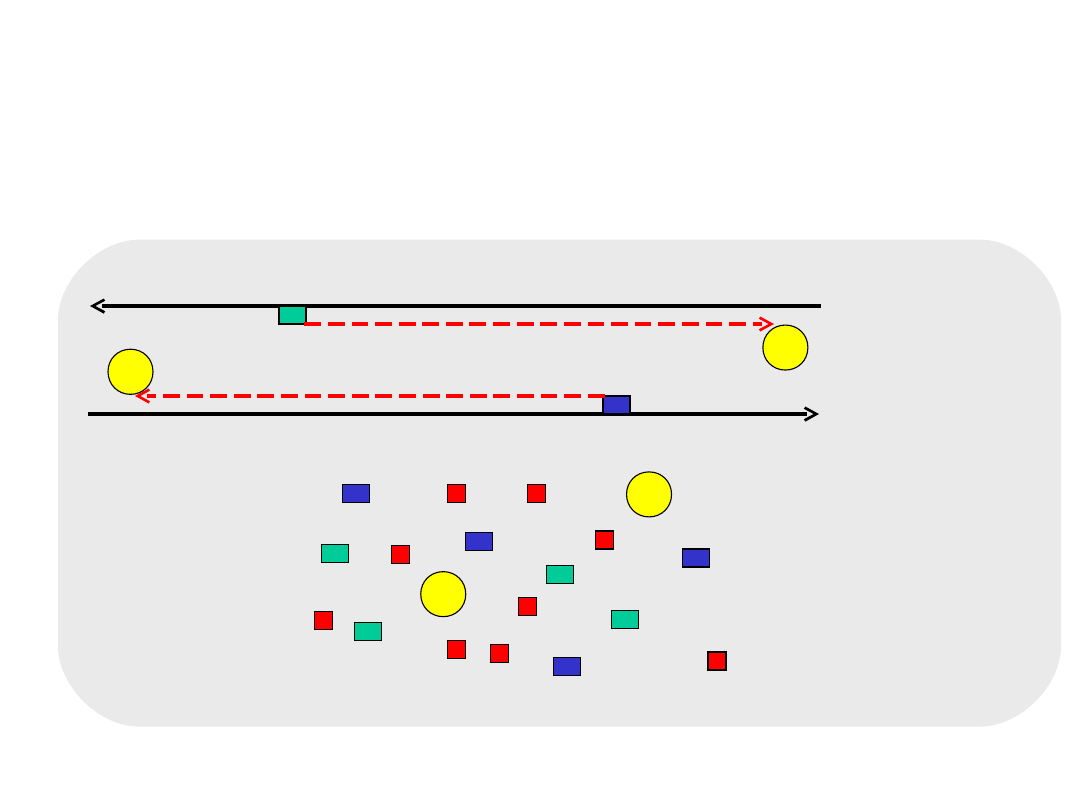
CYKL 1
wydłużanie startera 72C
starter 1
starter 2
powstają cząsteczki
dłuższe od sekwencji
docelowej
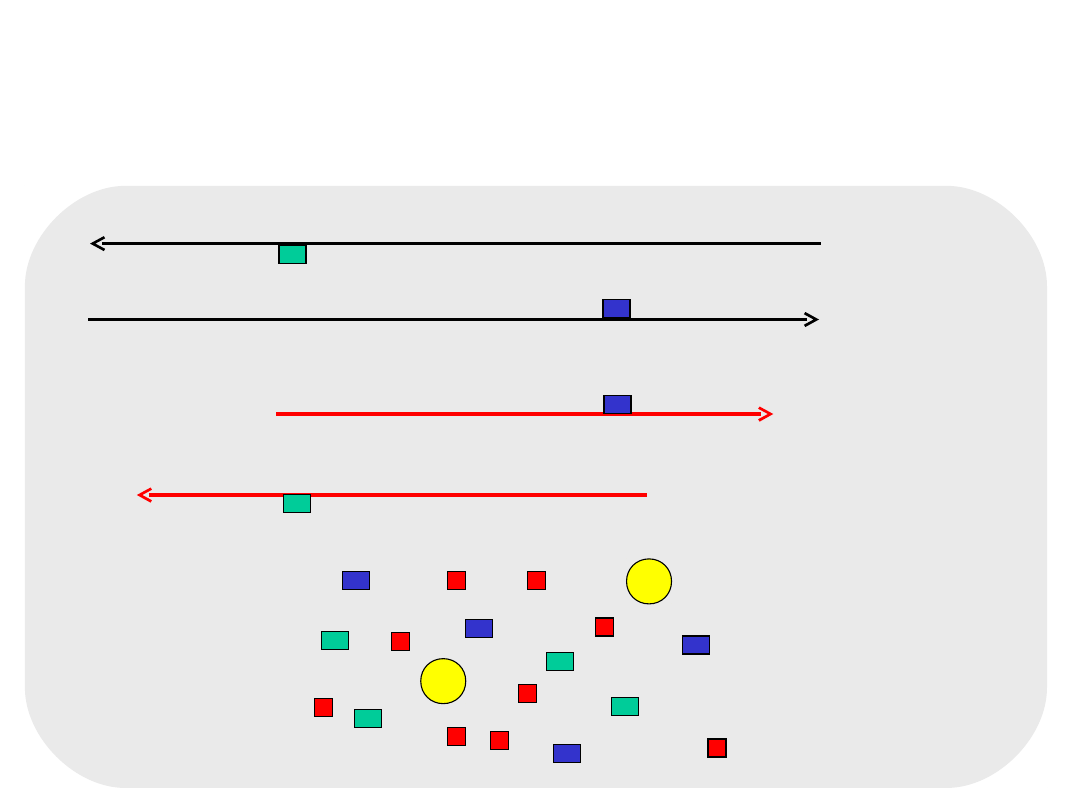
CYKL 2
denaturacja matrycy 94C
matryca
DNA, który powstał
w cyklu 1
CYKL 2
przyłączenie startera do matrycy 40-65C
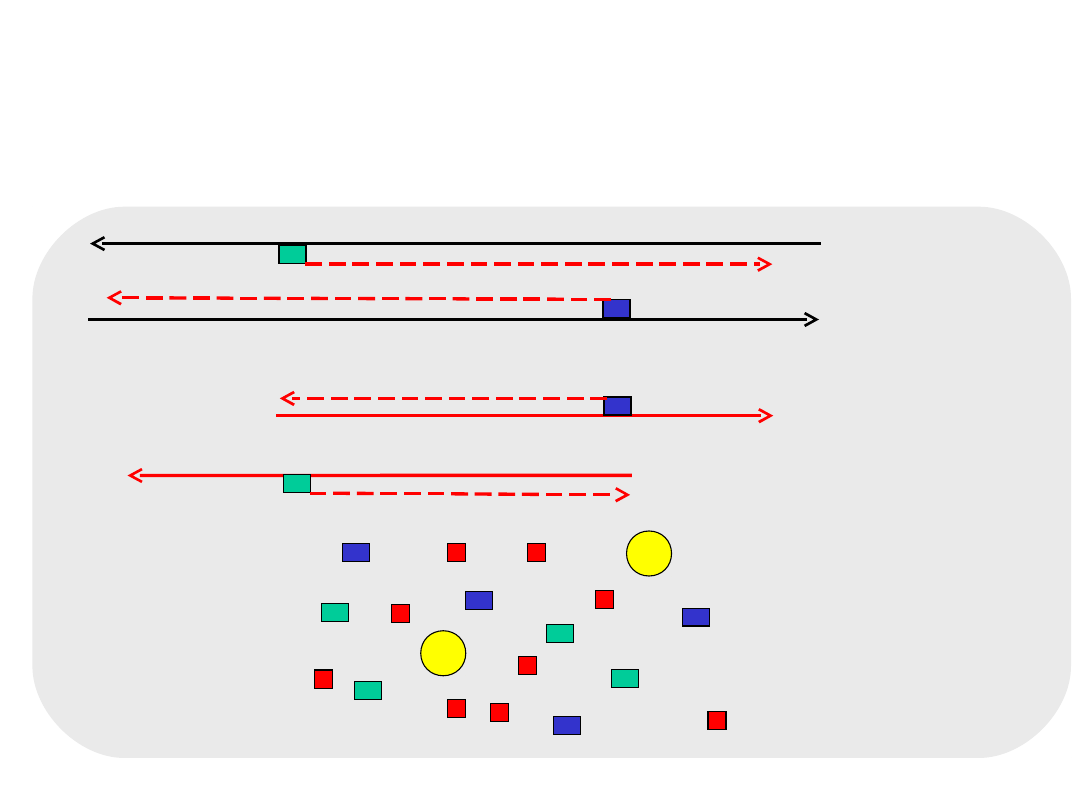
CYKL 2
wydłużanie starterów 72C
powstają cząsteczki
dłuższe od sekwencji
docelowej
powstają cząsteczki
o długości sekwencji
docelowej
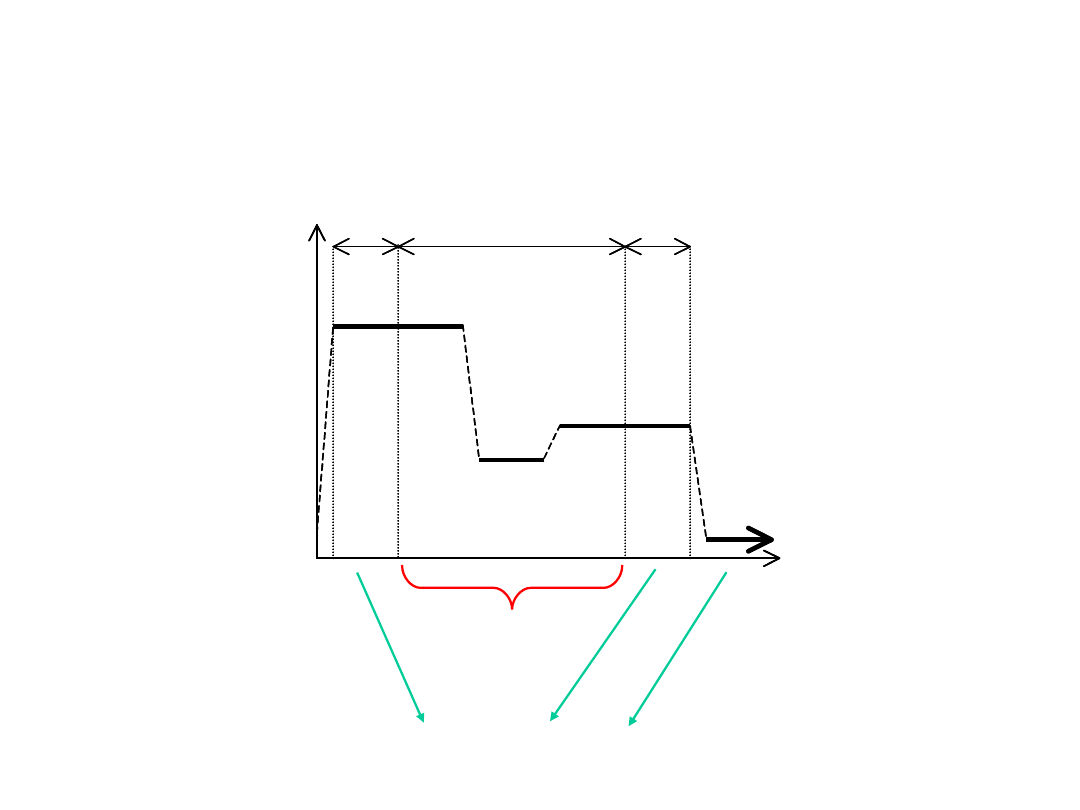
94°C
15s
94°C
3 min
65°C
15s
72°C
30s
te
m
pe
ra
tu
ra
czas
15x
72°C
30s
4°C
1x
1x
94°C
15s
94°C
3 min
65°C
15s
72°C
30s
te
m
pe
ra
tu
ra
czas
15x
72°C
30s
4°C
1x
1x
Profil termiczny reakcji PCR
cykl
podstawowy
cykle dodatkowe

Zastosowanie metody PCR (1)
DIAGNOSTYKA MIKROORGANIZMÓW (JAKOŚCIOWA I ILOŚCIOWA)
zakażenia bakterią
Chlamydia trachomatis (chlamydioza)
Chlamydia pneumoniae (chlamydioza układu
oddechowego)
Mycobacterium tuberculosis (gruźlica)
Borrelia burgdorferii (borelioza)
zakażenia pierwotniakiem Toxoplasma gondii
zakażenia wirusem HIV
CMV (cytomegalia)
Herpes simplex (opryszczka narządów płciowych)
zapalenia wątroby typu B (HBV) i typu C (HCV)
DIAGNOSTYKA CHORÓB NOWOTWOROWYCH
diagnostyka mutacji w genach BRCA1 i BRCA2
diagnostyka ryzyka nowotworowego - mutacje genów kodujących białka
naprawcze,
odpowiedzialne za nadwrażliwość na estrogeny
diagnostyka zagrożenia nowotworem szyjki macicy – wykrywanie wirusa
HPV (wirus
brodawczaka ludzkiego)

Zastosowanie metody PCR (2)
DIAGNOSTYKA CHORÓB GENETYCZNYCH
hemochromatoza
mukowiscydoza
dystrofia mięśniowa (Duchennea i Beckera)
choroba Alzheimera (genotypowanie ApoE)
dziedziczna skłonność miażdżycowa
Choroba Parkinsona
CCR5 (mutacja odpowiedzialna za odporność na HIV)
DIAGNOSTYKA NIEPŁODNOŚCI MĘSKIEJ O PODŁOŻU GENETYCZNYM
(polegającej na braku plemników w nasieniu - mikrodelecje na
chromosomie Y)
USTALANIE POKREWIEŃSTWA I OJCOSTWA
KRYMINALISTYKA
BADANIE ŻYWNOŚCI W KIERUNKU MODYFIKACJI GENETYCZNEJ GMO
(kukurzydza, soja, zboża, pomidory, ziemniaki, buraki cukrowe etc.) -
analiza jakościowa i ilościowa
BADANIA MIKROBIOLOGICZNE ŻYWNOŚCI
(Staphylococcus aureus, Salmonella species, Shigella species, Listeria
species)
BADANIA NAUKOWE
(biologia molekularna, biotechnologia, badania ewolucyjne, antropologia,
badania środowiskowe)

Ustalanie ojcostwa (1)
1. Pobranie materiału do badań
2. Kodowanie
próbek
3. Izolacja DNA
4. Amplifikacja DNA
5. Elektroforeza DNA
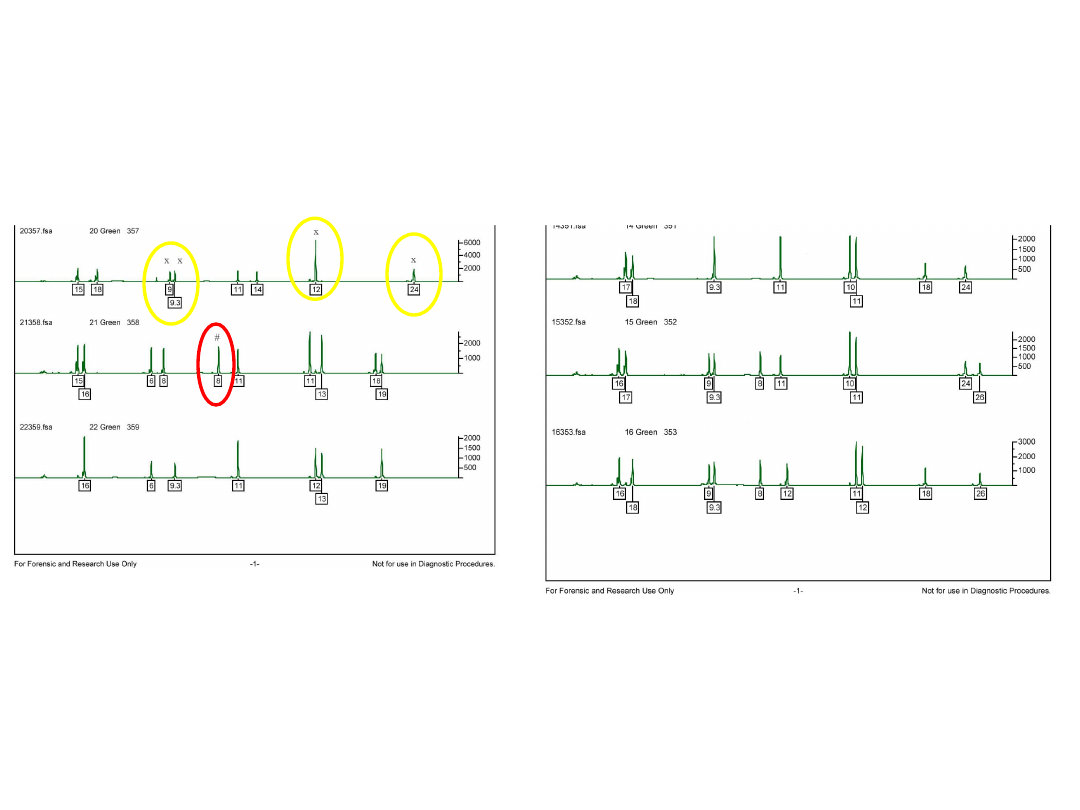
x – dziecko nie odziedziczyło
żadnego allela od mężczyzny
# - dziecko odziedziczyło allel
nieobecny u mężczyzny
wszystkie allele dziecka
pochodzą od matki i od
mężczyzny
7. Analiza statystyczna
Ustalanie ojcostwa (2)
6. Analiza genotypów badanych osób
wykluczenie
ojcostwa
potwierdzenie ojcostwa
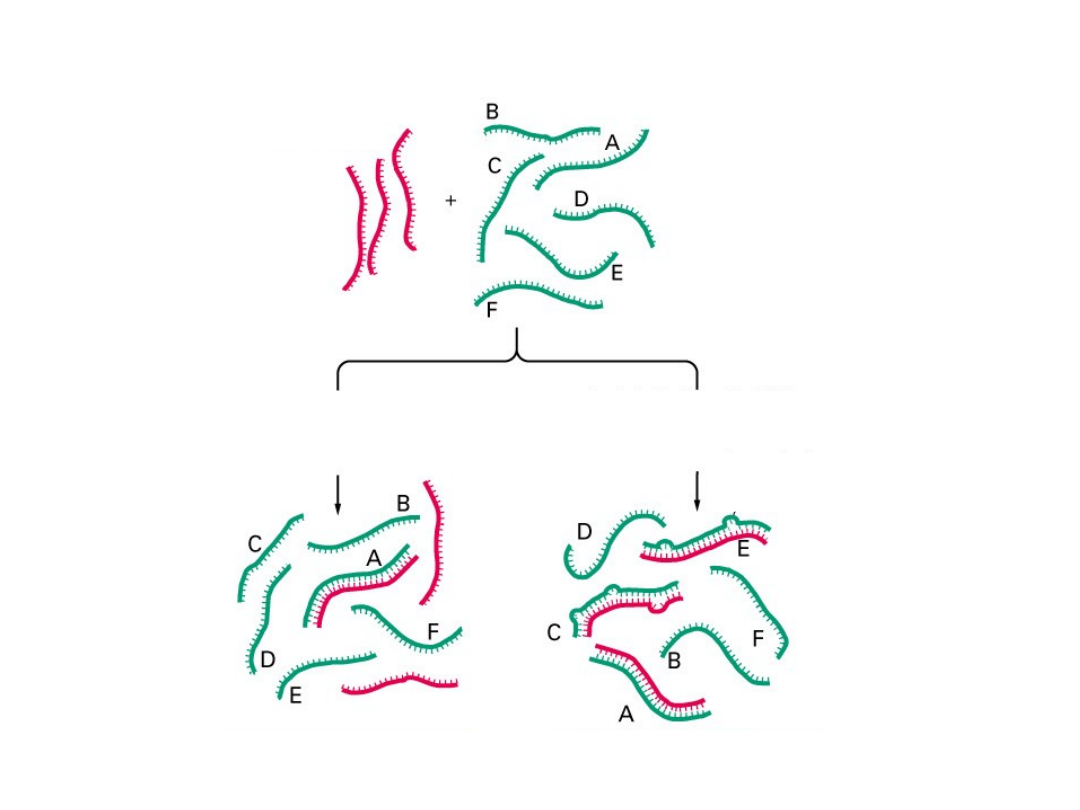
mieszanina
jednoniciowych
cząsteczek DNA
jednoniciowa wyznakowana
sonda komplementarna
do cząsteczki A
hybrydyzacja
w warunkach ostrych
hybrydyzacja
w warunkach łagodnych
tylko cząsteczka A tworzy
stabilną dwuniciową cząsteczkę
cząsteczki A, C i E tworzą
stabilne dwuniciowe cząsteczki
Hybrydyzacja
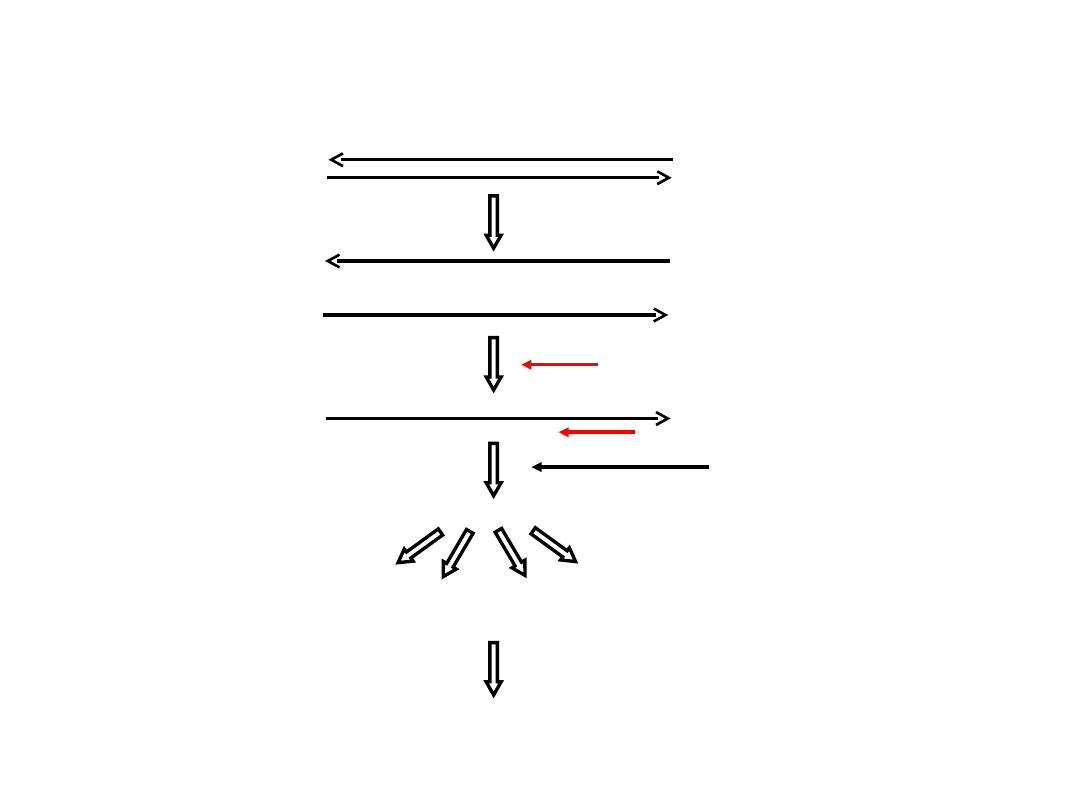
Sekwencjonowanie DNA
denaturacja
dwuniciowy DNA
jednoniciowy DNA
(matryca do sekwencjonowania)
przyłączenie znakowanego startera
starter
podział na 4 oddzielne reakcje
wydłużania startera
A T C G
rozdział produktów reakcji sekwencjonowania
w żelu poliakrylamidowym
pozostałe składniki reakcji
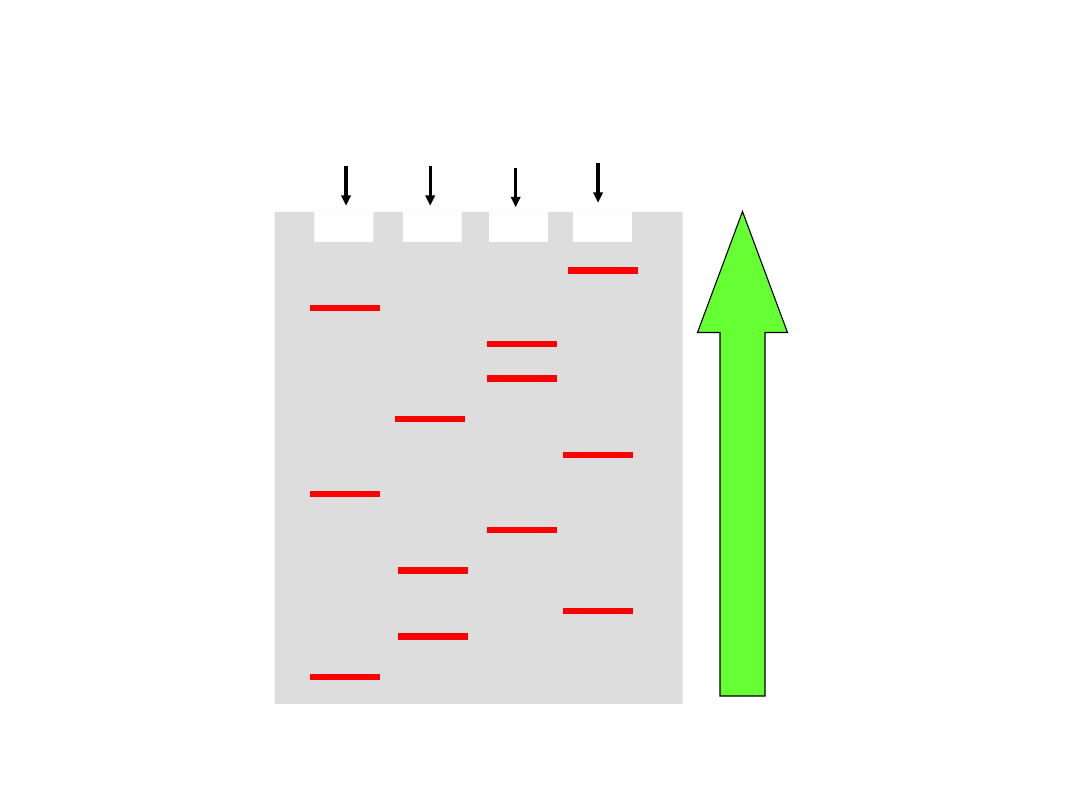
Sekwencjonowanie DNA
A T C G
G
A
C
C
T
G
A
C
T
G
T
A
5’
3’
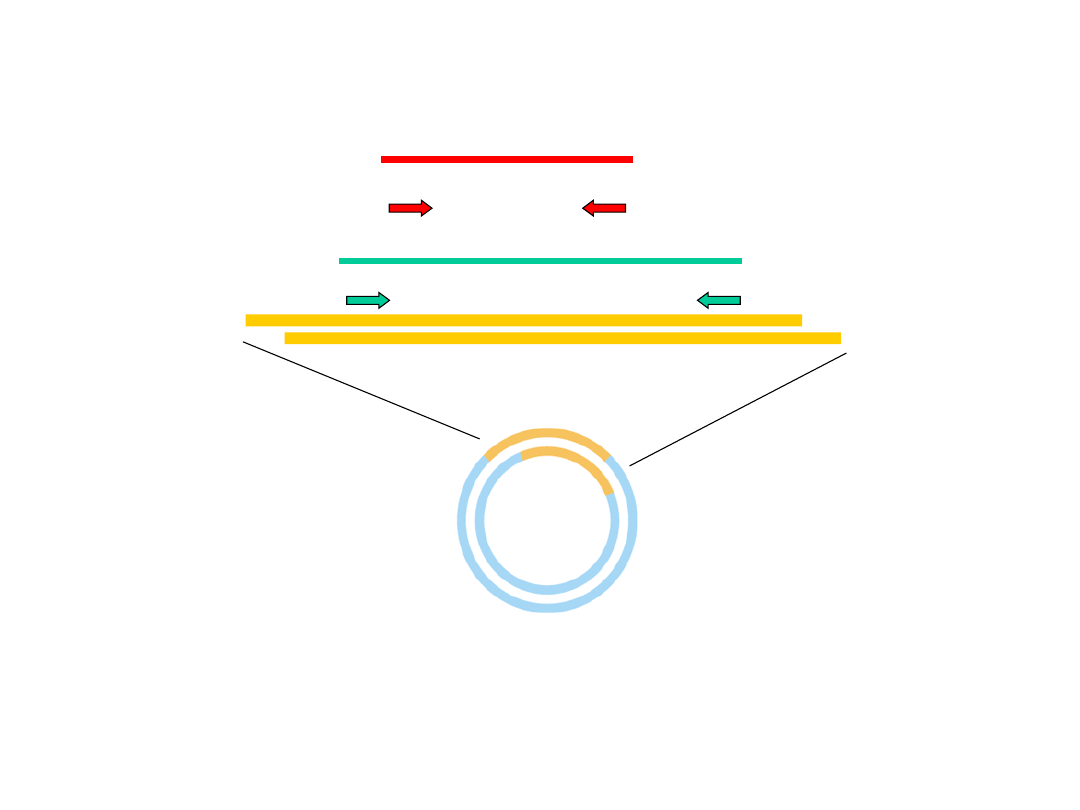
plazmid pUC18/cDNAPPRas2
ze wstawką 0,8 kp
mieszanina A:
startery A1 i A2
A1
A2
B2
B1
0,6 kb
0,35 kb
Część praktyczna
mieszanina B:
startery B1 i B2
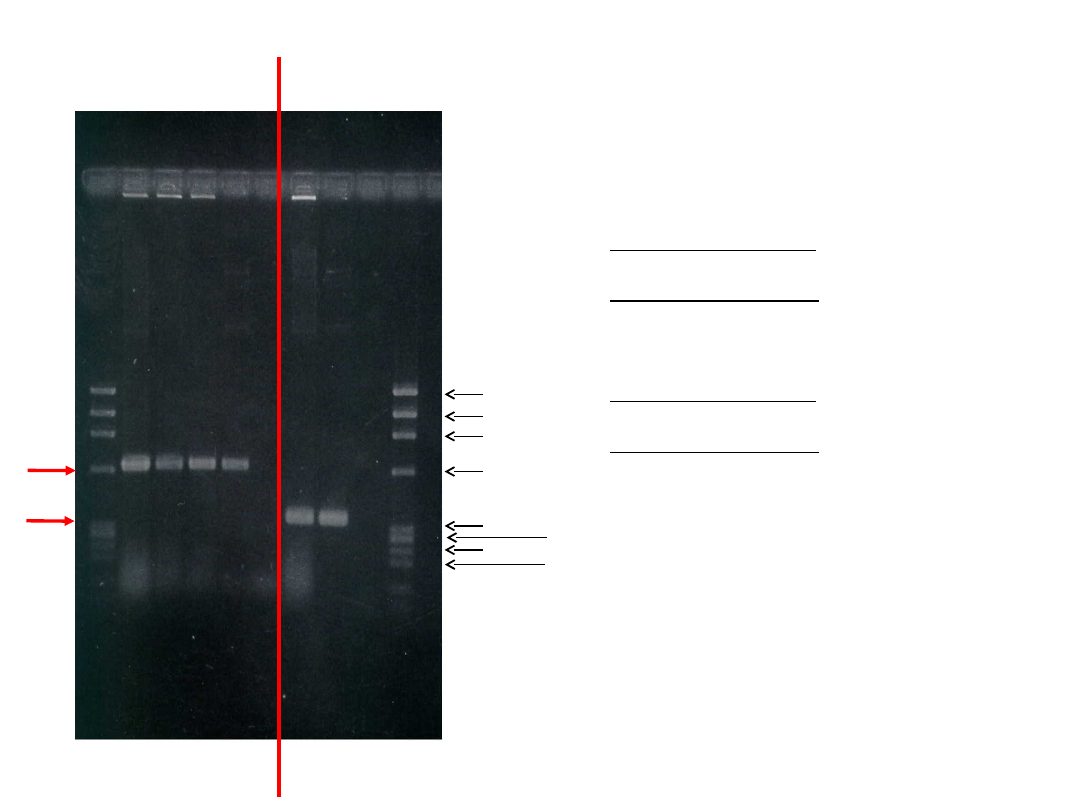
1 - wzorce wielkości
2, 3, 4 - produkty reakcji A, w której jako matrycy
użyto lizatu bakterii)
5 - kontrola pozytywna, czyli produkty reakcji A,
w której jako matrycy użyto czystego plazmidu
6 - kontrola negatywna, czyli produkty reakcji A,
do której nie dodano żadnej matrycy
7 - produkty reakcji B, w której jako matrycy
użyto lizatu bakterii)
8 - kontrola pozytywna, czyli produkty reakcji B,
w której jako matrycy użyto czystego plazmidu
9 - kontrola negatywna, czyli produkty reakcji B,
do której nie dodano żadnej matrycy
10 - wzorce wielkości
1 2 3 4 5 6 7 8 9 10
1,35 kb
1,08 kb
0,87 kb
0,6 kb
0,28 kb
0,23 kb
0,31kb
0,6 kb
0,35 kb
0,19 kb
A
B
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
Wyszukiwarka
Podobne podstrony:
nukleotydy i kwasy nukleinowe
Nukleotydy i kwasy nukleinowe
kwasy nukleinowe
13 Kwasy nukleinowe
wpływ leków na kwasy nukleinowe
kwasy nukleinowe
Biochmia 12 Kwasy nukleinowe
BW13 KWASY NUKLEINOWE id 95709 Nieznany
pkt1 kwasy nukleinowe-biochemia, Biochemia, Zagadnienia na kolokwia
kwasy nuklein
sprawozdanie kwasy nukleinowe
wszystko wyjście kwasy nukleinowe
kwasy nukleinowe opracowanie
Kwasy nukleinowe
KWASY NUKLEINOWE I STRUKTURA
więcej podobnych podstron